一种RNA-seq在线分析报告系统及其生成方法与流程
本发明涉及生物信息分析技术领域,特别是涉及一种rna-seq在线分析报告系统及其生成方法。
背景技术:
自人类基因组计划完成以来,基因测序成本以“超摩尔定律”的速度不断下降。基因测序在人类中应用越来越广泛,涵盖了dna水平的全基因组测序、外显子测序等,rna水平的rna-seq、环状rna测序等和微生物水平的宏基因组测序、meta16s测序等。随着新一代高通量测序技术的飞速发展,以高通量测序技术为基础的rna-seq测序技术已经成为研究rna的重要技术被广泛使用,可以检测基因的表达水平,而且也能对大量样品同时测序获得样品之间的表达差异等,应用于基础研究、临床研究和药物研发等领域。
然而,基因测序所生成的原始数据并不能反映任何有价值的信息,必须通过专业人员进行分析和解读,如何从海量测序数据中挖掘出有价值的核心信息,才是目前科研人员面临的关键问题。目前,绝大多数高通量测序如rna-seq测序等的生物信息分析结果都是以静态结题报告的形式呈现出来,数据展示不便于实时解析和关键数据挖掘。当科研人员对分析结果不满意或想进行修改时,如果自己无生物信息分析基础,这就需要生物信息分析人员按照个人需求重新进行数据分析,这样增加了数据分析的工作量,费时费力。面对大量的测序数据时,这样调整分析结果,也大大降低了生物信息分析人员的工作效率。
技术实现要素:
为克服上述现有技术存在的不足,本发明之目的在于提供一种rna-seq在线分析报告系统及方法,以使无编程基础的人员也能进行数据分析,并大大降低了生物信息分析人员的工作量,提高了工作效率。
为达上述及其它目的,本发明提出一种rna-seq在线分析报告系统,包括:
样本关系模块,用于对选择的样本进行分组,对选择的分组样本自动进行样本关系的分析;
基因筛选模块,用于根据所输入的基因名称或功能,筛选基因,并根据基因id得到基因的序列信息;
差异基因分析模块,用于对比较组样本的基因进行表达量差异分析,并输出差异分析结果;
基本功能分析模块,用于对选定的基因集或差异分析结果集进行go富集分析、kegg富集分析和韦恩图分析。
优选地,所述样本关系模块对样本关系的分析包括:
pca分析;
相关性热图分析;
样本聚类图分析,利用全部基因的表达量对所有样本的关系进行层级聚类,生成样本聚类图;
重复性散点图分析,用于根据计算两个样本表达量之间的相关性,生成重复性散点图。
优选地,所述基因筛选模块包括基因筛选和基因序列筛选,所述基因筛选为根据输入的基因名称或者功能,筛选一个或多个基因,所述基因序列筛选则根据输入的基因id,得到基因的序列信息。
优选地,所述差异基因分析模块还根据q值、差异倍数和基因丰度的设定,进行所选择比较组的差异分析,并输出筛选差异基因的实时热图或差异基因雷达图。
优选地,所述差异基因的表达情况采用柱状图或盒型图或火山图或散点图或折线图呈现。
优选地,所述系统还包括基本信息分析输出模块,用于分析项目的基本信息,并输出分析项目的基本信息以及相应的数据列表。
优选地,所述系统还包高级功能分析模块,用于根据选择基因集或差异分析结果集进行趋势分析。
优选地,所述高级功能分析模块还通过选择比对数据进行blast分析。
优选地,所述系统还包括任务列表模块,用于输出每个任务的类型、编号、开始时间和结束时间及任务进度,以便实时查看任务进展。
为达到上述目的,本发明还提供一种rna-seq在线分析报告方法,包括如下步骤:
步骤s1,获取样本数据;
步骤s2,对获取的样本数据进行分组,自动进行样本关系的分析;
步骤s3,根据所输入的基因名称或功能,筛选基因,并根据基因id,得到基因的序列信息;
步骤s4,对比较组样本的基因进行表达量差异分析,输出差异基因统计柱状图、基因统计表和聚类热图中的部分或全部,并根据q值、差异倍数和基因丰度进行筛选关注的比较组的具体差异基因的分析情况;
步骤s5,对选定的基因集或差异分析结果集进行go富集分析、kegg富集分析和韦恩图分析。
与现有技术相比,本发明一种rna-seq在线分析报告系统及其生成方法通过设计样本关系模块、基因筛选模块、差异基因分析模块以及基本功能分析模块,使得用户可以按需进行样本分组,挑选关注的基因进行分析,也可以自主设置过滤参数,筛选目标差异基因,对于分析结果可以根据个人需求进行图形修改、字体修改、图形下载等操作,这样使无编程基础的人员也能进行数据分析,具有操作简单、数据安全性高及分析结果动态化等优点,给用户得到满意的分析结果带来了便捷,同时也大大降低了生物信息分析人员的工作量,提高了工作效率,达到了双赢的效果。
附图说明
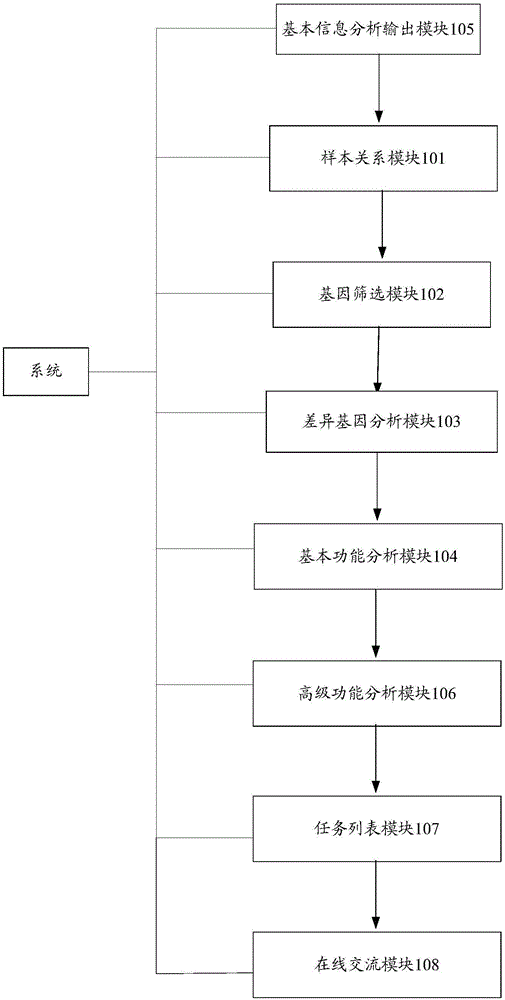
图1为本发明一种rna-seq在线分析报告系统的系统架构图;
图2为本发明一种rna-seq在线分析报告的生成方法的系统架构图
图3为本发明具体实施例中rna-seq在线分析报告系统的示意图;
图4为本发明具体实施例中差异基因分析模块整体图;
图5为本发明具体实施例中差异基因统计柱状图的示意图;
图6为本发明实施例中差异分析分组详细图。
具体实施方式
以下通过特定的具体实例并结合附图说明本发明的实施方式,本领域技术人员可由本说明书所揭示的内容轻易地了解本发明的其它优点与功效。本发明亦可通过其它不同的具体实例加以施行或应用,本说明书中的各项细节亦可基于不同观点与应用,在不背离本发明的精神下进行各种修饰与变更。
图1为本发明一种rna-seq在线分析报告系统的系统架构图。如图1所示,本发明一种rna-seq在线分析报告系统,包括:
样本关系模块101,用于对选择的样本进行分组,自动进行样本关系的分析,在本发明具体实施例中,样本关系的分析包括pca分析、相关性热图分析、样本聚类图分析和重复性散点图分析等。具体地,样本关系模块101可以通过添加样本关系单按钮进行分组信息选择和组间比较的选择,生成新的任务,由样本关系模块101对选择的分组样本进行pca分析、相关性热图、样本聚类图和重复性散点图等,在pca分析时,可以选择是否显示样本名、颜色选择、字体修改、图形下载等按钮,可以根据需要自行修改,由于具体的pca分析采用的是现有技术,在此不予赘述;相关性热图,可以选择是否显示相关性系数,同样也可对颜色、字体大小、图标题等进行自定义,这里具体地生成相关性热图采用的是现有技术,在此不予赘述;样本聚类图分析是利用全部基因的表达量对所有样本的关系进行层级聚类,生成样本聚类图,图片可以自行下载;重复性散点图分析则用于根据计算两个样本表达量之间的相关性,生成重复性散点图。
基因筛选模块102,用于根据所输入的基因名称或功能,筛选基因,并根据基因id,得到基因的序列信息。也就是说,基因筛选模块102包括基因筛选和基因序列筛选,其中基因筛选是根据输入所关心的基因名称或者功能,例如geneid、description、pathway和go等,筛选一个或多个基因,基因序列筛选则是通过输入一个或多个基因id,得到基因的序列信息,如基因的碱基序列、cds序列(如果有)和蛋白序列(如果有)等。
差异基因分析模块103,用于对比较组样本的基因进行表达量差异分析,输出差异基因统计柱状图、基因统计表和聚类热图中的部分或全部,并可以根据q值、差异倍数和基因丰度进行筛选关注的比较组的具体差异基因的分析情况。在本发明具体实施例中,差异基因分析模块103的输出包括总览和分组详细,其中总览中有差异基因统计柱状图、基因统计表和聚类热图,差异基因统计柱状图可选择是否显示上调基因、下调基因的具体数量,进行字体、标题、x轴和y轴的自定义进行结果的修改,基因统计表显示了所有分组样本之间上调基因和下调基因的具体数量,聚类热图展现了所有分组样本之间的基因聚类情况,也可以根据需要每一组样本的聚类情况并进行图片下载;分组详细部分可以分别展示各个比较组的差异表达情况,可以进行q值、差异倍数和基因丰度的自主设定,进行所选择比较组的差异分析,并绘制出筛选差异基因的实时热图,也可以通过绘制差异基因雷达图,差异基因的表达情况可以用柱状图、盒型图、火山图、散点图和折线图等形式呈现,图形的颜色、字体等可以进行修改。这里需说明是,差异基因分析模块的表达量差异分析方法采用的也是现有方法,这里不予赘述。
基本功能分析模块104,用于对选定的基因集或差异分析结果集进行go富集分析、kegg富集分析和韦恩图分析。也就是说,基本功能分析模块104可以通过选择基因集或差异分析结果集,对差异基因进行go富集分析和kegg富集分析,也可以对特定的基因集进行相关分析,同时也可以通过选择基因集或差异基因结果集进行比较分析,绘制出韦恩图输出。
优选地,本发明之rna-seq在线分析报告系统还包括:基本信息分析输出模块105,用于分析项目的基本信息,并输出分析项目的基本信息以及相应的数据列表,其中基本信息包括有项目编号、样品名称、样品间差异方案、分组方案和分组间差异方案等详细信息,数据列表则是展示样本中所有基因的表达量。
优选地,本发明之rna-seq在线分析报告系统还包括:高级功能分析模块106,用于根据选择基因集或差异分析结果集进行趋势分析。也就是说,高级功能分析模块106可通过选择基因集或差异分析结果集,并进行分组方案选择、趋势数量、显著趋势的p值和基因筛选的最小变化倍数等参数设置,进行趋势分析并输出趋势分析结果。较佳地,高级功能分析模块106还可根据通过选择比对数据进行blast分析,即通过上传或者输入query序列的fasta序列,并可以选择比对程序和比对数据库等,进行blast分析,分析完成,就会跳转到blast分析结果,同时也可以自主添加筛选条件对比对结果进行筛选。高级功能分析模块105还可以提供选择进行基因组浏览,即用户可以根据选择展示出cds、exon、gene、mrna等信息。
优选地,本发明之rna-seq在线分析报告系统还包括:任务列表模块107,用于输出每个任务的类型、编号、开始时间和结束时间及任务进度,能实时查看任务进展,在本发明具体实施例中任务列表模块107可提供用户选择任务,根据用户的选择输出每个任务的类型、编号、开始时间和结束时间及任务进度,并能实时查看任务进展。
优选地,本发明之rna-seq在线分析报告系统还包括在线交流模块108,以提供在线交流的信息,例如提供用户查看生物信息学教学视频、生物信息分析的工具以及一些精华的帖子。
图2为本发明一种rna-seq在线分析报告的生成方法的步骤流程图。如图2所示,本发明一种rna-seq在线分析报告的生成方法,包括如下步骤:
步骤s1,获取样本数据。在本发明具体实施例中,获取样本数据的方式可以是输入样本数据,也可以是从已有的样本数据中选择相应的样本数据。
步骤s2,对获取的样本数据进行分组,自动进行样本关系的分析,在本发明具体实施例中,样本关系的分析包括pca分析、相关性热图分析、样本聚类图分析和重复性散点图分析等。具体地,于步骤s2中,可以通过添加样本关系单按钮进行分组信息选择和组间比较的选择,生成新的任务,对选择的分组样本进行pca分析、相关性热图、样本聚类图和重复性散点图等,在pca分析时,可以选择是否显示样本名、颜色选择、字体修改、图形下载等按钮,可以根据需要自行修改;相关性热图,可以选择是否显示相关性系数,同样也可对颜色、字体大小、图标题等进行自定义;样本聚类图分析是利用全部基因的表达量对所有样本的关系进行层级聚类,生成样本聚类图,图片可以自行下载;重复性散点图分析则用于根据计算两个样本表达量之间的相关性,生成重复性散点图。
步骤s3,根据所输入的基因名称或功能,筛选基因,并根据基因id,得到基因的序列信息。在步骤s3中,基因筛选是根据输入所关心的基因名称或者功能,例如geneid、description、pathway和go等,筛选一个或多个基因,基因序列筛选则是通过输入一个或多个基因id,得到基因的序列信息,如基因的碱基序列、cds序列(如果有)和蛋白序列(如果有)等。
步骤s4,对比较组样本的基因进行表达量差异分析,输出差异基因统计柱状图、基因统计表和聚类热图中的部分或全部,并根据q值、差异倍数和基因丰度进行筛选关注的比较组的具体差异基因的分析情况。
步骤s5,对选定的基因集或差异分析结果集进行go富集分析、kegg富集分析和韦恩图分析。也就是说,可以通过选择基因集或差异分析结果集,对差异基因进行go富集分析和kegg富集分析,也可以对特定的基因集进行相关分析,同时也可以通过选择基因集或差异基因结果集进行比较分析,绘制出韦恩图输出。
优选地,本发明之rna-seq在线分析报告方法还包括:
根据选择基因集或差异分析结果集进行趋势分析,即可通过选择基因集或差异分析结果集,并进行分组方案选择、趋势数量、显著趋势的p值和基因筛选的最小变化倍数等参数设置,进行趋势分析并输出趋势分析结果。较佳地,还可根据通过选择比对数据进行blast分析,即通过上传或者输入query序列的fasta序列,并可以选择比对程序和比对数据库等,进行blast分析,分析完成,就会跳转到blast分析结果,同时也可以自主添加筛选条件对比对结果进行筛选。同时本发明还可以提供选择进行基因组浏览,即用户可以根据选择展示出cds、exon、gene、mrna等信息。
图3为本发明具体实施例中rna-seq在线分析报告系统的示意图。其主要包括:
(1)总览模块,即基本信息分析输出模块:输出分析项目的基本信息和数据列表两部分,其中基本信息包括有项目编号、样品名称、样品间差异方案、分组方案和分组间差异方案等详细信息;数据列表是展示样本中所有基因的表达量;
(2)样本关系模块:通过添加样本关系单按钮进行分组信息选择和组间比较的选择,生成一个新的任务,对选择的分组样本进行pca分析、相关性热图、样本聚类图和重复性散点图等,在pca分析时,可以选择是否显示样本名、颜色选择、字体修改、图形下载等按钮,可以根据需要自行修改;相关性热图,可以选择是否显示相关性系数,同样也可对颜色、字体大小、图标题等进行自定义;样本聚类图是利用全部基因的表达量对所有样本的关系进行层级聚类,图片可以自行下载;重复性散点图,是计算两个样本表达量之间的相关性;
(3)基因筛选模块:包括了基因筛选和基因序列筛选,基因筛选是通过输入所关心的geneid、description、pathway和go等查询出基因信息,基因序列筛选则是通过输入一个或多个基因id,点击“查询序列”即可得到基因的碱基序列、cds序列(如果有)和蛋白序列(如果有)等,可以进行fa格式文件的下载;
(4)差差异基因分析模块:对所选择的比较组样本的基因进行表达量差异分析。如图4所示,其中“总览”部分展示了样本关系单中所有比较组的差异表达结果,包括了差异基因统计柱状图、基因统计表和聚类热图。差异基因统计柱状图,如图5所示,可以选择是否显示上调基因、下调基因的具体数量,进行字体、标题、x轴和y轴的自定义进行结果的修改;基因统计表显示了所有分组样本之间上调基因和下调基因的具体数量;聚类热图是展现了所有分组样本之间的基因聚类情况,也可以根据需要每一组样本的聚类情况并进行图片下载。“分组详细”部分可以分别展示各个比较组的差异表达情况,可以进行q值、差异倍数和基因丰度的自主设定,进行所选择比较组的差异分析,并绘制出筛选差异基因的实时热图,也可以通过绘制差异基因雷达图,差异基因的表达情况可以用柱状图、盒型图、火山图、散点图和折线图等形式呈现,图形的颜色、字体等可以进行修改,如图6所示;
(5)基本功能分析模块:通过选择基因集或差异分析结果集,对差异基因进行go富集分析和kegg富集分析。也可以对特定的基因集进行相关分析,同时也可以通过选择基因集或差异基因结果集进行比较分析,绘制出韦恩图进行下载;
(6)高级功能分析模块:包括趋势分析、blast分析和基因组浏览,可以通过选择基因集或差异分析结果集,并进行分组方案选择、趋势数量、显著趋势的p值和基因筛选的最小变化倍数等参数设置,进行趋势分析并进行趋势分析结果的下载。blast分析,可以通过上传或者输入query序列的fasta序列,并可以选择比对程序和比对数据库等,进行blast分析。分析完成,就会跳转到blast分析结果,同时也可以自主添加筛选条件对比对结果进行筛选,根据个人需要下载原始比对结果或者筛选后的比对结果。基因组浏览,可以根据选择展示出cds、exon、gene、mrna等信息。
(7)任务列表模块:可以清楚地展示每个任务的类型、编号、开始时间和结束时间及任务进度,能实时查看任务进展。
(8)专属omicshare板块:可以查看生物信息学教学视频、生物信息分析的工具以及一些精华的帖子等。
综上所述,本发明一种rna-seq在线分析报告系统通过设计样本关系模块、基因筛选模块、差异基因分析模块以及基本功能分析模块,使得用户可以按需进行样本分组,挑选关注的基因进行分析,也可以自主设置过滤参数,筛选目标差异基因,对于分析结果可以根据个人需求进行图形修改、字体修改、图形下载等操作,这样使无编程基础的人员也能进行数据分析,具有操作简单、数据安全性高及分析结果动态化等优点,给用户得到满意的分析结果带来了便捷,同时也大大降低了生物信息分析人员的工作量,提高了工作效率,达到了双赢的效果。
与现有技术相比,本发明具有如下有益效果:
(1)通过云端服务平台封装式的设计,将后台复杂的实验、分析流程以简单、交互式的方式呈现给终端用户。使非生物信息背景的用户,也能够通过平台简单的操作界面完成复杂的生物信息分析。而这些工作过去只能由专业的生物信息人员才能完成。
(2)取代了传统的静态结题报告,可以根据个人需要自由挑选样本、设定样本分组、调整筛选参数等,具有动态挖掘数据的意义;同时,分析内容丰富,涵盖了rna-seq测序需要的差异基因筛选、基本功能分析和高级功能分析等,可以自主调整图片的颜色、字体大小、字体颜色、不同格式图片下载等,实现结果的个性化、动态化和可视化。
(3)用户可以自行根据需要进行分析调整,得到满意的结果,而不需要生物信息分析人员去重新分析,这样省时省力,提高了工作效率,达到客户和分析人员双赢的效果。
上述实施例仅例示性说明本发明的原理及其功效,而非用于限制本发明。任何本领域技术人员均可在不违背本发明的精神及范畴下,对上述实施例进行修饰与改变。因此,本发明的权利保护范围,应如权利要求书所列。
- 还没有人留言评论。精彩留言会获得点赞!