MoRFs预测模型的生成方法、装置、设备和存储介质与流程
本申请涉及生物信息技术领域,特别是涉及一种分子识别特征(英文:molecularrecognitionfeatures,简称:morfs)的预测模型生成方法和装置、以及一种morfs预测方法、装置、设备和存储介质。
背景技术:
通常,一些蛋白质在天然条件下能够折叠缠绕形成特定的空间三维结构,可以根据蛋白质的空间三维结构分析和确定出该蛋白质的生物功能;而另一些蛋白质则在天然条件下无法形成确定的空间三维结构,该类蛋白质称为本征无序蛋白质(英文:intrinsicallydisorderedproteins,简称:idps)。该idps由于空间三维结构的不确定性,所以无法通过分析其空间三维结构确定生物功能。
在idps中,morfs能够将无序的蛋白质序列转换为有序的蛋白质序列,体现出该idps和其他蛋白质结合的位点,分析该idps的生物功能,所以,确定idps中的morfs对分析该idps的生物功能具有重要意义。基于此,目前亟待提供一种能够快速、准确的识别idps中morfs的方法,以便分析和确定idps的生物功能。
技术实现要素:
为了解决上述技术问题,本申请实施例提供了一种morfs的预测模型生成方法、装置设备和存储介质,通过morfs的预测模型能够方便、快速和准确的识别出idps中的各位点是否属于morfs片段。
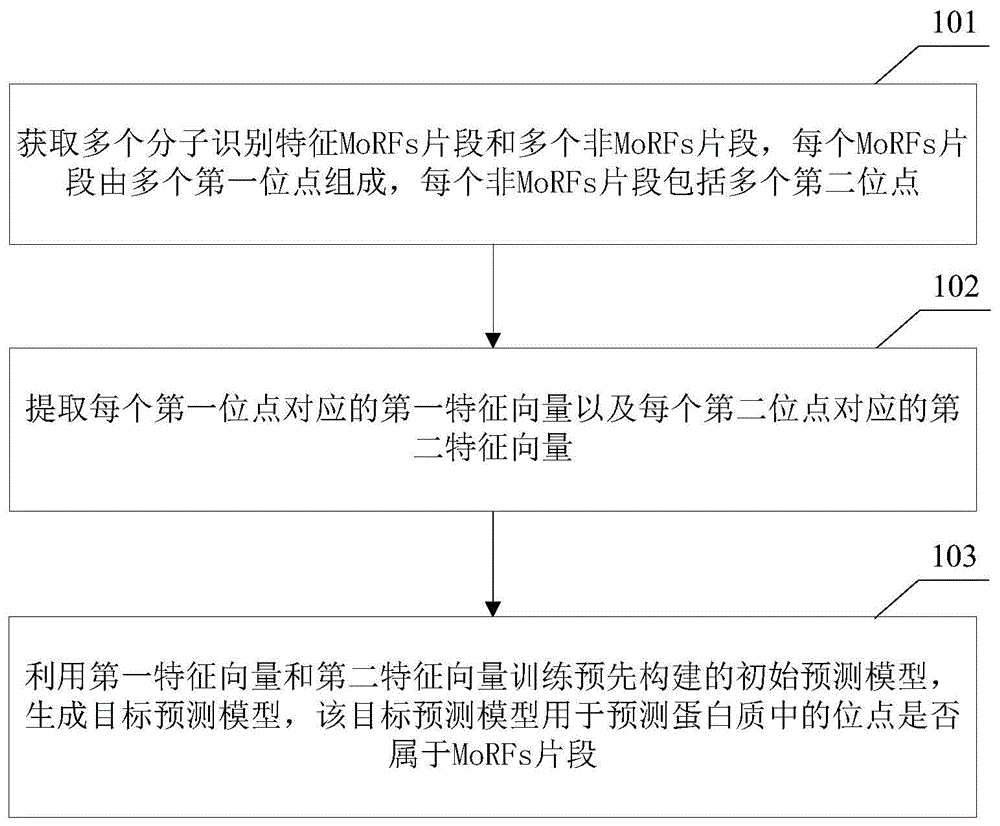
第一方面,提供了一种morfs预测模型的生成方法,包括:
获取多个分子识别特征morfs片段和多个非morfs片段,每个所述morfs片段由多个第一位点组成,每个所述非morfs片段包括多个第二位点;
提取每个所述第一位点对应的第一特征向量以及每个所述第二位点对应的第二特征向量;
利用所述第一特征向量和所述第二特征向量训练预先构建的初始预测模型,生成目标预测模型,所述目标预测模型用于预测蛋白质中的位点是否属于morfs片段。
可选地,所述第一特征向量的数量和所述第二特征向量的数量相同。
可选地,所述获取多个morfs片段和多个非morfs片段,包括:
从本征无序蛋白质idps序列库中,筛选多个所述morfs片段;
选取与各所述morfs片段相隔第一预设长度以外的多个所述非morfs片段。
可选地,所述提取每个所述第一位点对应的第一特征向量以及每个所述第二位点对应的第二特征向量,包括:
对于各所述morfs片段,利用蛋白质对比工具获得该morfs片段所在蛋白质对应的第一位置特异性得分矩阵pssm;
以该morfs片段中的每个所述第一位点为中心,基于所述第一pssm向外扩展第二预设长度,得到各所述第一位点对应的第一子特征向量;并且,以该非morfs片段中的每个所述第二位点为中心,基于所述第一pssm向外扩展所述第二预设长度,得到各所述第二位点对应的第二子特征向量;
根据各所述morfs片段所在蛋白质的氨基酸出现频率和理化性质,获得该morfs片段所在蛋白质的第三子特征向量;
基于所述第三子特征向量和每个所述第一位点对应的所述第一子特征向量,获得每个所述第一位点对应的所述第一特征向量;并且,基于所述第三子特征向量和每个所述第二位点对应的所述第二子特征向量,获得每个所述第二位点对应的所述第二特征向量。
可选地,所述方法还包括:
获取待预测蛋白质,所述待预测蛋白质包括n个位点,n为大于1的整数;
提取所述待预测蛋白质的第i位点对应的第i特征向量,其中,i=1,2,…,n;
根据所述第i特征向量和所述目标预测模型,获得第i预测结果,所述第i预测结果用于表征所述第i位点是否属于morfs片段。
可选地,所述提取所述待预测蛋白质的第i位点对应的第i特征向量,包括:
利用所述蛋白质对比工具获得所述待预测蛋白质对应的第二pssm,并以所述第i位点为中心,基于所述第二pssm向外扩展所述第二预设长度,得到所述第i位点对应的第四子特征向量;
根据所述待预测蛋白质的氨基酸出现频率和理化性质,获得所述待预测蛋白质的第五子特征向量;
基于所述第四子特征向量和所述第五子特征向量,获得所述第i位点对应的所述第i特征向量;
所述根据所述第i特征向量和所述目标预测模型,获得第i预测结果,具体为:
将所述第i特征向量输入所述目标预测模型,输出所述第i预测模型。
第二方面,还提供了一种morfs预测模型的生成装置,包括:
第一获取模块,用于获取多个分子识别特征morfs片段和多个非morfs片段,每个所述morfs片段由多个第一位点组成,每个所述非morfs片段包括多个第二位点;
第一提取模块,用于提取每个所述第一位点对应的第一特征向量以及每个所述第二位点对应的第二特征向量;
生成模块,用于利用所述第一特征向量和所述第二特征向量训练预先构建的初始预测模型,生成目标预测模型,所述目标预测模型用于预测蛋白质中的位点是否属于morfs片段。
可选地,所述第一特征向量的数量和所述第二特征向量的数量相同。
可选地,所述第一获取模块,包括:
第一获取单元,用于从本征无序蛋白质idps序列库中,筛选多个所述morfs片段;
第二获取单元,用于选取与各所述morfs片段相隔第一预设长度以外的多个所述非morfs片段。
可选地,所述第一提取模块,包括:
第三获取单元,用于对于各所述morfs片段,利用蛋白质对比工具获得该morfs片段所在蛋白质对应的第一位置特异性得分矩阵pssm;
第四获取单元,用于以该morfs片段中的每个所述第一位点为中心,基于所述第一pssm向外扩展第二预设长度,得到各所述第一位点对应的第一子特征向量;并且,以该非morfs片段中的每个所述第二位点为中心,基于所述第一pssm向外扩展所述第二预设长度,得到各所述第二位点对应的第二子特征向量;
第五获取单元,用于根据各所述morfs片段所在蛋白质的氨基酸出现频率和理化性质,获得该morfs片段所在蛋白质的第三子特征向量;
第六获取单元,用于基于所述第三子特征向量和每个所述第一位点对应的所述第一子特征向量,获得每个所述第一位点对应的所述第一特征向量;并且,基于所述第三子特征向量和每个所述第二位点对应的所述第二子特征向量,获得每个所述第二位点对应的所述第二特征向量。
可选地,所述装置还包括:
第二获取模块,用于获取待预测蛋白质,所述待预测蛋白质包括n个位点,n为大于1的整数;
第二提取模块,用于提取所述待预测蛋白质的第i位点对应的第i特征向量,其中,i=1,2,…,n;
第三获取模块,用于根据所述第i特征向量和所述目标预测模型,获得第i预测结果,所述第i预测结果用于表征所述第i位点是否属于morfs片段。
可选地,所述第二提取模块,包括:
第七获取单元,用于利用所述蛋白质对比工具获得所述待预测蛋白质对应的第二pssm,并以所述第i位点为中心,基于所述第二pssm向外扩展所述第二预设长度,得到所述第i位点对应的第四子特征向量;
第八获取单元,用于根据所述待预测蛋白质的氨基酸出现频率和理化性质,获得所述待预测蛋白质的第五子特征向量;
第九获取单元,用于基于所述第四子特征向量和所述第五子特征向量,获得所述第i位点对应的所述第i特征向量;
所述第三获取模块,具体用于:
将所述第i特征向量输入所述目标预测模型,输出所述第i预测模型。
第三方面,还提供了一种morfs预测模型的生成设备,所述设备包括处理器以及存储器:
所述存储器用于存储程序代码,并将所述程序代码传输给所述处理器;
所述处理器用于根据所述程序代码中的指令执行上述第一方面提供的所述方法。
第四方面,还提供了一种存储介质,所述存储介质用于存储程序代码,所述程序代码用于执行上述第一方面提供的所述方法。
与现有技术相比,本申请至少具有以下优点:
在本申请实施例中,首先,获取多个morfs片段和多个非morfs片段,每个morfs片段由多个第一位点组成,每个非morfs片段包括多个第二位点;然后,提取每个第一位点对应的第一特征向量以及每个第二位点对应的第二特征向量;接着,利用多个第一特征向量和多个第二特征向量训练预先构建的初始预测模型,生成目标预测模型,该目标预测模型用于预测蛋白质中的位点是否属于morfs片段。可见,通过本申请实施例提供的方法,仅需要提取该蛋白质上各位点的特征向量,借助训练完成的该目标预测模型,即可方便、快速和准确的预测出其上属于morfs片段的位点,从而识别出morfs片段,尤其对空间三维结构不确定的idps,由于该idps上morfs片段能够确定出该idps的生物功能,所以,通过本申请实施例提供的方法为快速、准确的确定出idps的生物功能提供了数据基础。
附图说明
为了更清楚地说明本申请实施例中的技术方案,下面将对实施例描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本申请中记载的一些实施例,对于本领域普通技术人员来讲,还可以根据这些附图获得其他的附图。
图1为本申请实施例提供的一种morfs预测模型的生成方法的流程示意图;
图2为本申请实施例提供的实现步骤101的一示例的流程示意图;
图3为本申请实施例提供的实现步骤102的一示例的流程示意图;
图4为本申请实施例提供的一种morfs预测方法的流程示意图;
图5为本申请实施例提供的实现步骤402的一示例的流程示意图;
图6为本申请实施例提供的一种morfs预测模型的生成装置的结构示意图;
图7为本申请实施例提供的一种morfs预测模型的生成设备的结构示意图。
具体实施方式
为了使本技术领域的人员更好地理解本申请方案,下面将结合本申请实施例中的附图,对本申请实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅是本申请一部分实施例,而不是全部的实施例。基于本申请中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本申请保护的范围。
目前,通常通过分析蛋白质的空间三维结构确定蛋白质的生物功能,但是对于空间三维结构不确定的idps,则无法通过对三维空间结构的分析确定出其生物功能。发明人经过研究发现,idps上通常存在morfs片段,该morfs片段能够将无确定顺序的idps序列转换为有序序列,从而揭示出idps的生物功能。可见,识别morfs片段对idps的分析、分类以及其他研究具有重要意义。
但是,目前还无法准确的识别出蛋白质上的morfs片段,基于此,本申请实施例提供了一种morfs预测模型的生成方法,通过获取多个morfs片段和多个非morfs片段,每个morfs片段由多个第一位点组成,每个非morfs片段包括多个第二位点;并提取每个第一位点对应的第一特征向量和每个第二位点对应的第二特征向量;接着,利用多个第一特征向量和多个第二特征向量训练预先构建的初始预测模型,生成目标预测模型,该目标预测模型用于预测蛋白质中的位点是否属于morfs片段。这样,通过本申请实施例提供的方法,仅需要提取该蛋白质上各位点的特征向量,借助训练完成的该目标预测模型,即可方便、快速和准确的预测出其上属于morfs片段的位点,从而识别出morfs片段,尤其对空间三维结构不确定的idps,由于该idps上morfs片段能够确定出该idps的生物功能,所以,通过本申请实施例提供的方法为快速、准确的确定出idps的生物功能提供了数据基础。
需要说明的是,本申请实施例中,位点特指蛋白质序列上的氨基酸,即,将蛋白质序列上的每个氨基酸都称为一个位点(也可以称为一个残基)。
下面结合附图,详细说明本申请实施例中的各种非限制性实施方式。
图1为本申请实施例提供的一种morfs预测模型的生成方法的流程示意图。参见图1,在本实施例中,该方法具体可以包括下述步骤101~步骤103:
步骤101,获取多个分子识别特征morfs片段和多个非morfs片段,每个morfs片段由多个第一位点组成,每个非morfs片段包括多个第二位点。
可以理解的是,可以通过分析蛋白质上的morfs片段确定蛋白质的生物功能,通常,一个idps上可以包括一个或多个morfs片段,每个morfs片段通常包括10到70个第一位点。多个morfs片段可以作为生成训练初始预测模型的训练样本的数据基础。
为了确保训练样本的全面性,除了需要有morfs片段上的第一位点参与训练之前,还需要有不在morfs片段上的其他位点参与训练。所以,步骤101中还获取了多条非morfs片段,非morfs片段是指蛋白质上除morfs片段以外的其他片段,该非morfs片段包括的位点记作第二位点。
可以理解的是,为了确保训练样本的均衡性,使得训练所得的目标预测模型鲁棒性更强,可以在步骤101中获取多个morfs片段和多个非morfs片段时,保证所有morfs片段包括的第一位点的总数和所有非morfs片段包括的第二位点的总数相同。这样,确保用于训练morfs预测模型的数据来源中,二分之一属于已知属于morfs片段的第一位点,另外二分之一属于已知不属于morfs片段的第二位点,一定程度上使得训练完成的morfs预测模型能够更加准确的完成morfs位点的预测。
作为一个示例,步骤101中获取多个morfs片段和多个非morfs片段,具体可以通过下述图2所示的方式实现。参见图2,例如可以包括下述步骤1011~步骤1012:
步骤1011,从本征无序蛋白质idps序列库中,筛选多个morfs片段;
可以理解的是,由于识别morfs片段对idps尤其关键,所以,本实施例中从idps序列库中选取参与训练的样本。idps序列库例如可以是:disprotversion8.0固有无序蛋白质序列库。
具体实现时,可以先在idps序列库中,通过人工实验以及生物文献校对的方式,获得idps;再从这些idps中确定出morfs片段。例如:可以从disprotversion8.0固有无序蛋白质序列库中,获得364条idps,并从该364条idps确定702个morfs片段,这702个morfs片段共包括15,542个位点。
步骤1012,选取与各morfs片段相隔第一预设长度以外的多个非morfs片段。
其中,第一预设长度例如可以取12个位点,具体可以根据需求针对性的设置,在本实施例中不作限定。
例如:假设idps1、idps2和idps3均包括200个位点,基于步骤1011获得3个morfs片段:morfs片段1、morfs片段2和morfs片段3,其中,morfs片段1包括第10到第39个共30位点,morfs片段2包括第50到第100个共51位点,morfs片段3包括第130到第166个共37位点,那么,假设第一预测长度为24个位点,则,通过步骤1012可以确定:对于idps1,非morfs片段1为第64到第200个位点;对于idps2,非morfs片段2为第1到第25个位点以及第125到第200个位点;对于idps3,非morfs片段3为第1到第105个位点以及第191到第200个位点。这样,确定的多个morfs片段共包括(30+51+37)=118个第一位点,多个非morfs片段共包括(137+25+76+105+10)=353个第二位点。
其中,为了确保训练样本的均衡,还可以统计多个morfs片段共包括的第一位点的总数,从第二位点中随机筛选出和第一位点总数相同的位点,作为参与训练morfs预测模型的第二位点,并将该筛选确定的第二位点所在的片段记作步骤1012中获得的非morfs片段。例如,将上述例子中353个第二位点,通过随机筛选,确定118个第二位点参与morfs预测模型的训练。
可见,通过上述图2所示的实现方式,可以实现上述步骤101,获得包括第一位点的多个morfs片段和包括第二位点的多个非morfs片段,为后续提供丰富和完善的训练样本以及训练出准确的morfs预测模型,提供了数据基础。
步骤102,提取每个第一位点对应的第一特征向量以及每个第二位点对应的第二特征向量。
可以理解的是,第一特征向量用于表征其对应的第一位点的特征,该第一特征向量和第一位点一一对应,两者的数量相同。第二特征向量用于表征其对应的第二位点的特征,该第二特征向量和第二位点一一对应,两者的数量相同。若第一位点和第二位点的数量相同,则,第一特征向量的数量和第二特征向量的数量也相同。
具体实现时,考虑到morfs的特点,为了准确的描述出morfs片段上位点的特征,本申请实施例将融合morfs片段或非morfs所在蛋白质序列的氨基酸出现频率和理化性质特征以及各第一位点或第二位点的同源进化特征,获得能够更加丰富的表征各位点的特征向量,为训练morfs预测模型提供了具有针对性、更加丰富和完善的训练样本。
作为一个示例,步骤102具体可以通过下述图3所示的方式实现。参见图3,例如可以包括下述步骤1021~步骤1024:
步骤1021,对于各morfs片段,利用蛋白质对比工具获得该morfs片段所在蛋白质对应的第一位置特异性得分矩阵(英文:positionspecificscoringmatrix,简称:pssm)。
可以理解的是,为了体现蛋白质的同源进化特征,采用pssm对morfs片段所在蛋白质进行分析和处理,可以大大的提高处理结果的准确性。需要说明的是,由于步骤101中获取非morfs片段时,在morfs片段所在的蛋白质中进行筛选,则可以确定选取的morfs片段和非morfs片段属于同一个蛋白质,morfs片段所在的蛋白质涵盖了所有morfs片段和非morfs片段。
具体实现时,步骤1021的实现过程具体可以包括:s11,在蛋白质序列数据库中查找该morfs片段所在蛋白质的同源蛋白质;s12,将morfs片段所在蛋白质与同源蛋白质的氨基酸序列进行多序列比对,得到该morfs片段所在蛋白质的第一pssm。
其中,蛋白质序列数据库,是指应用计算机功能分析生物学信息的数据库。应用计算机的运算法则,比较氨基酸序列而预测蛋白质的结构和功能。例如:该蛋白质序列数据库可以是非冗余蛋白质序列数据库,该数据库包含152,910,397条蛋白质,每条蛋白质对应的氨基酸序列的信息例如可以包括:标定好的该氨基酸序列中各个位置的氨基酸,以及该氨基酸序列是否具备和糖类结合的功能,是否具备和脂质结合的功能等。
可以理解的是,同源蛋白质,是指来自不同种类生物、而对应的氨基酸序列类似的蛋白质。
作为一个示例,可以采用同源检测迭代的hmm-hmm的比较(英文:homologydetectionbyiterativehmm-hmmcomparison,简称:hhblits)工具,作为该步骤1021中的“蛋白质对比工具”,获得各morfs片段所在蛋白质对应的第一pssm。
作为另一个示例,获得该morfs片段所在蛋白质的第一pssm的具体过程也可以包括:首先,从蛋白质序列数据库中检索出该morfs片段所在蛋白质的多个同源蛋白质,并从该蛋白质序列数据库中获取这些同源蛋白质的氨基酸序列;接着,可以将检索到的同源蛋白质的氨基酸序列和morfs片段所在蛋白质的氨基酸序列进行多序列比对,得到该morfs片段所在蛋白质的氨基酸序列的第一pssm。
其中,多序列比对具体可以采用位置特定的迭代-基本局部对齐搜索工具(英文:position-specificiterated-basiclocalalignmentsearchtool,简称:psi-blast)。假设psi-blast设置最大迭代次数为4,e值的临界值为0.001,采用该psi-blast进行多序列比对该同源蛋白质的氨基酸序列和morfs片段所在蛋白质的氨基酸序列,得到的第一pssm例如可以如下表1所示:
表1第一pssm
其中,横向的“a、r、n、d、c、q、e、……、v”表示组成蛋白质的氨基酸序列的20中氨基酸;纵向的“1m、2k、3i、4s、5f、6h、……”表示该类同源的蛋白质的氨基酸序列中每个位置的位置序号和该位置上的氨基酸;中间的数字为位置特异性得分,用于表示该氨基酸出现在该位置的可能性大小(也称为倾向程度或者保守程度),一般该位置特异性得分的取值范围为-13到+13。例如:上述表格中第二行和第三列交叉位置处的“-6”(字体加粗且加下划线的得分),表示该morfs片段所在蛋白质的氨基酸序列中,第一个位置出现氨基酸r的可能性得分为-6;再例如:上述表格中第五行和第五列交叉位置处的“-4”(字体加粗且加下划线的得分),表示该morfs片段所在蛋白质的氨基酸序列中,第四个位置出现氨基酸d的可能性得分为-4。
可以理解的是,若morfs片段所在蛋白质的长度为n,则,获得该morfs片段所在蛋白质对应的第一pssm为n×20的矩阵。
步骤1022,以该morfs片段中的每个第一位点为中心,基于第一pssm向外扩展第二预设长度,得到各第一位点对应的第一子特征向量;并且,以该非morfs片段中的每个第二位点为中心,基于第一pssm向外扩展第二预设长度,得到各第二位点对应的第二子特征向量。
可以理解的是,对于每个morfs片段中每个第一位点,都执行一次步骤1022,获得该第一位点对应的第一子特征向量。同理,对于每个非morfs片段中每个第二位点,都执行一次步骤1022,获得该第二位点对应的第二子特征向量。
例如,假设morfs片段所在蛋白质的长度为20,第一pssm为:
其中,第二预设长度也可以取5,那么,第一子特征向量为(5*2+1)*20=220维的特征向量。第二预设程度可以根据技术人员的经验进行设计,在本申请实施例中不作具体限定。
其中,“以该非morfs片段中的每个第二位点为中心,基于第一pssm向外扩展第二预设长度,得到各第二位点对应的第二子特征向量”的实现方式参见上述关于确定第一位点对应的第一子特征向量的相关描述,在此不再赘述。
第一子特征向量和第二子特征向量的维数相同,具体可以根据对蛋白质特征需求的不同,设置不同的维数,例如:第一子特征向量和第二子特征向量均可以是220维的特征向量。
由于第一位点已知为morfs片段上的位点,所以,该第一位点对应的第一子特征向量,能够表现出该位点为morfs片段上的位点。同理,由于第二位点已知为非morfs片段上的位点,所以,该第二位点对应的第二子特征向量,能够表现出该位点为非morfs片段上的位点,不属于morfs片段。
步骤1023,根据各morfs片段所在蛋白质的氨基酸出现频率和理化性质,获得该morfs片段所在蛋白质的第三子特征向量。
其中,理化性质,即蛋白质的物理化学性质。氨基酸出现频率,是指某种氨基酸在蛋白质序列中出现的频率。
具体实现时,步骤1023具体可以包括:
s21,根据下述公式(1)计算20种氨基酸的亲水性和疏水性的归一化结果:
其中,r表示20种氨基酸中的任意一种,pr,k用于表示氨基酸r的亲水性或疏水性,例如:k=0,pr,k表示氨基酸r的亲水性,k=1,pr,k表示氨基酸r的疏水性;pk用于表示20种氨基酸的亲水性平均值或疏水性平均值;sk表示20种氨基酸的亲水性标准差或疏水性标准差;nr,k表示20种氨基酸中每种氨基酸的亲水性归一化结果或疏水性归一化结果。
s22,设置氨基酸的距离lag=1、2、……、λ,其中,λ为预设参数,例如可以取10。计算各lag下morfs片段所在蛋白质的理化参数θlag,具体可以通过下述公式(2)计算θlag:
其中,n表示morfs片段所在蛋白质的长度,i表示蛋白质序列的序号,xi表示序号i所对应的氨基酸,nxi,k表示氨基酸xi的亲水性归一化结果或疏水性归一化结果。
s23,根据下述公式(3)计算morfs片段所在蛋白质的特征xu:
其中,u表示生成特征向量的维度,fj表示氨基酸j的频率,20种氨基酸对应的出现频率均已知;w为预设参数,例如可以取w=0.05,对于xu而言,一种情况下,对于该特征向量的前20维特征,即,u在1到20之间时,采用公式(3)中的第一个公式计算xu,即,fu表示第u维氨基酸在该蛋白质中出现的频率,对于确定的蛋白质序列,fu是已知的,通过20种氨基酸的出现频率体现morfs片段所在蛋白质的特征;另一种情况下,对于该特征向量的第21维到第(20+λ)维特征,即,u在21到(20+λ)之间时,采用公式(3)中的第二个公式计算xu,即,通过λ个理化参数体现morfs片段所在蛋白质的特征。
可见,当u=1、2、……、20+λ中的每个值时,均可以通过上述公式(3)得到对应的特征xu。
s24,基于morfs片段所在蛋白质的特征xu,获得morfs片段所在蛋白质的第三子特征向量φ,具体可以表示为:
φ(morfs片段所在蛋白质)=[x1,x2,……,x20+λ]
其中,当λ=10时,该第三子特征向量为(20+10)=30维的特征向量。该第三子特征向量用于表征morfs片段所在蛋白质的氨基酸出现频率和理化性质。
如此,通过上述s21~s24能够实现步骤1023,得到能够表现morfs片段的各位点在其所在的蛋白质的氨基酸序列中的特征,为构建更加丰富和针对性的训练样本提供了数据基础。
步骤1024,基于第三子特征向量和每个第一位点对应的第一子特征向量,获得每个第一位点对应的第一特征向量;并且,基于第三子特征向量和每个第二位点对应的第二子特征向量,获得每个第二位点对应的第二特征向量。
对于每个morfs片段所在蛋白质对应一个第三子特征向量,该morfs片段上的每个第一位点对应一个第一子特征向量,那么,以每个第一位点为对象,可以将其对应的第一子特征向量和所属morfs片段所在的蛋白质对应的第三子特征向量进行融合,获得该第一位点对应的第一特征向量。同理,对于每个非morfs片段所在蛋白质对应一个第三子特征向量,该非morfs片段上的每个第二位点对应一个第二子特征向量,那么,以每个第二位点为对象,可以将其对应的第二子特征向量和所属非morfs片段所在的蛋白质对应的第三子特征向量进行融合,获得该第二位点对应的第二特征向量。
例如:对于morfs片段1,其上包括:第一位点1、第一位点2和第一位点3,根据步骤1023获得morfs片段1所在蛋白质1对应的第三子特征向量1,根据步骤1021~步骤1022分别获得第一位点1对应的第一子特征向量1、第一位点2对应的第一子特征向量2和第一位点3对应的第一子特征向量3;那么,在步骤1024中,可以将第一子特征向量1和第三子特征向量1融合,得到第一位点1对应的第一特征向量1;将第一子特征向量2和第三子特征向量1融合,得到第一位点2对应的第一特征向量2;将第一子特征向量3和第三子特征向量1融合,得到第一位点3对应的第一特征向量3。
又例如:对于非morfs片段1,其上包括:第二位点1、第二位点2和第二位点3,根据步骤1023获得非morfs片段1所在蛋白质2对应的第三子特征向量1,根据步骤1021~步骤1022分别获得第二位点1对应的第二子特征向量1、第二位点2对应的第二子特征向量2和第二位点3对应的第二子特征向量3;那么,在步骤1024中,可以将第二子特征向量1和第三子特征向量1融合,得到第二位点1对应的第二特征向量1;将第二子特征向量2和第三子特征向量1融合,得到第二位点2对应的第二特征向量2;将第二子特征向量3和第三子特征向量1融合,得到第二位点3对应的第二特征向量3。
其中,将第一子特征向量和第三子特征向量融合,获得第一特征向量,具体可以是:将第一子特征向量和第三子特征向量进行拼接,获得第一特征向量,拼接的顺序可以不作具体限定。例如:假设第一子特征向量为220维的特征向量a,第三子特征向量为30维的特征向量b,那么,第一特征向量为250维特征向量c,特征向量c可以表示为[a,b]或者[b,a]。
如此,通过上述图3所示的方式,可以提取到已知属于morfs片段的第一位点对应的第一特征向量,以及已知不属于morfs片段的第二位点对应的第二特征向量,作为训练morfs预测模型的训练样本,为进行后续morfs预测模型的训练做好了准备。
步骤103,利用第一特征向量和第二特征向量训练预先构建的初始预测模型,生成目标预测模型,该目标预测模型用于预测蛋白质中的位点是否属于morfs片段。
可以理解的是,初始预测模型,可以预先构建的、用于预测蛋白质中位点是否属于morfs片段的模型。该初始预测模型具体可以是分类模型,那么,该初始预测模型的输入为蛋白质上位点对应的特征向量,输出包括两种情况,一种情况,输出用于表征该位点属于morfs片段,例如:输出“是”,另一种情况,输出用于表征该位点不属于morfs片段,例如:输出“否”。
具体实现时,可以将各第一位点对应的第一特征向量和各第二位点对应的第二特征向量,输入到该初始预测模型中,通过对比实际输出结果和目标输出结果的差异,调整该初始预测模型。需要说明的是,当采用训练样本中的下一个特征向量1进行训练时,需要将该下一个特征向量1输入至最新调整后的初始预测模型1,获得该次训练的实际输出结果,用该次训练的实际输出结果和目标输出结果的差异继续调整该最新调整后的初始预测模型1,获得最新调整后的初始预测模型2;当采用训练样本中的下一个特征向量2进行训练时,需要将该下一个特征向量2输入至最新调整后的初始预测模型2,获得该次训练的实际输出结果,用该次训练的实际输出结果和目标输出结果的差异继续调整该最新调整后的初始预测模型2,获得最新调整后的初始预测模型3;以此类推,直至训练样本中所有的特征向量(即所有的第一位点对应的第一特征向量和所有的第二位点对应的第二特征向量)都参与初始预测模型的训练为止,或者,直至最新调整后的初始预测模型的预测准确率达到预设准确率阈值(例如:98%)为止,此时,该最新调整的初始预测模型即为目标预测模型。
作为一个示例,如果将第一特征向量输入初始预测模型,已知目标输出结果为该第一位点属于morfs片段,若实际输出结果表征该第一位点属于morfs片段,则认为实际输出结果和目标输出结果一致,不调整初始预测模型;若实际输出结果表征该第一位点不属于morfs片段,则认为实际输出结果和目标输出结果不一致,调整初始预测模型。
作为另一个示例,如果将第二特征向量输入初始预测模型,已知目标输出结果为该第二位点不属于morfs片段,若实际输出结果表征该第二位点属于morfs片段,则认为实际输出结果和目标输出结果不一致,则调整初始预测模型;若实际输出结果表征该第二位点不属于morfs片段,则认为实际输出结果和目标输出结果一致,不对初始预测模型进行调整。
例如,由于支持向量机(英文:supportvectormachine,简称:svm),是一种按监督学习方式对数据进行二元分类的广义线性分类器,使用损失函数计算经验风险并在求解中加入了正则化项以优化结构风险,具有较强的鲁棒性,而且,svm可以还通过核方法(英文:kernelmethod)进行非线性分类,准确度较高,所以,本申请实施例中的初始预测模型可以采用svm。
为了使训练svm的过程更加准确,训练得到的svm的泛化能力尽可能好,防止在训练过程中svm的过拟合,可以使用软间隔技术和核函数技术来不断优化svm算法本身的泛化性能。可以理解的是,svm中核函数用于解决低维数据线性不可分的问题,能够将低维数据映射到高维空间从而达到可分的目的。本申请实施例中,svm的核函数可以采用径向基函数(英文:radialbasisfunction,简称:rbf),具体如下公式(4)所示:
其中,x和z分别为训练样本中的两个特征向量,gamma为核函数rbf自带的一个参数,决定了数据映射到新的高维空间后的分布,参数gamma主要是对低维数据进行高维空间的映射。
其中,在线性不可分问题中使用硬边距svm将产生分类误差,因此可在最大化边距的基础上引入损失函数构造新的优化问题。svm使用铰链损失函数,并使用松弛变量ξi处理铰链损失函数的分段取值后,沿用硬边界svm的优化问题形式,软边距svm的优化问题有如下表示:
s.t.,yi(wtxi+b)≥1-ξi,ξi≥0,i=1,…,n……公式(6)
公式(6)是公式(5)寻优求解的情况下的约束条件。其中,公式(5)和公式(6)中的w表示超平面的法向量;公式(6)中的b表示该超平面的截距,xi表示训练样本中的任意特征向量,yi表示该特征向量xi已知应该被分的类别,即,目标输出结果;c表示正则化系数。
可见,svm算法参数分别为c和gamma,c越大,表示该svm的分类越严格,不能有错误;反之,c越小,意味着有更大的错误容忍度;gamma值越大,映射到的高维空间的维度越高,表示训练的结果越好,但是,也越容易引起过拟合,即泛化能力低。
基于此,本申请实施例中,通过不断的调整gamma和c的值,不断地进行交叉验证,从而确定出合适的gamma和c的值,再进入训练样本中下一个特征向量的预测。其中,评估对初始预测模型的训练效果时,可以使用5-fold交叉验证,将训练样本集合(即包括所有的第一特征向量和第二特征向量的集合)中分为5份训练样本子集合,选取其中4份用作训练,1份用作测试,测试获得马修斯相关系数(英文:matthewscorrelationcoefficient,简称:mcc)评估指标得分。5份训练样本子集合得到的mcc的取平均,作为本次交叉验证的最终评估得分。
其中,mcc考虑到真阳性、假阳性、真阴性和假阴性,通常被视为一种平衡的措施,即使真阳性、假阳性、真阴性和假阴性这些类别的规模大小不同也可以使用。从混淆矩阵中计算mcc的公式如下:
其中,tp称为真阳性,即,目标输出结果表示该位点属于morfs片段,实际输出结果表示该位点也属于morfs片段;fp称为假阴性,即,目标输出结果表示该位点不属于morfs片段,实际输出结果表示该位点属于morfs片段;tn称为真阴性,即,目标输出结果表示该位点不属于morfs片段,实际输出结果表示该位点也不属于morfs片段;fn称为假阳性,即,目标输出结果表示该位点属于morfs片段,实际输出结果表示该位点不属于morfs片段。
可见,通过本申请实施例提供的morfs预测模型的生成方法,构建并训练完成目标预测模型(也即生成的morfs预测模型),该目标预测模型用于预测蛋白质上的位点是否属于morfs片段。这样,仅需要提取该蛋白质上各位点的特征向量,借助训练完成的该目标预测模型,即可方便、快速和准确的预测出其上属于morfs片段的位点,从而识别出morfs片段,尤其对空间三维结构不确定的idps,由于该idps上morfs片段能够确定出该idps的生物功能,所以,通过本申请实施例提供的方法为快速、准确的确定出idps的生物功能提供了数据基础。
在图1所示实施例的基础上,本申请实施例还提供了借助步骤103中生成的目标预测模型对蛋白质上morfs的预测方法。图4示出了一种morfs预测方法的流程示意图,参见图4,该方法例如可以包括下述步骤401~步骤403:
步骤401,获取待预测蛋白质,待预测蛋白质包括n个位点,n为大于1的整数;
步骤402,提取待预测蛋白质的第i位点对应的第i特征向量,其中,i=1,2,…,n;
步骤403,根据第i特征向量和目标预测模型,获得第i预测结果,第i预测结果用于表征第i位点是否属于morfs片段。
可以理解的是,对于待预测蛋白质,其上包括n个位点,若需要利用图1生成的目标预测模型预测该待预测蛋白质上各位点是否为morfs,则,首先,需要提取该待预测蛋白质上第i位点的第i特征向量。参见图5,步骤402具体可以包括:
步骤4021,利用蛋白质对比工具获得待预测蛋白质对应的第二pssm,并以第i位点为中心,基于第二pssm向外扩展第二预设长度,得到第i位点对应的第四子特征向量;
步骤4022,根据待预测蛋白质的氨基酸出现频率和理化性质,获得待预测蛋白质的第五子特征向量;
步骤4023,基于第四子特征向量和第五子特征向量,获得第i位点对应的第i特征向量。
其中,蛋白质对比工具采用与图3所示实施例中相同的蛋白质对比工具,步骤4022的实现方式具体可以参见图3所示实施例中步骤1023的“s21~s24”;第二预设长度也采用与图3所示实施例中相同的第二预设长度。具体实现方式可以参见图3所示实施例的相关说明,在此不再赘述。
需要说明的是,步骤4023中“基于第四子特征向量和第五子特征向量,获得第i位点对应的第i特征向量”采用的方式,应该与步骤1024中“基于第三子特征向量和每个第一位点对应的第一子特征向量,获得每个第一位点对应的第一特征向量”以及“基于第三子特征向量和每个第二位点对应的第二子特征向量,获得每个第二位点对应的第二特征向量”的方式一致。
其中,步骤403中根据第i特征向量和目标预测模型,获得第i预测结果,具体为:将第i特征向量输入目标预测模型,输出该第i预测模型。
在一些具体的实现方式中,对于待预测蛋白质的n个位点,可以都执行上述步骤402~步骤403,即,待预测蛋白质上的每个位点,均得到一个对应的特征向量,并借助目标预测模型,均获得对应的预测结果,那么,当n个预测结果中,存在m个连续的位点对应的预测结果均表征其对应的位点属于morfs片段,则可以确定这m个位点组成该待预测蛋白质的一个morfs片段,其中,m通常大于等于10个位点且小于等于70个位点。
可见,通过本申请实施例提供的morfs预测方法,不仅可以生成目标预测模型,而且仅需要提取待预测蛋白质上各位点对应的特征向量,借助训练完成的该目标预测模型,即可准确的预测出该蛋白质上的位点是否属于morfs片段,从而识别出morfs片段,尤其对空间三维结构不确定的idps,由于该idps上morfs片段能够确定出该idps的生物功能,所以,通过本申请实施例提供的方法为快速、准确的确定出idps的生物功能提供了数据基础。
相应的,本申请实施例还提供了一种morfs预测模型的生成装置,如图6所示,该装置具体可以包括:
第一获取模块601,用于获取多个分子识别特征morfs片段和多个非morfs片段,每个所述morfs片段由多个第一位点组成,每个所述非morfs片段包括多个第二位点;
第一提取模块602,用于提取每个所述第一位点对应的第一特征向量以及每个所述第二位点对应的第二特征向量;
生成模块603,用于利用所述第一特征向量和所述第二特征向量训练预先构建的初始预测模型,生成目标预测模型,所述目标预测模型用于预测蛋白质中的位点是否属于morfs片段。
可选地,所述第一特征向量的数量和所述第二特征向量的数量相同。
可选地,所述第一获取模块601,包括:
第一获取单元,用于从本征无序蛋白质idps序列库中,筛选多个所述morfs片段;
第二获取单元,用于选取与各所述morfs片段相隔第一预设长度以外的多个所述非morfs片段。
可选地,所述第一提取模块602,包括:
第三获取单元,用于对于各所述morfs片段,利用蛋白质对比工具获得该morfs片段所在蛋白质对应的第一位置特异性得分矩阵pssm;
第四获取单元,用于以该morfs片段中的每个所述第一位点为中心,基于所述第一pssm向外扩展第二预设长度,得到各所述第一位点对应的第一子特征向量;并且,以该非morfs片段中的每个所述第二位点为中心,基于所述第一pssm向外扩展所述第二预设长度,得到各所述第二位点对应的第二子特征向量;
第五获取单元,用于根据各所述morfs片段所在蛋白质的氨基酸出现频率和理化性质,获得该morfs片段所在蛋白质的第三子特征向量;
第六获取单元,用于基于所述第三子特征向量和每个所述第一位点对应的所述第一子特征向量,获得每个所述第一位点对应的所述第一特征向量;并且,基于所述第三子特征向量和每个所述第二位点对应的所述第二子特征向量,获得每个所述第二位点对应的所述第二特征向量。
可选地,所述装置还包括:
第二获取模块,用于获取待预测蛋白质,所述待预测蛋白质包括n个位点,n为大于1的整数;
第二提取模块,用于提取所述待预测蛋白质的第i位点对应的第i特征向量,其中,i=1,2,…,n;
第三获取模块,用于根据所述第i特征向量和所述目标预测模型,获得第i预测结果,所述第i预测结果用于表征所述第i位点是否属于morfs片段。
可选地,所述第二提取模块,包括:
第七获取单元,用于利用所述蛋白质对比工具获得所述待预测蛋白质对应的第二pssm,并以所述第i位点为中心,基于所述第二pssm向外扩展所述第二预设长度,得到所述第i位点对应的第四子特征向量;
第八获取单元,用于根据所述待预测蛋白质的氨基酸出现频率和理化性质,获得所述待预测蛋白质的第五子特征向量;
第九获取单元,用于基于所述第四子特征向量和所述第五子特征向量,获得所述第i位点对应的所述第i特征向量;
所述第三获取模块,具体用于:
将所述第i特征向量输入所述目标预测模型,输出所述第i预测模型。
上述描述为morfs预测模型的生成装置的相关描述,其中,具体实现方式以及达到的效果,可以参见上述morfs预测模型的生成方法实施例的描述,这里不再赘述。
此外,本申请实施例还提供了一种morfs预测模型的生成设备,如图7所示,该设备包括处理器701以及存储器702:
所述存储器702用于存储程序代码,并将所述程序代码传输给所述处理器701;
所述处理器701用于根据所述程序代码中的指令执行上述morfs预测模型的生成方法。
该morfs预测模型的生成设备的具体实现方式以及达到的效果,可以参见上述morfs预测模型的生成方法实施例的描述,这里不再赘述。
此外,本申请实施例还提供了一种存储介质,所述存储介质用于存储程序代码,所述程序代码用于执行上述morfs预测模型的生成方法。
本申请实施例中提到的“第一位点”、“第一特征向量”等名称中的“第一”只是用来做名字标识,并不代表顺序上的第一。该规则同样适用于“第二”等。
通过以上的实施方式的描述可知,本领域的技术人员可以清楚地了解到上述实施例方法中的全部或部分步骤可借助软件加通用硬件平台的方式来实现。基于这样的理解,本申请的技术方案可以以软件产品的形式体现出来,该计算机软件产品可以存储在存储介质中,如只读存储器(英文:read-onlymemory,rom)/ram、磁碟、光盘等,包括若干指令用以使得一台计算机设备(可以是个人计算机,服务器,或者诸如路由器等网络通信设备)执行本申请各个实施例或者实施例的某些部分所述的方法。
本说明书中的各个实施例均采用递进的方式描述,各个实施例之间相同相似的部分互相参见即可,每个实施例重点说明的都是与其他实施例的不同之处。尤其,对于装置实施例和设备实施例而言,由于其基本相似于方法实施例,所以描述得比较简单,相关之处参见方法实施例的部分说明即可。以上所描述的设备及装置实施例仅仅是示意性的,其中作为分离部件说明的模块可以是或者也可以不是物理上分开的,作为模块显示的部件可以是或者也可以不是物理模块,即可以位于一个地方,或者也可以分布到多个网络单元上。可以根据实际的需要选择其中的部分或者全部模块来实现本实施例方案的目的。本领域普通技术人员在不付出创造性劳动的情况下,即可以理解并实施。
以上所述仅是本申请的优选实施方式,并非用于限定本申请的保护范围。应当指出,对于本技术领域的普通技术人员来说,在不脱离本申请的前提下,还可以作出若干改进和润饰,这些改进和润饰也应视为本申请的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!