一种确定个性化药剂的方法与流程
一种确定个性化药剂的方法
1.本发明涉及一种方法,用于鉴定一种或多种与个体患者的给定患病组织(diseased tissue)的靶结构进行特异性结合的化合物。所述方法包括确定一组化合物对一个或多个与在该患者中确定的突变基因的一个或多个对接空间进行结合的亲和力,以及鉴定一种或多种与突变蛋白质特异性结合的化合物。此外,本发明还涉及一种计算机程序,其包括数个指令使计算机执行所述方法的几个步骤。
2.尽管在过去的几十年中已经开发了多种治疗策略,但基于患病组织的疾病通常仍然严重。特别是,肿瘤,例如癌症,仍然是一种危及生命的疾病。用于治疗肿瘤的治疗手段一般具有严重的不良副作用且疗效通常有限。抗肿瘤剂和辐射疗法通常也会对健康组织产生负面影响。尽管在过去几十年中抗肿瘤治疗取得了相当大的进步,但化疗的成功仍然受到严重和甚至危及生命的副作用的阻碍,这些副作用使得不能应用足够高的药物剂量来杀死反应性较低的肿瘤细胞。此外,肿瘤,例如恶性肿瘤,通常至少部分地对抗肿瘤剂具有抗性。因此,经常出现对抗癌药物的耐药性。这可能最终导致化疗的失败,对许多患者造成致命的后果。因此,现在仍然有需要鉴定可用于抗肿瘤治疗的其他药剂及其组合。
3.进一步的抗肿瘤剂的开发通常是非常昂贵和费力的。需要全面的临床前和临床试验。分子诊断的进步必须得到新药的同步支持。药物开发和营销是一个时间和成本密集的过程。
4.尽管近年来药物研发(r&d)的时间和支出持续增加,近几十年来新药批准的数量(例如fda批准的药物)却下降,主要是因为在临床ii期试验中的失败率高[mullard,2011;kola and landis,2004]。
[0005]
传统的肿瘤化疗基于官方标准治疗指南定义的治疗方案。这些化疗方案基于前瞻性、随机、双盲i-iii期研究的结果。然而,每个肿瘤的表现可能不同。因此,尽管可以从临床试验的结果估计大组患者的治疗反应的统计概率,但仍然无法可靠地预测个体患者的治疗成功。原因是,即使是同一起源和组织学的肿瘤,也常常因患者的个体生物学行为而异。尽管越来越多的新型靶向药物进入市场(例如小分子抑制剂和治疗性抗体),但其中许多药物的作用不足,无法持续和持久地改善肿瘤疾病。
[0006]
因此,人们经常尝试鉴定可用于新治疗用途的药物活性物质。即,将药学上用于另一治疗领域的已知药物进一步用于抗肿瘤治疗。进而,出现了一个令人惊讶的概念[aronson,2007]。
[0007]
wo 2001/035316描述了基于遗传多态性(genetic polymorphisms)的基于计算机的药物设计方法,重点是治疗病毒感染。然而,该文献并未提及病变组织,特别是未提及肿瘤组织。所描述的多态性可能会影响抗病毒药物的可用性。wo
[0008]
2003/057173描述了一种鉴定广谱抑制剂的方法,该抑制剂能够结合已知的野生型(wild-type)靶结构以及已知的变异(variant)靶结构。该文的目的是证明抑制剂对不同的变体可能都有活性。这种方法侧重于抗病毒疗法。然而,该方法不可能用来鉴定可以选择性与病变组织的目标结构相结合的化合物。
[0009]
对某些疾病具有熟知的安全性和药代动力学特征的现有药物可能作为有价值的
候选药物,用于治疗受相同途径影响的其他疾病。这种现象被描述为“药物重新定位”(也称为:“药物重新利用”、“靶外使用”或“标签外使用”)[ashburn和thor,2004年]。
[0010]
沙利度胺(thalidomide)是药物具有重新定位潜力的一个有趣例子,由于其致畸作用而被禁止用作为巴比妥类药物[vargesson,2015]。后来,沙利度胺被确定为治疗多发性骨髓瘤的有效药物[moehler,2012]。使用蛋白质化学计量学方法描述了药物再利用[dakshanamurthy et al.,2012]。然而,这是一种费力的方法,需要有关化学修饰的知识和专门用于这种方法的特殊计算机程序。医务人员在选择合适的药剂或药剂的某种组合进行脱靶/靶外(off-target)使用仍然面临严重困难。
[0011]
原则上,这需要个性化医疗。在选择特定的脱靶用途之前,必须对患者进行分析。
[0012]
目前,这通常是通过费力、昂贵和耗时的方式来完成的。过去,人们试图确定耐药性的分子基础,并先验地预测单个肿瘤是否会对标准药物治疗产生反应[volm and efferth,2015]。目的是根据事先预测的肿瘤个体药物敏感性特征调整临床治疗[walther and sklar,2011]。这个概念的任务是制定出个体化的有效治疗策略,这优于标准化肿瘤治疗的传统概念[schmidt and efferth,2016;efferth et al.,2017;mbaveng et al.,2017;hientz et al.,2017]。希望基于个体肿瘤基因组分子结构的新兴技术将有助于为市场产生新型抗癌药物。
[0013]
有人开发了一种基于计算机的方法,使用该方法发现一个单一植物来源的非药物物质冬凌草甲素(oridonin),对细胞有影响,并被怀疑与给定的细胞靶结构结合[kadioglu et al.,2018]。其目的是为冬凌草甲素在肿瘤细胞中的可用性提供进一步的证据。该方法模拟了这种特定物质与目标结构的结合。然而,这种基于计算机的方法不适合药物再利用(drug-repurposing),因为其既不使用一组选定的化合物,也不使用经批准的药物化合物。
[0014]
如上所述,个性化医疗面临着几个挑战。特别是药物再利用在技术上具有挑战性。通常,它需要相当复杂和费力的分析步骤。此外,在最后一步,医生必须仅根据科学上通常没有根据的经验做出选择。因此,仍然需要有改进的方法,用于鉴定与给定患病组织的靶结构特异性结合的一种或多种化合物。
[0015]
令人惊讶地,本发明发现,通过分子对接(molecular docketing)的方法,对一组化合物与患病组织中的突变基因的一个或多个对接空间的结合亲和力进行测量,就可以有效且容易地筛选与给定患病组织,特别是给定肿瘤组织,特异性结合的化合物。本发明涉及一种方法,该方法允许根据患者(例如癌症和其他遗传疾病患者)的个体突变和突变模式来预测药物的有效性。
[0016]
本发明第一个方面涉及一种用于鉴定一种或多种与给定患病组织的靶结构特异性结合的化合物的方法,所述方法包括:
[0017]
(i)鉴定所述患病组织的转录组(transcriptome)中的突变基因,并鉴定包含在所述突变基因中的至少一个突变;
[0018]
(ii)提供由对应于步骤(i)中鉴定的突变基因的野生型或同源基因表达的野生型或同源蛋白的三维(3d)结构;
[0019]
(iii)确定突变蛋白的3d结构,该突变蛋白是步骤(i)中鉴定的突变基因的表达产物或其一个或多个对接空间,包括:
[0020]
(a)使步骤(ii)的野生型或同源蛋白的3d结构的氨基酸序列适应(adapting)步骤
(i)中鉴定的突变基因的表达产物,并定义所述突变蛋白的3d结构的一个或多个对接空间,或
[0021]
(b)定义步骤(ii)的野生型或同源蛋白的3d结构的一个或多个对接空间,并使所述一个或多个对接空间的氨基酸序列适应步骤(i)中鉴定的突变基因的表达产物;
[0022]
(iv)提供所选的一组化合物的3d结构,并将每种化合物的每个3d结构与步骤(iii)的一个或多个对接空间拟合;
[0023]
(v)确定每种化合物对一个或多个对接空间的结合亲和力;和
[0024]
(vi)鉴定一种或多种与突变蛋白质特异性结合的化合物。
[0025]
本发明的方法涉及基于患者特异性突变谱将药物个体分配(individual allocation)在患病组织,特别是对于肿瘤疾病如癌症和其他遗传疾病。
[0026]
在一个优选的实施方案中,突变基因、突变蛋白或其组合与肿瘤相关。因此,在一个优选的实施方案中,突变基因与肿瘤的发生或进展有关。另外地或可选地,突变的蛋白质与肿瘤的发生或进展有关。在一个优选的实施方案中,突变基因和突变蛋白质与肿瘤(neoplasm),特别是炎症肿瘤(tumor),的发生或进展有关。
[0027]
在一个优选的实施方案中,本发明涉及一种方法,该方法是基于测定转录基因全基因组中的突变,以及鉴定针对每个个体中的特定突变起作用的药物。在一个优选的实施方案中,本发明侧重于用于其他疾病的已知药物,并将其重新用于个体化肿瘤治疗(也称为“药物重新定位”或“脱靶使用”)。
[0028]
优选地,本发明的方法是在个体体外进行的体外方法。在优选实施例中,本发明的方法是经计算机实施的方法。换句话说,该方法的一些或全部计算是以计算机辅助的方式进行的。可选地,计算机辅助方式可以包括在超级计算机上执行一个或多个程序步骤。适用的超级计算机的一个例子是mogon ii(德国美因茨)。
[0029]
在优选实施例中,步骤(ii)至(vi)中的一个、一些或全部,均部分地或完全地由计算机进行。
[0030]
在优选实施例中,步骤(ii)、(iii)、(iv)、(v)和/或(vi)中的至少一个以计算机辅助方式进行。在优选的实施方案中,选自步骤(ii)、(iii)、(iv)、(v)和/或(vi)的至少两个步骤以计算机辅助方式进行。在一个优选的实施方案中,至少步骤(ii)和(iii),至少步骤(ii)和(iv),至少步骤(ii)和(v),至少步骤(ii)和(vi),在至少步骤(iii)和(iv),至少步骤(iii)和(v),至少步骤(iii)和(vi),至少步骤(iv)和(v),至少步骤(iv)和(vi)或至少步骤(v)和(vi)是以计算机辅助方式进行。在优选实施例中,选自步骤(ii)、(iii)、(iv)、(v)和/或(vi)的至少三个步骤以计算机辅助方式进行。在一个优选的实施方案中,至少步骤(iii)、(iv)和(v),至少步骤(iii)、(iv)和(vi),至少步骤(ii)、(iv)和(v),至少步骤(ii)、(iv)和(vi),至少步骤(ii)、(iii)和(v),至少步骤(ii)、(iii)和(vi),至少步骤(ii))、(iii)和(iv),或至少步骤(ii)、(iii)和(vi)以计算机辅助方式进行。在优选实施例中,选自步骤(ii)、(iii)、(iv)、(v)和/或(vi)的至少四个步骤以计算机辅助方式进行。在一个优选的实施方案中,至少步骤(iii)-(vi),至少步骤(ii)、(iv)、(v)和(vi),至少步骤(ii)、(iii)、(v)和(vi)、至少步骤(ii)、(iii)、(iv)和(vi),至少步骤(ii)、(iii)、(iv)和(v)以计算机辅助方式进行。在优选实施例中,步骤(ii)-(vi)以计算机辅助方式进行。
[0031]
在高度优选的实施方案中,至少步骤(ii)-(v)以计算机辅助方式进行。
[0032]
如本文所用,术语“患病组织”可以从最广泛的意义上理解为具有患病特性的任何组织,例如过度生长、细胞外基质的不健康分泌或不健康的分泌物。在一个优选的实施方案中,这样的患病组织带有至少一个基因突变。在一个优选的实施方案中,患病组织由于其基因组中的至少一个突变而患病。这种患病组织也可以在最广泛的意义上被称为“遗传病”。这些遗传性疾病不一定是先天性疾病,也可能是由一种或多种出生后突变获得的疾病。在一个优选的实施方案中,患病组织的特征在于它带有一个或多个突变,特别是一个或多个与患病组织的疾病状态相关的突变,特别是与肿瘤相关的突变,因此,优选地,其与一种肿瘤发生或进展相关的突变。在一个优选的实施方案中,患病组织的特征在于它带有一个或多个与肿瘤相关的突变,因此,优选与肿瘤的发生或进展相关的突变。换言之,突变优选地是驱动突变(driver mutation)。与驱动突变相反的是被动突变(passenger mutation),其不会影响相关疾病的患病组织的状态。
[0033]
如在本发明中使用的,术语“与疾病状态相关”可以从最广泛的意义上理解为(潜在地)是组织变为患病组织的因素/原因。换言之,术语“与疾病状态相关”也可以优选地与“引起疾病状态”或“影响健康状态”互换理解。它可能是唯一的因素/原因,也可能是因素/原因之一。优选地,与疾病状态的关联意味着如果这些与疾病状态相关的因素不存在,组织就不会患病。
[0034]
在一个优选的实施方案中,患病组织被鉴定为遗传变异体,特别是与相应的健康组织相比,具有一个或多个突变、一个或多个(不同的)等位基因、一个或多个多态性(polymorphisms)、或其中两个或多个变异的组合。在一个优选的实施方案中,与相应的健康组织相比,患病组织被鉴定为一个或多个突变。在一个优选的实施方案中,与相应的健康组织相比,患病组织被鉴定为与患病组织的疾病状态相关的一个或多个突变,特别是与瘤形成的发生或进展相关的突变。这里,相应的健康组织可以是与患病组织具有相同组织类型来源的组织。相应的健康组织可以来自与患病组织相同物种的另一个个体的相同组织。
[0035]
患病组织可能遍布整个个体,也可能是位于局部病变组织。也就是说,可以是所有的体细胞都基本上带有某种突变,或者只有特定病变的细胞可能带有某种突变。在一个优选的实施方案中,病变组织是特定病变组织(lesion)。应当理解,患病组织通常源自寻求治疗的个体。因此,通常不是病原体,因此不是病毒,不是细菌,不是真菌,也不是致病性原生动物(理解为不能形成组织的单细胞生物)。
[0036]
如本文所用,术语“突变”可以从最广泛的意义上理解为核酸(即核糖核酸rna或脱氧核糖核酸dna)的核苷酸序列的任何改变。优选地,突变还导致由突变基因的翻译产生的蛋白质氨基酸序列的改变。优选地,突变与患病组织的疾病状态相关。换言之,突变优选地是驱动突变。优选地,突变与肿瘤相关。它可能与肿瘤的发生或进展有关。
[0037]
如本文所用,术语“突变基因”可以从最广泛的意义上理解为患病组织的细胞基因携带有核苷酸序列的永久性突变,特别是肿瘤细胞(即,形成一种肿瘤的细胞)。
[0038]
如本文所用,术语“转录组”可以从最广泛的意义上理解为患病细胞(特别是肿瘤细胞)中所有核糖核酸(rna)分子的集合,尤其是患病细胞(特别是肿瘤细胞)中的信使rna(mrna)分子。从其提供的信息可知,基因组的哪一部分,特别是外显子组,可以被转录成mrna。通常,转录组提供的信息还表明在患病细胞(特别是肿瘤细胞)中产生了哪些蛋白质,从而对蛋白质组(proteome)给出了提示。转录组也可能反映转录前和/或转录后剪接。或
者,mrna也可以是非编码rna和/或表观遗传改变的dna序列。
[0039]
如本文所用,“等位基因”可以理解为给定基因的变体形式。等位基因可能与患病组织的疾病状态相关或不相关。
[0040]
如本文所用,“多态性”可以被理解为在一个物种的种群中出现两种或更多种不同的遗传形式,任选地也导致不同的表型。多态性可能与患病组织的疾病状态相关或不相关。多态性也可以是与发生在基因组中特定位置的单个核苷酸的取代相关的单核苷酸多态性(snp)。优选地,这种snp变异在群体中的存在率大于1%。
[0041]
在一个优选的实施方案中,患病组织是肿瘤。
[0042]
如本文所用,术语“肿瘤”可以从最广泛的意义上理解为组织的任何异常和过度生长。赘生物可以是良性或恶性肿瘤。在一个优选的实施方案中,肿瘤是恶性肿瘤。在优选的实施方案中,赘生物是(癌性)肿瘤,换言之,该个体患有癌症。恶性肿瘤可以是原发性肿瘤、继发性或再次继发性肿瘤和/或可以是转移瘤。应当理解,个体还可以任选地携带不止一种相同类型和/或不同类型的肿瘤。
[0043]
如本文所用,术语“个体”可以从最广泛的意义上理解为可以携带患病组织,特别是肿瘤的任何动物或人类。在一个优选的实施方案中,个体是哺乳动物,包括人,例如人类、家养哺乳动物(例如狗、猫、马、骆驼、牛、绵羊、山羊、驴、等)或野生动物。在一个高度优选的实施方案中,个体是人。个体也可以被指定为“患者”或“受试者”。通常,个体具有至少一处损伤的患病组织,特别是至少一种赘生物。个体可能会或可能不会患有病变组织的病变,例如肿瘤。病变组织例如肿瘤也可以任选地不引起任何症状。
[0044]
在本发明中,术语“蛋白质”和“多肽”在最广泛的意义上可以互换理解为主要由通过酰胺键连续缀合的天然氨基酸组成的化合物。应当理解,本发明意义上的蛋白质可以进行或不进行一个或多个翻译后修饰和/或与一个或多个非氨基酸缀合。蛋白质的末端可以任选地通过本领域已知的任何方式加帽,例如酰胺化、乙酰化、甲基化、酰化。翻译后修饰是本领域众所周知的并且可以是但不限于脂化、磷酸化、硫酸化、糖基化、截短、氧化、还原、脱羧、乙酰化、酰胺化、脱酰胺、二硫键形成、氨基酸添加、辅因子添加(例如,生物素化、血红素添加、类二十烷酸添加、类固醇添加)和金属离子、非金属离子、肽或小分子的络合、以及硫化铁簇添加。此外,任选地,可以将辅助因子例如环磷酸胍(cgmp)、atp、adp、nad+、nadh
+h+
、nadp
+
、nadph
+h+
、金属离子、阴离子、脂质等结合到蛋白质上,不管这些辅助因素是否有生物学影响。
[0045]
步骤(i)中,突变基因和至少一个突变的鉴定,可以通过任何方式进行。鉴定所述患病组织特别是肿瘤的转录组中的突变基因的子步骤和鉴定包含在所述突变基因中的至少一种突变的子步骤可以任选地在单个步骤中同时进行。换言之,可以在基因中鉴定突变。该基因随后也被鉴定为突变基因。
[0046]
在一个优选的实施方案中,鉴定突变基因和至少一个突变的步骤(i)包括:
[0047]
(a)提供来自患病组织的含有mrna的样本;
[0048]
(b)任选地分离和/或纯化mrna;
[0049]
(c)任选地通过聚合酶链反应从mrna产生cdna;和
[0050]
(d)通过选自由以下组成的组中的至少一个步骤,鉴定至少一种突变:
[0051]-对mrna和/或cdna进行测序;
[0052]-将mrna和/或cdna与包含各种单链核苷酸的芯片杂交,其中包括突变和非突变序列;和
[0053]-用许多引物进行聚合酶链反应,包括那些针对特定突变的引物。
[0054]
作为编码mrna的替代方案,还可以使用非编码rna和表观遗传改变的dna序列,以及蛋白质、肽、脂质和所有其他代谢化学物质。
[0055]
可以通过任何方式进行从患病组织,特别是肿瘤,提供含有mrna的样品的步骤。通常,样品来自患病组织,特别是肿瘤。任选地,可以在移入rna稳定溶液后直接取出测试样品。它可以是储存的样品或先前从患病组织尤其是肿瘤解剖的样品。如上所述,优选地,本发明方法的所有步骤(i)-(vi)均在体外进行,即在个体体外进行。本领域技术人员熟知分离和纯化mrna的技术。互补dna(cdna)可以通过公知的方式从分离的和任选地纯化的mrna通过逆转录酶产生,任选地同时使用聚合酶链反应(pcr)的方法。本领域技术人员通常知道这些的方法。
[0056]
在一个优选的实施方案中,mrna是从总rna中分离和任选地纯化出来的。因此,在第一步中,可以进行总rna分离,例如,使用基于柱的提取程序,以获得纯rna,而无需进行dna消化。基因组dna可以通过特定的裂解步骤选择性地去除。这种方法适用于细胞、实体组织、血液和其他体液。总rna的质量和数量可以通过基于微流体的平台进行评估。
[0057]
加载后,样品可以通过微通道迁移以电泳分离样品成分。荧光探针可以插入到rna链中,进行荧光记录。分离、分馏poly a+rna,并合成双链cdna。如果随着时间的推移,有新的rna分离方法出现,它们可以用在整个实施方案中或取代目前的方法。这在下面的实验部分中进一步举例说明。可以测试rna的质量,并且设置阈值集,用于rna完整性的评分,例如,3或更高、4或更高、5或更高、6或更高,例如6.8或更高。在进一步分析前,为了除去核糖体rna片段,rna可以任选地与真核核糖体rna生物素标记的寡核苷酸探针杂交,以从总rna中去除核糖体rna。对于poly a+rna的制备,可以使用与oligo-dt偶联的链霉亲和素包被的磁珠。
[0058]
将几微克,例如1到10微克,如(大约)5微克的总rna与rna纯化珠粒混合并温育。温育几分钟后(例如,最多30分钟,如5-10分钟),可以将珠粒沉淀并弃去上清液。任选地,洗涤珠粒。可以将珠粒重新悬浮在洗脱缓冲液中以从其洗脱rna。然后,珠粒可以再次在结合缓冲中进行结合,并再次洗脱rna。可以在大约50至90℃或60至75℃下热处理数分钟,将rna片段化。洗脱和初始混合物可能包含具有随机序列的六聚体和逆转录酶,其可用于从rna模板开始cdna合成,上清液可转移到主混合物中并放入带有条形编码的pcr板。热循环完成后,可以将rna链移除,并用第二条cdna链取代。使用特定的珠粒,可以选择性地将双链cdna与rna和反应混合物分离。片段的悬垂链末端最后用3'-5'核酸外切酶消化成平末端。5'突出端可以被聚合酶填充至平末端。
[0059]
突变的蛋白质可以是本领域已知的任何突变。在一个优选的实施方案中,突变不是移码突变(frame-shift mutation.)。在一个优选的实施方案中,与相应的非突变(野生型)蛋白质相比,突变基本上保持了整个突变蛋白质的3d结构。在一个优选的实施方案中,突变蛋白质与非突变蛋白质的不同之处在于:
[0060]
(a)单个氨基酸残基(点突变)或两个、三个或更多个氨基酸残基;
[0061]
(b)1、2、3、4、5、多达10个、多达20个、多达50个、多达100个或甚至超过100个末端
氨基酸残基的截断;
[0062]
(c)1、2、3、4、5、多达10个、多达20个、多达50个、多达100个或甚至超过100个末端氨基酸残基的截断;
[0063]
(d)1、2、3、4、5、多达10个、多达20个、多达50个、多达100个或甚至超过100个末端氨基酸残基的延长;或
[0064]
(e)上述两种或更多种的组合。
[0065]
在一个优选的实施方案中,突变是点突变并且突变的蛋白质与非突变的蛋白质仅是单个氨基酸的不同,并且各自的对接空间包含该单个氨基酸。
[0066]
本领域技术人员知道许多用于鉴定突变,特别是点突变的手段和程序步骤。对于本发明,突变可以通过任何方式确定。例如,它可以通过rna测序进行。末端修复的、带a尾的和接头连接的cdna可以用pcr扩增,例如进行5到20个,如10个循环。可以使用商业rna测序系统以双端模式(2
×
100bp)对文库进行测序。任选地,所测得的序列可以与参考基因组比对,记录点突变、缺失、扩增、插入等的差异。可选地,标准化的rna表达可以使用rpkm测量进行量化。可以考虑转录本的rpkm值和转录本的比率来计算每个基因的总体rpkm值。这在下面的实验部分中进一步举例说明。
[0067]
在本发明方法的步骤(ii)中,鉴定与步骤(i)中发现的突变基因相对应的野生型或同源基因,即找到对应于突变基因的野生型或同源基因。在优选的实施方案中,鉴定对应于步骤(i)中发现的突变基因的野生型基因。野生型基因可以从最广泛的意义上理解为个体物种相应的健康,即未患病(如非肿瘤)组织中的典型基因。如本发明通篇所用,基因上下文中的术语“同源物”可以从最广泛的意义上理解为另一物种的相应基因,优选其相应的野生型基因。
[0068]
优选地,该物种与个体的相关相当密切。例如,当个体是人时,同源基因优选地来自另一哺乳动物的基因。优选地,如本文所用的同源物也是野生型基因或具有已知三维(3d)结构的突变基因。
[0069]
在本发明的一个可选实施例中,将患病组织与可比较的健康组织进行比较。可比较的健康组织优选地从同一个体(即患病组织的个体)获得,更优选地,所述病变组织是肿瘤组织,而可比较的健康组织是相同个体的相应非肿瘤组织。或者,可比较的健康组织可以从同一物种的另一个个体(即不是该患病组织的个体)获得,其中任选地且优选地,病变组织是赘生物(肿瘤组织)且可比较的健康组织是另一个个体的相应的非赘生性组织。患病组织与可比较的健康组织之间的比较可以优选地是目标结构的比较,其特征在于目标结构选自一个或多个个体突变、一个或多个(不同的)等位基因、一个或多个多态性,或它们其中的两个或更多个的组合,特别是其中每个目标结构可以与肿瘤组织例如炎症肿瘤相关。
[0070]
在一个优选的实施方案中,与相应的健康组织相比,患病组织具有一种或多种遗传变异,选自一种或多种突变、一种或多种(不同的)等位基因、一种或多种多态性,或其中两种或多种的组合。
[0071]
在一个优选的实施方案中,与相应的健康组织相比,患病组织带有一个或多个与患病组织的疾病状态相关的突变(也可称之为驱动突变)。
[0072]
患病组织与可比较的健康组织之间的比较可以是比较一种或多种化合物与一种或多种目标结构(靶结构)的特异性结合。优选地,患病组织与可比较的健康组织之间的比
较可以是,将一种或多种化合物与给定患病组织的一种或多种靶结构的特异性结合与所述一种或多种化合物与健康组织相应的一种或多种靶结构的结合进行比较。作为例子,突变基因可选自:
[0073][0074]
从步骤(i)中鉴定的突变基因,本领域技术人员可以容易且直接地推断出相应的蛋白质。这种蛋白质是基因的表达产物。许多野生型蛋白质的3d结构可从数据库中获得。作为对应于步骤(i)中鉴定的突变基因的野生型或同源基因的3d结构。3d结构可以是任何3d结构。
[0075]
在一个优选的实施方案中,步骤(ii)的野生型或同源蛋白的3d结构是晶体结构、3dnmr结构或计算出来的假定三维结构,可任选地从结构数据库中获得。
[0076]
通过比较从rna测序获得的突变谱与相应受影响蛋白质的蛋白质晶体结构,可以检查是否可以获得由发现突变的基因编码的三维蛋白质晶体结构。如果目标蛋白的同种型或剪接变体可用,则可选地并行建立几个同源模型。蛋白质的螺旋变化、二硫键二级环结构、β-折叠等的变形可能会改变蛋白质构象,因此可能会改变药物的结合特性。在选定的情况下,可以对来自不同物种的蛋白质进行序列比对,因为种间比较可以提供有关共同保守和独特的序列基序、药效团结构域中的关键氨基酸位置、螺旋弯曲残基的相同位置等感兴趣的信息。此外,可以任选地使用靶蛋白与其他结合蛋白、小分子、抗体、肽等的共结晶,因为它们不仅可以稳定被研究的蛋白质,而且可以将其构象从无活性状态变为活性状态,或者与其相反的转化。另外或可选地,可以计算静电势图(electrostatic potential maps)以确定电子密度热点,其可能干扰氨基酸残基的结合特性。这些信息可能有助于找到最合适的小分子抑制剂药物。
[0077]
本发明的方法可以在至少一台(高性能)计算机上运行,该计算机运行例如linux等操作系统,可以满足蛋白质建模的多阶段运行操作的要求。人类目标蛋白的晶体学的结构或来自其他物种的相应晶体结构可用于同源性建模。
[0078]
计算对接方法可用于预测配体(例如药物)与其受体(例如靶蛋白)的自由结合能(kcal/mol)和pki值(μm)。力场电位可用于计算给定结合构象下的自由结合能,并可估计配体和受体之间的构象空间。
[0079]
通过计算机检索数据库(例如,基于互联网或本地存储的数据库),可以寻找现有的蛋白三维(3d)结构,其可作为创建患者特异性突变的模型。如果某个物种(例如人类)没有可用的蛋白质晶体结构,在这种情况下,来自其他物种(同源物)的相应蛋白质结构可以作为模板来生成人类蛋白质同源模型。
[0080]
如前所述,如果不知道蛋白质的晶体结构,同源建模可以基于根据该蛋白质已知氨基酸序列创建的蛋白质的三维模型。
[0081]
同源建模的先决条件可能是相关蛋白质的晶体结构的存在。有了可用的晶体结构(例如野生型蛋白质),可用(野生型)蛋白质的序列与其突变对应物(其3d结构未知)的序列进行比对。根据野生型蛋白质的已知晶体结构,可以计算其相应突变蛋白质的假设3d结构。应当理解,已知和未知蛋白质的氨基酸序列越保守,所建的这种同源性模型就越好。作为第一步,可以从相应的网站(例如uniprot)以fasta格式下载蛋白质序列。然后下载相关蛋白质的已知3d结构,用作模板。最后使用blast(基本局部比对搜索工具)和clustalw2比对两个蛋白质序列。
[0082]
确定突变蛋白质的3d结构的步骤(iii)可以通过任何方式进行。优选地,是基于相应野生型蛋白质或同源物的3d结构,这种3d结构因氨基酸残基的改变而改变。
[0083]
在一个优选的实施方案中,这是通过针对具体突变建立的蛋白质同源性模型来进行,该模型模拟个体患病组织(患病组织损伤部位),特别是肿瘤中的突变基因。野生型蛋白质的3d结构或来自其他物种的同源3d结构均可用于同源建模。然后可以通过插入氨基酸变换来修饰野生型蛋白质的3d结构或同源模型,该氨基酸变换取决于特定患病组织,特别是肿瘤的rna测序。可以使用具有适当比对程序的比对文件来创建突变蛋白质的后续同源模型。然后可以使用swiss-model结构评估工具来选择最佳的分子对接同源模型。模型评估可以借助于多种工具(anolea、gromos、qmean、dfire等)。在细胞环境中,蛋白质通常以水合形式存在。因此,可以向asn和gln残基添加氢。这将在下面的实验部分中进一步举例说明。
[0084]
在步骤(iv)中,提供所选择的一组化合物的3d结构并且将每个化合物的每个3d结构与步骤(iii)的一个或多个对接空间拟合,这个步骤可以通过任何方式进行。该步骤也可称为“生物信息学筛选”或“虚拟药物筛选”。
[0085]
本领域技术人员可直接且明确地理解,术语“选择的一组化合物”通常可在最广泛的意义上与术语如“多种化合物”或“多种化合物”互换理解为一组多于一个的化合物,换句话说,不止一种类型的化合物。因此,化合物的选择通常包括至少两种(不同的)化合物。它也可以是化合物库。应当理解,在本发明方法的上下文中,化合物的选择不一定意味着物理存在的组合物,其中不同的化合物相互混合在一起。相反,选择的化合物的每个化合物的每个3d结构可以优选地单独地与步骤(iii)的一个或多个对接空间匹配/拟合(参见本发明的步骤(iv))。
[0086]
在一个优选的实施方案中,步骤(iv)中所选择的一组化合物包括至少5种化合物、至少10种化合物、至少25种化合物、至少50种化合物、至少100种化合物、至少250种化合物、至少500种化合物,或至少1000个化合物。根据步骤(iv),提供这些化合物中的每一种的3d
结构,并且每种化合物的每一种3d结构都与步骤(iii)的一个或多个对接空间分别进行匹配/拟合。
[0087]
在步骤(iv)中所选择的一组化合物可以具有任何分子量。在一个优选的实施方案中,步骤(iv)中的所述化合物中的至少一种其分子量不超过5000da、不超过2000da、不超过1000da或不超过的小分子超过750da。
[0088]
在步骤(iv)中所选择的一组化合物可能是改进的抗肿瘤剂或可能不是被批准使用的抗肿瘤剂。它可能有,也可能没有已知的药代动力学特性。在一个优选的实施方案中,在步骤(iv)中所选择的一组化合物不是经批准的抗肿瘤剂但具有已知的药代动力学特性。
[0089]
在一个优选的实施方案中,所述化合物被批准用于抗肿瘤活性之外的一种或多种药物用途。
[0090]
在优选的实施方案中,所述化合物是分子量不超过1000da或不超过2000da的小分子,并且被批准用于抗肿瘤活性以外的一种或多种药物用途。
[0091]
在一个优选的实施方案中,该方法是生物信息学筛选方法。优选地,测试(也称筛选)含若干化合物的文库。那么,该化合物也可以被指定为候选化合物。例如,一个包含几种化合物、几十种化合物、几百种化合物甚至超过1000种化合物(例如,fda-批准的药物)的库可用于研究药物与突变特异性蛋白质同源模型之间的结合,这可以通过特定的虚拟药物筛选程序进行。优选地,这些化合物包括未被批准作为抗肿瘤化合物(未被批准作为抗癌药物)的化合物。这个想法是基于药物通常不以单一特异性方式发挥作用,但具有更宽的活性谱。因此,针对其他特定疾病适应症的药物也可能抑制患病组织中的相关突变蛋白,特别是肿瘤。这些抑制性药物可以通过药物-蛋白质结合亲和力的生物信息学计算来识别。通过这种方法,可以使用已批准的药物,根据个体的突变,作为标签外的应用,用于治疗个体的患病组织,特别是肿瘤。
[0092]
重新利用fda批准的药物的优势是它们(已经证明)的生物活性和可接受的安全性/毒性特征。对于未经批准用于人类的化合物库,通常情况并非如此。
[0093]
确定每种化合物对一个或多个对接空间的结合亲和力的步骤(v)可以通过任何方式进行。有几种计算机算法可以独立地用来识别具有最佳结合的药物。在一个优选的例子中,可以选择具有最高亲和力的多种化合物中的前10个。
[0094]
可以使用所谓的“无偏”(unbiased)方法,其中程序从随机位置开始,探索蛋白质表面以实现配体的最佳结合。在第一次筛选中,可以使用程序计算柔性化学药物与刚性蛋白质表面的结合力(“刚性对接”)。当通过这种方法识别出感兴趣的药物时,可以应用对接程序来计算柔性药物结构与柔性蛋白质表面的对接(“柔性对接”)。
[0095]
同源建模的突变患者特异性蛋白质可以设置为刚性受体分子。计算的输出文件提供的信息是关于原子的部分变化、扭转自由度和不同原子类型的添加,例如脂肪族和芳香族碳或极性原子形成氢键,以pdqt格式输出。在目标蛋白质包含已知药效团位点的情况下,可以使用围绕该药效团的选定氨基酸残基的网格来计算药物结合(定义对接方法)。在这些情况下,如果目标蛋白的药物结合位点未知,则可以首先计算整个蛋白质的相互作用能量(盲对接方法)。然后可以使用显示具有最高结合亲和力的区域来设置网格,并作为第二步进行定义对接法。然后可以构造一个网格框来定义对接空间(docking space)。
[0096]
对于每个网格点,配体和受体之间的相互作用能量可以将在所有蛋白质原子上的
能量相加得到并将其保存。可以对配体中的每个原子进行单独的计算,得到它们的结合能,包括静电、氢结合能、扩散/排斥、去溶剂化和扭转熵作为关键参数。
[0097]
基于力场势(force field potential)的“亲和网格”可以考虑用于范德华力和静电相互作用以及“能量网格”,其中考虑配体的完整原子细节,而配体结合域则被简化。
[0098]
网格框的尺寸可以围绕整个蛋白质(盲对接方法)或围绕定义的药效团位点(定义的对接方法)设置,以使配体可以在对接空间中自由移动和旋转。网格框(grid box)可以由例如至少25个、50到10000之间、60到1000之间、70到500之间、80到300之间、90到200之间或100到150个分布在三个维度(x、y和z轴)中的网格点(grid points)组成,例如,使用126个网格点。这将在下面的实验部分中进一步举例说明。
[0099]
然后可以针对配体中存在的每种原子类型评估每个网格点处的能量,这些值用于预测特定配体构型的能量。可以进行三个独立的对接计算,进行至少100次、至少1000次、至少10,000次、至少100,000次、至少1,000,000次或至少10,000,000次的能量评估。可以进行三个独立的对接计算,进行至少2次、至少5次、至少10次、至少50次、至少100次或至少200次运行。可以采用拉马克(lamarckian)遗传算法。在优选的实施方案中,使用拉马克遗传算法确定每种化合物对一个或多个对接空间的结合亲和力。例如,可以使用拉马克遗传算法进行25,000,000次能量评估和250次运行。这在下面的实验部分中进一步举例说明。
[0100]
一次“运行”通常是单个对接过程,其由一个unix系统的指令启动并由单个对接参数文件控制。计算对接方法通常有高达2kcal/mol的标准偏差。因此,单次计算往往是不够的。在优选实施例中,执行至少三个具有25,000,000次能量评估和250次运行的独立对接活动以产生可靠稳定的结果。
[0101]
每个簇中相应的结合能和构象数量可以从对接计算日志文件(dlg)中获得。从中也可以获得相应的最低结合能(lbe),并且可以计算其平均值(可选地伴随标准偏差,
±
sd)。对接结果可以视化以证明药物与突变肿瘤蛋白的相关药物结合位点的正确结合。
[0102]
可以使用适当的软件程序将二维化学结构转换为三维化学结构。化合物的能量可以最小化并且新结构可以保存在计算机上(例如,作为摩尔文件,mol file)。对于随后的分子对接,配体文件可以以适合进一步处理的另一种格式准备(例如,以pdbqt格式、gpf、glg或dpf文件格式)。然后,可以准备运行对接的脚本。每个计算的最长运行时间可能是几小时到几天(例如,2小时到30天之间、5小时到20天之间、12小时到10天之间、1到9天之间、2到8天之间、3到7天之间,例如大约5天(=7200分钟)。可以使用脚本启动每次计算。可以保存运行作业的结果(例如,在配体的目录中)。完成作业后,结果可以选择性地复制到(个人)计算机。对于更多配体的对接活动,可以使用一个节点长的脚本(a node-long script)。
[0103]
通过上述程序鉴定的fda批准的与突变蛋白质结合的药物也可以与相应野生型蛋白质的晶体结构或同源模型对接。
[0104]
通常如果可获得已知配体及其受体的共结晶结构,可以使用共结晶构象作为对接模板,通过上述程序在新鉴定的配体与其受体之间进行对接。尽管进行了所有计算预测,对所有对接结果仍然进行了视觉检查,以排除明显的假阳性结果并提高已被fda批准的药物在癌症治疗中重新利用的成功率。
[0105]
此外,可以考虑到,已鉴定出与个体患病组织(尤其是肿瘤组织)基因组中发现的给定靶蛋白结合的药物可能不仅与该蛋白结合,而且还与多种其他蛋白结合。与非靶蛋白
的结合可能是正常组织中出现非特异性副作用的一个原因。为此,可以可选地使用基于网络服务器的药物靶标识别算法。
[0106]
使用这种策略,可以估计已识别的候选药物是否与相应的靶蛋白特异性结合。本技术所述的虚拟药物筛选程序可能主要基于刚性对接方法,即优选不考虑药物与其靶蛋白结合期间的构象变化。出于这个原因,也可以考虑将柔性对接技术包括在此筛选程序中(例如,分子动力学模拟)。在选定的情况下,可以通过实验验证通过这种虚拟筛选过程获得的结果。使用重组蛋白,可以通过适当的技术,如微尺度热分离、表面等离子体共振光谱、等温量热法等来研究有前景的候选药物的结合。
[0107]
在一个优选的实施方案中,确定每种化合物对一个或多个对接空间的结合亲和力的步骤(v)包括:
[0108]
(a)建立突变蛋白质和每个化合物的每个对接空间的3d网格框,其中每个网格框包含在所有三个维度中定义的网格点,这些网格点提供各种信息,如关于电荷、部分电荷、形成氢键的能力,形成π-π-电子相互作用的能力,以及形成范德华力的能力;
[0109]
(b)以化合物的3d结构可以在每个对接空间上旋转和移动的方式将化合物的每个3d结构与一个或多个对接空间拟合;
[0110]
(c)在每个网格点确定每个化合物和每个对接空间之间的结合能量,并计算每个化合物的每个3d构像与每个对接空间的结合亲和力;和
[0111]
(d)确定每种化合物-蛋白质相互作用的最低结合亲和力。
[0112]
在一个优选实施例中,该方法还包括以下步骤:
[0113]
定义步骤(ii)的野生型或同源蛋白的结构的一个或多个对接空间,每个对接空间对应于步骤(iii)的突变蛋白质的结构的相应对接空间;
[0114]
将化合物与这些一个或多个对接空间相匹配/拟合;
[0115]
确定每种化合物对这些一个或多个对接空间的最低结合能,从而确定其结合亲和力;
[0116]
比较每种化合物与突变和野生型或同源蛋白的对接空间的结合亲和力;和
[0117]
鉴定一种或多种对野生型或同源蛋白的对接空间比对突变蛋白的相应对接空间具有更高结合亲和力的化合物。
[0118]
感兴趣的突变蛋白质的一个或多个对接空间可以包含整个蛋白质结构或其一部分。
[0119]
在一个优选的实施方案中,对接空间包含整个蛋白质,整个蛋白质的表面任选地包括一个或多个潜在的结合袋或药效团结合位点的周围区域。
[0120]
在一个优选实施例中,该方法可以包括以下步骤:
[0121]-从患病组织,特别是来自患者的肿瘤、细胞或组织中分离rna
[0122]-通过rna测序确定突变谱(mutational profile)
[0123]-检查是否有现存的突变基因编码的三维蛋白质晶体结构。只是通过比较从rna测序获得的突变谱与相应受影响的患病组织,特别是肿瘤,的蛋白质的蛋白质晶体结构,-建立突变特异性蛋白质同源性模型,其类似于个体患病组织,特别是肿瘤中的突变基因。
[0124]-对所有fda批准的药物和其他物质进行生物信息学筛选,以发现与这些突变蛋白具有高亲和力的化合物。
[0125]-检查科学文献数据库,是否已有报道排名靠前的药物对癌细胞具有细胞毒性。
[0126]-主治医师决定可以选择哪种药物来治疗具有特定基因突变的个体患病组织,特别是肿瘤。
[0127]
通常,该技术程序适用于所有患病组织,特别是肿瘤的实体(例如炎症肿瘤实体,例如造血肿瘤、癌、肉瘤、转移瘤、腹水、胸膜积液等),以及其他疾病。
[0128]
然后,可以鉴定一种或多种与突变蛋白质特异性结合的化合物(步骤(vi))。任选地,可以设置一个或多个阈值水平以将候选化合物与不太适合的化合物区分开来。应当理解,这样的阈值水平应因人而异。
[0129]
如果合适,本领域技术人员应相应地选择阈值水平。通常,具有良好的可用性前景的化合物与感兴趣的突变蛋白质的(选择性)结合的亲和力最高。
[0130]
任选地,该方法可以包括一个或多个进一步的步骤,以进一步确保在步骤(vi)中鉴定的一种或多种候选化合物具有药用可用性前景。任选地,可以通过使用数据库和计算机算法,评估所确定的候选药物化合物的毒性特征以及它们与其他潜在联合用药的潜在相互作用。
[0131]
在一个优选的实施方案中,该方法还包括步骤(vii),其根据一个或多个数据库查找的信息确定步骤(vi)中鉴定的化合物的毒理学和药理学特性,并从中鉴别毒性低且(可选地)抗肿瘤活性高的化合物。
[0132]
该可选的步骤(vii)可以通过任何方式进行。例如,根据科学文献数据库的检索,确认排名靠前的药物是否有对癌细胞具有细胞毒性的报道。
[0133]
在许多情况下,对于获批准用于治疗非患病组织(特别是非肿瘤)疾病的药物,文献已有报道其也对肿瘤(例如,炎症肿瘤)细胞发挥细胞毒活性。这些公布的数据可作为佐证用本发明方法鉴定的化合物确实可能具有抗肿瘤作用。在该评估步骤中,常用的数据库如pubmed、scopus、scifinder,google scholar等,以及专业数据挖掘工具和软件可用来对已发表的文献进行高通量筛选。该步骤也可以以计算机辅助的方式进行。例如,筛选互联网或一个或多个本地存储的数据库以查找所要搜索的信息。
[0134]
在一个优选的实施方案中,本发明方法步骤(iv)选择的化合物组的化合物已经获得批准用于一种或多种治疗目的。在一个优选的实施方案中,本发明方法步骤(iv)选择的化合物组的化合物被批准用于一种或多种非抗肿瘤的治疗目的,但没有被批准作为抗肿瘤药。换句话说,本发明方法步骤(iv)选择的化合物组的化合物可以优选具有一种或多种上述特性。在一个优选的实施方案中,所述化合物是合成或半合成来源的。或者,化合物也可以是天然来源的。
[0135]
最后的决定是选择哪种化合物或哪些化合物的组合用于治疗。该步骤可以以计算机辅助的方式进行。为此目的,可对步骤(vi)中鉴定的化合物的所述作用和/或不良副作用进行进一步评估。
[0136]
在优选实施例中,所获得的信息然后可以用于自动化设备(如生物过程的信息模拟器)的决策,根据具体患者的临床、实验室和其他信息以及可用性、毒性特征、副作用和药物相互作用的风险等指标,用作为肿瘤学和其他领域个体精准医学候选药物的生成器。这里也可以设置阈值水平。应当理解,这样的阈值水平应因人而异。如果需要,本领域技术人员应相应地选择阈值水平。一般来说,一个化合物可用性的良好前景反映在其对感兴趣的
突变蛋白质的最高(选择性)结合亲和力,低的不良副作用(根据一个或多个数据库的检索)和以及任选地有抗肿瘤活性的报道(根据一个或多个数据库的检索)。
[0137]
本发明的方法还可提供信息,用于确定一种或多种化合物的个体化使用剂量。这里也可以设置阈值水平。应当理解,这样的阈值水平应因人而异。如果需要,本领域技术人员应相应地选择阈值水平。一般需要考虑良好的药物活性与相当低的毒性之间的平衡。该步骤也可以以计算机辅助的方式进行。
[0138]
如上所述,优选提供一种抗肿瘤剂或两种或更多种抗肿瘤剂的组合。在一个优选的实施方案中,本发明的方法是鉴定对肿瘤具有抗肿瘤活性的抗肿瘤药物的方法,其中所述抗肿瘤药物是(或包含在)步骤(vi)或(vii)中鉴定的一种或多种化合物。应当理解,本发明还提供新的和特别有益的抗肿瘤剂或其两种或更多种的组合,特别是用于个体化治疗患病组织(特别是肿瘤)的抗肿瘤剂或其两种或更多种的组合。应当理解,本发明还提供新的和特别有益的药物组合物。在一个优选的实施方案中,本发明的方法进一步包括制备药物组合物的步骤。该步骤包括将步骤(vi)或(vii)中鉴定的化合物与药学上可接受的载体进行组合。
[0139]
因此,本发明的另一方面涉及一种药物组合物,其包含步骤(vi)或(vii)中鉴定的一种或多种化合物以及药学上可接受的载体。
[0140]
如本文所用,术语“药学上可接受的载体”可指可支持抑制剂的药理学可接受性的任何物质。
[0141]
药物组合物可以制备用于任何类型的剂型,例如口服给药、鼻给药、通过注射给药(例如静脉内(iv)、动脉内(ia)、腹膜内(ip)、肌肉内(im)、皮下(sc)、鞘内和/或玻璃体内注射)、皮下给药、直肠给药和/或通过吸入给药。药物组合物可以以干燥形式(例如,作为粉末、片剂、丸剂、胶囊、可咀嚼胶囊等)或液体(例如,喷雾剂、糖浆、果汁、凝胶、液体、糊剂、注射液、气雾剂、灌肠剂等)
[0142]
药学上可接受的载体可以是无毒或低毒的溶剂,例如水性缓冲液、盐水、水、二甲亚砜(dmso)、乙醇、植物油、石蜡油或其组合。此外,药学上可接受的载体可包含一种或多种去污剂、一种或多种起泡剂(例如,十二烷基硫酸钠(sls)、二十二烷基硫酸钠(sds))、一种或多种着色剂(例如,二氧化钛、食用色素)、一种或多种维生素、一种或多种盐(例如钠盐、钾盐、钙盐、锌盐),一种或多种保湿剂(例如山梨糖醇、甘油)、甘露醇、丙二醇、聚葡萄糖)、一种或多种酶、一种或多种防腐剂(例如苯甲酸、对羟基苯甲酸甲酯)、一种或多种质地剂(例如羧甲基纤维素(cmc),聚乙二醇(peg)、山梨糖醇)、一种或多种乳化剂、一种或多种填充剂、一种或多种上光剂、一种或多种分离剂、一种或多种抗氧化剂)、一种或多种草药和植物提取物、一种或多种稳定剂、一种或多种聚合物(例如羟丙基甲基丙烯酰胺(hpma)、聚乙烯亚胺(pei)、羧甲基纤维素(cmc)、聚乙二醇(peg))、一种或多种摄取介质(例如聚乙烯亚胺(pei)、二甲基亚砜(dmso)、细胞穿透肽(cpp)、蛋白质转导结构域(ptd)、抗菌肽等),一种或多种抗体、一种或多种甜味剂(例如蔗糖、乙酰磺胺酸钾、糖精钠、甜菊糖)、一种或多种复染染料(例如荧光素、荧光素衍生物、cy染料、alexa氟染料、s染料、罗丹明、量子点等)、一种或多种味觉物质和/或一种或多种香料。
[0143]
如上所述,本发明步骤(vi)或(vii)中鉴定的化合物、本发明的抗肿瘤剂和本发明的药物组合物,可以特别好地用于个体化治疗病患组织,特别是肿瘤。
[0144]
本发明步另一方面涉及步骤(vi)或(vii)中鉴定的化合物、本发明的抗肿瘤剂或本发明的药物组合物在个体化治疗病患组织,特别是肿瘤中的用途。
[0145]
换言之,本发明涉及步骤(vi)或(vii)中鉴定的化合物、本发明的抗肿瘤剂或本发明的药物组合物,其用于治疗个体患有患病组织,特别是肿瘤,尤其是癌症。
[0146]
再换言之,本发明涉及一种治疗方法,用于治疗患有患病组织,特别是肿瘤,特别是癌症的个体,所述方法包括施用药学有效量的步骤(vi)或(vii)鉴定的任何一个化合物,本发明的抗肿瘤剂,或本发明的药物组合物。
[0147]
如本文所用,术语“抗肿瘤剂”、“抗癌剂”、“抗肿瘤药”、“抗癌药”、“抗癌化合物”、“抗肿瘤化合物”可以在最广泛的意义上被互换地理解为:适用于治疗恶性肿瘤(即癌症)的任何药剂。示例性地,此类抗肿瘤剂可选自化疗剂、激素及其类似物和其他抗肿瘤剂。
[0148]
示例性地,此类抗肿瘤剂可选自铂类(例如顺铂、卡铂、奥沙利铂)、抗代谢物(例如硫唑嘌呤、6-巯基嘌呤、巯基嘌呤、5-氟尿嘧啶、嘧啶、硫鸟嘌呤、氟达拉滨、氟尿嘧啶阿糖胞苷(阿糖胞苷)、培美曲塞、雷替曲塞、普拉曲沙、甲氨蝶呤)、其他烷化剂(例如苯丁酸氮芥、异环磷酰胺、甲氯乙胺、环磷酰胺)、他汀类药物(例如,西立伐他汀、辛伐他汀、洛伐他汀、氟伐他汀、索伐他汀、索伐他汀、索伐他汀、匹伐他汀、喷司他汀)、萜类和植物生物碱(例如,长春花生物碱(长春新碱、长春碱、长春瑞滨、长春地辛)、紫杉烷类(例如,紫杉醇)、胞嘧啶)、拓扑异构酶抑制剂(例如,喜树碱、依立替康、依立替康)磷酸盐、替尼泊苷)、马法兰、其他抗肿瘤药(如阿霉素(阿霉素)、阿霉素脂质体、表柔比星、博来霉素)、放线菌素d、氨基鲁米特、安吖啶、阿那曲唑、嘌呤和嘧啶碱的拮抗剂、蒽环类、芳香酶抑制剂、天冬酰胺酶、抗雌激素、贝沙罗汀、布舍瑞林、白消安、喜树碱衍生物、卡培他滨、卡莫司汀、克拉屈滨、阿糖胞苷、阿糖胞苷、阿糖胞苷、阿糖胞苷,多西紫杉醇,表柔比星,雌莫司汀,依托泊苷,依西美坦,氟达拉滨,氟尿嘧啶,叶酸拮抗剂,福美坦,吉西他滨,糖皮质激素,戈舍瑞林,激素和激素拮抗剂,hycamtin,羟基脲,伊达比星,伊立替康,来曲苯丙胺,来曲莫司汀丝裂霉素、有丝分裂抑制剂、米托蒽醌、尼莫司汀、丙卡巴肼、他莫昔芬、替莫唑胺、替尼泊苷、睾酮内酯、噻替哌、拓扑异构酶抑制剂、硫丹、维甲酸、曲普瑞林、三环磷酰胺、抑制细胞活性的抗生素、依维莫司、司克莫司霉素、西罗莫司、多吡胺,罗红霉素,子囊菌在,巴弗洛霉素,红霉素,麦地霉素,交沙霉素,刀豆霉素,克拉霉素,金兰霉素,叶霉素,妥布霉素,变霉素,更生霉素,更生霉素,瑞贝霉素,4-羟氧基环磷酰胺,苯达莫司汀,胸腺嘧啶-羟甲双氧嘧啶,5-氟霉素、阿地白介素、聚乙二醇天冬酰胺酶、头孢菌素、埃坡霉素a和b、硫唑嘌呤、霉酚酸酯、c-myc反义、b-myc反义、桦木酸、喜树碱、促黑素细胞激素(α-msh)、活化蛋白c、il-1β抑制剂,富马酸及其酯类、皮素、卡泊三醇、他拉糖醇、拉帕酚、β-拉帕酮、鬼臼毒素、桦木脑、鬼臼酸2-乙基酰肼、沙格司亭、(rhugm-csf)、聚乙二醇干扰素α-2b、来格司亭(r-hug-csf),非格司亭,聚乙二醇,头孢甘露碱,选择素(细胞因子拮抗剂),cetp抑制剂,钙粘蛋白,细胞分裂素抑制剂,农杆菌素、17-羟基农杆菌素、卵磷脂、4,7-氧基环茴香酸、杆菌素b1、b2、b3和b7、土贝母甙、鸦胆子醇a、b和c、鸦胆子苷c、矢车菊苷n和p、异脱氧叶黄素、托马托品a和b、冠蛋白a、b、c和d、熊果酸、cox抑制剂(例如,cox-2和/或cox-3抑制剂)、血管肽素、环丙沙星、荧光素、bfgf拮抗剂、普罗布考、前列腺素、1,11-二甲氧基菊素-6-one、1-羟基-11-甲氧基角蛋白-6-酮、东莨菪碱、秋水仙碱、no供体、季戊四醇四硝酸酯、辛多胺衍生物、星形孢菌素、β-雌二醇、α-雌二醇、雌三醇、雌酮、乙炔雌二醇、磷雌酚、甲羟孕酮、环戊丙酸雌二醇、苦参黄酮a、姜黄素、二
氢亚硝基丁烷、氯化亚硝胺、2-β-羟基孕二烯-3,20-双酮双糖、银杏醇、银杏酸、海兰素、茚苁素、茚-n-氧化物、紫果芸香碱,炔诺酮,糖苷1a,正义素(justicidin)a和b,拉瑞坦,马洛替林、马洛替铬醇、异丁基马洛替铬醇、马汉坦a,马可可碱,鹅掌楸,bisparthe诺丽定、牛心碱、马兜铃内酰胺-aii、雌二醇苯甲酸酯、曲尼司特、龟甲藻、维拉帕米、环孢菌素a、紫杉醇及其衍生物如6-α-羟基紫杉醇、浆果赤霉素、泰素帝、吗非泰酮、双氯芬他林、醋美他星、-苯氧乙酸、利多卡因、酮洛芬、甲芬那酸、吡罗昔康、美洛昔康、磷酸氯喹、青霉胺、羟氯喹、金诺芬、硫代苹果酸钠、奥塞洛尔、塞来昔布、β-谷甾醇、腺苷蛋氨酸、桃金娘、苯乙醇胺、多糖多辛,玫瑰树碱,d-24851(calbiochem),秋水仙碱,细胞松弛素ae,茚满碱,诺考达唑,杆菌肽,玻连蛋白受体拮抗剂,氮卓斯汀,游离核酸,核酸掺入病毒递质,dna和rna片段,纤溶酶原激活剂抑制剂-1,纤溶酶原激活剂抑制剂-2、反义寡核苷酸、vegf抑制剂、igf-1,来自抗生素组的活性剂,例如头孢羟氨苄、头孢唑啉、头孢克洛、头孢西丁、庆大霉素、青霉素、双氯西林、苯唑西林、磺胺类、甲硝唑、抗血栓药、阿加曲班、阿司匹林、阿昔洛班、恩昔单抗、合成抗凝胺gpiib/iiia血小板膜受体、xa因子抑制剂抗体、肝素、水蛭素、r-水蛭素、ppack、鱼精蛋白、尿激酶原、链激酶、华法林、尿激酶、血管扩张剂、双嘧啶、曲匹地尔、硝普钠、pdgf拮抗剂、三唑并嘧啶、、卡托普利、西拉普利、赖诺普利、依那普利、氯沙坦、硫蛋白酶抑制剂、前列环素、伐前列素、干扰素α、β和γ、组胺拮抗剂、5-羟色胺阻滞剂、凋亡抑制剂、凋亡调节剂、nf-kb、bcl-xl反义卤代寡核苷酸、卤代辛酮,生育酚,吗西多明,茶多酚,表儿茶素没食子酸酯,表没食子儿茶素没食子酸酯,乳香酸及其衍生物,lefluno米德、阿那白滞素、依那西普、柳氮磺吡啶、四环素、曲安西龙、普鲁卡因亚胺、视黄酸、奎尼丁、丙吡胺、氟卡尼、普罗帕酮、索他洛尔、胺碘酮、天然和合成获得的类固醇,如睡茄素a、苔藓茶素a、甘露醇二甙、苦杏仁甙,氢化可的松,倍他米松,地塞米松,非诺洛芬,布洛芬,吲哚美辛,萘普生,保泰松,阿昔洛韦,更昔洛韦,齐多夫定,抗真菌药,克霉唑,氟胞嘧啶,灰黄霉素,酮康唑、咪康唑、特比萘芬、氯喹、甲氟喹、奎宁、天然萜类化合物、海马素,巴灵通醇-c21-安吉酸,14-脱氢农杆菌素、农用地角蛋白、庚二酸a、齐奥林,马钱子碱,乌桑巴林,乌桑碱,瑞香素、落叶松树脂醇、甲氧基落叶松树脂醇、丁香树脂醇、伞形酮、afromoson,乙酰视素b,去乙酰视素a,vismione a和b,异碘曼,美藤叶酚、依芙桑素a、切除素a和b,龙胆素b,龙胆素c,卡梅鲍宁,白细胞介素a和b,13,18-脱氢-6-α-硒酰氧基查巴林,紫杉素a和b、雷根尼洛、雷公藤内酯、西马林、夹竹桃苷、马兜铃酸、胭脂虫红素、羟翅片甙、白头翁素、原白头翁素、小檗碱、氯化石蜡碱、马鞭草毒素、中华菊碱、康布瑞汀a和b,周叶甙a,加拉金苷,脱氧精胺,精神红素、蓖麻毒蛋白a、血根碱、曼乌小麦酸、甲基山梨糖醇、绿萝色酮、紫草素,赤影,二氢桑葚碱、羟基桑葚碱、马钱子五胺、及其药学上可接受的盐、或其两种或更多种的组合,或其两种或更多种药学上可接受的盐的组合。
[0149]
抗肿瘤剂也可以是适用于恶性肿瘤免疫治疗的药剂。适用于恶性肿瘤免疫治疗的药剂可以在最广泛的意义上理解为任何适合刺激免疫系统以治疗恶性肿瘤的药剂。它可以是主动的、被动的或两者的混合(二者兼有)。在这种情况下,免疫疗法可能基于与疾病组织(特别是肿瘤)相关的抗原(通常也称为肿瘤相关抗原(taa))的可检测性。主动免疫疗法可以通过靶向患病组织(特别是肿瘤)相关的抗原,引导免疫系统攻击患病细胞,特别是肿瘤细胞。被动免疫疗法可以增强现有的抗肿瘤反应,包括使用抗体或其片段或变体、免疫细胞(例如淋巴细胞,如t淋巴细胞、b淋巴细胞)、自然杀伤细胞、淋巴因子激活的杀伤细胞、细胞
因子激活的杀伤细胞、细胞毒性t细胞和树突细胞)和/或细胞因子,特别是(任选人源化的)单克隆抗体或其片段。根据个体体重,此类抗体或其片段或变体、免疫细胞和/或细胞因子可能导致抗体依赖性细胞介导的细胞毒性,可能激活补体系统,和/或可能阻止受体与其配体相互作用。因此,在某些个体中,目标细胞可能会被触发进入细胞凋亡。
[0150]
可用于免疫治疗的抗体的例子包括阿仑单抗、易普利姆玛、纳武单抗、奥法木单抗和利妥昔单抗。抗体或其片段或变体也可以任选地缀合(例如,通过放射性离子)。附加地或替代地,也可以使用树突细胞疗法(dendritic cell therapy)。此外或可选地,还可以使用细胞因子、锁孔血蓝蛋白、弗氏佐剂、卡介苗(bcg)疫苗和/或聚乙二醇干扰素α-2a。或者或另外,还可以使用抗肿瘤疫苗,例如由患病组织,特别是肿瘤的组织制成的疫苗或人工疫苗(例如,基于多肽的、基于多核苷酸的、基于糖苷的等)。本领域技术人员会知道在本发明上下文中可用的适用于恶性肿瘤的免疫治疗的其它药剂。
[0151]
如上所述,本发明方法中至少一些步骤优选地以计算机辅助的方式进行。应当理解,为此目的需要特殊的指令组合或算法(combination or algorithms)。因此,指令组合或算法产生特殊的技术效果。
[0152]
因此,本发明的另一方面涉及计算机指令程序,当该程序由计算机执行时,该程序的指令会使计算机至少完成本发明方法中的步骤(iv)和(v)。
[0153]
所述计算机程序可以存储在任何存储设备上,例如计算机硬盘、工作存储器、usb棒、cd rom等。因此,本发明还涉及一种存储设备,其上存储有包含指令的计算机程序(当该程序由计算机执行时)使计算机执行至少本发明方法的步骤(iv)和(v)。
[0154]
在优选实施例中,计算机程序包括指令,当程序由计算机执行时,该指令使计算机执行至少本发明的步骤(iii)-(v)、至少步骤(iii)-(vi),至少步骤(iv)-(vi)、至少步骤(ii)-(v)、至少步骤(ii)-(vi)或至少步骤(ii)和(iv)-(vi)
[0155]
以下实施例和权利要求旨在提供本发明的说明性实施方案。
实施例
[0156]
材料和方法
[0157]
1.从来自患者的肿瘤细胞或组织中分离rna
[0158]
在移入rna稳定溶液后直接取出测试样品。总rna分离是通过基于柱的提取程序进行的,以获得没有进行dna消化的纯rna。证实rna的质量,阈值设置为6.8或更高的rna完整性评分。为了有助于进一步分析,排除其中的核糖体rna序列,将rna与真核核糖体rna生物素标记的寡核苷酸探针杂交,以从总rna中去除核糖体rna。为了制备聚a+rna,使用了与oligo-dt偶联的链霉亲和素包被的磁珠。将5微克总rna与磁珠混合并温育。孵育5-10分钟后,将珠粒放在磁力架上,弃去上清液。用洗涤缓冲液洗涤珠粒后,将珠粒重新悬浮在洗脱缓冲液中以从珠粒上洗脱rna。然后,再次在结合缓冲液中进行结合/绑定过程。再次洗脱rna 珠粒合物,并通过在65℃下热处理5-10分钟使rna片段化。洗脱液和引物混合物包含具有随机序列和逆转录酶的六聚体,用于从rna模板开始cdna合成,将上清液转移到主混合物中,并放入带有条形码编码的pcr板中。当热循环完成时,rna链被移除并被第二条cdna链取代。使用特定的珠粒,从rna和反应混合物中分离出双链cdna。断裂的悬垂链末端被3'-5'核酸外切酶消化成平末端,且聚合酶将5'突出端填充到平末端。
[0159]
该提取方法适用于细胞、实体组织、血液和其他体液。通过基于微流体的平台评估总rna的质量和数量。加载后,样品通过微通道迁移以电泳分离样品成分。荧光探针插入rna链并记录荧光。该示例不仅涉及编码mrna,还涉及非编码rna和表观遗传改变的dna序列,以及蛋白质、肽、脂质和所有其他代谢化学物质。
[0160]
2.通过rna测序确定突变谱和转录本丰度
[0161]
末端修复的、a尾和接头连接的cdna用pcr扩增10个循环。使用商业rna测序系统以双端模式(2
×
100bp)对文库进行测序。所得序列与参考基因组比对。记录有关点突变、缺失、扩增、插入等差异。使用rpkm测量对标准化的rna表达进行量化。根据转录本的rpkm值和转录本的比率,计算每个基因的整体rpkm值。
[0162]
3.检索是否有现存的三维蛋白质晶体结构
[0163]
通过比较从rna测序获得的突变谱与相应蛋白质的蛋白质晶体结构,发现了几种基因编码的蛋白质发生了突变。如果目标蛋白有同种型或剪接变体可用,可以平行制备几个同源模型。螺旋型变化、二硫键二级环结构、β-折叠变形可能会改变蛋白质构象,因此可能会改变药物的结合特性。在某些情况下,对来自不同物种的蛋白质进行序列比对,因为种间比对可以提供有关共同保守序列部位和独特序列部位、药效团结构域中的关键氨基酸位置、螺旋弯曲残基的相同位置等有用的信息。此外,还考虑了靶蛋白与其他结合蛋白、小分子、抗体、肽等的共结晶,因为它们不仅可以稳定感兴趣的蛋白质,而且还可以将其构象从非活性状态变为活性状态,或从活性状态变为非活性状态。
[0164]
此外,计算静电势图以确定可能干扰氨基酸残基结合特性的电子密度热点。这些信息可能有助于找到最合适的小分子抑制剂药物。
[0165]
4.建立与个体肿瘤中突变基因相似的突变特异性蛋白质同源模型
[0166]
本文描述的方法在运行linux等操作系统的高性能计算机上进行,以满足蛋白质建模的多阶段计算进程的要求。对于一些计算,使用了超级计算机mogon ii(德国美因茨)。根据人类靶蛋白基于晶体学的结构或其他物种相应靶蛋白的晶体结构,进行同源性建模。首先检索基于互联网的蛋白质晶体结构数据库,寻找可用来作为模板创建患者特异性突变模型的蛋白质三维结构。在无法获得人类蛋白质晶体结构的情况下,来自其他物种的相应蛋白质结构可以作为模板来建立人类蛋白质同源模型。同源建模基于氨基酸序列已知但晶体结构未知的蛋白质的三维模型。同源建模的先决条件是有相关蛋白质的晶体结构。有了可用的晶体结构(例如野生型蛋白质),已知(野生型)蛋白质的序列可以与3d结构仍然未知的蛋白质(例如对应于野生型蛋白质的突变物)的序列进行比对.
[0167]
基于野生型蛋白质的已知晶体结构,可以计算出相应突变蛋白质的假设3d结构。已知和未知蛋白质的氨基酸序列越保守,所建立的同源性模型就更好。作为第一步,蛋白质序列以fasta格式从相应的网站(例如uniprot)下载。然后,下载作为模板的相关蛋白质的已知3d结构,并使用blast(基本局部比对搜索工具)和clustalw2比较两种蛋白质序列。然后通过插入从特定患者肿瘤的rna测序得到的氨基酸交换信息来修改野生型蛋白质的晶体结构或同源模型。然后,用适当的比对程序根据比对文件创建突变蛋白质的同源模型。
[0168]
然后使用swiss-model结构评估工具选择最佳的分子对接同源模型。模型评估是在多种工具(anolea、gromos、qmean、dfire等)的帮助下完成的。在细胞环境中,蛋白质以水合形式存在。因此,向asn和gln残基添加了氢。
[0169]
5.fda批准的药物库的生物信息学筛选,寻找其中与突变蛋白具有高亲和力的药物
[0170]
需要基于linux的高性能计算机集群在足够短的时间内运行虚拟药物筛选任务,以便为决策医生提供结果。基于fda批准的药物库(》1500种化合物),用虚拟药物筛选的方法研究药物与药物与突变特异性蛋白质同源性模型的结合。这些fda批准的药物不仅包含抗癌药物,还包含用于治疗各种疾病的药物。这个想法是基于药物通常不以单一特异性方式起作用,而是具有更广泛的活性谱。因此,针对特定疾病适应症的药物也可能抑制癌症中的相关突变蛋白。这些抑制性药物是通过药物-蛋白质结合亲和力的生物信息学计算来鉴定的。通过这种方法,已批准的药物可以在标签外使用,根据个体的突变来个体化治疗肿瘤。这是本发明现有药物再利用的主要概念。使用了几种相互独立的算法来识别具有最佳结合力的药物。例如,从超过1500种fda批准的药物中选择最高亲和力排名的前10种。同源建模的突变患者特异性蛋白质被设置为刚性受体分子。
[0171]
输出文件的信息包括原子的部分变化、扭转自由度和不同原子类型的添加,例如脂肪族和芳香族碳或形成氢键的极性原子,输出文件的格式例如为pdqt。在靶蛋白包含已知药效团位点的情况下,定义围绕该药效团的选定氨基酸残基的网格,用于计算药物结合(定义对接方法)。对于靶蛋白无已知药物结合位点的情况,首先计算整个蛋白质的相互作用能(盲对接方法)。然后将显示出最高结合亲和力的区域设置为网格,然后作为第二步进行定义对接。构建网格框以定义对接空间。
[0172]
网格框的设置可以围绕整个蛋白质(盲对接方法)或仅围绕药效团位点(定义对接方法),使得配体可以在对接空间中自由移动和旋转。网格框含有例如在所有三个维度(x、y和z轴)上的126个网格点,每个网格点之间的距离例如为1。然后针对配体中存在的每种原子类型估算每个网格点处的能量,这些能量值用于预测特定配体构型的能量。对接计算独立地重复三次,使用拉马克遗传算法进行了25,000,000次能量评估和250次运行。相应的结合能和每个簇中的构象数量可以从对接日志文件(dlg)中得到。从对接日志文件(dlg)中还可获得相应的最低结合能(lbe),并计算其平均值
±
sd。
[0173]
对接结果可视化以证明药物与突变肿瘤蛋白的相关药物结合位点的正确结合。通过使用数据库和计算机算法,对确定的候选药物的毒性特征以及与其他潜在联合用药的潜在相互作用进行了检查。为了证明所鉴定出的候选药物对给定突变靶蛋白的特异性,进行了该药物分别与突变型和野生型蛋白质模型的结合的试验。如果有更多模型(剪接变体、来自其他物种的蛋白质),它们也会被包含在对接程序中,以获得有关该药物与目标蛋白质结合的最佳信息。
[0174]
为了建立分子对接,首先将数据复制到配体对接程序的相应文件夹中。在此之前,使用适当的软件程序将二维化学结构转换为三维化学结构。化合物的能量被最小化,新结构保存为mol文件。对于随后的分子对接,配体文件以pdbqt格式输入,目标蛋白文件为gpf、glg和dpf文件格式。然后,准备运行对接的脚本。每个计算的最长运行时间为五天(=7200分钟)。每次计算都是使用脚本开启。运行作业的结果保存在配体的目录中。完成作业后,可以将结果复制到个人计算机。对于超过64个配体的对接作业,使用了一个节点长的脚本(node-long scrip)。
[0175]
此外,还考虑到一种已被鉴定为与患者肿瘤基因组中发现的给定靶蛋白结合的药
物,不仅可以与该蛋白质结合,还可能与其他几种蛋白质结合。与非靶蛋白的结合可能是正常组织中出现非特异性副作用的一个原因。为此,使用了基于网络服务器的药物靶标识别算法。使用这种策略,可以估计一个候选药物与相应的靶蛋白的结合是否是特异性结合。本技术所述的虚拟药物筛选程序主要基于刚性对接方法,即未考虑药物与其靶蛋白结合期间的构象变化。出于这个原因,也应考虑将柔性对接技术包含在这个筛选程序中(例如分子动力学模拟)。在某些情况下,通过这种虚拟筛选过程获得的结果得到了实验的验证。使用重组蛋白,通过适当的技术,如微尺度热分离、表面等离子体共振光谱、等温量热法等,研究了有希望的候选药物的结合。
[0176]
6.检索科学文献数据库,是否有关于排名靠前的药物对癌细胞具有细胞毒性的报道
[0177]
在许多情况下,文献中已经描述了获准用于癌症以外疾病的药物也对肿瘤细胞发挥细胞毒活性。这些公布的数据可以佐证,通过本发明的技术程序所确定的药物可能确实能够杀死癌细胞。利用常用数据库如pubmed、scopus、scifinder、google scholar等,以及专业数据挖掘工具和软件,可以对已经发表的文献进行高通量的筛选。
[0178]
7.主治医师决定选择哪种药物治疗具有特定基因突变的个体肿瘤
[0179]
根据本发明方法所获得的信息可以用作为主治医师、肿瘤委员会、其他决策者或自动化设备(如生物过程的信息模拟器)的决策依据。根据具体患者的临床、实验室和其他信息以及可用性、毒性特征、副作用和药物相互作用的风险等指标,用作为肿瘤学和其他领域个体精准医学候选药物的生成器。
[0180]
结果
[0181]
乳腺癌肝转移的活检材料来自一名50岁的患者。十多年来,患者接受了各种化疗,并显示出扩大的转移。当肿瘤标志物ca15.3升至22,230单位/毫升时,肿瘤呈进展性且不再对当前的化疗产生反应。pdl抗体疗法(keytruda,100毫克)没有改变对照组的肿瘤标志物。相关肿瘤委员会推荐了nap-paclitaxel,但几乎没有希望这会显着改变疾病的进程。
[0182]
为了获得进一步的治疗选择,进行了肝转移活检,并获得了如下所示的测试结果。根据测试结果,伊立替康(irinotecan)被确定为治疗候选药物,并根据标准方案每两周输注一次。之后,ca15.3下降到1513个单位,恶性腹水明显减少。半年后,患者病情稳定,临床健康。她去法国休了两周假,感觉很好。
[0183]
对具有多于20,000个mrna种类的完整转录组进行了测序。
[0184]
患者的rna测序显示总共有47,562个突变。
[0185]
建立了文献中描述为与癌症相关的2483种蛋白质的数据库。
[0186]
患者所呈现的611个rna突变可以导致该数据库收录的癌症相关蛋白的氨基酸的变化。
[0187]
从这2483种蛋白质中,排除了85种dna修复蛋白,因为无法从药理学上恢复突变的dna修复功能。
[0188]
从剩余的2398个蛋白质中,561个氨基酸突变在蛋白质中分布如下:
[0189]
253种蛋白质具有1个氨基酸突变;
[0190]
69种蛋白质,具有2个氨基酸突变;
[0191]
18种蛋白质,具有3个氨基酸突变;
[0192]
19种蛋白质,具有4个或更多氨基酸突变的。
[0193]
在受影响的359种蛋白质中,有12种有现存的三维晶体结构。随着人类蛋白质组越来越多的晶体结构被确定,可测试的蛋白质的数量会随着时间的推移而增加。这意味着识别有效再利用药物的能力会随着现存的三维蛋白质结构的增多而增加。
[0194]
使用这12种蛋白质的野生型序列,制备相应的突变蛋白质的三维同源模型。
[0195]
10种突变蛋白质各自携带一种氨基酸变化。另外两种蛋白质携带两个氨基酸突变:
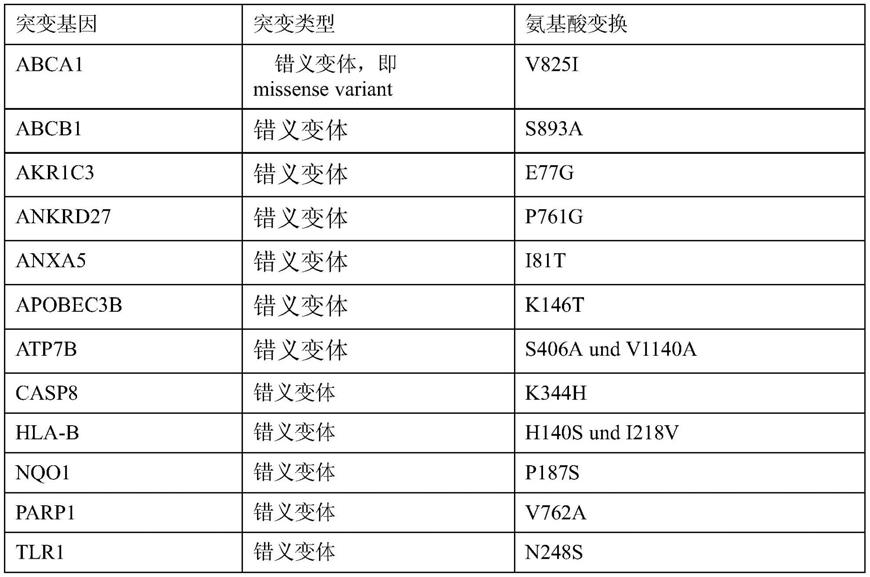
[0196][0197][0198]
所有12个同源模型均使用超过1500种fda批准的药物进行虚拟药物筛选。该筛选活动产生了12个药物排名列表。对所有12个药物排名列表中的前10个药物进行检查,以查找出现在多于一个列表的那些药物:
[0199][0200]
由于所有这些药物都以高亲和力与一种以上的突变蛋白结合,因此它们具有多特异性靶点特异性。可以预期,它们比仅结合一个单靶的单一特异性药物更具活性。
[0201]
我们也将这种方法应用于其他疾病(具有一种特定驱动突变的癌症、突变介导的遗传病和体细胞遗传病)。
[0202]
参考文献
[0203]
aronson jk,旧药-新用途。br.j clin.pharmacol.2007;64:563-565。
[0204]
ashburn tt,thor kb.药物重新定位:识别和开发现有药物的新用途。纳特。nat.rev.drug discov.2004;3:673-683。
[0205]
dakshanamurthy s,issa nt,assefnia s,seshasayee a,peters oj,madhavan s,uren a,brown ml,byers sw.使用蛋白质化学计量学方法预测已批准药物的新适应症。j med chem.2012;55(15):6832-6848。
[0206]
efferth t,saeed mem,mirghani e,alim a,yassin z,saeed e,khalid he,daak s.将植物化学物质和植物疗法整合到癌症精准医学中。oncotarget 2017;8:50284-50304。
[0207]
hientz k,mohr a,bhakta-guha d,efferth t.p53在癌症耐药性和靶向化疗中的作用。oncotarget 2017;8:8921-8946。
[0208]
kadioglu o,saeed m,kuete v,greten hj,efferth t.oridonin靶向多种耐药性肿瘤细胞,如硅片和体外分析所确定。frontier in pharmacology 2018;9:355.doi:10.3389/fphar.2018.00355。
[0209]
kola i,landis j.制药行业能否降低流失率?nat.rev.drug discov.2004;3:711-715。
[0210]
marin jjg,lozano e,herraez e,asensio m,di giacomo s,romero mr,briz o,serrano ma,efferth t,macias rir.胆管癌的化学抗性和化学增敏作用。biochim.biophys.acta 2018;1864:1444-1453。
[0211]
mbaveng at,kuete v,efferth t.中非、东非和西非用于癌症治疗的药用植物的潜力:聚焦抗性细胞和分子靶标。front.pharmacol.2017;8:343。
[0212]
moehler t.沙利度胺和来那度胺治疗多发性骨髓瘤的临床经验。curr.cancer drug targets 2012;12:372-390。
[0213]
mullard a.fda药物审批。nat.rev.drug discov.2012;11:91-94。
[0214]
schmidt f,efferth t.肿瘤异质性、单细胞测序和耐药性。pharmaceuticals(basel)2016;9.pii:e33。
[0215]
vargesson n.沙利度胺诱导的致畸:历史和机制。birth def.res.part c,embryo today:rev.2015;105:140-156。
[0216]
volm m,efferth t.癌症耐药性的预测和对个性化医学的影响。front.oncol.2015;5:282。
[0217]
walther z,sklar j.用于预测抗癌治疗反应的分子肿瘤分析。cancer j.2011;17:71-79。
[0218]
wo 2003/057173。
[0219]
wo 2001/035316。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1