一种结合多任务学习和自注意力机制的蛋白质功能预测方法与流程
1.本发明涉及蛋白质功能预测领域,更具体地,涉及一种结合多任务学习和自注意力机制的蛋白质功能预测方法。
背景技术:
2.蛋白质功能预测在生物学领域是一项的非常重要的任务,对于新药研发、病理学理解等方面发挥关键的作用。早期主要通过体内或体外实验对蛋白质进行功能注释,但其费时且昂贵的特点无法适应当前高通量测序技术的发展速度,因此基于计算的方法凭借其成本低、速度快的特点逐渐成为了重点的研究方向。
3.蛋白质功能可通过基因本体论(gene ontology)进行标注,其包含超过40000个功能项,可分为三大类别:分子功能(molecular function,mf)、生物学过程(biological process,bp)和细胞组件(cellular component,cc)。因此,蛋白质功能预测可以看作是大规模的多标签分类问题。
4.目前基于计算主要包括以下三种方法。第一种方法从蛋白质序列中提取特征并进行功能分类(如深度学习)。第二种方法使用blast,psi
‑
blast或diamond等软件对每一个查询蛋白从训练集中搜索相似序列,并基于序列相似性进行标注迁移。然而在uniprot数据库中仅有约1%的蛋白质序列进行了实验标注,因此对于许多未被标注的蛋白质可能难以找到与其具有高度序列相似性的已标注蛋白。前两种方法都利用了蛋白质序列作为模型的输入,但是,由于功能类别规模大及层次结构较为复杂,构建一个准确揭示蛋白序列和功能之间复杂关系的映射是一项艰巨的任务。因此,第三种方法应用了其他蛋白质元数据,例如蛋白质结构,蛋白质
‑
蛋白质相互作用网络,基因表达,遗传相互作用和生物医学文献等。实验证明,结合这些辅助信息能够进一步提高预测性能。然而,对于大量新发现的蛋白质会缺乏足够的辅助信息,只有它们的序列信息可供使用,因此基于序列的精确预测方法更需引起我们的关注。
5.以往的工作主要采用两种训练的方案,一种是根据三大类别划分标签并分别构造三个模型单独训练,另一种是将所有go类别结合成最终的标签并建立一个模型进行训练。考虑到go类别可分为三大分支(mf,bp和cc),一方面,三大分支从不同的角度描述蛋白质功能,即各个分支间的功能拥有不同的特性;另一方面,不同分支的功能之间也存在一定的语义关系(如is a,part of等)。因此,蛋白质功能预测也可视为三个不同任务的分类问题。多任务学习可通过硬参数共享、软参数共享、分级共享等方式提高模型的预测性能和泛化能力。但是,很少有工作集中在多任务学习方法上,以更准确地预测蛋白质功能。
6.在现有技术中,公开号为cn106126972a的中国发明专利,于2016年11月16日公开了一种用于蛋白质功能预测的层级多标签分类方法,包括以下步骤:一、训练阶段:在训练阶段针对类标签层级结构中的每个节点的数据集采用一个svm分类器进行训练,得到一组基础分类器;二、预测阶段:在预测阶段首先使用训练阶段获得的这组基础分类器得出未知样本的初步结果,而后采用带权重的tpr算法对结果进行处理,得到满足层级约束条件的最
终结果,实现对蛋白质功能的预测。虽然该方案能够在一定程度上解决现有分类方法用于预测蛋白质功能时存在的多标签问题,但是并未采用多任务学习方法,难以准确进行蛋白质的功能预测,因此,急需一种结合多任务学习和自注意力机制进行蛋白质功能预测方法。
技术实现要素:
7.本发明为解决现有技术中缺乏多任务学习方法的使用,难以准确进行蛋白质功能预测,提供了一种结合多任务学习和自注意力机制的蛋白质功能预测方法。
8.本发明的首要目的是为解决上述技术问题,本发明的技术方案如下:
9.一种结合多任务学习和自注意力机制的蛋白质功能预测方法,包括以下步骤:
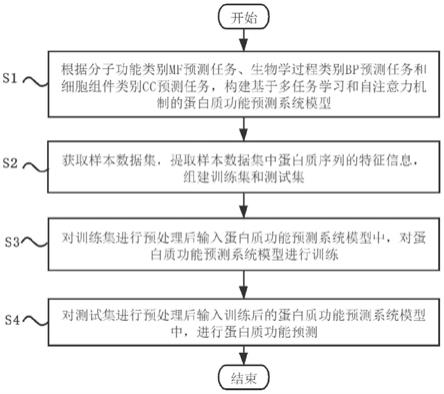
10.s1:根据分子功能类别mf预测任务、生物学过程类别bp预测任务和细胞组件类别cc预测任务,构建基于多任务学习和自注意力机制的蛋白质功能预测系统模型;
11.s2:获取样本数据集,提取样本数据集中蛋白质序列的特征信息,组建训练集和测试集;
12.s3:对训练集进行预处理后输入蛋白质功能预测系统模型中,对蛋白质功能预测系统模型进行训练;
13.s4:对测试集进行预处理后输入训练后的蛋白质功能预测系统模型中,进行蛋白质功能预测。
14.上述方案中,基因本体论(go)可分为三个主要类别(即mf、bp和cc),且每个类别都有其特性,因而可将它们视为三个不同的预测任务进行多任务学习;根据三个不同的预测任务,构建基于多任务学习和自注意力机制的蛋白质功能预测系统模型后进行训练,训练完成后用于进行蛋白质功能预测。
15.优选地,所述步骤s1具体为:
16.s101:根据分子功能类别mf预测任务构建基于自注意力机制的mf子网络;
17.s102:根据分子功能类别mf预测任务构建基于自注意力机制的bp子网络;
18.s103:根据分子功能类别mf预测任务构建基于自注意力机制的cc子网络;
19.s104:在mf子网络、bp子网络和cc子网络之间设置十字绣单元,实现子网络之间的连接和参数共享,以构建基于多任务学习和自注意力机制的蛋白质功能预测系统模型。
20.上述方案中,为每个预测任务构建具有相同结构的独立子网络(mf子网络、bp子网络和cc子网络),并通过十字绣单元对每个子网络的参数施加约束。
21.优选地,所述mf子网络、bp子网络和cc子网络均包括一维卷积层、残差卷积层、多头自注意力层、全连接层;其中:所述一维卷积层的输入作为所述蛋白质功能预测系统模型的输入;所述一维卷积层输出端与所述残差卷积层输入端连接;所述残差卷积层输出端与所述多头自注意力层输入端连接;所述多头自注意力层输出端与所述全连接层输入端连接;所述全连接层的输出作为所述蛋白质功能预测系统模型的输出;所述一维卷积层与所述残差卷积层之间、所述多头自注意力层与所述全连接层之间均设置有十字绣单元。
22.上述方案中,在每个子网络中,输入蛋白质的特征信息(l
×
84,其中l为最大蛋白质长度)后,由残差卷积层进行编码,以提取蛋白质抽象特征(得到大小为l
×
n
d
的特征,n
d
为每个卷积层中的卷积核数目),并将其作为多头自注意力层的输入,多头自注意力层中使用20个注意力头进行自注意力学习;经过前面的模块,模型得到大小为20
×
n
d
的输出,最后通
过全连接层来预测每种本体对应子网络的蛋白质功能的倾向。另外,在三个子网络之间使用十字绣单元来实现参数共享,在一维卷积层、多头自注意力层之后使用十字绣单元对子网参数施加约束。
23.优选地,所述残差卷积层包括若干一维残差卷积块,其中:每个所述一维残差卷积块之间均设置有十字绣单元;首个所述一维残差卷积块输入端与所述一维卷积层输出端连接;最后一个所述一维残差卷积块与所述多头自注意力层之间设置有十字绣单元,且所述一维残差卷积块输出端与所述多头自注意力层输入端连接。
24.上述方案中,在每个一维残差卷积块之后使用十字绣单元对子网络参数施加约束。
25.优选地,每个所述一维残差卷积块的结构均相同,包括第一卷积层、第一归一化层、第一激活层、第二卷积层、第二归一化层、第二激活层;其中:所述第一卷积层输入端接收所述一维残差卷积块的输入;所述第一卷积层输出端与所述第一归一化层输入端连接;所述第一归一化层输出端与所述激活层输入端连接;所述激活层输出端与所述第二卷积层输入端连接;所述第二卷积层输出端与第二归一化层输入端连接;所述第二激活层输入端接收所述一维残差卷积块的输入、第二归一化层的输出。
26.上述方案中,每个一维残差卷积块都具有相同的结构,均由两个卷积层,两个激活层和两个归一化层组成;在每个激活层前使用归一化层,以提高训练速度;所述一维残差卷积块可定义为:y=δ(f(x)+x)
27.其中x和y分别表示一维残差卷积块的输入和输出,f表示残差映射函数,δ表示激活函数。
28.优选地,所述多头自注意力层通过softmax函数进行归一化,使得蛋白质序列中各个位置的权重之和为1,获取y判断蛋白质序列中各个位置的重要性:y所占的比重越大,表示注意力权重越高;其具体表示为:
29.y=ax
30.a=softmax(w2tanh(w1x
t
))
31.其中:y代表多头自注意力层的输出;a代表注意力权重矩阵;x代表特征矩阵,作为多头自注意力层的输入;w1和w2代表权重矩阵。
32.上述方案中,自注意力机制在自然语言处理领域得到了广泛应用,使句子嵌入变得可视化和具有可解释性,此外自注意力机制可以计算出每个单词在整个句子中的重要性,并根据单词的注意力程度对单词特征进行加权求和,生成最终的句子嵌入。而具有相同功能的蛋白质往往包含相同的基序,具体地,基序可以与特定功能相关联,因此学习序列基序会有利于蛋白质功能的预测。根据这点,本方案利用多头自注意力层学习基序,对蛋白质残基特征的不同部分进行独立关注,以得到更好的蛋白质嵌入表示。
33.优选地,所述十字绣单元用于学习三个所述预测任务之间的权重,并根据权重进行计算,获取更优的特征图,其具体表示为:
[0034][0035]
其中,x
a
、x
b
和x
c
代表十字绣单元输入的特征图;和代表十字绣单元输出
的特征图;α代表十字绣单元;i代表特征图中的位置;α
aa
、α
bb
、α
cc
代表每个任务对自身的权重;α
ab
、α
ac
、α
ba
、α
bc
、α
ca
、α
cb
代表任务之间共享的权重。
[0036]
上述方案中,特征图的每个通道将编码不同的输入特征,为充分利用十字绣单元,对不同的通道应用独立的α参数,可自动学习任务之间的共享权重(该权重对于特定问题难以手动设置),从而得到更好的特征表示。结果表明,在多任务学习中,使用十字绣单元比不使用十字绣单元获得的预测性能更优。
[0037]
优选地,在所述步骤s2中,所提取的蛋白质序列的特征信息包括氨基酸序列seq、位置特异性打分矩阵pssm、隐藏马尔科夫模型的序列谱hmm、由spider3预测的蛋白质的结构信息spider3。
[0038]
上述方案中,蛋白质的序列的特征信息包括氨基酸序列、位置特异性打分矩阵、隐藏马尔科夫模型的序列谱、由spider3预测的蛋白质的结构信息,分别记作“seq”,“pssm”,“hmm”和“spider3”,其中:seq:为氨基酸序列的one
‑
hot编码,可表示为l
×
20的矩阵,其中l为序列长度;pssm:通过在uniref90数据库上运行psi
‑
blast进行3次迭代搜索,并为每个蛋白质生成pssm特征,可表示为l
×
20的矩阵;当无法通过psi
‑
blast生成特征时,可采用blosum矩阵进行替代;hmm:通过在uniclust30数据库(通过在序列一致性为30%对uniprotkb蛋白序列进行聚类而产生)上使用默认参数运行hhblits v3.0.0(一种用于序列搜索和比对的开源工具包)以生成隐藏马尔科夫模型的序列谱,可表示为l
×
30大小的矩阵;spider3:由spider3软件生成,包含蛋白质的结构信息,spider3软件的输入包括蛋白质序列以及通过psi
‑
blast和hhblits获得的pssm和hmm特征,spider3的输出可分为四部分:溶剂可及表面积(asa)、四个骨架夹角(即θ,τ,φ和ψ)的正弦值和余弦值、半球暴露(hse)和三个二级结构(即α
‑
螺旋,β
‑
折叠和无规卷曲)的预测概率,即通过spider3生成14个(1+8+2+3)结构性特征,可表示为l
×
14的矩阵。生成上述特征后,获得84(20+20+30+14)个序列性特征作为蛋白质功能预测系统模型的输入;考虑到数据集中的大多数蛋白质序列长度小于2000,通过零填充可形成长度恰好为2000的序列,最终得到大小为2000
×
84的矩阵,作为模型的输入特征。
[0039]
优选地,在所述步骤s3中,对蛋白质功能预测系统模型进行训练,所采用的损失函数为:
[0040][0041][0042]
p
i
=||(a
i
a
it
‑
i)||
f2
[0043]
其中,m代表任务数;n代表样本数;|go|代表所考虑的go类别数目;t
ijk
代表任务i的样本j中功能类别k的标签值;t
ijk
代表任务i的样本j中功能类别k的标签值;y
ijk
代表任务i的样本j中功能类别k的预测概率;e(t
i
,y
i
)代表二元交叉熵代价函数;a
i
代表任务i的注意力权重矩阵;i代表单位矩阵;‖
·
‖
f
代表矩阵的frobenius范数;p
i
代表任务i的多个注意力头的惩罚项;c代表惩罚系数。
[0044]
上述方案中,p
i
被添加到损失函数中,以增强多个注意力头之间的差异,以得到更好的蛋白质特征表示。
[0045]
优选地,在所述步骤s3、s4中,所述预处理为z
‑
score归一化处理。
[0046]
上述方案中,常用的归一化手段有min
‑
max、函数转换、z
‑
score等,本发明在预处理过程中采用z
‑
score归一化手段。
[0047]
与现有技术相比,本发明技术方案的有益效果是:
[0048]
本发明将三大本体论的预测视作三个预测任务,通过建立基于多任务学习和自注意力机制的蛋白质功能预测系统模型进行预测,提升了蛋白质功能预测的准确率。
附图说明
[0049]
图1为本发明方法流程图;
[0050]
图2为所述蛋白质功能预测系统模型结构示意图;
[0051]
图3为所述一维残差卷积块结构示意图;
[0052]
图4为实施例1中训练样本“p17121”在本发明方法(蛋白质序列输入)(a)和本发明方法(b)的模型中学习到的注意分数以及mast搜索到的对应基序(c)的示意图;
[0053]
图5为实施例1中测试样本“t96060014484”在本发明方法(蛋白质序列输入)(a)和本发明方法(b)的模型中学习到的注意分数以及mast搜索到的对应基序(c)的示意图。
具体实施方式
[0054]
为了能够更清楚地理解本发明的上述目的、特征和优点,下面结合附图和具体实施方式对本发明进行进一步的详细描述。需要说明的是,在不冲突的情况下,本申请的实施例及实施例中的特征可以相互组合。
[0055]
在下面的描述中阐述了很多具体细节以便于充分理解本发明,但是,本发明还可以采用其他不同于在此描述的其他方式来实施,因此,本发明的保护范围并不受下面公开的具体实施例的限制。
[0056]
实施例1
[0057]
如图1所示,一种结合多任务学习和自注意力机制的蛋白质功能预测方法,包括以下步骤:
[0058]
s1:根据分子功能类别mf预测任务、生物学过程类别bp预测任务和细胞组件类别cc预测任务,构建基于多任务学习和自注意力机制的蛋白质功能预测系统模型;
[0059]
s2:获取样本数据集,提取样本数据集中蛋白质序列的特征信息,组建训练集和测试集;
[0060]
s3:对训练集进行预处理后输入蛋白质功能预测系统模型中,对蛋白质功能预测系统模型进行训练;
[0061]
s4:对测试集进行预处理后输入训练后的蛋白质功能预测系统模型中,进行蛋白质功能预测。
[0062]
进一步地,所述步骤s1具体为:
[0063]
s101:根据分子功能类别mf预测任务构建基于自注意力机制的mf子网络;
[0064]
s102:根据分子功能类别mf预测任务构建基于自注意力机制的bp子网络;
[0065]
s103:根据分子功能类别mf预测任务构建基于自注意力机制的cc子网络;
[0066]
s104:在mf子网络、bp子网络和cc子网络之间设置十字绣单元,实现子网络之间的连接和参数共享,以构建基于多任务学习和自注意力机制的蛋白质功能预测系统模型。
[0067]
如图2所示,进一步地,所述mf子网络、bp子网络和cc子网络均包括一维卷积层、残差卷积层、多头自注意力层、全连接层;其中:所述一维卷积层的输入作为所述蛋白质功能预测系统模型的输入;所述一维卷积层输出端与所述残差卷积层输入端连接;所述残差卷积层输出端与所述多头自注意力层输入端连接;所述多头自注意力层输出端与所述全连接层输入端连接;所述全连接层的输出作为所述蛋白质功能预测系统模型的输出;所述一维卷积层与所述残差卷积层之间、所述多头自注意力层与所述全连接层之间均设置有十字绣单元。
[0068]
其中,多头自注意力层、全连接层之间进行了压平处理。
[0069]
进一步地,所述残差卷积层包括若干一维残差卷积块,其中:每个所述一维残差卷积块之间均设置有十字绣单元;首个所述一维残差卷积块输入端与所述一维卷积层输出端连接;最后一个所述一维残差卷积块与所述多头自注意力层之间设置有十字绣单元,且所述一维残差卷积块输出端与所述多头自注意力层输入端连接。
[0070]
进一步地,每个所述一维残差卷积块的结构均相同,包括第一卷积层、第一归一化层、第一激活层、第二卷积层、第二归一化层、第二激活层;其中:所述第一卷积层输入端接收所述一维残差卷积块的输入;所述第一卷积层输出端与所述第一归一化层输入端连接;所述第一归一化层输出端与所述激活层输入端连接;所述激活层输出端与所述第二卷积层输入端连接;所述第二卷积层输出端与第二归一化层输入端连接;所述第二激活层输入端接收所述一维残差卷积块的输入、第二归一化层的输出。
[0071]
进一步地,所述多头自注意力层通过softmax函数进行归一化,使得蛋白质序列中各个位置的权重之和为1,获取y判断蛋白质序列中各个位置的重要性:y所占的比重越大,表示注意力权重越高;其具体表示为:
[0072]
y=ax
[0073]
a=softmax(w2tanh(w1x
t
))
[0074]
其中:y代表多头自注意力层的输出;a代表注意力权重矩阵;x代表特征矩阵,作为多头自注意力层的输入;w1和w2代表权重矩阵。
[0075]
进一步地,所述十字绣单元用于学习三个所述预测任务之间的权重,并根据权重进行计算,获取更优的特征图,其具体表示为:
[0076][0077]
其中,x
a
、x
b
和x
c
代表十字绣单元输入的特征图;和代表十字绣单元输出的特征图;α代表十字绣单元;i代表特征图中的位置;α
aa
、α
bb
、α
cc
代表每个任务对自身的权重;α
ab
、α
ac
、α
ba
、α
bc
、α
ca
、α
cb
代表任务之间共享的权重。
[0078]
进一步地,在所述步骤s2中,所提取的蛋白质序列的特征信息包括氨基酸序列seq、位置特异性打分矩阵pssm、隐藏马尔科夫模型的序列谱hmm、由spider3预测的蛋白质的结构信息spider3。
[0079]
进一步地,在所述步骤s3中,对蛋白质功能预测系统模型进行训练,所采用的损失函数为:
[0080][0081][0082]
p
i
=||(a
i
a
it
‑
i)||
f2
[0083]
其中,m代表任务数;n代表样本数;|go|代表所考虑的go类别数目;t
ijk
代表任务i的样本j中功能类别k的标签值;t
ijk
代表任务i的样本j中功能类别k的标签值;y
ijk
代表任务i的样本j中功能类别k的预测概率;e(t
i
,y
i
)代表二元交叉熵代价函数;a
i
代表任务i的注意力权重矩阵;i代表单位矩阵;‖
·
‖
f
代表矩阵的frobenius范数;p
i
代表任务i的多个注意力头的惩罚项;c代表惩罚系数。
[0084]
进一步地,在所述步骤s3、s4中,所述预处理为z
‑
score归一化处理。
[0085]
在本实施例中,使用f
max
、s
min
和aupr作为评估模型性能的指标(f
max
和aupr越大、s
min
越小,表示模型获得更准确的预测结果)。为防止过拟合,随机选取样本数据集中的10%作为训练集,并通过早停法提前结束神经网络的训练,然后根据训练集的预测结果进行超参数调优。本发明中所述蛋白质功能预测系统模型基于深度学习框架pytorch实现,运行在ubuntu linux 16和nvidia gp102 gpu上。
[0086]
在本实施例中,从http://deepgoplus.bio2vec.net/data/下载两个样本数据集,分别为cafa3数据集和swissprot 2016数据集。由于更关注与训练集相似度较低的测试集的预测性能,所以对上述两个数据集使用diamond(e
‑
value为0.001)去除与训练集相似的样本。如表1所示,总结了去掉相似样本后cafa3数据集和swissprot 2016数据集中三种本体(mf、bp和cc)的训练集、测试集的大小和go项的数目。
[0087][0088]
表1
[0089]
(一)模型比较
[0090]
在本实施例中,采用三种对比模型验证本发明所构建的模型在蛋白质功能预测中的有效性。对比模型一:仅使用三个独立的子网络进行预测,未进行参数共享;对比模型二:使用硬参数共享机制,即对三个任务使用共享的卷积神经网络,每个任务都有独立的自注
意力模块和线性分类层;对比模型三:用均值池化替换自注意力模块,以探究自注意力机制是否能提高预测性能。
[0091]
如表2所示,在cafa3和swissprot 2016数据集上本发明方法和每种对比模型之间的预测结果进行了比较。可以看出,在这两个数据集上,本发明模型均取得了最好的平均f
max
、s
min
和aupr。结果显示,与不使用参数共享的网络相比,共享网络的使用带来了预测性能的提高(本发明模型和对比模型二)。而与使用硬参数共享策略相比,本发明模型在三个平均评价指标中取得了较大的提高,说明在多任务预测中利用十字绣单元实现软参数共享的有效性。同时,结果显示去掉自注意力模块会导致性能下降,因为使用自注意力机制的主要目的是学习序列的基序。综上所述,由于采用了自注意力机制和十字绣单元,本发明模型取得了最佳的预测性能。
[0092][0093]
表2
[0094]
(二)与其他方法的比较
[0095]
在本实施例中,还与其他基于序列的具有代表性的方法(包括naive、deepgo
‑
seq和deepgocnn)在两个数据集上进行对比。由于其他对比方法均将蛋白质序列作为模型输入,因此本实施例在本方法的基础上,以蛋白质序列作为输入,增加了一组对比。如表3所示,为各种方法在cafa3和swissprot 2016的测试集预测性能的对比。结果表明,在两个数据集中,以相同的特征作为模型的输入,本发明方法(蛋白质序列输入)在三个子本体上的平均评估指标上均优于naive、deepgo
‑
seq和deepgocnn。虽然本发明方法(蛋白质序列输入)的预测性能仅略优于deepgocnn,但其预测速度得到了显著提高。使用单个intel(r)xeon(r)e5
‑
2650 v4 cpu和nvidia gp102 gpu,deepgocnn每秒可以预测43个蛋白序列的功能,而本发明方法(蛋白质序列输入)每秒可以对81个蛋白序列进行功能注释,速度大约是deepgocnn的两倍,说明本发明方法是一种既精确又快速的蛋白质功能预测方法。
[0096]
值得注意的是,在使用所有特征信息(seq+pssm+hmm+spider3)时,本发明方法取得了十分显著的性能提高。在cafa3数据集中,本方法得到的平均f
max
=0.512,平均s
min
=13.649,平均aupr=0.480,显著优于deepgocnn(分别为0.469,13.984,0.432)和其他的对比方法;同样地,在swissprot 2016数据集上也取得了一致的结果。综上所述,本发明方法是一种更有效的蛋白质功能预测方法。
[0097][0098]
表3
[0099]
(三)自注意力机制
[0100]
由于具有相同功能的蛋白质序列往往包含相同的基序,所以学习基序有助于预测蛋白质的功能。因此,对自注意力分值和特定功能的序列基序以可视化的方式进行比较,以展现自注意力机制在蛋白质功能预测上的有效性。
[0101]
在本实施例中,使用mast对一组具有特定功能的蛋白质序列进行基序搜索,然后将mast搜索到的基序与本发明中模型获得的注意分数进行比较。以“酶活化剂活性”功能(go:0008047)为例。cafa3的测试集中有470个训练蛋白和5个测试蛋白具有该功能,将这475个蛋白序列输入于mast中进行基序搜索。如图4、图5所示,分别为训练样本“p17121”和测试样本“t96060014484”在本发明方法(蛋白质序列输入)(a)、本发明方法(b)模型中学习到的注意分数,以及mast搜索到的对应基序(c)。结果表明,两种模型在基序区域周围的注意分数相比其他区域明显偏高,说明本发明方法能够有效学习到序列基序。值得注意的是,本发明方法学习的基序区域比本发明方法(蛋白质序列输入)学习的基序区域位置更准确,且能获得更高的注意力分值,这说明使用的蛋白质特征有利于基序的学习,可以提高蛋白质功能预测的准确率。
[0102]
可见,本发明方法通过将对三大本体论(mf,bp,cc)的预测问题看作三个不同的任务,采用多任务学习的方法进行预测;并利用了三大本体论之间的区别和联系关系,使用十字绣单元实现参数共享以得到更好的嵌入表示,提高蛋白质功能预测准确率;采用自注意力机制学习序列的基序并进行可视化,增加神经网络的可解释性。
[0103]
本方法还可推广到其他与多标签分类相关的任务中,如基因功能预测。
[0104]
相同或相似的标号对应相同或相似的部件;
[0105]
附图中描述位置关系的用语仅用于示例性说明,不能理解为对本专利的限制;
[0106]
显然,本发明的上述实施例仅仅是为清楚地说明本发明所作的举例,而并非是对本发明的实施方式的限定。对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动。这里无需也无法对所有的实施方式予以穷举。凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明权利要求的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1