抗稻瘟病基因的分子标记及其应用
1.本发明属于基因检测领域,具体涉及一种抗稻瘟病基因的分子标记及其应用。
背景技术:
2.水稻(oryza sativa l.)是世界上最重要的粮食作物之一,世界上超过一半的人口以稻米为食。在中国,水稻种植约占全国耕地面积的25%,产量约占全国粮食总产量的50%,水稻是我国粮食安全和农业可持续发展的重要战略资源。稻瘟病是一种世界性的水稻病害,由稻瘟病菌(magnaporthe grisea)引起,稻瘟病既降低水稻的产量,同时也降低水稻的品质,稻瘟病严重危害水稻的产量和品质。
3.选育抗稻瘟病水稻品种是控制稻瘟病的有效途径。空育131和龙粳31是黑龙江水稻主栽品种,但是种植时间长了后,水稻的抗稻瘟病能力有所下降。为了提高空育131和龙粳31对稻瘟病的抗性,解决农业生产中对种质的需求,保证水稻的高产和稳产,需要对空育131和龙粳31进行品种改良以提高他们抗稻瘟病的能力。
技术实现要素:
4.我们通过杂交方法将抗稻瘟病基因导入到空育131和龙粳31,利用与水稻抗稻瘟病基因连锁的分子标记筛选含有pi-i、pi-b、pi-zt、pi-z、pi-kp、pi-km和pi-a等7个抗稻瘟病基因的个体,通过多代的杂交及回交,最终得到抗稻瘟病的空育131和龙粳31。上述研究的成果构成了本发明的基础。具体而言,本发明包括如下技术方案。
5.一种抗稻瘟病基因的分子标记,其为选自包含用于筛选抗稻瘟病基因pi-i、pi-b、pi-zt、pi-z、pi-kp、pi-km和pi-a的基因panel的snp(single nucleotide polymorphism,单核苷酸多态性)位点序列配对。具体而言,其为选自如下基因panel的snp位点序列配对:
6.判断无/有抗稻瘟病基因pi-i的snp序列seq id no:1/seq id no:2,本文中命名为“s0313_2k”;或者seq id no:3/seq id no:4,本文中命名为“s1879_4k”;
7.判断无/有抗稻瘟病基因pi-b的snp序列seq id no:5/seq id no:6,本文中命名为“s0380_300r”;或者seq id no:7/seq id no:8,本文中命名为“s1899-1k”;
8.判断无/有抗稻瘟病基因pi-zt的snp序列seq id no:9/seq id no:10,本文中命名为“pizt-shy-3”;或者seq id no:11/seq id no:12,本文中命名为“s0268_2”;
9.判断无/有抗稻瘟病基因pi-z的snp序列seq id no:13/seq id no:14,本文中命名为“piz-shy-1”;或者seq id no:15/seq id no:16,本文中命名为“piz-xy-2”;
10.判断无/有抗稻瘟病基因pi-kp的snp序列seq id no:17/seq id no:18,本文中命名为“pik-shy-4”;或者seq id no:19/seq id no:20,本文中命名为“s0712_6k”;
11.判断无/有抗稻瘟病基因pi-km的snp序列seq id no:21/seq id no:22,本文中命名为“pik-xy-2”;
12.判断无/有抗稻瘟病基因pi-a的snp序列seq id no:23/seq id no:24,本文中命名为“pia-shy-1”;或者seq id no:25/seq id no:26,本文中命名为“s0346_8k”。
13.当以水稻基因组dna为模板,进行pcr扩增所述分子标记时,使用的引物对分别为:
14.扩增snp序列seq id no:1/seq id no:2即“s0313_2k”的正/反向引物seq id no:27/seq id no:28(s0313_2k_f、s0313_2k_r);
15.扩增snp序列seq id no:3/seq id no:4即“s1879_4k”的正/反向引物seq id no:29/seq id no:30(s1879_4k_f、s1879_4k_r);
16.扩增snp序列seq id no:5/seq id no:6即“s0380_300r”的正/反向引物seq id no:31/seq id no:32(s0380_300r_f、s0380_300r_r);
17.扩增snp序列seq id no:7/seq id no:8即“s1899-1k_f”的正/反向引物seq id no:33/seq id no:34(s1899-1k_f、s1899-1k_r);
18.扩增snp序列seq id no:9/seq id no:10即“pizt-shy-3”的正/反向引物seq id no:35/seq id no:36(pizt-shy-3_f、pizt-shy-3_r);
19.扩增snp序列seq id no:11/seq id no:12即“s0268_2”的正/反向引物seq id no:37/seq id no:38(s0268_2_f、s0268_2_r);
20.扩增snp序列seq id no:13/seq id no:14即“piz-shy-1”的正/反向引物seq id no:39/seq id no:40(piz-shy-1、piz-shy-1_r);
21.扩增snp序列seq id no:15/seq id no:16即“piz-xy-2”的正/反向引物seq id no:41/seq id no:42(piz-xy-2_f、piz-xy-2_r);
22.扩增snp序列seq id no:17/seq id no:18即“pik-shy-4”的正/反向引物seq id no:43/seq id no:44(pik-shy-4_f、pik-shy-4_r);
23.扩增snp序列seq id no:19/seq id no:20即“s0712_6k”的正/反向引物seq id no:45/seq id no:46(s0712_6k_f、s0712_6k_r);
24.扩增snp序列seq id no:21/seq id no:22即“pik-xy-2”的正/反向引物seq id no:47/seq id no:48(pik-xy-2_f、pik-xy-2_r);
25.扩增snp序列seq id no:23/seq id no:24即“pia-shy-1”的正/反向引物seq id no:49/seq id no:50(pia-shy-1_f、pia-shy-1_r);
26.扩增snp序列seq id no:25/seq id no:26即“s0346_8k”的正/反向引物seq id no:51/seq id no:52(s0346_8k_f、s0346_8k_r)。
27.上述的分子标记可用于构建判断水稻是否存在抗稻瘟病基因的检测模型。
28.因此,本发明的第二个方面在于提供一种判断水稻是否存在抗稻瘟病基因的检测模型,其包括上述的分子标记的判断标准,即使用所述引物对水稻基因组dna进行pcr扩增后,若检测的某一个抗稻瘟病基因的snp位点序列号为奇数n(n=1-25),则缺少该抗稻瘟病基因;相反,若检测的某一个抗稻瘟病基因的snp位点序列号为偶数n+1(2-26),则含有该抗稻瘟病基因。具体而言,
29.seq id no:1或seq id no:3提示水稻不含抗稻瘟病基因pi-i;
30.seq id no:5或seq id no:7提示水稻不含抗稻瘟病基因pi-b;
31.seq id no:9或seq id no:11提示水稻不含抗稻瘟病基因pi-zt;
32.seq id no:13或seq id no:15提示水稻不含抗稻瘟病基因pi-z;
33.seq id no:17或seq id no:19提示水稻不含抗稻瘟病基因pi-kp;
34.seq id no:17或seq id no:21提示水稻不含抗稻瘟病基因pi-km;
35.seq id no:23或seq id no:25提示水稻不含抗稻瘟病基因pi-a;
36.seq id no:2或seq id no:4提示水稻含有抗稻瘟病基因pi-i;
37.seq id no:6或seq id no:8提示水稻含有抗稻瘟病基因pi-b;
38.seq id no:10或seq id no:12提示水稻含有抗稻瘟病基因pi-zt;
39.seq id no:14或seq id no:16提示水稻含有抗稻瘟病基因pi-z;
40.seq id no:18或seq id no:20提示水稻含有抗稻瘟病基因pi-kp;
41.seq id no:18或seq id no:22提示水稻含有抗稻瘟病基因pi-km;
42.seq id no:24或seq id no:26提示水稻含有抗稻瘟病基因pi-a。
43.上述的判断水稻是否存在抗稻瘟病基因的检测模型可以通过编程、数学软件包形式被输入计算机、基因检测装置比如abi 3730测序仪或illumina测序仪等、或者基因测序平台。
44.本发明的第三个方面在于提供一种用于检测抗稻瘟病基因的试剂盒,其包括用于检测上述snp位点序列的引物、尤其是seq id nos:27-52。
45.上述的试剂盒还可以包括用于确定判断水稻是否存在抗稻瘟病基因的标准对照表。
46.本发明的另一个方面在于提供上述的分子标记、检测模型、试剂盒在水稻改良中的用途。
47.例如,可以通过判断水稻是否存在某一种抗稻瘟病基因,指导将该抗稻瘟病基因通过基因工程导入缺少该抗稻瘟病基因的水稻中、或者通过与含有该抗稻瘟病基因的水稻品种进行杂交,形成转基因水稻或者杂交水稻。
48.本发明构建的抗稻瘟病基因的分子标记通过在水稻品种越光、空育131和龙粳31中的测试,证明能够准确定判断水稻是否存在抗稻瘟病基因pi-i、pi-b、pi-zt、pi-z、pi-kp、pi-km和pi-a,有效地指导水稻品种的改良,用于水稻抗稻瘟病育种,加快育种筛选速度,在育种中筛选含有抗稻瘟病基因的植株,最终育成含有稻瘟病抗性的水稻品种,对于提高水稻产量、保障粮食供应安全具有重要的经济意义。
具体实施方式
49.水稻品种越光含有pi-i、pi-b、pi-zt、pi-z、pi-kp、pi-km和pi-a等7个抗稻瘟病基因,而空育131和龙粳31缺乏这些基因。本发明基于水稻品种越光、空育131和龙粳31的全基因组测序,然后对基因组序列进行组装和序列比对分析,最终筛选得到13个与抗稻瘟病基因连锁的分子标记,建立了一种快速、准确检测抗稻瘟病基因pi-i、pi-b、pi-zt、pi-z、pi-kp、pi-km和pi-a的基因检测panel。
50.基因(检测)panel是高通量基因检测和基因测序发展起来后用的一个词语,它是指在检测中将若干基因对应的探针设计到同一张捕获芯片上以捕获目标dna并用于后续的基因测序。在检测中不只是检测一个位点、一个基因,而是同时检测多个位点、多个基因、多个位点,这些位点和基因需要按照一个标准进行选择和组合,需要一个检测panel。
51.本文中的基因panel又可称为基因集(gene set),是所有与抗稻瘟病检测相关的基因集合或基因组合。也可以包括针对抗稻瘟病基因或者是水稻基因组区域进行检测的一种探针捕获方案。
52.本发明的基因panel包括针对7个水稻抗稻瘟病基因pi-i、pi-b、pi-zt、pi-z、pi-kp、pi-km和pi-a构建的特异性snp序列集合,包含了13种分子标记。
53.snp是单核苷酸多态性(single nucleotide polymorphism)指由于单个核苷酸碱基的改变(包括单碱基的转换、颠换、以及单碱基的插入/缺失等)而导致的核酸序列的多态性。这是本技术领域所公知的。
54.利用该基因panel,本领域技术人员在抗稻瘟病水稻的育种中,可以方便地确定水稻杂交后代是否含有相应的抗稻瘟病基因pi-i、pi-b、pi-zt、pi-z、pi-kp、pi-km和pi-a,在苗期就可以鉴定水稻植株的稻瘟病抗性,从而提高水稻育种的效率,大大节约时间成本、人力成本和土地利用率。
55.本发明的抗稻瘟病基因的检测模型是以检测pi-i、pi-b、pi-zt、pi-z、pi-kp、pi-km和pi-a的分子标记即snp位点序列配对为变量建立起来的离散型数学模型。为描述方便起见,本文中也可将该模型简称为“抗稻瘟病基因模型”或者“分子标记模型”。
56.在一种实施方式中,当本发明抗稻瘟病基因的分子标记以试剂盒形式提供时,该试剂盒除了各种引物外,还可分别包括下述物品中的至少之一:携带工具,其空间划分为可以收容一种或多种容器、96孔板或板条的限定空间,该容器例如是试剂盒、药瓶、试管、和类似物,每样容器都含有一个单独的用于本发明方法的组分;说明书,其可以写在瓶子、试管和类似物上,或者写在一张单独的纸上,或者在容器的外部或内部,例如是带有操作演示视频app下载窗口比如二维码的纸件,说明书也可以是多媒体的形式,比如cd、u盘、网盘等。
57.本发明在筛选抗稻瘟病水稻植株方面、具有如下优势。第一,与传统的抗病育种中使用接种病原菌的方法鉴定水稻植株的抗性的方法相比,本发明通过分子标记方法筛选抗稻瘟病水稻植株的实验结果稳定性更高、可靠性更好。原因在于分子标记方法受环境因素干扰少,所以结果更可靠。第二,传统方法中接种实验周期长、成本高、操作过程复杂;而本发明的分子标记方法实验周期短,操作方便。第三,分子标记鉴定方法可在苗期取样、提取dna、通过pcr扩增和测序,找到抗病相应的snp,鉴定出抗稻瘟病的单株,淘汰其它植株,提高抗稻瘟病个体的筛选效率,可以满足高通量分子标记辅助育种的需求,实现抗稻瘟病基因的产业化分子育种。
58.如下以抗稻瘟病基因的分子标记的筛选和验证实施例说明本发明的技术效果。应理解,以下实施例仅用于说明本发明而非用于限定本发明的范围。
59.实施例中涉及到的百分含量,除特别说明外(比如明示为体积百分比率或比例),皆指质量百分含量。
60.实施例1:水稻抗稻瘟病基因的分子标记筛选
61.仪器:pcr仪、qubit荧光定量仪、abi 3730测序仪。
62.水稻基因供体品种:越光、空育131和龙粳31。
63.分子标记筛选包括以下步骤:
64.1)提取待测水稻植株的基因组dna;
65.2)使用选自下表1中的一对或多对引物,对水稻基因组dna进行pcr扩增;
66.pcr反应体系以20μl计为:10x pcr反应缓冲液2μ1,25mm mgso
4 0.8μl,2mm dntp2μ1,5μm正向引物f和反向引物r各1.2μ1,基因组dna 20ng,kod-plus聚合酶0.44μl,加ddh2o补足至20μ1。
67.pcr反应条件为:94℃预变性2分钟,94℃变性15秒,60℃退火30秒,68℃延伸1分钟,共35个循环;68℃保温5分钟。
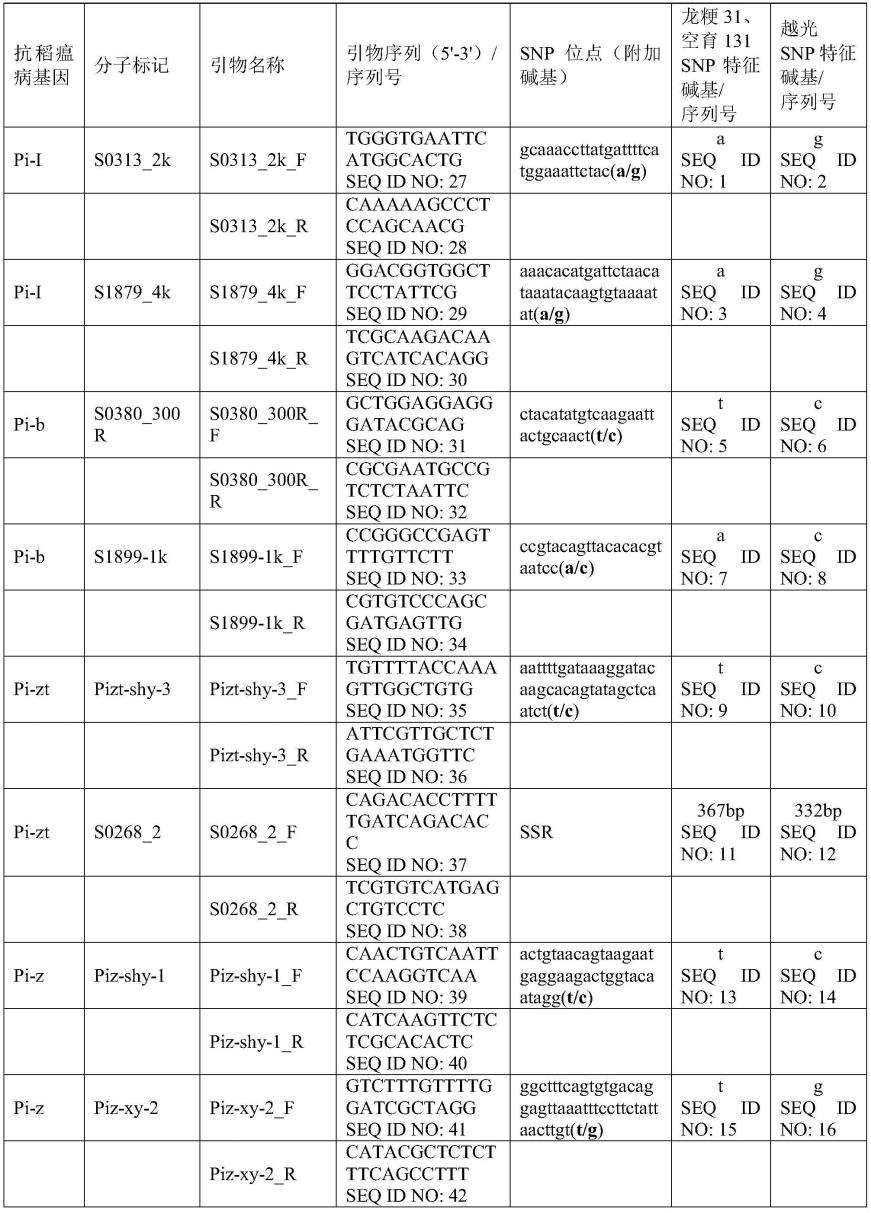
68.3)抗稻瘟病基因的snp分子标记。pcr产物进行纯化,然后进行测序反应,用abi 3730测序仪测序。单核苷酸多态性位点如果为越光的snp,则表明含有相应的抗稻瘟病基因。
69.例如,对于抗稻瘟病基因pi-zt的ssr分子标记(s0268_2)检测方法,s0268_2_f采用fam进行荧光标记,pcr扩增产物通过abi 3730测序仪检测特异性片段的大小,如果扩增出332bp特异性片段,则表明含有抗稻瘟病基因pi-zt。如果扩增出367bp特异性片段,则表明不含由抗稻瘟病基因pi-zt。也可以采用普通pcr引物,不加荧光标记,pcr产物通过琼脂糖凝胶电泳鉴定片段大小。
70.其中ssr是指简单重复序,也称微卫星dna,其串联重复的核心序列为1-6bp,其中最常见是双核苷酸重复,即(ca)n和(tg)n每个微卫星dna的核心序列结构相同,重复单位数目10-60个,其高度多态性主要来源于串联数目的不同。
71.三种水稻品种越光、空育131和龙粳31的基因测序结果参见表1。
72.表1、水稻抗稻瘟病基因的分子标记、引物对、及识别的snp位点
73.[0074][0075]
表1中,引物名称中的
“‑
f”代表正向;
“‑
r”代表反向。
[0076]
针对7个水稻抗稻瘟病基因pi-i、pi-b、pi-zt、pi-z、pi-kp、pi-km和pi-a,共得到了13种分子标记。
[0077]
实施例2:空育131的抗稻瘟病育种
[0078]
借助于全基因组测序分析,实施例1中建立的基因panel可以应用于水稻的抗稻瘟病育种,包括黑龙江主栽品种空育131、龙粳31的改良,将抗稻瘟病基因pi-i,pi-b,pi-zt,pi-z,pi-kp,km,pi-a导入空育131、龙粳31基因组中,提高空育131、龙粳31对稻瘟病的抗性。
[0079]
例如,含有抗稻瘟病基因的供体品种为抗稻瘟病越光品种,受体品种为空育131。我们的目标是将抗稻瘟病基因由越光品种导入空育131,增强空育131对稻瘟病的抗性,实现空育131的高产和稳产。
[0080]
1.含有抗稻瘟病基因pi-zt的空育131的育种
[0081]
首先,将含有抗稻瘟病基因pi-zt的越光品种与空育131进行杂交。然后,利用与水稻抗稻瘟病基因连锁的分子标记筛选含有pi-zt抗稻瘟病病基因的植株。其后,将此抗稻瘟病植株与空育131进行回交。回交的目的是将其背景回复为空育131。在每一代回交过程中,我们均挑选含有pi-zt抗稻瘟病病基因的植株与空育131进行回交。经过四到五代的回交后,植株背景基本已经回复到空育131。然后,将材料进行自交,再通过分子标记筛选含有pi-zt抗稻瘟病病基因的纯合个体,即为我们培育成功的含有抗稻瘟病基因pi-zt的空育131。材料经过繁种,可以用于田间种植。
[0082]
2.含有抗稻瘟病基因pi-i的空育131的育种
[0083]
空育131与含有抗稻瘟病基因pi-zt的越光品种进行杂交。其后,杂交、回交及自交
bt的纯合个体,纯合个体经过繁种,可以用于田间种植。
[0106]
实施例3:龙粳31的抗稻瘟病育种
[0107]
含有抗稻瘟病基因的供体品种为抗稻瘟病越光品种,受体品种为龙粳31。我们的目标是将抗稻瘟病基因由越光品种导入龙粳31,增强龙粳31对稻瘟病的抗性,实现水稻的高产和稳产。
[0108]
1.利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-i的龙粳31
[0109]
首先,将含有抗稻瘟病基因pi-i的越光品种与龙粳31进行杂交。然后,利用与水稻抗稻瘟病基因连锁的分子标记筛选含有pi-i抗稻瘟病病基因的植株。其后,将此抗稻瘟病植株与龙粳31进行回交。回交的目的是将其背景回复为龙粳31。在每一代回交过程中,我们均挑选含有pi-i抗稻瘟病病基因的植株与龙粳31进行回交。经过四到五代的回交后,植株背景基本已经回复到龙粳31。然后,将材料进行自交,再通过分子标记筛选含有pi-i抗稻瘟病病基因的纯合个体,即为我们培育成功的含有抗稻瘟病基因pi-i的龙粳31。材料经过繁种,可以用于田间种植。
[0110]
2.利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-b的龙粳31
[0111]
含有抗稻瘟病基因pi-b的越光品种与龙粳31进行杂交。其后,回交及自交的过程同“利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-i的龙粳31”,仅抗稻瘟病筛选的分子标记不同。
[0112]
3.利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-zt的龙粳31
[0113]
含有抗稻瘟病基因pi-zt的越光品种与龙粳31进行杂交。其后,回交及自交的过程同“利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-i的龙粳31”,仅抗稻瘟病筛选的分子标记不同。
[0114]
4.利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-z的龙粳31
[0115]
含有抗稻瘟病基因pi-z的越光品种与龙粳31进行杂交。其后,回交及自交的过程同“利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-i的龙粳31”,仅抗稻瘟病筛选的分子标记不同。
[0116]
5.利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-kp,km的龙粳31
[0117]
含有抗稻瘟病基因pi-kp,km的越光品种与龙粳31进行杂交。其后,回交及自交的过程同“利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-i的龙粳31”,仅抗稻瘟病筛选的分子标记不同。
[0118]
6.利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-a的龙粳31
[0119]
含有抗稻瘟病基因pi-a的越光品种与龙粳31进行杂交。其后,回交及自交的过程同“利用与水稻抗稻瘟病基因连锁的分子标记筛选的方法培育含有抗稻瘟病基因pi-i的龙粳31”,仅抗稻瘟病筛选的分子标记不同。
[0120]
以上所述仅为本发明的较佳实施例,并非对本发明任何形式上和实质上的限制,应当指出,对于本技术领域的普通技术人员,在不脱离本发明方法的前提下,还将可以做出若干改进和补充,这些改进和补充也应该视为本发明的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1