从单一样品预估肿瘤纯度的制作方法
从单一样品预估肿瘤纯度
相关申请的交叉引用
1.本技术要求2019年11月5日提交的第62/931,096号美国临时专利申请的优先权,该申请在此通过引用整体并入本文中,用于所有目的。
技术领域
2.本公开总体上涉及用于从单一样品预估肿瘤纯度的系统和方法。更具体地,但并非通过限制的方式,本公开涉及通过使用训练的机器学习模型处理b等位基因频率分布来预估生物样品的肿瘤纯度。
背景技术:
3.肿瘤细胞性,也称为“肿瘤纯度”,可鉴定样品中癌细胞的比例。生物样品中肿瘤纯度的准确估计可能有助于提高检测体细胞突变量和/或拷贝数变化的准确性。这是因为肿瘤纯度表明生物样品中存在的体细胞突变的等位基因频率。体细胞突变和拷贝数变异的检测又可用于确定对象的癌症阶段或评估特定癌症治疗是否有效。因此,肿瘤纯度可以告知确定癌症阶段和/或评估治疗效果。
4.虽然肿瘤纯度可以是一个有效的指标,但它也可能是几种生物信息学分析中的一个混杂变量。例如,预估肿瘤纯度的常规技术可能需要病理学家通过手动检查样本图像以预估肿瘤纯度来进行组织病理学评估。然而,包括手动检查样本图像在内的组织病理学评估可能是主观的和不准确的。用于预估肿瘤纯度的其他常规技术需要将源自给定肿瘤样品的核酸序列数据的值(例如,推定的体细胞变异)与源自匹配的正常对照样品的核酸测序数据的其他值进行比较。然而,这样的正常对照样品可能不可用。
5.例如,常规技术将样品的肿瘤纯度估计为个体肿瘤特有的体细胞突变的等位基因部分的函数。在没有匹配的正常样本的情况下,这些体细胞突变的鉴定不太精确,且预估纯度的准确性大大降低。在一些情况下,如果样本提供者没有收集或对正常对照(例如)测序,则匹配的正常对照不可用。
6.因此,需要准确预估样品中的肿瘤纯度以促进检测,而不依赖于主观分析(例如,组织病理学评估)或正常对照样品的存在。发明概述
7.在一些实施方案中,提供了预估肿瘤纯度的方法。方法可以包括获得代表对象肿瘤样品的多个核酸分子的核酸序列数据。方法还可以包括将核酸序列数据与参考基因组比对。方法还可以包括基于比对的核酸序列数据鉴定一组基因组区域。在一些情况下,该组基因组区域的每个基因组区域包括相对于参考基因组的相应基因组区域的一个或多个核苷酸序列变体。
8.方法还可以包括确定该组基因组区域的每个基因组区域的b等位基因频率。方法还可以包括基于该组基因组区域的b等位基因频率确定生物样品的b等位基因频率分布。方法还可以包括使用经过训练的机器学习模型处理b等位基因频率分布,以估计鉴定生物样
品的肿瘤纯度的度量。方法还可以包括输出度量。
9.在一些实施方案中,提供了系统,其包括一个或多个数据处理器和包含指令的非暂时性计算机可读存储介质,当在一个或多个数据处理器上执行这些指令时,使一个或多个数据处理器执行本文公开的一种或多种方法的一部分或全部。
10.在一些实施方案中,提供了计算机程序产品,其有形地体现在非暂时性机器可读存储介质中,并且包括配置为使一个或多个数据处理器执行本文公开的一种或多种方法的一部分或全部的指令。
11.本公开的一些实施方案包括系统,其包括一个或多个数据处理器。在一些实施方案中,系统包括包含指令的非暂时性计算机可读存储介质,当在一个或多个数据处理器上执行这些指令时,使一个或多个数据处理器执行本文公开的一种或多种方法的一部分或全部,和/或本文公开的一个或多个过程的一部分或全部。本公开的一些实施方案包括有形地体现在非暂时性机器可读存储介质中的计算机程序产品,包括配置为使一个或多个数据处理器执行本文公开的一种或多种方法的一部分或全部和/或本文公开的一个或多个过程的一部分或全部的指令。
12.已使用的术语和表达被用作描述性术语而不是限制性术语,并且在使用这些术语和表达时无意排除所示和描述的特征或其一部分的任何等同物,但应认识到,可能在要求保护的本发明的范围内进行各种修改。因此,应当理解,尽管要求保护的本发明已通过实施方案和任选特征具体公开,但是本领域技术人员可以对本文公开的概念进行修改和变化,并且这样的修改和变化被认为在由所附权利要求限定的本发明范围内。附图的简要说明
13.当参考以下附图阅读以下详细描述时,将更好地理解本公开的特征、实施方案和优点。
14.图1示出了根据一些实施方案的用于预估肿瘤纯度的全连接神经网络的示意图。
15.图2示出了根据一些实施方案的用于预估肿瘤纯度的一维卷积神经网络的示意图。
16.图3示出了根据一些实施方案的用于预估肿瘤纯度的二维卷积神经网络的示意图。
17.图4a显示了根据一些实施方案的用vcf级数据集训练的全连接神经网络、一维卷积神经网络和二维卷积神经网络的损失相对于epoch图。
18.图4b显示了根据一些实施方案的用vcf级数据集训练的全连接神经网络、一维卷积神经网络和二维卷积神经网络的均方根误差(rsme)和平均绝对误差(mae)数据。
19.图4c显示了根据一些实施方案的用于测试fastq级数据集的均方根误差(rsme)和平均绝对误差(mae)数据。
20.图5a显示了根据一些实施方案的将真实肿瘤纯度与通过训练的全连接神经网络预测的肿瘤纯度进行比较的图500a。
21.图5b显示了根据一些实施方案的将真实肿瘤纯度与通过训练的二维卷积神经网络预测的肿瘤纯度进行比较的图500b。
22.图5c显示了根据一些实施方案的将真实肿瘤纯度与通过一维卷积神经网络预测的肿瘤纯度进行比较的图500c。
23.图6示出了根据一些实施方案的用于使用经过训练的机器学习模型来估计肿瘤纯度的示意图的实例。
24.图7a至图7e提供了根据一些实施方案的绘制的baf分布的实例。
25.图8显示了根据一些实施方案的用于使用b等位基因频率特征对生物样品进行分类的前两个主要成分的图800。
26.图9包括的流程图示出了根据某些实施方案预估生物样品的肿瘤纯度的方法的实例。
27.图10示出了用于实施本文公开的一些实施方案的计算机系统的实例。发明详述i.概述
28.为了至少解决常规系统的上述缺陷,本技术可用于通过使用训练的机器学习模型处理测序数据中杂合位点的b等位基因频率分布来预估肿瘤纯度。经过训练的机器学习模型可以生成预估的度量,其可以鉴定生物样品中的肿瘤纯度,即使在没有正常对照样品的匹配测序数据的情况下也是如此。因此,本技术可以在没有匹配正常对照的情况下准确预估生物样品中的肿瘤纯度,这有助于更准确地分析来自仅肿瘤样品的核酸测序数据。
29.可以获得代表对象肿瘤样品的多个核酸分子的核酸序列数据。在一些实施方案中,核酸序列数据是全外显子组测序数据。核酸序列数据可以是全基因组测序数据。在一些实施方案中,测序数据来自肿瘤样品。肿瘤样品可以来自人类对象。核酸序列数据可以从鸟枪法测序产生。在一些实施方案中,可以通过对基因组或外显子组的选定部分进行测序来产生核酸序列数据。
30.核酸序列数据可以与参考基因组比对。基于比对的核酸序列数据,可以鉴定一组基因组区域。在一些情况下,该组基因组区域的每个基因组区域包括相对于参考基因组的相应基因组区域的一个或多个核苷酸序列变体。为了鉴定核苷酸序列变体,可以鉴定候选变体,并且可以计算候选变体的参考和替换读段深度。
31.可以确定该组基因组区域的每个基因组区域的ab等位基因频率。b等位基因频率是指两个等位基因(a和b)的等位基因强度比的标准化测量,使得baf为1或0表示两个等位基因之一(例如aa或bb)完全不存在,并且baf为0.5表示两个等位基因(例如ab)的存在相同。基于该组基因组区域的b等位基因频率,可以确定生物样品的b等位基因频率分布。在一些实施方案中,b等位基因频率分布是标准化的。
32.可以使用经过训练的机器学习模型处理b等位基因频率分布,以预估鉴定生物样品的肿瘤纯度的度量。如本文所用,肿瘤纯度或肿瘤细胞性是指肿瘤样品中癌细胞的比例。肿瘤纯度可以是各种技术用来预估与直接临床相关对应的肿瘤特征的度量。例如,预估的肿瘤纯度可能直接改变鉴定拷贝数改变所需的证据阈值,包括与批准的伴随诊断相关的那些(例如nsclc中的egfr外显子19缺失)。通过提供此类变体的预期等位基因部分的先验,肿瘤纯度估计还可用于提高体细胞变体调用的质量(敏感性,ppv)。例如,60%的肿瘤纯度值可能表明肿瘤样本包括60%的肿瘤细胞和40%的正常细胞。另外或可选地,肿瘤纯度可以指示肿瘤样品中癌细胞的数量。应注意,无论何时提及术语“肿瘤纯度”,术语“肿瘤细胞性”在本文自始至终可互换使用。
33.可以在计算机生成的数据集(例如,经由计算机模拟生成的核酸序列数据)上训练
经过训练的机器学习模型。在一些实施方案中,在训练数据集上训练经过训练的机器学习模型,该训练数据集从源自在正常细胞中稀释的肿瘤细胞的测序数据产生。经过训练的机器学习模型可以实现小于约0.2的平均绝对误差。在一些实施方案中,经过训练的机器学习模型实现了小于约0.2的均方根误差。
34.在一些实施方案中,经过训练的机器学习模型包括完全连接的神经网络。全连接网络可以包括具有整流线性单元(relu)激活函数的全连接层。在一些实施方案中,全连接神经网络的输出激活函数是sigmoid函数。全连接神经网络的损失函数可以配置为计算均方误差(mse)。在一些实施方案中,完全连接的神经网络通过使用随机采样经由超参数搜索来调整,其中具有层上的线性搜索、大小上的线性搜索、学习率上的对数搜索或以上的组合。
35.在一些情况下,经过训练的机器学习模型包括一维卷积神经网络。可以配置一维卷积神经网络,使得b等位基因频率分布可以用作输入,并且可以编码为高度为25、宽度为1且深度为100的输入大小。在一些实施方案中,一维卷积神经网络的每一层执行l x 1卷积,然后是relu激活函数。
36.在一些实施方案中,经过训练的机器学习模型包括二维卷积神经网络。在一些实施方案中,二维卷积神经网络的b等位基因频率分布被用作输入并且被编码为由高度为25、宽度为100且深度为1定义的输入大小。在一些实施方案中,二维卷积神经网络的每个卷积层之后是relu层。二维卷积神经网络的输出可以是具有sigmoid激活函数的密集连接层。另外,可以通过调整层数、滤波器大小、滤波器数量或以上的组合来调整二维卷积神经网络的超参数。
37.可以输出鉴定肿瘤纯度的估计度量。例如,可以输出包括估计度量的报告。在一些实施方案中,报告包括鉴定b等位基因频率分布的信息。报告还可以包括鉴定至少一种诊断标志物和/或至少一种预后标志物的信息。在一些实施方案中,报告包括鉴定预测的体细胞变体的信息。报告还可以包括治疗建议。例如,预估的肿瘤纯度可以表明o-6-甲基鸟嘌呤-dna甲基转移酶的甲基化水平增加,其为胶质母细胞瘤的预后生物标志物。在另一实例中,预估的肿瘤纯度可用于确定癌细胞的突变负荷量,其随后可用于确定某些类型的免疫疗法。取决于预估的肿瘤纯度的量,可以建议是否应该开始治疗胶质母细胞瘤。在一些实施方案中,治疗建议包括向人类对象给予治疗的建议。治疗建议可以包括不向人类对象给予治疗的建议。
38.因此,本公开的实施方案通过更准确地进行不需要依赖于源自匹配的正常对照样品的数据的肿瘤纯度的估计来提供优于常规系统的技术优势。预估的肿瘤纯度可用于提高来自肿瘤样本的测序数据分析和注释的准确性。可以生成一份或多份报告,说明估计的肿瘤纯度(例如,诊断和/或预后报告)。例如,肿瘤纯度的估计可用于提高诊断技术的准确性,以鉴定体细胞突变和/或拷贝数变化,并且可以生成具有预测的体细胞突变和/或拷贝数变化的详细信息的报告。
39.基于肿瘤纯度的估计和/或由肿瘤纯度的估计促成的报告,可以向患者给予一种或多种治疗或对患者不进行治疗。例如,可以将预测的体细胞变异与一个或多个已知癌症突变数据库进行比较,以诊断或表征癌症。可以鉴定与对某些癌症治疗的反应性或无反应性相关的变体,并且可以提供治疗建议。可以根据建议治疗癌症。
40.提供以下实例以介绍某些实施方案。在以下描述中,出于解释的目的,阐述了具体细节以提供对本公开实例的透彻理解。然而,显然可以在没有这些具体细节的情况下实践各种实例。例如,装置、系统、结构、组件、方法和其他组件可以显示为框图形式的组件,以免在不必要的细节中模糊实例。在其他情况下,可以在没有必要细节的情况下示出众所周知的装置、过程、系统、结构和技术,以避免混淆实例。附图和描述并非旨在为限制性的。本公开中已使用的术语和表达被用作描述性术语而不是限制性术语,并且在使用这些术语和表达时无意排除所示和描述的特征或其一部分的任何等同物。“实例”一词在本文中用于表示“作为实例、例子或说明”。本文作为“实例”描述的任何实施方案或设计不一定被解释为优选于或优于其他实施方案或设计。ii.用于从单一样品预估肿瘤纯度的机器学习模型a.用于预估生物样品的肿瘤纯度的示例性机器学习模型
41.如本文所讨论的,生物样品中的肿瘤纯度可以通过使用经过训练的机器学习模型来估计。经过训练的机器学习模型可以对应于经过训练以估计生物样品的肿瘤纯度的各种机器学习模型之一。在一些实施方案中,经过训练的机器学习模型包括多于一个模型(例如,1、2、3、4、5、6、7、8、9或10个机器学习模型)。例如,可以训练三种机器学习模型之一来估计核酸测序数据中的肿瘤纯度,包括全连接神经网络、一维卷积神经网络和二维卷积神经网络。在一些情况下,经过训练的机器学习模型包括深度神经网络。深度神经网络可用于捕获越来越大和高维数据集(例如核酸序列数据)的内部结构。深度神经网络可以鉴定高级特征,提高传统统计模型的性能,增加可解释性,并提供关于核酸序列数据结构的额外理解。
42.训练的机器学习模型可以包括超参数。超参数可以是模型外部的配置,且其值不是从数据(例如,训练数据、输入数据)估计的。在一些情况下,调整超参数,例如,调整以解决给定的预测建模问题。在一些情况下,超参数倍用于帮助估计模型参数。超参数可以由用户指定。在一些情况下,可以使用一组启发式算法来确定超参数。
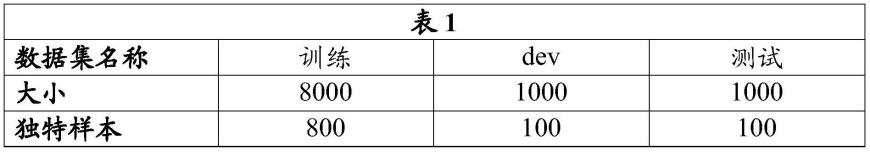
43.图1示出了根据一些实施方案的用于预估肿瘤纯度的全连接神经网络的示意图100。左侧显示了全连接神经网络的输入层105,然后是一组隐藏层110。右侧示出了输出层115。对于全连接网络,输入特征可以包括在生物样品中鉴定的核苷酸序列变体的全外显子组、b等位基因频率(baf)分布。全连接神经网络可能包括一系列具有relu激活函数的全连接层,并且输出激活函数可以是sigmoid函数。在一些情况下,全连接神经网络的损失函数被配置为生成均方误差(mse)。全连接神经网络可以经由使用随机采样的超参数搜索进行优化,包括对层和大小的线性搜索,以及对学习率的对数搜索。全连接神经网络的每个输出维度可以依赖于每个输入维度。在一些情况下,全连接神经网络可以是前馈神经网络。
44.可以训练卷积神经网络来预估生物样品的肿瘤纯度。卷积神经网络可以依赖于单元之间的局部连接和绑定权重,然后进行特征池(子采样)以获得平移不变描述符。基本的卷积神经网络架构可以包括一个卷积和池化层,任选地,后跟一个用于监督预测的全连接层。在一些情况下,卷积神经网络由多个(例如》10个)卷积和池化层组成,以更好地对输入空间进行建模。卷积神经网络可能需要大量数据集才能得到良好的训练。在一些实施方案中,卷积神经网络通过在输入空间的小区域上计算卷积并通过在区域之间共享参数来使用比完全连接的神经网络更少的参数。卷积神经网络可以是一维卷积神经网络。卷积神经网
络可以是二维卷积神经网络。在一些实施方案中,卷积神经网络包括三个或更多个维度。
45.图2示出了根据一些实施方案的用于预估肿瘤纯度的一维卷积神经网络的示意图200。对于一维卷积神经网络,输入特征可能包括染色体baf分布,可以将其编码为定义高度为25、宽度为1,且深度为100的输入大小。一维卷积神经网络的每一层都可以执行l x 1卷积,然后是一个relu激活函数。在一些情况下,使用“网络中的网络”深层网络结构。有效地,使用了在扁平化输入上每层深度跨度的一维卷积。在一些情况下,对应于深度和层大小的超参数是从完全连接的神经网络中获得的(参见图1),此时可以调整与一维卷积神经网络的学习率对应的超参数以进行优化。
46.图3示出了根据一些实施方案的用于预估肿瘤纯度的二维卷积神经网络的示意图300。对于二维卷积神经网络,输入特征可能包括染色体baf分布,可以将其编码为定义高度为25、宽度为100,且深度为1的输入大小。每层可能对应一个二维卷积层(二维卷积神经网络层),后跟一个relu激活函数。输出层可能对应于具有sigmoid激活函数的密集连接层。在一些实施方案中,用于二维卷积神经网络的超参数通过搜索层数、滤波器大小和滤波器数来优化。可以通过执行dropout正则化和/或l2正则化来减少训练集上的初始过拟合。
47.可以训练其他类型的机器学习模型来估计生物样品的肿瘤纯度。在一些实施方案中,机器学习模型对应于梯度提升决策树(例如,xgboost框架、lightgbm框架)、装袋程序、提升程序和/或随机森林算法中的一种或多种。例如,可以训练梯度提升决策树来预估生物样品的肿瘤纯度。梯度提升对应于一类机器学习技术,其可用于回归和分类问题,并用于生成可能包括弱预测模型集合的预测模型,例如决策树。在一些情况下,梯度提升决策树可以包括例如xgboost框架或lightgbm框架。b.用于训练预估生物样品的肿瘤纯度的机器学习模型的训练数据集
48.用于预估生物样品的肿瘤纯度的机器学习模型可以使用监督训练算法进行训练。监督训练算法可用于训练机器学习模型,以生成对应于肿瘤纯度的线性和/或逻辑回归值的输出。机器学习模型可以基于训练数据集进行训练。在一些情况下,训练数据集包括来自纯肿瘤样本、纯正常样本和正常细胞中的肿瘤细胞稀释系列的测序数据集。测序数据集可以来源于其他对象的生物样品,包括来自被诊断患有癌症的对象的生物样品。训练数据集的生物样本可以对应于恶性组织、良性组织或它们的混合物。在一些实施方案中,在没有匹配的正常样品的情况下获得包括癌组织的生物样品。另外或可选地,获得匹配的正常样本用于机器学习模型的训练和测试(例如)。
49.在一些实施方案中,训练数据集包括训练核苷酸序列变体数据集。训练变体数据集可以对应于核酸序列数据,其中鉴定了核苷酸序列变体。核苷酸序列变异可能包括单核苷酸多态性(snp)、一个或多个单核苷酸变异、插入-缺失突变(indel)、小插入、小缺失、结构变异连接、可变长度串联重复、侧翼序列和以上的组合。在一些情况下,使用从训练样本(包括肿瘤和正常样本)生成的vcf文件的人工组合来生成有偏差的“变体调用格式(vcf)级别”数据集。因此,训练数据集可以包括肿瘤序列的计算机“稀释”。可以使用表1中列出的特征生成vcf级别的数据集。为了训练机器学习模型,机器学习模型可以安装在“训练”集上,超参数可以根据“训练”和“dev”集上的性能进行调整,并且最终性能可以在“测试”集上进行评估。
50.在一些实施方案中,训练数据集包括训练核酸序列数据。训练核酸序列数据可以对应于无偏的“fastq级”数据集,这些数据集是经由下采样和组合来自训练样本的读数产生的。训练样本可以包括正常样本和肿瘤样本。fastq级别的数据集可以生成99的大小和11个独特的样本,并且可能来自肿瘤细胞和匹配的正常细胞的体外稀释测序。与vcf级别的数据集相比,fastq级别的数据集可以处于读段级别而不是变体级别,并因此更接近原始数据。在一些情况下,fastq级别的数据集用于使用更真实的数据来改进或优化性能。由于生成大型fastq级数据集在计算上很困难,因此可以使用fastq级数据集使用迁移学习来训练机器学习模型。迁移学习是使用在vcf级数据集上预训练的机器学习模型执行的,并进行5重交叉验证。在一些情况下,每个机器学习模型可以在4/5的训练数据集上进行训练,并在每次迭代的1/5的训练数据集上进行测试。c.用于预估生物样品的肿瘤纯度的机器学习模型的训练和测试
51.机器学习模型(例如,完全连接的神经网络)可以在训练数据集(例如,vcf级数据集)上进行训练。在一些情况下,机器学习模型首先在vcf级数据集上进行训练。机器学习模型可以安装在“训练”集上,超参数可以根据“训练”和“dev”集上的性能进行调整,并且最终性能可以在“测试”集上进行评估。图4a显示了根据一些实施方案的用vcf级数据集训练的全连接神经网络、一维卷积神经网络和二维卷积神经网络的损失相对于epoch图表400a。损失相对于epoch图表400a显示每个训练的机器学习模型的性能水平在每个epoch都增加,从而达到范围在约0.01至0.025之间的误差值(例如,均方误差)。
52.图4b显示了根据一些实施方案的用vcf级数据集训练的全连接神经网络、一维卷积神经网络和二维卷积神经网络的均方根误差(rsme)和平均绝对误差(mae)数据400b。所有模型在vcf级数据集上的性能相当,其中基于训练数据集的均方根误差值范围介于0.081(二维卷积神经网络)和0.088(全连接神经网络)之间。如图4a和图4b所示,利用vcf级训练和测试数据集训练的所有机器学习模型间的一致性能可以表明拟合良好,可能接近预估样品肿瘤纯度的最佳值。
53.在一些实施方案中,fastq级数据集用于改进或优化训练的机器学习模型的性能。由于生成大型fastq级数据集在计算上很困难,因此可以使用fastq级数据集使用迁移学习来训练机器学习模型。可以使用在vcf级数据集上预训练的机器学习模型来执行迁移学习。可以使用5重交叉验证策略来评估经过训练的机器学习模型的性能。在一些情况下,每个机器学习模型可以在4/5的训练数据集上进行训练,并在每次迭代的1/5的训练数据集上进行测试。图4c显示了根据一些实施方案的用fastq级数据集训练的全连接神经网络、一维卷积神经网络和二维卷积神经网络的rsme和mae数据400c。rmse和mae数据400c表明经过训练的一维卷积神经网络(例如,0.057rmse)和经过训练的二维卷积神经网络(例如,0.067rmse)之间的性能水平相当。另外,rmse和mae数据400c表明经过训练的机器学习模型在fastq级数据集上进行迁移学习后实现了较低的rsme和mae。
54.评估了每个经过训练的机器学习模型的性能水平。图5a显示了根据一些实施方案
0.99、0.9-0.98、0.9-0.97、0.9-0.96或0.9-0.95的fl评分训练经过训练的机器学习模型以预估肿瘤纯度。iii.预估的肿瘤纯度
57.图6示出了根据一些实施方案的用于使用经过训练的机器学习模型来估计肿瘤纯度的示意图600的实例。可以使用经过训练的机器学习模型(例如回归模型)来估计来自未匹配生物样品测序的肿瘤纯度。在一些情况下,深度神经网络用于根据未配对生物样品中核苷酸序列变体的等位基因分数分布(例如,b等位基因频率分布)来预估肿瘤纯度。回归模型可用于从全外显子组测序数据的b等位基因频率(baf)预测肿瘤纯度。
58.在框605中,可以从癌症患者获得不匹配的肿瘤样品(即,没有匹配的正常样品)。在框610中,从肿瘤样品中提取dna、处理并进行全外显子组测序。在一些情况下,对测序读数进行质量控制处理(例如,经由fastqc)以提供fastq文件。将fastq文件与参考基因组对齐以生成bam文件。
59.在框615中,gatk haplotypecaller可用于从bam文件中调用变体,并生成包含变体信息的vcf文件。可以鉴定样本外显子组中的杂合位点,并且可以过滤vcf文件以获得所有杂合位点的参考和替换读段深度。该信息用于计算b等位基因频率(baf)。为跨外显子组的杂合位点计算标准化的baf分布。
60.在框620中,可以使用经过训练的机器学习模型从标准化的baf分布估计肿瘤纯度。尽管缺乏匹配的正常样本,但经过训练的机器学习模型可以因此生成对肿瘤纯度的准确估计。a.对象和样品
61.为了预估肿瘤纯度,可以从对象的生物样品中获得代表多个核酸分子的核酸序列数据。对象可以是人。对象可以是男性或女性。受试者可以是胎儿、婴儿、儿童、青春期孩子、青少年或成人。对象可以是任何年龄的患者。例如,对象可以是小于约10岁的患者。例如,对象可以是至少约0、5、10、20、30、40、50、60、70、80、90或100岁的患者。对象可以是正在接受治疗方案或正在评估治疗方案(例如,癌症治疗)的患者或其他个体。然而,在一些情况下,对象没有接受治疗方案。
62.在一些情况下,对象可以是哺乳动物或非哺乳动物。在一些情况下,对象是哺乳动物,如人、非人灵长类动物(例如猿、猴、黑猩猩)、猫、狗、兔、山羊、马、牛、猪、啮齿动物、小鼠、scid小鼠、大鼠、豚鼠或羊。在一些实施方案中,这些基因的物种变体或同源物用于非人类动物模型中。物种变体可以是不同物种的基因,它们在功能特性上具有最大的彼此序列同一性和相似性。许多此类物种变体人类基因可能会列在swiss-prot数据库中。
63.某些实施方案可以包括从对象如人类对象获得样品。在一些情况下,获得来自患者的临床样本。例如,可以从患者抽取血液。某些实施方案可包括特异性检测、分析或定量生物样品内的分子(例如,核酸、dna、rna等)。
64.样品可以是组织样品或体液。在一些情况下,样品是组织样品或器官样品,如活组织检查。在一些情况下,样品包括癌细胞。在一些情况下,样品包括癌细胞和正常细胞。在一些情况下,样品是肿瘤活检。体液可以是汗液、唾液、眼泪、尿液、血液、月经血、精液和/或脊髓液。在一些情况下,样品是血液样品。样品可以包括一种或多种外周血淋巴细胞。样品可以是全血样品。血样可以是外周血样。在一些情况下,样品包括外周血单核细胞(pbmc);在
一些情况下,样本包括外周血淋巴细胞(pbl)。样品可以是血清样品。
65.可以使用可以提供适用于本文所述分析方法的样品的任何方法获得样品。样品可以通过非侵入性方法获得,如咽拭子、口腔拭子、支气管灌洗、尿液采集、皮肤或子宫颈刮擦、脸颊拭子、唾液采集、粪便采集、月经血采集或精液采集。样品可以通过诸如抽血的微创方法获得。样本可以通过静脉穿刺获得。在其他情况下,样品通过侵入性程序获得,包括但不限于:活检、肺泡或肺灌洗或针吸。活检的方法可以包括手术活检、切开活检、切除活检、穿孔活检、剃毛活检或皮肤活检。样品可以是福尔马林固定切片。针抽吸的方法还可以包括细针抽吸、核心针活检、真空辅助活检或大核心活检。在一些情况下,可以通过本文的方法获得多个样品以确保足够量的生物材料。在一些情况下,样本不是通过活检获得的。在一些情况下,样本不是肾活检。
66.本公开的方法可用于预估样品中的肿瘤纯度,包括至少约1%、5%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%或更多的肿瘤细胞。在一些实施方案中,本公开的方法可用于预估样品中的肿瘤纯度,包括至多约10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%或更多的肿瘤细胞。b.产生核酸测序数据
67.在一些实施方案中,处理样品以获得核酸序列数据。“核酸”或“核酸分子”可以对应于任何长度的核苷酸的聚合形式,无论是核糖核苷酸、脱氧核糖核苷酸还是肽核酸(pna),其包括嘌呤和嘧啶碱基,或其他天然的、化学或生物化学修饰的非天然或衍生的核苷酸碱基。多核苷酸的骨架可以包括糖和磷酸基团,如通常在rna或dna中发现的,或者修饰或取代的糖或磷酸基团。多核苷酸可以包括修饰的核苷酸,如甲基化核苷酸和核苷酸类似物。核苷酸序列可以被非核苷酸成分打断。因此,术语核苷、核苷酸、脱氧核苷和脱氧核苷酸通常包括类似物,如本文所述的那些。这些类似物是与天然存在的核苷或核苷酸具有一些共同结构特征的那些分子,使得当掺入核酸或寡核苷序列中时,它们允许与溶液中的天然存在的核酸序列杂交。通常,这些类似物通过替换和/或修饰碱基、核糖或磷酸二酯部分而衍生自天然存在的核苷和核苷酸。可以定制改变,以根据需要使杂交形成稳定或不稳定或增强与互补核酸序列杂交的特异性。核酸分子可以是dna分子。核酸分子可以是rna分子。
68.样品处理包括核酸样品处理和后续的核酸样品测序。可以对一些或所有生物样品进行测序以提供核酸序列数据,这些数据可以存储或以其他方式保持在电子、磁性或光学存储位置中。可以在计算机处理器的帮助下分析序列信息,并且可以将分析的序列信息存储在电子存储位置中。电子存储位置可以包括从核酸样品产生的序列信息和分析的序列信息的库或集合。在一些实施方案中,生物样品取自患有或怀疑患有癌症的对象。
69.在一些实施方案中,核酸测序数据由纯肿瘤和纯正常样品产生。配对细胞系可以从另一来源(例如,美国典型培养物保藏中心)获得。每个匹配对可以包括来自同一对象的肿瘤细胞系和正常细胞系。细胞系可以在体外培养和扩增,以获得合适数量的细胞用于dna提取。dna被提取、处理并进行全外显子组或全基因组测序。序列读段可以进行质量控制处理(例如,经由fastqc)以提供fastq文件。
70.在一些情况下,核酸序列数据是使用全基因组测序产生的。在一些情况下,全基因组测序用于鉴定人中的变异。在一些情况下,测序可以包括对一小部分基因组进行深度测
序。例如,基因组的分数可能至少约为50;75;100;125;150;175;200;225;250;275;300;350;400;450;500;550;600;650;700;750;800;850;900;950;1,000;1100;1200;1300;1400;1500;1600;1700;1800;1900;2,000;3,000;4,000;5,000;6,000;7,000;8,000;9,000;10,000;15,000;20,000;30,000;40,000;50,000;60,000;70,000;80,000;90,000;100,000个或更多个碱基或碱基对。在一些情况下,基因组可以被测序超过100万、200万、300万、400万、500万、600万、700万、800万、900万、1000万或超过1000万个碱基或碱基对。在一些情况下,基因组可以在整个外显子组上进行测序(例如,全外显子组测序)。在一些情况下,深度测序可能包括获取基因组部分的多个读段。例如,获取多个读段可以包括基因组部分的至少2、3、4、5、6、7、8、9、10、20、30、40、50、60、70、80、90、100、200、300、400、500、600、700、800、900、1000、10,000个读段或超过10,000个读段。
71.在一些情况下,生成核酸序列数据包括通过深度测序检测低等位基因部分。在一些情况下,通过下一代测序完成深度测序。在一些情况下,通过避免容易出错的区域来执行深度测序。在一些情况下,容易出错的区域可以包括接近序列重复的区域、异常高或低%gc的区域、接近均聚物、二核苷酸和三核苷酸的区域,以及接近其他短重复的区域。在一些情况下,易错区域可能包括导致dna测序错误(例如,均聚物序列中的聚合酶滑移)的区域。
72.在一些情况下,产生核酸序列数据包括对样品中的一种或多种核酸分子进行一种或多种测序反应。某些实施方案可以包括样品中的一种或多种核酸分子进行1个或多个、2个或更多个、3个或更多个、4个或更多个、5个或更多个、6个或更多个、7个或更多个、8个或更多个、9个或更多个、10个或更多个、15个或更多个、20个或更多个、30个或更多个、40个或更多个、50个或更多个、60个或更多个、70个或更多个、80个或更多个、90个或更多个、100个或更多个、200个或更多个、300个或更多个、400个或更多个、500个或更多个、600个或更多个、700个或更多个、800个或更多个、900个或更多个,或者1000个或更多个测序反应。测序反应可以同时、依次或以它们的组合进行。测序反应可以包括全基因组测序或外显子组测序。测序反应可能包括maxim-gilbert、链终止或高通量系统。可选地或另外,测序反应可包括helioscopetm单分子测序、纳米孔dna测序、lynx therapeutics的大规模平行特征测序(mpss)、454焦磷酸测序、单分子实时(rnap)测序、illumina(solexa)测序、solid测序、ion torrenttm、离子半导体测序、单分子smrt(tm)测序、polony测序、dna纳米球测序、visigen biotechnologies方法或以上的组合。可选地或另外,测序反应可包括一个或多个测序平台,包括但不限于illumina提供的genome analyzer iix、hiseq和miseq、单分子实时(smrttm)技术,如由pacific biosciences(california)和solexa sequencer提供的pacbio rs系统、真正的单分子测序(tsmstm)技术,如由helicos inc.(cambridge,ma)提供的heliscopetm sequencer。测序反应还可能包括电子显微镜或化学敏感场效应晶体管(chemfet)阵列。在本公开的一些方面,测序反应包括毛细管测序、下一代测序、sanger测序、合成测序、连接测序、杂交测序、单分子测序,或以上的组合。合成测序可包括可逆终止子测序、进行性单分子测序、顺序流动测序或以上的组合。顺序流动测序可包括焦磷酸测序、ph介导的测序、半导体测序或以上的组合。
73.在一些情况下,产生核酸序列数据包括进行至少一次长读段测序反应和至少一次短读段测序反应。可以对核酸分子子集的至少一部分进行长读段测序反应和/或短读段测序反应。长读段测序反应和/或短读段测序反应可以在两个或更多个核酸分子亚组的至少
一部分上进行。可以对一个或多个核酸分子亚组的至少一部分进行长读段测序反应和短读段测序反应。
74.一个或多个核酸分子或其亚组的测序可以包括至少约5;10;15;20;25;30;35;40;45;50;60;70;80;90;100;200;300;400;500;600;700;800;900;1,000;1500;2,000;2500;3,000;3500;4000;4500;5,000;5500;6,000;6500;7,000;7500;8,000;8500;9,000;10,000;25,000;50,000;75,000;100,000;250,000;500,000;750,000;10,000,000;25,000,000;50,000,000;100,000,000;250,000,000;500,000,000;750,000,000;1,000,000,000个或更多个测序读段。
75.测序反应可以包括测序一个或多个核酸分子的至少约50;60;70;80;90;100;110;120;130;140;150;160;170;180;190;200;210;220;230;240;250;260;270;280;290;300;325;350;375;400;425;450;475;500;600;700;800;900;1,000;1500;2,000;2500;3,000;3500;4,000;4500;5,000;5500;6,000;6500;7,000;7500;8,000;8500;9,000;10,000;20,000;30,000;40,000;50,000;60,000;70,000;80,000;90,000;100,000个或更多个碱基或碱基对。测序反应可以包括测序一个或多个核酸分子的至少约50;60;70;80;90;100;110;120;130;140;150;160;170;180;190;200;210;220;230;240;250;260;270;280;290;300;325;350;375;400;425;450;475;500;600;700;800;900;1,000;1500;2,000;2500;3,000;3500;4,000;4500;5,000;5500;6,000;6500;7,000;7500;8,000;8500;9,000;10,000;20,000;30,000;40,000;50,000;60,000;70,000;80,000;90,000;100,000或更多个连续碱基或碱基对。
76.在一些情况下,测序技术每次运行产生至少100个读段、每次运行产生至少200个读段、每次运行产生至少300个读段、每次运行产生至少400个读段、每次运行产生至少500个读段、每次运行产生至少600个读段、每次运行产生至少700个读段、每次运行产生至少800个读段、每次运行产生至少900个读段、每次运行产生至少1000个读段、每次运行产生至少5,000个读段、每次运行产生至少10,000个读段、每次运行产生至少50,000个读段、每次运行产生至少100,000个读段、每次运行产生至少500,000个读段,或每次运行产生至少1,000,000个读段。可选地,测序技术每次运行产生至少1,500,000个读段、每次运行产生至少2,000,000个读段、每次运行产生至少2,500,000个读段、每次运行产生至少3,000,000个读段、每次运行产生至少3,500,000个读段、每次运行产生至少4,000,000个读段、每次运行产生4,500,000个读段,或每次运行产生至少5,000,000个读段。
77.在一些情况下,测序技术产生至少约30个碱基对、至少约40个碱基对、至少约50个碱基对、至少约60个碱基对、至少约70个碱基对、至少约80个碱基对、至少约90个碱基对、至少约100个碱基对、至少约110个、至少约120个碱基对/读段、至少约150个碱基对、至少约200个碱基对、至少约250个碱基对、至少约300个碱基对、至少约350个碱基对、至少约400个碱基对、至少约450个碱基对、至少约500个碱基对、至少约550个碱基对、至少约600个碱基对、至少约700个碱基对、至少约800个碱基对、至少约900个碱基对,或至少约1,000个碱基对/读段。另外或可选地,测序技术可以产生长测序读段。在一些情况下,测序技术可以产生至少约1,200个碱基对/读段、至少约1,500个碱基对/读段、至少约1,800个碱基对/读段、至少约2,000个碱基对/读段、至少约2,500个碱基对/读段、至少约3,000个碱基对/读段、至少约3,500个碱基对/读段、至少约4,000个碱基对/读段、至少约4,500个碱基对/读段、至少约
5,000个碱基对/读段、至少约6,000个碱基对/读段、至少约7,000个碱基对/读段、至少约8,000个碱基对/读段、至少约9,000个碱基对/读段、至少约10,000个碱基对/读段、20,000个碱基对/读段、30,000个碱基对/读段、40,000个碱基对/读段、50,000个碱基对/读段、60,000个碱基对/读段、70,000个碱基对/读段、80,000个碱基对/读段、90,000个碱基对/读段,或100,000个碱基对/读段。
78.高通量测序系统可以允许在其掺入生长链之后或在掺入时立即检测测序的核苷酸,即实时或基本实时地检测序列。在一些情况下,高通量测序每小时产生至少1,000个、至少5,000个、至少10,000个、至少20,000个、至少30,000个、至少40,000个、至少50,000个、至少100,000个或至少500,000个序列读段;每个读段为每读段至少50个、至少60个、至少70个、至少80个、至少90个、至少100个、至少120个、至少150个、至少200个、至少250个、至少300个、至少350个、至少400个、至少450个或至少500个碱基。可以使用本文所述的核酸如基因组dna、源自rna转录物的cdna或rna作为模板进行测序。c.核苷酸序列变体的鉴定
79.为了预估生物样品的肿瘤纯度,可以确定生物样品的b等位基因频率(baf)。为了确定baf,可以鉴定一组基因组区域,其中每组基因组区域可以包括相对于参考基因组的相应基因组区域的核苷酸序列变体。为了确定一组基因组区域,可以将核酸序列数据与参考基因组进行比对。例如,对应于核酸序列数据的fastq文件可以与参考基因组比对以生成一个或多个bam文件。一个或多个bam文件可以由另一个模块(例如,gatk haplotypecaller)处理以鉴定一组基因组区域。在一些情况下,会生成包含一组基因组区域的vcf文件。另外或可选地,可以为匹配的细胞系获得vcf文件。vcf文件可以基于核酸序列数据和参考基因组的比较来鉴定具有核苷酸序列变异的一组基因组区域。
80.某些实施方案可以包括包含一个或多个基因组区域的核酸分子。某些实施方案可以包括包含一组或多组基因组区域的核酸分子。一个或多个基因组区域可以包括一个或多个基因组区域特征。基因组区域特征可以包括整个基因组或其一部分。基因组区域特征可以包括整个外显子组或其一部分。基因组区域特征可以包括一组或多组基因。基因组区域特征可以包括一个或多个基因。基因组区域特征可以包括一组或多组调控元件。基因组区域特征可以包括一个或多个调控元件。
81.基因组区域特征可以包括一组多态性。基因组区域特征可以包括一种或多种多态性。基因组区域特征可能与一种或多种核酸分子的gc含量、复杂性和/或可映射性有关。基因组区域特征可以包括一个或多个简单串联重复(str)、不稳定的扩展重复、节段重复、单个和成对的读段简并性映射分数、grch37补丁或以上的组合。基因组区域特征可以包括来自全基因组测序(wgs)的一个或多个低平均覆盖区域、来自wgs的零平均覆盖区域、经验证的压缩或以上的组合。基因组区域特征可以包括一个或多个替代或非参考序列。基因组区域特征可以包括一种或多种基因定相和重组基因。在本公开的一些方面,一个或多个基因组区域特征不是相互排斥的。例如,包括整个基因组或其一部分的基因组区域特征可以与另外的基因组区域特征重叠,如整个外显子组或其一部分、一个或多个基因、一个或多个调节元件等。可选地,一种或多种基因组区域特征是相互排斥的。例如,包括整个基因组的非编码部分的基因组区域不会与基因组区域特征重叠,如外显子组或其一部分或基因的编码部分。可选地或另外,一个或多个基因组区域特征是部分排斥的或部分包含的。例如,包括
整个外显子组或其一部分的基因组区域可以与包括基因的外显子部分的基因组区域部分重叠。然而,包括整个外显子组或其一部分的基因组区域不会与包括基因的内含子部分的基因组区域重叠。因此,包括基因或其一部分的基因组区域特征可以部分排除和/或部分包括包含整个外显子组或其一部分的基因组区域特征。
82.某些实施方案可以包括包含一个或多个基因组区域的核酸样品或分子,其中一个或多个基因组区域中的至少一个包括基因组区域特征,该基因组区域特征包括整个基因组或其一部分。整个基因组或其一部分可以包括基因组的一个或多个编码部分、基因组的一个或多个非编码部分或以上的组合。基因组的编码部分可以包括编码一种或多种蛋白质的基因的一个或多个编码部分。基因组的一个或多个编码部分可以包括整个外显子组或其一部分。可选地或另外,基因组的一个或多个编码部分可以包括一个或多个外显子。
83.基因组的一个或多个非编码部分可以包括一种或多种非编码分子或其一部分。非编码分子可以包括一种或多种非编码rna、一种或多种调节元件、一种或多种内含子、一种或多种假基因、一种或多种重复序列、一种或多种转座子、一种或多种病毒元件、一种或多种端粒、以上的一部分,或以上的组合。非编码rna可能是不翻译成蛋白质的功能性rna分子。非编码rna的实例包括但不限于核糖体rna、转移rna、piwi相互作用rna、microrna、sirna、shrna、snorna、sncrna和lncrna。假基因可能与已知基因有关,且通常不再表达。重复序列可以包括一个或多个串联重复、一个或多个散布重复或以上的组合。串联重复可包括一种或多种卫星dna、一种或多种小卫星、一种或多种微卫星或以上的组合。
84.散布重复可以包括一个或多个转座子。转座子可能是可移动的遗传元件。移动遗传元件通常能够改变它们在基因组中的位置。转座子可分为i类转座因子(i类te)或ii类转座因子(ii类te)。i类te(例如,反转录转座子)通常分两个阶段进行自我复制,首先通过转录从dna到rna,然后通过逆转录从rna回复到dna。然后可以将dna拷贝插入基因组中的新位置。i类te可包括一个或多个长末端重复序列(ltr)、一个或多个长散布核元件(line)、一个或多个短散布核元件(sine)或以上的组合。ltr的实例包括但不限于人内源性逆转录病毒(herv)、中度重复序列4(medium reiterated repeats 4,mer4)和逆转录转座子。line的实例包括但不限于line1和line2。sine可以包括一种或多种alu序列、一种或多种哺乳动物范围的散布重复(mir)或以上的组合。ii类te(例如dna转座子)通常不涉及rna中间体。dna转座子通常从一个位点切割并插入基因组中的另一个位点中。可选地,dna转座子被复制并插入基因组的新位置中。dna转座子的实例包括但不限于mer1、mer2和mariners。病毒元件可以包括一种或多种内源性逆转录病毒序列。端粒通常是染色体末端处的重复dna区域。
85.某些实施方案可以包括核酸样品或核酸分子的子集,其包括一个或多个基因组区域,其中一个或多个基因组区域中的至少一个包括基因组区域特征,该基因组区域特征包括整个外显子组或其一部分。外显子组通常是由外显子形成的基因组的一部分。外显子组可由非翻译区(utr)、剪接位点和/或内含子区形成。整个外显子组或其一部分可以包括蛋白质编码基因的一个或多个外显子。整个外显子组或其一部分可以包括一个或多个非翻译区(utr)、剪接位点和内含子。
86.某些实施方案可以包括包含一个或多个基因组区域的核酸样品或分子,其中一个或多个基因组区域中的至少一个包括基因组区域特征,该基因组区域特征包括基因或其一部分。通常,基因包括编码多肽或功能性rna的核酸片段。基因可以包括一个或多个外显子、
一个或多个内含子、一个或多个非翻译区(utr)或以上的组合。外显子通常是基因的编码部分,被转录成前体mrna序列,并在基因的最终成熟rna产物中。内含子通常是基因的非编码部分,被转录成前体mrna序列,并通过rna剪接去除。utr可以指mrna链上编码序列每一侧的部分。位于编码序列5'侧的utr可称为5'utr(或前导序列)。位于编码序列3'侧的utr可称为3'utr(或尾序列)。utr可以包含一种或多种用于控制基因表达的元件。元件,如调控元件,可能位于5'utr中。调控序列,如多腺苷酸化信号、蛋白质结合位点和mirna结合位点,可能位于3'utr中。位于3'utr中的蛋白质的结合位点可包括但不限于硒代半胱氨酸插入序列(secis)元件和富含au的元件(are)。secis元件可以指导核糖体将密码子uga翻译为硒代半胱氨酸而不是终止密码子。are通常是主要由腺嘌呤和尿嘧啶核苷酸组成的片段,其可能会影响mrna的稳定性。
87.某些实施方案可以包括核酸样品或核酸分子子集,其包括一个或多个基因组区域,其中一个或多个基因组区域中的至少一个包括包含一组基因的基因组区域特征。该组基因可以包括但不限于孟德尔db基因、人类基因突变数据库(hgmd)基因、癌症基因普查基因、在线人类孟德尔遗传(omim)孟德尔基因、hgmd孟德尔基因和人类白细胞抗原(hla)基因。该组基因可以具有一种或多种已知孟德尔特征、一种或多种已知疾病特征、一种或多种已知药物特征、一种或多种已知生物医学可解释变体或以上的组合。孟德尔性状可能由单个基因座控制,并可能表现出孟德尔遗传模式。一组具有已知孟德尔特征的基因可能包括一个或多个编码孟德尔特征的基因,包括但不限于品尝苯硫脲的能力(显性)、闻(苦杏仁样)氰化氢的能力(隐性)、白化病(隐性)、短指(手指和脚趾短)和湿(显性)或干(隐性)耳垢。疾病特征会导致或增加疾病风险,并且可能以孟德尔或复杂模式遗传。一组具有已知疾病特征的基因可以包括一个或多个编码疾病特征的基因,包括但不限于囊性纤维化、血友病和林奇综合征。
88.药物特性可能会改变一种或多种药物或药物家族的代谢、最佳剂量、不良反应和副作用。一组具有已知药物性状的基因可以包括一个或多个编码药物性状的基因,包括但不限于cyp2d6、ugt1a1和adrb1。生物医学可解释的变体可以是与疾病或适应症相关的基因中的多态性。一组具有已知生物医学可解释变异的基因可以包括一个或多个编码生物医学可解释变异的基因,包括但不限于囊性纤维化(cf)突变、肌营养不良症突变、p53突变、rb突变、细胞周期调节剂、受体和激酶。可选地或另外,一组具有已知生物医学可解释变异的基因可以包括与亨廷顿氏病、癌症、囊性纤维化、肌营养不良症(例如,杜氏肌营养不良症)相关的一个或多个基因。
89.某些实施方案可以包括包含一个或多个基因组区域的核酸样品或分子,其中一个或多个基因组区域中的至少一个包括基因组区域特征,该基因组区域特征包括调控元件或其一部分。调控元件可以是顺式调控元件或反式调控元件。顺式调控元件可能是控制附近基因转录的序列。顺式调控元件可能位于5'或3'非翻译区(utr)中或内含子内。反式调控元件可以控制远距离基因的转录。调控元件可以包括一个或多个启动子、一个或多个增强子或以上组合。启动子可以促进特定基因的转录,并且可以在编码区的上游发现。增强子可能对基因的转录水平产生远距离影响。
90.某些实施方案可以包括核酸样品或核酸分子子集,其包括一个或多个基因组区域,其中一个或多个基因组区域中的至少一个包括基因组区域特征,该基因组区域特征包
括多态性或其一部分。通常,多态性是指基因型中的突变。多态性可以是生殖系变体或体细胞变体。多态性可以包括一个或多个碱基改变、一个或多个碱基的插入、重复或缺失。拷贝数变异(cnv)、颠换和其他重排也是遗传变异的形式。多态性标记包括限制性片段长度多态性、可变数目的串联重复(vntr)、高变区、小卫星、二核苷酸重复、三核苷酸重复、四核苷酸重复、简单序列重复和插入元件如alu。在选定群体中最常出现的等位基因形式有时被称为野生型形式。二倍体生物对于等位基因形式可以是纯合的或杂合的。二等位基因多态性有两种形式。三等位基因多态性有三种形式。单核苷酸多态性(snp)是多态性的一种形式。在本公开的一些方面,一种或多种多态性包括一种或多种单核苷酸变异、插入缺失(indel)、小插入、小缺失、结构变体连接、可变长度串联重复、侧翼序列或以上的组合。一种或多种多态性可以位于编码和/或非编码区内。一种或多种多态性可位于基因、外显子、内含子、剪接位点、非翻译区或以上的组合之内、周围或附近。一种或多种多态性可能可以跨越基因、外显子、内含子、非翻译区的至少一部分。
91.某些实施方案可包括包含一个或多个基因组区域的核酸样品或分子,其中所述一个或多个基因组区域中的至少一个包括基因组区域特征,所述基因组区域特征包括一个或多个简单串联重复(str)、不稳定扩展重复、节段重复、单个和成对的读段简并性映射分数、grch37补丁或以上的组合。一种或多种str可包括一种或多种均聚物、一种或多种二核苷酸重复、一种或多种三核苷酸重复或以上的组合。一种或多种均聚物可以是约7、8、9、10、11、12、13、14、15、16、17、18、19、20个或更多个碱基或碱基对。二核苷酸重复和/或三核苷酸重复可以是约15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、35、40、45、50个或更多个碱基或碱基对。单个和成对的读段简并性映射分数可以基于或衍生自来自encode/crg(guigo)的gem的100mer的可对齐性、来自encode/crg(guigo)的gem的75mer的可对齐性、对于信号映射能力的100碱基对box car均值、基因座的最大值和配对读段分数的可能对,或以上的组合。
92.基因组区域特征可以包括来自全基因组测序(wgs)的一个或多个低平均覆盖区域、来自wgs的零平均覆盖区域、经验证的压缩或以上的组合。来自wgs的低平均覆盖区域可以包括从illumina v3化学生成的区域、低于基于平均覆盖的泊松分布的第一百分位数的区域,或以上的组合。来自wgs的零均值覆盖区域可能包括由illumina v3化学生成的区域。经验证的压缩可包括高映射深度的区域、具有两个或更多个观察到的单倍型的区域、预期在参考中缺失重复的区域或以上的组合。基因组区域特征可以包括一个或多个替代或非参考序列。一种或多种替代或非参考序列可以包括已知的结构变体连接、已知的插入、已知的缺失、替代的单倍型或以上的组合。基因组区域特征可以包括一种或多种基因定相和重组基因。定相和重组基因的实例包括但不限于一种或多种主要组织相容性复合物、血型和淀粉酶基因家族。一种或多种主要组织相容性复合物可包括一种或多种hla i类、hla ii类或以上的组合。一种或多种hla i类可包括hla-a、hla-b、hla-c或以上的组合。一种或多种hla ii类可以包括hla-dp、hla-dm、hla-doa、hla-dob、hla-dq、hla-dr或以上的组合。血型基因可以包括abo、rhd、rhce或以上的组合。
93.某些实施方案可以包括包含一个或多个基因组区域的核酸样品或分子,其中一个或多个基因组区域中的至少一个包括与一个或多个核酸分子的gc含量相关的基因组区域特征。gc含量可以指核酸分子的gc含量。可选地,gc含量可以指一种或多种核酸分子的gc含
量,并且可以称为平均gc含量。如本文所用,术语“gc含量”和“平均gc含量”可以互换使用。基因组区域的gc含量可以是高gc含量。通常,高gc含量是指大于或等于约65%、70%、75%、80%、85%、90%、95%、97%或更多的gc含量。在本公开的一些方面,高gc含量可以指大于或等于约70%的gc含量。基因组区域的gc含量可以是低gc含量。通常,低gc含量是指gc含量小于或等于约65%、60%、55%、50%、45%、40%、35%、30%、25%、20%、15%、10%、5%、2%或更少。
94.某些实施方案可以包括包含一个或多个基因组区域的核酸样品或分子,其中一个或多个基因组区域中的至少一个包括与一个或多个核酸分子的复杂性相关的基因组区域特征。核酸分子的复杂性可以指核苷酸序列的随机性。低复杂性可以指序列中一种或多种核苷酸种类的模式、重复和/或耗尽。
95.某些实施方案可以包括包含一个或多个基因组区域的核酸样品或分子,其中一个或多个基因组区域中的至少一个包括与一个或多个核酸分子的可映射性相关的基因组区域特征。核酸分子的可映射性可以指其与参考序列比对的唯一性。具有低可映射性的核酸分子可能与参考序列具有较差的比对。d.baf分布的计算
96.对于一组基因组区域中的每个基因组区域,可以确定b等位基因频率(baf)。baf是指两个等位基因(a和b)的等位基因强度比的标准化测量。baf为100或0表示完全不存在两个等位基因之一(例如aa或bb)。例如,当样本的两条染色体都包含相对于参考基因组的核苷酸序列变体时,给定基因组区域的baf可以是100。因此,变体等位基因应该存在于该样本接近100%的读段中。在一些情况下,baf分布被标准化以产生密度图,使得曲线下的面积总和为1。具体来说,生成所有变体的baf直方图,计算该直方图的面积,并将直方图的每个箱除以计算的面积。
97.对于样本中的杂合等位基因,变体将出现在序列读段的子集中。因此,baf为50表明两个等位基因的存在相同(例如ab)。在没有拷贝数变异的情况下,正常样本的baf可能为0、50或100。然而,在肿瘤样本中,突变可以改变baf值。例如,复制b等位基因的拷贝数事件可导致b等位基因频率为—67%。在另一实例中,当只有一条染色体包含变异时,变体等位基因应该存在于该样本接近50%的读段中。数据生成过程固有的随机变化会引入噪声,导致观察到的baf测量值偏离肿瘤和正常样本中给定拷贝数的理想值。
98.在一些情况下,鉴定对应于生物样品(例如,外显子组样品)中杂合位点的基因组区域子集,并且量化每个基因组区域子集的等位基因频率。因此,可以过滤vcf文件以获得所有具有核苷酸序列变异的杂合位点的参考和替换读段深度。此类信息可用于计算具有杂合性的基因组区域子集中的每个基因组区域的baf。
99.基于为每组基因组区域确定的baf,可以计算标准化的baf分布。baf分布可以指示对应于该组基因组区域的每个基因组区域的baf的绝对量、百分比和/或标准化量。在一些情况下,标准化的baf分布是从对应于基因组区域子集(例如,外显子组间的杂合位点)的baf确定的。因为baf值通常介于0和100之间,所以标准化的baf分布可以包括多达101个baf值,每个baf值指示对应的标准化频率。另外或可选地,可以修改标准化的baf分布,使得可以将对应的baf值填充到预定数量的箱中。例如,每个箱可以对应于baf值的非重叠范围(例如,1-9、10-19),并且baf值可以分配给相应的箱。然后可以基于对应于每个预定箱的值来
确定baf分布。
100.图7a-7e提供了根据一些实施方案的绘制的baf分布的实例。对于图7a和7b,x轴代表范围从0%(a等位基因纯合)到100%(b等位基因纯合)的baf值。如上文解释的,baf为50表示杂合性,这对应于两个等位基因的相等存在(例如ab)。正常样品的baf预期可能为0、50或100。然而,在肿瘤样本中,突变可以改变baf值。例如,复制b等位基因的拷贝数事件可导致b等位基因频率为—67%。y轴表示对应于x轴上每个baf值的标准化频率值。标准化的频率值可以鉴定对应于特定baf值的多个基因组区域。例如,50%的baf的标准化频率可以是4以上的值。
101.图7a是来自纯正常样品染色体中杂合位点的b等位基因频率的直方图。直方图呈现正态分布,其中大多数观察到的b等位基因频率下降至接近50%。图7b是来自纯肿瘤样品中杂合位点的b等位基因频率的直方图。直方图显示出改变的分布,其中b等位基因频率的更多观察值从50%进一步下降。如图7b所示,可以显示体细胞突变数量的增加可能导致baf频率从50%改变。
102.图7c是热图,其中每一行代表来自纯正常样品中染色体的baf分布。热图呈现正态分布,其中大多数观察到的b等位基因频率下降到接近50%。图7e是热图,其中每一行代表来自纯正常样品中染色体的baf分布。热图显示出改变的分布,其中b等位基因频率的更多观察值从50%进一步下降。图7d是来自50%正常和50%肿瘤的样品的热图。热图展示了落入图7c和图7e中展示的分布之间的中间分布。e.使用baf对纯肿瘤和纯正常样品进行分类
103.在一些情况下,生物样品的baf特征用于将生物样品分类为正常或具有肿瘤。例如,对于多个纯肿瘤和纯正常样本,可以在外显子组间的杂合位点处计算标准化的baf分布。可以使用具有全外显子组baf特征的逻辑回归对纯肿瘤和纯正常样品进行分类。
104.图8显示了根据一些实施方案的用于使用b等位基因频率特征对生物样品进行分类的前两个主要成分(pc1和pc2)的图表800。在图8中,前两个主成分显示肿瘤和正常的近线性可分性,表明baf特征可能适用于估计样品的肿瘤含量。另外,可以证明可以使用具有全外显子组baf特征的逻辑回归对纯肿瘤和纯正常样本进行分类。f.基于baf分布的肿瘤纯度预估
105.可以使用经过训练的机器学习模型来处理生物样品的baf分布,以预估鉴定生物样品的肿瘤纯度的指标。在一些实施方案中,训练的机器学习模型包括全连接神经网络。全连接神经网络可以包括具有整流线性单元(relu)激活函数的全连接层。在一些实施方案中,全连接神经网络的输出激活函数是sigmoid函数。全连接神经网络的损失函数可以配置为生成均方误差(mse)。在一些实施方案中,完全连接的神经网络通过使用随机采样经由超参数搜索来调整,其中具有层上的线性搜索、大小上的线性搜索、学习率上的对数搜索或以上的组合。
106.在一些情况下,经过训练的机器学习模型包括一维卷积神经网络。可以配置一维卷积神经网络,使得b等位基因频率分布可以用作输入,并且编码为高度为25、宽度为1且深度为100的输入大小。在一些实施方案中,一维卷积神经网络的每一层执行l x 1卷积,然后是relu激活函数。
107.在一些实施方案中,经过训练的机器学习模型包括二维卷积神经网络。在一些实
施方案中,二维卷积神经网络的b等位基因频率分布被用作输入并且被编码为由高度为25、宽度为100且深度为1定义的输入大小。在一些实施方案中,二维卷积神经网络的每个卷积层之后是relu层。二维卷积神经网络的输出可以是具有sigmoid激活函数的密集连接层。另外,可以通过调整层数、滤波器大小、滤波器数量或以上的组合来调整二维卷积神经网络的超参数。
108.可以输出鉴定肿瘤纯度的估计度量。例如,可以输出包括估计度量的报告。在一些实施方案中,报告包括鉴定b等位基因频率分布的信息。报告还可以包括鉴定至少一种诊断标志物和/或至少一种预后标志物的信息。在一些实施方案中,报告包括鉴定预测的体细胞变体的信息。报告还可以包括治疗建议。在一些实施方案中,治疗建议包括向人类对象给予治疗的建议。治疗建议可以包括不向人类对象给予治疗的建议。iv.用于从单一样品预估肿瘤纯度的示例性过程
109.图9包括的流程图900示出了根据某些实施方案预估生物样品的肿瘤纯度的方法的实例。流程图900中描述的操作可以由例如实现经过训练的机器学习模型的计算机系统执行,如经过训练的一维或二维卷积神经网络。尽管流程图900可以将操作描述为顺序过程,但是在各种实施方案中,许多操作可以并行或同时执行。另外,可以重新排列操作的顺序。操作可能具有图中未显示的另外的步骤。此外,方法的实施方案可以通过硬件、软件、固件、中间件、微代码、硬件描述语言或以上的任意组合来实现。当以软件、固件、中间件或微代码实现时,用于执行相关任务的程序代码或代码部分可以存储在诸如存储介质的计算机可读介质中。
110.在操作910处,计算机系统获得代表对象的生物样品的多个核酸分子的核酸序列数据。核酸序列数据可以通过对肿瘤样品的多个核酸分子进行测序来产生。在一些情况下,在测序之前分离多个核酸分子。核酸序列数据可以对应于整个外显子组序列数据。可选地或另外,核酸序列数据是全基因组测序数据。
111.在操作920处,计算机系统将核酸序列数据与参考基因组进行比对。例如,对应于核酸序列数据的fastq文件可以与参考基因组比对以生成一个或多个bam文件。
112.在操作930处,计算机系统基于比对的核酸序列数据鉴定一组基因组区域。该组基因组区域的每个基因组区域可以包括相对于参考基因组的相应基因组区域的一个或多个核苷酸序列变体。在一些情况下,计算机系统鉴定核酸序列数据中的一个或多个候选核苷酸序列变体并计算一个或多个候选核苷酸序列变体中的每一个的参考和替换读段深度。
113.在操作940处,计算机系统确定该组基因组区域中每个基因组区域的baf。baf是指两个等位基因(a和b)的等位基因强度比的标准化测量。在一些情况下,baf为100或0表示完全不存在两个等位基因之一(例如aa或bb),并且baf为50表示两个等位基因的相等存在(例如ab)。正常样品的baf预期可能为0、50或100。然而,在肿瘤样本中,突变可以改变baf值。
114.在操作950处,计算机系统基于该组基因组区域的baf确定生物样品的baf分布。在一些情况下,b等位基因频率被标准化。
115.在操作960处,计算机系统使用经过训练的机器学习模型处理b等位基因频率分布,以预估鉴定生物样品的肿瘤纯度的度量。在一些情况下,经过训练的机器学习模型在训练数据集上进行训练,该训练数据集由源自一个或多个稀释成正常细胞的肿瘤细胞的核酸序列数据生成。经过训练的机器学习模型可能具有小于约0.2的平均绝对误差。
116.在操作970处,计算机系统输出度量。在一些情况下,计算机系统输出包括鉴定肿瘤纯度的预估度量的报告。除了预估的度量之外,报告还可以包括确定的b等位基因频率分布。该报告还可以包括其他类型的信息,包括但不限于至少一种生物标志物、至少一种预后标志物、预测的体细胞变异和治疗建议(例如,是否应该向对象给予治疗的建议)。v.另外的考虑因素a.探测技术
117.某些实施方案可以包括一种或多种标记。一种或多种标记可附接至一种或多种捕获探针、核酸分子、珠子、引物或以上的组合。标记的实例包括但不限于可检测标记,如放射性同位素、荧光团、化学发光团、发色团、发光团、酶、胶体颗粒和荧光微粒、量子点,以及抗原、抗体、半抗原、抗生物素蛋白/链霉抗生物素蛋白、生物素、半抗原、酶辅因子/底物、猝灭系统的一个或多个成员、色原、半抗原、磁性粒子、表现出非线性光学的材料、半导体纳米晶体、金属纳米粒子、酶、适体和结合对的一个或多个成员。
118.某些实施方案可以包括一种或多种捕获探针、多种捕获探针或一种或多种捕获探针组。通常,捕获探针包含核酸结合位点。捕获探针还可包含一种或多种接头。捕获探针还可包含一种或多种标记。一种或多种接头可以将一种或多种标记附接至核酸结合位点。
119.捕获探针可以与样品中的一种或多种核酸分子杂交。捕获探针可以与一个或多个基因组区域杂交。捕获探针可以与一个或多个基因、外显子、内含子、utr或以上的组合内、其周围、其附近或跨越一个或多个基因、外显子、内含子、utr或以上的组合的一个或多个基因组区域杂交。捕获探针可以与跨越一个或多个基因、外显子、内含子、utr或以上的组合的一个或多个基因组区域杂交。捕获探针可以与一种或多种已知的indel杂交。捕获探针可以与一种或多种已知的结构变体杂交。
120.某些实施方案可以包括1个或多个、2个或更多个、3个或更多个、4个或更多个、5个或更多个、6个或更多个、7个或更多个、8个或更多个、9个或更多个、10个或更多个、20个或更多个、30个或更多个、40个或更多个、50个或更多个、60个或更多个、70个或更多个、80个或更多个、90个或更多个、100个或更多个、125个或更多个、150个或更多个、175个或更多个、200个或更多个、250个或更多个、300个或更多个、350个或更多个、400个或更多个、500个或更多个、600个或更多个、700个或更多个、800个或更多个、900个或更多个,或者1000个或更多个一个或多个捕获探针或捕获探针组。一种或多种捕获探针或捕获探针组可以是不同的、相似的、相同的或以上的组合。
121.一种或多种捕获探针可包含与样品或核酸分子子集中的一种或多种核酸分子或其变体或衍生物的至少一部分杂交的核酸结合位点。捕获探针可以包含与一个或多个基因组区域杂交的核酸结合位点。捕获探针可以与不同、相似和/或相同的基因组区域杂交。一种或多种捕获探针可以与一种或多种核酸分子或其变体或衍生物具有至少约50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、97%、99%或更多的互补性。
122.捕获探针可以包含一种或多种核苷酸。捕获探针可以包含1个或多个、2个或更多个、3个或更多个、4个或更多个、5个或更多个、6个或更多个、7个或更多个、8个或更多个、9个或更多个、10个或更多个、20个或更多个、30个或更多个、40个或更多个、50个或更多个、60个或更多个、70个或更多个、80个或更多个、90个或更多个、100个或更多个、125个或更多个、150个或更多个、175个或更多个、200个或更多个、250个或更多个、300个或更多个、350
个或更多个、400个或更多个、500个或更多个、600个或更多个、700个或更多个、800个或更多个、900个或更多个,或者1000个或更多个核苷酸。捕获探针可以包含约100个核苷酸。捕获探针可包含约10至约500个核苷酸、约20至约450个核苷酸、约30至约400个核苷酸、约40至约350个核苷酸、约50至约300个核苷酸、约60至约250个核苷酸,约70至约200个核苷酸,或约80至约150个核苷酸。在本公开的一些方面,捕获探针包含约80个核苷酸至约100个核苷酸。
123.多个捕获探针或捕获探针组可以包含两个或更多个具有相同、相似和/或不同核酸结合位点序列、接头和/或标记的捕获探针。例如,两个或更多个捕获探针包含相同的核酸结合位点。在另一实例中,两个或更多个捕获探针包含相似的核酸结合位点。在又一实例中,两个或更多个捕获探针包含不同的核酸结合位点。两个或更多个捕获探针可以进一步包含一个或多个接头。两个或更多个捕获探针可以进一步包含不同的接头。两个或更多个捕获探针可以进一步包含相似的接头。两个或更多个捕获探针可以进一步包含相同的接头。两个或更多个捕获探针可以进一步包含一种或多种标记。两个或更多个捕获探针可以进一步包含不同的标记。两个或更多个捕获探针可以进一步包含相似的标记。两个或更多个捕获探针可以进一步包含相同的标记。b.测定和扩增技术
124.某些实施方案可以包括对包含一种或多种核酸分子的样品进行一种或多种测定。产生两个或更多个核酸分子亚组可以包括进行一种或多种测定。可以对来自样品的核酸分子亚组进行测定。可以对来自样品的一种或多种核酸分子进行测定。可以对核酸分子亚组的至少一部分进行测定。测定可以包括一种或多种用于检测、定量和/或分析一种或多种核酸分子的技术、试剂、捕获探针、引物、标记和/或组分。
125.测定可包括但不限于一种或多种核酸分子的测序、扩增、杂交、富集、分离、洗脱、片段化、检测、定量。测定可以包括制备一种或多种核酸分子的方法。
126.某些实施方案可以包括对样品中的一种或多种核酸分子进行一种或多种扩增反应。术语“扩增”是指产生至少一个核酸分子拷贝的任何过程。术语“扩增子”和“扩增的核酸分子”是指核酸分子的拷贝并且可以互换使用。扩增反应可包括基于pcr的方法、非基于pcr的方法或以上的组合。非基于pcr的方法的实例包括但不限于多重置换扩增(mda)、转录介导的扩增(tma)、基于核酸序列的扩增(nasba)、链置换扩增(sda)、实时sda、滚环扩增或循环到循环扩增。基于pcr的方法可以包括但不限于pcr、hd-pcr、下一代pcr、数字rta或以上的任何组合。另外的pcr方法包括但不限于线性扩增、等位基因特异性pcr、alu pcr、组装pcr、不对称pcr、液滴pcr、乳液pcr、解旋酶依赖性扩增hda、热启动pcr、反向pcr、线性后-指数(late)-pcr、长pcr、多重pcr、巢式pcr、半巢式pcr、定量pcr、rt-pcr、实时pcr、单细胞pcr和降落pcr。
127.某些实施方案可以包括对样品中的一种或多种核酸分子进行一种或多种杂交反应。杂交反应可以包括一种或多种捕获探针与样品或核酸分子亚组中的一种或多种核酸分子的杂交。杂交反应可以包括将一种或多种捕获探针组与样品或核酸分子子集中的一种或多种核酸分子杂交。杂交反应可包括一个或多个杂交阵列、多重杂交反应、杂交链式反应、等温杂交反应、核酸杂交反应或以上的组合。一个或多个杂交阵列可包括杂交阵列基因分型、杂交阵列比例传感、dna杂交阵列、宏阵列、微阵列、高密度寡核苷酸阵列、基因组杂交阵
列、比较杂交阵列或以上的组合。杂交反应可包含一种或多种捕获探针、一种或多种珠子、一种或多种标记、一个或多个核酸分子亚组、一个或多个核酸样品、一种或多种试剂、一种或多种洗涤缓冲液、一种或多种洗脱缓冲液、一种或多种杂交缓冲液、一个或多个杂交室、一个或多个孵育器、一种或多种分离器或以上的组合。
128.某些实施方案可以包括对样品中的一种或多种核酸分子进行一种或多种富集反应。富集反应可以包括使样品与一个或多个珠子或珠子组接触。富集反应可以包括基于一个或多个基因组区域特征的两个或更多个核酸分子亚组的差异扩增。例如,富集反应包括基于gc含量的两个或更多个核酸分子亚组的差异扩增。可选地或另外,富集反应包括基于甲基化状态的核酸分子的两个或更多个亚组的差异扩增。富集反应可以包括一种或多种杂交反应。富集反应可以进一步包括分离和/或纯化一种或多种杂交核酸分子、一种或多种珠结合核酸分子、一种或多种游离核酸分子(例如,捕获探针游离核酸分子、珠游离核酸分子)、一种或多种标记的核酸分子、一种或多种未标记的核酸分子、一种或多种扩增子、一种或多种未扩增的核酸分子或以上的组合。可选地或另外,富集反应可包括富集样品中的一种或多种细胞类型。可以通过流式细胞术富集一种或多种细胞类型。
129.一种或多种富集反应可以产生一种或多种富集的核酸分子。富集的核酸分子可以包括核酸分子或其变体或衍生物。例如,富集的核酸分子包含一种或多种杂交核酸分子、一种或多种珠结合核酸分子、一种或多种游离核酸分子(例如,捕获探针游离核酸分子、珠游离核酸分子),一种或多种标记的核酸分子、一种或多种未标记的核酸分子、一种或多种扩增子、一种或多种未扩增的核酸分子或以上的组合。富集的核酸分子可以通过gc含量、分子大小、基因组区域、基因组区域特征或以上的组合与非富集核酸分子区分开来。富集的核酸分子可以来源于一种或多种测定、上清液、洗脱液或以上的组合。富集的核酸分子可以通过平均大小、平均gc含量、基因组区域或以上的组合而不同于非富集的核酸分子。
130.某些实施方案可以包括对样品中的一种或多种核酸分子进行一种或多种分离或纯化反应。分离或纯化反应可以包括使样品与一个或多个珠子或珠子组接触。分离或纯化反应可包括一种或多种杂交反应、富集反应、扩增反应、测序反应或以上的组合。分离或纯化反应可以包括使用一个或多个分离器。一个或多个分离器可以包括磁性分离器。分离或纯化反应可以包括将珠结合的核酸分子与珠游离核酸分子分离。分离或纯化反应可以包括将捕获探针杂交的核酸分子与捕获探针游离的核酸分子分离。分离或纯化反应可以包括将核酸分子的第一子集与核酸分子的第二子集分离,其中核酸分子的第一子集与核酸分子上的第二子集的区别在于平均大小、平均gc含量、基因组区域,或以上的组合。
131.某些实施方案可以包括对样品中的一种或多种核酸分子进行一种或多种洗脱反应。洗脱反应可以包括使样品与一个或多个珠子或珠子组接触。洗脱反应可以包括将珠结合的核酸分子与珠游离核酸分子分离。洗脱反应可以包括将捕获探针杂交的核酸分子与捕获探针游离的核酸分子分离。洗脱反应可以包括将核酸分子的第一子集与核酸分子的第二子集分离,其中核酸分子的第一子集与核酸分子上的第二子集的区别在于平均大小、平均gc含量、基因组区域,或以上的组合。
132.某些实施方案可以包括一种或多种片段化反应。片段化反应可以包括片段化样品或核酸分子亚组中的一种或多种核酸分子以产生一种或多种片段化的核酸分子。一种或多种核酸分子可以通过超声处理、针剪切、雾化、剪切(例如,声剪切、机械剪切、点槽剪切)、通
过french压力室或酶消化来片段化。酶消化可通过核酸酶消化(例如,微球菌核酸酶消化、内切核酸酶、外切核酸酶、rna酶h或dna酶i)进行。一种或多种核酸分子的片段化可导致片段大小为约100个碱基对至约2000个碱基对、约200个碱基对至约1500个碱基对、约200个碱基对至约1000个碱基对、约200个碱基对至约500个碱基对,约500个碱基对至约1500个碱基对,以及约500个碱基对至约1000个碱基对。一种或多种片段化反应可导致大小为约50个碱基对至约1000个碱基对的片段。一种或多种片段化反应可产生约100个碱基对、150个碱基对、200个碱基对、250个碱基对、300个碱基对、350个碱基对、400个碱基对、450个碱基对、500个碱基对、550个碱基对、600个碱基对、650个碱基对、700个碱基对、750个碱基对、800个碱基对、850个碱基对、900个碱基对、950个碱基对、1000个碱基对或更多的片段大小。
133.使一种或多种核酸分子片段化可包括对样品中的一种或多种核酸分子进行一段时间的机械剪切。片段化反应可发生至少约10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100、125、150、175、200、225、250、275、300、325、350、375、400、425、450、475、500秒或更多秒。
134.使一种或多种核酸分子片段化可包括使核酸样品与一个或多个珠子接触。使一种或多种核酸分子片段化可包括使核酸样品与多个珠子接触,其中多个珠子的体积与核酸样品的体积之比为约0.10、0.20、0.30、0.40、0.50、0.60、0.70、0.80、0.90、1.00、1.10、1.20、1.30、1.40、1.50、1.60、1.70、1.80、1.90、2.00或更多。使一种或多种核酸分子片段化可包括使核酸样品与多个珠子接触,其中多个珠子的体积与核酸样品的体积之比为约2.00、1.90、1.80、1.70、1.60、1.50、1.40、1.30、1.20、1.10、1.00、0.90、0.80、0.70、0.60、0.50、0.40、0.30、0.20、0.10、0.05、0.04、0.03、0.02、0.01或更小。
135.某些实施方案可以包括对样品中的一种或多种核酸分子进行一种或多种检测反应。检测反应可以包括一个或多个测序反应。可选地,进行检测反应包括光学传感、电传感或以上的组合。光学传感可以包括光致发光光子发射、荧光光子发射、焦磷酸光子发射、化学发光光子发射或以上的组合的光学传感。电传感可包括离子浓度、离子电流调制、核苷酸电场、核苷酸隧穿电流或以上的组合的电感测。
136.某些实施方案可以包括对样品中的一种或多种核酸分子进行一种或多种量化反应。量化反应可包括测序、pcr、qpcr、数字pcr或以上的组合。
137.某些实施方案可以包括一个或多个样品。某些实施方案可包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100个或更多个样品。样品可以来源于对象。两个或更多个样品可以来源于单个对象。两个或更多个样本可以来源于2、3、4、5、6、7、8、9、10、11、12、13、14、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100个或更多个不同的对象。对象可以是哺乳动物、爬行动物、两栖动物、鸟类和鱼类。哺乳动物可以是人、猿、猩猩、猴子、黑猩猩、牛、猪、马、啮齿动物、鸟、爬行动物、狗、猫或其他动物。爬行动物可能是蜥蜴、蛇、短吻鳄、乌龟、鳄鱼和陆龟。两栖动物可能是蟾蜍、青蛙、蝾螈和火蝾螈。鸟类的实例包括但不限于鸭、鹅、企鹅、鸵鸟和猫头鹰。鱼的实例包括但不限于鲶鱼、鳗鱼、鲨鱼和箭鱼。优选地,对象是人。对象可能患有疾病或病况(例如,癌症)。
138.两个或更多个样本可以在1、2、3、4、5、6、7、8、9、10、11、12、15、20、30、40、50、60、70、80、90、100、200、300、400、500、600、700、800、900、1000个或时间点采集。时间点可以在
1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60或更多小时时间段内发生。时间点可以在1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60或更多天时间段内发生。时间点可以在1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60或更多周时间段内发生。时间点可以在1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60或更多月时间段内发生。时间点可以在1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60或更多年时间段内发生。
139.样品可以来自体液、细胞、皮肤、组织、器官或以上的组合。样品可以是血液、血浆、血液部分、唾液、痰液、尿液、精液、经阴道液、脑脊液、粪便、细胞或组织活检。样本可以来自肾上腺、阑尾、膀胱、脑、耳朵、食道、眼睛、胆囊、心脏、肾脏、大肠、肝脏、肺、口腔、肌肉、鼻子、胰腺、甲状旁腺、松果体、垂体腺体、皮肤、小肠、脾脏、胃、胸腺、甲状腺、气管、子宫、阑尾、角膜、皮肤、心脏瓣膜、动脉或静脉。
140.样品可以包含一种或多种核酸分子。核酸分子可以是dna分子、rna分子(例如mrna、crna或mirna)和dna/rna杂合体。dna分子的实例包括但不限于双链dna、单链dna、单链dna发夹、cdna、基因组dna。核酸可以是rna分子,如双链rna、单链rna、ncrna、rna发夹和mrna。ncrna的实例包括但不限于sirna、mirna、snorna、pirna、tirna、pasr、tasr、atasr、tssa-rna、snrna、re-rna、uarna、x-ncrna、hy rna、usrna、snar和vtrna。
141.某些实施方案可以包括一个或多个容器。某些实施方案可以包括1个或多个、2个或更多个、3个或更多个、4个或更多个、5个或更多个、6个或更多个、7个或更多个、8个或更多个、9个或更多个、10个或更多个、20个或更多个、30个或更多个、40个或更多个、50个或更多个、60个或更多个、70个或更多个、80个或更多个、90个或更多个、100个或更多个、125个或更多个、150个或更多个、175个或更多个、200个或更多个、250个或更多个、300个或更多个、350个或更多个、400个或更多个、500个或更多个、600个或更多个、700个或更多个、800个或更多个、900个或更多个,或者1000个或更多个容器。一个或多个容器可以是不同的、相似的、相同的或以上的组合。容器的实例包括但不限于板、微孔板、pcr板、孔、微孔、管、eppendorf管、小瓶、阵列、微阵列和芯片。
142.某些实施方案可以包括一种或多种试剂。某些实施方案可以包括1种或多种、2种或更多种、3种或更多种、4种或更多种、5种或更多种、6种或更多种、7种或更多种、8种或更多种、9种或更多种、10种或更多种、20种或更多种、30种或更多种、40种或更多种、50种或更多种、60种或更多种、70种或更多种、80种或更多种、90种或更多种、100种或更多种、125种或更多种、150种或更多种、175种或更多种、200种或更多种、250种或更多种、300种或更多种、350种或更多种、400种或更多种、500种或更多种、600种或更多种、700种或更多种、800种或更多种、900种或更多种,或者1000种或更多种试剂。一种或多种试剂可以是不同的、相似的、相同的或以上的组合。试剂可以提高一种或多种测定的效率。试剂可以提高核酸分子或其变体或衍生物的稳定性。试剂可以包括但不限于酶、蛋白酶、核酸酶、分子、聚合酶、逆转录酶、连接酶和化合物。某些实施方案可以包括进行包含一种或多种抗氧化剂的测定。通常,抗氧化剂是抑制另一分子氧化的分子。抗氧化剂的实例包括但不限于抗坏血酸(例如维生素c)、谷胱甘肽、硫辛酸、尿酸、胡萝卜素、α-生育酚(例如维生素e)、泛醇(例如辅酶q)和
维生素a。
143.某些实施方案可以包括一种或多种缓冲液或溶液。一种或多种缓冲液或溶液可以是不同的、相似的、相同的或以上的组合。缓冲液或溶液可以提高一种或多种测定的效率。缓冲液或溶液可以提高核酸分子或其变体或衍生物的稳定性。缓冲液或溶液可包括但不限于洗涤缓冲液、洗脱缓冲液和杂交缓冲液。
144.某些实施方案可以包括一个或多个珠子、多个珠子或一个或多个珠子组。某些实施方案可以包括样品中的一种或多种核酸分子进行1个或多个、2个或更多个、3个或更多个、4个或更多个、5个或更多个、6个或更多个、7个或更多个、8个或更多个、9个或更多个、10个或更多个、15个或更多个、20个或更多个、30个或更多个、40个或更多个、50个或更多个、60个或更多个、70个或更多个、80个或更多个、90个或更多个、100个或更多个、200个或更多个、300个或更多个、400个或更多个、500个或更多个、600个或更多个、700个或更多个、800个或更多个、900个或更多个,或者1000个或更多个一个或多个珠子或珠子组。一个或多个珠子或珠子组可以是不同的、相似的、相同的或以上的组合。珠子可以是磁性的、抗体包被的、蛋白a交联的、蛋白g交联的、链霉抗生物素蛋白包被的、寡核苷酸缀合的、二氧化硅包被的或以上的组合。珠子的实例包括但不限于:ampure珠子、ampure xp珠子、链霉抗生物素蛋白珠子、琼脂糖珠子、磁珠、微珠、抗体缀合的珠子(例如,抗免疫球蛋白微珠)、蛋白a缀合的珠子、蛋白g缀合的珠子、蛋白a/g缀合的珠子、蛋白l缀合的珠子、寡聚dt缀合的珠子、二氧化硅珠子、二氧化硅样珠子、抗生物素微珠、抗荧光染料微珠和bcmagtm羧基末端磁珠。在本公开的一些方面,一种或多种珠子包括一种或多种ampure珠子。可选地或另外,一种或多种珠子包含ampure xp珠子。
145.某些实施方案可以包括一种或多种引物、多种引物或一种或多种引物组。引物还可包含一种或多种接头。引物还可包含或更多标记。引物可用于一种或多种测定。例如,引物用于一种或多种测序反应、扩增反应或以上的组合。某些实施方案可以包括1种或多种、2种或更多种、3种或更多种、4种或更多种、5种或更多种、6种或更多种、7种或更多种、8种或更多种、9种或更多种、10种或更多种、20种或更多种、30种或更多种、40种或更多种、50种或更多种、60种或更多种、70种或更多种、80种或更多种、90种或更多种、100种或更多种、125种或更多种、150种或更多种、175种或更多种、200种或更多种、250种或更多种、300种或更多种、350种或更多种、400种或更多种、500种或更多种、600种或更多种、700种或更多种、800种或更多种、900种或更多种,或者1000种或更多种一种或多种引物或引物组。引物可以包含约100个核苷酸。引物可包含约10至约500个核苷酸、约20至约450个核苷酸、约30至约400个核苷酸、约40至约350个核苷酸、约50至约300个核苷酸、约60至约250个核苷酸,约70至约200个核苷酸,或约80至约150个核苷酸。在本公开的一些方面,引物包含约80个核苷酸至约100个核苷酸。一种或多种引物或引物组可以是不同的、相似的、相同的或以上的组合。
146.引物可以与样品或核酸分子子集中的一种或多种核酸分子或其变体或衍生物的至少一部分杂交。引物可以与一个或多个基因组区域杂交。引物可以与不同、相似和/或相同的基因组区域杂交。一种或多种引物可以与一种或多种核酸分子或其变体或衍生物具有至少约50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、97%、99%或更多的互补性。
147.引物可以包含一个或多个核苷酸。引物可以包含1个或多个、2个或更多个、3个或
更多个、4个或更多个、5个或更多个、6个或更多个、7个或更多个、8个或更多个、9个或更多个、10个或更多个、20个或更多个、30个或更多个、40个或更多个、50个或更多个、60个或更多个、70个或更多个、80个或更多个、90个或更多个、100个或更多个、125个或更多个、150个或更多个、175个或更多个、200个或更多个、250个或更多个、300个或更多个、350个或更多个、400个或更多个、500个或更多个、600个或更多个、700个或更多个、800个或更多个、900个或更多个,或者1000个或更多个核苷酸。引物可以包含约100个核苷酸。引物可包含约10至约500个核苷酸、约20至约450个核苷酸、约30至约400个核苷酸、约40至约350个核苷酸、约50至约300个核苷酸、约60至约250个核苷酸,约70至约200个核苷酸,或约80至约150个核苷酸。在本公开的一些方面,引物包含约80个核苷酸至约100个核苷酸。
148.多个引物或引物组可以包含两种或更多种具有相同、相似和/或不同序列、接头和/或标记的引物。例如,两种或更多种引物包含相同的序列。在另一实例中,两种或更多种引物包含相似的序列。在又一实例中,两种或更多种引物包含不同的序列。两种或更多种引物可以进一步包含一个或多个接头。两种或更多种引物可以进一步包含不同的接头。两种或更多种引物可以进一步包含相似的接头。两种或更多种引物可以进一步包含相同的接头。两种或更多种引物可以进一步包含一种或多种标记。两种或更多种引物可以进一步包含不同的标记。两种或更多种引物可以进一步包含相似的标记。两种或更多种引物可以进一步包含相同的标记。
149.捕获探针、引物、标记和/或珠子可以包含一种或多种核苷酸。一种或多种核苷酸可包含rna、dna、dna和rna残基的混合物或其修饰的类似物,如2'-0me或2'-氟代(2'-f)、锁核酸(lna)或无碱基的位点。
150.某些实施方案可以包括一种或多种标记。某些实施方案可以包括1种或多种、2种或更多种、3种或更多种、4种或更多种、5种或更多种、6种或更多种、7种或更多种、8种或更多种、9种或更多种、10种或更多种、20种或更多种、30种或更多种、40种或更多种、50种或更多种、60种或更多种、70种或更多种、80种或更多种、90种或更多种、100种或更多种、125种或更多种、150种或更多种、175种或更多种、200种或更多种、250种或更多种、300种或更多种、350种或更多种、400种或更多种、500种或更多种、600种或更多种、700种或更多种、800种或更多种、900种或更多种,或者1000种或更多种一种或多种标记。一种或多种标记可以是不同的、相似的、相同的或以上的组合。
151.标记的实例包括但不限于本领域熟知的化学、生物化学、生物、比色、酶、荧光和发光标记。标记包括染料、光交联剂、细胞毒性化合物、药物、亲和标记、光亲和标记、反应性化合物、抗体或抗体片段、生物材料、纳米颗粒、自旋标记、荧光团、含金属部分、放射性部分、新官能团、与其他分子共价或非共价相互作用的基团、光笼部分、光化辐射可激发部分、配体、光致异构部分、生物素、生物素类似物、掺入了重原子的部分、化学可裂解基团、光裂解基团、氧化还原活性剂、同位素标记部分、生物物理探针、磷光基团、化学发光基团、电子致密基团、磁性基团、嵌入基团、发色团、能量转移剂、生物活性剂、可检测标记或以上的组合。
152.标记可以是化学标记。化学标记的实例可以包括但不限于生物素和放射亚型(例如,碘、碳、磷酸盐、氢)。
153.本文公开的方法、试剂盒和组合物可包含生物标记。生物标记可包括代谢标记,包括但不限于生物正交叠氮修饰的氨基酸、糖和其他化合物。
154.本文公开的方法、试剂盒和组合物可包含酶标记。酶标记可以包括但不限于辣根过氧化物酶(hrp)、碱性磷酸酶(ap)、葡萄糖氧化酶和0-半乳糖苷酶。酶标记可以是萤光素酶。
155.本文公开的方法、试剂盒和组合物可包含荧光标记。荧光标记可以是有机染料(例如,fitc)、生物荧光团(例如,绿色荧光蛋白)或量子点。荧光标记的非限制性列表包括异硫氰酸荧光素(fitc)、dylight fluors、荧光素、罗丹明(异硫氰酸四甲基罗丹明,tritc)、香豆素、荧光黄和bodipy。标记可以是荧光团。示例性荧光团包括但不限于吲哚羰花青(c3)、吲哚二羰花青(c5)、cy3、cy3.5、cy5、cy5.5、cy7、德克萨斯红、太平洋蓝、俄勒冈绿488、alexa-355、alexa fluor 488、alexa fluor 532、alexa fluor 546、alexa fluor-555、alexa fluor 568、alexa fluor 594、alexa fluor 647、alexa fluor 660、alexa fluor 680、joe、丽丝胺、罗丹明绿、bodipy、异硫氰酸荧光素(fitc)、羧基-荧光素(fam)、藻红蛋白、罗丹明、二氯罗丹明(drhodamine)、羧基四甲基罗丹明(tamra)、羧基-x-罗丹明(roxtm)、liztm、victm nedtm pettm、sybr、picogreen、ribogreen等。荧光标记可以是绿色荧光蛋白(gfp)、红色荧光蛋白(rfp)、黄色荧光蛋白、藻胆蛋白(例如别藻蓝蛋白、藻蓝蛋白、藻红蛋白和藻红蓝蛋白)。
156.某些实施方案可以包括一个或多个接头。某些实施方案可以包括1个或多个、2个或更多个、3个或更多个、4个或更多个、5个或更多个、6个或更多个、7个或更多个、8个或更多个、9个或更多个、10个或更多个、20个或更多个、30个或更多个、40个或更多个、50个或更多个、60个或更多个、70个或更多个、80个或更多个、90个或更多个、100个或更多个、125个或更多个、150个或更多个、175个或更多个、200个或更多个、250个或更多个、300个或更多个、350个或更多个、400个或更多个、500个或更多个、600个或更多个、700个或更多个、800个或更多个、900个或更多个,或者1000个或更多个一个或多个接头。一个或多个接头可以是不同的、相似的、相同的或以上的组合。
157.合适的接头包括能够附接至本文公开的标记、引物和/或捕获探针的任何化学或生物化合物。如果接头附接至标记和引物或捕获探针上,那么合适的接头将能够充分分离标记和引物或捕获探针。合适的接头不会显著干扰引物和/或捕获探针与核酸分子、其一部分或其变体或衍生物杂交的能力。合适的接头不会显著干扰待检测标记的能力。接头可以是刚性的。接头可以是柔性的。接头可以是半刚性的。接头可以是蛋白水解稳定的(例如,抗蛋白水解切割)。接头可以是蛋白水解不稳定的(例如,对蛋白水解切割敏感)。接头可以是螺旋的。接头可以是非螺旋的。接头可以是卷曲的。接头可以是(3-链。接头可以包含转角构象。接头可以是单链。接头可以是长链。接头可以是短链。接头可以包含至少约5个残基、至少约10个残基、至少约15个残基、至少约20个残基、至少约25个残基、至少约30个残基,或至少约40个残基或更多。
158.接头的实例包括但不限于腙、二硫化物、硫醚和肽接头。接头可以是肽接头。肽接头可以包含脯氨酸残基。肽接头可包含精氨酸、苯丙氨酸、苏氨酸、谷氨酰胺、谷氨酸或以上的任何组合。接头可以是异双功能交联剂。
159.某些实施方案可以包括进行对包括一个或多个核酸分子的样品进行1种或多种、2种或更多种、3种或更多种、4种或更多种、5种或更多种、6种或更多种、7种或更多种、8种或更多种、9种或更多种、10种或更多种、11种或更多种、12种或更多种、13种或更多种、14种或
更多种、15种或更多种、20种或更多种、25种或更多种、30种或更多种、35种或更多种、40种或更多种、45种或更多种,或者50种或更多种测定。两种或更多种测定可以是不同的、相似的、相同的或以上的组合。例如,某些实施方案包括进行两个或更多个测序反应。在另一实例中,某些实施方案包括进行两种或更多种测定,其中两种或更多种测定中的至少一种包括测序反应。在又一实例中,某些实施方案包括进行两种或更多种测定,其中两种或更多种测定中的至少两种包括测序反应和杂交反应。两种或更多种测定可以顺序地、同时地或以上的组合进行。例如,两种或更多种测序反应可以同时进行。在另一实例中,某些实施方案包括进行杂交反应,然后进行测序反应。在又一实例中,某些实施方案包括同时进行两种或更多种杂交反应,然后同时进行两种或更多种测序反应。两种或更多种测定可以由一台或多台装置进行。例如,两种或更多种扩增反应可以由pcr机器进行。在另一实例中,两种或更多种测序反应可以由两台或更多台测序仪进行。c.装置
160.某些实施方案可以包括一个或多个装置。某些实施方案可以包括一种或多种测定,该测定包括一个或多个装置。某些实施方案可以包括使用一个或多个装置来执行一个或多个步骤或测定。某些实施方案可以包括在一个或多个步骤或测定中使用一个或多个装置。例如,进行测序反应可以包括一台或多台测序仪。在另一实例中,产生核酸分子子集可以包括使用一个或多个磁性分离器。在又一实例中,一个或多个处理器可用于分析一个或多个核酸样品。示例性装置包括但不限于测序仪、热循环仪、实时pcr仪器、磁分离器、传输装置、杂交室、电泳装置、离心机、显微镜、成像仪、荧光计、光度计、读板机、计算机、处理器和生物分析仪。
161.某些实施方案可以包括一台或多台测序仪。一台或多台测序仪可包括一种或多种hiseq、miseq、hiscan、genome analyzer iix、solid测序仪、ion torrent pgm、454gs junior、pac bio rs或以上的组合。一台或多台测序仪可以包括一种或多种测序平台。一种或多种测序平台可以包括454life technologies/roche的gs flx、solexa/illumina的genome analyzer、applied biosystems的solid、complete genomics的cga platform、pacific biosciences的pacbio rs,或以上的组合。
162.某些实施方案可以包括一台或多台热循环仪。一台或多台热循环仪可用于扩增一种或多种核酸分子。某些实施方案可以包括一种或多种实时pcr仪器。一种或多种实时pcr仪器可以包括热循环仪和荧光计。一种或多种热循环仪可用于扩增和检测一种或多种核酸分子。
163.某些实施方案可以包括一个或多个磁性分离器。一个或多个磁性分离器可用于从悬浮液中分离顺磁性和铁磁性颗粒。一个或多个磁性分离器可以包括一个或多个lifesteptm生物磁性分离器、spherotm fleximag分离器、spherotm micromag分离器、spherotm handimag分离器、spherotm minitube mag分离器、spherotm ultramag分离器、dynamagtm磁体、dynamagtm-2磁体或以上的组合。
164.某些实施方案可以包括一个或多个生物分析仪。一般来说,生物分析仪是一种基于芯片的毛细管电泳仪,其可以分析rna、dna和蛋白质。一种或多种生物分析仪可以包括agilent 2100生物分析仪。
165.某些实施方案可以包括一个或多个处理器。一个或多个处理器可以分析、编译、存
储、分选、组合、评估或以其他方式处理来自一种或多种测定的一个或多个数据和/或结果、基于或源自一种或多种测定的一个或多个数据和/或结果、来自一种或多种测定的一个或多个输出、基于或源自一种或多种测定的一个或多个输出、来自一种或数据和/或结果的一个或多个输出、基于或源自一种或多种数据和/或结果的一个或多个输出,或以上的组合。一个或多个处理器可以传输来自一种或多种测定的一个或多个数据、结果或输出、基于或源自一种或多种测定的一个或多个数据、结果或输出、来自一个或多个数据或结果的一个或多个输出、基于或源自一个或多个数据或结果的一个或多个输出,或以上的组合。一个或多个处理器可以接收和/或存储来自用户的请求。一个或多个处理器可以产生或生成一个或多个数据、结果、输出。一个或多个处理器可以产生或生成一份或多份生物医学报告。一个或多个处理器可以传输一份或多份生物医学报告。一个或多个处理器可以分析、编译、存储、分选、组合、评估或以其他方式处理来自一个或多个数据库的信息、一个或多个数据或结果、一个或多个输出或以上的组合。一个或多个处理器可以分析、编译、存储、分选、组合、评估或以其他方式处理来自1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、30个或更多数据库的信息。一个或多个处理器可以将一个或多个请求、数据、结果、输出和/或信息传输到一个或多个用户、处理器、计算机、计算机系统、存储器位置、装置、数据库或以上的组合。一个或多个处理器可以从一个或多个用户、处理器、计算机、计算机系统、存储器位置、装置、数据库或以上的组合接收一个或多个请求、数据、结果、输出和/或信息。一个或多个处理器可从一个或多个用户、处理器、计算机、计算机系统、存储器位置、装置、数据库或以上的组合检索一个或多个请求、数据、结果、输出和/或信息。
166.某些实施方案可以包括一个或多个存储器位置。一个或多个存储器位置可以存储信息、数据、结果、输出、请求或以上的组合。一个或多个存储器位置可从一个或多个用户、处理器、计算机、计算机系统、装置或以上的组合接收信息、数据、结果、输出、请求或以上的组合。
167.本文所述的方法可以借助一台或多台计算机和/或计算机系统来实施。计算机或计算机系统可以包括电子存储位置(例如,数据库、存储器),其具有用于实现本文提供的方法的机器可执行代码,以及用于执行机器可执行代码的一个或多个处理器。
168.代码可以预编译和配置为与具有适合执行代码的处理器的机器一起使用,或者可以在运行时编译。可以在编程语言中提供代码,所述编程语言可被选择以使代码能够以预编译或编译后的方式执行。
169.一台或多台计算机和/或计算机系统可以分析、编译、存储、分选、组合、评估或以其他方式处理来自一种或多种测定的一个或多个数据和/或结果、基于或源自一种或多种测定的一个或多个数据和/或结果、来自一种或多种测定的一个或多个输出、基于或源自一种或多种测定的一个或多个输出、来自一个或数据和/或结果的一个或多个输出、基于或源自一个或多个数据和/或结果的一个或多个输出,或以上的组合。一台或多台计算机和/或计算机系统可以传输来自一种或多种测定的一个或多个数据、结果或输出、基于或源自一种或多种测定的一个或多个数据、结果或输出、来自一个或多个数据或结果的一个或多个输出、基于或源自一个或多个数据或结果的一个或多个输出,或以上的组合。一台或多台计算机和/或计算机系统可以接收和/或存储来自用户的请求。一台或多台计算机和/或计算机系统可以产生或生成一个或多个数据、结果、输出。一台或多台计算机和/或计算机系统
可以产生或生成一份或多份生物医学报告。一台或多台计算机和/或计算机系统可以传输一份或多份生物医学报告。一台或多台计算机和/或计算机系统可以分析、编译、存储、分选、组合、评估或以其他方式处理来自一个或多个数据库的信息、一个或多个数据或结果、一个或多个输出或以上的组合。一台或多台计算机和/或计算机系统可以分析、编译、存储、分选、组合、评估或以其他方式处理来自1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、30个或更多个数据库的信息。一台或多台计算机和/或计算机系统可以将一个或多个请求、数据、结果、输出和/或信息传输到一个或多个用户、处理器、计算机、计算机系统、存储器位置、装置或以上的组合。一台或多台计算机和/或计算机系统可以从一个或多个用户、处理器、计算机、计算机系统、存储器位置、装置或以上的组合接收一个或多个请求、数据、结果、输出和/或信息。一台或多台计算机和/或计算机系统可以从一个或多个用户、处理器、计算机、计算机系统、存储器位置、装置、数据库或以上的组合检索一个或多个请求、数据、结果、输出和/或信息。d.数据库
170.某些实施方案可以包括一个或多个数据库。某些实施方案可包括至少约1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、30个或更多个数据库。数据库可以包括基因组、蛋白质组、药物基因组、生物医学和科学数据库。数据库可以是公开可用的数据库。可选地或另外,数据库可以包括专有数据库。数据库可以是商业可用的数据库。数据库包括但不限于cosmic、gnomad、dbsnp、mills indels、mendeldb、pharmgkb、varimed、regulome、curated breakseq junctions、人类孟德尔遗传在线(omim)、人类基因组突变数据库(hgmd)、ncbi db snp、ncbi refseq、gencode、go(基因本体)和京都基因和基因组百科全书(kegg)。
171.某些实施方案可以包括分析一个或多个数据库。某些实施方案可包括分析至少约1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、30个或更多个数据库。分析一个或多个数据库可以包括一个或多个算法、计算机、处理器、存储器位置、装置或以上的组合。
172.某些实施方案可以包括基于来自一个或多个数据库的数据和/或信息鉴定一个或多个核酸区域。某些实施方案可以包括基于来自一个或多个数据库的数据和/或信息鉴定一组或多组核酸区域。某些实施方案可以包括基于来自至少约2个或更多个数据库的数据和/或信息鉴定一个或多个核酸区域和/或一组或多组核酸区域。某些实施方案可以包括基于来自至少约3个或更多个数据库的数据和/或信息鉴定一个或多个核酸区域和/或一组或多组核酸区域。某些实施方案可以包括基于来自至少约4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、30个或更多个数据库的数据和/或信息鉴定一个或多个核酸区域和/或一组或多组核酸区域。
173.某些实施方案可以包括基于来自一个或多个数据库的数据和/或信息分析一个或多个结果。某些实施方案可以包括基于来自一个或多个数据库的数据和/或信息分析一组或多组结果。某些实施方案可以包括基于来自一个或多个数据库的数据和/或信息分析一个或多个合并的结果。某些实施例可以包括基于来自至少约2个或更多个数据库的数据和/或信息分析一个或多个结果、结果集和/或合并的结果。某些实施例可以包括基于来自至少约3个或更多个数据库的数据和/或信息分析一个或多个结果、结果集和/或合并的结果。某
些实施方案可以包括基于来自至少约4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、30个或更多个数据库的数据和/或信息分析一个或多个结果、结果集和/或合并的结果。
174.某些实施方案可以包括基于来自一个或多个数据库的数据和/或信息比较一个或多个结果。某些实施方案可以包括基于来自一个或多个数据库的数据和/或信息比较一组或多组结果。某些实施方案可以包括基于来自一个或多个数据库的数据和/或信息比较一个或多个合并的结果。某些实施例可以包括基于来自至少约2个或更多个数据库的数据和/或信息比较一个或多个结果、结果集和/或合并的结果。某些实施例可以包括基于来自至少约3个或更多个数据库的数据和/或信息比较一个或多个结果、结果集和/或合并的结果。某些实施方案可以包括基于来自至少约4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、30个或更多个数据库的数据和/或信息比较一个或多个结果、结果集和/或合并的结果。
175.某些实施方案可以包括生物医学数据库、基因组数据库、生物医学报告、疾病报告、病例对照分析和基于来自一个或多个数据库的数据和/或信息的罕见变异发现分析、一种或多种测定、一个或多个数据或结果、基于或源自一种或多种测定的一个或多个输出、基于或源自一个或多个数据或结果的一个或多个输出,或以上的组合。e.数据集和分析
176.某些实施方案可以包括一个或多个数据、一个或多个数据集、一个或多个合并的数据、一个或多个合并的数据集、一个或多个结果、一组或多组结果、一个或多个合并的结果,或以上的组合。数据和/或结果可以基于或源自一种或多种测定、一个或多个数据库或以上的组合。某些实施方案可以包括分析一个或多个数据、一个或多个数据集、一个或多个合并的数据、一个或多个合并的数据集、一个或多个结果、一组或多组结果、一个或多个合并的结果,或以上的组合。某些实施方案可以包括处理一个或多个数据、一个或多个数据集、一个或多个合并的数据、一个或多个合并的数据集、一个或多个结果、一组或多组结果、一个或多个合并的结果,或以上的组合。
177.某些实施方案可以包括至少一项分析和至少一项处理一个或多个数据、一个或多个数据集、一个或多个合并的数据、一个或多个合并的数据集、一个或多个结果、一组或多组结果、一个或多个合并的结果,或以上的组合。某些实施方案可以包括一项或多项分析和一项或多项处理一个或多个数据、一个或多个数据集、一个或多个合并的数据、一个或多个合并的数据集、一个或多个结果、一组或多组结果、一个或多个合并的结果,或以上的组合。某些实施方案可包括至少1、2、3、4、5、6、7、8、9、10、15、20、30、40、50、60、70、80、90、100、200、300、400、500、600、700、800、900、1000项或更多项独特分析一个或多个数据、一个或多个数据集、一个或多个合并的数据、一个或多个合并的数据集、一个或多个结果、一组或多组结果、一个或多个合并的结果,或以上的组合。某些实施方案可包括至少1、2、3、4、5、6、7、8、9、10、15、20、30、40、50、60、70、80、90、100、200、300、400、500、600、700、800、900、1000项或更多项独特处理一个或多个数据、一个或多个数据集、一个或多个合并的数据、一个或多个合并的数据集、一个或多个结果、一组或多组结果、一个或多个合并的结果,或以上的组合。一项或多项分析和/或一项或多项处理可以同时、顺序或以上的组合发生。
178.一项或多项分析和/或一项或多项处理可在1、2、3、4、5、6、7、8、9、10、11、12、15、20、30、40、50、60、70、80、90、100、200、300、400、500、600、700、800、900、1000个或时间点内发生。时间点可以在1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、
24、25、30、35、40、45、50、55、60或更多小时时间段内发生。时间点可以在1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60或更多天时间段内发生。时间点可以在1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60或更多周时间段内发生。时间点可以在1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60或更多月时间段内发生。时间点可以在1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50、55、60或更多年时间段内发生。
179.某些实施方案可以包括一个或多个数据。一个或多个数据可以包括基于或源自一种或多种测定的一个或多个原始数据。一个或多个数据可以包括基于或源自一个或多个数据库的一个或多个原始数据。一个或多个数据可以包括至少部分地基于或源自一个或多个原始数据分析的数据。一个或多个数据可以包括至少部分地基于或源自一个或多个原始数据处理的数据。一个或多个数据可以包括完全基于或源自一个或多个原始数据分析的数据。一个或多个数据可以包括完全基于或源自一个或多个原始数据处理的数据。数据可以包括测序读段数据或表达数据。数据可以包括生物医学、科学、药理学和/或遗传信息。
180.某些实施方案可以包括一个或多个合并的数据。一个或多个合并的数据可以包括两个或更多个数据。一个或多个合并的数据可以包括两个或更多个数据集。一个或多个合并的数据可以包括一种或多种基于或源自一种或多种测定的原始数据。一个或多个合并的数据可以包括一个或多个基于或源自一个或多个数据库的原始数据。一个或多个合并的数据可以包括至少部分基于或源自一个或多个原始数据分析的数据。一个或多个合并的数据可以包括至少部分基于或源自一个或多个原始数据处理的数据。一个或多个合并的数据可以包括完全基于或源自一个或多个原始数据分析的数据。一个或多个合并的数据可以包括完全基于或源自一个或多个原始数据处理的数据。一个或多个合并的数据可以包括测序读段数据或表达数据。一个或多个合并的数据可以包括生物医学、科学、药理学和/或遗传信息。
181.某些实施方案可以包括一个或多个数据集。一个或多个数据集可以包括一个或多个数据。一个或多个数据集可以包括一个或多个合并的数据。一个或多个数据集可以包括一个或多个基于或源自一种或多种测定的原始数据。一个或多个数据集可以包括基于或源自一个或多个数据库的一个或多个原始数据。一个或多个数据集可以包括至少部分基于或源自一个或多个原始数据分析的数据。一个或多个数据集可以包括至少部分基于或源自一个或多个原始数据处理的数据。一个或多个数据集可以包括完全基于或源自一个或多个原始数据分析的数据。一个或多个数据集可以包括完全基于或源自一个或多个原始数据处理的数据。数据集可以包括测序读段数据或表达数据。数据集可以包括生物医学、科学、药理学和/或遗传信息。
182.某些实施方案可以包括一个或多个合并的数据集。一个或多个合并的数据集可以包括两个或更多个数据。一个或多个合并的数据集可以包括两个或更多个合并的数据。一个或多个合并的数据集可以包括两个或更多个数据集。一个或多个合并的数据集可以包括一个或多个基于或源自一种或多种测定的原始数据。一个或多个合并的数据集可以包括一个或多个基于或源自一个或多个数据库的原始数据。一个或多个合并的数据集可以包括至少部分基于或源自一个或多个原始数据分析的数据。一个或多个合并的数据集可以包括至
少部分基于或源自一个或多个原始数据处理的数据。一个或多个合并的数据集可以包括完全基于或源自一个或多个原始数据分析的数据。一个或多个合并的数据集可以包括完全基于或源自一个或多个原始数据处理的数据。某些实施方案还可以包括对合并的数据集的进一步处理和/或分析。一个或多个合并的数据集可以包括测序读段数据或表达数据。一个或多个合并的数据集可以包括生物医学、科学、药理学和/或遗传信息。
183.某些实施方案可以包括一个或多个结果。一个或多个结果可以包括一个或多个数据、数据集、合并的数据和/或合并的数据集。一个或多个结果可以基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集。一个或多个结果可以从一种或多种测定产生。一个或多个结果可以基于或源自一种或多种测定。一个或多个结果可以基于或源自一个或多个数据库。一个或多个结果可以包括至少部分基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集分析的结果。一个或多个结果可以包括至少部分基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集处理的结果。一个或多个结果可以包括完全基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集分析的结果。一个或多个结果可以包括完全基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集处理的结果。结果可以包括测序读段数据或表达数据。结果可以包括生物医学、科学、药理学和/或遗传信息。
184.某些实施方案可以包括一组或多组结果。一组或多组结果可以包括一个或多个数据、数据集、合并的数据和/或合并的数据集。一组或多组结果可以基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集。一组或多组结果可以从一种或多种测定产生。一组或多组结果可以基于或源自一种或多种测定。一组或多组结果可以基于或源自一个或多个数据库。一组或多组结果可以包括至少部分基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集分析的结果集。一组或多组结果可以包括至少部分基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集处理的结果集。一组或多组结果可以包括完全基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集分析的结果集。一组或多组结果可以包括完全基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集处理的结果集。结果集可以包括测序读段数据或表达数据。结果集可以包括生物医学、科学、药理学和/或遗传信息。
185.某些实施方案可以包括一个或多个合并的结果。合并的结果可以包括一个或多个结果、结果集和/或合并的结果集。合并的结果可以基于或源自一个或多个结果、结果集和/或合并的结果集。一个或多个合并的结果可以包括一个或多个数据、数据集、合并的数据和/或合并的数据集。一个或多个合并的结果可以基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集。一个或多个合并的结果可以从一种或多种测定产生。一个或多个合并的结果可以基于或源自一种或多种测定。一个或多个合并的结果可以基于或源自一个或多个数据库。一个或多个合并的结果可以包括至少部分基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集分析的合并的结果。一个或多个合并的结果可以包括至少部分基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集处理的合并的结果。一个或多个合并的结果可以包括完全基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集分析的合并的结果。一个或多个合并的结果可以包括完全基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集处理的合并的结果。
合并的结果可以包括测序读段数据或表达数据。合并的结果可以包括生物医学、科学、药理学和/或遗传信息。
186.某些实施方案可以包括一组或多组合并的结果。合并的结果集可以包括一个或多个结果、结果集和/或合并的结果。合并的结果集可以基于或源自一个或多个结果、结果集和/或合并的结果。一组或多组合并的结果可以包括一个或多个数据、数据集、合并的数据和/或合并的数据集。一组或多组合并的结果可以基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集。一组或多组合并的结果可以从一种或多种测定产生。一组或多组合并的结果可以基于或源自一种或多种测定。一组或多组合并的结果可以基于或源自一个或多个数据库。一组或多组合并的结果可以包括至少部分基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集分析的合并的结果集。一组或多组合并的结果可以包括至少部分基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集处理的合并的结果集。一组或多组合并的结果可以包括完全基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集分析的合并的结果集。一组或多组合并的结果可以包括完全基于或源自一个或多个数据、数据集、合并的数据和/或合并的数据集处理的合并的结果集。合并的结果集可以包括测序读段数据或表达数据。合并的结果集可以包括生物医学、科学、药理学和/或遗传信息。
187.某些实施方案可以包括一个或多个输出、输出集、合并的输出和/或合并的输出集。本文的方法、文库、试剂盒和系统可以包括产生一个或多个输出、输出集、合并的输出和/或合并的输出集。输出集可以包括一个或多个输出、一个或多个合并的输出或以上的组合。合并的输出可包括一个或多个输出、一组或多组输出、一组或多组合并的输出或以上的组合。合并的输出集可包括一个或多个输出、一组或多组输出、一个或多个合并的输出或以上的组合。一个或多个输出、输出集、合并的输出和/或合并的输出集可以基于或源自一个或多个数据、一个或多个数据集、一个或多个合并的数据、一个或多个合并的数据集、一个或多个结果、一组或多组结果、一个或多个合并的结果或以上的组合。一个或多个输出、输出集、合并的输出和/或合并的输出集可以基于或源自一个或多个数据库。一个或多个输出、输出集、合并的输出和/或合并的输出集可以包括一个或多个生物医学报告、生物医学输出、罕见变异输出、药物遗传学输出、群体研究输出、病例对照输出、生物医学数据库、基因组数据库、疾病数据库、网络内容。
188.某些实施方案可以包括一个或多个生物医学输出、一组或多组生物医学输出、一个或多个合并的生物医学输出、一组或多组合并的生物医学输出。本文的方法、文库、试剂盒和系统可以包括产生一个或多个生物医学输出、一组或多组生物医学输出、一个或多个合并的生物医学输出、一组或多组合并的生物医学输出。生物医学输出的集合可以包括一个或多个生物医学输出、一个或多个合并的生物医学输出或以上的组合。合并的生物医学输出可以包括一个或多个生物医学输出、一组或多组生物医学输出、一组或多组合并的生物医学输出或以上的组合。合并的生物医学输出的集合可以包括一个或多个生物医学输出、一组或多组生物医学输出、一个或多个合并的生物医学输出或以上的组合。一个或多个生物医学输出、一组或多组生物医学输出、一个或多个合并的生物医学输出、一组或多组合并的生物医学输出可以基于或源自一个或多个数据、一个或多个数据集、一个或多个合并的数据、一个或多个合并的数据集、一个或多个结果、一组或多组结果、一个或多个合并的
结果、一个或多个输出、一组或多组输出、一个或多个合并的输出、一组或多组合并的输出,或以上的组合。一个或多个生物医学输出可以包括对象的生物医学信息。对象的生物医学信息可以预测、诊断和/或预后一种或多种生物医学特征。一种或多种生物医学特征可以包括疾病或病况的状态、疾病或病况的遗传风险、生殖风险、对胎儿的遗传风险、药物不良反应的风险、药物治疗的功效、最佳药物剂量的预测、移植耐受性或以上的组合。
189.某些实施方案可以包括一份或多份生物医学报告。本文的方法、文库、试剂盒和系统可以包括产生一份或多份生物医学报告。一份或多份生物医学报告可以基于或源自一个或多个数据、一个或多个数据集、一个或多个合并的数据、一个或多个合并的数据集、一个或多个结果、一组或多组结果、一个或多个合并的结果、一个或多个输出、一组或多组输出、一个或多个合并的输出、一组或多组合并的输出、一个或多个生物医学输出、一组或多组生物医学输出、合并的生物医学输出、一组或多组生物医学输出,或以上的组合。生物医学报告可以预测、诊断和/或预后一种或多种生物医学特征。一种或多种生物医学特征可以包括疾病或病况的状态、疾病或病况的遗传风险、生殖风险、对胎儿的遗传风险、药物不良反应的风险、药物治疗的功效、最佳药物剂量的预测、移植耐受性或以上的组合。
190.某些实施方案还可以包括传输一个或多个数据、信息、结果、输出、报告或以上的组合。例如,基于或源自一个或多个测定的数据/信息被传输到另一装置和/或仪器。在另一实例中,将数据、结果、输出、生物医学输出、生物医学报告或以上的组合传输到另一装置和/或仪器。从算法获得的信息也可以传输到另一装置和/或仪器。基于一个或多个数据库的分析的信息可以被传输到另一装置和/或仪器。数据/信息的传输可以包括数据/信息从第一源到第二源的传输。第一和第二源可以在相同的大致位置(例如,在相同的房间、建筑物、街区、校园内)。可选地,第一和第二源可以在多个位置(例如,多个城市、州、国家、大陆等)。数据、结果、输出、生物医学输出、生物医学报告可以传输给患者和/或医疗保健提供者。
191.传输可以基于对一个或多个数据、结果、信息、数据库、输出、报告或以上的组合的分析。例如,第二报告的传输是基于对第一报告的分析。可选地,报告的传输基于对一个或多个数据或结果的分析。传输可以基于接收一个或多个请求。例如,报告的传输可以基于接收来自用户(例如,患者、医疗保健提供者、个人)的请求。
192.数据/信息的传输可以包括数字传输或模拟传输。数字传输可以包括在点对点或点对多点通信信道上的数据的物理传输(数字比特流)。这种信道的实例是铜线、光纤、无线通信信和存储介质。数据可以表示为电磁信号,如电压、无线电波、微波或红外信号。
193.模拟传输可以包括连续变化的模拟信号的传输。消息可以通过线路代码(基带传输)方式的脉冲序列来表示,也可以通过使用数字调制方法的一组有限的连续变化波形(通带传输)来表示。通带调制和相应的解调(也称为检测)可以通过调制解调器装置进行。根据最常见的数字信号定义,表示比特流的基带和通带信号都被视为数字传输,而一种替代的定义仅将基带信号视为数字信号,并将数字数据的通带传输视为数字模拟转换的一种形式。
194.某些实施方案可以包括一个或多个样品标识符。样品标识符可以包括标记、条形码和其他可以与一个或多个样品和/或核酸分子子集连接的指示物。某些实施方案可以包括一个或多个处理器、一个或多个存储器位置、一个或多个计算机、一个或多个监视器、一
个或多个计算机软件、一种或多种用于将数据、结果、输出、生物医学输出和/或生物医学报告与样品链接的算法。
195.某些实施方案可以包括用于将一种或多种核酸分子的表达水平与疾病结果的预后相关联的处理器。某些实施方案可以包括多种相关技术中的一种或多种,包括查找表、算法、多变量模型以及表达模型或算法的线性或非线性组合。表达水平可以转换为一个或多个可能性分数,反映提供样品的患者可能表现出特定疾病结果的可能性。模型和/或算法可以机器可读格式提供,并且可以任选地进一步指定患者或患者类别的治疗方式。
196.在一些情况下,本文所述的方法和系统用于产生输出,包括检测和/或定量基因组dna区域,如含有dna多态性的区域(例如,生殖系变体或体细胞变体)。在一些情况下,一个或多个基因组区域的检测基于一种或多种算法,这取决于本说明书其他地方描述的数据输入或数据库的来源。可以使用一种或多种算法中的每一种来接收、组合和生成包括基因组区域检测(即多态性)的数据。在一些实施方案中,本方法和系统可包括基于一种或多种、两种或更多种、三种或更多种、四种或更多种、五种或更多种、六种或更多种、七种或更多种、八种或更多种、九种或更多种或者十种或更多种算法的基因组区域的检测。算法可以是机器学习算法、计算机实现的算法、机器执行的算法、自动算法等。
197.可以使用特征选择技术分析每个核酸样品所得的数据,包括通过检查数据的内在属性来评估特征相关性的过滤技术、将模型假设嵌入特征子集搜索内的包装方法,以及嵌入技术,其中对一组最优特征的搜索被内置到算法或模型中。
198.在一些情况下,一种或多种基因组区域的检测基于一种或多种统计模型。可用于本发明方法的统计模型或过滤技术包括(1)参数方法,如使用两个样本t检验、anova分析、贝叶斯框架和gamma分布模型,(2)无模型方法,如使用wilcoxon秩和检验、类间平方和检验、秩积方法、随机排列方法或tnom,其涉及为两个数据集之间表达的倍数变化差异设置阈值点,然后检测每个基因中使错误分类次数最小化的阈值点,以及(3)多变量方法,如双变量方法、基于相关性的特征选择方法(cfs)、最小冗余最大相关性方法(mrmr)、马尔可夫毯式过滤器方法、马尔可夫模型、隐马尔可夫模型(hmm),以及不相关的收缩质心方法。在一些情况下,隐马尔可夫模型(hmm)被赋予内部状态,其中内部状态根据第一或第二核酸样品中染色体的总拷贝数设置。在实例中,对于二倍体染色体,hmm的内部状态可以是纯合缺失(局部零拷贝)、杂合缺失(局部一个拷贝)、正常(局部两个拷贝)、重复(多于两个拷贝)和参考间隙(作为一种状态存在以区分间隙与纯合缺失)。在另一种情况下,对于单倍体染色体(例如x或yin男性)、hmim的内部状态可以是纯合缺失(局部零拷贝)、正常(局部两个拷贝)、重复(多于两个拷贝)和参考间隙(作为一种状态存在以区分间隙与纯合缺失)。例如,对于单倍体染色体,可能没有可用的杂合缺失状态。在另一种情况下,对于三体和/或四体,hmm状态的额外中间物可能具有额外的中间状态,其中中间状态可以解释各种cnv可能性。在另一实施方案中,隐马尔可夫模型用于通过检查在检测到的特征的断点附近的读数的测量插入大小来过滤输出。
199.在本发明的方法中有用的其他模型或算法包括顺序搜索方法、遗传算法、分布估计算法、随机森林算法、支持向量机算法的权重向量、逻辑回归算法的权重等。bioinformatics.2007年10月1日;23(19):2507-17提供了上述用于数据分析的算法或模型的相对优点的概述。说明性算法包括但不限于减少变量数量的方法,如主成分分析算法、偏
最小二乘法、独立成分分析算法、直接处理大量变量的方法如统计方法和基于机器学习技术的方法。统计方法包括惩罚逻辑回归、微阵列预测分析(pam)、基于收缩质心的方法、支持向量机分析和正则化线性判别分析。
200.在一些实施方案中,基于hmm的检测算法可以“分段”检测大的或相当大的cnv。在一些情况下,由于覆盖信号的波动,沿真实cnv的长度可能存在较小的检测间隙。在实例中,可以将1兆碱基对(mbp)缺失检测为少量单独的标称检测,它们之间具有小间隙。为了减轻这种情况,可以采用合并操作来鉴定相邻检测对,这些检测之间的间隙小于两个包围检测中的任何一个。然后合并操作测量间隙中的中值覆盖水平。如果中值覆盖率超过预定义的阈值,则将两个检测合并为单个跨越两个原始检测(包括封闭的检测间隙)的大型检测。在实例中,真实特征跨越两个检测,并且间隙是统计伪影。使用已知具有较大cnv的样本的真实测序数据,这种合并操作可以允许相对于cnv的真实属性显著更好的保真度。
201.本文提供的方法和系统还可包括使用本文提供的特征选择算法。在本发明的一些实施方案中,通过使用limma软件包(smyth,g.k.(2005).limma:linear models for microarray data.bioinformatics and computational biology solutions using r and bioconductor,r.gentleman,v.carey,s.dudoit,r.irizarry,w.huber(eds.),springer,new york,第397-420页)来提供特征选择。
202.在本发明的一些实施方案中,提供了对角线性判别分析、k最近邻算法、支持向量机(svm)算法、线性支持向量机、随机森林算法或基于概率模型的方法或以上的组合,以用于检测一个或多个基因组区域。在一些实施方案中,区分样品(例如,患病相对于正常)或区分基因组区域(例如,拷贝数变异相对于正常)的已鉴定标记是基于目标类别之间表达水平差异的统计学显著性来选择的。在一些情况下,通过应用benjamini hochberg或其他错误发现率(fdr)校正来调整统计显著性。
203.在一些情况下,该算法可能补充有诸如fishel和kaufman et al.2007bioinformatics 23(13):1599-606描述的meta分析方法。在一些情况下,该算法可能补充有meta分析方法,如可重复性分析。在一些情况下,可重复性分析选择出现在至少一个预测性表达产物标记组中的标记。
204.基因组区域检测的统计评估可以提供指示以下一项或多项的一个或多个定量值:诊断准确性的可能性;病症、疾病、病况等的可能性;特定病症、疾病或病况的可能性;以及特定治疗干预成功的可能性。因此,不太可能接受过遗传学或分子生物学培训的医生不需要理解原始数据。相反,数据以定量值的形式直接呈现给医生,以指导患者护理。可以使用本领域已知的许多方法对结果进行统计评估,包括但不限于:学生t检验、双边t检验、pearson秩和分析、隐马尔可夫模型分析、q-q图表分析、主成分分析、单因素anova、双因素anova、limma等。f.疾病或病况
205.某些实施方案可以包括基于一个或多个生物医学输出预测、诊断和/或预测对象的疾病或病况的状态或结果。预测、诊断和/或预后对象中疾病的状态或结果可包括诊断疾病或病况、鉴定疾病或病况、确定疾病或病况的阶段、评估疾病或病况的风险、评估疾病复发的风险、评估药物的功效、评估药物不良反应的风险、预测最佳药物剂量、预测耐药性或以上的组合。
206.本文公开的样品可以来自患有癌症的对象。样品可以包括恶性组织、良性组织或以上的混合物。癌症可以是复发性和/或难治性癌症。癌症的实例包括但不限于肉瘤、癌、淋巴瘤或白血病。在一些情况下,获得了包含癌组织的样品,但没有获得匹配的正常样品。在一些情况下,没有匹配的正常样品可用。在一些情况下,获得匹配的正常样本(例如,用于训练和测试本文公开的模型)。
207.肉瘤是骨骼、软骨、脂肪、肌肉、血管或其他结缔或支持组织的癌症。肉瘤包括但不限于骨癌、纤维肉瘤、软骨肉瘤、尤文氏肉瘤、恶性血管内皮瘤、恶性神经鞘瘤、双侧前庭神经鞘瘤、骨肉瘤、软组织肉瘤(例如,肺泡软部分肉瘤、血管肉瘤、叶状囊肉瘤、皮肤纤维肉瘤、硬纤维瘤、上皮样肉瘤、骨骼外骨肉瘤、纤维肉瘤、血管外皮细胞瘤、血管肉瘤、卡波西肉瘤、平滑肌肉瘤、脂肪肉瘤、淋巴管肉瘤、淋巴肉瘤、恶性纤维组织细胞瘤、神经纤维肉瘤、横纹肌肉瘤和滑膜肉瘤)。
208.癌是从上皮细胞开始的癌症,上皮细胞是覆盖身体表面、产生激素和构成腺体的细胞。通过非限制性实例的方式,癌包括乳腺癌、胰腺癌、肺癌、结肠癌、结肠直肠癌、直肠癌、肾癌、膀胱癌、胃癌、前列腺癌、肝癌、卵巢癌、脑癌、阴道癌、外阴癌、子宫癌、口腔癌、阴茎癌、睾丸癌、食道癌、皮肤癌、输卵管癌、头颈癌、胃肠道间质癌、腺癌、皮肤或眼内黑色素瘤、肛门部癌、小肠癌、内分泌系统癌、甲状腺癌、甲状旁腺癌、肾上腺癌、尿道癌、肾盂癌、输尿管癌、子宫内膜癌、子宫颈癌、垂体腺癌、中枢神经系统(cns)肿瘤、原发性cns淋巴瘤、脑干胶质瘤和脊髓轴肿瘤。癌症可以是皮肤癌,如基底细胞癌、鳞状细胞癌、黑色素瘤、非黑色素瘤或光化性(日光性)角化病。
209.癌症可以是肺癌。肺癌可以从气管分支以供应肺(支气管)或肺的小气囊(肺泡)的气道开始。肺癌包括非小细胞肺癌(nsclc)、小细胞肺癌和间皮瘤。nsclc的实例包括鳞状细胞癌、腺癌和大细胞癌。间皮瘤可能是肺内膜和胸腔(胸膜)或腹部内膜(腹膜)的癌性肿瘤。间皮瘤可能是由于石棉所致。癌症可以是脑癌,如胶质母细胞瘤。
210.癌症可以是中枢神经系统(cns)肿瘤。cns肿瘤可分为神经胶质瘤或非神经胶质瘤。胶质瘤可以是恶性胶质瘤、高级别胶质瘤、弥漫性固有脑桥胶质瘤。神经胶质瘤的实例包括星形细胞瘤、少突神经胶质瘤(或少突神经胶质瘤和星形细胞瘤成分的混合物)和室管膜瘤。星形细胞瘤包括但不限于低级别星形细胞瘤、间变性星形细胞瘤、多形性胶质母细胞瘤、毛细胞星形细胞瘤、多形性黄色星形细胞瘤和室管膜下巨细胞星形细胞瘤。少突胶质细胞瘤包括低级别少突胶质细胞瘤(或少突星形细胞瘤)和间变性少突胶质细胞瘤。非神经胶质瘤包括脑膜瘤、垂体腺瘤、原发性cns淋巴瘤和髓母细胞瘤。癌症可能是脑膜瘤。
211.白血病可以是急性淋巴细胞白血病、急性髓细胞白血病、慢性淋巴细胞白血病或慢性髓细胞白血病。其他类型的白血病包括毛细胞白血病、慢性粒单核细胞白血病和幼年粒单核细胞白血病。
212.淋巴瘤是淋巴细胞的癌症,并且可以从b或t淋巴细胞发展而来。淋巴瘤的两种主要类型是霍奇金淋巴瘤(以前称为霍奇金病)和非霍奇金淋巴瘤。霍奇金淋巴瘤的特征是存在reed-sternberg细胞。非霍奇金淋巴瘤是非霍奇金淋巴瘤的所有淋巴瘤。非霍奇金淋巴瘤可能是惰性淋巴瘤和侵袭性淋巴瘤。非霍奇金淋巴瘤包括但不限于弥漫性大b细胞淋巴瘤、滤泡性淋巴瘤、粘膜相关淋巴组织淋巴瘤(malt)、小细胞淋巴细胞淋巴瘤、套细胞淋巴瘤、burkitt淋巴瘤、纵隔大b细胞淋巴瘤、waldenstrom巨球蛋白血症、淋巴结边缘区b细胞
淋巴瘤(nmzl)、脾边缘区淋巴瘤(smzl)、结外边缘区b细胞淋巴瘤、血管内大b细胞淋巴瘤、原发性渗出性淋巴瘤和淋巴瘤样肉芽肿。
213.某些实施方案可包括基于一个或多个生物医学输出治疗和/或预防对象中的疾病或病况。一种或多种生物医学输出可以推荐一种或多种疗法。一个或多个生物医学输出可以建议、选择、指定、推荐或以其他方式确定疾病或病况的治疗和/或预防过程。一种或多种生物医学输出可建议修改或继续一种或多种疗法。修改一种或多种疗法可以包括给予、开始、减少、增加和/或终止一种或多种疗法。一种或多种疗法包括抗癌、抗病毒、抗菌、抗真菌、免疫抑制疗法或以上的组合。一种或多种疗法可以治疗、减轻或预防一种或多种疾病或适应症。
214.抗癌疗法的实例包括但不限于手术、化学疗法、放射疗法、免疫疗法/生物疗法、光动力疗法。抗癌疗法可包括化学疗法、单克隆抗体(例如,利妥昔单抗、曲妥珠单抗)、癌症疫苗(例如,治疗性疫苗、预防性疫苗)、基因疗法或以上的组合。g.系统、试剂盒和文库
215.本公开的方法可以通过系统、试剂盒、文库或以上的组合的方式来实施。本发明的方法可以包括一种或多种系统。本公开的系统可以通过试剂盒、文库或两者的方式来实现。系统可以包括一个或多个组件以执行某些实施方案的任何方法或任何步骤。例如,系统可以包括一个或多个试剂盒、装置、文库或以上的组合。系统可以包括一个或多个测序仪、处理器、存储器位置、计算机、计算机系统或以上的组合。系统可以包括传输装置。
216.试剂盒可以包括用于实施本文公开的各种操作的各种试剂,包括样品处理和/或分析操作。试剂盒可以包括用于实施至少一些本文公开的操作的说明。试剂盒可包含一种或多种捕获探针、一种或多种珠子、一种或多种标记、一种或多种接头、一种或多种装置、一种或多种试剂、一种或多种缓冲液、一个或多个样品、一个或多个数据库,或以上的组合。
217.文库可以包含一种或多种捕获探针。文库可以包含一个或多个核酸分子子集。文库可以包括一个或多个数据库。文库可以通过本文公开的任何方法、试剂盒或系统产生或生成。数据库文库可以从一个或多个数据库产生。产生一个或多个文库的方法可以包括(a)聚合来自一个或多个数据库的信息以产生聚合数据集;(b)分析聚合的数据集;和(c)从聚合的数据集生成一个或多个数据库文库。vi.计算环境
218.图10示出了用于实施本文公开的一些实施方案的计算机系统1000的实例。计算机系统1000可以具有分布式架构,其中一些组件(例如,存储器和处理器)是终端用户装置的一部分,且一些其他类似组件(例如,存储器和处理器)是计算机服务器的一部分。计算机系统1000至少包括处理器1002、存储器1004、存储装置1006、输入/输出(i/o)外围装置1008、通信外围装置1010和接口总线1012。接口总线1012配置为在计算机系统1000的各种组件之间通信、传输和传输数据、控制和命令。处理器1002可以包括一个或多个处理单元,如cpu、gpu、tpu、脉动阵列或simd处理器。存储器1004和存储装置1006包括计算机可读存储介质,如ram、rom、电可擦可编程只读存储器(eeprom)、硬盘驱动器、cd-rom、光存储装置、磁存储装置、电子非易失性计算机存储,例如存储器和其他有形存储介质。任何这样的计算机可读存储介质可以配置为存储体现本公开的方面的指令或程序代码。存储器1004和存储装置1006还包括计算机可读信号介质。计算机可读信号介质包括传播的数据信号,其中
包含计算机可读程序代码。这种传播的信号采用多种形式中的任何一种,包括但不限于电磁、光学或以上的任何组合。计算机可读信号介质包括不是计算机可读存储介质并且可以通信、传播或传输用于与计算机系统1000结合使用的程序的任何计算机可读介质。
219.此外,存储器1004包括操作系统、程序和应用程序。处理器1002配置为执行存储的指令并且包括例如逻辑处理单元、微处理器、数字信号处理器和其他处理器。存储器1004和/或处理器1002可以被虚拟化并且可以托管在例如云网络或数据中心的另一计算系统中。i/o外围装置1008包括用户界面,如键盘、屏幕(例如,触摸屏)、麦克风、扬声器、其他输入/输出装置,以及计算组件,如图形处理单元、串行端口、并行端口、通用串行总线和其他输入/输出外设。i/o外围装置1008通过耦合到接口总线1012的任何端口连接到处理器1002。通信外围装置1010配置为便于通过通信网络在计算机系统1000和其他计算装置之间进行通信,并且包括例如网络接口控制器、调制解调器、无线和有线接口卡、天线和其他通信外围装置。
220.尽管本主题已针对其特定实施方案进行了详细描述,但应当理解,本领域技术人员在获得对前述内容的理解后,可以容易地产生对这些实施方案的改变、变化和等价物。因此,应当理解,本公开是为了示例而不是限制的目的而呈现的,并且不排除包含对本主题的这样的修改、变化和/或添加,这些修改、变化和/或添加对于本领域普通技术人员来说是显而易见的。实际上,本文描述的方法和系统可以以多种其他形式体现;此外,在不背离本公开的精神的情况下,可以对本文描述的方法和系统的形式进行各种省略、替换和改变。所附权利要求及其等价物旨在覆盖落入本公开的范围和精神内的此类形式或修改。
221.除非另有明确说明,否则应了解,贯穿本说明书的讨论利用诸如“处理(processing)”、“计算(computing)”、“计算(calculating)”、“确定(determining)”和“鉴定(identifying)”等术语是指计算装置的动作或过程,如一台或多台计算机或类似的一台或多肽电子计算装置,它们在计算平台的存储器、寄存器或其他信息存储装置、传输装置或显示装置中操纵或转换表示为物理电子或磁量的数据。
222.本文讨论的一个或多个系统不限于任何特定的硬件架构或配置。计算装置可以包括提供以一个或多个输入为条件的结果的任何合适的组件布置。合适的计算装置包括访问存储的软件的基于微处理器的多用途计算系统,所述存储的软件从通用计算装置到实现本主题的一个或多个实施方案的专用计算装置对计算系统进行编程或配置。任何合适的编程、脚本或其他类型的语言或语言的组合可用于在用于编程或配置计算装置的软件中实施本文中包含的教导。
223.本文公开的方法的实施方案可以在这样的计算装置的操作中执行。以上实例中呈现的框的顺序可以改变——例如,框可以被重新排序、组合和/或分解成子框。某些框或过程可以并行执行。
224.本文使用的条件性语言,如“可能(can)”、“可以(could)”、“可能(might)”、“可以(may)”、“例如(e.g.)”等,除非另有明确说明,或在所使用的上下文中以其他方式理解,通常旨在传达某些实例包括,而其他实例不包括某些特征、元素和/或步骤。因此,这种条件性语言通常不旨在暗示一个或多个实例以任何方式需要特征、元素和/或步骤,或者一个或多个实例必须包括用于在有或没有作者输入或提示的情况下决定是否这些特征、元素和/或步骤被包括或将在任何特定实例中执行的逻辑。
225.术语“包含(comprising)”、“包括(including)”、“具有(having)”等是同义词,并且以开放式的方式包括性地使用,并且不排除另外的元素、特征、动作、操作等。此外,术语“或”以其包括性含义(且不是以其排除性含义)使用,使得例如,当用于连接元素列表时,术语“或”意指列表中的一个、一些或全部元素。本文使用的“适用于”或“配置为”意指开放和包括性的语言,它不排除适用于或配置为执行附加任务或步骤的装置。另外,“基于”的使用意味着是开放性和包括性的,因为“基于”一个或多个列举的条件或值的过程、步骤、计算或其他动作实际上可能基于另外的条件或超出所列举的值。类似地,“至少部分基于”的使用意味着开放性和包括性的,因为“至少部分基于”一个或多个列举的条件或值的过程、步骤、计算或其他动作实际上可以基于另外的条件或超出所列举的值。本文包括的标题、列表和编号仅是为了便于解释,且不意味着是限制性的。
226.上述各种特征和过程可以彼此独立地使用,或者可以以各种方式组合。所有可能的组合和子组合旨在落入本公开的范围内。另外,在一些实施方式中可以省略某些方法或过程框。本文描述的方法和过程也不限于任何特定的顺序,并且与其相关的框或状态可以以其他适当的顺序来执行。例如,所描述的框或状态可以以不同于具体公开的顺序执行,或者多个框或状态可以组合在单个框或状态中。示例性的框或状态可以串行、并行或以一些其他的方式执行。可以将框或状态添加到所公开的实例中或从所公开的实例中去除。类似地,本文描述的示例性系统和组件可以配置为与所描述的不同。例如,与所公开的实例相比,可以将元素添加、移除或重新排列。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1