一种性状相关基因的筛选方法及其系统
1.本发明涉及基因测序技术领域,更具体地,涉及一种性状相关基因的筛选方法及其系统。
背景技术:
2.利用单细胞rna测序(scrna-seq)技术识别与复杂疾病或特征相关的关键细胞亚群,对于理解复杂疾病机制至关重要。但scrna-seq数据因其高成本和低通量的特性不允许对其进行大规模测序,且目前大多数基于单细胞的研究样本不超过20个,导致统计效能有限,无法准确揭示细胞亚群中与疾病或特征相关的风险子集。此外,scrna-seq数据在基因水平上具有高稀疏性、技术噪声和方差不稳定性的特点。
3.全基因组关联研究(gwas)广泛应用于研究不同的复杂疾病或性状,将scrna-seq数据与来自大规模样本的gwas的表型相关遗传信息相关联,被认为是一种实用而有效的方法,可以在单细胞分辨率下揭示复杂疾病或性状的遗传机制。
4.将gwas与scrna-seq数据相结合用来识别与复杂疾病相关的细胞类型的方法,包括诸如ldsc-seg,magma,rolypoly,但是上述方法需要大量调整参数,以便用已知标记基因注释细胞类型,且在很大程度上忽略了每种细胞类型的内部异质性。此外,现有技术可以识别高表达水平的基因,但是其潜在缺陷是,过度关注高表达基因会低估表达水平相对较低但对揭示细胞命运很重要的基因的功能作用。
技术实现要素:
5.本发明旨在至少解决现有技术中存在的技术问题之一。为此,本发明提供一种性状相关基因的筛选方法及其系统;本发明方法通过发明基于单细胞通路的评分方法,结合scrna-seq数据和遗传关联数据推断性状相关的基因、细胞等,从深层次挖掘隐含在单细胞数据背后的生命规律,解决相关的生命科学问题。
6.本技术公开一种性状相关基因的筛选方法,包括:
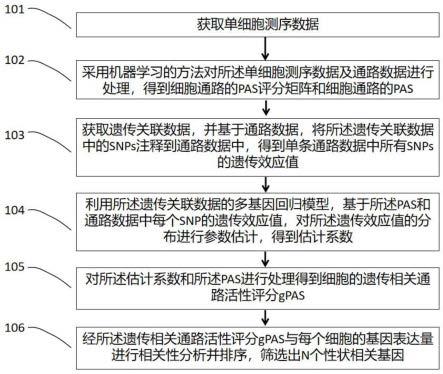
7.获取单细胞测序数据;
8.采用机器学习的方法对所述单细胞测序数据及通路数据进行处理,得到细胞通路的pas评分矩阵和细胞通路的pas;
9.获取遗传关联数据,并基于通路数据,将所述遗传关联数据中的snps注释到通路数据中,得到单条通路数据中所有snps的遗传效应值;
10.利用所述遗传关联数据的多基因回归模型,基于所述pas和通路数据中每个snp的遗传效应值,对所述遗传效应值的分布进行参数估计,得到估计系数;
11.对所述估计系数和所述pas进行处理得到细胞的遗传相关通路活性评分gpas;
12.对所述遗传相关通路活性评分gpas与每个细胞的基因表达量进行相关性分析并排序,筛选出n个性状相关基因。
13.所述遗传效应值的获取公式为:其中,β表示m个snps的理论效应大
小向量,ε表示随机环境误差,r代表ld矩阵,x
t
表示遗传关联数据样本中snps的标准基因型;
14.可选的,所述估计系数的获取方式包括:
15.其中,τ
i,j
表示细胞j中的通路i的估计系数,τ0表示截距项,σ2表示通路中snp效应大小的方差,表示加权pas;
16.所述对所述估计系数和所述pas进行处理得到细胞的遗传相关通路活性评分gpas的步骤包括:将所述估计系数乘以所述pas再求和得到细胞的遗传相关通路活性评分gpas;
17.所述遗传相关通路活性评分gpas(gpj)的获取方式为:
18.其中,gpj为遗传相关通路活性评分gpas,所述为优化后的估计系数;
19.所述采用机器学习的方法对所述单细胞测序数据及通路数据进行处理,得到细胞通路的pas评分矩阵和细胞通路的pas的获取步骤包括:
20.获取单细胞测序数据和通路数据;
21.对所述单细胞测序数据中的基因-细胞矩阵进行标准化处理,得到经标准化处理后的基因-细胞矩阵;
22.基于所述通路数据,利用机器学习的方法将所述经标准化处理后的基因-细胞矩阵转换为通路-细胞矩阵,利用通路-细胞矩阵得到细胞通路的pas评分矩阵,pas评分矩阵包括单条通路中单个细胞的通路活性评分pas;
23.可选的,对所述通路活性评分pas进行优化处理,得到所述加权pas;
24.所述加权pas的获取方式包括:
25.其中,表示加权pas,表示优化后的细胞i中基因g的标准化表达,s
i,j
表示细胞j通路i的通路活性评分pas;
26.可选的,所述的获取方式包括:
[0027][0028]
可选的,所述机器学习的方法包括奇异值分解svd的方法。
[0029]
将所述遗传关联数据中的snp注释到通路数据中的步骤包括:
[0030]
从所述遗传关联数据中进行筛选得到单个基因的snps,基于所述通路数据,将所述单个基因的snps映射到对应通路中,得到带有snps注释的通路;
[0031]
可选的,所述单个基因的snps的获取步骤包括:获取所述遗传关联数据中基因的snps后,分别对snps基因对进行分配,得到分配结果;
[0032]
将分配结果中数个单个snps对应多个基因的重复基因分别作为独立的snp基因关联处理;保留分配结果中的次要等位基因频率(maf)大于0.1的snps;删除性染色体上的snps;得到所述单个基因的snps;
[0033]
将所述单个基因的snps汇总后即为所有基因的snps。
[0034]
所述方法还包括:根据所述n个性状相关基因计算每个细胞的性状相关分数trs;
可选的,利用细胞评分方法计算所述n个性状相关基因的性状相关分数trs。
[0035]
可选的,对所述遗传相关通路活性评分gpas与每个细胞的基因表达量进行相关性分析并排序的方法包括:通过皮尔逊相关系数(pcc)确定单个基因的表达与所述gpas之间的相关性,根据相关性对基因进行排序,得到所述n个性状相关基因;
[0036]
可选的,所述n个性状相关基因为按照相关性降序或者升序规则进行排序后的前1000或后1000个性状相关基因。
[0037]
一种应用,所述应用包括以下任一项:
[0038]
基于每个细胞的性状相关分数trs和每个细胞的水平p值,得到性状相关细胞;可选的,基于区块拔靴法block bootstrap method得到性状相关细胞类型或亚群;可选的,对所述遗传相关通路活性评分gpas进行排序,根据排序结果和细胞类型水平上通路的p值,得到性状相关通路;
[0039]
一种性状相关基因的筛选设备,所述设备包括:存储器和处理器;
[0040]
所述存储器用于存储程序指令;所述处理器用于调用程序指令,当程序指令被执行时,用于执行上述的性状相关基因的筛选方法。
[0041]
一种性状相关基因的筛选系统,包括:
[0042]
获取单元,用于获取单细胞测序数据;
[0043]
第一处理单元,用于采用机器学习的方法对所述单细胞测序数据及通路数据进行处理,得到细胞通路的pas评分矩阵和细胞通路的pas;
[0044]
第二处理单元,用于获取遗传关联数据,并基于通路数据,将所述遗传关联数据中的snps注释到通路数据中,得到单条通路数据中所有snps的遗传效应值;
[0045]
第三处理单元,用于利用所述遗传关联数据的多基因回归模型,基于所述pas和通路数据中每个snp的遗传效应值,对所述遗传效应值的分布进行参数估计,得到估计系数;
[0046]
第四处理单元,用于对所述估计系数和所述pas进行处理得到细胞的遗传相关通路活性评分gpas;
[0047]
第五处理单元,用于对所述遗传相关通路活性评分gpas与每个细胞的基因表达量进行相关性分析并排序,筛选出n个性状相关基因。
[0048]
一种计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现上述的性状相关基因的筛选方法。
[0049]
本技术具有以下有益效果:
[0050]
1、本技术创新性的公开一种基于单细胞通路的评分方法,采用多基因回归模型,通过利用通路活性转化的scrna-seq数据和遗传关联研究数据揭示与性状相关的基因、细胞亚群;有效克服了目前与复杂疾病多基因风险相关的基因、细胞亚群的鉴定在很大程度上受到scrna-seq数据中样本量小和高度稀疏性的阻碍,导致统计效能有限,无法准确揭示细胞亚群中与疾病或性状相关的风险子集的问题。该方法从深层次挖掘隐含在单细胞测序数据背后的生命规律,从群体遗传学突变与疾病关系和单细胞测序基因丰度信息等多个维度深度分析,大大提高数据分析的精度和深度。
[0051]
2、本技术基于大规模模拟和真实数据,利用上述评分方法将scrna-seq数据和遗传关联数据相结合,可以有效克服现有技术中为了方便用已知标记基因注释细胞类型需要大量调整参数,且在很大程度上会忽略每种细胞类型的内部异质性的问题;不会存在因过
度关注高表达基因而低估表达水平相对较低但对揭示细胞命运很重要的基因的功能作用,有助于通过聚集平均表达水平较低的基因的作用来识别疾病相关的早期发育事件或祖细胞,例如与细胞发育相关的关键转录因子;同时可以有效降低scrna-seq数据的稀疏性和技术噪声,并在识别特征相关的细胞类型和亚群方面表现出很好的稳健性和能力。
[0052]
3、本技术创新性的公开了基于单细胞评分通路的性状相关基因的筛选方法,融合了参与相同生物通路的不同基因的功能作用以获得稳定的细胞状态,显著增加了统计效能、生物学可解释性和结果可重复性;克服了已知注释细胞类型的限制,并可能发现新的遗传相关亚群和细胞类型的关键基因或通路,应用广泛,实用性强。
附图说明
[0053]
为了更清楚地说明本发明实施例中的技术方案,下面将对实施例描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获取其他的附图。
[0054]
图1是本发明实施例提供的性状相关基因的筛选方法的分析示意流程图;
[0055]
图2是本发明实施例提供的性状相关基因的筛选设备示意图;
[0056]
图3是本发明实施例提供的性状相关基因的筛选系统示意流程图;
[0057]
图4是本发明实施例提供的基于单细胞通路的评分方法获得gpas,以及利用gpas输出trs、性状相关基因、性状相关细胞、性状相关细胞类型/亚群、性状型管通路的概述图。
具体实施方式
[0058]
为了使本技术领域的人员更好地理解本发明方案,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述。
[0059]
在本发明的说明书和权利要求书及上述附图中的描述的一些流程中,包含了按照特定顺序出现的多个操作,但是应该清楚了解,这些操作可以不按照其在本文中出现的顺序来执行或并行执行,操作的序号如101、102等,仅仅是用于区分开各个不同的操作,序号本身不代表任何的执行顺序。另外,这些流程可以包括更多或更少的操作,并且这些操作可以按顺序执行或并行执行。需要说明的是,本文中的“第一”、“第二”等描述,是用于区分不同的消息、设备、模块等,不代表先后顺序,也不限定“第一”和“第二”是不同的类型。
[0060]
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域技术人员在没有做出创造性劳动前提下所获取的所有其他实施例,都属于本发明保护的范围。
[0061]
图1是本发明实施例提供的一种性状相关基因的筛选方法示意流程图,具体地,所述方法包括如下步骤:
[0062]
101:获取单细胞测序数据;
[0063]
在一个实施例中,单细胞测序数据包括七个独立的单细胞rna-seq(scrna-seq)或单核rna-seq(snrna-seq)数据集,涵盖来自人类(homo sapiens)和小鼠(mus musculus)的139万个细胞。针对血细胞,收集了两个基于人类bmmc(n=35,582个细胞)和人类pbmc(n=
97,039个细胞)的scrna-seq数据集,以揭示性状相关的细胞亚群或类型。对于与免疫/代谢相关的疾病/特征,利用来自人类细胞的scrna-seq数据集(hcl,n=35个成人组织中的513,707个细胞)为每个组织构建一个伪组织(psudo-bulk)表达谱和与疾病/特征相关的优先风险组织。
[0064]
在一个实施例中,对于脑相关疾病,收集了三个单细胞数据集:小鼠脑scrnaseq数据集(n=160,796个细胞)、人脑内嗅皮质snrna-seq数据集(n=11,786个细胞)和包含内嗅皮层和体感皮层两个区域的人类大脑snrna-seq数据集(n=101,906个细胞)。
[0065]
在一个实施例中,为了发现与重症covid-19相关的免疫细胞群,收集了大规模的pbmc scrna-seq数据集(n=469,453个细胞),其中包含254个具有不同covid-19严重程度的外周血样本(轻度n=109个样本,中度n=102个样本,重度n=50)和16个健康对照。
[0066]
102:采用机器学习的方法对单细胞测序数据及通路数据进行处理,得到细胞通路的pas评分矩阵和细胞通路的pas;
[0067]
在一个实施例中,采用机器学习的方法对单细胞测序数据及通路数据进行处理,得到细胞通路的pas评分矩阵和细胞通路的pas的获取步骤包括:
[0068]
获取单细胞测序数据和通路数据;
[0069]
对单细胞测序数据中的基因-细胞矩阵进行标准化处理,得到经标准化处理后的基因-细胞矩阵;具体地,使用比例因子为10,000的方差稳定化变换参数将scrna-seq数据中的稀疏基因-细胞矩阵进行标准化,得到在单个细胞中单个基因的标准化表达;标准化的公式为:其中,a
g,j
表示细胞j中基因g的原始表达,e
g,j
表示细胞j中基因g的标准化表达;
[0070]
基于通路数据,利用机器学习的方法将经标准化处理后的基因-细胞矩阵转换为通路-细胞矩阵,利用通路-细胞矩阵得到细胞通路的pas评分矩阵,pas评分矩阵包括单条通路中单个细胞的通路活性评分pas;
[0071]
在一个实施例中,通路数据为kegg通路数据,来自kegg数据库的通路作为评估pas的默认基因集,利用奇异值分解svd的方法将经标准化处理后的基因-细胞矩阵转换为通路-细胞矩阵;使用pi表示通路i中的基因集,对于每条通路i,从标准化的基因-细胞矩阵a选择矩阵ai,其中矩阵ai的列是所有n个细胞,行是通路基因集pi中|pi|基因,根据svd得到的公式,其中,u表示n
×
n正交矩阵,σ表示除主对角线元素外具有全零的对角矩阵,v
t
表示|pi|
×
|pi|正交矩阵;对于右正交矩阵v=(v1,v2,...,v
|pi|
),第t列向量v
t
表示第t主成分,反映了单细胞数据中基因在通路中的协同表达变异性;由于第一主成分pc1代表最大的方差变异,因此细胞j特征在pc1的投影代表了通路i的pass
i,j
;对于细胞j,利用通路i中所有的表达方差作为权重调整原始pass
i,j
;对于通路i中的基因g,使用最小-最大值缩放法重新调整基因表达e
g,j
调整后的基因表达为
[0072]
在一个实施例中,对通路活性评分pas进行优化处理,得到加权pas;
[0073]
加权pas的获取方式包括:
[0074]
其中,表示加权pas,表示优化后的细胞i中基因g的标准化表达,s
i,j
表示细
胞j通路i的通路活性评分pas;
[0075]
在一个实施例中,的获取方式包括:
[0076][0077]
其中,表示细胞i中基因g的标准化表达,max(e
g,j
)表示通路i中基因表达最大值,min(e
g,j
)表示通路i中基因表达最小值。
[0078]
可选的,机器学习的方法包括奇异值分解svd的方法;奇异值分解svd方法大大提高了分析稀疏矩阵的计算效率,且可以在不计算方差矩阵的情况下获得特征值;利用奇异值分解方法将标准化的基因-细胞矩阵换华为低维空间的通路-细胞矩阵。
[0079]
103:获取遗传关联数据,并基于通路数据,将遗传关联数据中的snps注释到通路数据中,得到单条通路数据中所有snps的遗传效应值;
[0080]
在一个实施例中,将遗传关联数据中的snp注释到通路数据中的步骤包括:
[0081]
从遗传关联数据中进行筛选得到单个基因的snps,基于通路数据,将单个基因的snps映射到对应通路中,得到带有snps注释的通路;
[0082]
可选的,单个基因的snps的获取步骤包括:获取遗传关联数据中基因的snps后,分别对snps基因对进行分配,得到分配结果;
[0083]
将分配结果中数个单个snps对应多个基因的重复基因分别作为独立的snp基因关联处理;保留分配结果中的次要等位基因频率(maf)大于0.1的snps;删除性染色体上的snps;得到单个基因的snps;
[0084]
将单个基因的snps汇总后即为所有基因的snps。具体地,遗传关联数据为gwas数据,以20kb作为默认参数将gwas汇总统计数据中的snp分配给相关基因;使用符号g(k)表示带有snp k的基因g,通过snp基因对的分配,存在数个单个snps对应着多个基因;由于整个过程需要从成千上万个snp中推断参数,但上述单个snps对应着多个基因的snps对推断过程无作用,因此需要将上述重复基因作为独立的snp基因关联处理;保留次要等位基因频率(maf)大于0.1的snps,删除性染色体上的snps,最终得到相关基因的snps;
[0085]
基于kegg数据库中的通路,将具有关联snps的基因注释到通路中,并使用si=公式2表示通路i中的snps集合;利用千人基因组项目第3阶段数据对gwas汇总数据提取的snps计算连锁不平衡ld(linkage disequilibrium);本方案提供了例如go、reactome、和msigdb的功能基因集合作为替换选项。另外,删除存在广泛性ld的主要组织相容性复合物区域chr6:25-35mbp。
[0086]
在一个实施例中,gwas数据已给定表型,给定表型的表型注释包括二分法,连续依赖性特征或者内表型和中心测量。
[0087]
104:利用遗传关联数据的多基因回归模型,基于pas和通路数据中每个snp的遗传效应值,对遗传效应值的分布进行参数估计,得到估计系数;
[0088]
在一个实施例中,遗传效应值的获取公式为:其中,β表示m个snps的理论效应大小向量,ε表示随机环境误差,r代表ld矩阵,x
t
表示遗传关联数据样本中snps的标准基因型;
[0089]
在一个实施例中,si表示每条通路i的定位基因中包含所有snps的snp集合,多基
因模型假设先验通路i的所有snps的效应大小遵循多变量正态分布,其中σ2表示通路中snps效应大小的方差,i表示|si|
×
|si|单位矩阵;
[0090]
在一个实施例中,估计系数的获取方式包括:
[0091]
其中,τ
i,j
表示细胞j中的通路i的估计系数,估计系数反映了细胞特异性pas对gwas效应大小方差的影响,即遗传对反应的影响;τ0表示截距项,σ2表示通路中snp效应大小的方差,表示加权pas;
[0092]
在一个实施例中,基于先前假设,对遗传效应值的分布进行估计,采用的公式如下:利用此公式对估计系数进行优化;
[0093]
在一个实施例中,为了优化多基因回归模型中每条通路的估计系数,采用能够显著提高计算效率和估计一致收敛性的矩量法(method-of-moments approach)对多基因回归模型进行优化;然后,拟合与每条通路相关的snps的观察和预期平方效应,并通过如下公式估计预期值:其中,tr代表矩阵轨迹。
[0094]
105:对估计系数和pas进行处理得到细胞的遗传相关通路活性评分gpas;
[0095]
在一个实施例中,对估计系数和pas进行处理得到细胞的遗传相关通路活性评分gpas的步骤包括:将估计系数乘以pas再求和得到细胞的遗传相关通路活性评分gpas;
[0096]
遗传相关通路活性评分gpas(gpj)的获取方式为:
[0097][0098]
其中,为优化后的估计系数;
[0099]
106:对遗传相关通路活性评分gpas与每个细胞的基因表达量进行相关性分析并排序,筛选出n个性状相关基因。
[0100]
可选的,对遗传相关通路活性评分gpas与每个细胞的基因表达量进行相关性分析并排序的方法包括:通过皮尔逊相关系数(pcc)确定单个基因的表达与gpas总分之间的相关性,根据相关性对基因进行排序,得到n个性状相关基因;具体地,为了最大限度提高功效,每个基因g的表达都由其基因特定的技术噪声水平反向加权,该噪声水平通过在scrna-seq数据中建模基因间的平均方差关系估计;
[0101]
可选的,n个性状相关基因为按照相关性降序或者升序规则进行排序后的前1000或后1000个性状相关基因;n不限于1000,n为自然数整数。
[0102]
在一个实施例中,方法还包括:根据n个性状相关基因计算每个细胞的性状相关分数trs;可选的,利用seurat中的addmodulescore函数的细胞评分方法计算n个基因的性状相关分数trs;每个细胞的性状相关分数trs的获取公式为:trs=average re(gs)-average re(cg);其中,average re(gs)为给定细胞中n个性状相关基因集的平均相对表达值,average re(cg)为从现有基因库随机抽取的相同数量的对照基因集的平均相对表达值;re为relative expression;gs为gene set;cg为control gene set;
[0103]
在一个实施例中,通过计算性状相关基因的等级分布确定单个细胞的统计显著性,以进一步评估细胞是否与感兴趣的形状显著相关;具体地,得出性状相关基因在细胞中
的百分比等级,其中,r
g,j
表示细胞j中基因g的表达等级,g代表指定性状相关基因的数量;基因百分比等级遵循正态分布u(0,1),在基因的百分比等级之间无关联的无效假设下,获得每个细胞的统计值tj,获得公式如下:
[0104]
基于单细胞数据中的大量细胞,使用中心极限定理推导tj的分布:其中n是细胞总数;显著性检验的假设为:h0:tj=0 vs h1:tj>0;每个细胞j的p值为:pj=pr(tj≤t)。
[0105]
一种应用,应用包括以下任一项:
[0106]
基于每个细胞的性状相关分数trs和每个细胞的水平p值(代表单个细胞水平是否相关),得到性状相关细胞;
[0107]
可选的,基于区块拔靴法block bootstrap method得到性状相关细胞类型或亚群(明确单个细胞所属细胞类型是否相关);具体地,将一组细胞视为伪组织(psudo-bulk)转录组谱,并平均化给定的细胞类型内跨细胞的基因表达量;对于关联的细胞类型,用block bootstrap method估计标准误差并计算每个细胞类型对应p值的t统计值。鉴于区块拔靴法的目标是在从经验分布采样时保持数据结构,利用kegg数据库的通路将基因组划分为多个生物学意义的块,并对上述基于通路的块进行替换取样。在默认参数下,为每个细胞类型关联分析执行200次块引导迭代,具体执行时可修改默认参数。
[0108]
可选的,对遗传相关通路活性评分gpas进行排序,根据排序结果(选取排序结果中排名靠前的通路)和细胞类型水平上通路的p值,得到性状相关通路;具体地,基于中心极限定理对gpas进行排序;使用符号c
t
表示细胞类型t,利用如下公式计算c
t
内每个细胞j的通路百分比等级:其中,表示细胞j中通路i的gpas等级,m表示通路总数;类似地,利用如下公式计算细胞类型t中每条通路i的统计重要值t
it
:其中,假设为:h0:t
it
=0 vs h1:t
it
>0;细胞类型t中每条通路i的p值为:
[0109]
图2是本发明实施例提供的一种性状相关基因的筛选设备示意流程图,设备包括:存储器和处理器;
[0110]
存储器用于存储程序指令;处理器用于调用程序指令,当程序指令被执行时,用于执行上述的性状相关基因的筛选方法。
[0111]
图3是本发明实施例提供的一种性状相关基因的筛选系统示意流程图,包括:
[0112]
获取单元301,用于获取单细胞测序数据;
[0113]
第一处理单元302,用于采用机器学习的方法对单细胞测序数据及通路数据进行
处理,得到细胞通路的pas评分矩阵和细胞通路的pas;
[0114]
第二处理单元303,用于获取遗传关联数据,并基于通路数据,将遗传关联数据中的snps注释到通路数据中,得到单条通路数据中所有snps的遗传效应值;
[0115]
第三处理单元304,用于利用遗传关联数据的多基因回归模型,基于pas和通路数据中每个snp的遗传效应值,对遗传效应值的分布进行参数估计,得到估计系数;
[0116]
第四处理单元305,用于对估计系数和pas进行处理得到细胞的遗传相关通路活性评分gpas;
[0117]
第五处理单元306,用于对遗传相关通路活性评分gpas与每个细胞的基因表达量进行相关性分析并排序,筛选出n个性状相关基因。
[0118]
一种计算机可读存储介质,其上存储有计算机程序,计算机程序被处理器执行时实现上述的性状相关基因的筛选方法。
[0119]
图4是本发明实施例提供的基于单细胞通路的评分方法获得gpas,以及利用gpas输出trs、性状相关基因、性状相关细胞、性状相关细胞类型/亚群、性状相关通路的概述图;
[0120]
其中,a表示利用奇异值分解的方法将基因-细胞矩阵转换为通路-细胞矩阵,pc1表示每条通路的pas;b表示将gwas数据中的snp注释到对应通路中;c表示多基因回归模型;其中,位于顶部的图表示利用多基因回归模型推断每条通路中的估计系数,再使用估计系数和相应的pas进行计算得到gpas,位于底部的图表示pearson相关模型,用于将每个细胞的gpas与所有单个细胞的基因相关联,以便对性状相关基因进行排名;利用seurat中的addmodulescore函数得到前n个性状相关基因(默认前1000个)。以计算每个细胞的性状相关分数trs;d表示输出,分别包括四个输出:性状相关细胞、性状相关细胞类型、性状相关通路和性状相关基因。
[0121]
本验证实施例的验证结果表明,为适应症分配固有权重相对于默认设置来说可以适度改善本方法的性能。
[0122]
所属领域的技术人员可以清楚地了解到,为描述的方便和简洁,上述描述的系统,装置和单元的具体工作过程,可以参考前述方法实施例中的对应过程,在此不再赘述。
[0123]
在本技术所提供的几个实施例中,应该理解到,所揭露的系统,装置和方法,可以通过其它的方式实现。例如,以上所描述的装置实施例仅仅是示意性的,例如,所述单元的划分,仅仅为一种逻辑功能划分,实际实现时可以有另外的划分方式,例如多个单元或组件可以结合或者可以集成到另一个系统,或一些特征可以忽略,或不执行。另一点,所显示或讨论的相互之间的耦合或直接耦合或通信连接可以是通过一些接口,装置或单元的间接耦合或通信连接,可以是电性,机械或其它的形式。
[0124]
所述作为分离部件说明的单元可以是或者也可以不是物理上分开的,作为单元显示的部件可以是或者也可以不是物理单元,即可以位于一个地方,或者也可以分布到多个网络单元上。可以根据实际的需要选择其中的部分或者全部单元来实现本实施例方案的目的。
[0125]
另外,在本发明各个实施例中的各功能单元可以集成在一个处理单元中,也可以是各个单元单独物理存在,也可以两个或两个以上单元集成在一个单元中。上述集成的单元既可以采用硬件的形式实现,也可以采用软件功能单元的形式实现。
[0126]
本领域普通技术人员可以理解上述实施例的各种方法中的全部或部分步骤是可
以通过程序来指令相关的硬件来完成,该程序可以存储于一计算机可读存储介质中,存储介质可以包括:只读存储器(rom,read only memory)、随机存取存储器(ram,random access memory)、磁盘或光盘等。
[0127]
本领域普通技术人员可以理解实现上述实施例方法中的全部或部分步骤是可以通过程序来指令相关的硬件完成,所述的程序可以存储于一种计算机可读存储介质中,上述提到的存储介质可以是只读存储器,磁盘或光盘等。
[0128]
以上对本发明所提供的一种计算机设备进行了详细介绍,对于本领域的一般技术人员,依据本发明实施例的思想,在具体实施方式及应用范围上均会有改变之处,综上所述,本说明书内容不应理解为对本发明的限制。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1