用于治疗与诊断胃癌转移的生物标志物及其识别方法与流程
1.本发明涉及中医药与生物技术领域,具体涉及用于治疗与诊断胃癌转移的生物标志物及其识别方法。
背景技术:
2.胃癌是起源于胃黏膜上皮的恶性肿瘤,全球常见的恶性肿瘤之一,是严重危及人类健康的一类疾病。胃癌存在着特异的分子亚型与临床表现,虽然其早期治愈率很高,但由于大部分胃癌早期症状不明显,患者就诊时往往已处于中晚期,生存预后情况也不容乐观,临床上常用的胃癌生物标志物特异性并不高,在不同肿瘤、年龄、性别的患者间均有较大的差异,不利于诊断与治疗。其中导致胃癌患者死亡的最主要原因是胃癌转移,目前肿瘤转移是全世界科学家正在重点携手攻克的难题之一,由于其涉及的“侵袭-转移”级联机制极为复杂,参与该过程的靶点与通路尚未明确,因此寻找与胃癌转移相关的生物标志物,有利于胃癌诊断生物标志物的应用手册制定与靶向药物的设计,具有较大的现实意义和学术意义。
技术实现要素:
3.为此,本发明提供用于治疗与诊断胃癌转移的生物标志物及其识别方法,以实现与胃癌转移相关的生物标志物的识别,为胃癌临床上研发诊断胃癌转移的方法和治疗胃癌转移的药物提供新的分子靶标。
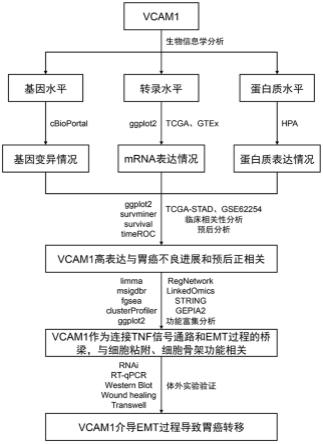
4.为了实现上述目的,本发明提供如下技术方案:根据本发明实施例的第一方面,提出了生物标志物在制备诊断和治疗胃癌转移的产品中的应用,所述生物标志物为参与上皮细胞向运动间质细胞转化过程的血管细胞粘附分子vcam1。
5.进一步地,所述产品包括治疗胃癌转移的药物组合物、诊断胃癌转移的试剂盒。
6.根据本发明实施例的第二方面,提出了一种胃癌转移相关的生物标志物的识别方法,所述方法包括:从tcga、geo和gtex数据库中获得胃癌患者的肿瘤组织、癌旁组织与正常组织的全转录组测序数据集;结合所述全转录组测序数据集,应用r语言与多个数据库对血管细胞粘附分子vcam1进行基因水平、转录水平、蛋白质水平的系统性生物信息学分析,所述生物信息学分析包括基因突变情况分析、mrna表达情况分析、蛋白质表达情况分析、预后分析、临床相关性分析、功能富集分析,根据生物信息学分析结果判断血管细胞粘附分子vcam1是否与胃癌转移相关;若判断血管细胞粘附分子vcam1与胃癌转移相关,则通过特定体外实验对结果进行验证。
7.进一步地,所述全转录组测序数据集,具体包括:
从tcga、geo和gtex数据库获得全转录组测序数据集,包括tcga-stad数据集、gtex-stomach数据集、gse62254数据集,tcga-stad数据集含有多个胃癌组织样本和多个癌旁组织样本,gtex-stomach数据集含有多个正常胃组织样本,gse62254数据集含有多个胃癌组织样本。
8.进一步地,所述生物信息学分析具体包括:通过用于癌症基因组学的cbioportal数据库对vcam1在胃癌患者中的基因突变情况进行分析,分析vcam1的基因突变情况及与患者临床信息的相关性;应用r软件中的“ggplot2”包,结合全转录组数据集中的表达谱矩阵和患者临床信息数据对胃癌组织与癌旁组织、正常组织中的vcam1进行mrna表达情况分析;通过人类蛋白图谱hpa对vcam1在胃癌组织与正常胃组织样本中的蛋白质表达水平进行分析。
9.进一步地,所述生物信息学分析具体还包括:应用r软件中的“survminer”、“survival”、“timeroc”、“ggplot2”包对tcga-stad数据集中vcam1的表达与胃癌患者的预后情况及临床信息相关性进行分析及可视化,并用gse62254数据集进行外部验证;将tcga-stad数据集的患者按照vcam1表达量的中位数进行1:1高低分组并根据患者的总生存期进行生存预测,获取vcam1的表达与胃癌患者1、3、5年的生存率auc面积结果;将vcam1的表达量与胃癌患者临床信息进行单因素cox、多因素cox回归分析,获取vcam1在胃癌患者中的预后分析结果。
10.进一步地,所述生物信息学分析具体还包括:应用r软件中的“limma”、“msigdbr”、“fgsea”、“clusterprofiler”、“ggplot2”包对vcam1表达量高低分组所得的患者分组表达谱矩阵进行差异表达分析,然后进行基因集富集分析并进行可视化;利用表达谱矩阵对vcam1与上皮标志物、间质标志物以及诱导上皮-间质转化过程的转录因子进行相关性分析;通过regnetwork调节网络数据库下载计算机预测或实验验证的基因及其基因调节子信息,筛选获得vcam1的基因调节子,包括转录因子和mirna两种,应用cytoscape软件绘制基因调节网络,获取直接调控vcam1的转录因子,结合富集分析结果获取vcam1介导胃癌转移的可能机制。
11.进一步地,所述生物信息学分析具体还包括:通过linkedomics链接域数据库下载tcga-stad数据集表达谱矩阵分析得到的与vcam1相关的基因集及相关系数数据,选择与vcam1正相关和负相关的前多个基因,并应用r软件中的“clusterprofiler”、“ggplot2”包对得到的基因进行go、kegg功能富集分析,结合富集分析结果获取vcam1介导胃癌转移的可能机制。
12.进一步地,所述生物信息学分析具体还包括:通过string弦数据库查找与vcam1有相互作用关系的蛋白质,并应用r软件中的“clusterprofiler”、“ggplot2”包对得到的蛋白质进行go、kegg功能富集分析,结合富集分析结果获取vcam1介导胃癌转移的可能机制;通过gepia2基因表达谱交互分析数据库对得到的有相互作用关系的蛋白质进行
表达量和预后分析,获取蛋白质在胃癌组织、癌旁组织、正常组织中的表达情况,结合蛋白质表达情况及胃癌患者生存期获取胃癌患者的风险蛋白质因素。
13.进一步地,若判断vcam1与胃癌转移相关,则通过特定体外实验对结果进行验证,具体包括:通过rt-qpcr、western blot实验检测vcam1在不同胃癌细胞系和胃正常细胞系中的mrna和蛋白质表达量;通过rnai、woundhealing、transwell实验检测vcam1敲低后胃癌细胞系的迁移、侵袭能力;通过rt-qpcr、western blot实验检测vcam1敲低后上皮-间质转化过程中的上皮标志物、间质标志物表达情况变化。
14.本发明具有如下优点:本发明提出的一种胃癌转移相关的生物标志物的识别方法,所述生物标志物为参与上皮细胞向运动间质细胞转化过程的血管细胞粘附分子vcam1,从数据库中获得胃癌患者的肿瘤组织、癌旁组织与正常组织的全转录组测序数据集;结合所述全转录组测序数据集,应用r语言与多个数据库对vcam1进行基因水平、转录水平、蛋白质水平的系统性生物信息学分析,根据生物信息学分析结果判断vcam1是否与胃癌转移相关;若判断vcam1与胃癌转移相关,则通过特定体外实验对结果进行验证。可实现与胃癌转移相关的生物标志物的识别,为胃癌临床上研发诊断胃癌转移的方法和治疗胃癌转移的药物提供了新的分子靶标。
附图说明
15.为了更清楚地说明本发明的实施方式或现有技术中的技术方案,下面将对实施方式或现有技术描述中所需要使用的附图作简单地介绍。显而易见地,下面描述中的附图仅仅是示例性的,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据提供的附图引伸获得其它的实施附图。
16.本说明书所绘示的结构、比例、大小等,均仅用以配合说明书所揭示的内容,以供熟悉此技术的人士了解与阅读,并非用以限定本发明可实施的限定条件,故不具技术上的实质意义,任何结构的修饰、比例关系的改变或大小的调整,在不影响本发明所能产生的功效及所能达成的目的下,均应仍落在本发明所揭示的技术内容得能涵盖的范围内。
17.图1为本发明实施例提供的一种胃癌转移相关的生物标志物的识别方法的流程示意图;图2为本发明实施例提供的一种胃癌转移相关的生物标志物的识别方法的具体实施流程示意图。
具体实施方式
18.以下由特定的具体实施例说明本发明的实施方式,熟悉此技术的人士可由本说明书所揭露的内容轻易地了解本发明的其他优点及功效,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
19.本发明实施例提供生物标志物在制备诊断和治疗胃癌转移的产品中的应用,生物标志物为参与上皮细胞向运动间质细胞转化过程的血管细胞粘附分子vcam1。本实施例中,产品包括治疗胃癌转移的药物组合物、诊断胃癌转移的试剂盒本发明实施例提供一种胃癌转移相关的生物标志物的识别方法,如图1和图2所示,该方法包括:s100、从tcga、geo和gtex数据库中获得胃癌患者的肿瘤组织、癌旁组织与正常组织的全转录组测序数据集;s200、结合所述全转录组测序数据集,应用r语言与多个数据库对血管细胞粘附分子vcam1进行基因水平、转录水平、蛋白质水平的系统性生物信息学分析,所述生物信息学分析包括基因突变情况分析、mrna表达情况分析、蛋白质表达情况分析、预后分析、临床相关性分析、功能富集分析,根据生物信息学分析结果判断血管细胞粘附分子vcam1是否与胃癌转移相关;s300、若判断血管细胞粘附分子vcam1与胃癌转移相关,则通过特定体外实验对结果进行验证。
20.应用生物信息学,可实现与胃癌转移相关的生物标志物的识别,为胃癌临床上研发诊断胃癌转移的方法和治疗胃癌转移的药物提供了新的分子靶标。具体实施内容如下:通过文献检索,上皮-间质转化,即上皮细胞向运动间质细胞的转分化过程,近年来研究表明上皮-间质转化在上皮来源恶性肿瘤中有助于癌细胞的转移,其中主要的参与者是细胞粘附分子。本发明实施例提出的胃癌转移相关的生物标志物vcam1的识别方法,根据生物信息学分析结果判断vcam1是否与胃癌转移相关,并可获知其具体参与胃癌转移的机制,以及是否有应用于临床的潜质进行评估。
21.从tcga、geo和gtex数据库获得胃癌患者的肿瘤组织、癌旁组织与正常组织的全转录组测序数据集,纳入的数据集需满足以下条件:1)使用人类组织样本,包括:人类正常组织样本,肿瘤组织样本和癌旁组织样本;2)每个数据集至少包含5个样本。全转录组测序数据集包括tcga-stad、gtex-stomach、gse62254。全转录组测序数据集tcga-stad含有375个胃癌组织样本和32个癌旁组织样本,gtex-stomach含有217个正常胃组织样本,gse62254含有300个胃癌组织样本。
22.通过用于癌症基因组学的cbioportal数据库(cbioportal,http://www.cbioportal.org/)对vcam1在胃癌患者中的基因突变情况进行分析,选择“stomach adenocarcinoma (tcga, nature 2014)”样本分析vcam1的基因突变情况及与患者临床信息的相关性,“oncoprint”模块结果表明vcam1的基因改变和过度表达与胃癌患者的tnm分期和不良预后呈正相关关系。
23.应用r软件中的“ggplot2”包,结合全转录组数据集中的表达谱矩阵和患者临床信息数据对胃癌组织与癌旁组织、正常组织中的vcam1进行mrna表达分析,结果显示vcam1在胃癌组织中高表达,在癌旁组织和正常组织中低表达,且vcam1的mrna高表达与不良病理阶段、组织学分级、t分期、n分期正相关。
24.通过人类蛋白图谱(hpa,https://www.proteinatlas.org/)对vcam1在胃癌组织与正常胃组织样本中的蛋白质表达水平进行分析,通过vcam1搜索“tissue”和“pathology”模块中胃癌组织和正常组织的免疫组化图片,免疫组化染色分析结果证明vcam1在胃癌组
织较正常胃组织更深,即vcam1的蛋白质表达水平在胃癌组织中更高。
25.应用r软件中的“survminer”、“survival”、“timeroc”、“ggplot2”包对tcga-stad数据集中vcam1的表达与胃癌患者的预后情况(总生存期,os)及临床信息相关性(年龄、性别、tnm分期等)进行分析及可视化,并用gse62254数据集进行外部验证,结果表明vcam1高表达的胃癌患者总生存期短,即vcam1是胃癌患者的风险因素。
26.将tcga-stad数据集的患者按照vcam1表达量的中位数进行1:1高低分组并根据患者的总生存期进行生存预测,结果显示vcam1的表达与胃癌患者1、3、5年的生存率auc面积均大于0.5,即具有一定的生存预测价值。
27.将vcam1的表达量与胃癌患者临床信息(年龄、性别、tnm分期等)进行单因素cox、多因素cox回归分析,只有vcam1在单、多因素cox回归分析中p《 0.05,即具有统计学意义,证明vcam1的表达是一个独立的预后因素。
28.应用r软件中的“limma”、“msigdbr”、“fgsea”、“clusterprofiler”、“ggplot2”包对vcam1表达量高低分组(1:1)所得的患者分组表达谱矩阵进行差异表达分析,然后进行基因集富集分析(gsea)并进行可视化,结果表明vcam1的高表达与上皮-间质转化(emt)过程显著正相关,利用表达谱矩阵对vcam1与上皮标志物cdh1,间质标志物cdh2、vim以及诱导emt的转录因子(tf)twist1、snai1、snai2、zeb1、zeb2进行相关性分析,结果表明vcam1的表达与上皮标志物cdh1负相关,与间质标志物cdh2、vim正相关,与诱导emt的转录因子twist1、snai1、snai2、zeb1、zeb2正相关。
29.通过调节网络数据库(regnetwork,http://www.regnetworkweb.org/)下载计算机预测或实验验证的基因及其基因调节子信息,筛选获得vcam1的基因调节子,包括tf和mirna两种,应用cytoscape软件绘制基因调节网络,直接调控vcam1的tf为jun、nfkb1、nfkb2、rela、cebpb、ets1、pou2f1、spi1,其中jun、nfkb1、cebpb、rela富集在tnf信号通路中,促进介导细胞粘附的vcam1转录生成,综上所述,vcam1可能作为连接tnf信号通路和emt过程的桥梁,进而介导胃癌转移。
30.通过链接域数据库(linkedomics,http://www.linkedomics.org/)下载tcga-stad数据集表达谱矩阵分析得到的与vcam1相关的基因集及相关系数数据,选择与vcam1正相关和负相关的前50个基因,并应用r软件中的“clusterprofiler”、“ggplot2”包对它们进行go、kegg功能富集分析,这些基因主要富集在细胞粘附功能上。
31.通过弦数据库(string,https://cn.string-db.org/)中“proteinbyname”模块查找与vcam1有相互作用关系的蛋白质,并应用r软件中的“clusterprofiler”、“ggplot2”包对它们进行go、kegg功能富集分析,这些蛋白质主要富集在细胞粘附和细胞骨架功能上,以上结果表明vcam1可能与肿瘤细胞的转移相关。
32.通过基因表达谱交互分析数据库(gepia2,http://gepia2.cancer-pku.cn/)中的“expression analysis”和“survival analysis”模块对这些有相互作用关系的蛋白质进行表达量和预后分析,结果表明itga4、itgax、itgb1、itgb2在胃癌组织中较癌旁组织、正常组织高表达,且itgb1高表达的胃癌患者总生存期短,即itgb1是胃癌患者的风险因素。
33.对上述生物信息学分析结果进行体外实验验证,通过rt-qpcr、western blot实验检测vcam1在不同胃癌细胞系和胃正常细胞系中的mrna和蛋白质表达量,结果表明vcam1在胃癌细胞系中高表达,与生物信息学分析结果一致。通过rnai、woundhealing、transwell实
验发现vcam1敲低后胃癌细胞系的迁移、侵袭能力减弱,通过rt-qpcr、western blot实验发现vcam1敲低后emt过程中的上皮标志物e-cadherin表达上升,间质标志物n-cadherin、vimentin表达下降,推测vcam1可介导emt过程,从而导致胃癌转移。
34.虽然,上文中已经用一般性说明及具体实施例对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1