融合DNA形状特征的蛋白质编码区域预测方法、介质和装置
本发明属于生物信息学领域,结合了结构生物学和基因组学的知识设计并实现了一套融合dna形状特征的蛋白质编码区域预测新方法。
背景技术:
1、蛋白质编码序列区域(cds)是成熟mrna中能够被翻译为蛋白质的编码序列区域。cds的识别是基因组研究中的重要问题之一,识别cds可以帮助挖掘基因组序列中的有效信息,从而更加深刻地理解基因组信息组成中蕴含的基本规律。面对急速增长的基因组序列信息数据,越来越多的研究通过采用计算的方法来代替传统生物学方法,高通量地对蛋白质编码区域进行预测。
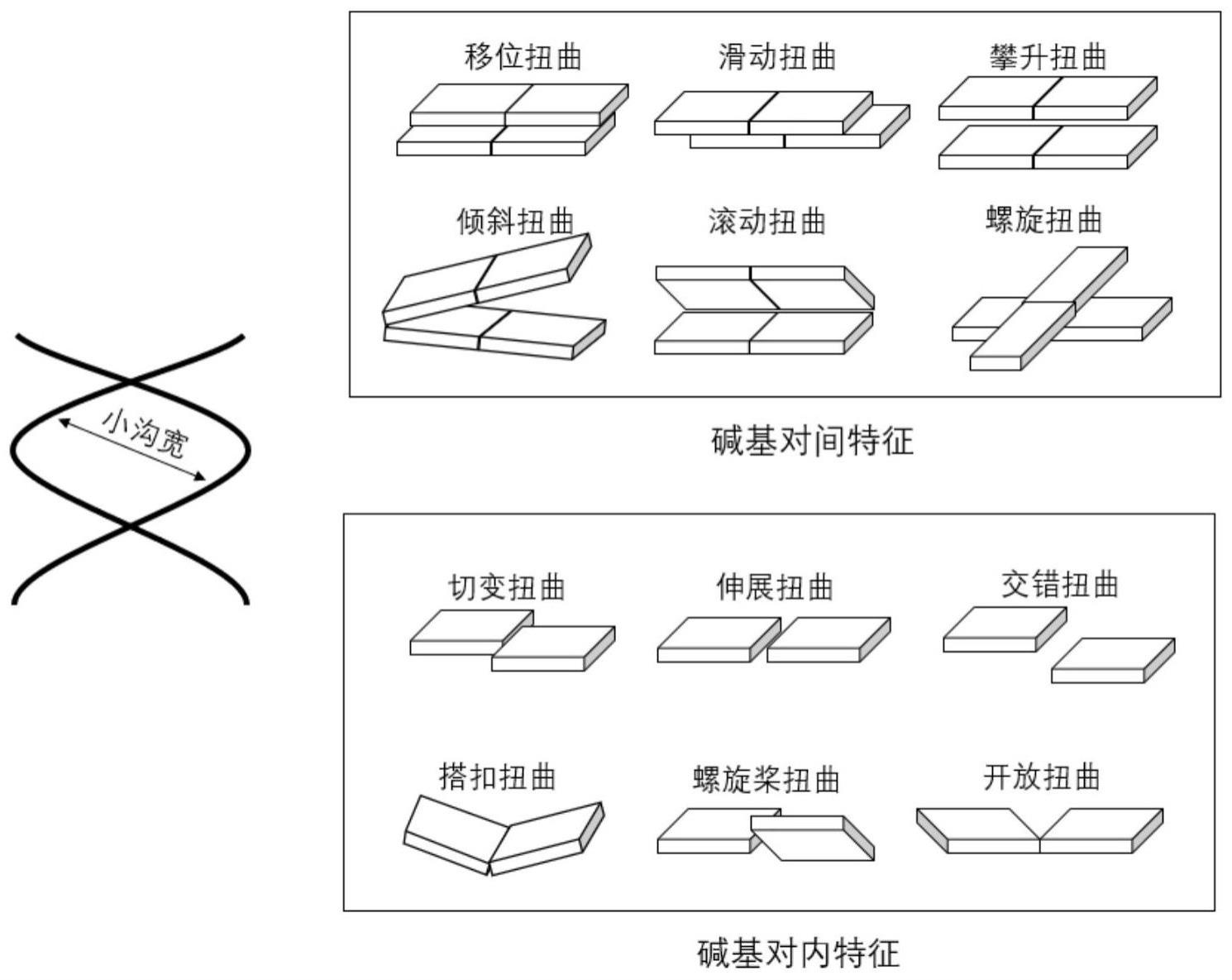
2、dna构象是转录因子与dna序列结合的重要影响因素,对于cds的预测同样具有重要作用。dna构象可以用dna形状特征来表示,其具体为碱基对内部和碱基对之间的平移、旋转所分配的数值(bp)或测量相反磷酸二酯骨架之间的凹槽宽度。这些数值可以通过curves或3dna等软件工具进行计算,将其合并到定量模型中,可用于蛋白质编码区域预测。dna形状特征信息可以从实验或者分子动力学模拟中获得,通过高通量方法获得整个基因组或者任意长度序列的大量形状信息。当前研究已经在dna构象信息中挖掘了13个dna形状(如图1所示)参数用以描述dna识别机制,并在高通量结合预测中得到了良好的结果,提高了结合预测的准确性。
3、然而当前生物dna序列中蛋白质编码区域的预测并未很好地将dna形状特征信息进行结合,在过去几十年中,已经出现了许多计算方法用于基因组或转录序列中蛋白质编码区域的预测。这些方法首先将生物dna序列信息编码为数值,将这些数值输入到分类器中进行最终的预测。主要使用的计算方法有顺序模型和离散模型两种。顺序模型保留了生物序列信息中碱基的原始顺序,如c4编码中使用4个二进制数编码对核苷酸进行编码(a-[1,0,0,0],c-[0,1,0,0]等);离散模型根据生物序列先验知识设计一组特征,如生物序列的3-mer表示,也能用于区分编码区域和非编码区域。
4、然而无论是顺序模型还是离散模型,都尚未将dna形状特征信息融入到蛋白质编码区域预测中,同时,预测的准确率有待进一步提高。
技术实现思路
1、本发明要解决的技术问题在于提供一种融合dna形状特征的蛋白质编码区域预测方法。
2、本发明要解决的技术问题在于提供一种融合dna形状特征的蛋白质编码区域预测方法,所述方法首先构建一个同时包含dna序列基序信息以及dna三维形状特征信息的可用于蛋白质编码区域预测的特殊数据集;然后提出一种新颖的可以同时融合dna形状特征与序列信息的转录因子结合位点预测模型,所述模型可以将dna的形状特征与序列信息相结合,从而提高蛋白质编码区域预测的准确性。
3、本发明通过如下技术方案实现:
4、一种融合dna形状特征的蛋白质编码区域预测方法,所述方法具体步骤如下所示:
5、1)构建数据库
6、根据现有公开数据库,设计构建一个包含dna形状特征数据和dna序列信息的特殊数据集,采用基于蒙特卡洛(mc)的dna shape方法预测dna的重要形状特征;
7、进一步,所述预测dna的重要形状特征包括六个碱基对间参数(shift、slide、rise、tilt、roll、helix twist)和六个碱基内部参数(shear、stretch、stagger、buckle、propeller twist、opening)以及小沟宽(minor groove width)。
8、2)dna原始序列数据及dna形状特征数据预处理;
9、使用基于mc的dna shape方法建立四聚体查询模型预测三维dna形状特征,获取形状数值,并将这些值组合成特征向量,输入包括dna序列信息和dna形状数值两部分,对于dna序列部分使用顺序编码和离散编码混合编码的形式,顺序编码使用c4编码,将碱基a、g、c、t分别编码为[1,0,0,0][0,1,0,0][0,0,1,0][0,0,0,1],输入为4×l的矩阵;离散编码根据dna序列的kmer特征,使用有间隙的kmer(gkm)编码,如使用f(xxaga)计算字段长度为5的拥有相同间隙三核苷酸(aga)的数值,输入为4×l的矩阵;对于dna形状特征部分,输入为4×l的矩阵,其中l表示序列长度,dna形状特征(shift、slide、rise、tilt、roll、helixtwist、shear、stretch、stagger、buckle、propeller twist、opening、minor groovewidth)用每个核苷酸位置的一个通道载体表示,共13个通道载体;
10、3)基于cnn-brnn的融合dna形状特征的蛋白质编码预测模型;
11、采用滑动窗口策略对编码区域和非编码区域进行区分;基于收集的样本dna序列信息、dna形状特征信息及其核苷酸位点上的标签数据,建立dna序列+形状特征模型;所述dna序列+形状特征模型为基于深度学习的cnn-brnn神经网络模型,对其中的dna序列信息进行c4编码与kmer特征编码,dna形状特征使用基于mc(蒙特卡洛算法)的dna shape方法提取,采取多输入并行卷积架构,cnn神经网络模型的输入为三个4×l的矩阵,分别为dna序列的c4编码信息、dna形状特征信息,然后分别进行卷积以及最大池化,其中卷积层使用的激活函数为relu(x)=max(0,x),最后经过flatten层将dna序列信息与dna形状特征进行结合,作为全连接层的输入,将cnn神经网络模型的输出结果与非重叠间隙kmer特征作为输入,分别输入至brnn神经网络模型进行计算,输出对核苷酸样本的蛋白质编码区域预测值,输出部分使用双层激活函数sigmoid函数以及softmax函数;
12、4)对步骤3)中的预测模型进行训练;
13、所提出的神经网络模型训练过程中,在tensorflow中使用tfrecord数据格式,学习率设置为常用值10-3,进行迭代训练。
14、一种计算机可读存储介质,所述计算机可读存储介质存储有计算机程序,所述计算机程序适用于由处理器加载并执行所述的融合dna形状特征的蛋白质编码区域预测方法。
15、一种蛋白质编码区域预测装置,所述装置运行所述的融合dna形状特征的蛋白质编码区域预测方法。
16、本发明与现有技术相比的有益效果:
17、1、构建了包含dna序列信息以及dna形状特征的数据集,将13个dna形状特征编码到数据集标签中。
18、本发明提出了包含丰富dna形状特征、融合dna序列信息的通用数据集,并提出了针对该数据集的cnn神经网络训练模型。该数据集同样适用于其他针对蛋白质编码区域预测方法的研究。
19、2、设计了使用cnn-brnn的深度学习模型,融合了混合编码框架以及dna shape编码,形成了蛋白质编码区域预测的新模型。
20、通过融合dna序列信息和dna形状特征信息,对dna序列信息进行c4和gkm混合编码,对dna形状特征信息进行基于mc的dna shape计算,经过cnn-brnn神经网络模型执行蛋白质编码区域预测任务,与其他模型相比具有更高的准确性和可用性。
- 还没有人留言评论。精彩留言会获得点赞!