一种基于固定框架的12sRNA二级结构可视化方法
本发明涉及算法设计和计算机科学、生物信息学以及计算机图形学领域,具体涉及一种基于固定框架的12srna二级结构可视化方法。
背景技术:
1、了解rna结构是理解其功能机制、设计合成rna和发现rna靶向制剂等研究的基础,但是高质量的二级结构观测和可靠的二级结构预测仍然十分困难。
2、rna二级结构可以通过应用能量最小化等生物物理学方法进行预测,但该方法在非常规配对和长序列从头预测等方面存在很高的错误率。基于机器学习的方法可以处理大规模的rna序列数据,但受限于已知rna结构很少,该方法对较为复杂的rna预测能力严重不足。目前准确度最高的rna二级结构预测方法仍然是同源建模法,但该方法对大规模构建类群驱动的模型及其可视化提出了较高的要求。
3、rna的二级结构可视化能够有效地显示更详尽的rna结构,使研究人员能够全面了解其组织和结构。同时rna分子可能存在多个不同的结构或伪结构,这些结构可能在不同的生物过程中起不同的作用。可视化工具能够同时表示多个结构或区分伪结构,以便研究人员可以比较它们并深入探索rna的多样性。rna二级结构可视化方法可以激发新的研究方向,帮助研究人员发现以前未察觉的rna结构特征,并在生物学领域开辟新的研究机会。通过可视化rna的二级结构,可以帮助人们更好地研究及理解rna在细胞中执行的各种生物学功能,如基因表达、调控和蛋白质合成。
4、目前rnastructure、viennarnapackage等软件均可以实现rna二级结构可视化,但是传统的rna二级结构静态图像无法直接用来进行引用,需要手动拖拽相关茎环结构至期望的rna二级结构相应位置,这大大增加了人力成本。现有可交互二级结构可视化方法虽然可以允许用户直观地操作和探索rna二级结构,从而更深入地了解其复杂性,但是依旧不能得到期望框架的rna二级结构可视化图,这使得开发出一种固定框架用于将rna序列得到期望的可视化图形显得愈加重要。
技术实现思路
1、针对现有的技术中的不足,本发明设计了一种基于固定框架的12srna二级结构可视化方法。
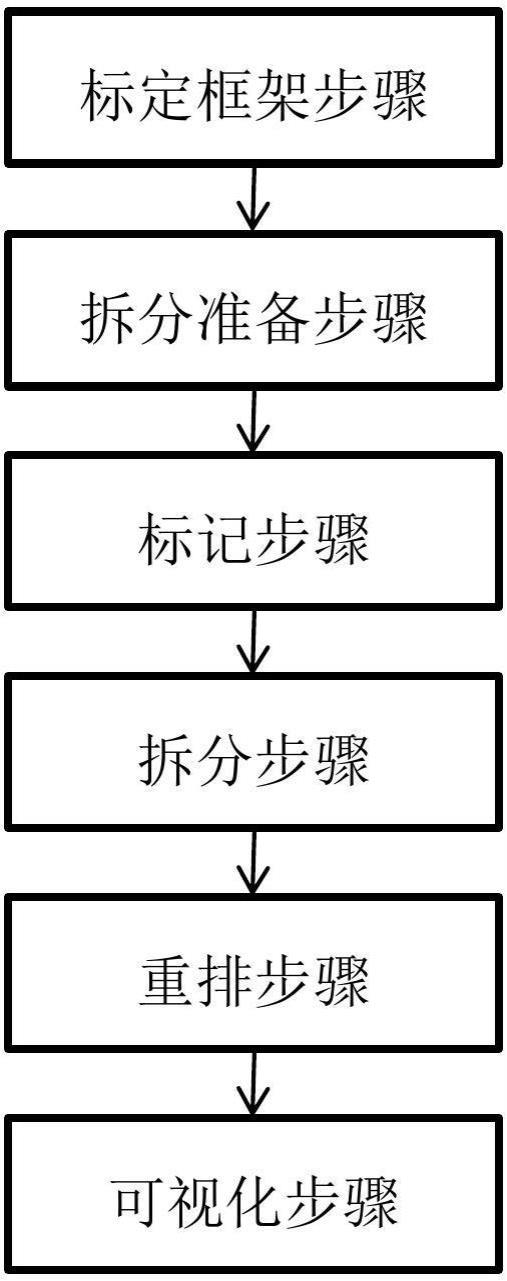
2、本发明提供的一种基于固定框架的12srna二级结构可视化方法,包括:
3、标定框架步骤:依据12srna序列的茎环结构标定12srna二级结构可视化图框架,包括给出用于放置茎-内环序列的茎环位置文档、用于放置发卡环序列的发卡环位置文档和用于放置多环序列的多环位置文档;
4、拆分准备步骤:根据12srna序列的点括号序列,提取发卡环、内环和多环的位置下标分别存入发卡环位置下标二维数组、内环位置下标二维数组、多环位置下标二维数组;
5、标记步骤:遍历12srna序列的点括号序列,给内环位置下标二维数组设置标记;
6、拆分步骤:根据发卡环位置下标二维数组提取碱基序列中的发卡环序列,根据多环位置下标二维数组提取碱基序列中的多环序列;根据带标记的内环位置下标二维数组将12srna序列的碱基序列一边拆分一边打上标记最后形成带标记的茎-内环序列的一维数组;
7、重排步骤:通过内环和相关茎的标记重排茎-内环序列的一维数组,得到重排后的茎-内环序列;
8、可视化步骤:根据茎-内环序列与茎环位置文档绘制12srna二级结构可视化图形的茎-内环部分;根据发卡环序列与发卡环位置文档绘制12srna二级结构可视化图形发卡环部分;根据多环序列与多环位置文档绘制12srna二级结构可视化图形的多环部分;最后形成12srna二级结构可视化图形。
9、进一步优选,所述茎环位置文档是用于摆放茎-内环序列的文档,包括存放茎-内环序列的矩形框架的4个顶点坐标信息;发卡环位置文档包括放置发卡环序列的初始位置和终点位置坐标信息以及半径信息,多环位置文档包括放置多环序列的初始位置和结束位置坐标信息以及半径信息。
10、进一步优选,所述拆分准备步骤:依据12srna序列的点括号序列,通过读取第一个点符号时查找下一个非点符号方法提取12srna序列中的发卡环结构,存储发卡环在12srna序列中的位置下标并放置发卡环位置下标二维数组中,通过查找配对符号方法和读取第一个点符号时查找下一个非点符号方法提取12srna序列中的内环加多环部分的位置下标放入三维数组中,通过三维数组中二维子数组的第一个一维子数组大小重排二维子数组中的一维数组,三维数组中长度小于3的二维子数组为内环位置下标二维数组,三维数组中长度大于2的二维子数组为多环位置下标二维数组。
11、进一步优选,所述标记步骤:给内环位置下标二维数组的第一个一维子数组打上标记,如果对应的12srna序列中内环两侧点括号序列符号为‘(’则在内环位置下标二维数组的第一个一维子数组添加‘left’标记;如果对应的12srna序列中内环两侧点括号序列符号为‘)’则在内环位置下标二维数组的第一个一维子数组添加‘rightn’标记;n为当前内环两侧点括号序列符号为‘)’的内环排序编号,n∈1,2,3,4,…,n;n表示12srna序列中内环两侧点括号序列符号为‘(’的内环总数。
12、进一步优选,所述标记步骤中:根据内环位置下标二维数组,将内环的前一个点括号序列为‘(’的内环打上‘l-left’标记;将内环的前一个点括号序列为‘)’的内环打上‘l-rightn’标记,n∈1,2,3,…,n。
13、进一步优选,所述拆分步骤依据12srna序列的点括号序列通过拆分(split)算法,将一条12srna序列拆分成茎、内环、发卡环和多环,并使用一维数组存放相对应碱基序列;所存放的碱基序列通过点括号序列中符号的位置下标索引映射相对应位置的碱基序列得到。
14、进一步优选,12srna序列包括碱基序列和点括号序列,根据发卡环位置下标二维数组提取碱基序列中的发卡环序列,发卡环序列为一维数组;根据多环位置下标二维数组提取碱基序列中的多环序列,多环序列为一维数组;根据带标记的内环位置下标二维数组将12srna序列的碱基序列一边拆分一边打上标记最后形成带标记的茎-内环序列。
15、进一步优选,根据带标记的内环位置下标二维数组将12srna序列的碱基序列一边拆分一边打上标记,最后形成带标记的茎-内环序列的方式如下:遍历12srna序列的点括号序列,首先通过读取连续的‘(’,在‘(’后通过查找配对方法找到配对的‘)’,得到‘()()()…()’形式,通过符号对应的位置下标找到12srna序列的对应位置,再使用调节字符串方法将‘()()()…()’对应的12srna序列转换成‘(…((()))…)’对应的12srna序列,至此可得到所有的茎,再根据已得到的带‘rightn’标记的内环位置下标二维数组对应的12srna序列中内环两侧相连的茎添加‘rightn’标记;在读取内环时根据内环位置下标二维数组中的‘left’标记给提取的对应内环打上‘l-left’标记,根据内环位置下标二维数组中的‘rightn’标记给提取的对应内环打上‘l-rightn’标记,由此得到带标记的茎-内环序列。
16、进一步优选,根据带标记的内环位置下标二维数组将12srna序列的碱基序列一边拆分一边打上标记,最后形成带标记的茎-内环序列使用的拆分算法如下:
17、读取‘.’时,当该‘.’前一序列符号不为‘.’时判断该‘.’的位置下标是否出现在带标记的内环中,如在带标记的内环中则继续判断在带标记的内环中的标记类型,如是‘l-left’则直接换行并给该‘.’打上‘l-left’标记输出位置下标对应的碱基序列,如是‘l-rightn’则直接换行并给该‘.’打上‘l-rightn’标记输出位置下标对应的碱基序列,当该‘.’前一序列符号为‘.’时,则照常读取输出位置下标对应的碱基序列;
18、读取‘(’时,对‘(’的前一符号进行判断,如前一序列为‘.’时,则判断该‘(’符号的配对符号下一个符号,当该‘(’符号的配对符号下一个符号为‘rightn’标记,则通过‘l-rightn’与该符号位置信息的映射给对应位置加上‘rightn’标记,输出位置下标对应的碱基序列以及该符号配对符号下标位置对应的碱基序列,否则直接输出位置下标对应的碱基序列以及该符号配对符号下标位置对应的碱基序列;如前一序列为‘(’时,判断该位置配对符号和该位置上一符号的配对符号相对位置,当相对位置在1以内时直接输出该位置下标对应的碱基序列以及该符号配对符号位置下标对应的碱基序列;否则判断该位置配对符号下一符号位置下标是否在‘rightn’标记与对应位置下标的映射中,如在则给对应位置加上‘l-rightn’标记输出该位置下标对应的碱基序列以及该符号配对符号下标位置对应的碱基序列,否则直接输出位置下标对应的碱基序列以及该符号配对符号位置下标对应的碱基序列;如前一序列为‘)’时,直接输出位置下标对应的碱基序列以及该符号配对符号位置下标对应的碱基序列;
19、读取‘)’时,直接跳至下一循环即可;
20、通过上述步骤拆分的茎点括号形式为‘()()()…()’,通过重排算法修改成‘(…((()))…)’形式,至此最终输出一个茎-内环序列的一维数组。
21、进一步优选,所述重排步骤:将已得到的带标记的茎-内环序列的一维数组依据‘l-rightn’以及‘l-left’标记进行重排将内环放置该内环两侧连接的茎之间。
22、进一步优选,所述可视化步骤:首先设置茎-内环序列、发卡环序列和多环序列的节点大小以及颜色,根据茎-内环序列的文档与茎环位置文档合成12srna二级结构可视化图形中的茎-内环图形;根据发卡环序列的文档与发卡环位置文档合成12srna二级结构可视化图形中的发卡环图形;根据多环序列的文档与多环位置文档合成12srna二级结构可视化图形中的多环图形;最后形成12srna二级结构可视化图形。
23、进一步优选,通过茎环位置文档以及茎-内环序列中每行茎的数量和内环的类别设定内环绘制的半径大小,确定茎、内环结构文档中每行茎所在位置,然后在茎所在位置上放置茎的碱基序列,最后在茎之间通过设定的内环绘制半径大小放置对应的内环的碱基序列,最终得到茎-内环图形;
24、通过发卡环位置文档中的起始位置、终点位置以及弧度信息确定发卡环摆放的位置,然后在位置上摆放发卡环的碱基序列,最终得到12srna二级结构可视化图形中发卡环图形;
25、通过多环位置文档中的起始位置、终点位置以及弧度信息确定多环摆放的位置,然后在位置上摆放多环的碱基序列,特别的,对于多环序列的文档中‘-’的部分代表该多环位置上无对应多环碱基序列,不进行多环碱基序列的放置,最终得到12srna二级结构可视化图形中多环图形。
26、与现有技术相比,本发明具有如下的有益效果:
27、1、能够根据12srna的碱基序列以及点括号序列可视化12srna序列的二级结构图片,方便人们更直观形象的观察12srna物种的序列信息以及用于文章引用图片。
28、2、本发明流程设计合理,使用方便,能够得到较为理想的12srna二级结构可视化图形;
29、3、使用设计拆分(split)算法得到准备绘制图形的茎环序列以及重排拆分的茎环结构,然后将拆分的序列与对应的结构文档结合得到一种固定结构的12srna二级结构可视化图形。使用该方法与传统的算法相比,具有固定结构框架,易于引用更加美观的优势。
- 还没有人留言评论。精彩留言会获得点赞!