一种基于混沌映射控制GC含量的双规则编码DNA存储方法
本发明涉及dna存储的,尤其涉及一种基于混沌映射控制gc含量的双规则编码dna存储方法。
背景技术:
1、随着互联网和物联网的迅速发展,不断产生和积累大量的数字数据,到2025年,预计将产生近175zb的数据。现代存储系统面临着高能耗,高成本,以及数据安全性不足等问题。dna是生物体中一种古老而高效的信息载体,作为一种具有长期稳定、低能耗、高安全性和巨大存储潜力的存储介质受到了研究人员的关注。与普通的信息载体相比,dna分子具有多种优势,包括超高的存储密度,文献[pavani yashodha de silva and gamage upekshaganegoda.“new trends of digitaldata storage indna”.in:biomed researchinternational 2016(2016).]发现每克dna的数据存储量能够达到215pb,约为2,2544,3840千兆字节(gb),相当于22万个1tb硬盘的数据存储量;其独特的稳定性能使dna分子在合适的环境下,比如-20℃以下的低温、20%-40%的湿度、避光以及真空条件下保存长达数千年的时间。
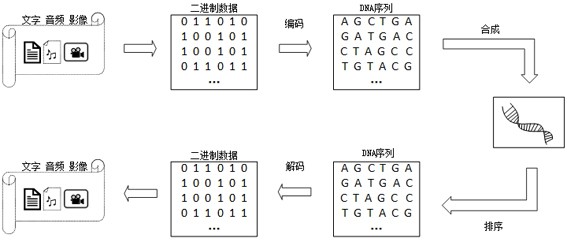
2、目前为止,已经提出了许多利用有机分子(dna,rna,寡肽,代谢体)进行信息存储的方法。dna测序技术的发展在过去几十年里取得了巨大的进展,使得基因组测序变得更加容易和普遍,因此dna信息存储成为备受推崇的存储策略。传统的dna存储如图1所示,其一般流程是将由文件转换来的二进制信息编码为dna碱基序列,合成并组装编码后的碱基序列,将其存储在适当的介质中以保证其稳定性和可访问性,利用测序技术检索并读取原始文件信息。
3、码本是dna存储过程中的一种编码方式,它反映的是二进制字符与碱基字符之间的映射关系。早期的码本([00,01,10,11]→[a,t,c,g])虽然很大程度地提高了dna的编码容量,但是会在碱基序列里产生一些特殊的结构,从而导致在合成和测序时面临某些困难[9,10,11]。比如,过长的重复碱基(n>4nt)会导致dna序列在合成或测序时发生碱基错误;dna序列中gc分布不均匀,含量过低或者过高会导致dna序列pcr扩增分布不均匀[12,13,14];dna序列存在的回文子序列会导致该序列在pcr扩增时出现发夹等二级结构,进而影响pcr扩增的效率。因此编码本应遵循一定的规则,以减少dna数据存储过程中的系统性错误。2012年8月,哈佛大学的church等在文献[g.m.church,y.gao,and s.kosuri,“next-generation digital information storage in dna,”science,vol.337,no.6102,pp.1628-1628,sep,2012.]中实现了对5.2mb数据的html文件、jpg图像和javascript程序的dna存储。他们首先提出了“位-基”映射码本,每比特对应一个碱基,这种码本避免了长度大于3的均聚物的出现,但由于测序深度有限和缺少纠错策略,数据并不能完全恢复。相比而言,goldman等[goldman,n.etal.towards practical,high-capacity,low-maintenanceinformation storage in synthesized dna.nature 494,77–80(2013).]加入了压缩策略和奇偶校验码,增加对dna存储系统错误的鲁棒性。2015年,grass等在文献[grass,r.n.,heckel,r.,puddu,m.,paunescu,d.&stark,w.j.robust chemical preservation ofdigital information on dna in silica with error-correctingcodes.angew.chem.int ed.engl.54,2552–2555(2015)]中提出了一种新的“符号-密码子”内码和外码编解码方法。这些策略都是以牺牲存储密度为代价的。erlich等于2017年在文献[erlich,y.&zielinski,d.dna fountain enables a robust and efcient storagearchitecture.science 355,950–954(2017).]中提出了一种基于fountain code(ltcode)的新型dna存储方法,并以较低的测序深度(平均覆盖率:~10.5×)实现了2.15mb数据文件(包括文本、操作系统、图像、pdf、电影和软件)的dna存储和完全准确恢复,与以往的编码译码方法相比,该方法不需要重复测序,显著减少了冗余度。dna fountain引入了低冗余、均聚物长度和gc含量的筛选约束,提高了信息保真度,并利用luby变换使存储密度达到了1.57bit/nt。然而,根据文献[feng,l.,foh,c.h.,jianfei,c.&chia,l.lt codesdecoding:design and analysis.in 2009ieee international symposium oninformation teory.2492–2496.(ieee,2009)]由于luby变换的基本问题,在处理特定的二进制时存在解码失败的风险。因此亟需提出一种满足均聚物和gc含量等约束,又不损失存储密度的编码算法[matange,k.,tuck,j.m.&keung,a.j.dna stability:a centraldesign consideration for dna data storage systems.nat.commun.12,1358(2021).]。
技术实现思路
1、针对早期编码方案追求高密度却忽视了dna序列的生物约束特性,导致dna合成和测序方面困难的技术问题,本发明提出一种基于混沌映射控制gc含量的双规则编码dna存储方法,具有较高的鲁棒性,能够保证数据的安全。
2、为了达到上述目的,本发明的技术方案是这样实现的:一种基于混沌映射控制gc含量的双规则编码dna存储方法,包括以下步骤:
3、s1:将数据压缩形成二进制流,利用混沌映射将二进制流的首位随机化,得到首位0/1平衡的二进制序列;
4、s2:根据gc含量约束和最大均聚物长度约束得到双规则编码表,以此对二进制序列进行编码映射,得到对应碱基序列;
5、s3:根据对应碱基序列对dna进行合成;
6、s4:对存储的dna序列进行测序,利用汉明距离与rs码对dna序列进行纠错并解码正确的碱基序列。
7、步骤s1所述将数据压缩形成二进制流的方法为利用霍夫曼编码对数据进行压缩。
8、步骤s1所述利用混沌映射将二进制流的首位随机化的方法为:
9、s11:将二进制流ⅰsrand以5bit为单位进行分组,每组第一位按顺序取出并排列成二进制序列ⅱsfirst;
10、s12:使用逻辑混沌映射生成一段由0和1组成的混沌序列schaotic;
11、s13:将二进制序列ⅱsfirst与混沌序列schaotic进行迭代异或运算得到新序列,直至新序列中0或1占总体的比例在40%-60%的范围内;
12、s14:利用新序列替换二进制流每组的首位,得到首位随机化后的二进制序列ⅲsrand。
13、步骤s12所述使用逻辑混沌映射生成混沌序列schaotic的方法为:设定辑混沌映射的控制参数,随机生成一段值在[0,1]的序列,将序列中属于区间[0,0.5]的值转为0,序列中属于区间[0.5,1]的值转为1,最终便生成一段由0和1组成的混沌序列schaotic。
14、所述二进制序列ⅲsrand为:
15、srand=(s11,s12,s13,s14,s15,…sn1,sn2,sn3,sn4,sn5)
16、以每5个为单位对二进制序列切片,其中n代表第n组切片,sn代表第n组切片的碱基。
17、所述双规则编码表包括0规则编码表和1规则编码表,0规则编码表为:
18、
19、;1规则编码表为:
20、。
21、所述对二进制序列进行编码映射,得到对应碱基序列的方法为:
22、s21:将二进制序列为srand以5bit为单位进行分组得到5bit二进制序列,则对于每组5bit二进制序列的映射过程为:{si1,si2,si3,si4,si5}→{ni1,ni2,ni3},1<i<n,ni1,ni2,ni3∈{a,t,c,g},i代表碱基或者二进制序列切片的组数,其中,a、t、c、g分别为构成dna的碱基序列的四种碱基;
23、s22:以每组5bit二进制序列首位数的值是0或是1来判断二进制序列片段si2,si3,si4,si5使用映射规则的条件;
24、s23:根据二进制序列部分片段si2,si3映射dna序列片段的第1位ni1:
25、
26、s24:根据二进制序列部分片段si4,si5结合0规则编码表或1规则编码表映射碱基序列的的第2位ni2和第3位ni3。
27、步骤s3所述根据对应碱基序列对dna进行合成的方法为:在碱基序列头尾两端添加引物,在碱基序列端部与引物之间添加索引,在碱基序列尾部与引物之间添加rs纠错码组成dna序列。
28、步骤s4所述利用汉明距离与rs码对dna进行纠错的方法为:
29、s411:将dna以3为单位分割为切片,并计算每个切片与编码表中码字之间的汉明距离,若汉明距离为0,则证明该碱基切片不存在错误,若汉明距离不为0,则该碱基切片存在错误;
30、s412:寻找第一个非零汉明距离切片,判定为第一个错误切片;如果发生插入错误,则删除该切片的第一个碱基;如果发生删除错误,则在切片的第一个位置加入任意一个碱基;如果发生替换错误,则利用rs码进行纠正;
31、s413:最后对整个序列再次以3碱基为单位切片,重新计算各个切片与编码表码字的汉明距离,并重复步骤s411-s413,直至所有切片的汉明距离均为0。
32、步骤s4所述解码正确的碱基序列的获得方法为:按照固定位数3分割测序得到的碱基序列切片,计算每个碱基序列片段的中g/c的数量之和nigc;当nigc=1时,用0规则对该碱基序列切片解码;当nigc=2时,用1规则对该碱基序列切片解码,将解码后的碱基序列按顺序拼接得到二进制序列ⅳ,将二进制序列ⅳ以5为单位分组并提取每组首位,将首位与混沌序列进行异或得到原首位序列,之后将原首位序列替换回每组的第一位,得到正确的碱基序列。
33、本发明提出了一种基于混沌映射控制gc含量的双规则dna存储系统(drrc),两种编码规则相辅相成,两者相结合能够灵活控制gc含量,引入了旋转编码模式有效地避免了均聚物的出现。在编码之前,对原始序列引入首位随机化,只有用相应的混沌序列参数才能解码出正确且完整的原始信息,提高了存储的安全性。通过控制gc含量,编码方案也有着良好的存储密度,并且支持huffman,算术编码等压缩方式,从而显著增加存储容量。
34、在鲁棒性方面,本发明引入了汉明距离的概念来检测错误片段,并采用插入或删除碱基的方法来切断错误片段对后续片段的误差传播。实验证明,加入这种阻止错误传播机制后,数据恢复率得到有效的提升。本发明在dna序列设计中还加入了rs纠错码,能够纠正整条dna序列上的最多8个错误。
35、通过实验和仿真证明,本发明的方案能够存储不同格式类型的源文件,编码容量为1.66bit/nt,并且具有较高的鲁棒性,能够保证数据的安全。
- 还没有人留言评论。精彩留言会获得点赞!