一种纳米孔测序中条形码的设计方法
本发明属于分子生物学和人工智能相结合的,具体涉及一种纳米孔测序中条形码的设计方法。
背景技术:
1、将多个样本汇集在一起以最大限度地利用资源是生物分子研究中的常见做法。为了识别不同的样本,实验平台通常把不同的条形码与不同的样本连接在一起。条形码在研究和生物技术中发挥着重要作用,例如单细胞测序、基因合成、药物发现和高通量测序等。然而,dna合成和测序过程中产生的错误(表现为碱基的插入、删除和替换)可能会破坏条形码,导致无法正确识别,降低实验的准确性。
2、纳米孔测序是现代测序技术的突破,由于其测序读数长的优势,极大地推动了基因组组装、单细胞转录组学和融合基因的研究。为了降低测序成本,平台通常会给每个样本链接独特的条形码来同时测序多个样本。然后,根据已知的条形码(短且唯一的核苷酸序列)将每个样本的测序读数分离。然而,oxford nanopore technologies(ont)官方提供的条形码套件中最多包含96个条形码,这意味着最多可以在单次运行中测序96个样本。当实验规模很大,就需要包含更多条形码的条形码套件。众所周知,与二代短读测序相比,纳米孔测序的通量低,这引入了一些权衡(如在单细胞研究中,将高准确性换为低读数深度)和高成本的问题。实现纳米孔测序的更高通量需要包含更多条形码的条形码套件。
3、目前有用纠错码方案设计dna条形码。纠错码主要用于计算机科学中的信息传输的错误检测和纠正。例如运用levenshtein码来设计条形码,它理论上可以处理所有类型的测序错误,但需要知道每个损坏的条形码的长度,另有人提出了tvns算法来生成条形码,并改进了条形码集合的最小长度下界。
4、据了解,这些现有的条形码设计方法是针对于具有低错误率的二代测序,而纳米孔测序的最大缺点是错误率高。因此,纳米孔测序中设计条形码的问题需要更精确的设计方法。
技术实现思路
1、本发明的目的在于提供一种纳米孔测序中条形码的设计方法,在纳米孔测序的高测序错误率情况下,从概率的角度设计包含较多条形码的条形码集合,并且能够实现高的解复用精度,以弥补现有技术的不足。
2、ont提供了一种同时测序多个样本的方法,该方法通过使用条形码(短dna序列)在单次运行中识别样本。根据条形码,解复用工具被用于在测序后将序列分配到特定的样本中。纳米孔官方目前提供的套件中只包含少量条形码。在大规模实验中,大量样本需要测序,这就需要更多的条形码来降低测序成本与实验难度。
3、为达到上述目的,本发明是通过以下技术方案实现的:
4、一种纳米孔测序中条形码的设计方法,包括以下步骤:
5、s1:利用碱基a、g、c、t形成一定长度的序列作为候选序列,对该候选序列进行预处理,初步筛选有差异性的序列;
6、s2:设定阈值t,保证设计出的条形码集合{barcode}中任意两个序列之间有足够大的差异性;
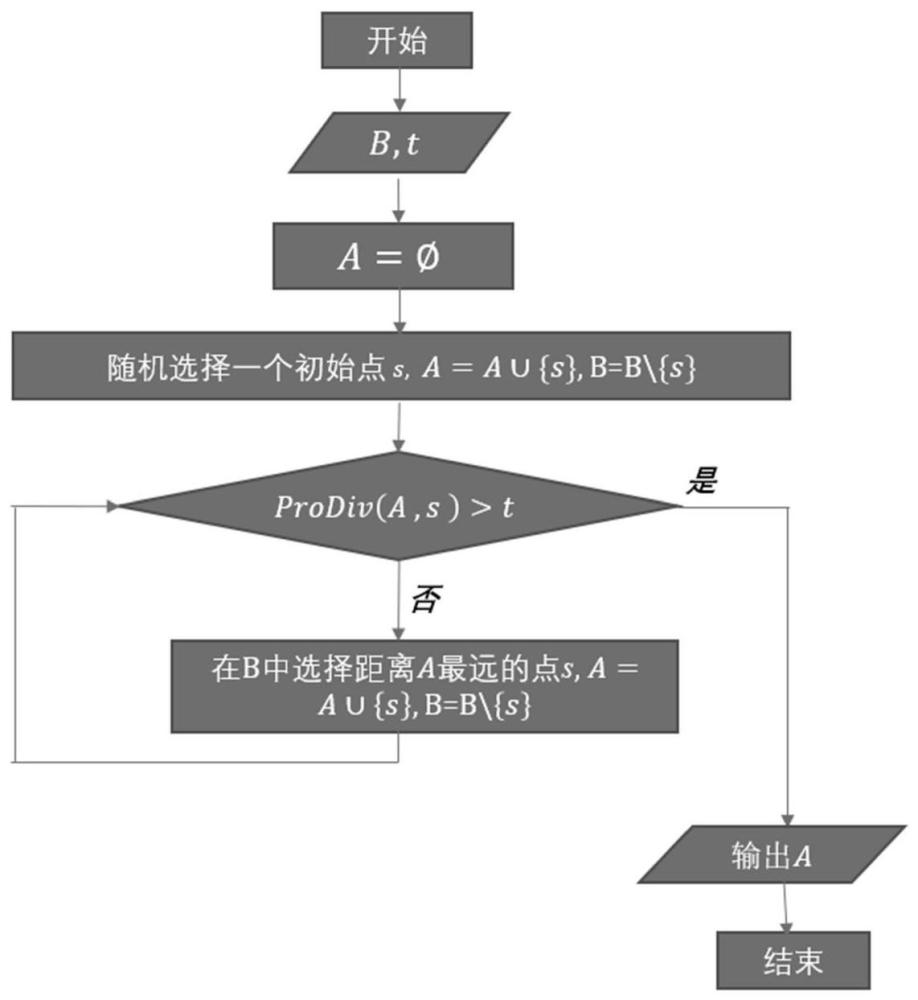
7、s3:针对最远点采样算法进行改进;
8、s4:在确定阈值t和候选的条形码集合{candidate}之后,采用改进的最远点采样算法来挑选条形码,输出采样集合{barcode}set。
9、进一步的,所述s1包括:
10、s1-1:记∑={a,g,c,t}是一个四元字母表.一个在字母表∑上的长度为k的序列seq=s1s2…sk,si∈∑,i=1,2…k;定义k-mer空间为所有长度为k的序列seq的集合,记作sk;所有k-mer空间的并集记为s,i.e.s=uk>0sk;
11、s1-2:对所有候选的dna序列进行预筛选,以粗略的方式选出有一定差异性的dna序列,减小算法输入的数据量;首先将长为n的dna序列通过哈希函数n:n(g)=0,n(a)=1,n(t)=2,n(c)=3转为整数;对于序列s1s2…sn∈sn,有n(s1s2...sn)=n(s1)40+n(s2)41+…+n(sn)4n-1;将所有整数从小到大排列,然后进行均匀采样;采样得到的数集对应的dna序列集合就是预处理后的dna序列集合。
12、进一步的,所述s2包括:
13、s2-1:对于给定的条形码长度和阈值t,记{barcode}为条形码集合,最大条形码集合{barcode}max满足以下条件:
14、
15、s2-2:确定阈值t即确定了在生成的{barcode}集合中任意两个条形码序列之间的最大概率差异;越小的阈值表示生成的{barcode}集合中两个序列之间的差异越大,最终挑
16、选出{barcode}集合中条形码的数目越少;
17、s2-3:如果阈值t要确保测序错误个数少于n的条形码能够被正确解复用,那么其中其中p为测序错误率。
18、进一步的,所述s3中,对最远点采样算法进行改进:(1)采样的终止条件由采样数目n变为控制由阈值t控制;(2)最远点采样算法中用概率差异来度量两个序列差异性;改进后的最远点采样算法不断迭代地挑选与已挑选集合{barcode}set差异最大的序列,将挑选的序列加入{barcode}set中,保证挑选到的序列集合{barcode}set中任意两条序列之间的差异性足够大。
19、进一步的,所述s4包括:
20、s4-1:首先从候选集合{candidate}中随机选取一条序列seq作为第一个采样序列,将其加入到采样集合中,并从候选集合中删除:{barcode}set={seq},{candidate}={candidate}\{seq};
21、s4-2:在剩余的候选序列集合{candidate}中选取与seq差异最大的点,也就是与seq概率差异最小的点,作为第二个采样点seq′,seq′满足
22、weight(seq′,seq)=min{weight(s,seq),s∈{candidate}}
23、将第二个采样点加入到采样集合中,并从候选集合中删除;
24、s4-3:在候选集合{candidate}中选取与已采样集合{barcode}set概率差异最小的点seq′,即seq′满足
25、weight(seq′,{barcode}set)=min{weight(s,{barcode}set),s∈{candidate}};
26、其中weight(s,{barcode}set)=max{weight(s,s′),s′∈{candidate}},
27、将采样点加入到采样集合中,并从候选集合中删除;
28、s4-4:重复s4-3,不断从候选集合{candidate}中选取采样点seq′,直到
29、weight({barcode}set,seq′)>t。
30、更进一步的,所述s4-2中,所述概率差异具体为:对于任意两个dna序列x,y,x到y概率差异定义为:
31、
32、其中,pm,pd,ps,pi分别为测序过程中碱基正确测量,发生删除,替换,插入的概率;显然,概率差异不满足对称性,定义两个序列x,y之间的权重:
33、weight(x,y)=max{prodiv(x,y),prodiv(y,x)}。
34、进一步的,上述方法还包括条形码解复用步骤:首先从测序后的序列中截取被破坏的条形码序列;然后在条形码套件中,选择与截取序列概率差异性最大的条形码,作为最后的解复用结果。
35、更进一步的,所述解复用方法分为两步:第一步从测序后的dna序列中提取在纳米孔测序进行文库制备时接入的条形码序列;借助对齐工具edlib识别测序建库时所用的左右两个侧翼序列的终止与起始位置endf与starts,这两个位置中间的序列为提取到的被测序破坏的条形码序列s′;第二步从{barcode}集合中挑选与s′最相似的序列,即在{barcode}中寻找满足以下条件的序列:
36、pro(s′,s0)=max{pro(s,s′),s∈{barcode}}
37、将s0作为最后的解复用结果。
38、与现有技术相比,本发明的优点和有益效果是:
39、本发明对条形码设计问题和解复用策略进行了全面的研究。从概率的角度进行了条形码的设计,两个序列之间的概率差异可以根据不同的测序技术的测序错误率改变,显著扩大了dna条形码套件的容量。本发明利用改进的最远点采样算法设计条形码套件,极大程度上保证了设计条形码之间的差异性,并且实现了较高的解复用精度。
40、本发明解决了纳米孔测序中给出的条形码数目少的问题,为提高纳米孔测序通量提供可能性,降低了大型实验的成本。
- 还没有人留言评论。精彩留言会获得点赞!