由自组装的含顶点的固定角度核酸结构形成的核酸多面体的制作方法
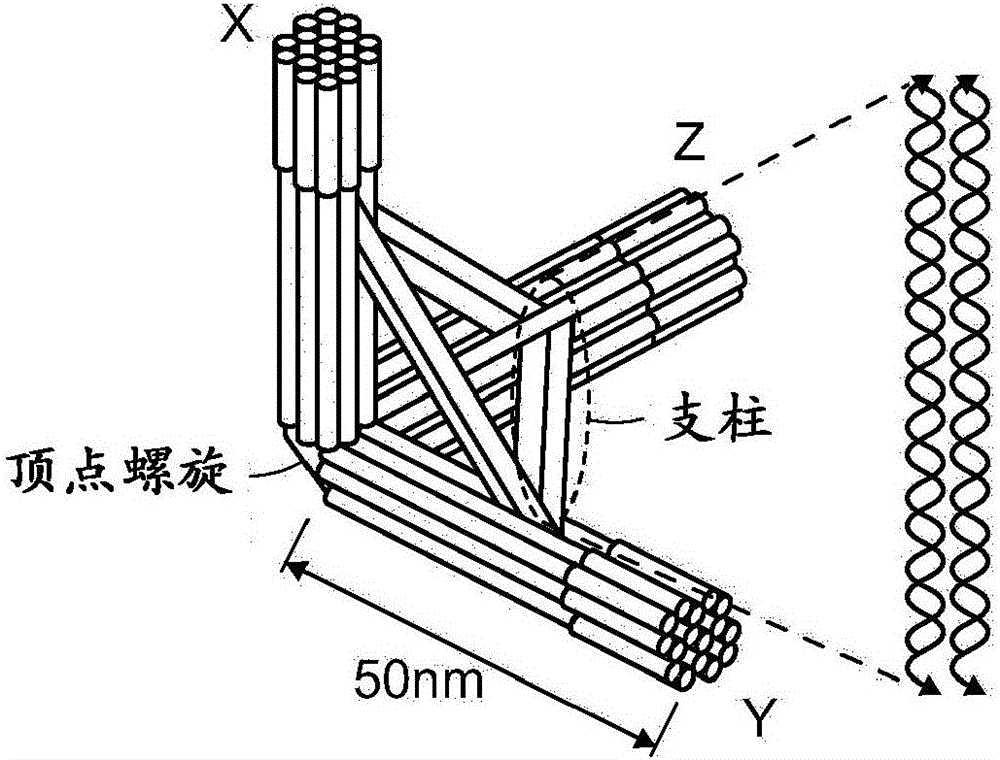
本申请根据35U.S.C.§119(e)要求2014年3月8日递交的美国临时申请号61/950098的权益,所述申请通过引用整体并入本文。联邦政府资助的研究本发明由美国政府支持在由海军研究办公室授予的基金号N000141110914、N000141010827和N00014130593;由陆军研究办公室授予的基金号W911NF1210238;由美国国立卫生研究院授予的基金号1DP2OD007292、1R01EB018659和5R21HD072481;和由美国国家科学基金会授予的基金号CCF1054898、CCF1317291、CCF1162459和CMM11333215下完成。美国政府对本发明具有一定的权利。发明领域本文提供了用于产生核酸结构例如DNA笼的新型组合物和方法。发明背景DNA纳米技术已经产生了广泛多样的形状受控的纳米结构(1-10)。中空多面体(1,5,11-26)是特别有趣的,因为它们类似于天然结构例如病毒衣壳,并且有希望用于支架和封装功能材料的应用。先前的工作已构建了多种多面体,例如四面体(13,16,20,24),立方体(1,19,23),双锥体(15),截短的八面体(11),八面体(12),十二面体(16,18),二十面体(17,21),纳米棱柱(14,22,25,26),和巴基球(16),具有低于80nm的尺寸和低于5兆道尔顿(MD)的分子量(例如图1A中的结构1-8)。组装策略包括逐步合成(1,11,21,22),长支架的折叠(12,19,20,24,25),个体链的协同组装(13-15,18,26),和分支DNA瓦片的分层组装(16,17,23)。按比例放大多面体的另一种途径是较大的单体的分层组装。使用小的三臂连接(16,21)(80kD)和五臂连接瓦片(17)(130kD)的先前工作已产生了几种低于5MD的多面体(例如图1A中的结构5-7)。此外,15MD二十面体(5)(图1A,结构9)由三个双三角形的折纸单体组装。然而,此二十面体以低产率生成(5),并且此方法尚未一般化至构建更复杂的多面体。发明概述本发明提供了用于任选地一步自组装线框DNA多面体的新颖的一般策略,所述多面体比以前的结构更大并且以比以前的结构更高的产率产生。硬的三臂连接瓦片模体被用于多面体的分层组装,所述硬的三臂连接瓦片模体可以使用例如DNA折纸以精确控制的角度和臂长制备。通过使用这些方法,有可能构建具有100纳米的边缘宽度的四面体(20兆道尔顿或MD)、三角柱(30MD)、立方体(40MD)、五角柱(50MD)和六角柱(60MD)。所述结构通过透射电子显微镜检查和通过解析的单分子的三维DNA-PAINT超分辨率荧光显微镜检查来可视化。因此,在一个方面,本文提供了核酸结构,所述核酸结构包含第一(x)、第二(y)和第三(z)核酸臂,其各自在一个末端连接至其它臂以形成顶点,和第一、第二和第三核酸支柱,其中所述第一核酸支柱将所述第一(x)核酸臂连接至所述第二(y)核酸臂,所述第二核酸支柱将所述第二(y)核酸臂连接至所述第三(z)核酸臂,并且所述第三核酸支柱将所述第三(z)臂连接至所述第一(x)核酸支柱。在另一个方面,本文提供了包含以固定的角度从顶点辐射的三个核酸臂的核酸结构。在另一个方面,本文提供了核酸结构,其包含从顶点辐射的N个核酸臂,其中N是核酸臂的数目并且为3或更多,和M个核酸支柱,每个支柱将两个核酸臂彼此连接,其中M是核酸支柱的数目并且为3或更多。在一些实施方案中,N等于M。在一些实施方案中,N小于M。现提供关于一个或多个前述方面的实施方案。在一些实施方案中,核酸结构包含4个核酸和至少4个核酸支柱,或5个核酸臂和5个核酸支柱。在一些实施方案中,核酸臂以相等的间隔彼此分开(或所述臂以相同的角度彼此分离)。在一些实施方案中,核酸臂以不相等的间隔彼此分开(或所述臂以不同的角度彼此分离)。在一些实施方案中,核酸结构包含以60°-60°-60°彼此分离的三个核酸臂。当四个这样的结构在它们的游离末端彼此连接时,它们形成四面体。在一些实施方案中,核酸结构包含以60°-90°-90°彼此分离的三个核酸臂。当六个这样的结构在它们的游离末端彼此连接时,它们形成三角柱。在一些实施方案中,核酸结构包含以90°-90°-90°彼此分离的三个核酸臂。当八个这样的结构在它们的游离末端彼此连接时,它们形成立方体。在一些实施方案中,核酸结构包含以108°-90°-90°彼此分离的三个核酸臂。当十个这样的结构在它们的游离末端彼此连接时,它们形成五角柱。在一些情况下,五角柱可通过连接限定为120°-90°-90°的核酸结构形成。在一些实施方案中,核酸结构包含以120°-90°-90°彼此分离的三个核酸臂。当十二个这样的结构在它们的游离末端彼此连接时,它们形成六角柱。在一些情况下,五角柱可通过连接限定为140°-90°-90°的核酸结构形成。在一些实施方案中,核酸结构还包含顶点核酸。在一些实施方案中,核酸结构还包含连接器核酸。在一些实施方案中,核酸臂、核酸支柱和/或顶点核酸包含平行的双螺旋。在一些实施方案中,核酸臂的长度相同。在一些实施方案中,核酸支柱的长度相同。在一些实施方案中,核酸支柱的长度不同。在一些实施方案中,至少一个核酸臂包含平末端。在一些实施方案中,至少一个核酸臂在其游离(非顶点)末端包含连接器核酸,所述连接器核酸的长度为多至16个核苷酸。在一些实施方案中,至少一个核酸臂在其游离(非顶点)末端包含连接器核酸,从而包含1或2个核苷酸突出端。在一些实施方案中,核酸结构的大小为多至5兆道尔顿(MD)。在一些实施方案中,核酸臂的长度为50nm。在另一个方面,本文提供了复合核酸结构,其包含选自任何前述核酸结构的L个核酸结构,其中L为核酸结构的偶数目,并且其中所述L个核酸结构在核酸臂的游离(非顶点)末端彼此连接。在一些实施方案中,两个以上的核酸结构是2、4、6、8、10、12个或更多个核酸结构。在一些实施方案中,复合核酸结构是四面体、三角柱、立方体、五角柱或六角柱。在一些实施方案中,复合核酸结构的大小为20兆道尔顿(MD)、30MD、40MD、50MD或60MD。在一些实施方案中,复合核酸结构具有100nm的边缘宽度,包含来自相邻核酸结构的两个核酸臂。在另一个方面中,本文提供了用于合成任何前述核酸结构和复合核酸结构的方法。在一些实施方案中,该方法包括在反应容器中组合核酸支架链与核酸订书钉链,其中所述核酸订书钉链被选择来在杂交至核酸支架链时形成任何前述核酸结构。在一些实施方案中,该方法进一步包括组合核酸支架链、核酸订书钉链和核酸连接器链,其中当所述核酸支架链、核酸订书钉链和核酸连接器链彼此杂交时,它们形成复合核酸结构,例如任何前述的复合核酸结构。本文中提供的这些和其它方面以及实施方案更详细地描述于本文中。附图简述图1A-1B。DNA-折纸多面体。(图1A)由DNA三脚架以可调的臂间角度自组装的多面体,以及其大小和分子量与选定的先前多面体(结构1-9;详细信息参见图5)的比较。(图1B)三脚架的设计图。圆柱代表DNA双螺旋。关于在顶点处的臂连接的细节参见图6。(图1C)显示两个三脚架单体之间的连接的圆柱体模型。(图1D和图1E)用于组装(图1E)四面体和(图1D)其它多面体(这里由立方体图样表示)的连接方案。图2A-2F。DNA三脚架和多面体的自组装。60°-60°-60°(凝胶中的泳道1)和90°-90°-90°(泳道2)三脚架的(图2A)凝胶电泳和(图2B)TEM图像。凝胶泳道3:1kb梯度标记物。凝胶电泳:1.5%天然琼脂糖凝胶,冰水浴。(图2C和2D)连接器设计的两种方案和相应的凝胶电泳结果。对于每一种方案,链模型描绘了两对DNA双链体之间的连接。凝胶泳道上方的数字表示两个相邻臂之间的连接的螺旋的数量。泳道L:1kb梯度标记物。泳道S:支架。箭头指示对应于组装的立方体的条带。(图2C)方案i:包含2nt粘性末端的长(30nt)连接器(红色)。完整的30nt连接器仅在左侧示出,其具有锚定在左螺旋上的28nt区段和可用于与90°-90°-90°右相邻物杂交(虚线圆圈描绘了杂交位点)的2nt的暴露粘性末端。(图2D)方案ii:包含2nt粘性末端的短(11nt)连接器。(图2E)立方体的组装产率,计算为立方体条带和相应的支架条带之间的强度比。(图2F)多面体的琼脂糖凝胶电泳。泳道1:90°-90°-90°单体。泳道2-6:多面体。泳道7:包含无支柱的三脚架的组装反应。泳道8:包含无顶点螺旋的90°-90°-90°的三脚架的组装反应。泳道9:1kb梯度标记物。相应于所需产物的凝胶条带标有箭头。凝胶电泳:0.8%天然琼脂糖凝胶,冰水浴。图3A-3E。多面体的TEM图像。显示了四面体(图3A)、三角柱(图3B)、立方体(图3C)、五角柱(图3D)和六角柱(图3E)的放大的(第1和2栏)和缩小的(第3栏)图像。四面体、三角柱和立方体的图像获自纯化的样品。五角柱和六角柱的图像从粗样品(用“*”表示)收集。比例尺在放大的TEM图像中为100nm且在缩小的图像中为500nm。需要注意的是对于未纯化样品可明显见到聚集体(在D的最右侧图中)。图4A1-4G。多面体的3DDNA-PAINT超分辨率荧光成像。(图4A1)将每一个多面体的顶点上的订书钉链用单链的对接序列延伸用于3DDNA-PAINT超高分辨率成像。(图4A1-4E1)多面体的示意图,其中突出显示了DNA-PAINT位点。(图4A2-4E2)以与A1-E1中所示的相同的角度显示的典型的多面体的3DDNA-PAINT超分辨率重建。(图4A3-4E3)2Dx-y投影。(图4A4-4E4)2Dx-z投影。(图2.4A5–4E5)从x-z-投影中的横截面直方图得到的多面体的高度测量。(图4F)四面体和漂移标记物(个体亮点)的较大的2D超高分辨率x-y投影视图。将衍射限制图像于上方施加在上半部分的超分辨率图像上。(图4G)四面体的较大视野图像的倾斜3D视图。漂移标记物以个体亮点出现。比例尺:200nm。颜色指示z方向上的高度。图5。20-60兆道尔顿DNA多面体。由可调的DNA折纸三脚架组装的20-60兆道尔顿DNA线框多面体。顶部:示意图示出了三脚架单体和多面体的组装过程;中间:多面体的TEM图像;底部:多面体的超分辨率荧光图像。这些多面体比图1A中的先前DNA多面体显著更大,其包含:(1)立方体(1)、截短的八面体(11)、四面体(13)、八面体(12),(2)四面体、十二面体和由三臂DNA瓦片组装的巴基球(16),(3)DNA折纸四面体(24),和(4)由三个DNA-折纸单体(5)组装的二十面体。图6。在顶点处三臂单体的连接。在顶点处连接的三个层:(1)第一层(最里层)连接仅通过支架链形成。在双链体之间没有额外的碱基。(2)第二层(中间)连接和(3)第三层(最外层)连接是通过订书钉链及其互补链形成的DNA双链体(即,顶点螺旋)。各多面体使用不同数目的具有不同长度的顶点螺旋(见表2),其根据顶点上的16-螺旋臂的末端之间的距离来估计。关于详细的设计和序列信息,参照图8至图13。“*”表示其中放置DNA柄用于DNA-PAINT的螺旋。图7A-7C。连接模式。(图7A)三臂三脚架单体。(图7B)三臂单体的臂的横截面。在A和B中的箭头指示相同的方向。虚线指示反射对称线。(图7C)在图2B至图2E中实施的连接模式。关于设计和序列的详细信息参见图8至图13。图8。四面体的线图。所使用的序列在表4中提供。水平轴提供了螺旋从其第一碱基的位置或长度。垂直轴提供了螺旋数。如所示的,存在螺旋的三个分组,每一个代表一个臂。在右侧的3个突出物对应于3个支柱。螺旋的右末端代表游离末端,而左末端代表在顶点处的末端。类似的绘制在图9-13中提供。图9。三角柱的线图。所使用的序列在表5中提供。图10。立方体(短连接器)线图。所使用的序列在表6中提供。图11。立方体(长连接器)线图。所使用的序列在表7中提供。图12。五角柱的线图。所使用的序列在表8中提供。图13。六角柱的线图。所使用的序列在表9中提供。图14A-14B。具有N个臂和N或更多个核酸支柱的核酸结构的示意图。发明详述本发明部分地基于用于从兆道尔顿单体使用DNA“三脚架”分层自组装多面体的一般策略的发现和开发,所述DNA“三脚架”是5MD三臂连接折纸瓦片,其是先前的三臂瓦片(16)的60倍大。三脚架模体的特征在于由支撑性支柱控制和由顶点螺旋巩固的臂间角度。本发明进一步提供了使用动态连接器设计将三脚架自组装成线框多面体。通过使用这种稳健的方法,我们构建了四面体(~20MD)、三角柱(~30MD)、立方体(~40MD)、五角柱(~50MD)和六角柱(~60MD)(图1A和图5)。这些结构具有多种用途,包括但不限于生物学应用。例如,当被产生为具有约100nm量级的边缘宽度时,这些多面体的尺寸可相当于细菌微区室例如羧基体。其它应用包括但不限于在光子器件、纳米电子产品和药物递送系统中使用或用作光子器件、纳米电子产品和药物递送系统。为了表征这些多面体的3D单分子形态学,我们使用了基于DNA的超分辨率荧光成像方法(在衍射极限以下的分辨率),被称为DNA-PAINT(28,29)(用于在纳米规模局部图中成像的点积累的变化(30))。不同于传统的透射电子显微镜检查(TEM)(其在干燥和染色条件下在真空中成像样品,从而可能不会使结构以其天然形式呈现),3DDNA-PAINT通过使结构处于更“天然”的水合成像环境中来对结构引入最小的失真。一般性三脚架设计和方法学本文公开了包含最少三个核酸臂(或臂)的核酸结构(可替代地在本文中被称为结构)。这样的三臂结构在本文中被称为三脚架。如将被理解的,假定三脚架的结构,所述三个臂在顶点处彼此相遇并且朝向每个臂的游离末端向外辐射。本公开内容考虑和提供了包含多于三个核酸臂的核酸结构,包括包含四、五、六、七个或更多个臂的结构。在图14中提供了这样的结构的实例。在图14A中,较长较粗的线对应于核酸臂并且较短较细的线对应于核酸支柱。在图14B和C中,仅示出核酸臂,但应理解的是,这样的核酸结构还包含核酸支柱。结构内(或复合结构内)的核酸臂通常具有相同的长度。然而它们不受这样的限制,并根据不同的实施方案长度可以不同。特别重要的和如本文所提供的,核酸臂以彼此固定的角度存在。这是通过使用定位在结构的臂之间的核酸来实现的;这些核酸被称为核酸支柱(或支柱)。每个核酸支柱连接至单一结构中的两个核酸臂,从而保持两个臂之间的角距离。核酸支柱可以定位在沿臂的长度上的任何地方。沿臂长度(从顶点)的支柱位置和支柱的长度一起可影响臂之间的角距离。臂之间的角距离还可部分地由顶点核酸和存在于顶点处的其它连接器(包括核酸连接器相互作用)控制。在表1中提供了许多核酸结构的支柱长度和从顶点沿臂的支柱位置的实例。如根据表格和根据其余公开内容将是清楚的,结构中(或复合结构内)的支柱可具有相同的长度或不同的长度。应当理解的是,根据本文所提供的方法学,可以产生具有在其臂之间的任何特别限定的角距离和任何数量的臂的核酸结构。在这方面,所述结构被认为是“可调”的,因为最终用户能够修改合成方法以获得所选择的结构。为清楚起见结构的臂可以在本文中被称为x、y和z臂,例如在三脚架结构的上下文中。在这种结构中,通常一个(但任选多于一个)支柱连接x和y臂,通常一个(但任选多于一个)支柱连接y和z臂,和通常一个(但任选多于一个)支柱连接z和x臂。再次为了清楚起见,这些支柱可被称为xy支柱、yz支柱和zx支柱。在三脚架的情况下,每个臂连接至结构中的每个其它臂。在具有多于三个臂的结构的情况下,所有的相邻臂将通常通过支柱彼此连接,并且任选地非相邻臂也可以通过支柱彼此连接。可期望的是包括非相邻臂之间的支柱以提供更大的结构完整性。作为实例,在图14A中,所示的第二结构包含四个臂和相邻臂之间的四个支柱。该结构还可以包含非相邻臂之间例如“北侧”和“南侧”臂和/或“西侧”和“东侧”臂之间的额外的支柱,为了解释的目的想象臂是指南针上的方向。因此,臂的最小数目为3,并且支柱的最小数目为3。本内容考虑了具有3个或更多个臂和3个或更多个支柱的结构。支柱的数目通常等于或大于臂的数量。因此,本文提供了核酸结构,所述核酸结构包含第一(x)、第二(y)和第三(z)核酸臂,其各自在一个末端连接至其它臂以形成顶点,和第一、第二和第三核酸支柱,其中所述第一核酸支柱将所述第一(x)核酸臂连接至所述第二(y)核酸臂,所述第二核酸支柱将所述第二(y)核酸臂连接至所述第三(z)核酸臂,并且所述第三核酸支柱将所述第三(z)臂连接至所述第一(x)核酸支柱。本文提供了包含以固定的角度从顶点辐射的三个核酸臂的核酸结构。这样的结构可以具有多于三个臂,包括4、5、6、7个或更多个臂。本文还提供了核酸结构,其包含从顶点辐射的N个核酸臂,其中N是核酸臂的数目并且为3或更多,和M个核酸支柱,每个支柱将两个核酸臂彼此连接,其中M是核酸支柱的数目并且为3或更多。N可以等于M或其可以小于M。实例包括包含4个核酸和至少4个核酸支柱的核酸结构,或包含5个核酸臂和5个核酸支柱的核酸结构。在一些实施方案中,结构内的核酸臂(包括相邻臂)以相等的间隔彼此分开。换句话说,臂以相同的角度彼此分离,或臂之间的角距离是相同的。这样的实例是其中相邻臂通过60℃的角度彼此分离的三臂结构。该三脚架被称为60℃-60℃-60℃。当彼此连接时,这种类型的三脚架将形成四面体。因此,将理解,臂之间的角距离还决定这样的结构将如何彼此连接以及将形成的最终的3D形状(或复合核酸结构)。另一个实例是其中相邻臂通过90℃的角度彼此分离的三臂结构。该三脚架被称为90℃-90℃-90℃。当彼此连接时,这种类型的三脚架将形成立方体。在一些实施方案中,结构内的核酸臂(包括相邻臂)以不相等的间隔彼此分开。换句话说,臂以不同的角度彼此分离,或臂之间的角距离是不同的。这样的实例是其中一些相邻臂通过60℃的角度彼此分离并且其它相邻臂通过90℃的角度彼此分离的三臂结构。这样三脚架可被称为90℃-90℃-60℃。当彼此连接时,这种类型的三脚架将形成三角柱。另一个实例是其中一些相邻臂通过108℃的角度彼此分离并且其它相邻臂通过90℃的角度彼此分离的三臂结构。这样三脚架被称为90℃-90℃-108℃。当彼此连接时,这种类型的三脚架将形成五角柱。另一个实例是其中一些相邻臂通过120℃的角度彼此分离并且其它相邻臂通过90℃的角度彼此分离的三臂结构。这样三脚架被称为90℃-90℃-120℃。当彼此连接时,这种类型的三脚架将形成六角柱。如根据本公开内容将理解的是,核酸结构排列其臂(其臂的三个或更多个)以形成顶点。存在于顶点的臂末端可以通过核酸螺旋或通过核酸连接器(或连接器链)或通过螺旋和连接器链的组合彼此连接。这样的实例在图6中示出。在表2中提供了第一和第二层中的顶点螺旋的长度。通常地0-6个顶点螺旋存在于结构中。因此,该结构可进一步包含顶点核酸例如顶点螺旋。一些复合结构可以不包含顶点螺旋。实例是可以由两个不具有顶点螺旋的三脚架结构的附接形成的四面体。所述结构还可以包含连接器核酸。这些连接器核酸可以位于顶点处和/或臂的游离末端处。在后一种情况下,这样的连接器核酸促进两个核酸结构的彼此附接,从而形成复合的核酸结构。因此结构中的每个核酸臂通常具有位于顶点处的一个末端和一个游离末端(即,不位于顶点处的末端)。游离末端可以是平末端,这意味着它缺乏任何单链核酸序列。或者,它可以是粘性末端,这意味着它包含单链核酸序列。该序列(被称为突出端)的长度可以为1或2个核苷酸。它可能是更长的,尽管1-2个核苷酸是合适的并且在一些情况下可能导致复合核酸更有效的合成(和从而这种复合材料的更大产率)。突出端可通过连接器核酸来提供。这样的连接器核酸可以存在于初始杂交反应中,或者它们可以在核酸结构的合成后添加,进行或不进行合成的结构的纯化。连接器核酸(在本文中也被称为连接器链)可以是任何长度的,虽然已发现较短的长度导致更高的复合核酸结构产率。图2C提供了较长的连接器链的示意图(在具有2个核苷酸突出端的30个核苷酸的量级上)。图2D提供了较短的连接器链的示意图(在具有2个核苷酸突出端的11个核苷酸的量级上)。图2C和2D的结构被用于形成是立方体的复合核酸结构。这样的立方体的产率示于图2E中。顶部线对应于较短的连接器并且底部线对应于较长的连接器。因此,较短的连接器导致其复合立方体的产量较高。虽然不希望受到任何理论的束缚,但使用较长的连接器链的较低的产率可能是因为包含较长连接器链的不匹配复合材料(或不匹配复合中间体)可能是更稳定的,而包含较短连接器的不匹配复合材料(或不匹配复合中间体)可能是较不稳定的从而更容易离解和重新缔合以形成适当匹配的复合材料和复合中间体。如本文所使用的,复合中间体包含形成复合结构所需的核酸结构的子集。例如,如果所需的复合材料是立方体(其需要4个结构),则中间体可以由2或3个结构组成。本公开内容考虑了连接器可以是任何长度,包括50个或更少的核苷酸、40个或更少的核苷酸、30个或更少的核苷酸、25个或更少的核苷酸、20个或更少的核苷酸、15个或更少的核苷酸、10个或更少的核苷酸、或5个或更少的核苷酸的长度。连接器可以为5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20个或更多个核苷酸。核酸结构可以是任何尺寸,尽管通常它们在多至约5兆道尔顿(MD)的范围内。因此,在一些实施方案中它们可以是3、4、5或6MD。核酸臂的长度由所需刚性和由它们的合成方法决定。例如,本文描述的结构具有由16个平行双螺旋构成的臂。由于它们使用DNA折纸技术以M13支架链起始制备,臂的长度通常为约50nm。应理解的是,如果使用不同长度的支架,或者如果臂被设计为具有不同数量的双螺旋(例如,如果期望更多或更少的刚性和强度),则臂的长度可以与本文所述的那些不同。假定核酸结构具有50nm的臂,并假定所有臂长度相等,那么将被理解的是,复合核酸结构将具有100nm数量级的边缘宽度。因此,可能根据本公开内容生成的复合材料可以被定义为具有至少100nm的边缘宽度(120,140,160,180,200nm或更多)。在一些情况下,复合材料可具有80nm或更多的边缘宽度。核酸臂、核酸支柱和顶点核酸可以包含双螺旋例如平行双螺旋。本文举例说明了各自包含16个平行双螺旋的臂,各自包含2个平行双螺旋的支柱,以及各自包含单个双螺旋的顶点核酸。当多于一个双螺旋存在时,通常存在交叉链,其杂交至平行螺旋,从而促进螺旋的靠近和最终其刚性。应进一步理解的是,本文所公开的核酸结构可以使用任何数目的核酸纳米结构物合成方法包括但不限于DNA折纸和DNA单链瓦片(SST)来合成。这些技术在本领域中是已知的,并且在美国专利号7,745,594和7,842,793;美国专利公开号2010/00696621;和Goodman等人NatureNanotechnology中更详细地描述。可使用核酸结构以产生在本文中被称为复合核酸结构(或复合材料或复合结构)的较大的结构。复合结构通过核酸结构的彼此连接形成。通常地,核酸结构在长度和角度定义上是相同的。因此多个相同的核酸结构被组合在单一反应容器中,并使其彼此附着以通过它们的游离臂末端的连接来形成更大的3D结构。这样的连接可通过连接器链的存在(或包含)来促进,尽管合成方法并不限于此。因此,本文公开和提供了复合核酸结构,其包含L个核酸结构,其中L为核酸结构的数目,并且其中所述L个核酸结构在核酸臂的游离(非顶点)末端彼此连接。制备复合材料所需的结构的数目将取决于所需的复合结构和用作组分的结构。在一些情况下,复合结构可包含2、4、6、8、10、12个或更多个核酸结构,其各自具有三个臂。如本文全文所举例说明的,该方法可用于产生四面体、三角柱、立方体、五角柱或六角柱的复合核酸结构。应该理解的是,任何的任意复合结构可以使用本文提供的方法制备。这些复合材料实际上可以是任何大小,包括但不限于。本文举例说明了20兆道尔顿(MD)、30MD、40MD、50MD和60MD大小的复合核酸结构。复合材料可以在核酸结构的产生后立即产生从而在相同的容器中被产生为结构。如果使用的话,连接器链可以存在于杂交反应的开始时或可以在形成结构时和在形成复合材料之前加入。这样的单一反应容器合成被称为“一锅”退火。下文是特定核酸结构和特定复合核酸结构以及它们的合成方法的更详细的和示例性的描述。这些描述意欲是示例性的而非限制本公开内容的范围。例如,应理解的是,虽然许多下文的描述和示例涉及3-臂“三脚架”核酸结构,但该教导内容可以一般化至如本文所描述的任何数量的臂的结构。示例性三脚架设计和方法学多面体的组装策略和三脚架的设计特征。在一锅退火中,支架和订书钉链首先组装成三脚架折纸单体,然后三脚架(无中间纯化)组装成多面体(图1A)。还考虑了三脚架单体可以在最终组装成复合核酸结构之前进行纯化。不同的多面体可以通过使用具有不同设计的臂间角度的三脚架来构造。三脚架具有三个通常长度相等(例如,~50nm)的硬的臂,其以受控的臂间角度在顶点连接(连接的细节参见图6)(图1B)。为了确保刚性,每个臂包含以双重旋转对称在蜂巢晶格(5)上装填的足够数量的(例如,16)的平行双螺旋。由两个双螺旋组成的支撑性“支柱”控制两个臂之间的角度。三脚架根据其三个臂间角度命名(例如四面体和立方体分别由60°-60°-60°和90°-90°-90°三脚架组装)。为了避免潜在的不想要的由DNA螺旋的平末端堆叠(5)引起的聚集,在顶点处包含多至六个短的DNA双螺旋(命名为“顶点螺旋”)以部分地掩盖其双链平末端(图1B;螺旋的数目和它们的长度对于不同的多面体不同,关于详细信息参见图6和表2)。此外,预期顶点螺旋通过增加顶点的刚性来帮助保持臂间的角度。两种连接策略被用于将三脚架组装成多面体。为了便于论述,将三个臂命名为X臂,Y臂,和Z臂(图1C)。连接X臂至X臂和连接Y臂至Z臂产生除四面体以外的多面体(例如立方体;图1D),四面体通过连接X至X、Y至Y和Z至Z来组装(图1E)。使用支柱的三脚架构象控制。首先,我们验证了臂间角度由支撑性支柱的长度控制。60°-60°-60°和90°-90°-90°三脚架的凝胶电泳揭示了每个三脚架的主要条带(图2A),从而确认它们的正确形成。与其更紧凑设计的构象一致,60°-60°-60°三脚架比90°-90°-90°三脚架迁移得略快。将两个三脚架条带各自纯化,通过TEM成像,并显示所设计的三脚架样形态(图2B)。测量的臂间角度略小于所设计的角度(60°-60°-60°三脚架为53±5°[SD,n=60];90°-90°-90°三脚架为87±4°[SD,n=60]),这可能反映了小程度的支柱弯曲。连接器设计。连接三脚架的链被称为“连接器”。连接器设计影响多面体组装产率。对于立方体测试了两个设计。在方案i中,每个30-碱基连接器横跨两个相邻的三脚架,其中28-碱基区段锚定在一个三脚架上并且另外的2-碱基(粘性末端)锚定在另一个三脚架上(图6;细节参见图7)。凝胶电泳(图2E中量化)揭示,组装产率受到连接的螺旋的数量(n)的影响:产物条带仅对于4≤n≤12观察到;对于n<4,主要条带是单体,可能反映了过于弱的单体间连接;对于n>12,主要是聚集体。在方案i中,在发生单体间连接之前将连接器稳定锚定(形成28个碱基对)在三脚架上。在方案ii中,将连接器从30缩短至11个碱基,以使得在组装的立方体中它只能通过9-碱基和2-碱基区段锚定至两个相邻的三脚架(图2D),并且仅动态结合至单体三脚架。与稳定附接的连接器设计相比,动态连接器设计预期减少在组装过程中可能发生的单体间不匹配,因为这样的不匹配不太可能在动态情况下一直存在。事实上,方案ii显示显著增加的组装产率(图2E)。因此它被用于后续的多面体设计(除了四面体以外,其中对于此相对简单的结构方案i产生足够的产率)。组装产率从凝胶估计(图2F)。90°-90°-90°单体样品(图2F,泳道1)显示强的单体条带和推定的二聚体条带(未通过TEM研究,相较于单体~27%的强度)。我们将多面体的组装产率定义为其产物条带强度与90°-90°-90°单体和二聚体条带(泳道1)的组合强度的比率,并获得四面体、三角柱、立方体、五角柱和六角柱分别45%、24%、20%、4.2%和0.11%的产率(图2F)。多面体组装。支柱的长度和连接点对于每个多面体不同(表1)。四面体、三角柱、立方体、五角柱以及六角柱应分别由具有所设计的60°-60°-60°、90°-90°-60°、90°-90°-90°、90°-90°-108°和90°-90°-120°角度的单体组装(图1B)。前三种单体确实产生四面体、三角柱和立方体[由凝胶电泳(图2F)和TEM成像(图3,A至C)核实],表明角度准确控制在90°内。然而,五角柱从具有90°-90°-120°(而非90°-90°-108°)的设计角度的单体组装,并且六角柱从90°-90°-140°(而非90°-90°-120°)组装。因此,这两个多面体的组装要求具有比设计标准更大的所设计的Y-Z角度的单体。这一要求可能反映了相关支柱的轻微弯曲,这可以通过使用较长的支柱来补偿。支柱和顶点螺旋对多面体组装的影响。接下来我们验证三脚架需要支柱和顶点螺旋来组装成设计的多面体。使用包含(i)支柱和顶点螺旋(图2F,泳道4)、(ii)顶点螺旋而非支柱(泳道7)和(iii)支柱而非顶点螺旋(泳道8;在退火后将样品进行凝胶电泳)的三脚架制备用于立方体组装的三个样品。第一样品显示对应于立方体的清晰强条带(通过TEM验证,图3B)。第二个没有产生任何明显的产物条带。第三个产生显著的聚集物和具有与三角柱相当的迁移率的清楚但弱的条带。该条带可对应于六聚物,但其分子形态未被研究。基于上述的实验,我们在三脚架中包含支柱和顶点螺旋用于随后的多面体组装。TEM表征。将产物条带纯化并在TEM下成像。对于四面体、三角柱和立方体,大多数结构出现为完整的多面体;小部分的破损结构(<20%)可能在纯化和成像期间破裂(图3,A至C)。相反,对于纯化的五角柱和六角柱观察到少量完整的结构(数据未显示)。因此,对于这两个的未纯化样品被直接成像并观察到预期的分子形态(对于示例性的图像,图3,D和E,还有另外的图像但未示出)。支柱在许多图像中清晰可见。3DDNA-PAINT超分辨率显微镜检查。基于定位的3D超高分辨率荧光显微镜检查(31-33)提供了最低限度侵袭的方式来获得DNA纳米结构在其“天然”水合环境中的真正的单分子3D图像。在随机重建显微镜检查(34)中,大多数的分子被切换至荧光暗(OFF)状态,只有少数发射荧光(ON状态)。每个分子通过将其发射拟合至2D高斯函数来以纳米精度定位。在DNA-PAINT中,ON-和OFF-状态之间的“转换”通过荧光标记的寡核苷酸(“成像”链)与互补“对接”链的反复、短暂结合来促进(24,28,29,35)。我们通过使用光学散光(31,36)将DNA-PAINT扩展至3D成像(29),其中当散焦成像时在成像路径中使用的柱面透镜将分子的球面点扩散函数(PSF)“转换”至椭圆PSF。椭圆PSF的程度和方向取决于点源从当前焦点成像平面的位移和方向,并用于确定其z位置(31,36)。我们采用3DDNA-PAINT来获得多面体的亚衍射分辨率单分子图像。为了确保将成像多面体的所有顶点,用对称排列的多个(约18个)9-nt对接链(订书钉-TTATCTACATA-3’;SEQIDNO:1)(图4A1)修饰每个顶点(图6)。对于表面固定,沿多面体边缘的一个子集的链用21-nt的延长序列(订书钉TTCGGTTGTACTGTGACCGATTC-3';SEQIDNO:2)修饰,其被杂交至附接至抗生蛋白链菌素覆盖的载玻片的生物素化的互补链(生物素-GAATCGGTCACAGTACAACCG-3';SEQIDNO:3)。通过使用3DDNA-PAINT显微镜检查,所有五个多面体显示具有预期高度(图4,A5-E5)的顶点的所设计的3D模式(图4,第1-4栏),这表明结构的解析形状在表面固定和成像期间得到保持。我们量化了四面体形成和成像产率(图4,F和G)。285个结构的253个(89%)包含预期四面体几何学中的4个点。高度测量得到82±15nm,与设计值(82nm)是一致的。单个DNA-PAINT结合事件以x-y中的5.4nm和z中的9.8nm的精度被定位[对于如何确定定位精度参见下文]。该z定位精度几乎完全占据了高度测量分布中的15nm扩展。计算出的定位精度转换为可获得的在x和y中的~13nm和在z中的~24nm的分辨率。以往的研究显示了从小的3-臂-连接瓦片(~80kD)(其由通过柔性的单链铰链连接的三个双螺旋臂组成)自组装的多样的DNA多面体(16)。然而,使用类似的柔性臂间铰链的兆道尔顿3-臂折纸瓦片(即没有支柱或顶点螺旋的三脚架)的直接应用没有产生良好形成的多面体(图2B,泳道7)。折纸三脚架包含比先前的3-臂-连接瓦片(从3个不同链形成)50倍更多的不同的链并且分子量大60倍。除了与从个体链更容易出错地构建更复杂的单体相关的挑战,这样的大单体至多面体的成功分层组装还需要克服慢得多的反应动力学(由三脚架单体的较大尺寸和较低的浓度引起)。硬的DNA三脚架(具有由支撑性支柱和顶点螺旋控制的合理设计的臂间角度)导致不同多面体的成功构建,这表明分支的兆道尔顿单体的构象控制能够促进它们成功组装成较高阶的结构。DNA三脚架的设计原理可以扩展至具有受控的臂间角度的硬的兆道尔顿n-臂(n≥4)分支模体。使用这样的n-臂模体的自组装可用于构建更复杂的多面体和潜在地扩展的具有低于100nm的可调腔的2D和3D晶格。这样的结构可潜在地用于针对不同应用的模板客体分子,例如空间排列多种酶至有效的反应级联(37)或纳米颗粒以实现有用的光子性质(38,39)。此外,此处构建的具有与细菌微区室相当的大小的DNA多面体可以潜在地用作骨架用于通过在其外表面周围包装脂膜制备具有精确控制的维度和形状的区室(40)。这样的膜包封微区室可潜在地用作生物反应器用于合成有用的产品或用作治疗货物的运载工具(25)。对于DNA纳米结构的3D表征,超分辨率荧光显微镜检查(例如3DDNA-PAINT)为现有的电子显微镜(例如cryo-EM(12,16,17,23))提供补充能力。虽然cryo-EM提供未标记的结构的更高的空间分辨率成像,但DNA-PAINT的实施牵涉较少的技术,其获得个体结构的真正的单分子图像(而不是依赖于类别平均),并保留荧光显微镜的多色能力(29)。此外,DNA-PAINT原则上允许观察纳米结构在其“天然”水合环境中的动态结构变化,目前适合用于在分钟时间尺度上的缓慢变化(例如合成DNA行走的移位)和潜在地用于具有进一步发展的较快的运动。表1。多面体的支柱设计。所有单位均为纳米。连接(i)Y臂和Z臂、(ii)X臂和Z臂或(iii)X臂和Y臂的支柱的设计长度。从顶点到(iv)X臂、(v)Y臂或(vi)Z臂上的支柱连接点的设计距离。iiiiiiivvvi四面体282828292929三角柱182626181818立方体303030212121五角柱322626191818六角柱372828202020表2核酸纳米结构一般方法学可以使用任何核酸折叠或杂交方法来形成本文中提供的核酸结构。一种这样的方法是DNA折纸(Rothemund,2006,Nature,440:297-302,其全文通过引用并入本文)。在DNA折纸方法中,结构通过较长的“支架”核酸链经由其杂交至多个较短的“订书钉”寡核苷酸(其每一个杂交至支架链内的两个或更多的非连续区域)的折叠产生。在一些实施方案中,支架链的长度为至少100个核苷酸。在一些实施方案中,支架链的长度为至少500,至少1000,至少2000,至少3000,至少4000,至少5000,至少6000,至少7000,或至少8000个核苷酸。支架链可以是天然或非天然存在的。通常使用M13mp18病毒基因组DNA(其为约7kb)中的支架。可使用其它单链支架,包括例如λ基因组DNA。订书钉链的长度通常小于100个核苷酸;然而,取决于应用并且取决于支架链的长度它们可以更长或者更短。在一些实施方案中,订书钉链的长度可以是约15至约100个核苷酸。在一些实施方案中,订书钉链的长度为约25至约50个核苷酸。在一些实施方案中,核酸结构可以在不存在支架链的情况下组装(例如,无支架结构)。例如,可组装许多寡核苷酸(例如,长度<200个核苷酸或小于100个核苷酸)以形成核酸纳米结构。这种方法在WO2013/022694和WO2014/018675(其每一个通过引用以其全文并入本文)中描述。用于组装核酸结构的其它方法在本领域中是已知的,其中的任何一个可被用于本文中。(参见例如KuzuyaandKomiyama,2010,Nanoscale,2:310-322)。还应理解,这些方法的组合或混合也可以用于产生本文所公开的核酸结构。这些方法可以基于本文提供的教导进行修改以获得本公开内容的固定角度的核酸结构。核酸核酸结构可以包含天然存在的和/或非天然存在的核酸。如果是天然存在的,核酸可以从天然来源中分离,或者它们可以被合成脱离其天然存在的来源。非天然存在的核酸是合成的。术语“核酸”、“寡核苷酸”和“链”可互换使用来表示以连续的方式彼此连接的多个核苷酸。核苷酸是包含与磷酸基团和可交换的有机碱基连接的糖(例如,脱氧核糖)的分子,所述碱基可以是嘧啶(例如,胞嘧啶(C),胸苷(T)或尿嘧啶(U))或嘌呤(例如,腺嘌呤(A)或鸟嘌呤(G))。在一些实施方案中,核酸可以是L-DNA。在一些实施方案中,核酸不是RNA或寡核糖核苷酸。在这些实施方案中,核酸结构可以被称为DNA结构。然而DNA结构仍然可包含碱基、糖和主链修饰。修饰核酸结构可以由DNA、修饰的DNA以及它们的组合制成。用于产生核酸结构或存在于核酸结构中的寡脱氧核糖核苷酸(本文中也称为寡核苷酸,并且其可以是订书钉链、连接器链等)可具有同质或异质(即,嵌合)主链。主链可以是天然存在的主链,例如磷酸二酯主链,或它可包含主链修饰。在一些情况下,主链修饰由于降低的核酸酶介导的降解而导致寡核苷酸的较长的半衰期。这进而引起较长的半衰期。合适的主链修饰的实例包括但不限于:硫代磷酸酯修饰,二硫代磷酸酯修饰,对乙氧基修饰,甲基膦酸酯修饰,甲基硫代磷酸酯修饰,烷基-和芳基-磷酸酯(其中带电荷的膦酸酯氧被烷基或芳基取代),烷基磷酸三酯(其中带电荷的氧部分是烷基化的),肽核酸(PNA)主链修饰,锁核酸(LNA)主链修饰,和类似的。这些修饰可以彼此组合使用和/或与磷酸二酯主链键组合使用。可替代地或另外地,寡核苷酸可以包含其它修饰,包括碱基或糖部分的修饰。实例包括具有共价附着至低分子量有机基团而非在3’位置上的羟基且非在5’位置上的磷酸基团的糖的核酸(例如2'-O-烷基化的核糖),具有糖例如阿拉伯糖而非核糖的核酸。核酸还包括取代的嘌呤和嘧啶例如C-5丙炔修饰的碱基(Wagner等人,NatureBiotechnology14:840-844,1996)。其它的嘌呤和嘧啶包括但不限于5-甲基胞嘧啶,2-氨基嘌呤,2-氨基-6-氯嘌呤,2,6-二氨基嘌呤,次黄嘌呤。其它这样的修饰是本领域的技术人员公知的。修饰的主链例如硫代磷酸酯可以使用采用氨基磷酸酯或H-膦酸酯化学的自动化技术合成。芳基-和烷基-膦酸酯可以例如如美国专利号4,469,863中所述的制备,并且烷基磷酸三酯(其中带电荷的氧部分是烷基化的,如在美国专利号5,023,243和欧洲专利号092574中所述的)可以通过自动固相合成使用市售试剂来制备。用于制备其它DNA主链修饰和取代的方法已被描述(Uhlmann,E.andPeyman,A.,Chem.Rev.90:544,1990;Goodchild,J.,BioconjugateChem.1:165,1990)。核酸可以使用任意数量的本领域已知的方法来从头合成,包括,例如,b-氰乙基亚磷酰胺法(BeaucageandCaruthersTet.Let.22:1859,1981),和核苷H-膦酸酯法(Garegg等人,Tet.Let.27:4051-4054,1986;Froehler等人,Nucl.Acid.Res.14:5399-5407,1986;Garegg等人,Tet.Let.27:4055-4058,1986,Gaffney等人,Tet.Let.29:2619-2622,1988)。这些化学法可以通过在市场上可获得的各种自动化核酸合成仪来进行。这些核酸被称为合成核酸。修饰和未修饰的核酸也可以从商业来源例如IDT和Bioneer购买。分离的核酸一般是指核酸与其通常天然关联的组分是分离的。作为实例,分离的核酸可以是从细胞、从细胞核、从线粒体或从染色质分离的核酸。核酸结构和复合核酸结构可以是分离的和/或纯化的。如本文所使用的“分离”是指所需的实体(例如,核酸结构等)从其通常或天然存在的环境中或从其被产生于环境中物理分离。分离可以是部分或完全的。核酸结构的分离可以通过在凝胶上电泳杂交反应混合物并分离在特定的分子量上迁移从而与核酸底物和杂交反应的伪产物区分开的核酸结构来进行。作为另一实例,核酸结构的分离可以利用浮力密度梯度、沉降梯度离心或通过过滤装置来进行。试剂复合核酸结构可以包含旨在用于在体内和/或体外、在生物或非生物应用中使用的试剂。例如,试剂可以是可以被用来给受试者提供益处(包括但不限于预防或治疗益处)、或者可用于体内诊断和/或检测(例如,成像)、或可以用于在体外环境(例如,组织或器官培养,清除过程,等等)中发挥作用的任何原子、分子或化合物。试剂可以是但不限于治疗剂和诊断剂。用于与本文所描述的任一实施方案使用的试剂的实例在下文描述。在一些方面,复合核酸结构被用于全身递送药剂或递送至局部区域,例如组织或细胞。任何试剂可以使用本发明的方法递送,只要它可以被装载到复合结构中。试剂可以是但不限于化学化合物,包括小分子,蛋白质,多肽,肽,核酸,病毒样颗粒,类固醇,蛋白聚糖,脂质,碳水化合物,和类似物,以及其衍生物、混合、融合物、组合或缀合物。试剂可以是被代谢从而在体内转化成其活性(和/或稳定)形式的前药。本发明还考虑了多于一个类型的试剂在复合结构中的装载和/或组合使用包含不同试剂的复合结构。一类试剂是基于肽的试剂例如(单或多链)蛋白质和肽。基于肽的试剂的实例包括但不限于抗体,单链抗体,抗体片段,酶,辅因子,受体,配体,转录因子和其它调节因子,一些抗原(如下面讨论的),细胞因子,趋化因子,激素,和类似物。另一类试剂包括非天然存在的化学化合物。当前用于治疗或诊断目的的各种试剂包括但不限于成像剂,免疫调节剂例如免疫刺激剂和免疫抑制剂(例如环孢菌素),抗原,佐剂,细胞因子,趋化因子,抗癌剂,抗感染剂,核酸,抗体及其片段,融合蛋白例如细胞因子-抗体融合蛋白、Fc-融合蛋白,镇痛药,阿片类药物,酶抑制剂,神经毒素,安眠药,抗组胺剂,润滑剂,镇静剂,抗惊厥药,肌肉松弛剂,抗帕金森剂,抗痉挛剂,肌肉收缩剂包括通道阻断剂、缩瞳药和抗胆碱能药,抗青光眼化合物,细胞-细胞外基质相互作用的调节剂包括细胞生长抑制剂和抗粘附分子,血管扩张剂,DNA、RNA或蛋白质合成的抑制剂,抗高血压药,解热剂,甾体和非甾体抗炎剂,抗血管生成因子,抗分泌因子,抗凝血剂和/或抗血栓剂,局部麻醉剂,眼药,前列腺素,靶向试剂,神经递质,蛋白质,细胞响应调节剂和疫苗。在一些实施方案中,试剂是诊断剂,例如成像剂。如本文中所使用的,成像剂是直接或间接发出信号从而允许其在体内检测的试剂。成像剂例如造影剂和放射性试剂可以使用医学成像技术例如核医学扫描和磁共振成像(MRI)来检测。用于磁共振成像(MRI)的成像剂包括Gd(DOTA),氧化铁或金纳米颗粒;用于核医学的成像剂包括201Tl,发射γ射线的放射性核素99mTc;用于正电子发射断层扫描(PET)的成像剂包括正电子发射同位素,(18)F-氟脱氧葡萄糖((18)FDG),(18)F-氟化物,铜-64,gadoamide和Pb(II)的放射性同位素例如203Pb,和11In;用于体内荧光成像的成像剂例如为荧光染料或染料缀合的纳米颗粒。本公开内容进一步提供了以下编号的实施方案:1.一种核酸结构,其包含第一(x)、第二(y)和第三(z)核酸臂,其各自在一个末端连接至其它臂以形成顶点,和第一、第二和第三核酸支柱,其中所述第一核酸支柱将所述第一(x)核酸臂连接至所述第二(y)核酸臂,所述第二核酸支柱将所述第二(y)核酸臂连接至所述第三(z)核酸臂,并且所述第三核酸支柱将所述第三(z)臂连接至所述第一(x)核酸支柱。2.一种核酸结构,其包含以固定的角度从顶点辐射的三个核酸臂。3.一种核酸结构,其包含从顶点辐射的N个核酸臂,其中N是核酸臂的数目并且为3或更多,和M个核酸支柱,每个支柱将两个核酸臂彼此连接,其中M是核酸支柱的数目并且为3或更多。4.实施方案3的核酸结构,其中N等于M。5.实施方案3的核酸结构,其中N小于M。6.实施方案1-5的任一个的核酸结构,其中所述核酸结构包含4个核酸和至少4个核酸支柱,或5个核酸臂和5个核酸支柱。7.实施方案1-6的任一个的核酸结构,其中所述核酸臂以相等的间隔彼此分开(或所述臂以相同的角度彼此分离)。8.实施方案1-7的任一个的核酸结构,其中所述核酸臂以不相等的间隔彼此分开(或所述臂以不同的角度彼此分离)。9.实施方案1-8的任一个的核酸结构,其还包含顶点核酸。10.实施方案1-9的任一个的核酸结构,其还包含连接器核酸。11.实施方案1-10的任一个的核酸结构,其中所述核酸臂、核酸支柱和/或顶点核酸包含平行的双螺旋。12.实施方案1-11的任一个的核酸结构,其中所述核酸臂的长度相同。13.实施方案1-12的任一个的核酸结构,其中所述核酸支柱的长度相同。14.实施方案1-13的任一个的核酸支柱,其中所述核酸支柱的长度不同。15.实施方案1-14的任一个的核酸结构,其中至少一个核酸臂包含平末端。16.实施方案1-15的任一个的核酸结构,其中至少一个核酸臂在其游离(非顶点)末端包含连接器核酸,所述连接器核酸的长度为多至16个核苷酸。17.实施方案1-16的任一个的核酸结构,其中至少一个核酸臂在其游离(非顶点)末端包含连接器核酸,从而包含1或2个核苷酸突出端。18.实施方案1-17的任一个的核酸结构,其中所述核酸结构的大小为多至5兆道尔顿(MD)。19.实施方案1-18的任一个的核酸结构,其中所述核酸臂的长度为50nm。20.实施方案1-19的任一个的核酸结构,其中所述核酸结构包含以60°-60°-60°彼此分离的三个核酸臂(四面体)。21.实施方案1-20的任一个的核酸结构,其中所述核酸结构包含以60°-90°-90°彼此分离的三个核酸臂(三角柱)。22.实施方案1-21的任一个的核酸结构,其中所述核酸结构包含以90°-90°-90°彼此分离的三个核酸臂(立方体)。23.实施方案1-22的任一个的核酸结构,其中所述核酸结构包含以108°-90°-90°彼此分离的三个核酸臂(五角柱)。24.实施方案1-23的任一个的核酸结构,其中所述核酸结构包含以120°-90°-90°彼此分离的三个核酸臂(六角柱)。25.一种复合核酸结构,其包含选自实施方案1-24任一个的核酸结构的L个核酸结构,其中L为核酸结构的偶数目,并且其中所述L个核酸结构在核酸臂的游离(非顶点)末端彼此连接。26.实施方案25的复合核酸结构,其中两个以上的核酸结构是2、4、6、8、10、12个或更多个核酸结构。27.实施方案25或26的复合核酸结构,其中所述复合核酸结构是四面体、三角柱、立方体、五角柱或六角柱。28.实施方案25-27的任一个的复合核酸结构,其中所述复合核酸结构的大小为20兆道尔顿(MD)、30MD、40MD、50MD或60MD。29.实施方案25-28的任一个的复合核酸结构,其中所述复合核酸结构具有100nm的边缘宽度,包含来自相邻核酸结构的两个核酸臂。实施例材料和样品制备。DNA链通过IntegratedDNATechnology,Inc.或BioneerCorporation合成。为了组装结构,在补充有12mMMgCl2的0.5×TE缓冲液(5mMTris,pH7.9,1mMEDTA)中以10:1的摩尔化学计量比混合未纯化的100μΜDNA链与p8064支架。p8064支架的最终浓度调节到10nM。Cy3b修饰的DNA寡核苷酸购自Biosynthesis(Lewisville,TX)(5’-TATGTAGATC-Cy3b;SEQIDNO:4)。抗生蛋白链菌素购自Invitrogen(S-888,Carlsbad,CA)。从SigmaAldrich(A8549,St.Louis,MO)获得牛血清白蛋白(BSA)和BSA-生物素。载玻片和盖玻片从VWR(Radnor,PA)购买。两个缓冲液被用于样品制备和用于超分辨率DNA-PAINT成像的成像:缓冲液A(10mMTris-HCl,100mMNaCl,0.05%Tween-20,pH7.5),缓冲液B(5mMTris-HCl,10mMMgCl2,1mMEDTA,0.05%Tween-20,pH8)。退火坡道。随后将链混合物在PCR热循环仪中使用在1小时内从80℃至65℃的快速线性冷却步骤进行退火,然后进行从64℃至24℃的42小时线性冷却坡道。琼脂糖凝胶电泳。退火样品在0.5%TBE缓冲液(包含10mM的MgCl2)中于90V在冰水浴中凝胶电泳3小时。凝胶在成像前使用Safe染色。TEM成像。对于成像,将2.5μL退火样品吸附至辉光放电、碳涂覆的TEM格栅上进行2分钟。然后使用含有25mMNaOH的2%甲酸双氧铀水溶液对格栅染色10秒。成像使用在80kV操作的JEOLJEM-1400TEM进行。超高分辨率成像。荧光成像在倒置的NikonEclipseTi显微镜(NikonInstruments,Melville,NY)上使用PerfectFocusSystem进行,应用使用具有油浸物镜(CFIApoTIRF100,NA1.49,Oil)的NikonTIRF照明器的物镜型TIRF配置。对于Cy3b激发,使用561nm激光(名义200mW,CoherentSapphire)。使激光束通过净化滤光器(ZET561/10,ChromaTechnology,BellowsFalls,VT)并偶联至使用多频带分束器(ZT488rdc/ZT561rdc/ZT640rdc,ChromaTechnology)的显微镜物镜中。荧光使用发射滤光器(ET600/50m,ChromaTechnology)光谱过滤并在EMCCD相机(iXonX3DU-897,AndorTechnologies,NorthIreland)上成像。成像在检测路径中无额外放大的情况下进行,产生160nm像素大小。样品制备和成像。对于样品制备,将一块盖玻片(No.1.5,18x18mm2,0.17mm厚)和载玻片(3x1英寸2,1mm厚)通过两条双面胶带夹心在一起,以形成具有20μL内部体积的流动室。首先,将20μL生物素标记的牛白蛋白(1mg/mL,溶解于缓冲液A中)流入腔室中并孵育2分钟。然后用40μL缓冲液A洗涤腔室。随后使20μL抗生蛋白链菌素(0.5mg/mL,溶解于缓冲液A中)流过腔室并允许结合2分钟。用40μL缓冲液A和随后用40μL缓冲液B洗涤后,最后使20μL在缓冲液B中的生物素标记的微管样DNA结构(≈300pM单体浓度)和DNA折纸漂移标记物(≈100pM)流入腔室并孵育5分钟。使用40μL缓冲器B洗涤腔室。最终的成像缓冲溶液包含在缓冲液B中的3nMCy3b标记的成像链。在随后的成像前使用环氧树脂密封腔室。CCD读出带宽被设置至14比特下的3MHz和5.1前置放大增益。没有使用EM增益。使用具有561nm处的~200W/cm2的激发强度的倾斜照明进行成像。3D图像使用检测路径中的柱面透镜(Nikon)获得。所有图像从以200ms积分时间获得的5000帧长的时间推移影片重建,导致≈17分钟的成像时间。图像处理和漂移校正。超分辨率DNA-PAINT图像使用斑点发现和编程在LabVIEW(Jungmann,R.,等人NatureMethods,提前在线发表,2014)中的2DGaussian拟合算法重建。该软件的简化版本可在“dna-paint”网站下载。将用于NISElements(Nikon)的N-STORM分析软件包用于数据处理。3D校准根据制造商的说明进行。DNA折纸漂移标记物(Lin,C.,等人NatureChemistry4,832-839,2012)用作基准标记物。高的结合位点密度增加观察每个图像帧中的每结构的一个结合的成像链的概率。另外,折纸漂移标记物的荧光强度类似于单个成像链结合事件并且标记物从不“漂除相片银影(bleach)”。这些特性使得DNA折纸结构成为理想的漂移标记物。漂移校正通过跟踪每个折纸漂移标记物结构在每个影片的整个持续时间内的位置来进行。随后平均所有检测的漂移标记物的轨迹,并用来校正最终超分辨率重建中的漂移。定位精度的测定。将拟合1D-高斯函数至从DNA折纸漂移标志物的z定位的分布和计算标准偏差用来确定z中的定位精度。折纸漂移标记物是2D结构,所有的结合事件发生在表面上的2D平面中,并因此在相同的z定位上。在x和y中的定位精度通过计算在相邻帧中的单分子定位的平均间隔来确定,这可以归因于成像链与单个对接链的结合。因为多个对接链用于多面体的各顶点中(每顶点~18条链),不能拟合每顶点的结合事件的分布,因为这会导致定位精度的过高估计。每个顶点的测量值会代表实际定位精度与在该顶点中的结合位点的空间幅度的卷积。空间相对时间的成像分辨率。在随机超分辨率显微镜检查例如DNA-PAINT中,可以通常认为的是,空间和时间分辨率之间存在折衷。更高的空间分辨率可通过收集每结合或光电开关事件的光子的较大量而获得。这可以通过增加荧光ON时间和将照相机积分时间匹配于这些ON时间来实现。在DNA-PAINT成像中,这可以通过增加成像/对接复合物的结合稳定性(即从9-nt相互作用区增加至10-nt相互作用区)和增加照相机积分时间以匹配较长的结合时间(这进而导致较长的图像采集时间)来完成。更高的时间分辨率可以通过降低成像/对接复合物的结合稳定性(即从9-nt相互作用区降低至8-nt相互作用区)和减小照相机积分时间以匹配较短的结合时间来获得。表3用于超分辨率DNA-PAINT成像的序列:表4四面体的序列。表5三角柱的序列表6具有长连接器订书钉的立方体的序列表7具有短的连接器订书钉的立方体的序列表8五角柱的序列表9六角柱的序列参考文献1.J.H.Chen,N.C.Seeman,SynthesisfromDNAofamoleculewiththeconnectivityofacube.Nature350,631-633(1991).2.E.Winfree,F.Liu,L.A.Wenzler,N.C.Seeman,Designandself-assemblyoftwo-dimensionalDNAcrystals.Nature394,539-544(1998).3.P.W.Rothemund,N.Papadakis,E.Winfree,Algorithmicself-assemblyofDNASierpinskitriangles.PLoSbiology2,e424(2004).4.P.W.Rothemund,FoldingDNAtocreatenanoscaleshapesandpatterns.Nature440,297-302(2006).5.S.M.Douglas,H.Dietz,T.Liedl,B.Hogberg,F.Graf,W.M.Shih,Self-assemblyofDNAintonanoscalethree-dimensionalshapes.Nature459,414-418(2009).6.J.Zheng,J.J.Birktoft,Y.Chen,T.Wang,R.Sha,P.E.Constantinou,S.L.Ginell,C.Mao,N.C.Seeman,Frommoleculartomacroscopicviatherationaldesignofaself-assembled3DDNAcrystal.Nature461,74-77(2009).7.B.Wei,M.Dai,P.Yin,Complexshapesself-assembledfromsingle-strandedDNAtiles.Nature485,623-626(2012).8.Y.Ke,L.L.Ong,W.M.Shih,P.Yin,Three-dimensionalstructuresself-assembledfromDNAbricks.Science338,1177-1183(2012).9.D.Han,S.Pal,Y.Yang,S.Jiang,J.Nangreave,Y.Liu,H.Yan,DNAgridironnanostructuresbasedonfour-armjunctions.Science339,1412-1415(2013).10.V.Linko,H.Dietz,TheenabledstateofDNAnanotechnology.Currentopinioninbiotechnology24,555-561(2013).11.Y.Zhang,N.C.Seeman,ConstructionofaDNA-TruncatedOctahedron.JournaloftheAmericanChemicalSociety116,1661-1669(1994).12.W.M.Shih,J.D.Quispe,G.F.Joyce,A1.7-kilobasesingle-strandedDNAthatfoldsintoananoscaleoctahedron.Nature427,618-621(2004).13.R.P.Goodman,I.A.Schaap,C.F.Tardin,C.M.Erben,R.M.Berry,C.F.Schmidt,A.J.Turberfield,RapidchiralassemblyofrigidDNAbuildingblocksformolecularnanofabrication.Science310,1661-1665(2005).14.F.A.Aldaye,H.F.Sleiman,Modularaccesstostructurallyswitchable3DdiscreteDNAassemblies.JournaloftheAmericanChemicalSociety129,13376-13377(2007).15.C.M.Erben,R.P.Goodman,A.J.Turberfield,Aself-assembledDNAbipyramid.JournaloftheAmericanChemicalSociety129,6992-6993(2007).16.Y.He,T.Ye,M.Su,C.Zhang,A.E.Ribbe,W.Jiang,C.Mao,Hierarchicalself-assemblyofDNAintosymmetricsupramolecularpolyhedra.Nature452,198-201(2008).17.C.Zhang,M.Su,Y.He,X.Zhao,P.A.Fang,A.E.Ribbe,W.Jiang,C.Mao,Conformationalflexibilityfacilitatesself-assemblyofcomplexDNAnanostructures.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica105,10665-10669(2008).18.J.Zimmermann,M.P.Cebulla,S.Monninghoff,G.vonKiedrowski,Self-assemblyofaDNAdodecahedronfrom20trisoligonucleotideswithC(3h)linkers.AngewandteChemie47,3626-3630(2008).19.E.S.Andersen,M.Dong,M.M.Nielsen,K.Jahn,R.Subramani,W.Mamdouh,M.M.Golas,B.Sander,H.Stark,C.L.Oliveira,J.S.Pedersen,V.Birkedal,F.Besenbacher,K.V.Gothelf,J.Kjems,Self-assemblyofananoscaleDNAboxwithacontrollablelid.Nature459,73-76(2009).20.Y.Ke,J.Sharma,M.Liu,K.Jahn,Y.Liu,H.Yan,ScaffoldedDNAorigamiofaDNAtetrahedronmolecularcontainer.Nanoletters9,2445-2447(2009).21.D.Bhatia,S.Mehtab,R.Krishnan,S.S.Indi,A.Basu,Y.Krishnan,IcosahedralDNAnanocapsulesbymodularassembly.AngewandteChemie48,4134-4137(2009).22.H.Yang,C.K.McLaughlin,F.A.Aldaye,G.D.Hamblin,A.Z.Rys,I.Rouiller,H.F.Sleiman,Metal-nucleicacidcages.Naturechemistry1,390-396(2009).23.C.Zhang,S.H.Ko,M.Su,Y.Leng,A.E.Ribbe,W.Jiang,C.Mao,SymmetrycontrolsthefacegeometryofDNApolyhedra.JournaloftheAmericanChemicalSociety131,1413-1415(2009).24.D.M.Smith,V.Schuller,C.Forthmann,R.Schreiber,P.Tinnefeld,T.Liedl,AstructurallyvariablehingedtetrahedronframeworkfromDNAorigami.Journalofnucleicacids2011,360954(2011).25.S.M.Douglas,I.Bachelet,G.M.Church,Alogic-gatednanorobotfortargetedtransportofmolecularpayloads.Science335,831-834(2012).26.Z.Nie,X.Li,Y.Li,C.Tian,P.Wang,C.Mao,Self-assemblyofDNAnanoprismswithonlytwocomponentstrands.Chemicalcommunications49,2807-2809(2013).27.MaterialsandmethodsareavailableassupplementarymaterialonScienceOnline.28.R.Jungmann,C.Steinhauer,M.Scheible,A.Kuzyk,P.Tinnefeld,F.C.Simmel,Single-moleculekineticsandsuper-resolutionmicroscopybyfluorescenceimagingoftransientbindingonDNAorigami.Nanoletters10,4756-4761(2010).29.R.Jungmann,M.S.Avendano,J.B.Woehrstein,M.Dai,W.M.Shih,P.Yin,Multiplexed3Dcellularsuper-resolutionimagingwithDNA-PAINTandExchange-PAINT.Naturemethods,(2014).30.A.Sharonov,R.M.Hochstrasser,Wide-fieldsubdiffractionimagingbyaccumulatedbindingofdiffusingprobes.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica103,18911-18916(2006).31.B.Huang,W.Wang,M.Bates,X.Zhuang,Three-dimensionalsuper-resolutionimagingbystochasticopticalreconstructionmicroscopy.Science319,810-813(2008).32.G.Shtengel,J.A.Galbraith,C.G.Galbraith,J.Lippincott-Schwartz,J.M.Gillette,S.Manley,R.Sougrat,C.M.Waterman,P.Kanchanawong,M.W.Davidson,R.D.Fetter,H.F.Hess,Interferometricfluorescentsuper-resolutionmicroscopyresolves3Dcellularultrastructure.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica106,3125-3130(2009).33.J.J.Schmied,C.Forthmann,E.Pibiri,B.Lalkens,P.Nickels,T.Liedl,P.Tinnefeld,DNAorigaminanopillarsasstandardsforthree-dimensionalsuperresolutionmicroscopy.Nanoletters13,781-785(2013).34.S.W.Hell,Far-fieldopticalnanoscopy.Science316,1153-1158(2007).35.C.Lin,R.Jungmann,A.M.Leifer,C.Li,D.Levner,G.M.Church,W.M.Shih,P.Yin,Submicrometregeometricallyencodedfluorescentbarcodesself-assembledfromDNA.Naturechemistry4,832-839(2012).36.H.P.Kao,A.S.Verkman,Trackingofsinglefluorescentparticlesinthreedimensions:useofcylindricalopticstoencodeparticleposition.Biophysicaljournal67,1291-1300(1994).37.J.Fu,M.Liu,Y.Liu,N.W.Woodbury,H.Yan,InterenzymesubstratediffusionforanenzymecascadeorganizedonspatiallyaddressableDNAnanostructures.JournaloftheAmericanChemicalSociety134,5516-5519(2012).38.G.P.Acuna,F.M.Moller,P.Holzmeister,S.Beater,B.Lalkens,P.Tinnefeld,FluorescenceenhancementatdockingsitesofDNA-directedself-assemblednanoantennas.Science338,506-510(2012).39.A.Kuzyk,R.Schreiber,Z.Fan,G.Pardatscher,E.M.Roller,A.Hogele,F.C.Simmel,A.O.Govorov,T.Liedl,DNA-basedself-assemblyofchiralplasmonicnanostructureswithtailoredopticalresponse.Nature483,311-314(2012).40.M.Langecker,V.Arnaut,T.G.Martin,J.List,S.Renner,M.Mayer,H.Dietz,F.C.Simmel,SyntheticlipidmembranechannelsformedbydesignedDNAnanostructures.Science338,932-936(2012).当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1