用于评估肺癌状态的方法与流程
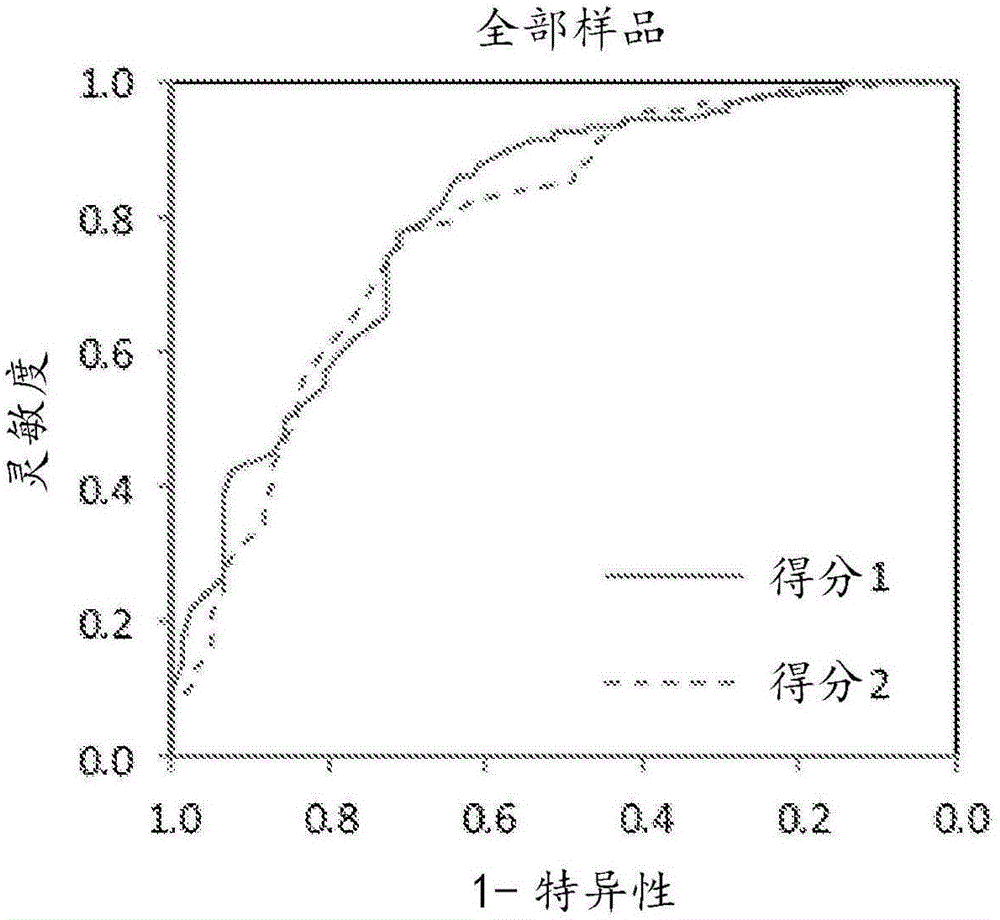
本申请要求2014年7月14日提交的美国临时申请No.62/024,456和2015年5月12日提交的美国临时申请No.62/160,403的优先权,据此通过援引完整收录它们的完整内容用于所有目的。电子提交的文本文件的描述通过援引将电子提交文本文件的内容完整收入本文:计算机可读格式拷贝的序列表(文件名:VRCT-008_02WO_ST25.txt,记录日期:2015年7月14日;文件大小:9千字节)。发明领域本公开文本一般涉及使用基因表达信息评估癌症的方法和组合物。发明背景诊断肺癌(特别是在它能最有效治疗的早期阶段)中的一项挑战是获取细胞来诊断疾病。早期阶段肺癌通常与小损害有关,它们还可能出现于特别难以通过标准技术诸如支气管镜检到达的肺气道的周围区域。发明概述本文中提供的是用于为受试者确立适宜的诊断性干预计划和/或治疗计划及用于在确立适宜的诊断性干预计划和/或治疗计划方面帮助健康护理提供者的方法。在一些实施方案中,该方法基于气道损伤场概念。在一些实施方案中,该方法涉及基于信息性基因的表达水平建立肺癌风险得分,它们对于评估受试者具有癌症的可能性是有用的。在一些实施方案中,本文中提供的方法涉及基于例行细胞或组织取样规程期间自受试者获得的生物学样品中信息性基因的表达水平进行评估。在一些实施方案中,该生物学样品包含组织学上正常的细胞。在一些实施方案中,本公开文本的多个方面至少部分基于确定自气道第一位置获得的表观组织学上正常的细胞中某些信息性基因的表达水平可用于评估气道中第二位置处(例如气道中与该组织学上正常的细胞取样的位置远离的位置处)的癌症的可能性。在一些实施方案中,与涉及获得可疑损害的组织(它们可能少得多地可重复取样)的规程相比,组织学上正常的细胞(例如支气管的细胞)的取样是有利的,因为含有此类细胞的组织一般容易获得,而且因此有可能可重复地获得有用的样品。在一些实施方案中,该方法涉及基于自受试者的支气管收集的细胞学上看来正常的细胞中信息性基因的表达水平进行肺癌评估。在一些实施方案中,表1,11,和26中提供对于预测肺癌的可能性有用的信息性基因。依照本公开文本的一些方面,提供确定受试者具有肺癌的可能性的方法,其涉及对自受试者获得的生物学样品进行基因表达分析,其中该基因表达分析包括测定该生物学样品中一种或多种与肺癌状态有关的信息性基因(例如选自表11的信息性基因)的mRNA表达水平。在一些实施方案中,该方法包括测定该生物学样品中与该受试者的一种或多种可自我报告特征有关的一种或多种基因组关联基因的mRNA表达水平。在一些实施方案中,该方法进一步包括将上文测定的表达水平转换成肺癌风险得分,其指示该受试者具有肺癌的可能性。在一些实施方案中,该受试者的一种或多种可自我报告特征选自:吸烟包年数,吸烟状态,年龄和性别。在一些实施方案中,依照具有大于90%的在预期使用人群中排除肺癌的负面预测值(NPV)的模型确定肺癌风险得分。在一些实施方案中,依照具有大于85%的诊断有COPD的受试者的负面预测值(NPV)的模型确定肺癌风险得分。在一些实施方案中,至少部分基于该肺癌风险得分确立适宜的诊断性干预计划。在一些实施方案中,该方法帮助健康护理提供者做出早期和准确诊断。在一些实施方案中,该方法帮助健康护理提供者基于住院患者的临床评估及早确立适宜的治疗性干预。在一些实施方案中,该方法涉及评估支气管镜检规程期间获得的生物学样品。在一些实施方案中,该方法相对于其它情况下非信息性支气管镜检是有益的,因为它们使得健康护理提供者能够关于患者诊断和/或治疗做出信息性决策。在一些实施方案中,该风险或可能性评估通向用于监测低风险损害的适宜监视。在一些实施方案中,该风险或可能性评估对于某些癌症通向更快的诊断及因此更快的疗法。本文中描述的某些方法单独或与其它方法组合为健康护理提供者提供有用的信息帮助他们为患者做出诊断性和治疗性决策。在其它方法关于患者的肺癌状态提供有用信息已经失败的情况中采用本文中公开的某些方法。本文中公开的某些方法为评估或诊断例行支气管镜检规程期间获得的细胞或组织样品提供替代或互补方法,且提高该规程会得出对于管理患者的护理有用的信息的可能性。本文中公开的方法是高度灵敏的,且自可以自远离恶性肺组织的位置获得的细胞或组织样品(例如组织学上正常的组织)产生关于受试者具有肺癌的可能性的信息。本文中描述的某些方法可用于通过评估例行细胞或组织取样规程(例如辅助性支气管镜检规程,诸如刷检,诸如使用细胞刷;活检;灌洗;和针穿刺)期间获得的组织学上正常的细胞或组织来评估受试者具有肺癌的可能性。然而,应当领会,可使用任何合适的组织或细胞样品。通常,通过该方法评估的细胞或组织在组织学上看来是正常的。在一些实施方案中,该受试者已经鉴定为支气管镜检的候选人和/或具有呼吸道中的可疑损害。在一些实施方案中,本文中公开的方法是有用的,因为它们使得健康护理提供者能够通过平衡受试者具有肺癌的风险同与目的在于确认受试者中肺癌的存在或缺失的某些侵入性诊断性规程有关的风险来确定适宜的诊断性干预和/或治疗计划。在一些实施方案中,一个目标是使用可能不能排除癌症但具有更低风险的干预排列具有低疾病概率的受试者。依照本公开文本的一些方面,提供方法利用基因表达信息来评估受试者的肺癌状态,其涉及一个或多个下面的动作:(a)自受试者的呼吸道获得生物学样品,其中该受试者已经求助于支气管镜检(例如,已经鉴定为具有呼吸道中的可疑损害且因此求助于支气管镜检以评估该损害),(b)对该生物学样品进行基因表达分析,其中该基因表达分析包括测定该生物学样品中多数信息性基因的表达水平,(c)基于该多数信息性基因的表达水平运算肺癌风险得分,(d)如果该肺癌风险得分的水平超过(例如在上)第一阈水平的话,确定该受试者需要第一诊断性干预来评估肺癌状态,并(e)如果该肺癌风险得分的水平超过(例如在下)第二阈水平的话,确定该受试者需要第二诊断性干预来评估肺癌状态。在一些实施方案中,该方法进一步包括(f)如果该肺癌风险得分的水平介于该第一阈和该第二阈水平之间的话,确定该受试者需要第三诊断性干预来评估肺癌状态。在特定的实施方案中,当受试者求助于支气管镜检且支气管镜检规程得出不确定性的或非诊断性的信息时可使用本文中的办法。因而,本文中公开的是用于将此类受试者指派至低风险的方法,其包括一个或多个下面的步骤:(a)自该受试者的呼吸道获得生物学样品,其中该受试者已经经历非诊断性支气管镜检规程,(b)对该生物学样品进行基因表达分析,其中该基因表达分析包括测定该生物学样品中多数信息性基因的表达水平,(c)基于该多数信息性基因的表达水平运算肺癌风险得分,并(d)如果该肺癌风险得分的水平超过(例如在下)第一阈水平的话,确定该受试者是低肺癌风险,并任选(e)对该低风险受试者指派一种或多种非侵入性跟踪规程;例如CT监视。此类办法容许一群受试者避免后续侵入性办法。对于不在该阈水平下的受试者,可以继以非诊断性支气管镜检继以传统办法。在一些实施方案中,该第一诊断性干预包括实施经胸针穿刺,纵隔镜检查术或开胸术。在一些实施方案中,该第二诊断性干预包括进行留置观察(watchfulwaiting)(例如定期监测)。在一些实施方案中,留置观察包括对呼吸道定期成像以评估可疑损害。在一些实施方案中,留置观察包括多至一年,两年,四年,五年或更多对呼吸道定期成像以评估可疑损害。在一些实施方案中,留置观察包括每年至少一次对呼吸道成像以评估可疑损害。在一些实施方案中,留置观察包括每年至少两次对呼吸道成像以评估可疑损害。在一些实施方案中,留置观察包括定期监测受试者,除非和直至该受试者诊断为没有癌症。在一些实施方案中,留置观察包括定期监测受试者,除非和直至该受试者诊断为具有癌症。在一些实施方案中,留置观察包括定期重复前一段中记录的步骤(a)至(f)中的一个或多个。在一些实施方案中,该第三诊断性干预包括实施支气管镜检规程。在一些实施方案中,该第三诊断性干预包括重复前一段中记录的步骤(a)至(e)。在某些实施方案中,该第三诊断性干预包括在确定该肺癌风险得分介于该第一阈和该第二阈水平之间六个月内重复步骤(a)至(e)。在某些实施方案中,该第三诊断性干预包括在确定该肺癌风险得分介于该第一阈和该第二阈水平之间三个月内重复步骤(a)至(e)。在一些实施方案中,该第三诊断性干预包括在确定该肺癌风险得分介于该第一阈和该第二阈水平之间一个月内重复步骤(a)至(e)。在一些实施方案中,该多数信息性基因选自表11中的基因的组。在一些实施方案中,评估这些基因的一个子集的表达水平并与参照表达水平(例如没有癌症的正常患者的)比较。在一些实施方案中,该子集包括a)其表达升高与肺癌或升高的肺癌风险有关的基因,b)其表达降低与肺癌或升高的肺癌风险有关的基因,或二者。在一些实施方案中,子集中至少5%,至少10%,至少20%,至少30%,至少40%,或约50%的基因具有与升高的肺癌风险有关的水平升高的表达。在一些实施方案中,子集中至少5%,至少10%,至少20%,至少30%,至少40%,或约50%的基因具有与升高的肺癌风险有关的水平降低的表达。在一些实施方案中,评估(例如测定或其它方式调查)选自表11中基因的10-80或更多种基因(例如5-10,10-20,20-30,30-40,40-50,50-60,60-70,70-80,约10,约15,约17,约25,约35,约45,约55,约65,约75,或更多种基因)每一种的表达水平。在一些实施方案中,还评估一种或多种对照基因(例如1,2,3,4,或5种对照基因)的表达水平。应当领会,测定法还能包括其它基因,例如参照基因或其它基因(不管它们的信息性如何)。然而,如果本文中描述的信息性基因子集任一的表达概况指示升高的肺癌风险,那么能如本文中描述的那样做出适宜的治疗性或诊断性推荐。在一些实施方案中,来自表11的基因的一个或多个子集的表达水平的变化的鉴定可以以任何合适的格式提供给内科医师或其它健康护理专业人员。在一些实施方案中,单独的这些基因表达概况和/或本文中公开的预测模型的结果可能足以做出诊断,提供预后,或推荐进一步的诊断或特定的治疗。然而,在一些实施方案中,基因表达概况和/或本文中公开的预测模型的结果可能连同其它信息(例如其它表达信息,和/或关于该受试者的其它物理或化学信息,包括家族史)一起帮助受试者的诊断,预后,和/或治疗。在一些实施方案中,通过对呼吸道成像,受试者鉴定为具有呼吸道中的可疑损害。在某些实施方案中,对呼吸道成像包括实施计算机辅助断层摄影术,磁共振成像,超声波扫描术或胸部X射线。在一些实施方案中,提供用于自患者获得生物学样品的方法。这些生物学样品中信息性基因的表达水平为评估该患者具有肺癌的可能性提供基础。提供用于处理生物学样品的方法。在一些实施方案中,该处理方法确保RNA质量和完整性,使得能够进行对信息性基因的下游分析及确保所获得的结果的质量。因而,在这些方法中可以采用各种质量控制步骤(例如RNA大小分析)。提供用于包装和保存生物学样品的方法。提供用于航运或运输生物学样品(例如到达可以处理该生物学样品和/或可以实施基因表达分析的测定实验室)的方法。提供用于对生物学样品实施基因表达分析以测定该样品中信息性基因的表达水平的方法。提供用于分析和解读信息性基因的基因表达分析的结果的方法。提供用于生成汇总基因表达分析的结果的报告及用于将测定法结果和/或测定法解读传输或传送给健康护理提供者(例如内科医师)的方法。而且,提供用于基于该基因表达测定法结果做出治疗决策的方法,包括对于进一步的治疗或侵入性诊断性规程做出推荐。在一些实施方案中,本公开文本的多个方面涉及确定受试者具有肺癌的可能性,其通过对自受试者获得的生物学样品进行基因表达分析,其中该基因表达分析包括测定该生物学样品中至少一种信息性基因(例如至少两种选自表11的基因)的表达水平,并使用该表达水平来帮助确定该受试者具有肺癌的可能性。在一些实施方案中,该测定步骤包括将该表达水平转换成肺癌风险得分,其指示该受试者具有肺癌的可能性。在一些实施方案中,该肺癌风险得分是加权表达水平的组合。在一些实施方案中,该肺癌风险得分是加权表达水平之和。在一些实施方案中,该表达水平是以它们对预测具有肺癌的可能性升高的相对贡献加权的。在一些实施方案中,本公开文本的多个方面涉及为受试者确定治疗过程,其通过对自该受试者获得的生物学样品进行基因表达分析,其中该基因表达分析包括测定该生物学样品中至少两种信息性基因(例如至少两种选自表11的mRNA)的表达水平,并基于该表达水平为该受试者确定治疗过程。在一些实施方案中,该治疗过程是基于自该表达水平推导的肺癌风险得分确定的。在一些实施方案中,基于指示该受试者具有相对高的具有肺癌的可能性的肺癌风险得分将该受试者鉴定为肺癌疗法的候选人。在一些实施方案中,基于指示该受试者具有相对高的具有肺癌的可能性的肺癌风险得分将该受试者鉴定为侵入性肺规程的候选人。在一些实施方案中,该侵入性肺规程是经胸针穿刺,纵隔镜检查术或开胸术。在一些实施方案中,基于指示该受试者具有相对低的具有肺癌的可能性的肺癌风险得分将该受试者鉴定为不是肺癌疗法或侵入性肺规程的候选人。在一些实施方案中,创建汇总该基因表达分析的结果的报告。在一些实施方案中,该报告指出该肺癌风险得分。在一些实施方案中,本公开文本的多个方面涉及确定受试者具有肺癌的可能性,其通过对自受试者获得的生物学样品进行基因表达分析,其中该基因表达分析包括测定该生物学样品中至少一种信息性基因(例如至少一种选自表11的信息性mRNA)的表达水平,并至少部分基于该表达水平确定该受试者具有肺癌的可能性。在一些实施方案中,本公开文本的多个方面涉及确定受试者具有肺癌的可能性,其通过对自受试者的呼吸上皮获得的生物学样品进行基因表达分析,其中该基因表达分析包括测定该生物学样品中至少一种信息性基因(例如至少一种选自表11的信息性mRNA)的表达水平,并至少部分基于该表达水平确定该受试者具有肺癌的可能性,其中该生物学样品包含组织学上正常的组织。在一些实施方案中,本公开文本的多个方面涉及一种处理基因组信息的计算机执行方法,其通过获得代表生物学样品中至少两种信息性基因(例如至少两种来自表11的信息性mRNA)的表达水平的数据,其中该生物学样品是自受试者获得,并使用该表达水平来帮助确定该受试者具有肺癌的可能性。计算机执行方法可包括经由用户界面输入数据,使用处理器运算(例如计算,比较,或其它方式分析),和/或经由显示器或其它用户界面输出结果。在一些实施方案中,该确定步骤包括计算风险得分,其指示该受试者具有肺癌的可能性。在一些实施方案中,运算风险得分涉及确定加权表达水平的组合(例如单独的或连同一种或多种基因组关联基因一起的一种或多种信息性基因的表达水平),其中该表达水平是以它们对预测具有肺癌的可能性升高的相对贡献加权的。在一些实施方案中,基因组关联基因是与特定临床变量(例如可自我报告变量)相关或有关的基因。在一些实施方案中,此类临床变量与癌症(例如肺癌)有关。在一些实施方案中,不是使用多种基因的表达水平,而是可以将在一群受试者内共线变化的多组相关基因(例如彼此相关的)组合或分解成单个数值(例如一组相关基因的均值)。在一些实施方案中,计算机执行方法包括生成指出该风险得分的报告。在一些实施方案中,将该报告传输给该受试者的健康护理提供者。在一些实施方案中,计算机执行方法包括获得代表生物学样品中至少2,3,4,5,6,7,8,9,或10种选自表11中在簇1,2,3,4,5,6,7,8,9,10,或11中鉴定的基因集合的基因的表达水平的数据。在一些实施方案中,该基因包含MYOT。应当领会,在本文中描述的任何实施方案或方面,可以自该受试者的呼吸上皮获得生物学样品。呼吸上皮可以是口,鼻,咽,气管,支气管,细支气管,或肺泡的。然而,也可以使用呼吸上皮的其它来源。该生物学样品可以包含组织学上正常的组织。该生物学样品可以使用支气管刷检,诸如细胞刷或组织刷;支气管-肺泡灌洗;支气管活检;口腔清洗;触摸制备;细针穿刺;或痰收集来获得。该受试者可以展现肺癌的一种或多种症状和/或具有通过计算机辅助断层摄影术或胸部X射线可观察的损害。在一些情况中,该受试者在通过本文中公开的方法评估之前尚未诊断有原发性肺癌。在本文中描述的任何实施方案或方面,该表达水平可以使用定量逆转录聚合酶链式反应,基于珠的核酸检测测定法或寡核苷酸阵列测定法(例如微阵列测定法)或其它技术来测定。在本文中描述的任何实施方案或方面,该肺癌可以是腺癌,鳞状细胞癌,小细胞癌或非小细胞癌。在一些实施方案中,本公开文本的多个方面涉及一种本质上由至少一种核酸探针组成的组合物,其中该至少一种核酸探针每一种与信息性基因(例如至少一种选自表11的信息性mRNA)特异性杂交。在一些实施方案中,本公开文本的多个方面涉及一种包含多至5种,多至10种,多至25种,多至50种,多至100种,或多至200种核酸探针的组合物,其中该核酸探针每一种与信息性基因(例如至少一种选自表1或11的信息性mRNA)特异性杂交。在一些实施方案中,组合物包含至少1,2,3,4,5,6,7,8,9,或10种核酸探针。在一些实施方案中,至少2,3,4,5,6,7,8,9,或10种该核酸探针与自选自表11簇1,2,3,4,5,6,7,8,9,10,或11的不同基因表达的mRNA杂交。在一些实施方案中,核酸探针是直接或间接缀合至珠的。在一些实施方案中,该珠是磁珠。在一些实施方案中,该核酸探针是固定化至固体支持物的。在一些实施方案中,该固体支持物是玻璃,塑料或硅芯片。在一些实施方案中,本公开文本的多个方面涉及一种试剂盒,其包含至少一个装有本文中描述的任何核酸探针组合物的容器或包装。在一些实施方案中,表达水平是使用定量逆转录聚合酶链式反应测定的。在一些实施方案中,本公开文本的多个方面涉及其表达水平可用于确定受试者(例如人受试者)具有肺癌的可能性的基因。在一些实施方案中,一种或多种本文中描述的基因的表达水平(例如mRNA水平)可以在气道样品(例如支气管镜检期间获得的上皮细胞或其它样品或来自适宜的支气管灌洗样品)中测定。在一些实施方案中,可以测定本文中描述的信息性基因(例如1-5,5-10,10-15,15-20,20-25,25-50,50-80,或更多种基因)的一个或多个子集的升高的和/或降低的mRNA表达水平的样式并用于诊断性,预后性,和/或治疗性目的。应当领会,一种或多种本文中描述的表达样式可以单独使用,或者可以连同一种或多种另外的患者特异性标记或症状一起有助于为患者提供个性化诊断性,预后性,和/或治疗性预测或推荐。在一些实施方案中,提供区分有和无肺癌的吸烟者(当前或从前)的信息性基因的集合,它们对于以高准确度预测肺癌风险是有用的。在一些实施方案中,该信息性基因选自表1或11。在一些实施方案中,本文中提供的方法用于确定受试者具有肺癌的可能性,其涉及对自受试者获得的生物学样品进行基因表达分析,包括测定该生物学样品中一种或多种与肺癌状态有关的信息性基因(例如选自表1或11的信息性基因)的mRNA表达水平。在一些实施方案中,该方法包括测定该生物学样品中与该受试者的一种或多种可自我报告特征有关的一种或多种基因组关联基因的mRNA表达水平。在一些实施方案中,该方法进一步包括将上文测定的表达水平转换成肺癌风险得分,其指示该受试者具有肺癌的可能性。在一些实施方案中,该受试者的一种或多种可自我报告特征选自:吸烟包年数,吸烟状态,年龄和性别。在一些实施方案中,该肺癌风险得分是依照下面的方程确定的:其中:X得分1=W0+W1xGG+W2xGS+W3xGPY+W4xGA+W5xC1A+W6xC1B+W7xC2+W8xC3+W9xC4A+W10xC4B且x得分2=W0+W1xGG+W2xGS+W3xGPY+W4x所报告的年龄+W5xC1A+W6xC1B+W7xC2+W8xC3+W9xC4A+W10xC4B,且其中GG,GS,GPY,GA,C1A,C1B,C2,C3,C4A和C4B是依照本文中公开的方程确定的。在一些实施方案中,信息性基因选自表1或11。在一些实施方案中,可以将在一群受试者内共线变化的多组相关基因(例如彼此相关的的)组合或分解成单个数值(例如一组相关基因的均值)。在一些实施方案中,多组相关基因是相关的,因为它们与相同细胞和/或分子途径有关。在一些实施方案中,在单个数值中将至少2,至少3,至少4,至少5或更多种相关基因(例如相关基因,一个共同簇内的基因)组合到一起。在一些实施方案中,通过对自多名受试者(例如2至100,2至500,2至1000或更多名受试者)获得的表达水平实施簇分析,鉴定多组相关基因。可以使用任何适宜的簇分析来鉴定此类相关基因,包括例如基于质心(centroid)的聚簇(例如k均值聚簇),基于连通性的聚簇(例如等级聚簇)和其它合适的办法。表11中鉴定此类簇的非限制性例子,栏2中的数值规定归于其中的每种基因的簇,使得有关基因(例如相关基因)在相同簇内。在一些实施方案中,反映相关基因的集合的表达状态的数值是相关基因的集合的均值表达水平。例如,可以在用于预测受试者具有癌的可能性的模型中使用一项或多项下面的数值:CIA,C1B,C2,C3,C4A,和C4B,其中CIA=(BST1,CD177.1,CD177.2)的均值,C1B=(ATP12A,TSPAN2)的均值,C2=(GABBRI,MCAM,NOVA1,SDC2)的均值,C3=(CDR1,CGREF1,CLND22,NKX3-1)的均值,C4A=(EPHX3,LYPD2)的均值,且C4B=(MIA,RNF150)的均值。在一些实施方案中,簇内的多种基因可以彼此替代。如此,在一些实施方案中,需要在预测模型中评估或使用一个簇内所有基因。在一些实施方案中,独立选择簇内的仅1,2,3,4,5,6,7,8,9,或10种基因进行本文中描述的分析。在一些实施方案中,鉴定表11的簇内的至少1,2,3,4,5,6,7,8,9,或10种基因。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇1的基因集合。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇2的基因集合。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇3的基因集合。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇4的基因集合。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇5的基因集合。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇6的基因集合。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇7的基因集合。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇8的基因集合。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇9的基因集合。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇10的基因集合。在一些实施方案中,一种或多种信息性基因选自表11中鉴定为簇11的基因集合。在一些实施方案中,该信息性基因包含MYOT。在一些实施方案中,将选自簇的多种基因简化成单个数值,诸如例如选定基因的表达水平的均值,中值,模式或其它汇总统计量。在一些实施方案中,本文中提供的是用于为受试者确立适宜的诊断性干预计划和/或治疗计划及用于帮助健康护理提供者确立适宜的诊断性干预计划和/或治疗计划的方法。在一些实施方案中,提供如下的方法,其涉及基于例行细胞或组织取样规程期间自受试者获得的生物学样品中信息性基因的表达水平做出风险评估。在一些实施方案中,提供如下的方法,其涉及基于信息性基因的表达水平确立肺癌风险得分。在一些实施方案中,至少部分基于该肺癌风险得分确立适宜的诊断性干预计划。在一些实施方案中,本文中提供的方法帮助健康护理提供者做出早期和准确诊断。在一些实施方案中,本文中提供的方法帮助健康护理提供者基于住院患者的临床评估及其确立适宜的治疗性干预。在一些实施方案中,本文中提供的方法涉及评估支气管镜检规程期间获得的生物学样品。在一些实施方案中,该方法相对于其它情况下非信息性支气管镜检是有益的,因为它们使得健康护理提供者能够关于患者诊断和/或治疗做出信息性决策。在一些实施方案中,该风险评估通向用于监测低风险损害的适宜监视。在一些实施方案中,该风险评估对于某些癌症通向更快的诊断及因此更快的疗法。本文中提供的是用于确定受试者具有肺癌(诸如腺癌,鳞状细胞癌,小细胞癌或非小细胞癌)的可能性的方法。该方法单独或与其它方法组合为健康护理提供者提供有用的信息来帮助他们为患者做出诊断性和治疗性决策。常常在其它方法关于患者的肺癌状态提供有用信息已经失败的情况中采用本文中公开的方法。例如,大约50%的支气管镜检规程得出不确定性的或非诊断性信息。不确定性的结果有多种来源,而且可能取决于不同医学中心处可得的训练和规程。然而,在某些实施方案中,分子方法与支气管镜检组合预期改善癌症检测准确度。在一些实施方案中,本文中提供的是确定受试者具有肺癌的可能性的方法。在一些实施方案中,提供如下的方法,其涉及对自受试者获得的生物学样品进行基因表达分析,其中该基因表达分析包括测量一种或多种与肺癌状态有关的信息性基因的cDNA水平,并测量与该受试者的一种或多种可自我报告特征有关的一种或多种基因组关联基因的cDNA水平;并基于(a)和(b)中测定的cDNA水平确定肺癌风险得分,其指示该受试者具有肺癌的可能性;其中该cDNA是自来自该生物学样品的mRNA制备的。在一些实施方案中,本公开文本的方法包括将mRNA转变成cDNA。在又一些实施方案中,扩增cDNA。本文中更为详细地描述了这些和其它方面并以非限制性附图和实施例例示。附图简述图1是来自所有合格的AEGISI样品的所有基因表达数据的成对相关性的相关系数的图的非限制性例子;相关系数<0.955的样品鉴定为异常值并从进一步分析中排除;保留总共597份样品;图2是基于一个训练样品集合的预测模型的ROC曲线的非限制性例子;和图3是基于一个支气管镜检阴性训练样品集合的预测模型的ROC曲线的非限制性例子。图4描绘以下颜色编码:满足研究纳入标准的患者(蓝色);排除的患者(黄色);包括在最终分析中的患者(绿色)。图5描绘在AEGIS1(左)和AEGIS2(右)分支中的全部患者(浅灰色)和具有非诊断性支气管镜检的患者子集(黑色)的ROC曲线。在AEGIS1中,两个组分别为AUC=0.78(95%CI,0.73至0.83)和AUC=0.76(95%CI,0.68至0.83)(p=0.31)。在AEGIS2中,两个组中的AUC=0.74(95%CI,0.68至0.80)和AUC=0.75(95%CI,0.68至0.82)(p=0.85)。对于具有非诊断性支气管镜检的患者,AUC就比较AEGIS1和AEGIS2而言也没有显著差异(p=0.61)。图6A和图6B。图6A描绘基于为分类器与支气管镜检组合计算的负分类器调用(实线;使用负似然比调整)和正分类器调用(虚线;使用正似然比调整),与测试前POM相关的测试后POM。负分类器调用曲线显示,对于测试前POM<66%的患者,当支气管镜检为阴性且分类器为负时,测试后POM<10%。对于阴性支气管镜检和正分类器得分的患者,当测试前可能性大于5%时,癌症的测试后可能性>10%。图6B描绘基于分类器和支气管镜检的测试前概率和负似然比的测试后癌症概率。测试后肺癌概率是与基于非诊断性支气管镜检检查和负分类器得分(使用负似然比调整)的测试前概率相关显示的。该曲线显示对于测试前癌症概率小于66%的患者(短垂直线),当支气管镜检发现为阴性且分类器得分为负时,测试后概率小于10%(虚线)。图7描绘具有癌症相关基因表达的基因的成对相关性。评估具有癌症相关基因表达的基因的所有可能对之间的相关性(n=232)以鉴定共享相似基因表达模式的基因的组。使用无监督分级聚簇将相关基因分组成11个簇,其中在y轴上指示建立簇的树状图阈水平(绿色线)。使用线性回归以节约方式自簇中选择基因以预测肺癌状态。分类器基因来自特定的簇(用蓝色表示),使用来自每个簇的2-4种基因。簇4和7含有在肺癌中上调的基因,而簇1,2,9,和10在肺癌中下调。图8描绘测试集合中具有非诊断性支气管镜检的患者的ROC曲线。支气管镜检没有导致肺癌诊断的123名患者的AUC=0.81(其中肺癌的流行度为31%)。图9描绘与训练集合中的所有患者对应的基因表达数据(黑线),而且使用锁定的分类器分析具有非诊断性支气管镜检的患者子集(灰线)。对于两个组,AUC分别计算为0.78(95%CI,0.73-0.82)和0.78(95%CI,0.71-0.85)。图10A和图10B描绘用于与由基因分类器CD177代表的核酸序列杂交的核酸探针。图10A公开了CD177.1中的19种核酸探针(SEQIDNO:24-42,从顶部到底部的次序),图10B公开了CD177.2中的4种核酸探针(SEQIDNO:43-46,从顶部到底部)。发明详述本文中公开的方法为评估通过支气管镜检规程(或用于评估呼吸组织的其它规程)获得的细胞或组织样品提供替代或互补办法,且提高该规程会得出对于管理该患者的护理有用的信息的可能性。本文中公开的方法是高度灵敏的,且自常常自气道中远离恶性肺组织的区域获得的细胞或组织样品(例如气道上皮细胞的支气管刷检)产生关于受试者具有肺癌的可能性的信息。一般而言,本文中公开的方法涉及对自受试者获得的生物学样品进行基因表达分析以评估基因表达水平。然而,在一些实施方案中,在别的部分中基于该生物学样品的组织学检查的结果或通过考虑其它诊断性指标诸如蛋白质水平,mRNA水平,成像结果,胸部X射线检查结果等来确定该受试者具有肺癌的可能性。如本文中使用的,术语“受试者”一般指哺乳动物。典型地,该受试者是人。然而,该术语涵盖其它物种,例如猪,小鼠,大鼠,犬,猫,或其它灵长类动物。在某些实施方案中,该受试者是实验受试者,诸如小鼠或大鼠。该受试者可以是雄性/男性或雌性/女性。该受试者可以是婴儿,幼儿,儿童,年轻成人,成人或老人。该受试者可以是吸烟者,以前的吸烟者或非吸烟者。该受试者可以具有癌症的个人或家族史。该受试者可以具有无癌症个人或家族史。该受试者可以展现肺癌或其它肺病症(例如肺气肿,COPD)的一种或多种症状。例如,该受试者可以具有新的或持久的咳嗽,已有的慢性咳嗽的恶化,痰中带血,持久的支气管炎或反复的呼吸道感染,胸痛,未解释的重量减轻和/或疲劳,或呼吸困难诸如呼吸急促或哮鸣。该受试者可以具有可以是通过计算机辅助断层摄影术或胸部X射线可观察的损害。该受试者可以是已经经历支气管镜检或已经鉴定为支气管镜检的候选人(例如由于存在可检测损害或可疑成像结果)的个体。在一些实施方案中,受试者具有或已经诊断有慢性阻塞性肺病(COPD)。在一些实施方案中,受试者不具有或尚未诊断有COPD。在内科医师或其它健康护理提供者护理下的受试者可称作“患者”。如本文中使用的,术语“约”指加或减“约”修饰的对象的10%。如此,短语“约10,20,或30”分别涵盖8-11,18-22,27-33。信息性基因本公开文本的基因的表达水平已经鉴定为关于受试者的肺癌状态提供有用的信息。这些基因在本文中称作“信息性基因”。信息性基因包括蛋白质编码基因和非蛋白质编码基因。熟练技术人员会领会,信息性基因的表达水平可以通过评估适宜的基因产物(例如mRNA,miRNA,蛋白质等)的水平来测定。因此,某些mRNA的表达水平已经鉴定为提供关于受试者的肺癌状态有用的信息。这些mRNA在本文中称作“信息性mRNA”。表11提供在癌症中差异表达的信息性基因的列表。在一些实施方案中,在肺癌中差异表达的信息性基因选自:BST1,CD177.1,CD177.2,ATP12A,TSPAN2,GABBR1,MCAM,NOVA1,SDC2,CDR1,CGREF1,CLND22,NKX3-1,EPHX3,LYPD2,MIA,RNF150。在一些实施方案中,在肺癌中差异表达的信息性基因选自:TMEM51,CR1L,PDZKlIP1,MICAL2,VWA5A,ACAD8,SAA4,GLYATL2,ETV6,CD177,CEACAM7,QPCT,CASP10,PI3,BST1,MTNR1A,STARD4,CFB,SLC26A8,VNN2,HDAC9,SLC26A4,和LCN2。在一些实施方案中,在肺癌中差异表达的信息性基因选自:CCDC18,FAM72D,NUF2,FBXO28,GPR137B,STIL,DEPDC1,TSPAN2,ASPM,KIF14,KIF20B,RAD51AP1,GAS2L3,SPIC,SMAGP,ATP12A,BRCA2,BORA,SKA3,DLGAP5,CASC5,LRRC28,PYCARD,TXNL4B,EFCAB5,SPAG5,ABCAl2,AURKA,SGOL1,BANK1,CENPE,CASP6,MAD2L1,CCNA2,CCNB1,KIF20A,CENPK,ERAP1,FAM54A,PHTF2,CLDN12,BPGM,PCMTD1,MELK,和MST4。在一些实施方案中,在肺癌中差异表达的信息性基因选自:CR1,GOS2,CSF3R,S100Al2,SELL,NCF2,LIPN,ZNF438,NAMPT,CBL,CASP5,CARD16,CARD17,CLEC4A,LRRK2,HMGN2P46,AQP9,BCL2A1,ITGAX,GPR97,CCL4,PSTPIP2,IFI30,FFAR2,EMR3,FPR1,LILRA5,PLEK,MXD1,TNFAIP6,CXCR2,IL1B,CXCR1,SIRPB1,NCF4,IRAK2,PROK2,TLR2,TREM1,SOD2,CREB5,TNFRSF10C,CSGALNACT1,和ASAP1。在一些实施方案中,在肺癌中差异表达的信息性基因选自:PLA2G2A,NFYC,RASSF10,GLB1L3,TRIM3,MCAM,MSRB3,SLITRK5,GAS6,NOVA1,GABRG3,ABCA3,LPO,FSCN2,RASD1,HILS1,SDK2,NTN5,KCNA7,ATOH8,KCNIP3,INHBB,VSTM2L,ZNRF3,PLEKHG4B,GNMT,GABBR1,ARHGEF10,SDC2,CRB2,GAS1,PNPLA7,和RAI2。本文中公开的某些方法涉及测定该生物学样品中至少一种信息性基因的表达水平。然而,在一些实施方案中,该表达分析涉及测定该生物学样品中至少2,至少3,至少4,至少5,至少6,至少7,至少8,至少9,至少10,至少20,至少30,至少40,至少50,至少60,至少70,或至少80种信息性基因的表达水平。在一些实施方案中,该表达分析涉及测定该生物学样品中1至5,1至10,5至10,5至15,10至15,10至20,15至20,15至25,20至30,25至50,25至75,50至100,50至200或更多种信息性基因,诸如表11中的那些的表达水平。在一些实施方案中,该表达分析涉及测定该生物学样品中至少1至5,1至10,2至10,5至10,5至15,10至15,10至20,15至20,15至25,20至30,25至50,25至75,50至100,50至200或更多种信息性基因,诸如表11中的那些的表达水平。在一些实施方案中,用于表达分析的信息性基因的数目足以提供预测结局中在临床上有用的置信度水平。这种置信度水平(例如预测模型的效能)可以通过多种性能参数来评估,包括但不限于准确度,灵敏度,特异性,和接受者操作特征(ROC)曲线的曲线下面积(AUC)。可以用不同数目的特征(例如基因,mRNA的数目)评估这些参数以确定信息性基因的最佳数目和集合。当单独或与其它信息组合使用时,至少60%,70%,80%,90%的准确度,灵敏度或特异性可能是有用的。可以使用任何适宜的系统或方法来测定信息性基因的表达水平。可以经由使用基于杂交的测定法来测定基因表达水平。如本文中使用的,术语“基于杂交的测定法”指任何涉及核酸杂交的测定法。基于杂交的测定法可以涉及或不涉及核酸的扩增。基于杂交的测定法是本领域公知的且包括但不限于基于阵列的测定法(例如寡核苷酸阵列,微阵列),寡核苷酸缀合的珠测定法(例如多路基于珠的测定法),分子倒置探针测定法,和定量RT-PCR测定法。多路系统(诸如寡核苷酸阵列或基于珠的核酸测定系统)对于同时评估多数基因的水平是特别有用的。其它用于测定核酸的水平的适宜方法对于熟练技术人员会是显而易见的。如本文中使用的,“水平”指指示物质(例如mRNA)的量或发生的数值。水平可以是绝对值,例如样品中mRNA的数量,或相对值,例如样品中mRNA的数量相对于参照样品(对照样品)中mRNA的数量。该水平还可以是二元值,指示物质的存在或缺失。例如,当样品中物质的数量的测量(例如来自PCR反应或微阵列的荧光测量)超出背景值时,可以鉴定为该样品中存在该物质。类似地,当样品中分子的数量的测量处于或不及背景值时,可以鉴定为该样品中缺失该物质(或该样品中检测不到该物质)。应当领会,可以直接或间接测定物质的水平。例如,下面的专利申请(通过援引将其内容完整收入本文中用于所有目的)中公开了信息性mRNA的别的非限制性例子:2006年5月12日提交的,题目为ISOLATIONOFNUCLEICACIDFROMMOUTHEPITHELIALCELLS的美国专利公开文本No.US2007/148650;2009年1月9日提交的,题目为ISOLATIONOFNUCLEICACIDFROMMOUTHEPITHELIALCELLS的美国专利公开文本No.US2009/311692;2010年9月17日提交的,题目为ISOLATIONOFNUCLEICACIDFROMMOUTHEPITHELIALCELLS的美国申请No.12/884,714;2005年12月6日提交的,题目为DETECTIONMETHODSFORDISORDEROFTHELUNG的美国专利公开文本No.US2006/154278;2008年2月8日提交的,题目为DIAGNOSTICFORLUNGDISORDERSUSINGCLASSPREDICTION的美国专利公开文本No.US2010/035244;2010年8月26日提交的,题目为DIAGNOSTICFORLUNGDISORDERSUSINGCLASSPREDICTION的美国申请No.12/869,525;2008年9月19日提交的,题目为BIOMARKERSFORSMOKEEXPOSURE的美国申请No.12/234,368;2010年10月154日提交的,题目为BIOMARKERSFORSMOKEEXPOSURE的美国申请No.12/905,897;2008年9月19日提交的,题目为IDENTIFICATIONOFNOVELPATHWAYSFORDRUGDEVELOPMENTFORLUNGDISEASE的美国专利申请No.US2009/186951;2008年9月9日提交的,题目为DIAGNOSTICANDPROGNOSTICMETHODSFORLUNGDISORDERSUSINGGENEEXPRESSIONPROFILES的美国公开文本No.US2009/061454;2010年11月5日提交的,题目为DIAGNOSTICANDPROGNOSTICMETHODSFORLUNGDISORDERSUSINGGENEEXPRESSIONPROFILES的美国申请No.12/940,840;2009年3月30日提交的,题目为MULTIFACTORIALMETHODSFORDETECTINGLUNGDISORDERS的美国公开文本No.US2010/055689;和2013年4月26日提交的,题目为METHODSFOREVALUATINGLUNGCANCERSTATUS的国际专利申请No.PCT/US13/38449。cDNAcDNA分子是由本领域普通技术人员自mRNA分子合成的非天然发生多核苷酸序列。在一些实施方案中,获得或获取本发明的cDNA分子。利用酶逆转录酶将RNA转变成cDNA创建cDNA,是缺乏内含子的非天然发生分子。依赖于cDNA的方法必然依赖于自然中并非天然发生的人工分子,例如cDNA分子的蛋白质表达或cDNA分子的杂交。在某些方面,利用生物学样品中的mRNA使用至少一种引物通过mRNA的逆转录自样品生成cDNA;使用多核苷酸作为有义和反义引物扩增cDNA以扩增其中的cDNA;并检测扩增cDNA的存在。在进一步的方面,可以通过任何合适的方法测定扩增cDNA的序列。在一个实施方案中,一旦自样品获得mRNA,就将它转变成互补DNA(cDNA)。cDNA在体内不存在且因此是非天然分子。在一个进一步的实施方案中,然后扩增cDNA,例如通过聚合酶链式反应(PCR)或本领域普通技术人员知道的其它扩增方法。这个扩增反应的产物(即扩增cDNA)必然是非天然产物。如上文提到的,cDNA是非天然分子。其次,在PCR的情况中,扩增过程用来为起始材料的每一个cDNA分子个体创建数以亿计的cDNA拷贝。所生成的拷贝的数目大大超出体内存在的mRNA的拷贝的数目。在一个实施方案中,用将另外的DNA序列(衔接头序列)引入到片段的引物(凭借衔接头特异性引物的使用)扩增cDNA。因此,扩增用来通过将条形码,衔接头和/或报告物序列引入到早就是非天然的cDNA上,自非天然单链cDNA创建非天然双链分子。在一个实施方案中,在用衔接头特异性引物扩增期间,将可检测标记物(例如荧光团)添加至单链cDNA分子。因此,扩增也用来创建自然中不发生的DNA复合物,至少因为(i)cDNA在体内不存在,(i)将衔接头序列添加至cDNA分子的末端以生成在体内不存在的DNA序列,(ii)与扩增有关的错误率进一步创建在体内不存在的DNA序列,(iii)cDNA分子与自然中存在的相比根本不同的结构和(iv)可检测标记物对cDNA分子的化学添加。在一个实施方案中,经由与探针的杂交(例如经由微阵列)在固体表面上固定化合成cDNA(例如扩增cDNA)。在另一个实施方案中,经由实时聚合酶链式反应(PCR)经由引入与cDNA产物杂交的荧光探针来检测DNA产物。例如,在一个实施方案中,通过定量发荧光RT-PCR(例如用探针)来评估生物标志物检测。对于PCR分析,本领域有公知方法用于确定供分析中使用的引物序列。在一个实施方案中,为了合成及扩增cDNA,可使用5’AmpliFINDERRACE试剂盒(由Clontech制造)和使用PCR的5’-RACE方法(Frohman,M.A.etal.,Proc.Natl.Acad.Sci.USA(1988)85:8998-9002;Belyaysky,A.etal.,NucleicAcidsResearch.(1989)17:2919-2932)。在此类cDNA合成过程中,可以将限制酶位点引入cDNA的两端。基因组关联如本文中公开的,某些基因的表达水平已经鉴定为与受试者的某些可自我报告特征有关(相关)。此类基因在本文中称作“基因组关联基因”或“基因组关联”且是有用的,因为它们为其它情况下可能不正确和/或不精确报告的受试者特征提供替代标志物。例如,在一些实施方案中,受试者可能不正确地评估诸如包年数,吸烟状态或年龄等信息(例如通过提供此类信息的低估)。在此类实施方案中,使用基于基因组关联基因的预测模型能降低或消除与不正确的报告有关的可变性,因为它基于基因组关联基因的表达而非受试者做出的关于报告什么信息的决策和/或受试者对情况的回忆。熟练技术人员会领会,可以通过评估适宜的基因产物(例如mRNA,miRNA,蛋白质等)的水平来测定此类基因组关联基因的表达水平。可以与肺癌状态的信息性基因(例如选自表11的信息性基因)平行地或与此类基因独立地测定基因组关联基因的表达水平。在一些实施方案中,基因组关联反映个体对环境危险(例如吸烟)的响应。在一些实施方案中,基因组关联反映对风险的暴露。在一些实施方案中,基于一种或多种基因组关联基因确定受试者的性别。在一些实施方案中,与性别相关的基因组关联基因是RPS4Y1。在一些实施方案中,如果RPS4Y1的表达在阈下,那么将该受试者鉴定为男性,且如果RPS4Y1的表达在阈上,那么将该受试者鉴定为女性。在一些实施方案中,对于感兴趣基因,阈是准确区分男性和女性的相对表达水平。在一些实施方案中,基于一种或多种基因组关联基因确定受试者的吸烟状态(例如当前的或以前的)。在一些实施方案中,与吸烟状态相关的基因组关联基因是SLC7A11,CLND10或TKT。在一些实施方案中,依照下面的模型来确定受试者的吸烟状态:吸烟状态(也称作基因组吸烟(GS))=exp(x)/(l+exp(x)),其中其中是回归模型的回归权重且基因符号代表每一种各自基因的相对表达强度。在一些实施方案中,吸烟者是一生中已经抽烟至少100根纸烟的受试者。在一些实施方案中,以前的吸烟者是戒掉或在支气管镜检之前1个月内没有抽吸一根纸烟的受试者。在一些实施方案中,基于一种或多种基因组关联基因确定受试者的吸烟史。在一些实施方案中,与吸烟史相关的基因组关联基因是AKR1C2或RUNX1T1。在一些实施方案中,依照下面的模型来确定受试者的吸烟史:吸烟史(也称作基因组包年数(GPY))=exp(x)/(l+exp(x)),其中其中是该模型的回归权重且基因符号代表每一种各自基因的相对表达强度。在一些实施方案中,基于一种或多种基因组关联基因确定受试者的年龄。在一些实施方案中,与年龄相关的基因组关联基因是CD52,SYT8,TNNT3,ALX1,KLRK1,RASA3,CERS3,ASPA,GRP,APOC1,EPHX3,REEP1,FAM198B,PCDHB4,PCDHB16,FOXD1,SPARC,NKAPL,或GPR110。在一些实施方案中,依照下面的模型来确定受试者的年龄:年龄(也称作基因组年龄其中是该模型的回归权重且基因符号代表每一种各自基因的相对表达强度。生物学样品该方法一般涉及获得自受试者生物学样品。如本文中使用的,短语“获得生物学样品”指任何用于直接或间接自受试者获取生物学样品的过程。例如,可以通过自受试者获取组织或流体样品(例如在护理点设施(point-of-carefacility),内科医师的办公室,医院处)来获得生物学样品。或者,可以通过自直接自受试者获取样品的一个或多个人接收样品(例如在实验室设施处)来获得生物学样品。术语“生物学样品”指自受试者(例如患者)衍生的样品。典型地,生物学样品包含组织,细胞和/或生物分子。在一些实施方案中,生物学样品是在它是组织学上正常(例如通过内窥镜检(例如支气管镜检)确定的)的基础上获得的。在一些实施方案中,生物学样品是自并非怀疑含有癌性细胞的区域(例如支气管或其它部位或区域)获得的。在一些实施方案中,实施组织学或细胞学检查。然而,应当领会,组织学或细胞学检查可以是任选地。在一些实施方案中,生物学样品是呼吸上皮的样品。呼吸上皮可以是受试者的口,鼻,咽,气管,支气管,细支气管,或肺泡的。生物学样品可以包含支气管的上皮。在一些实施方案中,生物学样品没有可检测的癌细胞,例如通过标准组织学或细胞学方法确定的。在一些实施方案中,获得组织学上正常的样品来进行评估。通常,通过刮检或刷检(例如支气管刷检)来获得生物学样品。然而,应当领会,可以使用其它规程,包括例如刷检,刮检,支气管-肺泡灌洗,支气管活检或经支气管针穿刺。要理解的是,可以以任何适宜的方式加工生物学样品以便于测定表达水平。例如,可以恰当地使用生物化学,机械和/或热处理方法自生物学样品分离感兴趣的生物分子,例如RNA。因而,可以通过使用本领域公知的方法处理样品自生物学样品分离RNA或其它分子。肺癌评估本文中公开的方法可以涉及比较信息性基因的表达水平与一种或多种适宜的参照。“适宜的参照”是特定信息性基因的指示已知肺癌状态的表达水平(或表达水平的范围)。可以由该方法的从业人员凭经验确定适宜的参照,或者可以是预先存在的数值或数值范围。适宜的参照代表指示肺癌的表达水平(或表达水平的范围)。例如,适宜的参照可以代表自已知具有肺癌的受试者获得的参照(对照)生物学样品中信息性基因的表达水平。当适宜的参照指示肺癌时,自需要表征或诊断肺癌的受试者测定的表达水平和适宜的参照之间缺乏可检测差异(例如缺乏统计学上显著的差异)可指示该受试者中的肺癌。当适宜的参照指示肺癌时,自需要表征或诊断肺癌的受试者测定的表达水平和适宜的参照之间的差异可指示该受试者没有肺癌。或者,适宜的参照可以是指示受试者没有肺癌的基因的表达水平(或表达水平的范围)。例如,适宜的参照可以代表自已知没有肺癌的受试者获得的参照(对照)生物学样品中特定信息性基因的表达水平。当适宜的参照指示受试者没有肺癌时,自需要诊断肺癌的受试者测定的表达水平和适宜的参照之间的差异可以指示该受试者中的肺癌。或者,当适宜的参照指示受试者没有肺癌时,自需要诊断肺癌的受试者测定的表达水平和适宜的参照水平之间缺乏可检测差异(例如缺乏统计学上显著的差异)可以指示该受试者没有肺癌。在一些实施方案中,参照标准提供变化的阈水平,使得如果样品中基因的表达水平在变化(升高或降低,取决于特定的标志物)的阈水平内,那么将该受试者鉴定为没有肺癌,但是如果该水平在该阈以上,那么将该受试者鉴定为有风险具有肺癌。在一些实施方案中,该方法涉及比较信息性基因的表达水平与代表鉴定为没有肺癌的对照受试者中信息性基因的表达水平的参照标准。这种参照标准可以是例如一群鉴定为没有肺癌的对照受试者中信息性基因的平均表达水平。统计学上显著的表达水平和适宜的参照之间差异的量级可以变化。例如,当生物学样品中信息性基因的表达水平比该基因的适宜参照要高或低至少1%,至少5%,至少10%,至少25%,至少50%,至少100%,至少250%,至少500%,或至少1000%时,可以检测到指示肺癌的显著差异。类似地,当生物学样品中信息性基因的表达水平比该基因的适宜参照要高或低至少1.1倍,1.2倍,1.5倍,2倍,至少3倍,至少4倍,至少5倍,至少6倍,至少7倍,至少8倍,至少9倍,至少10倍,至少20倍,至少30倍,至少40倍,至少50倍,至少100倍,或更多时,可以检测到显著差异。在一些实施方案中,信息性基因和适宜参照之间表达中至少20%至50%的差异是显著的。显著差异可以使用适宜的统计学检验来鉴定。用于统计学显著性的检验是本领域公知的且例示于AppliedStatisticsforEngineersandScientistsbyPetruccelli,ChenandNandram1999ReprintEd。要理解的是,为了评估受试者的肺癌状态,可以比较多数表达水平与多数适宜参照水平,例如在基因-接-基因的基础上。可以作为矢量差来进行该比较。在此类情况中,可以使用多变量检验(例如Hotelling氏T2检验)来评估观察到的差异的显著性。此类多变量检验是本领域公知的且例示于AppliedMultivariateStatisticalAnalysisbyRichardArnoldJohnsonandDeanW.WichernPrenticeHall;6thedition(April2,2007)。分类方法该方法还可以涉及比较自受试者获得的生物学样品中信息性基因的表达水平集合(称作表达样式或概况)与多数参照水平集合(称作参照样式),每一种参照样式与一种已知的肺癌状态有关,鉴定与该表达样式最密切相像的参照样式,并将该参照样式的已知肺癌状态与该表达样式关联起来,由此对该受试者的肺癌状态的分类(表征)。该方法还可以涉及建造或构建能用于对受试者的疾病状态分类的预测模型,其也可以称作分类器或预测器。如本文中使用的,“肺癌分类器”指基于在自受试者获得的生物学样品中测定的表达水平表征该受试者的肺癌状态的预测模型。典型地,该模型是使用分类(肺癌状态)早就确定的样品构造的。一旦构造该模型(分类器),就可以将它应用于自其肺癌状态未知的受试者的生物学样品获得的表达水平以预测该受试者的肺癌状态。如此,该方法可涉及将肺癌分类器应用于该表达水平,使得该肺癌分类器基于该表达水平表征受试者的肺癌状态。可以基于预测的肺癌状态进一步治疗或评估该受试者,例如由健康护理提供者进行。该分类方法可涉及将该表达水平转换成肺癌风险得分,其指示该受试者具有肺癌的可能性。在一些实施方案中,诸如例如当使用线性判别式分类器时,该肺癌风险得分可以作为加权表达水平的组合(例如和,积,或其它组合)获得,其中该表达水平是以它们对预测具有肺癌的可能性升高的相对贡献加权的。应当领会,可以使用本领域已知的多种预测模型作为肺癌分类器。例如,肺癌分类器可以包含选自逻辑回归,偏最小二乘,线性判别式分析,二次判别式分析,神经网络,朴素贝叶斯,C4.5决策树,k-最近邻,随机森林,支持向量机,或其它适宜方法的算法。可以在包含自鉴定为具有肺癌的多数受试者获得的生物学样品中多数信息性基因的表达水平的数据集合上训练肺癌分类器。例如,可以在包含自基于组织学发现鉴定为具有肺癌的多数受试者获得的生物学样品中多数信息性基因的表达水平的数据集合上训练肺癌分类器。典型地,训练集合还会包含鉴定为没有肺癌的对照受试者。正如熟练技术人员会领会的,训练数据集合的受试者群体可以故意具有多种特征,例如该群体的特征可取决于如下的受试者的特征,使用该分类器的诊断性方法对于该受试者可能是有用的。例如,该群体可以全部由男性组成,全部由女性组成或由男性和女性二者组成。该群体可以由具有癌症历史的受试者,没有癌症历史的受试者,或来自这两个范畴的受试者组成。该群体可以包括是吸烟者,以前的吸烟者,和/或非吸烟者的受试者。还可以测量类别预测力度以测定该模型对生物学样品分类的置信度程度。这种置信度程度可以用作该受试者属于由该模型预测的特定类别的可能性的估值。因而,该预测力度传达样品分类的置信度程度且评估何时不能对样品分类。可能有这样的情况,其中测试了样品,但是它不属于,或不能可靠地指派至特定类别。这可以例如通过利用阈或范围来实现,其中得分在所确定的阈以上或以下或者在特定范围内的样品不是不能分类的样品(例如“无调用”)。一旦构造了模型,就能使用本领域已知方法测试该模型的有效性。测试该模型的有效性的一种方式是通过数据集合的交叉验证。为了实施交叉验证,消除一份或一个子集的样品并在没有所消除样品的情况下如上文所述构造模型,形成“交叉验证模型”。然后如本文所述依照该模型对所消除样品分类。对初始数据集合的所有样品或子集进行这个过程并确定误差率。然后评估该模型的准确度。对于已知的类别或先前已经确认的类别,这种模型以高准确度对要测试的样品分类。验证模型的另一种方式是将模型应用于独立的数据集合,诸如新的具有未知肺癌状态的生物学样品。正如熟练技术人员会领会的,可以通过多种参数来评估模型的力度,包括但不限于准确度,灵敏度和特异性。用于运算准确度,灵敏度和特异性的方法是本领域已知且本文中有描述(见例如实施例)。肺癌分类器可以具有至少60%,至少65%,至少70%,至少75%,至少80%,至少85%,至少90%,至少95%,至少99%,或更多的准确度。肺癌分类器可以具有约60%至70%,70%至80%,80%至90%,或90%至100%的范围中的准确度。肺癌分类器可以具有至少60%,至少65%,至少70%,至少75%,至少80%,至少85%,至少90%,至少95%,至少99%,或更多的灵敏度。该肺癌分类器可以具有约60%至70%,70%至80%,80%至90%,或90%至100%的范围中的灵敏度。肺癌分类器可以具有至少60%,至少65%,至少70%,至少75%,至少80%,至少85%,至少90%,至少95%,至少99%,或更多的特异性。肺癌分类器可以具有约60%至70%,70%至80%,80%至90%,或90%至100%的范围中的特异性。在预期使用人群中排除肺癌的负面预测值(NegativePredictiveValue,NPV)可以大于40%,41%,42%,43%,44%,45%,46%,47%,48%,49%,50%,51%,52%,53%,54%,55%,56%,57%,58%,59%,60%,61%,62%,63%,64%,65%,66%,67%,68%,69%,70%,71%,72%,73%,74%,75%,76%,77%,78%,79%,80%,81%,82%,83%,84%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,或99%。预期使用人群可以具有处于或约40%,41%,42%,43%,44%,45%,46%,47%,48%,49%,50%,51%,52%,53%,54%,55%,56%,57%,58%,59%,60%,61%,62%,63%,64%,65%,66%,67%,68%,69%,70%,71%,72%,73%,74%,75%,76%,77%,78%,79%,80%,81%,82%,83%,84%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,或99%的癌症流行度。临床治疗/管理在某些方面,提供用于为受试者确定治疗过程的方法。典型地,该方法涉及测定自该受试者获得的生物学样品中一种或多种信息性基因的表达水平,并基于该表达水平为该受试者确定治疗过程。通常,治疗过程是基于自该表达水平推导的肺癌风险得分确定的。可以基于指示该受试者具有相对高的具有肺癌的可能性的肺癌风险得分将该受试者鉴定为肺癌疗法的候选人。可以基于指示该受试者具有相对高的具有肺癌的可能性的肺癌风险得分(例如大于60%,大于70%,大于80%,大于90%)将该受试者鉴定为侵入性肺规程(例如经胸针穿刺,纵隔镜检查术,或开胸术)的候选人。可以基于指示该受试者具有相对低的具有肺癌的可能性的肺癌风险得分(例如小于50%,小于40%,小于30%,小于20%)将该受试者鉴定为不是肺癌疗法或侵入性肺规程的候选人。在一些情况中,获得的是中等风险得分且不指示该受试者在高风险或低风险范畴中。在一些实施方案中,在做出风险确定后不久,健康护理提供者可参与“留置观察”并对在一个或多个稍晚的时间点采集的生物学样品重复分析,或采取进一步的诊断性规程以排除肺癌,或做出存在癌症的判定。在一个特定的例子中,由于非诊断性支气管镜检将受试者鉴定为中等风险,并遵循使用本文中的方法做出的该患者处于低癌症风险的判定,重新指派非侵入性监测(诸如CT监视)。在另一个特定的例子中,可以使用如本文所述测定的样品。该方法还可涉及创建汇总该基因表达分析的结果的报告。典型地,该报告还会包括指出该肺癌风险得分。计算机执行方法本文中公开的方法可以以众多方式任一执行。例如,某些实施方案可以使用硬件,软件或其组合执行。当以软件执行时,该软件代码可以在任何合适的处理器或处理器集合上执行,无论是在单一计算机中提供或者在分散在多台计算机间。此类处理器可以作为集成电路执行,其中一个或多个处理器在一个集成电路组件中。可是,处理器可以使用任何合适型式的电路执行。而且,应当领会,计算机可以体现为多种形式任一,诸如机架式计算机,台式计算机,膝上式计算机,或平板计算机。另外,计算机可以体现为一般不认为是计算机但具有合适的处理能力的设备,包括个人数字助理(PDA),智能电话或任何其它合适的便携式或固定式电子设备。还有,计算机可以具有一种或多种输入和输出设备。除其它事项外,这些设备可以用于呈现用户界面。能用于提供用户界面的输出设备的例子包括用于视觉呈现输出的打印机或显示屏和用于听觉呈现输出的扬声器或其它声音产生设备。能用于用户界面的输入设备的例子包括键盘,和指点设备,诸如鼠标,触摸板,和数字化平板。又例如,计算机可经由语音识别或以其它音频型式接收输入信息。此类计算机可以互联一种或多种任何合适形式的网络,包括作为局域网或广域网,诸如企业网络或因特网。此类网络可基于任何合适的技术,而且可以依照任何合适的协议操作,而且可包括无线网络,有线网络或光纤网络。还有,本文中概述的各种方法或过程可编码为在采用多种操作系统或平台任一的一个或多个处理器上可执行的软件。另外,此类软件可使用多种合适的编程语言和/或编程或脚本工具任一来编写,而且还可编译为在框架或虚拟机上执行的可执行机器语言代码或中间代码。在这方面,本公开文本的各方面可体现为计算机可读介质(或多种计算机可读介质)(例如计算机存储器,一个或多个软盘,压缩盘(CD),光盘,数字视频盘(DVD)),磁带,闪存,现场可编程门阵列或其它半导体器件中的电路配置,或其它非暂时性有形计算机存储介质),其编码有一种或多种程序,在一台或多台计算机或其它处理器上执行时,实施执行上文讨论的本公开文本的各种实施方案的方法。计算机可读介质可以是可传输的,使得存储在其上的程序能加载到一台或多台不同计算机或其它处理器上,以执行如上文讨论的本公开文本的各个方面。如本文中使用的,术语“非暂时性计算机可读存储介质”仅涵盖可认为是产品(即制品)或机器的计算机可读介质。术语“程序”或“软件”在本文中在一般意义上用于指可用于对计算机或其它处理器进行编程以执行如上文讨论的本公开文本的各个方面的任何类型的计算机代码或计算机可执行指令集合。另外,应当领会,依照此实施例的一个方面,在执行时实施本公开文本的方法的一种或多种计算机程序不需要驻留在单个计算机或处理器上,而是可以以模块方式分散在多台不同计算机或处理器上以执行本公开文本的各个方面。如本文中使用的,术语“数据库”一般指为了搜索和检索的容易和速度而安排的数据的集合。而且,数据库通常包含逻辑和物理数据结构。本领域技术人员会认识到,本文中描述的方法可以与包括关系数据库,对象关系数据库和基于XML的数据库(其中XML代表“可扩展标记语言”)的任何类型的数据库一起使用。例如,基因表达信息可以存储在数据库中并从数据库中检索。基因表达信息可以以将基因表达信息与多种其它相关信息(例如与创建帮助医生建立治疗方案和/或做出诊断确定的报告或文档相关的信息或帮助跟踪患者样品的信息)相关联的方式存储或索引。此类相关信息可以包括例如患者识别信息,订购医生识别信息,关于订购医生办公室的信息(例如地址,电话号码),关于生物学样品的来源的信息(例如组织类型,采样日期),生物学样品处理信息,样品质量控制信息,生物学样品储存信息,基因注释信息,肺癌风险分类器信息,肺癌风险因素信息,支付信息,订单日期信息,等。计算机可执行指令可以是由一台或多台计算机或其它设备执行的许多形式,诸如程序模块。一般而言,程序模块包括实施特定任务或执行特定抽象数据类型的例程,程序,对象,组件,数据结构,等。通常,程序模块的功能性可以依照各种实施方案中的需要组合或分配。在本公开文本的一些方面,提供了用于处理基因组信息的计算机执行方法。该方法一般涉及获得代表生物学样品中一种或多种信息性基因的表达水平的数据,并至少部分基于该表达水平确定受试者具有肺癌的可能性。本文中公开的任何统计或分类方法可以并入计算机执行方法中。在一些实施方案中,该方法涉及计算指示受试者具有肺癌的可能性的风险得分。计算风险得分可涉及确定加权表达水平的组合(例如和,积或其它组合),其中该表达水平是以它们对预测具有肺癌的可能性升高的相对贡献加权的。该计算机执行方法还可以涉及生成汇总该基因表达分析的结果的报告,诸如通过规定风险得分。此类方法还可以涉及将该报告传输给该受试者的健康护理提供者。Affymetrix阵列在一些方面中,使用Affymetrix人基因1.0ST阵列(Affymetrix产品目录号901087)来鉴定生物学样品中的mRNA或cDNA。Affymetrix人基因1.0ST阵列利用在www.affymetrix.com/site/include/byproduct.affx?product=hugene-1_0-st-v1处公开的众多探针。存在与特定基因的区段对应的多种探针,也就是说,不是每种基因一种探针的1:1比率,而是存在与一种基因的多个区段对应的多种探针。在一个例子中,LYPD2基因由人基因1.0ST阵列(版本32)中的三个探针集代表,探针基ID8153343,8153344,和8153345,如在Affymetrix人基因1.0ST阵列(HuGene-1_0-st-v1探针集注释。阵列的例示性合适构件包括版本32(09/30/2011),版本33(03/27/2013),版本34(04/07/2014),版本35(04/15/2015),和版本36)中公开的。还可以使用别的版本,包括未来的版本。关联探针集和核酸序列的信息可以见Affymetrix.com,包括www.affymetrix.com/site/include/byproduct.affx?product=hugene-1_0-st-v1。此外,数据集可在www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL6244处在NCBIGeneExpressionOmnibus网站上在PlatformGPL6244下获得,其详述可用于实施本公开文本的方法的探针和基因。这些文件(包括那些将探针集和基因符号相关联的)通过援引收入本文。组合物和试剂盒在一些方面,提供了可用于测定信息性基因的表达水平的组合物和相关方法。例如,提供了基本上由与信息性基因或与具有与信息性基因互补的序列的核酸特异性杂交的核酸探针组成的组合物。这些组合物还可以包括与对照基因或与其互补的核酸特异性杂交的探针。这些组合物还可以包括适宜的缓冲剂,盐或检测试剂。该核酸探针可以直接或间接固定至固体支持物(例如玻璃,塑料或硅芯片)或珠(例如磁珠)。可以定制核酸探针以用于基于珠的核酸检测测定法中。在一些实施方案中,提供了包含多至5,多至10,多至25,多至50,多至100,或多至200种核酸探针的组合物。在一些情况中,每种核酸探针与选自表11的mRNA或与具有与该mRNA互补的序列的核酸特异性杂交。在一些实施方案中,还包括检测信息性mRNA的探针。在一些情况中,至少2,至少3,至少4,至少5,至少6,至少7,至少8,至少9,至少10,或至少20种核酸探针每一种与选自表11的mRNA或与具有与该mRNA互补的序列的核酸特异性杂交。在一些实施方案中,制备该组合物用于在生物化学上分开的反应中检测不同基因,或用于在相同的生物化学反应中检测多种基因。在一些实施方案中,制备该组合物用于实施多重反应。本文中还提供的是在用于同时测定多种信息性基因的水平的方法中有用的寡核苷酸(核酸)阵列。此类阵列可以从商业来源获得或生产。用于生成核酸阵列的方法也是本领域公知的。例如,核酸阵列可以通过将能够与对应于基因或其部分的核酸杂交的大量寡核苷酸,多核苷酸,或cDNA固定化至固体支持物来构建。本领域技术人员可参见CurrentProtocolsInMolecularBiology(Eds.Ausubeletal.JohnWileyand#38;SonsNY,2000)第22章“NucleicAcidArrays”或LiuCG,etal.,Anoligonucleotidemicrochipforgenome-widemicroRNAprofilinginhumanandmousetissues.ProcNallAcadSciUSA.2004Jun29;101(26):9740-4,其提供与核酸阵列构建有关及在检测感兴趣核酸中使用的方法的非限制性例子。在一些实施方案中,该阵列包含至少2,至少5,至少10,至少20,至少50,至少60,至少70或更多种信息性基因的结合探针或基本上由其组成。在一些实施方案中,该阵列包含至多2,至多5,至多10,至多20,至多50,至多60,至多70或更多种信息性基因的结合探针或基本上由其组成。在一些实施方案中,阵列包含1,2,3,4,5,6,7,8,9,或10种选自表11的mRNA或由其组成。在一些实施方案中,阵列包含4,5,或6种选自表11的mRNA或由其组成。还提供了包含该寡核苷酸阵列的试剂盒。试剂盒可包括核酸标记试剂和使用该阵列测定表达水平的说明书。本文中描述的组合物可以作为试剂盒提供用于测定和评估信息性基因的表达水平。该组合物可以组装成诊断或研究试剂盒以便于它们在诊断或研究应用中的使用。试剂盒可以包括容纳本公开文本的成分的一个或多个容器和使用说明书。具体而言,此类试剂盒可以包括本文中描述的一种或多种组合物,连同描述这些组合物的预期应用和适当使用的说明书。试剂盒可以含有适宜浓度或数量的成分用于运行各种实验。该试剂盒可以设计成便于研究人员,健康护理提供者,诊断实验室,或其他实体使用本文中描述的方法,而且可以采取许多形式。该试剂盒的每种组合物(在适用时)可以以液体形式(例如以溶液)或以固体形式(例如干粉)提供。在某些情况中,一些组合物可以是可构建的或以其它方式可加工的,例如通过添加合适的溶剂或其它物质,其可以与或不与该试剂盒一起提供。如本文中使用的,“说明书”可以定义指令和/或宣传的组件,而且通常涉及在本公开文本的包装上或与其关联的书面说明书。说明书还可以包括以任何方式提供的任何口头或电子指令,使得用户会清楚地认识到该说明书与该试剂盒相关联,例如视听(例如视频磁带,DVD,等),因特网,和/或基于网络的通信,等。书面说明书可以是由管理诊断或生物学产品的制造,使用或销售的政府机构规定的形式,该说明书还可以反映该机构的批准。试剂盒可以在一个或多个容器中含有本文中描述的任何一种或多种成分。例如,在一个实施方案中,该试剂盒可包括关于混合该试剂盒的一种或多种成分和/或分离并混合样品并应用于受试者的说明书。该试剂盒可以包括容纳本文中描述的药剂的容器。该成分可以是液体,凝胶或固体(例如粉末)的形式。该成分可以无菌制备及冷藏运输。或者,它们可以容纳在管形瓶或其它容器中用于储存。第二容器可以具有无菌制备的其它成分。如本文中使用的,提到数值的术语“大约”或“约”一般认为包括在任一方向上(大于或小于)落入1%,5%,10%,15%,或20%范围内的数值,除非另有说明或从上下文另外明显可得的(除非此类数值会小于可能值的0%或超过可能值的100%的情况)。通过援引收录本文中描述的所有参考文献用于本文中描述的目的。会通过下面的实施例更为详细地描述本公开文本的例示性实施方案。这些实施方案是本公开文本的例示,本领域技术人员会认识到本公开文本不限于该例示性实施方案。实施例实施例1:基于微阵列的预测模型的开发导言此实施例描述一种用于开发预测算法的方法。描述了一种最终的经过优化的模型,包括该模型中使用的基因的组合。该方法使用临床因素基因组关联(CFGC)来帮助选择癌症特异性签名。目标是在通过派生基因组关联考虑更加特异性地归结于临床因素的基因表达信号后开发和表征一种新的有效预测肺癌状态的癌症预测模型。基因组关联本文中定义为预测特定临床特征,诸如受试者性别,吸烟状态,和吸烟史的基因表达算法。一个目标是开发满足下面的特定性能表征的预测算法:大于90%的在预期使用人群中排除肺癌的负面预测值(NPV),和大于85%的诊断有COPD的受试者的NPV。材料和方法样品加工和分析自因怀疑肺癌安排接受支气管镜检的患者收集临床标本。在安排的支气管镜检规程期间使用标准支气管刷自受试者的主干支气管收集支气管上皮细胞(BEC)。使用基因1.0ST微阵列(Affymetrix)在基因表达微阵列上分析样品。对阵列数据进行成对关联分析以鉴定离群值(本文中描述的)。保留总共597份样品作为最终的数据集合。然后以随机化方式将该数据集合拆分成等同大小的训练和验证集合。使用微阵列CEL文件来开发预测算法。首先将受试者指派至独立的训练和验证集合,并在训练集合内进行预测模型的基因选择和优化。优化并锁定该模型,之后预测验证集合样品的癌症状态。标准化/批次调整使用RMA来运算来自基因1.0ST(Affymetrix)CEL文件的基因表达值。使用ComBat批次调整来校正批次效应。在5项分开的微阵列实验(即批次)内分析所有样品。在RMA和ComBat预加工中组合训练和测试样品。如先前描述的那样,后续开发局限于训练集合。排除与RIN得分小于4的样品对应的CEL文件,亦如平均配对关联小于.955的样品(见图1)。基因组关联作为精确预测相应临床特征的基因表达签名,建立基因组关联。在预测算法中组合该基因组关联,与分开的预测癌症状态的基因组合。开发并评估下面的临床特征的关联:性别吸烟状态(当前的对以前的)吸烟史(包-年;PY)年龄该基因组关联是通过选择区分感兴趣的临床特征的排序头等的基因并使用逻辑回归将那些基因拟合至模型而开发的。临床特征的评分基于那些选定基因的基因表达。平行开发了两种模型以在验证集合中同时测试。第一模型(得分1)基于本文中描述的方法并将报告的年龄归为预测算法的因素与基因组信号一样。使用年龄的基因组关联开发第二模型(得分2),然后将其并入预测算法。初始基因选择使用癌症状态(0/1)对年龄,性别,吸烟状态,和包装年的逻辑回归来开发临床因素模型。使用来自临床因素模型的残差选择基因,使用经验贝叶斯线性模型测试每种基因与残差的关联。基于来自此模型的p值和倍数变化选择头等232种基因。该头等232种基因列于表11。聚簇/最终基因选择以自动化交叉验证办法进行基因选择和模型拟合,以便在选择过程期间使偏差最小化及提供稳健有力的最终选择。基因选择由以下步骤组成。使用分级聚簇将基因分成11个簇。每种基因的簇成员关系在表11的栏2中标识。对于每个簇,使用每个簇内的所有基因运算簇均值。在数据的重复随机子集合中使用LASSO和反向选择的组合,以鉴定一致地选择与癌症状态具有独立预测性关联的六个簇。然后使用交叉验证来确定每个簇内会保留簇均值的预测强度的基因的近似数目。进行基因滴定分析以确定模型对来自最终模型内所选择的簇的基因数量增加的灵敏度。这被包括作为优化的一部分以确定互补基因是否能为模型增加另外的临床灵敏度。软件使用R(版本3.01)进行分析,包括软件包rms,limma,verification,和sva。结果基因组关联的派生使用整个训练集合选择基因来代表临床特征。这些“基因组关联基因”基于最小限度的基因集合来尽可能准确地代表临床因素。使用单种基因以100%的准确度定义基因组性别(GG)。数值设置如下:如果RPS4Y1<阈值,则GG=1(男性),否则,GG=0(女性)。对于基因组吸烟状态,基于经验贝叶斯t检验筛选基因。将p值的头等基因包括在逻辑回归模型中,其中吸烟状态是因变量。所得预测的基因组吸烟(GS)值源自此模型(使用基因符号来代表相对表达强度),其中,其中是该基因组回归模型的回归权重,且GS=exp(x)/(1+exp(x))。对于基因组包年数,基于经验贝叶斯t检验筛选基因。将p值的头等基因包括在逻辑回归模型中,其中包年数<10是因变量。所得预测的基因组包年数(GPY)值源自此模型,其中,其中是该基因组包年数回归模型的回归权重,且GPY=exp(x)/(1+exp(x))。对于基因组年龄,基于经验贝叶斯线性模型筛选基因。将p值的头等基因包括在罚分线性回归模型(LASSO)中,其中年龄(以年计)是因变量。所得预测的基因组年龄(GA)值源自此模型,其中,其中是该基因组年龄回归模型的回归权重。癌症基因的派生最初选择头等232种基因,接着使用方法部分中描述的聚簇分析来缩减选择基因。然后使用交叉验证来确定每个簇内会保留簇均值的预测强度的基因的近似数目。发现此数目在每个簇2和4种基因之间。每个簇内的最终基因选择基于来自文献的癌症关联的p值,倍数变化,和证据强度。使用每个簇内简化的基因集合重新运算簇均值,如下:CIA=(BST1,CD177.1,CD177.2)的均值C1B=(ATP12A,TSPAN2)的均值C2=(GABBRI,MCAM,NOVA1,SDC2)的均值C3=(CDR1,CGREF1,CLND22,NKX3-1)的均值C4A=(EPHX3,LYPD2)的均值C4B=(MIA,RNF150)的均值。最终模型的描述为了评估最终的模型系数,使用了罚分逻辑回归模型,其中年龄(以年计),基因组性别(GG),基因组吸烟状态(GS),基因组包年(GPY),和6项缩减基因簇均值(标记为CIA,C1B,C2,C3,C4A,C4B)作为独立预测因子和癌症状态(0/1)作为因变量。惩罚因子(λ),对于临床/基因组关联为0,对于每个基因表达簇为10。第二种模型的构建使用相同的办法,但是用如上文定义的基因组年龄(GA)替换年龄。然后重新评估模型系数。最终的分类算法为如下形式的:x得分1=W0+W1xGG+W2xGS+W3xGPY+W4x所报告的年龄+W5xC1A+W6xC1B+W7xC2+W8xC3+W9xC4A+W10xC4BX得分2=W0+W1xGG+W2xGS+W3xGPY+W4xGA+W5xC1A+W6xC1B+W7xC2+W8xC3+W9xC4A+W10xC4B。然后使用如下方程将该逻辑回归得分转换成范围为0至1的预测得分:预测模型的评估使用交叉验证办法,其中分别将训练集合样品随机分拆(90:10)成训练和测试组。记录临床精确度,并将该过程重复100倍。以每份样本的总迭代的平均值报告训练集合的分数。以ROC曲线,AUC,及在定义得分阈以产生50%特异性及最大化临床灵敏度之后的灵敏度和特异性报告结果。生成所有训练集合样品的得分,并与记录的临床状态比较。对于得分1和得分2的ROC曲线,所有样品的显示于图2中,支气管镜检阴性样品的显示于图3中。对于AUC,所有样品的计算为得分1=0.803,得分2=0.785,支气管阴性集合的计算为得分1=0.808,得分2=0.778。表1提供了在预测得分模型中使用的信息性基因的列表。表1.1提供了用于检测此类基因的表达的探针序列的非限制性列表。表1:基因列表和功能表1.1:用于检测信息性基因的探针序列的非限制性例子下文表2提供了训练集合中用于支气管镜检阴性受试者的两种模型的性能特征的汇总。支气管阴性受试者的数目(N)对应于用于灵敏度的CA+受试者和用于特异性的CA-受试者。*NPV是假设50%的癌症流行度与观察到的支气管镜检和预测模型的灵敏度和特异性组合而计算的。模型性能使用特定得分阈来进行预测模型(基于得分1和2)的灵敏度和特异性的计算,以区分CA+和CA-样品的预测。在验证集合中使用在训练集合中为这两种模型选择的相同阈(模型得分=0.65)。首先在支气管镜检阴性样品(预期用途病例)中确定两种模型的预测准确度,并汇总于还针对验证集合内的数个亚组范畴计算预测模型的灵敏度。这两种模型的结果显示于表3-9中。子范畴含有影响置信区间的不同数目的样品。表3–作为观察到的损伤的块大小的函数的预测模型的灵敏度–得分1块大小NSensSens.Sens.PM+BRSens.<113100.00%83.30%100.00%1至23587.50%50.00%100.00%2至34190.60%53,10%96.90%>315585.10%79.40%95.00%浸润物32100.00%100.00%100.00%未知21100.00%80.00%100.00%PM=预测模型;BR=支气管镜检表4–作为观察到的损伤的块大小的函数的预测模型的灵敏度–得分2块大小NPMs.Senens.BRSens.PM+BRS<11366.70%83.30%100.00%1至23587.50%50.00%93.80%2至34181.30%53.10%93.80%>315583.00%79.40%95.70%浸润物3290.00%100.00%100.00%未知21100.00%80.00%100.00%表5–作为肺中观察到的损伤的定位的函数的预测模型的灵敏度(得分1)块大小NPMSensBRSensPM+BRSens中央9784.40%79.20%93.50%周围8690.70%61.10%96.30%二者9090.50%78.40%100.00%未知2586.70%80.00%93.30%表6–作为肺中观察到的损伤的定位的函数的预测模型的灵敏度(得分2)表7–作为如病理学中确定的癌症亚型的函数的预测模型的灵敏度(得分1)癌症类型NPMSens.BRSens.PM+BRSens.AC6783.60%70.10%94.00%SCC7294.40%75.00%95.80%LC771.40%85.70%100.00%NSCLC2692.30%73.10%76.90%SCLC3789.20%86.50%97.30%NSCLC/SCLC250.00%100.00%100,00%未知8675.00%37.50%100.00%表8–作为如病理学中确定的癌症亚型的函数的预测模型的灵敏度(得分2)癌症类型NPMSens.BRSens.PM+BRSens.AC6780,60%70.10%94,00%SCC7287.50%75.00%97.20%LC785.70%85.70%100.00%NSCLC2669.20%73.10%80.80%SCLC3789.20%86.50%97.30%NSCLC/SCLC20.00%100.00%100.00%未知8662.50%37.50%87.50%PM=预测模型;BR=支气管镜检表9–作为癌症阶段的函数的预测模型的灵敏度训练和验证集合中性能的比较在训练和验证集合中比较预测模型的总体预测准确度,以评估模型在独立分支中的再现性。结果提供于表10中。表10-训练和验证集合中两种模型的灵敏度,特异性和AUC的比较对于任一模型在训练和验证集合之间没有观察到灵敏度或特异性的显著差异(p>0.05)。同样,对于每种模型,两个分支之间的AUC的小差异不是统计学上显著的(基于p>0.05)。与所有样品(组合的支气管阴性和支气管阳性)相比,支气管阴性样品的灵敏度和特异性也是类似的。得分1和得分2之间的总体性能有相对较小的下降,得分2显示AUC与得分1相比降低2-4%点。表11:232种差异表达的基因和簇成员性实施例2:用于经历诊断性支气管镜检的肺癌患者的支气管基因组分类器的验证导言支气管镜检在具有疑似肺癌的肺部损害的患者中常常是非诊断性。这常常导致另外的侵入性测试,尽管许多损害是良性的。我们寻求验证能改善支气管镜检的诊断性性能的支气管基因表达分类器。在胸部成像中常常鉴定出疑似肺癌的损害。是否进行监视成像或需要组织采样的侵入性评估的决定是复杂的,而且需要评估恶性的可能性,活检的能力,手术风险,和患者偏好[1]。当需要活检时,该办法可以包括支气管镜检,经胸腔针活检(TTNB),或外科肺活检(SLB)。这些模态之间的选择通常由诸如损害大小和位置,腺病变的存在,规程的风险和当地专长的考虑来决定。支气管镜检是一种安全的规程,气胸并发少于1%[2]。在美国每年实施大约500,000例支气管镜检[3],其中大约一半用于肺癌的诊断性检查。然而,支气管镜检受到其灵敏度的限制,范围在34-88%之间,取决于损害的位置和大小[4]。即使使用较新的支气管镜引导技术,针对周围损害的灵敏度只有约70%[5]。具有非诊断性支气管镜检的患者常常经历进一步的侵入性测试以建立确定性诊断。鉴于固有风险,并发率为大约5%,30天死亡率为约1%,SLB不是首选的办法[6]。重要的是,在最终诊断为良性损害的患者中实施20-25%的SLB[7,8]。而且,TTNB与显著的发病有关,包括15%气胸率[9],其中6%需要胸管引流[10,11]。鉴于侵入性规程的缺陷,需要备选办法来鉴定适合于成像监视的恶性可能性较低的患者。使用临床标本的基因表达测量对生物学疾病状态(包括癌症)进行分类是良好建立的[12]。在肺癌的设置中,在自吸烟者的近端气道收集的细胞学正常上皮中存在独特的癌症相关基因表达样式[13]。最近,我们开发了经由支气管镜检自主干支气管收集的支气管上皮细胞(BEC)中在当前的和以前的吸烟者区分有和没有肺癌的患者的基因表达分类器(提交的手稿)。我们进行了本研究以在为疑似肺癌经历支气管镜检的患者的两项前瞻性多中心试验中验证这种分类器及评估这种分类器如何改变支气管镜检的诊断性能。材料和方法研究设计,群体,和方案为疑似肺癌经历支气管镜检的当前的和以前的吸烟者登记AEGIS1和AEGIS2,两项独立的,前瞻性的,多中心的观察性研究(NCT01309087和NCT00746759)。患者在美国,加拿大,和爱尔兰的28个地点登记。使用细胞刷自主干支气管收集看来正常的BEC并浸没在RNA防腐剂中。分类器的结果没有报告给医生或患者。在支气管镜检之前筛选患者以确定他们是否满足研究方案的要求。排除标准包括<21岁的受试者,从不吸烟者(定义为生命中吸烟<100支香烟),及具有并发的癌症或肺癌的历史的患者。排除了临支气管镜检前在呼吸机上>24小时,或不能同意或服从研究的患者。跟踪患者直至确立最终的诊断或直至支气管镜检后12个月。在指引支气管镜检时或通过随后使用TTNB,SLB,第二支气管镜检,或其它侵入性规程的活检建立肺癌的诊断。使用的具体支气管镜检方法和非诊断性支气管镜检后实施的后续监视成像或规程凭主治医师斟酌。诊断为无癌症的患者在12个月的随访中具有特定的良性诊断或放射照相术稳定性/分辨率。没有确定性癌症诊断,12个月随访中的特定良性诊断或稳定性/分辨率的患者排除在进一步分析之外。主治医师使用五级量表(<10%,10-39%,40-60%,61-85%,和>85%)在支气管镜检前评估每名患者的测试前恶性概率(POM)。该研究方案得到每个参与中心的机构审查委员会批准,而且所有患者在登记前提供知情同意书。在美国,加拿大,和爱尔兰的28个医学中心,AEGIS1中的总共855名患者和AEGIS2中的总共502名患者合格并在2009年1月至2012年8月之间登记(图4)(表12)。这些地点是三级/学术医学中心(n=20),社区医院(n=6),和退伍军人管理局的医院(n=2)的混合。在登记后,基于以下标准将另外的患者从研究中排除(见图4)。首先,AEGIS1中的总共111名患者和AEGIS2中的总共71名患者由于以下方案偏差而不合格:在AEGIS1中,24名患者或是在审查时不满足研究登记标准,收集的标本不满足进入的接受标准,或是登记了但没有收集标本。在RNA分离后,另外87份样品不符合最低QC标准,或是由于RIN得分<4或是RNA产量<1μg。在AEGIS2中,由于收集的标本不符合进入的接受标准,有3名不合格患者,及不符合RNA最低QC标准的68份样品。第二,排除诊断为非原发性肺癌的患者,包括AEGIS1中的40名患者和AEGIS2中的10名患者。此外,在12个月时审查时,没有最终诊断的患者也被排除在研究之外,包括AEGIS1中的91名受试者和AEGIS2中的71名患者。来自AEGIS1和2的这162名患者中的大约三分之二(n=106)在其初始支气管镜检后失去随访,而剩余的三分之一(n=56)在支气管镜检后12个月时没有确定性诊断。最后,我们排除了AEGIS1中的16名患者和AEGIS2中的9名患者,其中基因表达数据未能通过测定法QC标准(在下面微阵列处理方法下描述)。AEGIS1分支先前已经以随机化方式分成相等的训练和测试集合。使用训练集合(n=299名患者)来派生由23种基因加年龄构成的基因表达分类器,而且在别处已经描述(提交的手稿)。在训练集合中,发现分类器在支气管镜检未导致肺癌诊断(n=134)的患者中具有0.78(95%CI,0.71-0.85)的AUC,灵敏度为93%,特异性为57%。当前的研究聚焦于锁定的分类器在两个独立验证集合中的验证。在表1中比较了最终的AEGIS1和AEGIS2验证集合的基线人口统计学和临床特征。在表12中提供了每个分支内诊断为具有癌症和良性疾病的患者的单独比较。表12–AEGIS1和AEGIS2测试集合中的患者的特征。a)以中值(和四分位间范围;IQR)报告。使用Mann-Whitney检验计算P值。b)为白种人对非白种人计算P值。收集每名患者的临床和人口统计学数据并记录在研究临床报告表(CRF)上。在每名患者的医学记录中保存了另外的病理学和放射学报告,并且可用于审查。所有源文件都被监测并输入由研究发起人维护的数据库。肺部损害的大小和位置自CT扫描报告获得。对受试者随访至支气管镜检后12个月以收集用于临床诊断的数据。肺癌的诊断基于来自病理学的结果,并且自医学中心收集病理学报告的拷贝。导致癌症的临床诊断的标本或是在支气管镜检期间获得,或是当支气管镜检是非诊断性时自随后的侵入性获得。在研究期间收集在非诊断性支气管镜检后实施的所有临床规程的数据。随后用非诊断性支气管镜检对患者的评估凭每个研究中心的主治医师斟酌。在一些情况中,支气管镜检后需要多于一个规程来成功地做出诊断,但是在所有情况中,收集导致肺癌诊断的规程。侵入性随访规程定义为第二支气管镜检,TTNB,或手术肺活检(SLB),其可包括纵隔镜检查,胸腔镜检查,开胸手术,或视频辅助胸腔镜手术(VATS)。未诊断有癌症的患者的记录经历了判定过程,以宣布受试者是无癌症的。该过程由五名肺病学家的小组审查每名患者的可得医学记录组成。来自此小组的两名肺病学家独立审查了每个病例,如果他/她符合以下标准之一,那么确定患者无癌症:患者诊断为解释了最初的可疑异常的其它诊断,异常确定是稳定的,或异常解决了。在12个月随访期结束时不满足这些标准的患者没有最终的诊断,并且排除在研究的进一步分析之外。对诊断有原发性肺癌的患者分析组织学(表13)和癌症分期(表14)数据。从研究CRF中提取数据,并通过审查完整医学记录来确认。在全部癌症中,AEGIS1中的80%(175/220;95%CI,74-84%)和AEGIS2中的83%(222/267;95%CI,78-87%)是NSCLC。分别有17%(38/220;95%CI,13-23%)和16%(42/26795%CI,12-21%)SCLC癌症。在具有已知癌症分期数据的NSCLC患者中,AEGIS1中的40%(53/139;95%CI,32-48%)和AEGIS2中的37%(67/199;95%CI,30-44%)存在早期阶段(I和2期)。表13–研究参与者的人口统计学和临床特征的汇总:在所有临床特征间实施AEGIS1对AEGIS2患者的比较。显著性差异由*p<.05,**p<.001指示。计算白种人对非白种人的人种p值,以及使用NSCLC对SCLC的肺癌组织学p值。表14–诊断有原发性肺癌的患者的分期数据。自医学记录提取关于每例良性诊断的数据,并汇总在表15中。在两项研究中,大约三分之二的无癌症受试者具有其它诊断,每项研究的具体其它诊断在表S4中列举。具有异常的患者确定为已经解决或是稳定的诊断基于对医学记录的审查,包括支气管镜检后12个月时的随访成像,而且大约三分之一的无癌患者在两项研究中均在这个范畴中。表15–具有良性疾病的患者的其它诊断在临床指示的支气管镜检期间,研究参与者使用与支气管壁摩擦的标准一次性细胞刷经历右或左主干支气管的粘膜刷样的收集。自每名受试者获得两份刷样;训练医生获得看来正常的上皮组织的刷样,并避免对肿瘤组织或发育不良的细胞采样。所有样品储存在RNA防腐剂(RNAprotect;Qiagen)中,并运送至中心CLIA认证实验室用于进入和加工。中心被要求于4℃存储样品长达14天,并且于环境温度以下(4-20℃)使用由发起人提供的2天运输和隔热运输容器(NanoCool)运输。实验室方法在基因表达分析之前处理所有BEC标本以分离和分析RNA的质量和产量;只有RNA产量为至少1μg且RIN得分>4的标本在Gene-ST1.0微阵列上运行。所有微阵列数据已经作为GSE66499保存于GEO。裂解样品,并使用基于柱的方法(miRNeasy试剂盒;Qiagen)和制造商推荐的方案分离RNA。在分光光度计(Nanodrop;ThermoFisher)上分析大RNA级分以测定浓度,纯度,和产量。还分析样品的RNA完整性(Bioanalyzer),作为RIN得分报告。自研究中排除产量<1μg且RIN评分<4的样品。由于RNA质量或数量不足,排除了AEGIS1中大约10%的标本和AEGIS2中大约13%的标本。然后冷冻(-80℃)保存RNA,直至进一步处理。使用AmbionWT表达试剂盒(Lifetechnologies产品目录号4440536)将200ng总RNA转变成有义链cDNA,随后用Affymetrix基因芯片WT末端标记试剂盒(Affymetrix产品目录号900671)标记。对于杂交,使用杂交,清洗和染色试剂盒(Affymetrix产品目录号900720)制备杂交混合物并添加至经过标记的cDNA靶,应用于人基因1.0ST阵列(Affymetrix产品目录号901087),并于45℃温育16小时。杂交后,清洗阵列,并使用标准Affymetrix规程染色,之后在Affymetrix基因芯片扫描仪上扫描它们。使用ExpressionConsole软件(Affymetrix)提取数据。Affymetrix人基因1.0ST阵列(Affymetrix产品目录号901087)利用在www.affymetrix.com/site/include/byproduct.affx?product=hugene-1_0-st-v1处公开的众多探针。存在与特定基因的区段对应的多种探针,也就是说,不是每种基因一种探针的1:1比率,而是存在与一种基因的多个区段对应的多种探针。在一个例子中,LYPD2基因由人基因1.0ST阵列(版本32)中的三个探针集合代表,探针集ID8153343,8153344,和8153345,如在Affymetrix人基因1.0ST阵列(HuGene-1_0-st-v1探针集注释,2011年9月30日版本32,2013年3月27日版本33,和2014年4月7日版本34)中公开的。关联探针集和核酸序列的信息可以在Affymetrix.com处找到,包括在www.affymetrix.com/site/include/byproduct.affx?product=hugene-1_0-st-v1处。而且,数据集可在NCBIGeneExpressionOmnibus网站在PlatformGPL6244下获得,其详述www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL6244处可在实施本公开文本的方法中利用的探针和基因。这些文件(包括那些关联探针集和基因符号的)通过援引收入本文。使用RMA[22]标准化与每个分支的最终的数据集中的样品对应的微阵列数据(CEL文件)。AEGIS1样品以总共5个批次运行,并且使用ComBat[23]来校正批次效应。AEGIS2样品全部在一个微阵列批次中运行。在标准化后,作为在全局基因表达中具有小于0.955的全基因组成对相关性鉴定异常值。所有微阵列数据已经以编号GSE66499保存于GEO。微阵列数据的标准化和预处理在补充附录中描述。登记在AEGIS1中的受试者先前已经随机指派至独立的训练和验证集合(图4),并且如前所述,严格地在AEGIS1训练集内派生分类器算法并锁定。使用基于23种基因的表达和患者年龄的此预先规定的分类器产生AEGIS1验证集中的每份样品和所有AEGIS2样品的得分。使用预先规定的阈值将这些得分二分为测试阳性和测试阴性。使用接受者操作特征(ROC)曲线,曲线下面积(AUC)的计算[14],及灵敏度,特异性,阴性预测值(NPV),阳性预测值(PPV)和定义为(1-灵敏度)/特异性的负似然比(NLR)的估值来评估分类器的性能。将Mann-Whitney非参数检验用于连续变量的分析,并将Fisher氏精确检验用于分类变量。以双侧二项式95%置信区间(95%CI)报告所有置信区间。使用R软件(版本3.01)实现统计分析。要求医生基于专家意见和支气管镜检前可得的医学记录,使用以下范畴之一评估所有登记的受试者的测试前POM:<10%,10-39%,40-60%,61-85%,和>85%。结果分入<10%,10-60%,和>60%的范畴。然后,我们计算癌症在每个POM范畴中的实际流行度,以比较癌症在分层POM水平中的实际流行度(表16)。另外,我们发现非诊断性支气管镜检后的肺癌流行度在<10%,10-60%和>60%POM组中分别为3%,29%和80%。表16–内科医师评估的POM和癌症流行度的相关性。a)POM=恶性的概率;在支气管镜检之前由主治内科医师评估。b)作为每个POM范畴内诊断有肺癌的分数计算流行度。通过损害大小分层的支气管基因组分类器的预测准确度汇总于表2中,而且通过测试前POM的汇总于表3中。对于诊断有通过阶段和组织学分层的肺癌的患者,还在表S5和S6中报告分类器的灵敏度。分类器与支气管镜检组合导致所有范畴的灵敏度>90%。表17–根据放射学成像特征,分类器,支气管镜检,和组合办法的灵敏度。对于每个范畴中的肺癌患者确定支气管镜检的灵敏度。对于在支气管镜检期间没有诊断出的具有肺癌的患者确定分类器的灵敏度。对于每个范畴中的所有肺癌患者,计算分类器与支气管镜检组合的灵敏度。结果研究参与者的特征来自AEGIS1的298名患者充当第一验证集合,来自AEGIS2的满足研究标准的所有341名患者用作第二验证集合(图4和表13)。AEGIS1和AEGIS2分支的肺癌流行度分别为74%和78%。具有肺癌的患者年龄较大(p<0.001),与没有癌症的患者相比具有更高的累积烟草暴露(p<0.001),而且更有可能是当前的吸烟者(AEGIS1中p=0.07,AEGIS2中p=0.03;表12)。癌症阶段和良性诊断的范畴的汇总分别显示于表15和16中。支气管镜检的性能总共639名患者为疑似肺癌经历支气管镜检。其中,272例(43%;95%CI,39至46)是非诊断性的,包括最终诊断为肺癌的487名患者中的120例(25%;95%CI,21至29)。支气管镜检对肺癌的灵敏度分别为AEGIS1和AEGIS2中的74%(95%CI,68至79)和76%(95%CI,71至81)。具有非诊断性支气管镜检的患者中98%(267/272)的随访规程数据可得。在267名患者中的170例(64%,95%CI,58至69)中在非诊断性支气管镜检后实施了侵入性规程,包括具有良性损害的147例中的52例(35%;95%CI,24至38)和具有癌症的120例中的118例(98%;95%CI,94至99)。在76名患者中实施了SLB,其中27例(36%;95%CI,26至47)具有良性损害。基因表达分类器的性能在AEGIS1中,该分类器单独具有AUC=0.78(95%CI,0.73至0.83)且准确鉴定了220名具有癌症的患者中的194例(88%灵敏度;95%CI,83至92)和78名无癌症的患者中的37例(47%特异性;95%CI,37至58)(图5)。在AEGIS2中,该分类器具有AUC=0.74(95%CI,0.68至0.80)且正确鉴定了267名具有癌症的患者中的237例(89%灵敏度;95%CI,84至92)和74名无癌症的患者中的35例(47%特异性;95%,36至59)。该分类器与支气管镜检的组合分别将AEGIS1和2中的灵敏度提高至96%(95%CI,93至98)和98%(95%CI,96至99),相比之下单独的支气管镜检分别为74%和76%(p<0.001)。在具有非诊断性支气管镜检的患者中,该分类器准确地鉴定了AEGIS1中的57位患者中的49例(86%灵敏度;95%CI,74至94)中和AEGIS2中的67位患者中的62例(92%灵敏度;95%CI,82至97)中的癌症。由于在所有患者间(p=0.32)或对于具有非诊断性支气管镜检的患者(p=0.61)两个分支中的患者之间就分类器AUC而言没有显着差异(图5),我们组合两个分支用于后续亚组分析。单独的支气管镜检的灵敏度在<3cm(p<0.001)或位于周围(p<0.001)的损害中较低(表17)。相比之下,单独的分类器和与支气管镜检组合的分类器的灵敏度一致地较高,而且与损害的大小或位置(表17),癌症阶段(表18)或组织学亚型(表19)没有显著关联。表18–根据癌症阶段,分类器和支气管镜检的灵敏度。表19–根据组织学,分类器和支气管镜检的灵敏度。分类器在具有中等癌症概率的患者中的准确度我们将内科医师评估的恶性概率(POM)组合入低(<10%),中等(10-60%),和高(>60%)POM(表3)的范畴中,以与用于评估肺癌风险的指导方针建议比对[1]。支气管镜检在83%的具有中等测试前POM(n=101)的患者中对于癌症是非诊断性的,尽管具有41%的癌症流行率。在这组患者中,该分类器在那些具有非诊断性支气管镜检中实现91%(95%CI,75至98)的NPV和40%(95%CI,28至54)的PPV(表20)。表20–支气管镜检和分类器分层的测试前POM的性能。(a)在所显示的POM范畴间有总共639名患者。(b)支气管镜检对于639名患者中的272例(43%;95%CI,39至46)是非诊断性的,包括诊断有肺癌的487名患者中的120例(25%;95%CI,21至29)。(c)该分类器准确地预测了具有癌症的120名总体全部患者中的107例(89%;95%CI,82至94)。对于具有非诊断性支气管镜检规程的患者,报告了每个POM范畴中的灵敏度。(d)该分类器准确地预测了没有癌症的152名总体患者中的72例(47%;95%CI,40至55)。对于具有非诊断性支气管镜检规程的患者,报告了每个POM范畴中的特异性。对于具有非诊断性支气管镜检规程的患者,报告了(e)NPV和(f)PPV。虽然该分类器在具有非诊断性支气管镜检的患者中具有高NPV,但是存在13名具有肺癌和负分类器得分的具有非诊断性支气管镜检的患者(即假阴性)。大多数(13例中的10例)具有高(>60%)POM,只有3名患者在10-60%测试前POM组中。计算分类器与支气管镜检组合的NLR以确定测试前POM中测试后概率会是<10%的范围。当与分类器组合时,支气管镜检的NLR(0.244;95%CI,0.21至0.29)改善至0.056(95%CI,0.03至0.10)。结果是,当支气管镜检和分类器二者均为阴性时,对于具有高达66%的测试前POM的患者,测试后POM降低至<10%(图6)。结果的评估此研究描述一种在经历支气管镜检的患者中鉴定没有肺癌的那些的支气管基因组分类器在两个独立的前瞻性分支中的验证。我们发现该基因表达分类器在组合分支中在不同大小,位置,阶段,和细胞类型的肺癌间具有高灵敏度。该分类器和支气管镜检的组合在AEGIS-1和AEGIS-2验证分支中分别具有96%和98%的灵敏度。我们还报告了数项另外的发现,支持这种类型的分类器的临床需要。首先,我们的研究确认了先前报告的观察结果,即常常在最终发现没有肺癌的患者中,非诊断性支气管镜检是常见的(特别是在中等测试前POM的患者中)且导致进一步的侵入性测试,包括SLB。第二,与该分类器的高灵敏度相比,我们发现支气管镜检在小的,周围的或早期阶段的癌症中表现较差。第三,该分类器在具有中等POM和非诊断性支气管镜检的患者中具有高NPV。这些发现提示,这种分类器有潜力在具有中等POM的患者(其中肺癌的流行度为41%但支气管镜检的灵敏度仅为41%)中帮助做出临床决策。由于高NPV,在具有非诊断性支气管镜检和中等POM的患者中的负分类器得分批准更加保守的诊断策略,经由成像的主动监视。虽然该分类器的高NPV会有助于在具有分类器负的中等POM的患者中避免不必要的侵入性规程,但是在这组中存在少数具有肺癌的患者;负的基因表达分类器结果可能延迟这些患者中的进一步侵入性测试。然而,这组患者会经历经由成像进行的主动监测,这是当不采用立即侵入性策略时的标准实践[1,15]。这会容许鉴定损害生长,触发另外的侵入性测试以建立确定性诊断。与在具有中等POM的患者中观察到的高NPV相反,该分类器在此设置中具有40%的适度PPV。如此,该分类器的正结果不保证诊断策略的改变;进一步的测试会需要基于用于在侵入性与成像监视策略之间选择的传统因素。此基因表达分类器在近端BEC中,而不是自肺损害内的细胞测量。细胞学正常的近端气道中基因表达改变检测肺实质内肺癌的存在的能力直接源自“损伤场”范例[13]。Spira等人先前已经显示,在具有肺癌的当前的和以前的吸烟者中,细胞学正常的支气管上皮细胞中有基因表达改变的独特样式[13,16]。此外,致癌信号传导途径在具有肺癌的吸烟者和具有恶变前气道损害的吸烟者的近端气道上皮中被激活[17]。最近,Kadara等人[18]证明,在非小细胞肺癌本身和邻近的小气道上皮中表达改变的基因在那些在近端气道上皮中改变的基因中富集,提示近端气道内的基因表达变化部分地反映在肺肿瘤中观察到的改变的转录组。仅当支气管镜检产生肺癌诊断时,认为该规程是“诊断性的”。有相对较少数目的支气管镜检,潜在诊断为特定的良性病因,但大多数这些患者接受进一步的侵入性测试,包括最终诊断有肺癌的患者,提示尽管最初支气管镜检发现是良性的但对肺癌的担忧仍然升高。最后,我们没有评估并入与临床变量组合的分类器的模型的准确性。虽然已经为孤立性肺结节开发了临床风险预测模型[1,20,21],但是对于选择经历诊断性支气管镜检的患者(其包括具有较宽范围的发现的患者,包括较大的损害(即>3cm),浸润物或其它特征诸如淋巴结病)没有经过验证的模型。如此,基于内科医生对肺癌概率的定性评估,选择大多数患者进行支气管镜检。重要的是,我们证明我们的分类器在具有通过医生评估(一种并入可用临床风险因素的过程)确定的中等POM的患者中实施良好。这项工作的潜在影响得到其研究设计中的许多关键实力所支持。首先,这些是两项独立的前瞻性验证研究,其中在临床上会使用该测试的设置中(即在诊断之前)测量分类器。这是将分子生物标志物自发现研究转移至其最终临床应用的一个关键步骤。第二,大型多中心设计使得能够包括来自不同的实践设置和地理位置的经历支气管镜检的患者。第三,我们的数据证明肺癌在具有非诊断性支气管镜检和中等测试前POM的患者中的高流行度,并且显示该分类器在存在关于癌症状态的最大不确定性的这种设置中具有91%的NPV。在这种设置中,使用对肺癌具有高灵敏度的分类器的机会容许对没有肺癌的患者进行可信的鉴定,其后可以进行主动监视,由此避免潜在有害的侵入性跟踪测试。实施例3:经历诊断性支气管镜检的患者中用于肺癌患者的支气管基因组分类器的派生导言肺癌仍然是美国癌症死亡的首要原因,估计2014年有224,000例新诊断和160,000例死亡,其中90%是由于吸烟[24]。最近,国家肺癌筛查试验显示,低剂量计算机断层扫描(CT)筛查导致高风险个体中20%的相对死亡率降低[25]。然而,死亡率降低伴随着高比例(约96%)的假阳性CT结果,这继而引起了对侵入性诊断规程的过度利用的担忧[26]。具有疑似肺癌的患者常常被提及支气管镜检,其中主要目的是采集可疑肺部损害的样品用于病理学分析。据估计,美国每年实施500,000例支气管镜检[27],其中大约一半用于诊断肺癌。支气管镜检被认为比其它侵入性采样方法更加安全,诸如经胸针活检(TTNB)或手术技术。然而,支气管镜检的诊断灵敏度是亚最佳的,范围从34%(对于<2cm的周围结节)至88%(对于较大的,位于中央的损害)[28]。采用引导技术已经将支气管镜检的适用性扩展到更具挑战性的可疑损害(即常常在肺中周围的孤立性肺结节),但是支气管镜检对肺癌的整体临床敏感度没有实质性改善[29,30]。当支气管镜检是非诊断性的时,常常给内科医师留下模糊性,是否进行进一步的侵入性诊断规程,相关的并发症[31,32],或选择成像监视。在当前的实践中,当实施这些侵入性规程时,大约三分之一的患者被确定为具有良性疾病[33],提示这些规程是可避免的。通过实质性改善支气管镜检的诊断产率来减少这种模糊性的方法能改善患者护理。先前已经证明,香烟烟雾在整个呼吸道衬里的气道上皮细胞中产生损害的分子场[34]。已经表征了香烟烟雾对支气管气道转录组的可逆和不可逆影响,并且已经在具有肺癌的当前的和以前的吸烟者中鉴定了支气管上皮中的一组基因表达改变[35]。这些癌症相关的基因表达概况先前已经显示产生用于在支气管镜检是非诊断性的时候检测肺癌的灵敏分类器。在测试结果为阴性时,这种分类器的高灵敏度(在支气管镜检期间容易获得的生物学标本中测量)导致很低的肺癌概率,并且提示可以使得内科医师能够自信地在无肺癌的受试者中进行主动监视及减少危险的侵入性规程。材料和方法训练集合患者群体患者登记在AEGIS试验(肺癌的诊断中的气道上皮基因表达)中,其设计为作为其诊断工作的一部分经历支气管镜检的怀疑具有肺癌的当前的和以前的吸烟者的前瞻性,观察性,分支研究(登记为NCT01309087和NCT00746759)。选择来自分支之一(“AEGIS1”)的一组患者用于训练基因表达分类器的唯一目的。该研究得到每个参与的医学中心处的IRB批准,并且所有患者在登记前签署知情同意书。在支气管镜检后跟踪所有登记的患者,直到做出最终的诊断或12个月。基于在支气管镜检时或随后的肺活检(诸如当支气管镜检没有导致肺癌诊断时,TTNB或手术肺活检(SLB))获得的细胞病理学诊断患者为具有原发性肺癌。基于对医学记录的审查和在支气管镜检后12个月时的随访规程(在附加文件1中更加详细地描述),诊断患者为具有良性疾病。当在支气管镜检规程时收集的临床样品经由细胞学或病理学产生经过确认的肺癌诊断时,认为支气管镜检是“诊断性的”。在NIH注册的AEGIS研究(NCT01309087和NCT00746759)中登记的患者是为怀疑肺癌经历临床上指示的支气管镜检的患者,年龄为至少21岁且在其一生中吸烟至少100支香烟。研究排除包括先前已经诊断有原发性肺癌的,临支气管镜检之前已经连续使用呼吸机≥24小时的,或不能同意或遵守该研究的患者。在训练分类器之前排除另外的患者以排除具有除原发性肺癌以外的恶性肿瘤的患者。这包括排除具有任何恶性肿瘤历史,经过确认的对肺的转移性癌症,或在登记后发现具有活动性非肺原发性癌症的患者。还有,排除没有最终的确定性诊断的患者。最后,在标本处理后,那些RNA产量(<1μg)或质量(RIN<4)不足的排除在进一步分析之外。在支气管镜检后跟踪患者长达12个月,并审查记录以确认或确定最终的临床诊断。癌症的诊断基于在支气管镜检期间或当支气管镜检是非诊断性时在随访规程中收集的细胞/组织的细胞病理学。导致诊断的随访规程由第二支气管镜检,经胸针穿刺(TTNA),手术肺活检(SLB),或规程组合组成。未被诊断有癌症且已经跟踪了12个月的患者的记录由五名肺病学家的小组进行了判决过程。该过程由对可得医学记录的审查组成,并且如果患者满足以下标准之一,那么患者只宣布为无癌症:诊断有解释初始可疑异常的其它诊断,异常确定是稳定的,或异常消除了。在12个月随访期完成时不满足这些标准的患者标记为“不确定”,并且由于缺乏诊断性“事实”而排除在训练之外。样品收集指示25个参与医学中心中每一个处的内科医师使用标准支气管镜检细胞刷在支气管镜检期间自右主干支气管(或左侧,如果在右边观察到任何异常的话)收集看来正常的支气管上皮细胞(BEC)。收集后,在收集后立即切割细胞刷并置于RNA防腐剂(QiagenRNAProtect,产品目录76526)中,并保存于4℃。然后将标本于4-20℃运输至中心实验室进行进一步处理。向所有地点提供运输容器,使得能够在48小时时段内于4-20℃运输标本。要求各地点使用2天船运服务发送标本。在中心实验室接收后,检查标本并加入实验室信息系统。在RNA分离之前将接收的标本保存于4℃,RNA分离通常在接收后7天内进行。保留所有储存和运输时间的记录,并且标本收集和RNA分离之间的累积时间小于30天(与制造商对RNA防腐剂的建议一致)。RNA分离使用涡旋混合器自细胞刷分离BEC,然后使其形成团粒并使用QIAzol裂解试剂(Qiagen)处理。通过酚/氯仿提取来分离RNA,并依照制造商的建议在二氧化硅膜旋转柱(QiagenmiRNeasy试剂盒,产品目录号217004)上纯化。在NanoDropND-1000分光光度计(ThermoScientific)上分析RNA以测定浓度和纯度,并在2100Bioanalyzer(AgilentTechnologies)上测量RNA完整性(RIN)。然后将每份样品保存于-80℃直至在微阵列上进一步处理。cDNA制备将总RNA转变成有义链cDNA,使用设计成与Affymetrix微阵列一起使用的AmbionWT表达试剂盒(LifeTechnologies,产品目录号4440536)扩增。以200ng总RNA开始,使用T7启动子引物方案经由逆转录制备单链cDNA。使用DNA聚合酶将单链cDNA转变成双链cDNA。微阵列加工用AffymetrixGeneChipWT末端标记试剂盒(Affymetrix,产品目录号900671)标记自总RNA获得的cDNA。将经过标记的cDNA与Gene1.0ST微阵列(Affymetrix,产品目录号901085)杂交,并在AffymetrixGeneChip扫描仪上分析。使用标准AffymetrixGene1.0STCDF和RMA[38]对每份患者样品的个体CEL文件进行标准化。使用设计用于与Affymetrix微阵列一起使用的商业试剂盒(AmbionWT;LifeTechnologies,产品目录号4440536)将总RNA转变成有义链cDNA。以200ng总RNA开始,使用T7启动子引物方案经由逆转录制备单链cDNA。使用DNA聚合酶将单链cDNA转变成双链cDNA。双链cDNA充当模板用于cRNA的体外转录,然后将其纯化以去除酶,盐,无机磷酸盐和未掺入的核苷酸。使用UV吸收测量cRNA的产量,然后用随机引物和dUTP/dNTP混合物通过逆转录使用10μg经过纯化的cRNA产生经过标记的有义链cDNA,片段化,并使用GeneChipWT末端标记试剂盒(Affymetrix,产品目录号900671)标记。使用杂交,清洗和染色试剂盒(Affymetrix,产品目录号900720)将经过标记的cDNA与Gene1.0ST微阵列(Affymetrix,产品目录号901085)杂交,并于45℃温育16小时。杂交后,使用标准Affymetrix规程对阵列清洗和染色,之后在AffymetrixGeneChip扫描仪上扫描,并使用ExpressionConsole软件(Affymetrix)提取数据。如此,本文中描述的微阵列不测量天然分子的表达,而是该微阵列测量非天然发生的cDNA分子的表达。分类器开发在一个多步骤过程中派生了基因表达分类器。初始建模由使用训练数据来选择与三个临床协变量(性别,烟草使用,和吸烟史)相关联的基因(“基因表达关联”)以鉴定这些临床变量的基因表达关联组成。然后选择肺癌相关基因,并最后确定基于癌基因,基因表达关联,和患者年龄的组合预测肺癌可能性的分类器。使用交叉验证并仅使用来自训练集合样品的数据来确定此分类器开发规程的所有方面。临床因素基因表达关联(CFGC)模拟此研究群体中肺癌的协变量,包括性别(男性/女性),吸烟状态(当前的/以前的),和包年(<10/>10),以鉴定临床因素的基因表达关联。使用经验贝叶斯t检验来鉴定其表达与每个临床因素显著相关的基因。接下来,以可用于预测每个临床因素的价值的稀疏方式[36]选择基因。最后,对于每名患者计算来自性别(GG),吸烟状态(GS),和包-年(GPY)的基因表达关联的预测值,并用于选择具有肺癌相关基因表达的基因和下文描述的肺癌分类器。肺癌基因的选择使用训练数据,CFGC,和患者年龄作为预测因子,拟合以肺癌状态(1=癌症阳性且0=癌症阴性)作为因变量的逻辑回归模型。接下来,使用基因表达值作为自变量和逻辑回归模型残差作为因变量拟合经验贝叶斯线性模型。将这用于选择与疾病状态最直接相关而不依赖于临床协变量的基因。使用分级聚簇对来自此分析的头等肺癌相关基因进行分组。以迭代方式选择基因以使用交叉验证最大化AUC以评估预测准确度。目的是选择累积提供最佳分类器性能的簇,以及最好地代表每个簇的具体基因。还实施基因滴定分析以确定提供最佳性能的每个簇的基因数目。对于所选择的簇,对头等基因取平均,为每个患者/簇组合产生簇均值估值。使用DAVID[37]实施每个癌症簇内的基因的功能分析,以鉴定描述该分类器中的癌症相关基因的生物学项。肺癌分类器使用肺癌状态作为输出变量和癌症基因表达估值,患者年龄,和性别(GG),吸烟状态(GS),包年(GPY)的CFGC作为预测因子,开发了肺癌分类器。使用罚分逻辑回归模型拟合模型;对于临床/基因表达关联,惩罚因子(λ)为0,对于每种基因表达簇估值,惩罚因子(λ)为10。所得得分在0到1的度量上。建立了用于预测肺癌状态的得分阈,以对于具有非诊断性支气管镜检的患者实现大约90%的灵敏度。通过在逻辑回归模型中产生包括年龄,性别,吸烟状态,和包-年(临床确定)的预测肺癌状态的“临床模型”来实施基因表达分类器与单独的临床因素相比预测肺癌的益处的评价。通过在训练集合中比较每种模型的AUC来评估完全基因表达分类器和临床因素分类器之间预测肺癌状态的性能的差异。独立测试集合的分析使用来自先前研究[35]的数据作为独立测试集合以评估在此研究中派生的锁定分类器的性能。在该研究中,自为怀疑肺癌经历支气管镜检的患者在支气管镜检时收集了BEC,并在微阵列(AffymetrixHG-U133A)上分析RNA。使用BioconductorR包(版本1.28.1)中的RobustMultiarrayAverage(RMA)[38]将来自该研究的CEL文件(n=163)重新标准化以产生基因水平表达值。此处理使用EntrezGene特异性探针集芯片定义文件(CDF)[39]代替由Affymetrix提供的标准U133ACDF,以便于跨平台分析。使用用于统计运算的R环境(版本2.9.2)实施分析。将该分类器应用于测试集合中的患者,具有两项修改以考虑微阵列平台中的差异。首先,通过逐个基因常数调整HG-U133ARMA表达值,该常数将测试集合中每种基因的表达水平的均值移动至在训练集合中观察到的均值。其次,对于不存在相应的HG-U133A探针集的分类器基因(LYPD2和RNF150),将该基因在训练集合中的均值表达值用于所有测试集合样品。统计方法使用预测准确度的标准测量来评估分类器准确度:曲线下面积(AUC),灵敏度,特异性,NPV和PPV。使用10%样品抽取集合,在训练集合中使用交叉验证来评估使用这些办法产生的预测分类器的性能[40]。使用这些性能估值来指导分类器发现规程的开发。设置最终的模型,之后实施测试集合的一次性分析。使用Fisher氏精确检验来计算所有分类变量的统计学显著性,并将t检验用于连续变量。结果研究群体使用来自AEGIS1的299名患者的集合(由223名诊断有肺癌的患者和76名诊断有良性疾病的患者组成)(表1)来派生我们的基因表达分类器。独立测试集合的特征先前已有描述[错误!书签未定义。],并在此处汇总。虽然研究设计与此处描述的相似,但是研究群体中存在一些差异。与测试集合相比,训练集合中的患者平均而言年龄更大(p<0.001)(尽管对于诊断有肺癌的患者而言年龄没有显著差异(p=0.959))。训练集合也由较少的当前的吸烟者(p=0.050);和较低比例的具有<3cm损害的患者(p<0.001)组成。另外,肺癌的流行度在训练集合中更高(75%对48%;p<0.001)。表21–用于训练分类器的患者的临床和人口统计学特征。分类器的派生和性能的评估对于阵列上的大部分基因而言,基因表达与当前的吸烟状态相关(6477种基因,p<0.001;表22中报告的头10种基因)。基于交叉验证,选择三种排序头等的基因(SLC7A11,TKT,和CLND10)充当基于逻辑回归的吸烟状态分类器。此吸烟状态分类器在训练集合内具有0.93的AUC。不依赖于吸烟状态,为吸烟史派生另一个CFGC,并且基于以包-年测量的累积烟雾暴露。吸烟史(<10PY对>10PY)与531种基因的表达显著相关(p<0.001;表23中报告的头10种基因)。选择两种头等基因充当基于逻辑回归的吸烟史分类器(RUNX1T1,AKR1C2),其在训练集合内具有0.78的AUC。性别与339种基因显著相关(p<0.001;表24中报告的头10种基因)。排序头等的基因(RPS4Y1)是训练集合内性别的完美分类器(AUC=1)。表22:头等的与吸烟状态相关的差异表达的基因表23:头等的与吸烟史相关的差异表达的基因表24:头等的与性别相关的差异表达的基因如方法中描述的,我们鉴定了其表达与来自用于肺癌的CFGC模型的残差显著相关的基因。总共232种癌症相关基因(表25)满足显著性标准(T得分>2.7)。检查了232种基因的成对相关性,接着是分级聚簇,以鉴定具有相似表达样式的基因,并将基因划分成11个簇(图7)。由于基因关联在每个簇内,因此我们假设每个簇内的一小组基因的均值可以用于以稀疏方式代表该簇。我们优化了分类器,使用交叉验证来评估AUC。我们选择基因来代表其表达与肺癌最强相关的基因簇,并确定了包含簇1,2,4,7,9和10给出最佳的AUC。我们还确定了,每个簇超过2-4种基因,测试的性能没有改善。在交叉验证中,对于训练集合中的所有患者(n=299),AUC=0.80(95%CI0.75-0.84);对于具有非诊断性支气管镜检的患者子集(n=134),性能是类似的(AUC=0.81;95%CI0.74-0.87)。表25:包括在分类器中的与癌症相关的基因然后使用完成的分类器发现规程在整个训练集合上确定最终的肺癌分类器。该分类器由作为预测因子的六个癌症基因簇组合(由总共17种基因代表),患者年龄,和基因表达关联(GG,GS,GPY)(表26)组成。使用0.65的得分阈实施二分分类(得分>/=0.65的患者预测为癌症阳性且得分<0.65的患者预测为癌症阴性)。该分类器在训练集合中具有93%的灵敏度和57%的特异性,而且与其支气管镜检对肺癌是非诊断性的患者的子集(AUC=0.78;95%CI0.71-0.85)相比,对于整个训练集合(0.78;95%CI,0.73-0.82)而言,分类器的AUC没有差异(见图9)。表26–基因表达分类器的描述aa)若RPS4Y1<7.5,则基因组性别定义为GG=1(女性),否则为0(男性)。派生预测的基因组吸烟(GS)值,其中x=40.8579-0.4462*SLC7A11-2.1298*CLND10-1.8256*TKT,且基因组吸烟GS=ex/(1+ex)。派生预测的基因组包-年(GPY)值,其中x=-5.1429+2.1891*RUNX1T1-0.9506*AKR1C2,且基因组包-年GPY=exp(x)/(1+exp(x))。预测分类器的广义方程为:得分=ey/(1+ey),其中,y=b0+Σ(bi*xi),其中b0是截距,bi是系数,且xi是特征(如显示的)。b)特征包括如方法中描述的患者年龄(如报告的),GG,GS,GPY,和CA(i),肺癌基因簇(图7中显示)。在训练集合中,与单独使用临床因素的模型(AUC=0.72;95%CI,0.67-0.77)相比,该基因表达分类器的性能显著更好(AUC=0.78;95%CI,0.73-0.82)(p<0.001)。分开汇总17种癌症基因的功能分析(表27)。与癌症相关,9种基因下调,且8种上调。表27:分类器基因的生物学表征。在独立测试集合中的验证在独立测试集合的具有非诊断性支气管镜检的患者(n=123)中,该分类器的AUC为0.81(95%CI,0.73-0.88)(图8),其类似于在训练集合中具有非诊断性支气管镜检的患者中的性能(AUC=0.78;95%0.71-0.85;p=0.495)。灵敏度为92%,特异性为55%,NPV为94%(95%CI,83-99%)(见表28)。有趣的是,我们没有观察到癌症组织学或阶段(表29)或损害大小(表30)对分类器关于癌症的灵敏度的任何影响。此外,在测试集合中,该分类器具有当前吸烟者中0.79和以前吸烟者中0.82的AUC,提示吸烟状态对分类器性能没有显著影响(p=0.710)。当与单独的支气管镜检相比时,基因表达分类器与支气管镜检的组合将灵敏度自51%提高至95%(p<0.001)。表28–支气管镜检,分类器,和组合规程在测试集合中的性能。a)诊断有癌症的患者=CA+和良性疾病=CA-b)在支气管镜检未导致发现癌症的患者(n=123)中评估分类器的性能。表29–支气管镜检,分类器,和组合程序对测试集合中具有肺癌的患者的灵敏度。在为怀疑肺癌经历诊断性支气管镜检规程的163名患者中,78例诊断有癌症。在支气管镜检(a)时,在40名患者(51%;95%CI,40-62%)中做出了肺癌诊断,而且在支气管镜检时未做出诊断的剩余肺癌患者中,该分类器正确预测了他们中的34例(b)(89%;95%CI,75-96%)。分类器与支气管镜检组合产生了78名具有肺癌的患者中74例的检测(95%,95%CI,87-98%)(c)。还显示了支气管镜检,分类器,和组合程序对于肺癌依照亚型和阶段的灵敏度。表30–支气管镜检,分类器,和组合规程在通过可疑损害的大小分层的测试集合中的灵敏度。包括诊断有肺癌的患者和具有良性疾病的患者。表26列出了感兴趣的多个基因表达分类器。下面的表,表31,提供了在NCBI上可得的基因ID,提供了对基因表达分类器的描述性支持。基因分类器CD177在表31中用两个名称描述,即CD177.1和CD177.2。.1和.2标记标识在检测由基因分类器代表的基因的差异表达的阵列中使用两种不同的探针集。图10A公开了用于与CD177杂交的19种探针,说明CD177.1。图10B公开了用于与CD177杂交的4种探针,说明CD177.2。表31–与基因表达分类器对应的NCBI基因ID号。基因分类器基因ID号基因分类器基因ID号RPS4Y16192MCAM4162SLC7A1123657NOVA14857CLDN109071SDC16382TKT7086CDR11038AKR1C21646CGREF110669BST1683CLDN2253842CD177.157126(见图10A)NKX3-14824CD177.257126(见图10B)EPHX379852ATP12A479LYPD2137797TSPAN210100MIA8190GABBR12550RNF15057484RUNX1T1862结果的评估工作已经证明在来自支气管气道的正常上皮细胞中存在与暴露于香烟烟雾和当前和以前吸烟者中肺癌的存在相关的持久基因表达改变[32,41,42,43]。这些癌症相关差异可用于派生能够在支气管镜检期间获得的这些相对非侵入性收集的生物标本中准确检测肺癌的分类器[35]。在当前的实践中,当支气管镜检没有导致发现恶性,而且假阴性率的范围可为20-70%时,排除肺癌是具有挑战性的[28]。当前的指导方针建议,疾病风险升高的患者应当进行更具侵入性的随访诊断规程[28],其具有升高的并发症风险[31]。然而,由于不确定性,这些规程常常在发现具有良性疾病的患者中实施[33]。因此,我们的目标是在支气管镜检期间自近端气道派生基因表达分类器,以提高总体灵敏度,当支气管镜检是非诊断性时最小化模糊性,及减少对不必要的侵入性规程的需要。在这项研究中,我们利用了来自一项更大的多中心研究的为疑似肺癌经历支气管镜检的当前和以前吸烟者的分支以派生用于肺癌的基因表达分类器。该分类器是具有高灵敏度和高NPV的多变量逻辑回归模型。重要的是,我们已经在一个独立分支中验证了该分类器的性能,使用来自先前发表的自为疑似肺癌经历支气管镜检的吸烟者收集的气道样品的研究的数据。在具有55%的特异性的测试集合中,支气管镜检是非诊断性的患者中的灵敏度为92%。测试集合中的NPV为94%,比较而言,单独的支气管镜检的NPV为69%,提示该分类器能帮助内科医师在非诊断性支气管镜检后可靠地鉴定不太可能具有肺癌的患者。差异表达的基因在具有肺癌的当前和以前吸烟者中看来正常的气道上皮中的功能提供了对损害场根本的生物学的了解。在受到遏制的基因中,有多种涉及免疫应答,包括CD177和BST1,提示具有肺癌的吸烟者的气道中受损的免疫应答。基因TSPAN2(其表达受到p53敲减阻抑且与肺腺癌中的不良预后相关[44])在具有癌症的患者中也以较低水平表达。还有,EPHX3(一种涉及异型生物质代谢,烟草烟雾中致癌物的处理,和其它上皮癌中的癌发生的基因[45])下调。在肺癌中上调的分类器基因中,NOVA1和CDR1主要在神经元中表达,但是也在肿瘤中表达,而且在数种恶性肿瘤(包括小细胞肺癌)中与副肿瘤抗体(para-neoplasticantibodies)相关[46,47,48,49,50]。而且,在肺癌中上调的MCAM在基底支气管上皮细胞中表达[51]。MCAM在修复期间在气管上皮中也强烈且瞬时上调[52],是气管上皮再生所需的[53],而且在具有COPD[54]和哮喘[55]的患者的支气管上皮中上调。调节细胞生长和增殖的多种分类器基因在具有肺癌的患者中上调,包括SDC2和NKX3-1以及细胞周期阻滞介导物CGREF1。最后,选择用于在我们的分类器中预测吸烟状态(SLC7A11,CLDN10,TKT)和吸烟史(RUNX1T1,AKR1C2)的CFGC基因先前已经报告为通过烟雾暴露而改变,确认了吸烟对气道上皮生物学的稳健有力影响[11,18,33]。我们的发现办法扩展了早期关于基于基因表达的肺癌诊断的工作[35],主要是在选择具有肺癌相关表达的特征之前,作为预测性模型的成分,临床协变量的显式建模。已知对环境危害和其它临床因素的响应可以在个体间有实质性变化。因此,我们的办法是使用基因表达来捕获对环境危害(例如累积烟暴露)的患者水平生理反应,因为此反应可能比实际报告的值更能反映疾病风险[57]。我们的办法的另一个成分是选择在考虑建模临床因素后其表达与癌症相关的基因。我们假设这种办法会有助于确保由具有癌症相关基因表达的基因关于癌症的可能性捕获的信息独立于由建模的临床因素关于癌症捕获的信息。我们的分类器发现办法的另一个重要方面是我们通过聚簇,然后作为每个癌症相关基因表达模块的加性效应对癌症建模来鉴定独立的癌症相关基因表达的样式的办法。这与仅选择根据其与癌症的关联而全局排序头等的基因(这能潜在导致选择反映单一癌症相关分子过程的整组基因)形成对比。在看来正常的气道上皮细胞中派生基因表达分类器以预测肺癌风险的先前研究已经描述了当支气管镜检是非诊断性的时候具有高灵敏度和NPV的类似结果[35]。虽然该分类器与本文所述分类器相比没有共同的基因,但是我们相信,鉴于独立测试集合中的强性能,我们的新分类器代表类似的作用机制。然而,所选择的具体基因的差异这可能是由于特征选择过程的差异。我们已经使用来自近端气道的细胞派生出用于肺癌的基因表达分类器,其可以与支气管镜检联合用于疑似肺癌。我们已经在一个独立测试集合中验证了这个分类器的性能。该分类器对支气管镜检规程增加了实质性灵敏度,导致高NPV。通过鉴定处于低风险具有肺癌的患者,当支气管镜检是非诊断性的时候,此分类器可用于帮助做出决策。如此已经描述了本公开文本的至少一个实施方案的数个方面,要领会,本领域技术人员会容易想到各种改变,修改,和改进。此类改变,修改,和改进意图作为本公开文本的一部分,而且意图在本公开文本的精神和范围内。因而,前面的描述和附图仅仅是例示性的,而且通过所附权利要求书详细地描述本公开文本。在权利要求中使用诸如“第一”,“第二”,“第三”,等序数术语来修饰权利要求元素本身并不意味着任何优先,在先,或一个权利要求元素相对于另一个的次序或实施方法的动作的时间次序,而是仅仅用作标记来区分具有某个名称的一个权利要求元素与具有相同名称(但是使用该序数术语)的另一个元素以区分各权利要求元素。援引加入通过援引完整收录本文中引用的所有参考文献,论文,出版物,专利,专利公开文本,和专利申请用于所有目的。参考文献1.Gould,M.K.,Donington,J.,Lynch,W.R.,etal.Evaluationofindividualswithpulmonarynodules:Whenisitlungcancer?:Diagnosisandmanagementoflungcancer:AmericanCollegeofChestPhysiciansevidence-basedclinicalpracticeguidelines.CHESTJournal2013;143(5_suppl):e93S-e120S.2.Tukey,M.H.,&Wiener,R.S..Population-basedestimatesoftransbronchiallungbiopsyutilizationandcomplications.Respiratorymedicine2012;106(11):1559-1565.3.ErnstA,SilvestriG,JohnstoneD.InterventionalPulmonaryProcedures:GuidelinesfromtheAmericanCollegeofChestPhysicians.Chest2003;123;1693-17174.RiveraMP,MehtaAC,WahidiMM.EstablishingtheDiagnosisofLungCancer:DiagnosisandManagementofLungCancer,3rded:AmericanCollegeofChestPhysiciansEvidence-BasedClinicalPracticeGuidelines.Chest2013;143:e142S-65S.5.Memoli,J.S.W.,Nietert,P.J.,&Silvestri,G.A.Meta-analysisofguidedbronchoscopyfortheevaluationofthepulmonarynodule.CHESTJournal2012;142(2):385-393.6.Ost,D.,Fein,A.M.,&Feinsilver,S.H.(2003).Thesolitarypulmonarynodule.NewEnglandJournalofMedicine,348(25),2535-2542.7.Grogan,E.L.,Weinstein,J.J.,Deppen,S.A.,etal.Thoracicoperationsforpulmonarynodulesarefrequentlynotfutileinpatientswithbenigndisease.Journalofthoraciconcology:officialpublicationoftheInternationalAssociationfortheStudyofLungCancer2011,6(10):1720.8.Detterbeck,F.C.,Mazzone,P.J.,Naidich,D.P.,&Bach,P.B.Screeningforlungcancer:diagnosisandmanagementoflungcancer:AmericanCollegeofChestPhysiciansevidence-basedclinicalpracticeguidelines.CHESTJournal2013;143(5_suppl):e78S-e92S.9.Wiener,R.S.,Wiener,D.C.,&Gould,M.K.RisksofTransthoracicNeedleBiopsy:HowHigh?Clinicalpulmonarymedicine2013;20(1):29.10.Covey,A.M.,Gandhi,R.,Brody,L.A.,Getrajdman,G.,Thaler,H.T.,&Brown,K.T.Factorsassociatedwithpneumothoraxandpneumothoraxrequiringtreatmentafterpercutaneouslungbiopsyin443consecutivepatients.Journalofvascularandinterventionalradiology2004;15(5):479-483.11.Geraghty,P.R.,Kee,S.T.,McFarlane,G.,Razavi,M.K.,Sze,D.Y.,&Dake,M.D.CT-guidedTransthoracicNeedleAspirationBiopsyofPulmonaryNodules:NeedleSizeandPneumothoraxRate1.Radiology2003;229(2):475-481.12.Golub,T.R.,Slonim,D.K.,Tamayo,P.,etal.Molecularclassificationofcancer:classdiscoveryandclasspredictionbygeneexpressionmonitoring.Science1999;286(5439):531-537.13.SpiraA,BeaneJE,ShahV,etal.Airwayepithelialgeneexpressioninthediagnosticevaluationofsmokerswithsuspectlungcancer.Naturemedicine2007;13:361-6.14.HanleyJA,McNeilBJ.Themeaninganduseoftheareaunderareceiveroperatingcharacteristic(ROC)curve.Radiology1982;143:29-36.15.MacMahon,H.,Austin,J.H.,Gamsu,G.,etal.GuidelinesformanagementofsmallpulmonarynodulesdetectedonCTscans:astatementfromtheFleischnerSociety1.Radiology2005,237(2):395-400..16.Beane,J.,Sebastiani,P.,Whitfield,T.H.,etal.Apredictionmodelforlungcancerdiagnosisthatintegratesgenomicandclinicalfeatures.CancerPreventionResearch2008;1(1):56-64.17.Gustafson,A.M.,Soldi,R.,Anderlind,C.,etal.AirwayPI3Kpathwayactivationisanearlyandreversibleeventinlungcancerdevelopment.Sciencetranslationalmedicine2010;2(26):26ra25-26ra25.18.Kadara,H.,Fujimoto,J.,Yoo,S.Y.,Maki,etal.Transcriptomicarchitectureoftheadjacentairwayfieldcancerizationinnon–smallcelllungcancer.JournaloftheNationalCancerInstitute2014;106(3):dju004.19.Alexander,E.K.,Kennedy,G.C.,Baloch,etal.Preoperativediagnosisofbenignthyroidnoduleswithindeterminatecytology.NewEnglandJournalofMedicine2012,367(8):705-715.20.McWilliamsA,TammemagiMC,MayoJR,etal.ProbabilityofcancerinpulmonarynodulesdetectedonfirstscreeningCT.NEnglJMed2013;369:910-9.21.OstDE,GouldMK.Decisionmakinginpatientswithpulmonarynodules.Americanjournalofrespiratoryandcriticalcaremedicine2012;185:363-72.22.Irizarry,R.A.,Bolstad,B.M.,Collin,F.,Cope,L.M.,Hobbs,B.,&Speed,T.P.(2003).SummariesofAffymetrixGeneChipprobeleveldata.Nucleicacidsresearch,31(4),e15-e15.23.JohnsonWE,LiC,RabinovicA.AdjustingbatcheffectsinmicroarrayexpressiondatausingempiricalBayesmethods.Biostatistics2007;8:118–127.24.HowladerN,NooneAM,KrapchoM,etal.SEERstatfactsheets:lungandbronchus.Availableonline:http://seer.cancer.gov/statfacts/html/lungb.html.25.TheNationalLungScreeningTrialResearchTeam.ReducedLung-CancerMortalitywithLow-DoseComputedTomographicScreening.NEnglJMed2011;365:395-40926.TanoueLT,TannerNT,GouldMK,SilvestriGA.LungCancerScreening.AmJRespirCritCareMed.publishedonline04Nov201427.ErnstA,SilvestriG,JohnstoneD.InterventionalPulmonaryProcedures:GuidelinesfromtheAmericanCollegeofChestPhysicians.Chest2003;123;1693-171728.Rivera,M.P.,Mehta,A.C.,&Wahidi,M.M.(2013).EstablishingtheDiagnosisofLungCancerDiagnosisandManagementofLungCancer,3rded:AmericanCollegeofChestPhysiciansEvidence-BasedClinicalPracticeGuidelines.CHESTSupplement.Chest,143,e143S.29.Silvestri,G.A.,Feller-Kopman,D.,Chen,A.,Wahidi,M.,Yasufuku,K.,&Ernst,A.(2012).LatestAdvancesinAdvancedDiagnosticandTherapeuticPulmonaryProceduresUpdateonPulmonaryProcedures.CHESTJournal,142(6),1636-1644.30.Gildea,T.R.,Mazzone,P.J.,Karnak,D.,Meziane,M.,&Mehta,A.C.(2006).Electromagneticnavigationdiagnosticbronchoscopy:aprospectivestudy.Americanjournalofrespiratoryandcriticalcaremedicine,174(9),982-989.31.Wiener,R.S.,Schwartz,L.M.,Woloshin,S.,&Welch,H.G.(2011).Population-basedriskforcomplicationsaftertransthoracicneedlelungbiopsyofapulmonarynodule:ananalysisofdischargerecords.Annalsofinternalmedicine,155(3),137-144.32.Fontaine-Delaruelle,C.,Ferretti,G.,Gamondes,D.,Pradat,E.,Souquet,P.J.,&Couraud,S.(2014).IstransthoraciccoreneedlebiopsyunderCTscanagooddealforbenigndiseases′diagnosis?.EuropeanRespiratoryJournal,44(Suppl58),P679.33.SmithMA,BattafaranoRJ,MeyersBF,ZooleJB,CooperJD,PattersonGA.Prevalenceofbenigndiseaseinpatientsundergoingresectionforsuspectedlungcancer.TheAnnalsofthoracicsurgery2006;81:1824-1828;discussion1828-182934.Beane,J.,Sebastiani,P.,Liu,G.,Brody,J.S.,Lenburg,M.E.,&Spira,A.(2007).Reversibleandpermanenteffectsoftobaccosmokeexposureonairwayepithelialgeneexpression.GenomeBiol,8(9),R201.35.SpiraA,BeaneJE,ShahV,SteilingK,LiuG,SchembriF,GilmanS,DumasYM,CalnerP,SebastianiP,SridharS,BeamisJ,LambC,AndersonT,GerryN,KeaneJ,LenburgME,BrodyJ.Airwayepithelialgeneexpressioninthediagnosticevaluationofsmokerswithsuspectlungcancer.NatureMedicine2007;13(3):361-636.Tibshirani,R.(1996).Regressionshrinkageandselectionviathelasso.JournaloftheRoyalStatisticalSociety.SeriesB(Methodological),267-288.37.DaWeiHuang,B.T.S.,&Lempicki,R.A.(2008).SystematicandintegrativeanalysisoflargegenelistsusingDAVIDbioinformaticsresources.Natureprotocols,4(1),44-57.38.Irizarry,R.A.,Hobbs,B.,Collin,F.,Beazer‐Barclay,Y.D.,Antonellis,K.J.,Scherf,U.,&Speed,T.P.(2003).Exploration,normalization,andsummariesofhighdensityoligonucleotidearrayprobeleveldata.Biostatistics,4(2),249-264.39.Dai,M.,Wang,P.,Boyd,A.D.,Kostov,G.,Athey,B.,Jones,E.G.,etal.,(2005).Evolvinggene/transcriptdefinitionssignificantlyaltertheinterpretationofGeneChipdata.Nucleicacidsresearch,33(20),e175-e175.40.Golub,T.R.,Slonim,D.K.,Tamayo,P.,Huard,C.,Gaasenbeek,M.,Mesirov,J.P.,etal.,(1999).Molecularclassificationofcancer:classdiscoveryandclasspredictionbygeneexpressionmonitoring.Science,286(5439),531-537.41.Kadara,H.,Fujimoto,J.,Yoo,S.Y.,Maki,Y.,Gower,A.C.,Kabbout,M.,etal.,(2014).TranscriptomicArchitectureoftheAdjacentAirwayFieldCancerizationinNon–Smallcelllungcancer.JournaloftheNationalCancerInstitute,106(3)42.Steiling,K.,Ryan,J.,Brody,J.S.,&Spira,A.(2008).Thefieldoftissueinjuryinthelungandairway.Cancerpreventionresearch,1(6),396-403.43.Bossé,Y.,Postma,D.S.,Sin,D.D.,Lamontagne,M.,Couture,C.,Gaudreault,N.,etal.,(2012).Molecularsignatureofsmokinginhumanlungtissues.Cancerresearch,72(15),3753-3763.44.Otsubo,C.,Otomo,R.,Miyazaki,M.,Matsushima-Hibiya,Y.,Kohno,T.,Iwakawa,R.,etal.,(2014).TSPAN2IsInvolvedinCellInvasionandMotilityduringLungCancerProgression.Cellreports,7(2),527-538.45.B.,Thorsen,K.,Lamy,P.,Wojdacz,T.K.,Hansen,L.L.,Birkenkamp‐K.,etal.,(2011).IdentificationandvalidationofhighlyfrequentCpGislandhypermethylationincolorectaladenomasandcarcinomas.Internationaljournalofcancer,129(12),2855-2866.46.Knudsen,A.,Monstad,S.E.,A.,P.E.,Salvesen,H.B.,Drivsholm,L.,etal.,(2006).Riantibodiesinpatientswithbreast,ovarianorsmallcelllungcancerdeterminedbyasensitiveimmunoprecipitationtechnique.CancerImmunology,Immunotherapy,55(10),1280-1284.47.Buckanovich,R.J.,Posner,J.B.,&Darnell,R.B.(1993).Nova,theparaneoplasticRiantigen,ishomologoustoanRNA-bindingproteinandisspecificallyexpressedinthedevelopingmotorsystem.Neuron,11(4),657-672.48.Salemi,M.,Fraggetta,F.,Galia,A.,Pepe,P.,Cimino,L.,Condorelli,R.A.,&Calogero,A.E.(2013).Cerebellardegeneration-relatedautoantigen1(CDR1)geneexpressioninprostatecancercelllines.TheInternationaljournalofbiologicalmarkers,30;29(3):e288-9049.Tanaka,M.,Tanaka,K.,Onodera,O.,&Tsuji,S.(1995).Trialtoestablishananimalmodelofparaneoplasticcerebellardegenerationwithanti-Yoantibody:1.MousestrainsbearingdifferentMHCmoleculesproduceantibodiesonimmunizationwithrecombinantYoprotein,butdonotcausePurkinjecellloss.Clinicalneurologyandneurosurgery,97(1),95-100.50.Furneaux,H.M.,Rosenblum,M.K.,Dalmau,J.,Wong,E.,Woodruff,P.,Graus,F.,&Posner,J.B.(1990).SelectiveexpressionofPurkinje-cellantigenstumortissuefrompatientswithparaneoplasticcerebellardegeneration.NewEnglandJournalofMedicine,322(26),1844-1851.51.Shih,I.M.,Nesbit,M.,Herlyn,M.,&Kurman,R.J.(1998).AnewMel-CAM(CD146)-specificmonoclonalantibody,MN-4,onparaffin-embeddedtissue.Modernpathology:anofficialjournaloftheUnitedStatesandCanadianAcademyofPathology,Inc,11(11),1098-1106.52.Tsukamoto,Y.,Taira,E.,Miki,N.,&Sasaki,F.(2001).Theroleofgicerin,novelcelladhesionmolecule,indevelopment,regenerationandneoplasia.HistolHistopathol.2001;16(2):563-71.53.Tsukamoto,Y.,Taira,E.,Kotani,T.,Yamate,J.,Wada,S.,Takaha,N.,etal.,(1996).Involvementofgicerin,acelladhesionmolecule,intrachealdevelopmentandregeneration.CellGrowthDiffer.7(12),1761-1767.54.Schulz,C.,Petrig,V.,Wolf,K.,K.,M.,Becker,B.,&M.(2003).UpregulationofMCAMinprimarybronchialepithelialcellsfrompatientswithCOPD.EurRespirJ.2003;22(3):450-6.55.Simon,G.C.,Martin,R.J.,Smith,S.,Thaikoottathil,J.,Bowler,R.P.,Barenkamp,S.J.,&Chu,H.W.(2011).Up-regulationofMUC18inairwayepithelialcellsbyIL-13:implicationsinbacterialadherence.AmJRespCellMolBiol,44(5),606-613.56.Penning,T.M.,&Lerman,C.(2008).Genomicsofsmokingexposureandcessation:lessonsforcancerpreventionandtreatment.CancerPreventionResearch,1(2),80-83.57.Lampe,J.W.,Stepaniants,S.B.,Mao,M.,Radich,J.P.,Dai,H.,Linsley,P.S.,etal.,(2004).Signaturesofenvironmentalexposuresusingperipheralleukocytegeneexpression:tobaccosmoke.CancerEpidemiologyBiomarkers&Prevention,13(3),445-453.序列表<110>D·H·惠特尼M·埃拉斯霍夫<120>用于评估肺癌状态的方法<130>VRCT-008/02US<150>US62/024,456<151>2014-07-14<150>US62/160,403<151>2015-05-12<160>46<170>PatentInversion3.5<210>1<211>25<212>DNA<213>人工序列<220><223>TSPAN2探针序列<400>1tcaacattaagaagtcttaattcag25<210>2<211>25<212>DNA<213>人工序列<220><223>MCAM探针序列<400>2gctttaatccccatgaaggacagtg25<210>3<211>25<212>DNA<213>人工序列<220><223>ATP12A探针序列<400>3gcggtggagataccggcggcggcgc25<210>4<211>25<212>DNA<213>人工序列<220><223>NOVA1探针序列<400>4gacgaaattcagacatggagcatca25<210>5<211>25<212>DNA<213>人工序列<220><223>MIA探针序列<400>5atgacaccaaggcacaccagggacc25<210>6<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>6gacccgtctgtggctggtaatctct25<210>7<211>25<212>DNA<213>人工序列<220><223>EPHX3探针序列<400>7gctccactggaagagaggtataccc25<210>8<211>25<212>DNA<213>人工序列<220><223>CD177.2探针序列<400>8ttcctgtgtcccattgagcaggttg25<210>9<211>25<212>DNA<213>人工序列<220><223>CGREF1<400>9tagggtacagcacttaacgcaatct25<210>10<211>25<212>DNA<213>人工序列<220><223>BST1探针序列<400>10tgtttgccgtttcccgttccagaca25<210>11<211>25<212>DNA<213>人工序列<220><223>RNF150探针序列<400>11tggttaatccaagccgcagcctggt25<210>12<211>25<212>DNA<213>人工序列<220><223>CLDN22探针序列<400>12ggtgtttctcgtctccagttcttga25<210>13<211>25<212>DNA<213>人工序列<220><223>GABBR1探针序列<400>13tacggagccattacctgggcagtgc25<210>14<211>25<212>DNA<213>人工序列<220><223>SDC2探针序列<400>14tgagcctgcttctccgggctcccct25<210>15<211>25<212>DNA<213>人工序列<220><223>NKX3-1探针序列<400>15tttgtgctggctagtactccggtcg25<210>16<211>25<212>DNA<213>人工序列<220><223>LYPD2探针序列<400>16ttcttcaaggcattcggggctgggc25<210>17<211>25<212>DNA<213>人工序列<220><223>CDR1<400>17aacaactccgggtcttccagcgact25<210>18<211>25<212>DNA<213>人工序列<220><223>RPS4Y1探针序列<400>18taaaccgcaggaagtcagatgagtg25<210>19<211>25<212>DNA<213>人工序列<220><223>SLC7A11<400>19ggttgaagcaactagaagcgtgaca25<210>20<211>25<212>DNA<213>人工序列<220><223>CLDN10探针序列<400>20gacagcgtttcatgctcggatggcc25<210>21<211>25<212>DNA<213>人工序列<220><223>TKT探针序列<400>21ggtttattctctccagacggtcagg25<210>22<211>25<212>DNA<213>人工序列<220><223>RUNX1T1探针序列<400>22taacagggaggaggtcaaatctatc25<210>23<211>25<212>DNA<213>人工序列<220><223>AKR1C2探针序列<400>23tagctgtagcttactgaagtcgcca25<210>24<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>24gacgtgaaccagaccaatgaagggc25<210>25<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>25tcccatagggcaagtccggatgctc25<210>26<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>26agcaggaagggcaaaccactcccca25<210>27<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>27ccaagtgagagactccaggccctca25<210>28<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>28aggaggctgcacatcacgcttctca25<210>29<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>29ggaggctgcacatcacgcttctcac25<210>30<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>30acaagaagctggaaggttgggccac25<210>31<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>31aatgctcattttggtggacagccca25<210>32<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>32ctacatctaggagcagcagcgtctc25<210>33<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>33agcagcgtctcctgacacacctgcc25<210>34<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>34tcttgagccaagtttccgtgtgtca25<210>35<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>35tccgtgtgtcataatggtggtcccc25<210>36<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>36gttaacgaggttgttgcagaagtcc25<210>37<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>37ttggagagcaccaggctcacttggg25<210>38<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>38tctcaatgagcatcaacgtgtcctg25<210>39<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>39gcaggtcggacaccttccacacatg25<210>40<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>40agggccagcagtaataccgcgctca25<210>41<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>41gggccagcagtaataccgcgctcat25<210>42<211>25<212>DNA<213>人工序列<220><223>CD177.1探针序列<400>42gacccgtctgtggctggtaatctct25<210>43<211>25<212>DNA<213>人工序列<220><223>CD177.2探针序列<400>43ttcctgtgtcccattgagcaggttg25<210>44<211>25<212>DNA<213>人工序列<220><223>CD177.2探针序列<400>44tcccattgagcaggttgcaaactgg25<210>45<211>25<212>DNA<213>人工序列<220><223>CD177.2探针序列<400>45cctgaggaggccatcataacagtgt25<210>46<211>25<212>DNA<213>人工序列<220><223>CD177.2探针序列<400>46aggccatcataacagtgtgtggtcc25当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1