一种Keap1Kelch区多位点分子对接筛选食源性抗氧化寡肽的方法与流程
本发明涉及一种筛选食源性抗氧化寡肽的方法,特别涉及一种Keap1 Kelch区多位点分子对接筛选食源性抗氧化寡肽的方法。
背景技术:
当前,现代医学普遍认为,由自由基增加导致的氧化应激会造成组织细胞损伤,进而引起机体衰老,严重时甚至会诱发恶性肿瘤。因此,提高机体抗氧化能力就成了保持身体健康的一种重要手段。与其他提高机体抗氧化能力的方法相比,在日常饮食中食用含有天然抗氧化剂的食品,是一种成本低、效果好,性价比高的方法。而在天然抗氧化剂之中,食源性抗氧化肽占有重要的一席之地。
食源性肽是食物蛋白质降解的产物,按其大小可分为食源性寡肽(包含2-10个氨基酸残基)和食源性多肽(包含10个以上氨基酸残基)。一般而言,与食源性多肽相比,食源性寡肽具有更高的抗氧化性活性。抗氧化性作为食源性寡肽的重要生物活性之一,对其的研究也由来已久。
通常我们研究食源性抗氧化寡肽的方法由以下5个步骤构成:(1)酶解食源性蛋白;(2)按肽的分子量分离酶解粗肽;(3)利用体外化学试验、细胞试验和动物实验验证其抗氧化性;(4)对有抗氧化活性的粗肽进行质谱分析试验,得到其中寡肽的分子结构;(5)利用体外化学试验、细胞试验和动物实验验证这些寡肽的抗氧化性。这种方法效率低、耗资大且耗时长。同时,该方法不适于从大量寡肽中筛选高活性的抗氧化寡肽。因此,开发一种高效、经济、快速的食源性抗氧化寡肽的筛选方法已成当务之急。
技术实现要素:
本发明的目的是为了开发一种高效、经济、快速的分子对接筛选食源性抗氧化寡肽的方法而提供的一种Keap1 Kelch区多位点分子对接筛选食源性抗氧化寡肽的方法。
本发明提供的Keap1 Kelch区多位点分子对接筛选食源性抗氧化寡肽的方法,其方法如下所述:
步骤一、从Uniprot数据库中获得食源性蛋白的蛋白质一级结构,并以此为依据,构建食源性寡肽分子结构库;
步骤二、从PDB数据库中寻找能反映Keap1蛋白Kelch区与包含ETGE序列的Nrf2片段相互作用的PDB文件;
步骤三、分别以该PDB文件的Nrf2结合Keap1的结合位点、离Nrf2片段过近接触的Keap1上所有氨基酸残基和Nrf2片段关键位置ETGE序列这三个标准为参考,设计3个结合位点;
步骤四、以寡肽分子结构库的所有寡肽为配体,以PDB文件中Keap1蛋白Kelch区为受体,在步骤三中所设计的3个结合位点上进行分子对接;
步骤五、以结合能力为指标评价对接结果,比较并分析结合能力靠前的寡肽配体与活性中心的相互作用与PDB文件中原有Keap1蛋白Kelch区与包含ETGE序列的Nrf2片段相互作用,综合评价,初步筛选出食源性抗氧化寡肽。
步骤三通过设计3个结合位点,减少由于寡肽比PDB文件中包含ETGE序列的Nrf2片段小而造成的对对接试验的影响。
本发明的机理如下:
Keap1-Nrf2信号通路是机体应对氧化应激重要细胞防御机制之一。在正常生理条件下,Nrf2在机体内不断产生,而Keap1蛋白通过其BTB区结合形成同源二聚体并绑定在细胞的肌动蛋白上,并通过二聚体的两个Kelch区分别结合Nrf2蛋白Neh2区域的DLG与ETGE序列(其中Kelch区与ETGE序列之间的结合能力为Kelch区与DLG序列结合能力的100倍),进而使Nrf2蛋白泛素化和降解。而当机体处于氧化应激条件下,外来的氧化剂会改变Keap1蛋白BTB和IVR域的半胱氨酸残基构象,从而改变Keap1二聚体的构象,这种改变可能会干扰Keap1与低亲和力DLG序列相互作用,但不影响与ETGE序列相互作用。进而导致与Keap1结合的Nrf2不能被泛素化进而降解,最终导致细胞内Nrf2的积累。这部分Nrf2进入细胞核,并激活了一系列抗氧化和细胞保护作用的Ⅱ相解毒酶(包括HO-1,NQO1,SOD,和CAT)的相关基因的表达,因而提高机体的抗氧化能力。
因此,如果外来的抗氧化活性物质能够影响Keap1-Nrf2相互作用,从而调高细胞内Nrf2含量,就能激活通路,提高机体的抗氧化能力。这样影响Keap1-Nrf2相互作用的抗氧化活性物质被称为Keap1-Nrf2相互作用抑制剂。Keap1-Nrf2相互作用抑制剂可分为两大类:间接抑制剂和直接抑制剂。间接抑制剂就是改变Keap1蛋白BTB和IVR域的关键半胱氨酸残基构象,从而抑制Keap1-Nrf2相互作用进而激活通路。而直接抑制剂是直接与Keap1蛋白Kelch区结合,占据Keap1与Nrf2结合的位置,从而抑制Keap1-Nrf2相互作用进而激活通路。与间接抑制剂相比,直接抑制剂的特异性更强,且没有间接抑制剂由于其具有改变半胱氨酸残基构象能力而存在的影响体内其他含有关键半胱氨酸残基蛋白正常生理功能的副作用。因而直接抑制剂比间接抑制剂更适合去研究。
在Keap1-Nrf2相互作用中,其Keap1蛋白Kelch区与Nrf2蛋白ETGE序列的结合尤为关键,如果外来的抗氧化物质能占据Keap1蛋白Kelch区与Nrf2蛋白ETGE序列的结合位点,则其极大可能会直接抑制Keap1-Nrf2相互作用,从而激活Keap1-Nrf2信号通路,提高机体的抗氧化能力。
基于以上机理,本发明中,首先从Uniprot数据库中找到食源性蛋白的蛋白质一级结构,并以此为依据构建食源性寡肽分子结构库;其次,选用PDB数据库中能反映Keap1蛋白Kelch区与包含ETGE序列的Nrf2片段相互作用的PDB文件为受体文件,并分别以受体文件中包含ETGE序列的Nrf2片段与Keap1的结合位点、离Nrf2片段过近接触的Keap1上所有氨基酸残基和Nrf2片段关键位置ETGE序列这三个标准为参考,设计3个新的结合位点;然后,利用分子对接软件,分别在选出的3个位点进行受体和寡肽分子结构库的分子对接,初步选定具有抗氧化作用的食源性寡肽;最后利用体外化学试验进行再次筛选,验证其抗氧化性。
本发明的有益效果:
本发明通过基于Keap1蛋白Kelch区的分子对接筛选,能高效、快速地从几千个乃至几万个食源性寡肽中筛选出几十个具有较强抗氧化活性的食源性寡肽。同时,通过设计多个结合位点,减少由于寡肽比包含ETGE序列的Nrf2片段小而造成的对对接试验的影响。后续试验还能以筛选得到的食源性寡肽为蓝本,利用体外化学试验和细胞试验逐次筛选出具有抗氧化功能的食源性寡肽,从而有效提高试验的效率,减少试验周期。
附图说明
图1为本发明中PDB文件(PDB ID:2FLU)Keap1蛋白Kelch区与包含ETGE序列的Nrf2片段相互作用示意图。
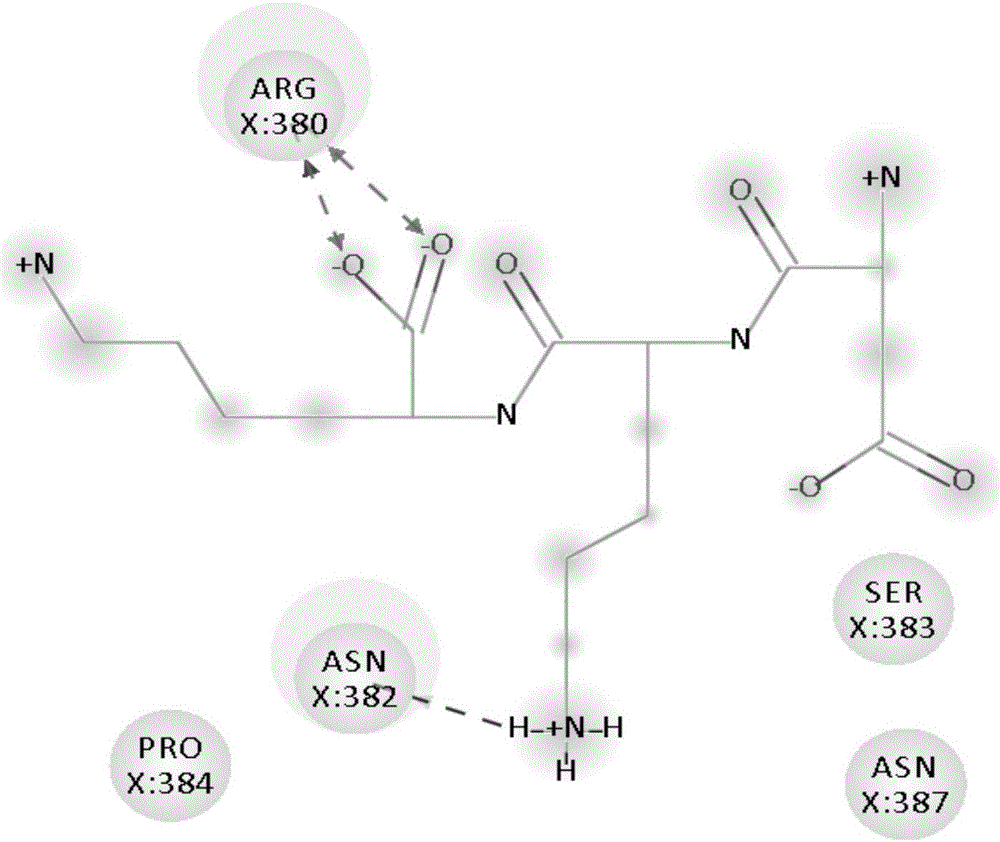
图2为本发明中Asp-Lys-Lys与Keap1蛋白Kelch区相互作用示意图。
图3为本发明中Asp-Lys-Lys荧光偏振试验结果示意图。
具体实施方式
实施例1一种基于Keap1 Kelch区,蛋清源抗氧化三肽的多位点分子对接筛选。
(一)分子对接筛选步骤如下:
1、从Uniprot数据库中获得蛋清蛋白的蛋白质一级结构,并以此为依据,构建蛋清源三肽分子结构库;
利用Uniprot蛋白数据库,得到鸡蛋蛋清蛋白,并获取其蛋白质一级结构,以此为依据建立蛋清源三肽分子结构库。
2、从PDB数据库中寻找能反映Keap1蛋白Kelch区与包含ETGE序列的Nrf2片段相互作用的PDB文件(PDB ID:2FLU);
利用PDB数据库,搜索包含人源性Keap1的全部PDB文件,从其中筛选能反映Keap1-Nrf2相互作用的PDB文件(PDB ID:2FLU),并下载下来。
3、分别以该PDB文件的Nrf2结合Keap1的结合位点、离Nrf2片段过近接触的Keap1上所有氨基酸残基和Nrf2片段关键位置ETGE序列这三个标准为参考,设计3个结合位点;
本步骤中,首先分析PDB文件(PDB ID:2FLU)中,Keap1蛋白Kelch区与包含ETGE序列的Nrf2片段的结合情况并找出其原始结合位点,在该PDB文件中,Keap1蛋白Kelch区与Nrf2片段(包含ETGE序列,由16个氨基酸残基组成),于位点(x:2.28,y:6.23,z:1.63;半径:)处结合;其次,由于该Nrf2片段由16个氨基酸残基组成,远远大于由3个氨基酸残基组成的三肽,为了减少这种差异所造成对对接试验的影响,分别以该位点、距离Nrf2片段过近接触的Keap1上所有氨基酸残基和Nrf2片段关键位置ETGE序列三个标准作为设计新结合位点构建的依据,经过调整,最终得到3个结合位点:结合位点1(中心坐标:x:-4,y:6,z:0;半径:)、结合位点2(中心坐标:x:5,y:9,z:1;半径:)和结合位点3(中心坐标:x:7.36,y:8.33,z:1.77;半径:)。
4、以蛋清源三肽分子结构库的所有三肽为配体,以PDB文件中Keap1蛋白Kelch区为受体,在步骤3所设计的3个结合位点上进行分子对接,主要步骤如下:
(1)对步骤3构建的分子结构库的所有三肽配体附加MMFF力场,并进行能量最小化直至配体分子构象不改变为止。
(2)对步骤1获得的2FLU文件进行去除水分子与Nrf2片段,加氢,处理,获得受体文件。
(3)对步骤(1)获得的受体文件,附加CHARMm力场。
(4)在附加CHARMm力场的受体文件上,分别设定步骤2得到的三个结合位点。
(5)以步骤(3)获得的受体为受体,分别在选出的三个位点,以步骤(1)获得的三肽为配体,进行分子对接筛选。
5、以结合能力为指标评价对接结果,综合评价,初步筛选出蛋清源抗氧化三肽。
对步骤4筛选所得到的对接结果排序,初步选定具有抗氧化作用的蛋清源三肽。为了进一步提高筛选结果的准确性,根据对接结果的排序,比较并分析排序靠前的三肽配体与活性中心的相互作用与PDB文件中原有Keap1蛋白Kelch区与包含ETGE序列的Nrf2片段相互作用(如图1所示),选定具有抗氧化作用的蛋清源三肽。
经过以上筛选,分别在三个位点中筛选排序前十的三肽。它们与Keap1蛋白Kelch区的部分氨基酸残基结合,产生相互作用。这些氨基酸残基也在Keap1蛋白Kelch区与包含ETGE序列的Nrf2片段相互作用中发挥作用。这进一步说明了其与Keap1蛋白Kelch区的结合能调节该通路,从而发挥其抗氧化作用。
(二)利用荧光偏振试验筛选具有抗氧化功能的蛋清源三肽。
通过上述的分子对接筛选,从蛋清源三肽配体库中筛选出一系列能和keap1于其Kelch区域新设定的三个位点处结合的三肽。然后对其进行荧光偏振试验,验证其抗氧化性。
1、我们从分子对接筛选得到的蛋清源三肽中,随意选择一个化合物:Asp-Lys-Lys,其分子结构如下式所示。其与Keap1蛋白Kelch区相互作用情况如图3所示。选用该化合物进行如下荧光偏振试验。
荧光偏振所用的孔板为黑色无结合能力的384孔板,每孔加40微升待测液。试验组待测液由10ul PBS,10ul 4mM Asp-Lys-Lys,10ul梯度的Keap1蛋白Kelch区和10ul 200nM探针构成。对照组待测液由20ul PBS,10ul梯度的Keap1蛋白Kelch区和10ul 200nM探针构成。探针为FITC标记的Nrf2片段(包含ETGE序列,由9个氨基酸残基组成)加完孔后,盖盖,于室温避光条件下震荡30min后测量Fp值,以Keap1蛋白Kelch区与包含ETGE序列的Nrf2片段的Kd值最为评价指标。结果如图4所示,加入Asp-Lys-Lys的试验组其Kd值远远高于对照组的Kd的值,这表明Asp-Lys-Lys能抑制keap1-Nrf2的相互作用。结论:证明了Asp-Lys-Lys在体外化学试验中具有结合keap1的能力,可进一步用于细胞试验。
2、通过分子对接筛选后得到的蛋清源三肽,都可以按照Asp-Lys-Lys的方法,进行荧光偏振试验,从而确定其在体外化学试验中是否具有结合keap1的能力,从而确定其抗氧化性。
实施例2一种基于Keap1 Kelch区,大豆源抗氧化四肽的多位点分子对接筛选。
方法与实施例1相同,不同点在于:建立分子结构库;从Uniprot数据库中找到大豆蛋白的蛋白质一级结构,以此为依据建立大豆源四肽分子结构库。
SEQUENCE LISTING
<110> 吉林大学
<120> 一种Keap1 Kelch 区多位点分子对接筛选食源性抗氧化寡肽的方法
<130> 2016
<160> 1
<170> PatentIn version 3.5
<210> 1
<211> 624
<212> PRT
<213> 人(human)
<400> 1
Met Gln Pro Asp Pro Arg Pro Ser Gly Ala Gly Ala Cys Cys Arg Phe
1 5 10 15
Leu Pro Leu Gln Ser Gln Cys Pro Glu Gly Ala Gly Asp Ala Val Met
20 25 30
Tyr Ala Ser Thr Glu Cys Lys Ala Glu Val Thr Pro Ser Gln His Gly
35 40 45
Asn Arg Thr Phe Ser Tyr Thr Leu Glu Asp His Thr Lys Gln Ala Phe
50 55 60
Gly Ile Met Asn Glu Leu Arg Leu Ser Gln Gln Leu Cys Asp Val Thr
65 70 75 80
Leu Gln Val Lys Tyr Gln Asp Ala Pro Ala Ala Gln Phe Met Ala His
85 90 95
Lys Val Val Leu Ala Ser Ser Ser Pro Val Phe Lys Ala Met Phe Thr
100 105 110
Asn Gly Leu Arg Glu Gln Gly Met Glu Val Val Ser Ile Glu Gly Ile
115 120 125
His Pro Lys Val Met Glu Arg Leu Ile Glu Phe Ala Tyr Thr Ala Ser
130 135 140
Ile Ser Met Gly Glu Lys Cys Val Leu His Val Met Asn Gly Ala Val
145 150 155 160
Met Tyr Gln Ile Asp Ser Val Val Arg Ala Cys Ser Asp Phe Leu Val
165 170 175
Gln Gln Leu Asp Pro Ser Asn Ala Ile Gly Ile Ala Asn Phe Ala Glu
180 185 190
Gln Ile Gly Cys Val Glu Leu His Gln Arg Ala Arg Glu Tyr Ile Tyr
195 200 205
Met His Phe Gly Glu Val Ala Lys Gln Glu Glu Phe Phe Asn Leu Ser
210 215 220
His Cys Gln Leu Val Thr Leu Ile Ser Arg Asp Asp Leu Asn Val Arg
225 230 235 240
Cys Glu Ser Glu Val Phe His Ala Cys Ile Asn Trp Val Lys Tyr Asp
245 250 255
Cys Glu Gln Arg Arg Phe Tyr Val Gln Ala Leu Leu Arg Ala Val Arg
260 265 270
Cys His Ser Leu Thr Pro Asn Phe Leu Gln Met Gln Leu Gln Lys Cys
275 280 285
Glu Ile Leu Gln Ser Asp Ser Arg Cys Lys Asp Tyr Leu Val Lys Ile
290 295 300
Phe Glu Glu Leu Thr Leu His Lys Pro Thr Gln Val Met Pro Cys Arg
305 310 315 320
Ala Pro Lys Val Gly Arg Leu Ile Tyr Thr Ala Gly Gly Tyr Phe Arg
325 330 335
Gln Ser Leu Ser Tyr Leu Glu Ala Tyr Asn Pro Ser Asp Gly Thr Trp
340 345 350
Leu Arg Leu Ala Asp Leu Gln Val Pro Arg Ser Gly Leu Ala Gly Cys
355 360 365
Val Val Gly Gly Leu Leu Tyr Ala Val Gly Gly Arg Asn Asn Ser Pro
370 375 380
Asp Gly Asn Thr Asp Ser Ser Ala Leu Asp Cys Tyr Asn Pro Met Thr
385 390 395 400
Asn Gln Trp Ser Pro Cys Ala Pro Met Ser Val Pro Arg Asn Arg Ile
405 410 415
Gly Val Gly Val Ile Asp Gly His Ile Tyr Ala Val Gly Gly Ser His
420 425 430
Gly Cys Ile His His Asn Ser Val Glu Arg Tyr Glu Pro Glu Arg Asp
435 440 445
Glu Trp His Leu Val Ala Pro Met Leu Thr Arg Arg Ile Gly Val Gly
450 455 460
Val Ala Val Leu Asn Arg Leu Leu Tyr Ala Val Gly Gly Phe Asp Gly
465 470 475 480
Thr Asn Arg Leu Asn Ser Ala Glu Cys Tyr Tyr Pro Glu Arg Asn Glu
485 490 495
Trp Arg Met Ile Thr Ala Met Asn Thr Ile Arg Ser Gly Ala Gly Val
500 505 510
Cys Val Leu His Asn Cys Ile Tyr Ala Ala Gly Gly Tyr Asp Gly Gln
515 520 525
Asp Gln Leu Asn Ser Val Glu Arg Tyr Asp Val Glu Thr Glu Thr Trp
530 535 540
Thr Phe Val Ala Pro Met Lys His Arg Arg Ser Ala Leu Gly Ile Thr
545 550 555 560
Val His Gln Gly Arg Ile Tyr Val Leu Gly Gly Tyr Asp Gly His Thr
565 570 575
Phe Leu Asp Ser Val Glu Cys Tyr Asp Pro Asp Thr Asp Thr Trp Ser
580 585 590
Glu Val Thr Arg Met Thr Ser Gly Arg Ser Gly Val Gly Val Ala Val
595 600 605
Thr Met Glu Pro Cys Arg Lys Gln Ile Asp Gln Gln Asn Cys Thr Cys
610 615 620
- 还没有人留言评论。精彩留言会获得点赞!