用于选择南方根结线虫抗性大豆植物的方法和组合物与流程
本申请是中国专利申请号200880105732.x,申请日2008年08月06日,发明名称“用于选择南方根结线虫抗性大豆植物的方法和组合物”的分案申请(申请号为201410020821.1,分案申请提交日为2014年1月13日,发明名称为“用于选择南方根结线虫抗性大豆植物的方法和组合物”)的分案申请。相关申请的交叉引用本申请要求于2008年5月23日提交的美国临时申请61/055519和2007年8月7日提交的美国临时申请60/963836的优先权。序列表的引入包含于2008年8月1日创建的、322千字节(在microsoft中计数)的名为“pa_01413.txt”的文件的序列表包括45个核苷酸序列,并且在这里全部引入作为参考。本发明属于植物育种和抗病性的领域。更具体地说,本发明包括培育含有与南方根结线虫(srkn)抗病性相关的数量性状基因座(qtl)的大豆植物的方法,该病是与病原体南方根结线虫(meloidogyneincognita)相关的病害。本发明进一步涉及遗传标记在确定病原体南方根结线虫抗病性的qtl中的应用。本发明进一步包括含有用于渗入优良种质中的赋予抗病性的qtl的种质和该种质在针对srkn抗性的育种计划中的应用。
背景技术:
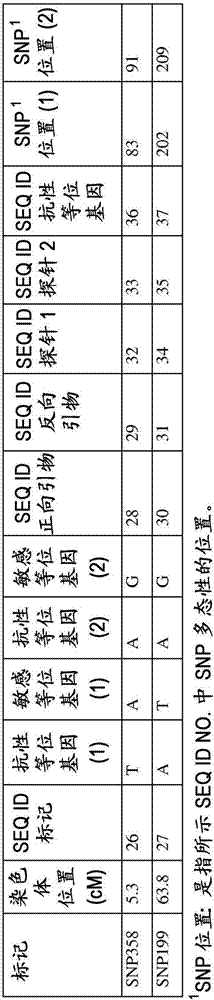
:根结线虫(根结线虫属(meloidogyne)的种)是感染许多宿主并且在世界上引起广泛的作物损害的植物病原体。在美国东南部,南方根结线虫(meloidogyneincognita),在本文中称为srkn,是大豆(glycinemax(l))的主要害虫。对使用杀线虫剂的限制,包括取消dbcp(1,2-二溴-3-氯丙烷)和edb(二溴乙烷),已经促进了替代的srkn控制方法的发展(harris等人.cropsci.43:1848-1851(2003))。培育srkn抗性大豆栽培种是最有效的降低寄生虫引起的产量和栽培者利益损失的方法(li等人.,theorapplgenet103:1167-1173(2001))。因此,已经开发了大量的srkn抗性栽培种(ha等人.,cropscience44:758-763(2004))。已经检查了优良大豆品种和保藏的种质的srkn抗性。srkn抗性的两种来源是pi96354和palmetto(美洲蒲葵)。对来自usda大豆种质保藏中心(urbana,il)的2,370种大豆保藏物的表型筛查确定了pi96354是该保藏中心抗性最高的来源(luzzi,等人.cropsci.27:258-262(1987))。高度srkn抗性品种pi96354与srkn敏感的bossier之间的杂交允许将srkn抗性的主要数量性状基因座(qtl)定位于可公开获得的大豆遗传连锁图的连锁群(lg)o并且将次要qtl定位于lg-g(tamulonis等人.,cropsci.37:1903-1909(1997))。forrest是显示srkn抗性并且在大豆品种开发中用作srkn抗性的亲本来源的另外一个大豆品种。forrest和许多其它优良品种中的srkn抗性的祖先来源被认为是palmetto(ha等人.cropsci.44:758-763(2004))。在forrest与敏感的bossier杂交的研究中鉴定了主要srkn抗性qtlrmi(luzzi等人.,jheredity85:484-486(1994))。对48个大豆品种的谱系分析和使用简单序列重复(ssr)标记的基因型分型提供了证据证明srkn抗性品种遗传了来自祖先抗性来源的连锁群o上的主要srkn抗性qtl(rmi)(ha等人.cropsci.44:758-763(2004))。通过使用对srkn抗性qtl的标记辅助的选择可以大大促进srkn抗性大豆的培育。单核苷酸多态性(snp)和(ssr)可以用作定位与srkn抗性有关的qtl的遗传标记。优选snp,因为可以获得使用snp标记平台的自动化、高通量筛选技术,这些技术可以缩短选择和向大豆植物中渗入srkn抗性的时间。此外,snp标记是理想的,因为特定snp等位基因来自于特定物种的现存群体中的独立来源的可能性非常低。因此,snp标记可用于跟踪和辅助srkn抗性等位基因的基因渗入,特别是在srkn抗性单元型的情况下。可以使用snp标记筛选大豆遗传连锁图的lg-o和lg-g上的srkn抗性qtl(ha等人.,cropsci.47(2)s73-s82(2007))。本发明提供并包括用于筛选和选择包含srkn抗性qtl的大豆植物的方法和组合物。技术实现要素:本发明提供一种将等位基因渗入大豆植物中的方法,包括以下步骤:(a)提供大豆植物群体;(b)根据选自seqidno:1-4的大豆基因组核酸标记,对该群体中的至少一个大豆植物进行基因型分型;(c)从该群体中选择至少一个包含至少一个与srkn抗性相关的等位基因的大豆植物,其中该srkn抗性等位基因选自seqidno:21-25。本发明进一步提供该大豆植物群体可以通过使至少一个srkn抗性大豆植物与至少一个srkn敏感性大豆植物杂交而产生。本发明进一步包括通过以下方法产生的优良大豆植物:(a)提供大豆植物群体;(b)根据选自seqidno:1-4的大豆基因组核酸标记,对该群体中的至少一个大豆植物进行基因型分型;(c)从该群体中选择至少一个包含至少一个与srkn抗性相关的等位基因的大豆植物,其中该srkn抗性等位基因选自seqidno:21-25。本发明进一步提供所述优良大豆植物可以显示转基因性状,其中该转基因性状选自除草剂耐受性、产量增加、昆虫控制、真菌病抗性、病毒抗性、线虫抗性、细菌病抗性、支原体病抗性、油产生改变、高产油量、高蛋白质产量、发芽和幼苗生长的控制、动物和人类营养增强、低棉子糖、环境应激抗性、可消化性提高、加工性状改善、风味改善、固氮、杂种种子的产生和变应原性降低。在进一步的实施方案中,赋予的除草剂耐受性选自对草甘膦、麦草畏、草丁膦、磺脲、溴苯腈、2,4-二氯苯氧基乙酸和达草灭除草剂的耐受性。本发明提供的方法可以进一步包括以下步骤:(d)测定选择的大豆植物对诱导srkn的病原体的抗性。在该方法的某些实施方案中,在步骤(b)中通过选自单碱基延伸(sbe)、等位基因特异性引物延伸测序(aspe)、dna测序、rna测序、基于微阵列的分析、通用pcr、等位基因特异性延伸、杂交、质谱法、连接、延伸-连接和瓣核酸内切酶介导的分析的分析方法实现基因型分型。本发明进一步提供一种将等位基因渗入大豆植物中的方法,包括:(a)提供大豆植物群体;(b)用至少一种核酸标记筛查该群体;和(c)从该群体中选择一个或多个包含srkn抗性等位基因的大豆植物,其中该srkn抗性等位基因是选自srkn抗性基因座的等位基因,其中在其一个或多个基因座处的一个或多个等位基因选自srkn抗性等位基因1、srkn抗性等位基因2、srkn抗性等位基因3和srkn抗性等位基因4。本发明进一步包括通过以下方法产生的优良大豆植物:(a)提供大豆植物群体;(b)用至少一种核酸标记筛查该群体;和(c)从该群体中选择一个或多个包含srkn抗性等位基因的大豆植物,其中该srkn抗性等位基因是选自srkn抗性基因座的等位基因,其中在其一个或多个基因座处的一个或多个等位基因选自srkn抗性等位基因1、srkn抗性等位基因2、srkn抗性等位基因3和srkn抗性等位基因4。通过本发明的任一种方法产生的大豆或优良大豆植物可以进一步包含转基因性状。该转基因性状可以选自除草剂耐受性、产量增加、昆虫控制、真菌病抗性、病毒抗性、线虫抗性、细菌病抗性、支原体病抗性、油产生改变、高产油量、高蛋白质产量、发芽和幼苗生长的控制、动物和人类营养增强、低棉子糖、环境应激抗性、可消化性提高、加工性状改善、风味改善、固氮、杂种种子的产生和变应原性降低。除草剂耐受性性状可以选自草甘膦、麦草畏、草丁膦、磺脲、溴苯腈、2,4-二氯苯氧基乙酸和达草灭除草剂耐受性。本发明包括基本上纯化的核酸分子,其包含选自seqidno:1-25、38-45及其互补序列的核酸序列。这些分离的核酸可以用于实施本发明的方法。用于检测代表大豆dna中多态性的分子标记的分离的核酸分子可以包含至少15个包括或者邻近该多态性的核苷酸,其中该核酸分子与包括或者邻近该多态性的dna任一条链中的相同数目连续核苷酸的序列至少90%相同,并且其中该分子标记选自seqidno:1-4和26-27。该分离的核酸可以进一步包含可检测标记或者提供可检测标记的掺入。这种可检测标记可以选自同位素、荧光团、抗氧化剂、还原剂、核苷酸和半抗原。这种可检测标记可以通过化学反应添加到核酸上或者通过酶反应掺入。此处提供的分离的核酸分子可以包含包括或者邻近该多态性的dna任一条链上的至少16、17、18或20个核苷酸。在其它实施方案中,该分离的核酸可以与所述分子标记的至少一个等位基因在严格杂交条件下杂交。与缺乏srkn抗性等位基因的大豆植物相比,通过本发明的方法选择的大豆植物在srkn的存在下可以进一步显示出提高的谷物产量。在这些方法中,与缺乏srkn抗性等位基因的植物相比,选择的一种或多种大豆植物在srkn的存在下可以显示出至少0.5bu/a、1.0bu/a或1.5bu/a的谷物产量提高。特别地,本发明涉及以下各项:1.一种将等位基因渗入大豆植物的方法,包括:a.提供大豆植物群体;b.根据选自seqidno:1-4的大豆基因组核酸标记,对该群体中的至少一个大豆植物进行基因型分型;c.从所述群体中选择至少一个包含至少一个与srkn抗性相关的等位基因的大豆植物,其中该srkn抗性等位基因选自seqidno:21-25。2.如第1项所述的方法,其中,所述群体是通过使至少一个srkn抗性大豆植物与至少一个srkn敏感性大豆植物杂交形成群体而产生的。3.如第1项所述的方法,其中,所述选择的大豆植物显示不差于大约3.5的srkn抗性反应等级。4.如第1项所述的方法,进一步包括步骤(d)测定所述选择的大豆植物对诱导srkn的病原体的抗性。5.如第1项所述的方法,其中所述基因型通过选自单碱基延伸(sbe)、等位基因特异性引物延伸测序(aspe)、dna测序、rna测序、基于微阵列的分析、通用pcr、等位基因特异性延伸、杂交、质谱法、连接、延伸-连接和瓣核酸内切酶介导的分析的分析方法来确定。6.如第1项所述的方法,其中,所述大豆基因组核酸标记是seqidno:3。7.通过以下步骤产生的优良大豆植物:a.提供大豆植物群体;b.根据选自seqidno:1-4的大豆基因组核酸标记,对该群体中的至少一个大豆植物进行基因型分型;c.从所述群体中选择至少一个包含至少一个与srkn抗性相关的等位基因的大豆植物,其中该srkn抗性等位基因选自seqidno:21-25。8.如第7项所述的优良大豆植物,其中,所述优良大豆植物显示转基因性状。9.如第8项所述的优良大豆植物,其中,所述转基因性状选自除草剂耐受性、产量增加、昆虫控制、真菌病抗性、病毒抗性、线虫抗性、细菌病抗性、支原体病抗性、油产生改变、高产油量、高蛋白质产量、发芽和幼苗生长的控制、动物和人类营养增强、低棉子糖、环境应激抗性、可消化性提高、加工性状改善、风味改善、固氮、杂种种子的产生和变应原性降低。10.如第9项所述的优良大豆植物,其中,所述除草剂耐受性是对至少一种选自草甘膦、麦草畏、草丁膦、磺脲、溴苯腈、2,4-二氯苯氧基乙酸和达草灭除草剂的除草剂的耐受性。11.一种将等位基因渗入大豆植物中的方法,包括:a.提供大豆植物群体;b.用至少一种核酸标记筛查该群体;c.从该群体中选择一个或多个包含srkn抗性等位基因的大豆植物,其中该srkn抗性等位基因是选自srkn抗性基因座的等位基因,其中在其一个或多个基因座处的一个或多个等位基因选自srkn抗性等位基因1、srkn抗性等位基因2、srkn抗性等位基因3和srkn抗性等位基因4。12.如第11项所述的方法,其中所述群体是通过使至少一个srkn抗性大豆植物与至少一个srkn敏感性大豆植物杂交形成群体而产生的。13.如第11项所述的方法,其中,所述选择的大豆植物显示不差于大约3.5的srkn抗性反应等级。14.如第11项所述的方法,进一步包括步骤(d)测定所述选择的大豆植物对诱导srkn的病原体的抗性。15.如第11项所述的方法,其中,基因型通过选自单碱基延伸(sbe)、等位基因特异性引物延伸测序(aspe)、dna测序、rna测序、基于微阵列的分析、通用pcr、等位基因特异性延伸、杂交、质谱法、连接、延伸-连接和瓣核酸内切酶介导的分析的分析方法来确定。16.如第11项所述的方法,其中,所述核酸标记选自seqidno:1-4。17.如第11项所述的方法,其中,与缺乏srkn抗性等位基因的大豆植物相比,所述选择的一个或多个大豆植物在srkn的存在下显示谷物产量增加。18.如第11项所述的方法,其中,与缺乏srkn抗性等位基因的植物相比,所述选择的一个或多个大豆植物在srkn的存在下显示至少0.5bu/a的谷物产量增加。19.如第11项所述的方法,其中,与缺乏srkn抗性等位基因的植物相比,所述选择的一个或多个大豆植物在srkn的存在下显示至少1.0bu/a的谷物产量增加。20.如第11项所述的方法,其中,与缺乏srkn抗性等位基因的植物相比,所述选择的一个或多个大豆植物在srkn的存在下显示至少1.5bu/a的谷物产量增加。21.通过以下步骤产生的优良大豆植物:a.提供大豆植物群体;b.用至少一种核酸标记筛查该群体;c.从该群体中选择一个或多个包含srkn抗性等位基因的大豆植物,其中该srkn抗性等位基因是选自srkn抗性基因座的等位基因,其中在其一个或多个基因座处的一个或多个等位基因选自srkn抗性等位基因1、srkn抗性等位基因2、srkn抗性等位基因3和srkn抗性等位基因4。22.如第21项所述的优良大豆植物,其中,所述优良大豆植物显示转基因性状。23.如第22项所述的优良大豆植物,其中,所述转基因性状选自除草剂耐受性、产量增加、昆虫控制、真菌病抗性、病毒抗性、线虫抗性、细菌病抗性、支原体病抗性、油产生改变、高产油量、高蛋白质产量、发芽和幼苗生长的控制、动物和人类营养增强、低棉子糖、环境应激抗性、可消化性提高、加工性状改善、风味改善、固氮、杂种种子的产生和变应原性降低。24.如第23项所述的优良大豆植物,其中,所述除草剂耐受性是对至少一种选自草甘膦、麦草畏、草丁膦、磺脲、溴苯腈、2,4-二氯苯氧基乙酸和达草灭除草剂的除草剂的耐受性。25.用于检测与srkn抗性相关的基因座的基本上纯化的核酸分子,包含选自seqidno:1-25、38-45及其互补序列的核酸序列。26.一种鉴定大豆植物中的srkn抗性等位基因的方法,包括检测与选自seqidno:1-4的snp标记相关的基因座。27.一种分离的核酸分子,用于检测代表大豆dna中的多态性的分子标记,其中所述核酸分子包含至少15个包括或者邻近所述多态性的核苷酸,其中所述核酸分子与包括或者邻近所述多态性的dna任一条链中的相同数目连续核苷酸的序列至少90%相同,并且其中所述分子标记选自seqidno:1-4。28.如第27项所述的分离的核酸,其中,所述核酸进一步包含可检测标记或者提供可检测标记的掺入。29.如第28项所述的分离的核酸,其中,所述可检测标记选自同位素、荧光团、氧化剂、还原剂、核苷酸和半抗原。附图说明引入说明书中并且作为其一部分的附图说明了本发明的实施方案,并且与说明书一起用来解释本发明的原理。在附图中:图1.用于srkn抗性基因座的snp标记的列表,指示出了每种标记的抗性和敏感等位基因。对于标记ns0102683,有两个抗性等位基因:a和g,其中a是抗性的,g是高度抗性的。图2.用于检测srkn抗性的snp标记。snp358的抗性单元型是ta,snp199的抗性单元型是aa。每个标记处来自两个多态性的单元型可以用于筛选srkn抗性。核酸序列的简要说明seqidno:1是来源于大豆的与srkn抗性基因座rmi相关的基因组序列。seqidno:2是来源于大豆的与srkn抗性基因座rmi相关的基因组序列。seqidno:3是来源于大豆的与srkn抗性基因座rmi相关的基因组序列。seqidno:4是来源于大豆的与srkn抗性基因座rmi相关的基因组序列。seqidno:5是用于扩增seqidno:1的正向pcr引物。seqidno:6是用于扩增seqidno:1的反向pcr引物。seqidno:7是用于扩增seqidno:2的正向pcr引物。seqidno:8是用于扩增seqidno:2的反向pcr引物。seqidno:9是用于扩增seqidno:3的正向pcr引物。seqidno:10是用于扩增seqidno:3的反向pcr引物。seqidno:11是用于扩增seqidno:4的正向pcr引物。seqidno:12是用于扩增seqidno:4的反向pcr引物。seqidno:13是用于检测seqidno:1的srkn抗性基因座的探针。seqidno:14是用于检测seqidno:1的srkn抗性基因座的第二探针。seqidno:15是用于检测seqidno:2的srkn抗性基因座的探针。seqidno:16是用于检测seqidno:2的srkn抗性基因座的第二探针。seqidno:17是用于检测seqidno:3的srkn抗性基因座的探针。seqidno:18是用于检测seqidno:3的srkn抗性基因座的第二探针。seqidno:19是用于检测seqidno:4的srkn抗性基因座的探针。seqidno:20是用于检测seqidno:4的srkn抗性基因座的第二探针。seqidno:21是对应于seqidno:1的srkn抗性等位基因1。seqidno:22是对应于seqidno:2的srkn抗性等位基因2。seqidno:23是对应于seqidno:3的srkn抗性等位基因3。seqidno:24是对应于seqidno:4的srkn抗性等位基因4。seqidno:25是对应于seqidno:4的第二srkn抗性等位基因基序。seqidno:26是来源于大豆的对应于srkn抗性基因座rmi的基因组序列。seqidno:27是来源于大豆的对应于lg-g上的srkn抗性基因座的基因组序列。seqidno:28是用于扩增seqidno:26的正向pcr引物。seqidno:29是用于扩增seqidno:26的反向pcr引物。seqidno:30是用于扩增seqidno:27的正向pcr引物。seqidno:31是用于扩增seqidno:27的反向pcr引物。seqidno:32是用于检测seqidno:26的srkn抗性基因座的探针。seqidno:33是用于检测seqidno:26的srkn抗性基因座的第二探针。seqidno:34是用于检测seqidno:27的srkn抗性基因座的探针。seqidno:35是用于检测seqidno:27的srkn抗性基因座的第二探针。seqidno:36是对应于seqidno:26的srkn抗性等位基因5。seqidno:37是对应于seqidno:27的srkn抗性等位基因6。seqidno:38是用于检测seqidno:2的srkn抗性基因座的杂交探针。seqidno:39是用于检测seqidno:2的srkn抗性基因座的第二杂交探针。seqidno:40是用于检测seqidno:3的srkn抗性基因座的杂交探针。seqidno:41是用于检测seqidno:3的srkn抗性基因座的第二杂交探针。seqidno:42是用于检测seqidno:2的srkn抗性基因座的正向单碱基延伸探针。seqidno:43是用于检测seqidno:2的srkn抗性基因座的反向单碱基延伸探针。seqidno:44是用于检测seqidno:3的srkn抗性基因座的正向单碱基延伸探针。seqidno:45是用于检测seqidno:3的srkn抗性基因座的反向单碱基延伸探针。发明详述本文提供的定义和方法限定本发明,并指导本领域普通技术人员实施本发明。除非另有说明,应当根据相关领域的普通技术人员的常规用法来理解术语。在分子生物学中的常用术语的定义也可以在以下文献中找到:alberts等人,molecularbiologyofthecell,第5版,garlandpublishing,inc.:newyork,2007;rieger等人,glossaryofgenetics:classicalandmolecular,第5版,springer-verlag:newyork,1991;king等人,adictionaryofgenetics,第6版,oxforduniversitypress:newyork,2002;和lewin,genesix,oxforduniversitypress:newyork,2007。使用如37cfr§1.822所述的dna碱基命名法。“等位基因”指特定的基因座处的替代序列;等位基因的长度可以小到1个核苷酸碱基,但通常较大。等位基因序列可以表示为核酸序列或由该核酸序列编码的氨基酸序列。“基因座”是在通常由参照点发现的基因组序列上的位置;例如,作为基因或基因部分或基因间区域的短dna序列。本发明的基因座在群体中包含一种或多种多态性,即,在一些个体中存在替代的等位基因。如本文所用,“多态性”是指在一个或多个个体的群体中存在一个或多个基因座处的核酸序列的一种或多种变异。变异可以包括但不限于一个或多个碱基的变化、一个或多个核苷酸的插入或一个或多个核苷酸的缺失。多态性的产生可能是由于核酸复制中的随机过程,通过诱变,由于移动的基因组元件,拷贝数变异,和减数分裂过程,如不等交换、基因组复制和染色体断裂和融合。在群体中,变异可能经常被发现或可能以低频率存在,前者在普通植物育种中具有更大的应用,而后者则可能与罕见但重要的表型变异有关。有用的多态性可包括单核苷酸多态性(snp)、dna序列中的插入或缺失(indel)、dna序列的简单序列重复(ssr)、限制性片段长度多态性以及标签snp。遗传标记、基因、dna衍生序列、单元型、rna衍生序列、启动子、基因的5'非翻译区、基因的3'非翻译区、microrna、sirna、qtl、卫星标记、转基因、mrna、dsmrna、转录概况以及甲基化模式可能包括多态性。另外,上述这些的存在、不存在或拷贝数的变化可能包括多态性。如本文所用,“标记”是指可用于区别生物体的可检测的特征。这样的特征的例子可包括遗传标记、蛋白质组成、蛋白质水平、油组成、油水平、碳水化合物组成、碳水化合物水平、脂肪酸组成、脂肪酸水平、氨基酸组成、氨基酸水平、生物聚合物、药品、淀粉组成、淀粉水平、可发酵的淀粉、发酵产量、发酵效率、能量产量、次生化合物、代谢物、形态特征和农艺学特征。如本文所用,“遗传标记”是指多态性核酸序列或核酸特征。遗传标记可由一个或多个特定的变异序列或共有序列代表。在另一种意义上,“遗传标记”是分离的变异体或这种序列的共有序列。如本文所用,“标记试验”指使用特定方法检测特定基因座处的多态性的方法,例如,至少一种表型的测定(如种子颜色、花的颜色或其它视觉可检测的性状)、限制性片段长度多态性(rflp)、单碱基延伸、电泳、序列比对、等位基因特异性寡核苷酸杂交(aso)、随机扩增的多态性dna(rapd)、基于微阵列的技术和核酸测序技术等。如本文所用,“分型”(“typing”)指确定给定的大豆基因组多态性的特定等位基因形式的任何方法。例如,通过确定存在哪种核苷酸(即,a、g、t或c),对单核苷酸多态性(snp)进行分型。通过确定是否存在indel来确定插入/缺失(indel)。通过包括但不仅限于标记分析的多种分析方法可以对indel进行分型。如本文所用,术语“邻近”,当用来描述与包含多态性的dna杂交的核酸分子时,指的是与直接紧靠该多态性核苷酸碱基位置的dna序列杂交的核酸。例如,可在单碱基延伸试验中使用的核酸分子“邻近”该多态性。如本文所用,“探询位置”是指固体载体上的物理位置,可以对其进行查询以获取一个或多个预定的基因组多态性的基因分型数据。如本文所用,“共有序列”是指构建的dna序列,其确定基因座处等位基因的snp和indel多态性。共有序列可以基于基因座处dna的任一链,并且表示该基因座中每种snp的任一个的核苷酸碱基及该基因座中的所有indel的核苷酸碱基。因此,虽然共有序列可能不是一个实际的dna序列的拷贝,但共有序列可用于精确设计用于基因座中的实际多态性的引物和探针。如本文所用,术语“单核苷酸多态性”,也缩写为“snp”,是指单个位点上的多态性,其中,所述多态性构成单碱基对改变、一个或多个碱基对的插入或一个或多个碱基对的缺失。如本文所用,术语“单元型”是指由至少一种多态性分子标记限定的单元型窗口内的染色体区域。每个单元型窗口中的独特的标记指纹组合限定了该窗口的各个单元型。此外,例如重组导致的单元型的变化可能导致单元型的修饰,使其只包含与性状可操作地连锁的初始(亲本)单元型的一部分,例如,通过与基因、qtl或转基因物理连锁。单元型中的任何这样的变化都包括在我们对于构成单元型的内容的定义中,只要该基因组区域的功能完整性不变或改善。如本文所用,术语“单元型窗口”是指通过本领域技术人员已知的统计分析建立的,且处于连锁不平衡的染色体区域。因此,将位于该区域内的一个或多个分子标记基因座处的两个近交个体(或两个配子)之间的状态同一性(identitybystate)作为整个区域的谱系同一性(identity-by-descent)的证据。每个单元型窗口包含至少一个多态性分子标记。单元型窗口可以沿基因组中的每个染色体作图。单元型窗口本身不是固定不变的,且考虑到逐渐增加的分子标记密度,本发明预计单元型窗口的数目和大小将会发展,窗口数目逐渐增加,其各自的大小逐渐减小,从而导致在根据标记基因座的状态同一性确定谱系同一性方面的置信度逐渐增加。如本文所用,“基因型”是指表型的遗传部分,且可以使用标记间接表征,或通过核酸测序直接表征。适当的标记包括表型特性、代谢谱、遗传标记或一些其它类型的标记。基因型可以构成至少一个遗传标记基因座的等位基因或至少一个单元型窗口的单元型。在某些实施方案中,基因型可以代表单个基因座,而在其它实施方案中,它可以代表整个基因组宽的一组基因座。在另一种实施方案中,基因型可以反映染色体的一部分、整个染色体、基因组的一部分和整个基因组的序列。如本文所用,“表型”是指可能受基因表达影响的细胞或生物体的可检测的特征。如本文所用,“连锁”是指杂交产生配子类型的相对频率。例如,如果基因座a具有基因“a”或“a”,基因座b具有基因“b”或“b”,具有aabb的亲本i与具有aabb的亲本b之间的杂交将产生4种可能的配子,其中基因分离为ab、ab、ab和ab。空预期为独立相等地分离成4个可能的基因型中的每一个,即,如果没有连锁,每个基因型将会有1/4的配子。配子分离成基因型不等于1/4是由于连锁。如本文所用,“连锁不平衡”,是针对一代的许多个体的群体中配子类型的相对频率而定义的。如果等位基因a的频率是p,a是p',b是q,b是q',那么基因型ab的预期频率(没有连锁不平衡)是pq,ab是pq’,ab是p’q,ab是p’q’。相对于预期频率的任何偏差称为连锁不平衡。当两个基因座处于连锁不平衡时,它们被称为是“遗传连锁的”。如本文所用,“数量性状基因座(qtl)”指在一定程度上控制通常连续分布的、可用数字表示的性状的基因座。如本文所用,“抗性等位基因”是指包括与srkn抗性相关的多态性等位基因的核酸序列。如本文所用,术语“大豆”是指大豆(glycinemax),且包括可用包括野生大豆种在内的大豆培育出的所有植物品种。如本文所用,术语“包括”指“包括但不限于”。如本文所用,术语“优良品系”指通过针对优异的农艺学表现进行育种和选择而产生的任何品系。农民或大豆育种者可以买到的优良大豆品种的非限制性的例子包括ag00802、a0868、ag0902、a1923、ag2403、a2824、a3704、a4324、a5404、ag5903和ag6202(asgrowseeds,desmoines,iowa,usa);bpr0144rr、bpr4077nrr和bpr4390nrr(bioplantresearch,camppoint,illinois,usa);dkb17-51和dkb37-51(dekalbgenetics,dekalb,illinois,usa);及dp4546rr和dp7870rr(delta&pinelandcompany,lubbock,texas,usa);jg03r501、jg32r606cadd和jg55r503c(jglinc.,greencastle,indiana,usa);nks13-k2(nkdivisionofsyngentaseeds,goldenvalley,minnesota,usa);90m01、91m30、92m33、93m11、94m30、95m30和97b52(pioneerhi-bredinternational,johnston,iowa,usa);sg4771nrr和sg5161nrr/sts(soygenetics,llc,lafayette,indiana,usa);s00-k5、s11-l2、s28-y2、s43-b1、s53-a1、s76-l9和s78-g6(syngentaseeds,henderson,kentucky,usa)。优良植物是来自优良品种的代表性的植物。本发明提供了可用于筛选位于连锁群o和连锁群g上的srkn抗性qtl的snpdna标记(cregan,等人cropsci.39:1464-1490(1999))。用来监测连锁群o上的srkn抗性qtlrmi的渗入的snp标记包括选自ns0094902、ns0097935、ns0135583、ns0102683和snp358的那些。snp标记snp199用来监测连锁群g上的srkn抗性qtl的渗入。在本发明中,说明性srkn抗性基因座rmisnp标记序列(seqidno:1)可以使用如seqidno:5和6所示的引物扩增,并且利用如seqidno:13或14所示的探针检测。说明性srkn抗性基因座rmisnp标记序列(seqidno:2)可以使用如seqidno:7和8所示的引物扩增,并且利用如seqidno:15、16、38、39、42或43所示的探针检测。说明性srkn抗性基因座rmisnp标记序列(seqidno:3)可以使用如seqidno:9和10所示的引物扩增,并且利用如seqidno:17、18、40、41、44或45所示的探针检测。说明性srkn抗性基因座rmisnp标记序列(seqidno:4)可以使用如seqidno:11和12所示的引物扩增,并且利用如seqidno:19或20所示的探针检测。说明性srkn抗性基因座rmisnp标记序列(seqidno:26)可以使用如seqidno:28和29所示的引物扩增,并且利用如seqidno:32或33所示的探针检测。说明性srkn抗性基因座rmisnp标记序列(seqidno:27)可以使用如seqidno:30和31所示的引物扩增,并且利用如seqidno:34或35所示的探针检测。本发明也提供了包含选自seqidno:21-25、36-37及其互补序列的核酸序列的大豆植物。本发明还提供了包含选自seqidno:1-4、25和26、其片段和它们的互补序列的核酸序列的大豆植物。本发明还提供了包含选自seqidno:5-20和27-34、它们的片段和它们的互补序列的核酸序列的大豆植物。在一个方案中,大豆植物包含选自seqidno:21-25、36-37及其互补序列的2、3或4个核酸序列。在另一方案中,大豆植物包含选自seqidno:1-4、26和27、它们的片段和它们的互补序列的2、3或4个核酸序列。在进一步的方案中,大豆植物包含选自seqidno:5-20和27-34、它们的片段和它们的互补序列的2、3或4个核酸序列。如本文使用的srkn是指任何srkn变种或分离种。本发明的大豆植物可以对一种或多种能够引起或诱导srkn的线虫具有抗性。一方面,本发明提供了srkn抗性植物以及用于根据对根结线虫属引起的srkn的抗性或敏感性筛选大豆植物的方法和组合物。在一个优选方面,本发明提供了用于根据对南方根结线虫的抗性或敏感性筛选大豆植物的方法和组合物。本发明进一步提供,选择的植物来自大豆属(glycine)的成员,更特别地来自glycinearenaria、glycineargyrea、glycinecanescens、glycineclandestine、glycinecurvata、glycinecyrtoloba、glycinefalcate、glycinelatifolia、glycinelatrobeana、glycinemax、glycinemicrophylla、glycinepescadrensis、glycinepindanica、glycinerubiginosa、野大豆(glycinesoja)、大豆属的种(glycinesp.)、glycinestenophita、glycinetabacina和短绒野大豆(glycinetomentella)。本发明的植物可以是具有非常高的抗性、抗性、基本抗性、中度抗性、相当的抗性、部分抗性、中度敏感性或敏感性的大豆植物。在一个优选方面,本发明提供了将要通过任何方法测定其对srkn的抗性或敏感性的大豆植物,以确定大豆植物是否具有非常高的抗性、抗性、基本抗性、中度抗性、相当的抗性、部分抗性、中度敏感性或敏感性。一方面,本发明提供了用于根据对南方根结线虫种引起的srkn的抗性、免疫性或敏感性筛选大豆植物的方法和组合物。在一个优选方面,本发明提供了用于根据对南方根结线虫的抗性、免疫性或敏感性筛选大豆植物的方法和组合物。针对对srkn的抗性、免疫性或敏感性,基于虫瘿(gall)或卵群(eggmass)的数目,对大豆植物进行表型分型和打分(taylor和sasser,biology,identificationandcontrolofroot-knotnematodes(meloidogynespecies):acooperativepublicationofthedepartmentofplantpathology.p.111(1978))。本发明的srkn抗性等位基因可以被引入优良大豆品系中。“优良品系”是通过针对优异的农艺学表现进行育种和选择而产生的任何品系。本发明的srkn抗性等位基因也可以被引入包含一种或多种以下基因的优良大豆转基因植物中:除草剂耐受性、产量增加、昆虫控制、真菌病抗性、病毒抗性、线虫抗性、细菌病抗性、支原体病抗性、油产生改变、高产油量、高蛋白质产量、发芽和幼苗生长的控制、动物和人类营养增强、低棉子糖、环境应激抗性、可消化性提高、工业酶、药用蛋白质、肽和小分子、加工性状改善、风味改善、固氮、杂种种子的产生、变应原性降低、生物聚合物和生物燃料等。这些农艺学性状可通过植物生物技术方法在大豆中作为转基因提供。一种或多种抗病性qtl等位基因可以从任何包含该等位基因的植物(供体)引入到任何接受体大豆植物中。一方面,接受体大豆植物可以包含另外的srkn抗性基因座。另一方面,接受体大豆植物可以包含转基因。另一方面,在保持引入的qtl的同时,可以通过回交或其它合适的方法来减少提供抗病性qtl的植物的遗传贡献。一方面,来自大豆植物中的供体材料的核遗传物质可能少于或是大约50%、少于或是大约25%、少于或是大约13%、少于或是大约5%、3%、2%或1%,但该遗传物质包含一种或多种感兴趣的srkn抗性基因座。进一步可以理解,本发明的大豆植物可能显示任何相对成熟组的特征。一方面,成熟组选自组000、00、0、1、2、3、4、5、6、7、8、9和10。qtl的等位基因可以包含多个基因或其它遗传因素,甚至在连续基因组区域或连锁群如单元型内。如本文所用的,抗性基因座的等位基因可以包含一个以上的基因或其它遗传因素,其中,每个单独的基因或遗传组分也能够显示等位基因变异,并且其中每个基因或遗传因素也能够引发对所述数量性状的表型效应。在本发明的一方面,qtl的等位基因包含也能够显示等位基因变异的一个或多个基因或其它遗传因素。术语“qtl的等位基因”的使用并不排除包含一个以上的基因或其它遗传因素的qtl。特别是,本发明中的“qtl的等位基因”可以表示单元型窗口中的单元型,其中表型可以是害虫抗性。单元型窗口是可以用一组一个或多个多态性标记定义和示踪的连续的基因组区域,其中,多态性指示谱系同一性。该窗口中的单元型可以通过每个标记处的等位基因的独特指纹确定。如本文所用的,等位基因是占据染色体上给定基因座的基因的几种替代形式之一。当染色体上的给定基因座处存在的所有等位基因相同时,该植物在该基因座处是纯合的。如果染色体上的给定基因座处存在的等位基因不同,该植物在该基因座处是杂合的。本发明的植物在任何特定的srkn抗性基因座处或对于特定的多态性标记,可能是纯合或杂合的。本发明也提供了本发明的植物的部分。植物部分包括但不限于,种子、胚乳、胚珠和花粉。在本发明的特别优选的方面,植物部分是种子。本发明也提供了大豆的容器,该容器中超过50%、60%、70%、80%、90%、95%或99%的种子包含srkn抗性基因座,其中该基因座处的一个或多个等位基因选自srkn抗性等位基因1、srkn抗性等位基因2、srkn抗性等位基因3或srkn抗性等位基因4。大豆种子的容器可以含有任何数量、重量或体积的种子。例如,容器可以含有至少或超过大约10、25、50、100、200、300、400、500、600、700、80、90、1000、1500、2000、2500、3000、3500、4000个或更多的种子。另一方面,容器可以含有大约或超过大约1克、5克、10克、15克、20克、25克、50克、100克、250克、500克或1000克种子。此外,容器可以含有至少或超过大约0盎司、1盎司、5盎司、10盎司、1磅、2磅、3磅、4磅、5磅、10磅、15磅、20磅、25磅或50磅或更多的种子。大豆种子的容器可以是本领域中可以获得的任何容器。例如,容器可以是盒子、袋子、罐、包、囊、卷带、桶或管。另一方面,大豆种子的容器中含有的种子可以是处理过的或未处理的大豆种子。一方面,种子可以经过处理以改善发芽,例如通过引发种子或者通过消毒以防御种子携带的病原体。另一方面,种子可以用任何可以使用的涂层涂覆,以改善例如可种植性、种子发芽和防御种子携带的病原体。种子涂层可以是任何形式的种子涂层,包括但不限于粒化、薄膜涂层和硬外层。本发明的植物或其部分可以在培养基中培养和再生。从各种组织类型再生大豆植物的方法和大豆的组织培养方法是本领域已知的(参见,例如,widholm等人,invitroselectionandculture-inducedvariationinsoybean,insoybean:genetics,molecularbiologyandbiotechnology,verma和shoemaker编,cabinternational,wallingford,oxon,england(1996))。如大豆的植物的再生技术可以使用多种组织或细胞类型作为起始原料。尤其是对于大豆,已经开发了再生方法,其开始于某些分化的组织类型,如,分生组织(cartha等人,can.j.bot.59:1671-1679(1981))、下胚轴节(cameya等人,plantscienceletters21:289-294(1981))和茎节点段(saka等人,plantscienceletters,19:193-201(1980);cheng等人,plantscienceletters,19:91-99(1980))。已报道了由未成熟大豆胚的外植体产生的体细胞胚再生完整的性成熟的大豆植物(ranch等人,invitrocellular&developmentalbiology21:653-658(1985))。也已报道了通过器官发生和胚发生从组织培养物再生成熟大豆植物(barwale等人,planta167:473-481(1986);wright等人,plantcellreports5:150-154(1986))。qtl的抗病性效果可能随各个基因型和测定抗病性效果的环境条件而变化。植物育种领域的技术人员不需要过多的实验就可以利用此处所述的方法从植物群体中或从亲本基因型集合中选择那些当含有病害基因座时导致相对于该基因型抗病性增强的植物。本发明包括一种将等位基因渗入大豆植物中的方法,包括:(a)使至少一个包含选自seqidno:21-25、36和37的核酸序列的第一大豆植物与至少一个第二大豆植物杂交,以形成群体,(b)用一种或多种核酸标记筛查该群体,以确定来自该群体的一个或多个大豆植物是否含有该核酸序列,和(c)从该群体中选择一个或多个包含选自seqidno:21-25、36和37的核酸序列的大豆植物。本发明包括一种将等位基因渗入大豆植物中的方法,包括:(a)使至少一个srkn抗性大豆植物与至少一个srkn敏感性植物杂交,以形成群体,(b)用一种或多种核酸标记筛查所述群体,以确定来自所述群体的一个或多个大豆植物是否在lg-o或lg-g上含有srkn抗性基因座。本发明包括分离的核酸分子。这些分子包括那些与srkn基因座遗传或物理连锁的能够检测多态性的核酸分子。这些分子可以被称为标记。可以通过现有技术获得与srkn抗性基因座连锁的另外的标记。一方面,核酸分子能够检测是否存在位于距离srkn抗性基因小于30、25、20、15、10、5、2或1厘摩(centimorgans)的标记。另一方面,使用qgeneversion2.23(1996)和默认参数进行srkn测定,标记显示2或更高、3或更高、4或更高的lod得分。另一方面,核酸分子能够检测选自srkn抗性基因座rmi的基因座中的标记。在另一方面,核酸分子能够检测srkn抗性qtl中的标记。在进一步的方面,核酸分子选自seqidno:1-4、26和27、其片段、其互补序列及能够与这些核酸分子中的一个或多个特异性杂交的核酸分子。在优选的一方面,本发明的核酸分子包括那些在中度严格条件(例如,大约2.0xssc和大约65℃)下与seqidno:1至seqidno:45所示的一个或多个核酸分子或其互补分子或其中任一个的片段特异性杂交的核酸分子。在特别优选的一方面,本发明的核酸在高严格条件下与seqidno:1至seqidno:45所示的一个或多个核酸分子或互补分子或其中任一个的片段特异性杂交。在本发明的一方面,本发明的优选的标记核酸分子具有seqidno:1至seqidno:45所示的核酸序列或其互补序列或其中任一个的片段。在本发明的另一方面,本发明的优选的标记核酸分子与seqidno:1至seqidno:45所示的核酸序列或其互补序列或其中任一个的片段共有80%-100%或90%-100%的序列同一性。在本发明的进一步的一方面,本发明的优选的标记核酸分子与seqidno:1至seqidno:45所示的核酸序列或其互补序列或其中任一个的片段共有95%-100%的序列同一性。在本发明的更优选的一方面,本发明的优选的标记核酸分子与seqidno:1至seqidno:45所示的核酸序列或其互补序列或其中任一个的片段共有98%-100%的序列同一性。核酸分子或其片段在某些情况下能够与其它核酸分子特异性杂交。如本文所用,如果两个核酸分子能够形成反平行双链核酸结构,那么这两个分子能够彼此特异性杂交。如果它们表现出完全的互补性,则一核酸分子与另一核酸分子“互补”。如本文所用,当一个分子的每个核苷酸都与另一分子的核苷酸互补时,这两个分子显示“完全互补”。如果两个分子在至少常规的“低严格”的条件下能互相杂交,具有足够的稳定性,从而允许它们保持彼此退火,那么这两个分子是“最低限度互补的”。类似地,如果两个分子在常规的“高严格”的条件下能互相杂交,具有足够的稳定性,从而允许它们保持彼此退火,那么这两个分子是“互补的”。sambrook等人,in:molecularcloning,alaboratorymanual,2ndedition,coldspringharborpress,coldspringharbor,newyork(1989)与haymes等人,in:nucleicacidhybridization,apracticalapproach,irlpress,washington,dc(1985)描述了常规的严格条件。因而偏离完全互补性是允许的,只要这种偏离没有完全消除该分子形成双链结构的能力。为了使核酸分子作为引物或探针,只需要在序列上充分互补,以在所采用的特定的溶剂和盐浓度下能够形成稳定的双链结构。如本文所用,基本上同源的序列是在高严格条件下与所比较的核酸序列的互补序列特异性杂交的核酸序列。本发明的核酸探针和引物可以在严格条件下与靶dna序列杂交。术语“严格的杂交条件”是指在该条件下,探针或引物特异性地与靶序列杂交而不与非靶序列杂交,这可以根据经验确定。术语“严格的条件”是功能上的定义,涉及通过sambrook等人,1989,9.52-9.55所述的具体的杂交程序,核酸探针与靶核酸(即,与感兴趣的特定核酸序列)的杂交。也参见sambrook等人,19899.47-9.52,9.56-9.58;kanehisa1984nucl.acidsres.12:203-213;wetmur等人1968j.mol.biol.31:349-370。促进dna杂交的合适的严格条件是,例如,大约45℃、6.0x氯化钠/柠檬酸钠(ssc),接着50℃、2.0xssc洗涤,这是本领域的技术人员已知的,或可以在currentprotocolsinmolecularbiology,johnwiley&sons,n.y.,1989,6.3.1-6.3.6中找到。例如,洗涤步骤中的盐浓度可以从大约2.0xssc、50℃的低严格条件到大约0.2xssc、50℃的高严格条件中选择。另外,洗涤步骤中的温度可以从低严格条件的室温大约22℃增加到高严格条件的大约65℃。温度和盐都可以变化,或者,温度或盐浓度可以保持不变,而另一个变量发生变化。本发明考虑,如果保持探针或引物与靶序列结合的特异性,可以使用如较低的杂交和/或洗涤温度的较低严格杂交条件,来确认具有较低的序列相似性的相关的序列。因此,本发明的核苷酸序列可用于选择性地与dna、rna或cdna片段的互补延伸形成双链分子的能力。核酸分子片段可以是任何大小的片段,且示例性的片段包括seqidno:1至seqidno:45所示的核酸序列及其互补序列的片段。一方面,片段可以是15-25、15-30、15-40、15-50、15-100、20-25、20-30、20-40、20-50、20-100、25-30、25-40、25-50、25-100、30-40、30-50及30-100。另一方面,片段可以是大于10、15、20、25、30、35、40、50、100或250个核苷酸。本发明的遗传标记包括“显性”或“共显性”标记。“共显性标记”揭示两个或多个等位基因的存在(每个二倍体个体中有两个)。“显性标记”揭示只存在一个等位基因。显性标记表型(例如,dna带)的存在表明在纯合或杂合条件下存在一个等位基因。不存在显性标记表型(例如,不存在dna带)仅能证明存在“某些其它”不明确的等位基因。在群体中的个体主要是纯合的且基因座主要是二态性(dimorphic)的情况下,显性和共显性标记可能具有同样的价值。随着群体变得越来越杂合和多等位基因化(multiallelic),共显性标记往往比显性标记提供更多的基因型信息。可以使用与本发明的qtl的等位基因遗传连锁或相关的标记,如单序列重复标记(ssr)、aflp标记、rflp标记、rapd标记、表型标记、snp、同工酶标记、微阵列、转录概况(walton,1993;burow等人.1988)。鉴定这些标记的方法是本领域中已知的。通过使用核酸扩增方法可有利于dna、rna或cdna样品中的多态性位点的检测。这些方法特异性地增加了跨越多态性位点或包含该位点和位于其远端或近端的序列的多核苷酸的浓度。这些扩增的分子可以通过凝胶电泳、荧光检测方法或其它方法很容易地检测。实现这种扩增的一种方法采用聚合酶链反应(pcr)(mullis等人1986coldspringharborsymp.quant.biol.51:263-273;欧洲专利50,424;欧洲专利84,796;欧洲专利258,017;欧洲专利237,362;欧洲专利201,184;美国专利4,683,202;美国专利4,582,788;和美国专利4,683,194),其中使用能够与以双链形式限定多态性的近端序列杂交的引物对。在一种优选的检测多态性的方法中,可以通过美国专利5,210,015、5,876,930和6,030,787中公开的方法检测snp和indel,其中,寡核苷酸探针具有与探针的5'和3'端共价连接的5'荧光报告染料3'猝灭染料。当探针完整时,报告染料接近猝灭染料导致报告染料荧光被抑制,例如通过由福斯特型能量转移(forster-typeenergytransfer)。在pcr正向和反向引物与位于多态性两侧的靶dna的特定序列杂交过程中,杂交探针与位于扩增的pcr产物中的含有多态性的序列杂交。在随后的pcr循环中,具有5’→3’核酸外切酶活性的dna聚合酶切开探针,并分离报告染料与猝灭染料,导致报告染料的荧光增强。一种有用的试验是可从abbiosystems获得的试验,其在一个反应中采用4种合成寡核苷酸,其同时扩增大豆基因组dna,区别存在的等位基因,并直接提供用于区别和检测的信号。4种寡核苷酸中的2种作为pcr引物,并产生包含待检测的多态性的pcr产物。另外两个是等位基因特异性荧光共振能量转移(fret)探针。在这种试验中,使用两种携带不同的荧光报告染料的fret探针,其中,将独特的染料掺入可以与两个等位基因中的仅仅一个高特异性退火的寡核苷酸中。有用的报告染料包括但不限于6-羧基-4,7,2’,7’-四氯荧光素(tet)、2'-氯-7'-苯基-1,4-二氯-6-羧基荧光素(vic)和6-羧基荧光素亚磷酰胺(fam)。一种有用的猝灭剂是6-羧基-n,n,n’,n’-四甲基罗丹明(tamra)。此外,化学封闭每个fret探针的3’末端,使得它不能作为pcr引物发挥作用。也提供了用作被动参比的第三荧光团,例如罗丹明x(rox),以帮助相关荧光值在随后的标准化(校正反应装配中的空间误差)。在每一个pcr循环中,fret探针以等位基因特异性的方式与模板dna分子退火。当酶遇到退火探针的5'端时,退火的(不是非退火的)fret探针被taqdna聚合酶降解,从而从猝灭剂附近释放出荧光团,其中使用荧光光度法确定两种荧光团各自的荧光,以及被动参比的荧光。两种染料各自的荧光的标准化强度与样本中最初存在的每种等位基因的量成比例,因此,可以推断出样本的基因型。为了qtl作图,包括的标记应当是来源特征性的,以对随后的群体作出推断。snp标记对于作图是理想的,因为特定的snp等位基因源自特定物种的现存群体中的独立来源的可能性较低。因此,snp标记可用于示踪和辅助qtl的渗入,特别是在为作为单元遗传的紧密连锁的等位基因的单元型的情况下。可以通过基因作图模型建立另外的标记分子的遗传连锁,所述基因作图模型例如,但不限于,lander等人(lander等人,genetics,121:185-199(1989))报道的侧翼标记模型,和区间作图(intervalmapping),其基于其中所述的最大似然法,并用软件包mapmaker/qtl执行(lincoln和lander,mappinggenescontrollingquantitativetraitsusingmapmaker/qtl,whiteheadinstituteforbiomedicalresearch,massachusetts,(1990))。另外的软件包括qgene,version2.23(1996),departmentofplantbreedingandbiometry,266emersonhall,cornelluniversity,ithaca,ny。使用qgene软件是一种特别优选的方法。对于标记的存在,计算最大似然估计值(mle),以及假设没有qtl效应的mle,以避免假阳性。然后计算优势率的log10(lod):lod=log10(存在qtl的mle/假定没有连锁qtl的mle)。lod得分基本上指示,假定存在qtl,相对于不存在qtl,数据增大的可能性有多大。对于给定的置信度,比如95%,为了避免假阳性的lod阈值取决于标记的数量和基因组的长度。lander等人(1989)说明了显示lod阈值的曲线图,且arús和moreno-gonzález,plantbreeding,hayward,bosemark,romagosa(编)chapman&hall,london,第314-331页(1993)进一步对其描述。可以使用另外的模型。已经报导了对于区间作图的许多修改和可供选择的方法,包括使用非参数法(kruglyak等人,1995genetics,139:1421-1428)。也可以使用多元回归方法或模型,其中,在大量标记上对性状进行回归(jansen,biometricsinplantbreed,vanoijen,jansen(编)proceedingsoftheninthmeetingoftheeucarpiasectionbiometricsinplantbreeding,thenetherlands,第116-124页(1994);weber和wricke,advancesinplantbreeding,blackwell,berlin,16(1994))。jansen等人(jansen等人,genetics,136:1447-1455(1994))和zeng(zeng,genetics136:1457-1468(1994))报道了组合了区间作图与回归分析的程序,由此将表型回归到给定的标记区间的单个假定的qtl上,且同时回归到作为'辅助因素'的标记数量上。一般来说,辅助因素的使用减少了估计的qtl位置的偏差和抽样误差(utz和melchinger,biometricsinplantbreeding,vanoijen,jansen(编)proceedingsoftheninthmeetingoftheeucarpiasectionbiometricsinplantbreeding,thenetherlands,第195-204页(1994)),从而提高qtl作图的精度和效率(zeng1994)。这些模型可以扩展到多环境实验,以分析基因型-环境的相互作用(jansen等人,1995theor.appl.genet.91:33-3)。合适的作图群体的选择对于图谱的构建是重要的。合适的作图群体的选择取决于所用的标记系统的类型(tanksley等人,molecularmappinginplantchromosomes.chromosomestructureandfunction:impactofnewconceptsj.p.gustafson和r.appels(编).plenumpress,newyork,第157-173页(1988))。必须考虑在作图群体中所用的亲本的来源(适应的相对于外来的)。染色体配对和重组率在远缘杂交(适应的×外来的)中会受到严重干扰(抑制),且一般产生大大降低的连锁距离。与近缘杂交(适应的×适应的)的后代相比,远缘杂交通常会提供具有相对较大的多态性阵列的分离群体。f2群体是自交的第一代。通常一个f1植物自交,产生所有基因都以孟德尔(1:2:1)方式分离的群体。使用共显性标记系统从完全分类的f2群体中获得最大遗传信息(mather,measurementoflinkageinheredity:methuenandco.,(1938))。在显性标记的情况中,需要后代测试(例如f3,bcf2)来确定杂合子,从而使其等同于完全分类的f2群体。但是由于后代测试的成本和时间原因,这一程序通常是不可行的。f2个体的后代测试通常在其中表型不能一贯地反映基因型(例如抗病性)或性状表达受qtl控制的图谱构建中使用。来自后代测试群体(例如f3或bcf2)的分离数据可以在图谱构建中使用。然后可以基于标记-性状图谱关联(f2,f3),对杂交后代进行标记辅助的选择,其中连锁群没有被重组事件完全分离(即最大不平衡)。重组近交系(ril)(遗传相关的系,通常是>f5,从继续自交的f2系向纯合性发展)可以作为作图群体使用。通过使用ril可以将从显性标记获得的信息最大化,因为所有的基因座都是纯合的或接近纯合。在紧密连锁的条件下(即,大约<10%的重组),对于每个个体,在ril群体中评估的显性和共显性标记比在回交群体中的每个标记类型提供更多的信息(reiter等人,1992proc.natl.acad.sci.(usa)89:1477-1481)。然而,由于标记之间的距离增大(即,基因座变得更加独立),ril群体中的信息明显减少。回交群体(例如,通过成功的品种(轮回亲本)和携带前者不具有的性状的另一品种(供体亲本)之间杂交产生)可作为作图群体来使用。可以与轮回亲本进行一系列回交,以恢复大部分其需要的性状。因此,产生由几乎类似于轮回亲本的个体组成的群体,但每个个体携带不同量的或嵌合的来自供体亲本的基因组区域。如果轮回亲本中的所有基因座都是纯合的,且供体亲本和轮回亲本具有截然不同的多态性标记等位基因,回交群体可以用于对显性标记作图(reiter等人1992)。使用共显性或显性标记从回交群体获得的信息少于从f2群体获得的信息,因为从每株植物采集一个而不是两个重组配子。然而,与ril相比,回交群体提供更多的信息(在低标记饱和度时),因为ril群体中的连锁的基因座之间的距离增加(即,大约0.15%的重组)。增加的重组对于紧密连锁的分析是有益的,但在构建具有低标记饱和度的图谱时可能是不需要的。为了产生除了接受探询的性状或基因组区域之外在遗传组成上几乎相同的个体的阵列,通过多次回交产生的接近等基因的品系(nil)可用作作图群体。在用nil作图时,预计仅有一部分多态性基因座将定位于选定的区域。集群分离分析法(bsa)是为快速鉴定标记和感兴趣的性状之间的连锁而开发的方法(michelmore等人1991proc.natl.acad.sci.(u.s.a.)88:9828-9832)。在bsa中,从一次杂交产生的分离群体中抽取两个集群dna样品。这些集群包含特定性状(对特定病害具有抗性或敏感性)或基因组区域相同但在非连锁的区域是任意的(即,杂合的)个体。与靶区域不连锁的区域在bsa中的许多个体的集群样品之间没有差异。本发明的植物可以是育种计划的一部分或由育种计划产生。育种方法的选择取决于植物繁殖方式、待改善的性状的遗传性以及商业使用的栽培种的类型(例如,f1杂种栽培种、纯系栽培种等)。栽培种是已经产生或有意挑选并通过栽培维持的植物种的生理小种或品种。下文说明了用于培育本发明的植物的选择的、非限制性的方法。对任何杂交的后代使用标记辅助的选择(mas)可以提高育种计划。可以理解:本发明核酸标记可以在mas(育种)计划中使用。进一步可以理解:可以在育种计划中使用任何商业和非商业的栽培种。例如,如发芽活力、植物生长活力、应激抗性、抗病性、分枝、开花、结实、种子的大小、种子密度、直立性(standability)和脱粒性(threshability)等各种因素通常指导选择。对于高度遗传的性状,在单一位置评估优良的单株植物的选择是有效的,然而,对于具有低遗传性的性状,选择应该基于通过相关植物的科的反复评估获得的平均值。流行的选择方法通常包括系谱选择、修改的系谱选择、混合选择(massselection)和轮回选择。在优选的一方面,采取回交或轮回育种计划。育种系可以进行测试,且可以与代表商业靶区域的环境中合适的标准比较两代或更多代。最好的品系是新的商业栽培种的候选品系;那些仍然为性状缺陷的品系可用作亲本,以产生新的群体用于进一步选择。系谱育种和轮回选择育种方法可用于从育种群体中开发栽培种。育种计划将来自两个或多个栽培种或各种广泛的来源的理想性状组合为育种池,从育种池中通过自交和所需的表型的选择开发栽培种。可以评估新栽培种以确定哪些具有商业潜力。回交育种已被用来将简单遗传的、高度遗传的性状的基因转移到作为轮回亲本的需要的纯合栽培种或自交系中。待转移的性状的来源称为供体亲本。在初始杂交后,选择具有供体亲本的表型的个体,并与轮回亲本反复杂交(回交)。产生的植物预计具有轮回亲本(例如,栽培种)的大部分属性,此外,还有从供体亲本转移来的理想的性状。严格意义上说,单种子遗传程序是指种植分离群体,每株植物收获一个种子的样品,并使用这一个种子样品种植下一代。当群体已从f2提高到理想的近交水平时,作为该品系的来源的植物均回溯到不同的f2个体。由于一些种子不能发芽或一些植物不能产生至少一个种子,种群中的植物数量每一代都在下降。结果,当完成了世代进步时,并非全部原先在群体中取样的f2植物都有后代代表。对于通常用于不同性状和农作物的其它育种方法的描述可在以下几种参考书中找到(allard,principlesofplantbreeding,johnwiley&sons,ny,u.ofca,davis,ca,50-981960;simmonds,principlesofcropimprovement,longman,inc.,ny,369-3991979;sneep和hendriksen,plantbreedingperspectives,wageningen(编),centerforagriculturalpublishinganddocumentation1979;fehr,in:soybeans:improvement,productionanduses,2ndedition,manograph.,16:2491987;fehr,“principlesofvarietydevelopment,”theoryandtechnique,(vol.1)和cropspeciessoybean(vol.2),iowastateuniv.,macmillanpub.co.,ny,360-3761987)。如本文所用,“核酸分子”,无论是自然产生的分子还是在需要时以其它方式“基本上纯化的”分子,指的是从基本上全部其它通常与其天然状态相关的分子中分离的分子。更优选地,基本上纯化的分子是制剂中存在的主要的种类。基本上纯化的分子可以大于60%、优选75%、更优选90%且最优选95%地不含天然混合物中存在的其它分子(不包括溶剂)的分子。术语“基本上纯化的”并非意在包括其天然状态存在的分子。本发明的材料优选是“生物活性的”,无论是在结构属性方面,如核酸与另一种核酸分子杂交的能力,还是在蛋白质被抗体结合(或与另一种分子竞争这种结合)的能力方面。或者,这种属性可以是催化性的,因此涉及该材料介导化学反应或应答的能力。本发明的材料也可以是重组的。如本文所用,术语重组是指通过核酸分子的人工操作间接产生的任何材料(例如,dna、肽等)。本发明的材料可以用便于检测该材料的试剂标记,例如荧光标记(prober等人,science238:336-340(1987);albarella等人,欧洲专利144914)、化学标记(sheldon等人,美国专利4,582,789;albarella等人,美国专利4,563,417)、修饰的碱基(miyoshi等人,欧洲专利119448)。现已一般性地描述了本发明,通过参考以下的由举例的方式提供的,并且除非另外指定,不限制本发明的实施例,会更容易理解本发明。实施例实施例1:srkn抗性标记的发现通过筛查srkn抗性和敏感性大豆品系中的连锁群(lg)o中位于rmi基因座侧翼的核酸序列,确定了srkn抗性qtlrmi的snp标记。图1总结了标记、它们的染色体位置、抗性和敏感性等位基因和每个标记的snp的位置。图1也提供了用于检测snp的引物和探针。实施例2:srkn抗性snp标记在可公开获得的种质中的验证根据植物根上存在的虫瘿或卵群的数目对大豆植物进行基因型分型和打分(表1)。虫瘿或卵群的数目越大,植物越敏感。然后计算平均虫瘿指数,并给予植物抗性、中度抗性、中度敏感或敏感的srkn等级(表2)。为了检测本发明的srkn抗性标记的应用和可预测性,对16个可公开获得的具有报道的srkn抗性和敏感性反应的大豆品系进行基因型分型(表3)。使用snp标记ns0094902、ns0097935、ns0135583和ns0102683发现已知抗性和敏感性品种的单元型是一致的。发现这4种snp标记可以预测可公开获得的大豆种质中的srkn抗性。表4提供了这4种snp标记的进一步的验证。对14个具有报道的srkn抗性反应的公开项进行基因型分型。另外,使用palmetto作为srkn抗性对照,并且使用srkn敏感的品系作为阴性对照。通过使用这4种snp标记,发现12个品种的单元型与抗性对照一致。一个品种,即g93-9223,只在一个标记ns0102683处具有不同的等位基因状态。两个品种,即musen和ls97-1610,具有对应于敏感类型的基因型。srkn反应是基于报道的反应而不是该实施例中的基因型分型。但是,snp标记可预测大多数品系的srkn反应,并且可用于预测大豆srkn反应。以palmetto作为srkn抗性对照,以敏感的品系作为阴性对照,对5个具有报道的srkn敏感性的公开项进行基因型分型(表5)。发现这5个公开项的单元型与敏感性对照一致。发现4种snp标记,即ns0094902、ns0097935、ns0135583和ns0102683,可预测公开大豆品种中的srknqtl(rmi)。表1.在srkn表型分析中使用的虫瘿指数(taylor和sasser,1978).表2.srkn病等级量表。等级是抗性(r)、中度抗性(mr)、中度敏感(ms)和敏感(s)。srkn等级虫瘿指数r平均虫瘿指数<3.2mr平均虫瘿指数3.2至<3.5ms平均虫瘿指数3.5至<4.0s平均虫瘿指数4.0+表3.用于检测lg-o上rmiqtl的存在或不存在和相应的srkn病表型应答的srkn抗性snp标记的验证。品种cns、s-100、palmetto和bragg用作抗性(r)和敏感(s)组,用来评价snp标记在区别历史r和s大豆品系中的抗性与敏感性中的效用。项srknns0094902ns0097935ns0135583ns0102683cnssttatcaattatccttaas-100sttatcaattatccttaapalmettor***********ggccaabraggr***********ggccaaleesttatcaattatccttaadyersttatcaattatccttaapickett71sttatcaattatccttaahutchesonsttatcaattatccttaaforrestr***********ggccaabraxtonr***********ggccaahartwigr***********ggccaamanokinr***********ggccaadillonr***********ggccaac6738r***********ggccaacookr***********ggccaabenningr***********ggccaa表4.用于检测lg-o上rmiqtl的存在或不存在和相应的srkn病表型应答的srkn抗性snp标记的验证,palmetto作为srkn抗性的对照,srkn敏感的品系作为阴性对照。项srknns0094902ns0097935ns0135583ns0102683palmettor***********ggccaa敏感型sttatcaattatccttaaaccomacr***********ggccaaboggsr***********ggccaabryanr***********ggccaadelsoy5710r***********ggccaag93-9106r***********ggccaag93-9223r***********ggccagls94-3207r***********ggccaals97-1610rttatcaattatccttaals97-3004r***********ggccaamusenrttatcaattatccttaas96-2692r***********ggccaasanteer***********ggccaastonewallr***********ggccaanks75-55r***********ggccaa表5.用于检测lg-o上rmiqtl的存在或不存在和相应的srkn病表型应答的srkn抗性snp标记的验证,palmetto作为srkn抗性的对照,srkn敏感的品系作为阴性对照。实施例3:srkn抗性snp标记在monsanto专利品系中的验证使用对srkn病具有已知的抗性和敏感性反应的monsanto专利品系验证了srkn抗性snp标记。使用了8个mr/r品种(表6)和17个敏感品种(表7)。病害等级基于如前面表2所述的平均虫瘿指数得分。在该实施例中,进行表型分型,并且srkn分类基于温室测试或温室测试与田间测试的结合。对8个monsanto专利品种进行了表型分型。基于虫瘿指数等级,将这些品系分类为抗性/中度抗性。对叶组织进行基因型分型。发现这8个品种的单元型与抗性对照palmetto一致(表6)。对17个monsanto专利品种进行了表型分型,并且基于虫瘿指数等级将其评定为敏感的。对叶组织进行基因型分型。发现这17个品种的单元型与敏感型对照一致(表7)。发现4种snp标记可用于检测monsanto专利品系中的srkn抗性qtl(rmi)。表6.用于检测lg-o上rmiqtl的存在或不存在和相应的srkn病表型应答的srkn抗性snp标记的验证,palmetto作为srkn抗性对照,srkn敏感品系作为阴性对照。项srknns0094902ns0097935ns0135583ns0102683palmettor***********ggccaa敏感型sttatcaattatccttaaag5402r/mr***********ggccaadkb64-51r/mr***********ggccaah5218r/mr***********ggccaah6372r/mr***********ggccagh7440r/mr***********ggccaah5050rrr/mr***********ggccaah6104rrr/mr***********ggccaah7242rrr/mr***********ggccaa表7.用于检测monsanto项的lg-o上rmiqtl的存在或不存在和相应的srkn表型应答的srkn抗性snp标记的验证,palmetto作为srkn抗性对照,srkn敏感的品系作为阴性对照。项srknns0094902ns0097935ns0135583ns0102683palmettor***********ggccaa敏感型sttatcaattatccttaamv0009sttatcaattatccttaamv0010sttatcaattatccttaamv0011sttatcaattatccttaamv0012sttatcaattatccttaamv0013sttatcaattatccttaamv0014sttatcaattatccttaamv0015sttatcaattatccttaamv0016sttatcaattatccttaamv0017sttatcaattatccttaamv0018sttatcaattatccttaamv0019sttatcaattatccttaamv0020sttatcaattatccttaamv0021sttatcaattatccttaamv0022sttatcaattatccttaamv0023sttatcaattatccttaamv0024sttatcaattatccttaamv0025sttatcaattatccttaa实施例4:区别srkn抗性单元型与不同抗性来源的能力srkn抗性的两个来源是palmetto和pi96354。本发明的snp标记可以用来区别作为srkn抗性qtl(rmi)的来源的palmetto和pi96354(表8)。该标记提供的信息可用于谱系分析。表8.用于区别srkn抗性的两个来源——palmetto和pi96354的srkn抗性snp标记的验证。项srknns0094902ns0097935ns0135583ns0102683敏感型sttatcaattatccttaapalmettor***********ggccaapi96354vrttatcaattatccttgg实施例5:用于srkn抗性的另外的snp标记通过使用细菌人工染色体(bac)末端和含ssr的基因组dna克隆开发了与lg-o和lg-g上的srkn抗性qtl连锁的snp标记。在位于lg-o上的主要srkn抗性qtl附近的satt358来源-序列中确定了三种snp。在位于lg-g上的次要srkn抗性qtl附近的satt199来源-序列中确定了三种snp。使用pi96354xbossier杂交产生的94株f2植物进行snp基因型分析。从各个亲代基因型和各个在田间生长的f2植物中提取dna。在温室中评价f2:3系的srkn虫瘿形成(galling)。使用来自lg-o上的satt358来源序列的snp358开发和优化了直接杂交(dh)试验。设计了对应于pi96354和bossier序列变体中每一个的等位基因特异性寡核苷酸探针,使得该探针在83和91碱基位点处包含两个多态性碱基。aso358-ta探针是对pi96354单元型特异性的,aso358-ag探针是对bossier单元型特异性的。对于lg-g上的次要srkn抗性qtl,使用来自lg-g的satt199来源序列的snp199开发了dh试验。aso199-aa探针是pi96354单元型特异性的,aso199-tg探针是对bossier单元型特异性的。通过测定12个srkn抗性和12个srkn敏感性大豆品系,将snp358和snp199的效果与各自的ssr标记satt358和satt199进行比较。通过多路测定进行基因型分型,其中反应特异性微球体以不同频率发荧光。表9提供了该试验的结果。对于pi96354等位基因(抗性)特异性aso358-ta探针,与bossier等位基因(敏感的)特异性aso358-ag探针相比,12个在satt358处携带200-bp等位基因的srkn抗性品系中的11个具有大约高10x的阳性信号。对于bossier-特异性aso358-ag探针,所有在satt358处携带192-bp等位基因的srkn敏感品系都具有阳性信号。敏感的大豆品系fc33243在snp358处具有独特的单元型(aa)。因此,snp358在24个大豆品系中具有三种snp单元型,即ta、ag和aa。图2提供了snp标记和抗性单元型。表9.在srkn抗性和敏感性大豆品系中连锁群-o(lg-o)和lg-g上的简单序列重复(ssr)标记与单核苷酸多态性(snp)标记之间的比较1mfi=平均荧光强度实施例6.用于检测具有srkn抗性基因座的大豆植物的寡核苷酸杂交探针通过基于杂交的snp检测方法,寡核苷酸也可用于检测与本文所公开的srkn抗性相关的多态性或对其进行分型。本发明提供了能够与包含多态性的分离的核酸序列杂交的寡核苷酸。本领域的技术人员能够设计具有经实验确定的严格性的试验,以区别本文提出的多态性的等位基因状态。示例性的试验包括southern印迹法、northern印迹法、微阵列、原位杂交和其它基于杂交的多态性检测方法。表10提供了示例性的用于基于杂交的snp检测的寡核苷酸。可以用放射性标记、荧光团或其它化学发光手段可检测地标记这些寡核苷酸,以有助于使用本领域已知的方法检测与源自一种或多种大豆植物的基因组或扩增核酸的样品的杂交。表10.寡核苷酸杂交探针实施例7.用于通过单碱基延伸方法检测具有srkn抗性基因座的大豆植物的寡核苷酸探针通过基于单碱基延伸(sbe)的snp检测方法,寡核苷酸也可用于检测与本文所公开的srkn抗性相关的多态性或对其进行分型。表11提供了示例性的用于基于sbe的snp检测的寡核苷酸。sbe方法基于与邻近多态性的序列杂交的核苷酸引物的延伸,以在引物延伸时掺入可检测的核苷酸残基。也预期sbe方法可以使用3种合成的寡核苷酸。其中两种寡核苷酸作为pcr引物发挥作用,并与位于包含待测多态性的区域两翼的基因座的序列互补。在图1的标注为“正向引物seqid”和“反向引物seqid”的栏中提供了可用于对本发明公开的多态性进行分型的示例性的pcr引物。在包含多态性的区域扩增之后,将pcr产物与延伸引物杂交,该延伸引物与邻近该多态性的扩增的dna退火。然后提供dna聚合酶和两种差别标记的双脱氧核苷三磷酸。如果模板上存在该多态性,可以在单碱基链延伸中将一个标记的双脱氧核苷三磷酸添加到引物中。然后通过确定两种差别标记中的哪一个添加到延伸引物中推断存在的等位基因。纯合的样品将导致两种标记的碱基中只有一种被掺入,因此两种标记中只有一种被检测到。杂合的样品存在两个等位基因,因此将直接掺入两种标记(进入延伸引物的不同的分子),因此这两种标记都被检测到。表11.用于单碱基延伸(sbe)分析的探针(延伸引物)标记标记seqidsnp位置探针(sbe)探针seqidns00979352346tgtcacaagagaaaagt42ns00979352346attacatacattaagat43ns01355833730cacattattagatgaac44ns01355833730gaatgtatgtacataca45已经说明和描述了本发明的原理,本领域的技术人员应该明白,在不背离这些原理的情况下,可以在排列和细节上修改本发明。我们要求保护在所附的权利要求书的精神和范围之内的所有修改。本说明书中提到的所有出版物和公布的专利文件都引入本文作为参考,如同每一单独出版物或专利申请具体且单独地引入作为参考。序列表<110>narvel,jamesconcibido,vergelcerny,liesatamulonis,johnphancock,floyddougherty,richardboerma,henryrogerha,bo-keun<120>用于选择南方根结线虫抗性大豆植物的方法和组合物<130>46-21(53775)<140><141><150>us61/055519<151>2008-05-23<150>us60/9638736<151>2007-08-07<160>45<210>1<211>532<212>dna<213>大豆<400>1gctcaatcaagtacacccatcatccttcttctcttcacgcctagcactcaaatttgtagc60gcatgctataactattaactaaccctctgtcccttcttgcaacaattgcaacctccttcc120ttccactcttttcccatcatcattccacacctacctttctctctcccccatgttttaact180ttcttcaacactcactgacttttccactatctcttccttcttccattcacatcattttct240cttccttcttcctccccctttttgttgttgttgttgttgtttatttatcaattatcacca300tgcccctcaaacaattcaaccaaagcttgcaactttgacaaccctttggcctcttcttct360tgttccccttaatgggtctctctccaattcccctcacaaagcctatctattctgatgtct420gaatttcactttggttttcatatgttcccaaaggctacacctttgcgggtgtagaagtgg480ataatttcagagtttttgggacaccccattttgaagaaagtgtgtgtgtgtg532<210>2<211>703<212>dna<213>大豆<220><221>不确定<222>(693)..(693)<223>n=a,t,c或g<220><221>不确定<222>(1)..(703)<223>所有n位置不确定<400>2attagatggtagttgaagcaatctgcctttgaactcttcttctggctcagcatgcatgct60tgtgttcatactattactatatccacctctctttatgtctcattctatttcgtaagtatt120tgatattgaattttggataatgattatttgctgcaatagccactttgagcttagggagaa180cttttctattctttgatggctgcaataactgcatatggcattttaatatagtttgataat240aatgtaagagatgggggacctggacggctggacccggtaatggcaaagacaatttctttt300agaaaaaattaattaggacatttcgttttgtcacaagagaaaagtgaatcttaatgtatg360taatagataatgccacaagaaaaaattaaaaaactagaaaaactaaggccttctatcggt420cagttgacctcaaaatacgtactcgagtactgtacattcggatgttggaggtataaatta480aaagtcatttttaatttatatttatcattagtgttgagataaaattgaattattcaactt540gagaaaatgattgataattcagttggattgcccctttgacttgtatgcttgcgtatattg600gtatgaaatcaaatatgccaaatattatcatcattgaaataaccttttgatattgatagg660gtacttaattatttcaagaattgttgtgtgctngttaatcctt703<210>3<211>792<212>dna<213>大豆<400>3tttccttgaaggtttacttcaacgtggtttggaaagatttgctacattcgccaaaagcta60cacctaaatttttattatgattgtcctgatttttatagcagggctacatttctaaaagtg120aacatacctgattattttcatggcgatctcattctgtctgaacattgattgaattactgt180tatcatgccaatttttggttgcatgaatggttatcccattttgaagtgttaattaattat240gtataactctgatagacggtcttaatgcaaaatgaacacatccgacataatgttaaaggt300ctgttcttaaatccgcaatcctaaatttgtaacgctgctttgagcacttcagaaatgatt360ggatttggatcctagcaagtaaaatttgccaaaccatctcgaaacttaagctccagagtc420ctatgcggagtatatagatacatgtcctaaaatgagtatatgtaattgctaacagataat480ggaatagaagctaagagattccatttattgacgcacaagaatctgacatgaatgtggcag540tcatagtaaaaattgactatagtacaaaataataatgtaacgacaacacatatttacacc600accatcctccaacccaggctacgtacatcaaccgaccacgtaaaaaaatgatggcagctt660tttccaatgtctaaactgaagcaaccgaaaacagtgtgtataatcccaacaacacattat720tagatgaaccgtgtatgtacatacattcagttgcttgcactgccttccaacaagctcctc780aagctacaaaga792<210>4<211>835<212>dna<213>大豆<220><221>不确定<222>(692)..(692)<223>n=a,t,c或g<220><221>不确定<222>(1)..(835)<223>所有n位置不确定<400>4gagaaactgccttagcaagaaatatggcacggagagcaaactgagctgtatgttgttggg60atggctccaattgccaatcatactctgtaactgaatatgtgaagagaacgaggttgaaaa120tatagaaaaccactttccacgtggcaaatgagcagaggtatagtattcgatcaagcacaa180tcaagaagaagatagcctgaacagaagaagtaactcttattaggagtgtatatgacatga240caactctagccacacttataaatgaaacatgcaataaatcattgcgtcactctaacatat300gtattcaacaaagaaacgtggataagaaatgtttcctaaacatgtcaatgatgaattaca360gtcattctctttcgaagtgctacaaattttaacctccaaattatgaaaaggaaaacaaaa420tcttaatgtttcattccatttggaaaacaaaatgtatcataaatgaaatcatttcaaagg480ttctggacaatttcttggcattataaacaacatgaattttatattagatctctaacatcc540cccctgtacaggccctttgggtttgaaaaagggaatatgctgacctaccatgtgctgaaa600ttcaacatttttttagaatgacagacaacaaggatcaaactctagactacatacttggtc660aaagaacctctaataccacatcaggaaccaancactccataagcttaagctgttagatga720agagatatgaatagttgtacactacgtctctaacaattggcacacttgacctgctttgtt780taccccataaaagtcaaatttgtctgaatgaattaaattctttttaagccctgtt835<210>5<211>21<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成引物<400>5tcctccccctttttgttgttg21<210>6<211>22<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成引物<400>6tgcaagctttggttgaattgtt22<210>7<211>28<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成引物<400>7aaattaattaggacatttcgttttgtca28<210>8<211>29<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成引物<400>8ctagttttttaattttttcttgtggcatt29<210>9<211>27<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成引物<400>9aaaacagtgtgtataatcccaacaaca27<210>10<211>20<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成引物<400>10ggaaggcagtgcaagcaact20<210>11<211>28<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成引物<400>11cattgcgtcactctaacatatgtattca28<210>12<211>28<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成引物<400>12tgtaattcatcattgacatgtttaggaa28<210>13<211>15<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>13tttatcaccatgccc15<210>14<211>19<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>14ttgttgttgtttatttatc19<210>15<211>18<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>15caagagaaaagtcaatct18<210>16<211>18<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>16caagagaaaagtgaatct18<210>17<211>15<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>17tacacggttcatcta15<210>18<211>19<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>18tgtacatacacagttcatc19<210>19<211>18<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>19caaagaaacgtagataag18<210>20<211>16<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>20aaacgtggataagaaa16<210>21<211>34<212>dna<213>人工序列<220><223>人工序列<400>21tgttgttgttgtttatcaccatgcccctcaaaca34<210>22<211>35<212>dna<213>人工序列<220><223>人工序列<400>22gtcacaagagaaaagtgaatcttaatgtatgtaat35<210>23<211>35<212>dna<213>人工序列<220><223>人工序列<400>23acattattagatgaaccgtgtatgtacatacattc35<210>24<211>35<212>dna<213>人工序列<220><223>人工序列<400>24ttcaacaaagaaacgtggataagaaatgtttccta35<210>25<211>35<212>dna<213>人工序列<220><223>人工序列<400>25ttcaacaaagaaacgtagataagaaatgtttccta35<210>26<211>439<212>dna<213>大豆<400>26gaattttattttgaacgcctttctgaagattcattaacttccattctggcaccagaaatc60attataaatgctatcctttaattcttagctatgcgctttatgtaacaatacgatttctat120tattattattattattattattattattattattattattattattattattattattat180tatattttttcctatttttggaaatatatttctattttcaaaataatatactttttattt240taattatatattttaagtttaattactccgcactctgcttttactaaaatttttaattag300ttctctaaatttttttaagtaagtttggattctagaactttacattaaattcatctaact360caattcacaagcatcctctgaatatatcattatttaaagtattcagtaaaaaaaacaagt420cttatttaaagtactgcca439<210>27<211>378<212>dna<213>大豆<400>27aaataggcaaaacaccgtatgaaaaataggttatcgttattcataatttacttcagacac60tttatatttctgttttctcttaagaaaaaaaaaactaaaacaaaatgtcgacagtagttg120tcaatacagtagcgtatgaataaaggttttctgttgaatataacattattattattatta180ttattattattattattattaatattagactgtaaacgttttaattactactactttttt240actttatgaattttgtgagtaagttgatgatggacccctacttcaatgagattttaacca300ttaagcccacgattgactaaaggaatttataggttcacacttgtgaatgatgtagtcctt360gcctgttaagttttgccc378<210>28<211>23<212>dna<213>人工序列<220><223>人工序列<400>28gaattttattttgaacgcctttc23<210>29<211>23<212>dna<213>人工序列<220><223>人工序列<400>29gcagagtgcggagtaattaaact23<210>30<211>24<212>dna<213>人工序列<220><223>人工序列<400>30aaataggcaaaacaccgtatgaaa24<210>31<211>21<212>dna<213>人工序列<220><223>人工序列<400>31gggcaaaacttaacaggcaag21<210>32<211>23<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>32agcgcatagctaagaattaaagg23<210>33<211>23<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>33agcgcacagctaagtattaaagg23<210>34<211>20<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>34taatattagactgtaaacgt20<210>35<211>19<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>35tattattaggctgtaaacg19<210>36<211>35<212>dna<213>人工序列<220><223>人工序列<400>36gctatcctttaattcttagctatgcgctttatgta35<210>37<211>33<212>dna<213>人工序列<220><223>人工序列<400>37ttattattattaatattagactgtaaacgtttt33<210>38<211>16<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>38aaaagtgaatcttaat16<210>39<211>16<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>39aaaagtcaatcttaat16<210>40<211>16<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>40atgaaccgtgtatgta16<210>41<211>16<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>41atgaactgtgtatgta16<210>42<211>17<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>42tgtcacaagagaaaagt17<210>43<211>17<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>43attacatacattaagat17<210>44<211>17<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>44cacattattagatgaac17<210>45<211>17<212>dna<213>人工序列<220><223>其它信息:人工序列的说明:合成探针<400>45gaatgtatgtacataca17当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1