具有扩大的光吸收能力和增加的光合活性的微生物的制作方法
本申请要求2015年7月10日提交的美国临时专利申请号62/191,171的优先权,所述专利申请如同在本文中完全阐述一样以引用方式整体并入本文。
公开领域
本公开提供具有扩大的光吸收能力和增加的光合活性的微生物。扩大的光吸收通过修饰所述微生物以利用非天然后胆色素来实现。增加的光合活性由扩大的光吸收引起,并且还可由自遮蔽的减少引起。所述微生物包括蓝细菌,包括遗传修饰的蓝细菌。
公开背景
光合作用是太阳能转化成化学键能的过程。光合作用过程最终导致生物质积累。生物质可用于生产能源、燃料、化学制品和食物。作为实例,生物乙醇可通过糖化碳水化合物的醇发酵来产生,并且生物柴油和生物喷气燃料可由中性脂质(诸如蜡酯和甘油三酯)来产生。此外,光合作用处理了环境二氧化碳。
光合作物诸如大豆、玉米和棕榈已被用作生产生物燃料和其他产品的原料。然而,将食用作物用于此类目的可能造成食物短缺。还已使用非食用作物诸如麻风树和亚麻荠,但这些作物的每单位面积产率低。
类似地可培养光合微生物以生产能源、燃料、化学制品和食物,以及处理环境二氧化碳。事实上,这些光合微生物中的许多均能够比植物产生更大量的油、脂肪和碳水化合物。
藻胆体是光合微生物用于捕获用于光合作用的光的蛋白质和后胆色素复合物。当每个光子均用于光化学时发生最佳量的光合作用。然而,天然光合微生物(诸如蓝细菌)利用有限类型的后胆色素,并且经常仅生产并利用一种类型的后胆色素。由于特定菌株所使用的这些有限的后胆色素类型,因此每种菌株均为光捕获专家;也就是说,它们仅优先使用可见光光谱的有限部分以进行光合作用。例如,细长聚球藻7942(Syn 7942)产生后胆色素藻蓝胆素(PCB),并且因此主要使用红/橙光以进行光合作用。
光合作用还可由自遮蔽限制。当培养物内的光合微生物对培养物内的其他光合微生物的光传递造成障碍时发生自遮蔽。当光照不再充分穿过培养物的厚度以便以最佳量达到培养物内的所有光合微生物时,培养物的生长是低效或受限制的。此类自遮蔽可限制生物质产率。
公开概述
当前公开提供具有扩大的光吸收能力和增加的光合活性的修饰的光合微生物。扩大的光吸收能力通过修饰光合微生物以利用另外的后胆色素,使得所述光合微生物可更有效地利用光谱的另外部分以进行光合作用来实现。增加的光合活性通过允许使用光谱的另外部分来实现。增加的光合活性还可由于天然后胆色素过表达所引起的自遮蔽减少而发生。光合活性的这些增加有可能增加总碳固定、含碳化合物的产生和生长(生物质积累)以及其他用途。当前公开另外提供了具有红色和紫色色素的后胆色素结合蛋白(即藻胆蛋白(phycobiloprotein))。
附图简述
图1提供用于藻蓝胆素(PCB)、藻红胆素(PEB)和藻尿胆素(PUB)生物合成的所选途径的示意图。
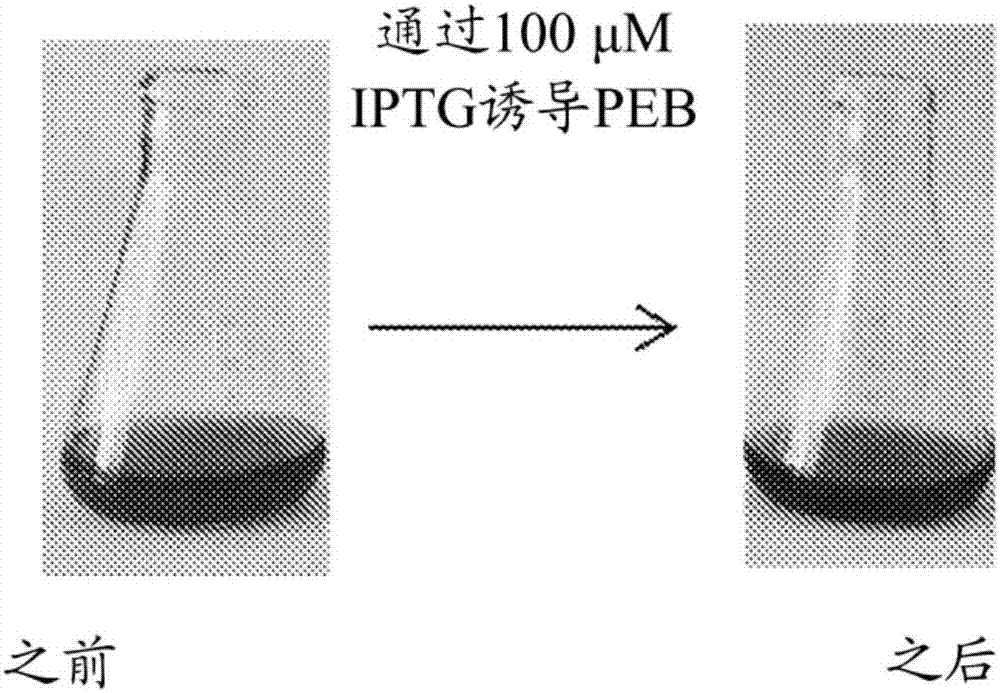
图2示出添加IPTG之前(左)和之后(右)的培养物Syn 7942菌株MX2037。如可在彩色复制物中看出,之前的培养物为浅翠绿色,而之后的培养物为深褐色/黑色。
图3示出由IPTG诱导后的修饰的Syn 7942菌株MX2037(短划线)和未诱导的MX2037培养物(实线)的吸收光谱。所述菌株利用PCB作为其天然后胆色素,并且经修饰以利用PEB作为非天然后胆色素。
图4示出藻胆体蛋白的SDS-PAGE分析。如可在彩色复制物中看出,PCB为蓝色色素,而PEB为粉红色色素。在诱导PEB后,α-亚基变成粉红色表明PCB已被PEB替换,并且β-亚基变成紫色表明它已结合PEB和PCB的混合物。α-亚基和β-亚基的迁移距离用箭头来标记。
图5A-图5G示出利用PEB和PUB的野生型和修饰的菌株中Syn7942的藻胆体组分的分析。(5A)野生型、(5B)PEB利用菌株(MX2037)和(5C)PUB利用菌株(2064)中的别藻蓝蛋白(短划线)和藻蓝蛋白(实线)的吸收光谱。照片示出不同菌株(5D)中别藻蓝蛋白和藻蓝蛋白的颜色。如可在数据的颜色复制物中看出,野生型色素为蓝色,PEB利用菌株中的藻蓝蛋白为紫色,PUB利用菌株中的藻蓝蛋白为深蓝色,而不依赖于PEB和PUB利用的所有菌株中的别藻蓝蛋白均保持蓝色。SDS-PAGE之后的蛋白质的目视(5E)、醋酸锌染色(5F)和帝王蓝(5G)分析。如可在彩色复制物中看出,醋酸锌导致PCB发出红色荧光,PEB发出黄色荧光,以及PUB发出绿色荧光。
图6A-图6E示出通过LC/MS分离藻蓝蛋白的α亚基和β亚基(6A)。野生型的吸收光谱显示,如由625nm附近的峰值吸光度指示,PCB结合至α亚基(6B)和β亚基(6C)。诱导的MX2037的吸收光谱显示,PEB结合至α亚基(6D),并且PCB和PEB两者均结合至β亚基(6E)。
图7A-图7D示出通过LC/MS鉴定PEB。PEB(7A)和PEB异构体的587分子离子的相对丰度由箭头指示。PEB的吸收光谱示出预期的626吸光度最大值(7B)。PEB异构体的色谱图(7C和7D)。
图8A和图8B示出未诱导(短划线)和诱导的(实线)PEB利用菌株中的绿光滴定(8A)以及计算为增加百分比的数据(8B)。
图9A和图9B示出经诱导(短划线)以利用PEB的培养物和未诱导的(实线)培养物的吸收光谱(9A)。实心箭头指示PEB的预期吸收最大值,并且轮廓箭头指示PCB的预期吸收值。响应于PEB利用菌株、野生型培养物和未诱导的培养物中的红光的氧气释放(9B)。响应于PEB利用菌株、野生型培养物和未诱导的培养物中的黄/琥珀光的氧气释放(9C)。
图10示出来自野生型(实线)、经诱导用于PEB利用(短划线)和诱导的PebAB+RpcG(虚线)培养物的全细胞裂解物的水溶性级分的吸收光谱。藻尿胆素的预期吸收最大值由箭头指示。
图11示出来自野生型(虚线)、PebAB利用菌株(实线)和ΔcpcE+pebAB+rpcG(短划线)的水溶性细胞裂解物的吸收光谱。箭头指示期望由PUB吸收的光的区域。
图12A-图12C示出使用可见光(12A)、醋酸锌染色(12B)和蛋白质染色(12C)的后胆色素的SDS-PAGE分析。如可在数据的目视复制物中看出,醋酸锌染色引起PCB的红色荧光,PEB的黄色荧光和PUB的绿色荧光。
图13A-图13C示出在MX2064突变体中通过LC/MS(13A)分离藻蓝蛋白的α亚基和β亚基(13A)。α亚基的吸收光谱(13B)示出两个吸收峰,493nm为PUB的预期吸光度,并且550为PEB的预期吸光度。β亚基(13C)在603nm处示出最大吸光度,所述最大吸光度不对应于PCB、PEB或PUB的已知吸光度光谱。
图14示出响应于蓝光的诱导和未诱导的PUB利用菌株(MX2064)中的氧气释放速率。所有数据点均为三次重复的平均值,并且误差棒表示一个标准偏差。
图15A和图15B示出野生型(15A)和PUB利用菌株(15B)的LC/MS分析。括号标记出现新峰的区域。
图16A和图16B示出其中由诱导型启动子(PEB菌株;16A;样品5和6;16B)表达pebA、pebB、rpcG和cpcAB的MX2479突变体的α-亚基的光谱。cpcA基因编码突变T130C。
图17A-图17C示出在使用77K荧光向PSII进行激子转移方面发挥作用的野生型(17A)、PEB(MX2037;17B)和PUB+PEB(MX2064;17C)。
图18A-图18C示出MX2479(Y130C)突变体的生长速率(18A;18B)和干重产率(18C)增加。
图19A和19B在示出白光滴定(19A)和100μE照射点(19B)下MX2479(Y130C)和MX2507(A26K)突变体的氧气释放增加。
图20A-图20C示出响应于不同波长的光的氧气释放的比较。与野生型(20A)相比,cpeS(MX2506;20B)和Y130C(MX2479;20C)突变体示出响应于520nm和505nm光的氧气释放增加。
图21示出结合至PCB(CpcA、CpcB、ApcA、ApcB);PEB(CpeA);和PUB(RpcA和MpeA)的后胆色素结合蛋白片段的氨基酸比对。在藻蓝蛋白和别藻蓝蛋白的β-亚基上方提供藻蓝蛋白(例如,CpcA(2种同系物);RpcA(2种同系物)等)和别藻蓝蛋白(ApcA(6种同系物)的的α-亚基。箭头指示结合后胆色素色素的普遍保守的半胱氨酸的位置(位置82/84)。
图22提供了支持本公开的示例性蛋白质和核苷酸序列。
详述
光合作用是太阳能转化成化学键能的过程。光合作用的总体反应为二氧化碳和水向葡萄糖和氧的光驱动转化:
6CO2+6H2O→C6H12O6+6O2
在植物以及细菌和蓝绿藻中观察到光合作用。
光合作用过程最终导致生物质积累。生物质可用于生产能源、燃料、化学制品和食物。作为实例,生物乙醇可通过糖化碳水化合物的醇发酵来产生,并且生物柴油和生物喷气燃料可由中性脂质(诸如蜡酯和甘油三酯)来产生。此外,光合作用处理了环境二氧化碳。
蓝细菌已开发了许多色素蛋白质以收集最佳用于光合作用的光能。大多数利用称为藻胆体的调谐天线,所述天线为由发色蛋白质和非发色蛋白质组成的超分子结构。发色组分,即藻胆蛋白,其携带负责这些蛋白质的光捕获性质的共价结合的线性四吡咯(后胆色素)。蓝细菌可利用至少四种不同类型的后胆色素:藻蓝胆素(PCB);藻红胆素(PEB);以及它们各自的Δ5至Δ2双键异构体、藻紫胆素(PVB)和藻尿胆素(PUB)。每种后胆色素能够吸收可见光谱的不同部分。例如,PCB吸收橙色/红色光谱(波长为550至650nm)的光;PEB吸收绿色光谱(波长为525至590nm)的光;PUB吸收蓝色光谱(波长为490至525nm)的光;以及PVB吸收黄光光谱(波长为550至590nm)的光。
当每个光子均用于光化学时发生最佳量的光合作用。然而,自然光合微生物诸如蓝细菌是光捕获专家,因为它们通常不利用每一种后胆色素类型。例如,蓝细菌菌株细长聚球藻7942(Syn 7942)仅天然利用PCB后胆色素,并且因此主要使用红/橙光以进行光合作用。相比之下,Syn 8102利用主要吸收绿光的PEB和主要吸收蓝光的PUB(Blot等人,(2009)J.Biol.Chem.,284(14):9290-8.doi:10.1074/jbc.M809784200)。
光合作用还可由自遮蔽限制。当培养物内的光合微生物对培养物内的其他光合微生物的光传递造成障碍时发生自遮蔽。当光照不再充分穿过培养物的厚度以便以最佳量达到培养物内的所有光合微生物时,培养物的生长是低效或受限制的。此类自遮蔽可限制生物质产率。
当前公开检查了光合微生物是否可经修饰以利用除了它们主要使用的那些后胆色素类型之外的后胆色素类型。为了评估这个问题,光合微生物经修饰以表达除了特定生物体的天然后胆色素类型之外的非天然后胆色素,并且评估另外的非天然后胆色素的表达是否将扩大用于光合作用的光的范围并增加光合活性(例如,扩展活性的光合光谱)。“非天然的”意指不是由未修饰的光合微生物天然产生的化合物(例如,后胆色素、蛋白质、核苷酸序列),或者如果产生,则与其引入的对应物(counterpart)相比以显著不同的水平或出于显著不同的目的产生。在详细描述方法和结果之前,提供关于后胆色素合成的背景信息。
所选后胆色素合成生产途径的示意图提供于图1中。细胞产生血红素,所述血红素经受血红素加氧酶(例如,HO1)以形成胆绿素。胆绿素进一步经受后胆色素还原酶(并且可能进一步经受细胞的另外的酶,诸如另外的还原酶)以形成所需的后胆色素。后胆色素然后通过裂解酶连接到所需的藻胆蛋白。然而,应指出,某些菌株利用可发挥多于一种功能的蛋白质。例如,RpcG(SEQ ID NO:1)为具有酶功能和裂解酶功能的融合蛋白,其可在利用PUB的菌株中发现。此外,某些菌株利用具有自我裂解酶活性的藻胆蛋白。也就是说,不需要不依赖于藻胆蛋白的裂解酶。因此,并且如本领域普通技术人员将理解,在实践当前公开的教义时,必须考虑所使用的菌株及其特定的后胆色素合成和利用方案。遵循当前公开的教义,可能需要一些实验以优化特定菌株中活性光合光谱的扩大。然而,当前的教义非常足以使普通技术人员成功地生产具有扩展的活性光合光谱的修饰的光合微生物。
示例性胆绿素还原酶包括将胆绿素转化为PCB的3Z-藻蓝胆素:铁氧还蛋白氧化还原酶(PcyA)。更具体地,PcyA执行两步反应:胆绿素IXα的乙烯基吡咯A环的还原和18-乙烯基的还原以产生PCB。胆绿素还原酶的另外实例包括3Z-藻红胆素:铁氧还蛋白氧化还原酶、PebA(SEQ ID NO:2)、PebB(SEQ ID NO:3)和PebS。PebA和PebB经常在相同的途径中发现。在许多蓝细菌中发现PebA-PebB途径,所述许多蓝细菌使用PebA以将胆绿素IXα还原成15,16二氢胆绿素(DHBV),然后使用PebB将15,16DHBV进一步还原成PEB。可替代地,来自肌尾病毒P-SSM4的PebS可以类似于通过PcyA进行的胆绿素至PCB的两步还原的方式执行两种反应。
通常,后胆色素通过裂解酶附接至藻胆蛋白。示例性裂解酶包括CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE和PecF。CpcE/CpcF是负责将PCB附接至CpcA的异二聚体。相关的裂解酶PecE/PecF催化由PCB形成PVB,并将此后胆色素附接至PecA。其他表征的裂解酶包括CpcS或CpcS/CpcU异二聚体裂解酶,这两者均可将PCB附接至CpcB、ApcA、ApcB、ApcD和ApcF的82/84位置处的保守Cys残基;以及CpcT裂解酶,其将PCB附接至CpcB的Cys153残基。似乎为PecE型结构域和PecF型结构域的融合体的RpcG据报道将PVB和PUB两者附接至RpcA(RpcG的同源的脱辅基蛋白)。与CpcS/CpcU/CpcT家族同源且可能负责附接藻红蛋白中的PEB的裂解酶已在许多物种的基因组中得以鉴定。这些酶大部分未经表征,但可能发挥类似的作用。藻红蛋白比藻蓝蛋白含有更多的后胆色素,并且因此它们可能需要另外的裂解酶以进行翻译后成熟。还应指出,PVB和PUB在蓝细菌细胞中不作为游离后胆色素出现;而是,这两种后胆色素通过异构化裂解酶来形成,所述异构化裂解酶将PCB和PEB分别转化为PVB和PUB并且将它们附接至适当藻胆蛋白的半胱氨酸。
如所指出,除了CpcE和CpcF之外的后胆色素裂解酶包括PecE和PecF,其催化将PCB添加至藻红蓝蛋白apo-α亚基并且将结合的后胆色素异构化为藻紫胆素(Jung,等人,.(1995)J.Biol.Chem.,270,12877-12884;Zhao,等人,(2000)FEBS Lett.,469,9-13)。CpeY加CpeZ据报道催化将藻红胆素添加至C-藻红蛋白的α亚基上的后胆色素附接位点中的一个(Kahn,等人,(1997)J.Bacteriol.,.179,998-1006)。裂解酶可提供任何所需的异构酶活性,或者这种活性可由独立的异构酶提供,所述异构酶可以是内源的或重组的。
因为大多数蓝细菌仅天然地利用一种类型的天然后胆色素和一种类型的天然后胆色素结合蛋白(藻胆蛋白),因此探索了蓝细菌可经修饰以利用另外的后胆色素类型的可能性。更具体地,探索了以下可能性:导致产生非天然后胆色素的非天然蛋白质的异源表达将导致与异源天然藻胆蛋白的结合以及活性光捕获以进行光合作用,从而扩展蓝细菌在较宽的光谱区域上吸收光以进行光合作用的能力。甚至更具体地,探索了两种非天然后胆色素增强Syn 7942中的光合作用的能力。
在Syn 7942中,仅产生主要吸收橙/红光的后胆色素PCB,并且因此主要在橙/红光中观察到最大光合速率。其他蓝细菌产生主要吸收绿色(PEB)和/或蓝色(PUB)光的后胆色素。例如,将编码用于PEB生物合成的途径的具有终止密码子的pebA和pebB基因(例如,分别为SEQ ID NO:4和SEQ ID NO:5)克隆到Syn 7942中,并且置于IPTG诱导型启动子的控制之下(对于整个插入序列,参见SEQ ID NO:6,其包含或编码lacI、pebA、pebB和链霉素抗性盒;SEQ ID NO:6被插入到中性位点1中;还参见SEQ ID NO:9)。当在IPTG存在下生长时,与未诱导的对照相比,培养物变暗(图2)。PEB的产生通过550nm处吸收峰的存在来确认(图3),并且PEB与藻胆体的蛋白质组分的结合通过SDS-PAGE分析来目视确认(图4)。因为PCB在可见光下为蓝色,并且PEB为粉红色,因此蛋白质凝胶的目视检查提供关于PEB定位至藻胆体的各种亚基的信息。在未诱导的对照中,由于结合的PCB,藻蓝蛋白的α-亚基和β-亚基均为蓝色。在诱导PEB后,α-亚基变成粉红色表明PCB已被PEB替换,并且β亚基变成紫色表明它已结合PEB和PCB的混合物。应指出,表达交替着色的藻蓝蛋白(红色和紫色)的能力是本公开的出乎意料的效用,因为藻蓝蛋白已经被高度评价为天然蓝色色素,并且红色和紫色版本具有类似值。关于PEB与藻胆体蛋白结合的进一步信息通过分离两种类型的后胆色素结合蛋白,别藻蓝蛋白和藻蓝蛋白来获得。别藻蓝蛋白在40%硫酸铵中沉淀,并且在650nm处具有吸收最大值。在PEB诱导后,别藻蓝蛋白在550处未显示吸收峰,表明其未结合PEB。相反,特别地在藻蓝蛋白级分中观察到PEB吸光度最大值(图5)。使用LC/MS分离藻蓝蛋白的α亚基和β亚基来探索后胆色素的结合模式(Kumar等人(2014)Ind J Plant Physiol.19,184-188)。发现PEB是α亚基上的唯一色素,并且β亚基含有大约相等丰度的PEB和PCB(图6A-图6E)。最后,为了确保观察到的后胆色素是PEB,使用LC/MS来确认其身份。可从藻蓝蛋白中提取作为2种异构体的PEB,所述2种异构体均具有587的母离子以及626nm处的吸收最大值(图7A-图7D,以及Fu,等人,(1979),J.Biochem.,.179:1-6)。
为了探索PEB表达对光合作用的影响,执行绿光滴定。当供应绿光以进行光合作用时,产生PEB的菌株总是比未诱导的对照产生更多的氧(图8A、8B)。表达PEB的菌株对低水平的绿光(100μE)非常敏感,并且显示优于未诱导的对照高达80%改善。
因为PEB表达对应于PCB的减少(图9A),并且PCB的过表达已被证明在红光下引起自遮蔽,所以探索了PEB表达将减少红光自遮蔽的可能性。当将细胞悬浮至OD750为2并且提供500μE的红光(630nm)时,与野生型或未诱导的对照相比,产生PEB的培养物显示氧气释放提高16%(图9B)。
作为对照,在PEB表达预期将不影响光吸收的光波长下重复实验。当提供590μE的黄光(590nm)时,PEB表达菌株显示与野生型和未诱导的对照相同的氧气释放速率(图9C)。
已报道了PUB在大肠杆菌中的异源表达(Alvey,等人,(2011a),Biochemistry,.50(22):4890-902.doi:10.1021/bi200307s)。如本文所公开,Syn 7942的菌株经修饰以表达用于PUB生物合成和附接至藻胆体的所有必需组分。SEQ ID NO:7表示编码具有终止密码子的RpcG的核苷酸序列。SEQ ID NO:8被插入到Syn 7942的中性位点4中,并且包含或编码lacI、rpcG和庆大霉素抗性盒。SEQ ID NO:9被插入到中性位点3中,并且包含或编码paraup1、pebA、pebB和潮霉素抗性盒。意外的是,在诱导表达后,在Syn 7942中未观察到PUB(图10)。
因为后胆色素通常通过裂解酶活性来附接至藻胆蛋白(例如藻蓝蛋白),并且PUB生物合成中的最后酶促步骤通过RpcG融合蛋白(具有裂解酶和生物合成两种作用)来进行,所以推断天然裂解酶CpcE与引入的裂解酶RpcG之间的竞争可能阻止PUB的表达。其中cpcE缺失(SEQ ID NO:10表示缺失的序列)并且被SEQ ID NO:11替换并且表达PUB生物合成途径(SEQ ID NO:8和9)的Syn 7942菌株的构建产生表达PUB的Syn 7942菌株(MX2064)(图11)。PUB与藻蓝蛋白α-亚基特异性的结合通过蛋白质的SDS-PAGE分析和醋酸锌菌株来确认,其中藻蓝蛋白在由PCB结合时发出红色荧光,在由PEB结合时发出黄色荧光,并且在由PUB结合时发出绿色荧光(图12)。LC/MS还允许分离藻蓝蛋白的α亚基和β亚基。发现藻蓝蛋白的α-亚基结合PEB和PUB的混合物,而β-亚基结合最大吸收为603nm的未知后胆色素(图13A-图13C)。诱导后,此突变体响应于蓝光显示出66%的光合作用增加(图14),并且响应于红光继续进行光合作用。
意想不到的结果是,产生PUB的菌株的LC/MS分析指示,产生了不仅仅是PCB、PEB和PUB的多种多样的后胆色素(图15)。不受理论的束缚,这些可能是来源于PCB、PEB和/或PUB的后胆色素异构体。就此而言,先前的研究已描述了apo-藻蓝蛋白α-亚基在不存在CpcE裂解酶时通过异构化反应将PCB转化成中胆绿素和鉴定的后胆色素的能力。通过类似的反应,PEB和PUB可异构化成新的未表征的后胆色素。因为apo-藻蓝蛋白是新后胆色素异构体的可能原因,所以使用定点诱变方法来限制此反应并且增强藻蓝蛋白α-亚基的PUB结合性质。将cpcA的A86K(MX2507;SEQ ID NO:61)和Y130C(MX2479;SEQ ID NO:62)变体形式表达为来自中性位点2中的IPTG诱导型启动子的cpcAB操纵子,如通过LC/MS所检测,当连同PebA、PebB和RpcG一起表达时,所述cpcAB操纵子在藻蓝蛋白的α-亚基上表达少量的PUB(图16A、16B;参见SEQ ID NO.57和58)。
还采取了第二种方法以解决后胆色素异构化问题。因为Syn 8102不形成中胆绿素,并且其基因组含有已知将后胆色素附接至藻蓝蛋白的β亚基的后胆色素裂解酶家族,所以探索了cpeS裂解酶的表达可在MX2064(ΔcpcE、RpcG、PebAB)背景中阻止中胆绿素形成的可能性。含有cpeS的菌株的诱导显示藻蓝蛋白的β亚基上的中胆绿素减少(参见,例如,SEQ ID NO.59、60、69和70)。数据与将PCB附接至藻蓝蛋白的β亚基并且防止中胆绿素形成的cpeS一致。
使用77K荧光建立PEB和PUB的功能性表达(图17A-图17C)。当将520nm的激发光施加至MX2037的诱导培养物时,观察到PSII荧光,这表明激子已从PEB转移至PSII。类似地,当将470nm的激发光施加至MX2064的诱导培养物时,再次观察到PSII荧光,这表明激子已经从PUB转移至PSII。
因为PEB和PUB在Syn 7942中功能性地表达,并且Syn 7942的突变菌株(A86K和Y130C)可表达PUB而不产生后胆色素异构体,所以探索PEB和PUB对生长的影响。如由干重所测量,在400μE白色LED灯和4%CO2下,Y130C菌株生长超过WT菌株16%(图18)。白光滴定指示,在100μE的低光照条件下,如通过氧气释放所测量,S7942的PUB表达菌株具有增加的光合作用(图19)。其中100μE光来自光的每个波长的作用光谱指示,表达PEB和PUB的菌株在520和505nm的光波长下具有增强的氧气释放(图20)。这些数据指示,PUB和PEB表达增强Syn 7942中的氧光合作用。
所述结果中的一些是出乎意料的。首先,预期PEB和PUB的功能性生产和利用将需要非天然藻胆体蛋白组分以及非天然后胆色素生物合成酶两者。相反,发现Syn 7942的天然藻蓝蛋白可与非天然后胆色素一起发挥作用。此外,Syn7942中发现的天然裂解酶活性可将非天然PEB附接至天然藻蓝蛋白。其次,先前从未证实非天然后胆色素的产生可响应于可见光的特定波长而增加光合作用(例如,扩展活性光合光谱)。第三,非天然PEB的产生减少了天然PCB产生,并且减轻红光的自遮蔽。虽然自遮蔽的减少先前已归因于PCB减少,但这是通过将后胆色素代谢汇集成替代性非天然后胆色素来实现PCB减少的新型方法。第四,先前的工作显示,大肠杆菌中PUB的异源生产通过表达生物合成酶(PebA、PebB、RpcG)、藻蓝蛋白蛋白(CpcA)和RpcG的裂解酶活性来达成(Blot,等人,(2009),J.Biol.Chem.,284(14):9290-8.doi:10.1074/jbc.M809784200)。所有这些基因在Syn 7942中的表达不足以达成PUB产生。不受理论的束缚,这可能是因为用于后胆色素生物合成和与藻蓝蛋白附接的天然途径与已在Syn 7942中表达的非天然途径(包括PebA、PebB和RpcG)竞争。通过下调将天然PCB附接至藻蓝蛋白的天然α-亚基的天然裂解酶CpcE,并且通过表达非天然RpcG补充天然CpcE的下调,这些途径之间的竞争减少,并且可产生PUB。第五,在产生PUB的菌株中出现了意想不到的并且可能先前未知的后胆色素异构体。PUB功能的优化可能需要对天然藻蓝蛋白的定点诱变,以便使藻蓝蛋白成为PUB的更好结合配偶体。可替代地,诱变可有利于产生一种优于其他后胆色素异构体的后胆色素异构体。
因为两种功能性非天然后胆色素是异源生产的,所以本文学习并且公开了可用于其他蓝细菌中非天然后胆色素的异源生产的多种方法和策略。本公开的教义对其他光合微生物和蓝细菌的应用由可在光合微生物(包括蓝细菌)中广泛变化的特定藻胆体蛋白和特定内源裂解酶基因来引导。藻蓝蛋白中PCB附接位点的共有氨基酸序列为(A/S)(K/A)C(I/L/A)RD(SEQ ID NO:12)。如果存在非天然PEB,则识别这类附接位点的裂解酶还将非天然PEB添加至藻蓝蛋白。表达含有此共有附接位点的藻蓝蛋白的蓝细菌的示例性物种包括Syn 7002和集胞藻属6803,并且是非天然PEB产生和/或并入藻胆体并因此扩展活性光合光谱的候选物。
PUB的功能性利用可能需要非天然裂解酶替换将天然PCB缀合到天然藻蓝蛋白上特异性位点的天然裂解酶中的一种;在本文所述的实例中,此为CpcE。这种方法可广泛适用于含有CpcE的同源物的其他蓝细菌物种,包括Syn 7002和集胞藻属6803;并且适用于表达控制PCB与藻蓝蛋白上特异性位点的附接的功能等效的裂解酶的其他蓝细菌物种。替代性方法是将藻蓝蛋白工程化以用共有PUB附接位点(A/S)(K/A)CSRD(SEQ ID NO:13)替换其共有PCB附接位点。这将增加通过非天然RpcG裂解酶进行的非天然PUB与现在非天然藻蓝蛋白的附接。这些结果和教义证实,光合微生物可经修饰以功能性地利用非天然后胆色素。
光合微生物。本公开的光合微生物可以是能够执行光合作用的任何类型的生物体,其中微生物已经修饰以利用非天然后胆色素来扩大其光吸收能力、扩展其活性光合光谱并且增加光合活性。“扩大其光吸收能力”意指微生物吸收其在未修饰状态下不吸收的可见光谱波长,或其显示出优于其未修饰状态的可见光谱波长的吸收的显著增加。光吸收能力的扩大可通过扩展微生物活性光合光谱来增加光合活性。也就是说,新吸收的光导致光合活性增加。
通常,利用天然PCB的天然存在的光合微生物吸收主要处于橙色/红色光谱中的光(峰吸收发生在620nm处);利用天然PEB的天然存在的光合微生物吸收主要处于绿色光谱中的光(峰吸收发生在550nm处);利用天然PUB的天然存在的光合微生物吸收主要处于蓝色光谱中的光(峰吸收发生在495nm处);并且利用PVB的转基因光合微生物吸收主要处于黄光光谱中的光(峰吸收发生在570nm处)。如先前所指出,本文公开的方法还可通过减少自遮蔽来增加光合活性。
天然光合或可经工程化为光合的示例性光合微生物包括细菌(例如,蓝细菌);真菌;古细菌;原生生物;真核生物,诸如绿藻;以及动物,诸如浮游生物、真涡虫和变形虫。天然存在的光合微生物的实例包括极大节旋藻(螺旋藻)(Arthrospira(Spirulina)maxima)、钝顶节旋藻(螺旋藻)(Arthrospira(Spirulina)platensis)、杜氏盐藻(Dunaliella salina)、布朗葡萄藻(Botrycoccus braunii)、普通小球藻(Chlorella vulgaris)、蛋白核小球藻(Chlorella pyrenoidosa)、羊角月牙藻(Serenastrum capricomutum)、四尾栅藻(Scenedesmus auadricauda)、紫球藻(Porphyridium cruentum)、Scenedesmus acutus、杜氏藻属(Dunaliella sp.)、斜生栅藻(Scenedesmus obliquus)、项圈藻属(Anabaenopsis)、管链藻属(Aulosira)、筒孢藻属(Cylindrospermum)、聚球藻属(Synechoccus sp.)、集胞藻属(Synechocystis sp.)、Cyanobacterium aponinum和单歧藻属(Tolypothrix sp.)。
蓝细菌(还称为蓝绿藻、蓝绿细菌或蓝藻门)是通过光合作用获得能量的细菌门。蓝细菌可从CO2、水、无机盐和光产生代谢物,诸如碳水化合物、蛋白质、脂质和核酸。可根据本公开使用任何蓝细菌。在特定实施方案中,蓝细菌必须是可遗传学操作的,例如,可允许引入并且表达外源(例如,非天然)遗传物质(例如,外源核苷酸序列)。
蓝细菌包括单细胞和菌落两种物种。菌落可形成丝状体、片状体或甚至空心球。一些丝状菌落显示出分化成以下各项的能力:若干不同细胞类型,诸如营养细胞,在有利的生长条件下形成的正常的光合细胞;厚壁孢子,可在环境条件变得恶劣时形成的气候抗性孢子;以及厚壁异形细胞,其含有对于固氮作用至关重要的酶固氮酶。
可根据本文所述的方法利用和/或修饰的蓝细菌的实例包括来自节旋藻属(Arthrospira)、隐球藻属(Aphanocapsa)、隐杆藻属(Aphanothece)、管孢藻属(Chamaesiphon)、色球藻属(Chroococcus)、Chroogloeocystis、腔球藻属(Coelosphaerium)、鳄球藻属(Crocosphaera)、蓝细菌属(Cyanobacterium)、蓝菌属(Cyanobium)、蓝网藻属(Cyanodictyon)、Cyanosarcina、蓝丝菌属(Cyanothece)、蓝纤维藻属(Dactylococcopsis)、粘球藻属(Gloecapsa)、粘杆藻属(Gloeothece)、碟状菌属(Merismopedia)、微囊藻属(Microcystis)、辐囊藻属(Radiocystis)、棒条藻属(Rhabdoderma)、Snowella、聚球藻属(Synychococcus)、集胞藻属(Synechocystis)、Thermosenechococcus和乌龙藻属(Woronichinia)的色球藻目(Chroococcales)蓝细菌;来自鱼腥藻属(Anabaena)、项圈藻属、束丝藻属(Aphanizomenon)、管链藻属、眉藻属(Calothrix)、Coleodesmium、蓝螺藻属(Cyanospira)、拟筒孢藻属(Cylindrospermosis)、筒孢藻属(Cylindrospermum)、夫列藻属(Fremyella)、胶刺藻属(Gleotrichia)、微毛藻属(Microchaete)、节球藻属(Nodularia)、念珠藻属(Nostoc)、Rexia、植生藻属(Richelia)、伪枝藻属(Scytonema)、Sprirestis和单歧藻属(Toypothrix)的念珠藻目(Nostacales)蓝细菌;来自节旋藻属、盖丝藻属(Geitlerinema)、Halomicronema、盐螺旋藻属(Halospirulina)、假膜藻属(Katagnymene)、瘦鞘丝藻属(Leptolyngbya)、湖丝藻属(Limnothrix)、鞘丝藻属(Lyngbya)、微鞘藻属(Microcoleus)、颤藻属(Oscillatoria)、席藻属(Phormidium)、拟浮丝藻属(Planktothricoides)、浮丝藻属(Planktothrix)、织线藻属(Plectonema)、假鱼腥藻属(Pseudoanabaena)/湖丝藻属、裂须藻属(Schizothrix)、束藻属(Symploca)、束毛藻属(Trichodesmium)和常丝藻属(Tychonema)的颤藻目(Oscillatoriales)蓝细菌;来自拟色球藻属(Chroococcidiopsis)、皮果藻属(Dermocarpa)、果菌属(Dermocarpella)、粘囊藻属(Myxosarcina)、宽球藻属(Pleurocapsa)、直毛藻属(Stanieria)和异球藻属(Xenococcus)的宽球藻目(Pleurocapsales)蓝细菌;来自原绿藻属(Prochloron)、原绿球藻属(Prochlorococcus)和原绿发藻属(Prochlorothrix)的原绿生物(Prochlorophytes)蓝细菌;以及来自蒴链藻属(Capsosira)、拟绿胶藻属(Chlorogeoepsis)、侧生藻属(Fischerella)、软管藻属(Hapalosiphon)、Mastigocladopsis、拟珠藻属(Nostochopsis)、真枝藻属(Stigonema)、聚线藻属(Symphyonema)、Symphonemopsis、Umezakia和拟惠氏蓝细菌属(Westiellopsis)的真枝藻目(Stigonematales)蓝细菌。在特定实施方案中,蓝细菌来自聚球藻(Synechococcus)属,包括聚蓝藻(Synechococcus bigranulatus)、细长聚球藻(Synechococcus elongatus)、聚球藻(Synechococcus leopoliensis)、铅色聚球藻(Synechococcus lividus)、Synechococcus nidulans和Synechococcus rubescens。还可使用蓝细菌嗜热聚球藻(Thermosynechococcus)和粘杆菌属(Gloeobacter)。
更特定实施方案包括或利用鱼腥藻属菌株PCC 7120、集胞藻属菌株PCC 6803、灰色念珠藻(Nostoc muscorum)、Nostoc ellipsosporum或念珠藻属菌株PCC 7120。在特定实施方案中,蓝细菌是细长聚球藻菌株PCC 7942。可利用的蓝细菌的另外实例包括聚球藻属菌株WH7803、WH8102、WH8103(通常通过接合来修饰)、形成Baeocyte的拟色球藻属物种(通常通过接合/电穿孔来修饰)、形成非异形胞的丝状菌株浮丝藻属、鲍氏织线藻M101(Plectonema boryanum M101)(通常通过电穿孔来修饰)、形成异形胞的鱼腥藻属ATCC 29413(通常通过接合来修饰)、单歧藻属菌株PCC 7601(通常通过接合/电穿孔来修饰)以及点状念珠藻菌株ATCC 29133(通常通过接合/电穿孔来修饰)。
在特定实施方案中,蓝细菌可以是例如海洋形式的蓝细菌或淡水形式的蓝细菌。海洋形式的蓝细菌的实例包括聚球藻属WH8102、聚球藻属RCC307、聚球藻属NKBG 15041c和束毛藻属。淡水形式的蓝细菌的实例包括细长聚球藻PCC 7942、集胞藻属PCC6803、鲍氏织线藻、Cyanobacterium aponinum和鱼腥藻属。
在其他实施方案中,修饰的蓝细菌可能能够在苦咸水或盐水中生长。当使用淡水形式的蓝细菌时,其使用的总体净成本将取决于使培养物生长所需的养分和淡水的价格两者。可预见,在未来淡水是一种有限的资源,并且在所述情况下,寻找淡水的替代物将更具成本效益。两种此类替代物包括:(1)来自处理厂的废水的使用;以及(2)盐水或苦咸水的使用。
海洋中咸水的盐度的范围可在3.1%与3.8%之间,平均为3.5%,并且这大部分但不是完全由氯化钠(NaCl)离子组成。另一方面,苦咸水与淡水相比具有较大的盐度,但没有海水那么大。苦咸水含有0.5%与3%之间的盐度,并且因此包括大范围的盐度体系(salinity regime),并且因此未进行精确定义。废水是已经历人影响的任何水。废水包括从家庭和商业物业、工业和/或农业释放的液体废物,并且可涵盖各种浓度的宽范围的可能的污染物。
蓝细菌在海洋中分布广泛,其中聚球藻属仅占一个小生境。具体而言,聚球藻属PCC 7002(以前称为阿格门氏藻(Agmenellum quadruplicatum)菌株PR-6)在苦咸水中生长,其是单细胞的并具有38℃的最佳生长温度。虽然这种菌株非常适合在高盐条件中生长,但其在淡水中将生长缓慢。在特定实施方案中,本公开包括使用以允许在废水或盐水/苦咸水中生长的方式改变的蓝细菌PCC 7942。已描述了抗氯化钠胁迫的细长聚球藻PCC 7942突变体(Bagchi,等人,(2007)Photosynth Res.,92:87-101),并且已描述了耐盐水生长的遗传修饰的细长聚球藻PCC 7942(Waditee,等人,(2002)PNAS,99:4109-4114)。耐盐水蓝细菌还可如美国专利号8,394,614的实施例中所述来制备。根据本公开,耐盐水菌株能够在盐度范围为0.5%至4.0%盐度的水或培养基中生长,但不一定能够在此范围所涵盖的所有盐度中生长。在特定实施方案中,耐盐菌株能够在盐度范围为1.0%至2.0%盐度的水或培养基中生长。在特定实施方案中,耐盐水菌株能够在盐度范围为2.0%至3.0%盐度的水或培养基中生长。
修饰生物体以利用非天然后胆色素的特定机制依赖于将外源核苷酸序列插入到所选择的光合微生物的基因组中。“外源的”是指其在野生型光合微生物的基因组所插入的特定位置中不天然存在的,而是通过分子生物学技术在所述特定位置处插入的核苷酸序列。外源核苷酸序列的实例包括载体、质粒和/或人造核酸构建体。
如本文所用,核苷酸序列可包括编码蛋白质(例如,PebA、PebB、PebS、PycA、RpcG、CpcA、CpcB、CpeS等)的基因。关于基因,此术语包括各种序列多态性、突变和/或序列变体。在特定实施方案中,序列多态性、突变和/或序列变体不影响编码的蛋白质的功能。基因不仅可包括编码序列,还可包括非编码调控区,诸如启动子、增强子和终止区。所述术语还可包括从mRNA转录物剪接的所有内含子和其他DNA序列,连同由可变剪接位点产生的变体。编码蛋白质的核酸序列可以是指导蛋白质或RNA表达的DNA或RNA。这些核酸序列可以是被转录成RNA的DNA链序列或被翻译成蛋白质的RNA序列。核酸序列包括全长核酸序列以及来源于全长蛋白质或RNA的非全长序列两者。序列还可包括天然序列的简并密码子或可被引入以提供密码子偏好的序列。因此,基因是指占据染色体上特定基因座的遗传单位,并且包括转录和/或翻译调控序列和/或编码区和/或非翻译序列(即内含子,5'和3'非翻译序列)。
编码序列是有助于编码基因的蛋白质产物的任何核苷酸序列(例如,SEQ ID NO.4、5、7和66-70)。因此,非编码序列是指不有助于编码基因的蛋白质产物的任何核酸序列。
除了所提供的特定序列之外,本文公开的蛋白质的序列以及编码它们的核苷酸序列可在可公开获得的数据库和出版物中获得。
“载体”是来源于例如,可插入或克隆核苷酸序列(例如,基因)的质粒、噬菌体、酵母或病毒的核苷酸分子(例如,DNA分子)。载体优选含有一个或多个独特的限制位点,并且可能能够在光合微生物中自主复制。自主复制载体包括作为染色体外实体存在的载体,其复制不依赖于染色体复制,例如,线性或闭合的环状质粒、染色体外元件、微型染色体或人工染色体。载体还可与光合微生物的基因组整合。这种类型的载体与其中已整合所述载体的染色体一起复制。这种载体可包含允许重组到宿主染色体的特定所需位点中的特异性序列。当前公开内使用的载体可包括用于确保自我复制的任何机制。载体可包括单个载体(或质粒)、两种或更多种载体、三种或更多种载体等,所述载体一起含有待在光合微生物中表达的感兴趣的核苷酸序列的表达所需的总DNA。
如所指示,待表达的编码序列可操作地连接至启动子;也就是说,它们被置于启动子的调控控制之下,然后所述启动子控制编码序列的转录并且任选地控制其翻译。在异源启动子/结构编码序列组合的构建中,通常优选将启动子定位在自编码序列转录起始位点的一段距离处,所述距离与天然环境中启动子与其控制的编码序列之间的距离大约相同。如本领域中已知,这个距离的一些变化可在没有失去功能的情况下进行调整。类似地,调控序列元件相对于待置于其控制下的编码序列的优选定位由元件在其天然环境;即其来源于的基因中的定位来限定。
“组成型启动子”通常在大多数条件下是活性的,即促进转录。“诱导型启动子”通常仅在某些条件下,诸如在给定分子因子(例如,IPTG)存在下或给定环境条件下是活性的。在不存在所述条件时,诱导型启动子通常不允许显著或可测量水平的转录活性。例如,诱导型启动子可根据温度、pH、激素、代谢物(例如,乳糖、甘露醇、氨基酸)、光(例如,特定波长)、渗透势(例如,盐诱导的)、重金属或抗生素来诱导。
在特定实施方案中,控制感兴趣的编码序列的转录的启动子可以是蓝细菌启动子。启动子对于修饰的光合微生物可以是内源的,或者可以是经修饰以便提高其效率的启动子。启动子还可以是来自不同光合微生物物质(诸如不同蓝细菌或细菌物种)的异源非天然启动子。
在特定实施方案中,将感兴趣的编码序列置于选自以下的启动子(P)的转录控制之下:PaztA(例如,来自鱼腥藻属(念珠藻属)菌株PCC7120);Pc1pB1;PcorT(例如,来自集胞藻属PCC6803);PcrhC;PcpcB(例如,来自蓝细菌ABICyano1(SEQ ID NO:14));PcpcBA(例如,来自集胞藻属PCC6803);PggpS(例如,来自蓝细菌ABICyano1:(SEQ ID NO:15));PhliB;PhspA;PhtpG;PisiA;PisiB;PIrtA(例如,来自蓝细菌ABICyano1;SEQ ID NO:16));PnarB;Pnb1A(例如,来自蓝细菌ABICyano1;(SEQ ID NO:17));PnirA;PntcA;PpetE;PpetJ(例如,来自蓝细菌ABICyano1;(SEQ ID NO:18));PpsbA2;PpsbD;PmrgA(例如,来自蓝细菌ABICyano1;(SEQ ID NO:19));PnbIA(例如,来自念珠藻属PCC7120);PnirA(例如,来自蓝细菌ABICyano1);PnrsB(例如,来自集胞藻属PCC6803);PnrtA;PntcA;PppsA(例如,来自蓝细菌ABICyano1(SEQ ID NO:20));PpsaA;PpsbD;PpstS(例如,来自蓝细菌ABICyano1(SEQ ID NO:21);PrbcL(例如,来自集胞藻属PCC6803);PrbcLS;PrnpA(例如,来自蓝细菌ABICyano1(SEQ ID NO:22);PrpoA;PrpsL;PsbA2(例如,来自集胞藻属PCC6803);PsigB;PsmtA(例如,来自聚球藻属PCC 7002和聚球藻属PCC 7942);以及PziaA(例如,来自集胞藻属PCC6803)。还可酌情使用来自其他物种(例如,细长聚球藻、极大节旋藻、钝顶螺旋藻和Cyanobacterium aponinum)的同源启动子。
PhspA、Pc1pB1和PhliB可通过热休克(例如,将光合微生物培养物(所述培养物)的生长温度从300℃提高至400℃)、冷休克(例如,将培养物的生长温度从300℃降低至20℃)、氧化胁迫(例如,通过向培养物中添加氧化剂,诸如过氧化氢)或渗透胁迫(例如,通过增加培养物的盐度)来诱导。PsigB可通过平稳生长、热休克和渗透胁迫来诱导。PntcA和PnblA可通过降低生长培养基中的氮浓度来诱导,并且PpsaA和PpsbA2可通过低光照或高光照条件来诱导。PhtpG可通过渗透胁迫和热休克来诱导。PcrhC可通过冷休克来诱导。铜浓度的增加可用于诱导PpetE,而PpetJ通过降低铜浓度来诱导。PaztA、PsmtA和PziaA可通过添加Zn2+来诱导。PnrsB可通过添加Ni2+来诱导。PcorT可通过添加钴来诱导。这些启动子的另外的细节可见于例如PCT/EP2009/060526。
可用的组成型或诱导型启动子还描述于例如:Samartzidou,等人,(1998)Plant Physiol.,117:225-234;Duran,等人,(2004)J.of Biol.Chem.,279:7229-7233;Singh,等人,(2006)Arch Microbiol.,186:273-286;Imamura,等人,(2003)FEBS Lett.,2003;554:357-362;Imamura,等人,(2006)J.Biol.Chem.,281:2668-2675;Agrawal,等人,(1999)Biochem.Biophys.Res.Commun.,255:47-53;Mohamed,等人,(1989)Plant Mol.Biol.,13:693-700;Muramatsu,等人,(2006)Plant Cell Physiol.,47:878-890;Marin,等人,(2004)Plant Physiol.,136:3290-3300;Marin,等人,(2002)J.Bacteriol.,184:2870-2877;Qi,等人,(2005)Appl.Environ.Microbiol.,71:5678-5684;Maeda等人,(1998)J.Bacteriol.;180:4080-4088;Herranen,等人,(2005)Plant Cell Physiol.,46:1484-1493;Buikema,等人,(2001)Proc.Natl.Acad.Sci.USA,98:2729-2734;Mary,等人,(2004)Microbiol.,150:1271-1281;He,等人,(2001)J.Biol.Chem.,276:306-314;Fang,等人,(2004)Curr.Microbiol.,49:192-198;以及Kappell,等人,(2007)Arch.Microbiol.,187:337-342中。
在存在多于一种感兴趣的编码序列的情况下,然后例如第一编码序列和第二编码序列可由一种启动子控制,从而形成转录操纵子。可替代地,第一编码序列和第二编码序列可分别可操作地连接至不同的第一启动子和第二启动子。当使用多于一种启动子时,其全部可以是组成型启动子、全部可以是诱导型启动子,或者可使用组成型启动子和诱导型启动子的组合。
当在TATA盒、操纵子序列和/或控制编码序列表达的启动子的核糖体结合位点(RBS)中引入突变时,可使表达控制变紧,使得启动子与修饰的光合微生物的内源启动子具有至少90%序列同一性。以下关于启动子PnirA、PcorT和PsmtA描述这些方法的实例。
在特定实施方案中,PnirA可具有广义核苷酸序列SEQ ID NO:23,其中每个核苷酸n独立地选自:a、t、c和g,并且其中启动子5'区域中的两个(atg)是NtcB结合位点的起始,gta是NtcA结合位点的起始,ccg表示RBS的起始,并且3'-atg是由此启动子转录性地控制的第一重组编码序列的起始密码子。
PnirA的另一种广义DNA序列包括RBS中的核苷酸变化,所述核苷酸变化导致广义DNA序列SEQ ID NO:24。在特定实施方案中,修饰的PnirA可包括操纵子区域(NtcB和NtcA的结合位点)和TATA盒中的变化,所述变化导致广义核苷酸序列SEQ ID NO:25。PnirA的另一种变体与RBS、操纵子区域和TATA盒中的变化组合以形成SEQ ID NO:26。
特定实施方案提供Co2+诱导型PcorT,其具有一般核苷酸序列SEQ ID NO:27,其中每个核苷酸n独立地选自:a、t、c和g,并且其中5'-cat是corR的起始密码子(反义取向)且3'-atg是由此启动子转录性地控制的第一重组编码序列的起始密码子。PcorT的修饰的变体包括具有SEQ ID NO:28的RBS中的变化。PcorT的另一种变体包括具有一般序列SEQ ID NO:29的TATA盒中的变化。第三种修饰的PcorT将RBS和TATA盒修饰组合到SEQ ID NO:30中。
此外,可使用来自聚球藻属PCC 7002的Zn2+-诱导型PsmtA,其具有广义核苷酸序列SEQ ID NO:31。RBS的变化可导致以下广义核苷酸序列SEQ ID NO:32或SEQ ID NO:33。同样,还可酌情使用来自其他物种(例如,细长聚球藻、极大节旋藻、钝顶螺旋藻和Cyanobacterium aponinum)的同源序列。
如所提出,特定实施方案包括密码子优化。可选择由特定光合微生物优选的密码子,以例如增加蛋白质表达的速率或产生具有期望性质(诸如与由天然存在序列产生的转录物相比更长的半衰期)的重组RNA转录物性质。此类核苷酸序列通常被称为“密码子优化的”。
待在修饰的光合微生物中表达的核苷酸序列中的至少一些可经密码子优化以在所选蓝细菌菌株中最佳表达。基本原理是,高度表达基因的密码子使用频率通常与宿主同源tRNA丰度相关。(Bulmer,Nature,1987;325:728-730)。在特定实施方案中,密码子优化是基于蓝细菌ABICyano1(以及其近亲物种)密码子使用频率(宿主密码子偏倚),以便实现期望的异源基因表达(Sharp,等人,(1987);Nucleic Acids Res.,15:1281-1295)。在特定实施方案中,密码子优化可基于细长聚球藻PCC 7942。
密码子优化可在可公开获得的软件(诸如基因设计器(DNA 2.0))的帮助下执行。还可执行使不需要的限制位点、内部Shine-Dalgarno序列和其他序列(诸如内部终止序列和重复序列)最小化的另外修饰。已显示这些一般的密码子优化方法导致异源非天然基因在目标生物体中的表达高达1000倍更高(Welch,等人,(2009)PLoS One 4,e7002;以及Welch等人,(2009)J.of the Royal Society,Interface 6(增刊4):S467-S476。
在特定实施方案中,可将编码后胆色素产生和功能途径中的一种或多种蛋白质(例如,酶、裂解酶和/或藻胆蛋白)的基因置于中性位点(例如,NS1、NS3、NS4)中的诱导型启动子之后以驱动蛋白质的表达。
在特定实施方案中,可将与SEQ ID NO:4、5、6、7、8、9、57、58、59、60、66、67、68、69或70具有至少85%序列同一性;86%序列同一性;87%序列同一性;88%序列同一性;89%序列同一性;90%序列同一性;91%序列同一性;92%序列同一性;93%序列同一性;94%序列同一性;95%序列同一性;96%序列同一性;97%序列同一性;98%序列同一性;或99%的序列同一性的基因置于启动子之后以分别驱动PebA、PebB、RpcG、变体CpcA、CpcB或CpeS的表达。还可使用与其他可公开获得的相关基因序列具有这些序列同一性的基因(例如,编码参与后胆色素产生或功能的其他酶、裂解酶或藻胆蛋白的基因)。
在特定实施方案中,可将编码酶、裂解酶或藻胆蛋白的变体的基因置于启动子之后以驱动变体的表达。
本文公开的蛋白质变体(例如,PebA、PebB、变体CpcA、CpcB、CpeS、PebS、PycA、RpcG、藻蓝蛋白)包括与参考序列相比具有一个或多个氨基酸添加、缺失、停止位置或取代的蛋白质。蛋白质变体与参考序列具有至少85%序列同一性;86%序列同一性;87%序列同一性;88%序列同一性;89%序列同一性;90%序列同一性;91%序列同一性;92%序列同一性;93%序列同一性;94%序列同一性;95%序列同一性;96%序列同一性;97%序列同一性;98%序列同一性;或99%序列同一性,并且与尚未经修饰以利用变体蛋白质以产生非天然后胆色素的光合微生物相比,响应于可见光谱的一部分引起光合微生物光合活性的统计学上显著的增加。
氨基酸取代可以是保守取代或非保守取代。“保守取代”涉及在以下保守替代组中的一种中发现的取代:第1组:丙氨酸(Ala;A)、甘氨酸(Gly;G)、丝氨酸(Ser;S)、苏氨酸(Thr;T);第2组:天冬氨酸(Asp;D)、谷氨酸(Glu;E);第3组:天冬酰胺(Asn;N)、谷氨酰胺(Gln;Q);第4组:精氨酸(Arg;R)、赖氨酸(Lys;K)、组氨酸(His;H);第5组:异亮氨酸(Ile;I)、亮氨酸(Leu;L)、甲硫氨酸(Met;M)、缬氨酸(Val;V);以及第6组:苯丙氨酸(Phe;F)、酪氨酸(Tyr;Y)、色氨酸(Trp;W)。
另外,氨基酸可通过类似的功能、化学结构或组成(例如,酸性、碱性、脂族、芳香族、含硫的)来分组为保守取代组。例如,出于取代的目的,脂族组可包括Gly、Ala、Val、Leu和Ile。含有被认为是保守取代的氨基酸的其他组包括:含硫的:Met和Cys;酸性:Asp、Glu、Asn和Gln;小的脂族非极性或轻微极性的残基:Ala、Ser、Thr、Pro和Gly;极性、带负电荷的残基及其酰胺:Asp、Asn、Glu和Gln;极性、带正电的残基:His、Arg和Lys;大的脂族非极性残基:Met、Leu、Ile、Val和Cys;以及大的芳香族残基:Phe、Tyr和Trp。如所指示,在特定实施方案中,保守取代可包括用Glu、Ser、Thr或Tyr取代Asp56。
非保守取代包括以统计学上显著的方式影响参考蛋白的功能的那些取代。非保守取代包括其中(i)亲水性残基(例如,Ser或Thr)被疏水性残基(例如,Leu、Ile、Phe、Val或Ala)取代;(ii)Cys或Pro被任何其他残基取代;(iii)具有正电性侧链的残基(例如,Lys、Arg或His)被负电性残基(例如,Gln或Asp)取代;或(iv)具有庞大侧链的残基(例如Phe)被不具有庞大侧链的残基(例如Gly)取代的那些取代。另外的信息见于Creighton(1984)Proteins,W.H.Freeman and Company。
一般而言,非保守取代将不在酶、裂解酶或藻胆蛋白中保守的位置(例如,SEQ ID NO.12和13以及保守的半胱氨酸残基)中进行。
在特定实施方案中,可将编码与参考蛋白质变体具有至少90%序列同一性;91%序列同一性;92%序列同一性;93%序列同一性;94%序列同一性;95%序列同一性;96%序列同一性;97%序列同一性;98%序列同一性;99%序列同一性;或100%序列同一性的蛋白质的基因置于启动子之后以驱动非天然后胆色素产生和/或功能途径中的蛋白质的表达。
“序列同一性%”是指如通过比较序列所确定的两种或更多种序列之间的关系。在本领域中,“同一性”还意指如通过此类序列串之间的匹配所确定的序列之间的序列相关性程度。“同一性”(经常称为“相似性”)可通过已知方法来容易地计算,所述方法包括在以下中描述的那些:Computational Molecular Biology(Lesk,A.M.编)Oxford University Press,NY(1988);Biocomputing:Informatics and Genome Projects(Smith,D.W.编)Academic Press,NY(1994);Computer Analysis of Sequence Data,第I部分(Griffin,A.M.和Griffin,H.G.编)Humana Press,NJ(1994);Sequence Analysis in Molecular Biology(Von Heijne,G.编)Academic Press(1987);以及Sequence Analysis Primer(Gribskov,M.和Devereux,J.编)Oxford University Press,NY(1992)。确定序列同一性的优选方法经设计以得到所测试序列之间的最佳匹配。确定序列同一性和相似性的方法编写于可公开获得的计算机程序中。序列比对和同一性百分比计算可使用LASERGENE生物信息学计算套件(DNASTAR,Inc.,Madison,Wisconsin)的Megalign程序来执行。序列的多重比对还可使用Clustal比对方法(Higgins和Sharp,CABIOS,1989;5:151-153以默认参数(缺口罚分=10,缺口长度罚分=10)来执行。相关程序还包括GCG程序套件(Wisconsin Package第9.0版,Genetics Computer Group(GCG),Madison,Wisconsin);BLASTP、BLASTN、BLASTX(Altschul,等人,(1990)J.Mol.Biol.,215:403-410;DNASTAR(DNASTAR,Inc.,Madison,Wisconsin);以及并入Smith-Waterman算法的FASTA程序(Pearson,Comput.Methods Genome Res.,[Proc.Int.Symp.](1994),会议日期1992,111-20.编者:Suhai,Sandor.出版者:Plenum,New York,N.Y.)。在本公开的上下文内,将理解在将序列分析软件用于分析的情况下,分析的结果是基于所引用的程序的“默认值”。如本文所用,“默认值”将意指在首次初始化时用软件最初加载的值或参数的任何集合。
并入终止位置的变体可以是生物活性片段。生物活性片段具有参考序列的活性的0.1%、0.5%、1%、2%、5%、10%、12%、14%、16%、18%、20%、22%、24%、26%、28%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%、100%、110%、120%、150%、200%、300%、400%、500%、600%、700%、800%、900%、1000%或更多。参考序列通常是指表达如本文所述的非天然后胆色素合成或功能途径活性中的蛋白质的氨基酸序列或核酸编码序列。SEQ ID NO.1、2、3、61、62、63、64和65提供示例性参考序列。
将核苷酸序列(例如,载体)插入(例如,转化)到光合微生物中可使用任何适当方法来实现,所述方法包括例如天然转化(例如,天然DNA摄取;参见,例如Chung,等人,(1998)FEMS Microbiol.Lett.,164:353-361;Frigaard,等人,(2004)Methods Mol.Biol.,;274:325-40;Zang,等人,(2007)J.Microbiol.,2007;45:241-245);接合(例如,双亲本或三亲本配对)、转导、玻璃珠转化(参见,例如Kindle,等人,(1989)J.Cell Biol.,109:2589-601;Feng,等人,(2009)Mol.Biol.Rep.,36:1433-9;美国专利号5,661,017)、碳化硅晶须转化(参见,例如Dunahay,等人,(1997)Methods Mol.Biol.,62:503-9)、基因枪(参见,例如Dawson,等人,(1997)Curr.Microbiol.,35:356-62;Hallmann,等人,(1997)Proc.Natl.Acad.USA,94:7469-7474;Doestch,等人,(2001)Curr.Genet.,39:49-60;Jakobiak,等人,(2004)Protist,155:381-93;Ramesh,等人,(2004)Methods Mol.Biol.,274:355-307;Tan,等人,(2005)J.Microbiol.,43:361-365;Steinbrenner,等人,(2006)Appl Environ.Microbiol.,72:7477-7484;Kroth,(2007)Methods Mol.Biol.,390:257-267;美国专利号5,661,017);电穿孔(参见,例如Kjaerulff,等人,(1994)Photosynth.Res.,41:277-283;Iwai,等人,(2004)Plant Cell Physiol.,45:171-5;Ravindran,等人,(2006)J.Microbiol.Methods,66:174-6;Sun,等人,(2006)Gene,377:140-149;Wang,等人,(2007)Appl.Microbiol.Biotechnol.,76:651-657;Chaurasia,等人,(2008)J.Microbiol.Methods,73:133-141;Ludwig,等人,(2008)Appl.Microbiol.Biotechnol.,78:729-35)、激光介导的转化或在任何聚(酰氨基胺)树枝状聚合物存在下或在用其预处理之后与DNA一起孵育(参见,例如Pasupathy,等人,(2008)J.Biotechnol.,3:1078-82)、聚乙二醇(参见,例如Ohnuma,等人,(2008)Plant Cell Physiol.,49:117-120)、阳离子脂质(参见,例如Muradawa,等人,(2008)J.Biosci.Bioeng.,105:77-80)、葡聚糖、磷酸钙或氯化钙(参见,例如Mendez-Alvarez,等人,(1994)J.Bacteriol.,176:7395-7397)、任选地在用细胞壁降解酶处理细胞之后(参见,例如Perrone,等人,(1998)Mol.Biol.Cell,9:3351-3365)。
此外,载体可通过添加与光合微生物基因组的目标区域同源的适当DNA序列或者通过将镶嵌末端(mosaic end,ME)引入到载体进行体内转座来修饰以允许整合到染色体中。一旦质粒在光合微生物中得以建立,则其可以例如每细胞1至许多拷贝的范围存在。
上述插入方法可用于将核苷酸序列(例如,载体)引入带有细胞外聚合物层(EPS)的蓝细菌细胞中。具有EPS的蓝细菌的非限制性实例包括若干念珠藻属和鱼腥藻属菌株,诸如普通念珠藻(Nostoc commune)和圆筒状鱼腥藻(Anabanena cylindrica)以及若干蓝丝菌属菌株,诸如蓝丝菌属PCC9224、蓝丝菌属CA 3、蓝丝菌属CE 4、蓝丝菌属ET5、蓝丝菌属ET 2和蓝螺藻(Cyanospira capsulata)ATCC43193。具有EPS的蓝细菌的其他实例包括隐球藻属、蓝细菌、倒囊藻属(Anacystis)、色球藻属、粘杆藻属、微囊藻属、集胞藻属、鞘丝藻属、微鞘藻属、颤藻属、席藻属、节旋藻属、鱼腥藻属、蓝螺藻属、念珠藻属、伪枝藻属、单歧藻属、拟绿胶藻属(Chlorogloeopsis)、侧生藻属和鞭枝藻属(Mastigocladus)(参见,例如:De Philippis等人,J.of Applied Phycology,2001;13:293-299;De Philippis,等人,(1998)FEMS Microbiol.Reviews,22:151-175)。
在蓝细菌中,限制系统可产生对引入外源核苷酸序列的阻碍。限制系统包括限制酶和特异性DNA甲基转移酶。限制酶识别序列的特异性甲基化保护光合微生物中的DNA免受相应限制酶的降解。特定细菌细胞类型中的特定限制系统的知识可允许通过在特定位点处将外源核苷酸序列甲基化来保护外源核苷酸序列以防止光合微生物的限制系统限制酶的降解。因此,理解这些限制系统可能有助于为特定细菌选择适当的转化方案。不同蓝细菌细胞的特定限制系统可见于rebase.neb.com。
如所指示,本文使用的核苷酸序列可包括可选标记以鉴定遗传修饰的光合微生物。可选标记可以是任何鉴定因子,通常为抗生素或化学抗性基因,其能够基于标记基因的作用进行选择,诸如对抗生素的抗性、对除草剂的抗性、比色标记、酶、荧光标记等,其中所述作用用于追踪感兴趣的核苷酸序列的转化和/或鉴定已经遗传感兴趣的核苷酸序列的遗传修饰的光合微生物。本领域已知并且使用的可选标记基因的实例包括:提供对氨苄青霉素、庆大霉素、潮霉素、卡那霉素、壮观霉素、链霉素、荧光蛋白(例如,来自Promega Corporation、Invitrogen、Clontech、Stratagene、BD Biosciences Pharmingen、Evrogen JSC)等的抗性的基因。
除了基于光生物反应器的技术(诸如Nedbal,等人,(2008)Biotechnol.Bioeng.,100:902-10中描述的那些技术)之外,修饰的光合微生物(包括蓝细菌)可根据本领域已知的技术来培养或培育,所述技术诸如Acreman,等人,(1994)J.of Industrial Microbiol.and Biotechnol.,13:193-194)中描述的那些。蓝细菌的典型实验室培养条件的一个实例为在BG-11培养基(ATCC培养基616)中在30℃下在通风的培养瓶中,在不断搅拌且恒定照射为30-100μmole光子m-2sec-1的情况下生长。
用于培养蓝细菌的另外培养基包括Aiba和Ogawa(AO)培养基、Allen和Amon培养基加硝酸盐(ATCC培养基1142)、Antia氏(ANT)培养基、Aquil培养基、Ashbey氏无氮琼脂、ASN-III培养基、ASP 2培养基、ASW培养基(人造海水及衍生物)、ATCC培养基617(用于海洋蓝绿藻的BG-11;改良的ATCC培养基616[BG-11培养基])、ATCC培养基819(蓝绿色固氮培养基、没有NO3的ATCC培养基616[BG-11培养基])、ATCC培养基854(具有维生素B12的ATCC培养基616[BG-11培养基])、ATCC培养基1047(具有维生素B12的ATCC培养基957[MN海洋培养基])、ATCC培养基1077(固氮海洋培养基;没有NO3的ATCC培养基957[MN海洋培养基])、ATCC培养基1234(BG-11尿嘧啶培养基;具有尿嘧啶的ATCC培养基616[BG-11培养基])、贝日阿托氏菌培养基(ATCC培养基138)、贝日阿托氏菌培养基2(ATCC培养基1193)、用于蓝绿藻的BG-11培养基(ATCC培养基616)、蓝绿色(BG)培养基、Bold氏基础培养基(BB)培养基、Castenholtz D培养基、改良的Castenholtz D培养基(嗜盐蓝细菌)、Castenholtz DG培养基、Castenholtz DGN培养基、Castenholtz ND培养基、绿屈挠菌(Chloroflexus)肉汤、绿屈挠菌培养基(ATCC培养基920)、Chu氏#10培养基(ATCC培养基341)、改良的Chu氏#10培养基、改良的Chu氏#11培养基、DCM培养基、DYIV培养基、E27培养基、E31培养基及衍生物、f/2培养基、f/2培养基衍生物、Fraquil培养基(淡水微量金属缓冲培养基)、用于藻类的Gorham氏培养基(ATCC培养基625)、h/2培养基、Jaworski氏(JM)培养基、K培养基、L1培养基及衍生物、MN海洋培养基(ATCC培养基957)、Plymouth Erdschreiber(PE)培养基、原绿球藻PC培养基、蛋白胨(PP)培养基、Prov培养基、Prov培养基衍生物、S77加维生素培养基、S88加维生素培养基、盐水营养琼脂(SNA)培养基及衍生物、SES培养基、SN培养基、改良的SN培养基、SNAX培养基、土壤/水两相(S/W)培养基及衍生物、用于节旋藻属(螺旋藻属)的SOT培养基:ATCC培养基1679、螺旋藻属(SP)培养基、van Rijn和Cohen(RC)培养基、Walsby氏培养基、Yopp培养基和Z8Medium以及其他培养基。
特定实施方案依赖于上调或下调修饰的光合微生物基因组的一部分,以在特定实施方案中降低或去除编码的蛋白质(例如,后胆色素合成或利用途径中的蛋白质)的活性。下调可通过各种机制来实现。下调可通过以下来实现:例如,减少基因的拷贝数、将外来的碱基对集合插入到基因中(例如,插入到编码区中)、缺失基因的任何部分(例如,编码区的全部或一部分)、取代基因内的碱基对(例如,至编码区中)、干扰编码的RNA转录物、存在干扰基因转录或翻译的反义序列;翻译不完整的蛋白质;不正确地折叠蛋白质;表达不稳定的蛋白质;基因转录减少;基因转录不完全或通过导致促进特定后胆色素的产生或使用的途径中蛋白质的存在、表达或活性降低的任何其他活性。
上调可通过例如增加基因的拷贝数、引入强启动子和/或诱导型启动子、防止编码核苷酸或表达的蛋白质降解的机制或其他机制来实现。
如本领域普通技术人员所理解,基因和蛋白质表达的“上调”和“下调”以及扩大的光吸收能力和增加的光合活性可针对相关的对照条件来测量,所述相关的对照条件包括相对于未修饰的光合微生物或具有不同修饰(诸如与利用非天然后胆色素无关的修饰)的光合微生物的表达或活性。
在特定实施方案中,扩大的光吸收能力意味着修饰的生物体吸收为其未修饰形式时不吸收的可见光谱部分中的光的波长。在特定实施方案中,扩大的光吸收能力意味着修饰的生物体与其未修饰形式相比吸收显著更多的可见光谱部分中的光的波长。在特定实施方案中,增加的光合活性意指特定波长的光下的氧气释放的统计学上显著的增加。在特定实施方案中,与相关对照的相关参考水平相比,光合活性的增加可以是统计学上显著的增加。如果差异处于预期将单独基于机会发生的水平内,则测量不是统计学上显著不同的。相比之下,统计学上显著的差异或增加是比预期将单独偶然发生的差异或增加更大的差异或增加。统计学显著性或其缺乏可通过本领域中使用的各种系统和方法中的任何一种来确定。统计学显著性的常用量度的实例为p值。p值表示获得等效于特定数据点的给定结果的概率,其中数据点是单独的随机机会的结果。在p值小于或等于0.05时的结果经常被认为是显著的(不是随机的机会)。
以下实例证实本公开的特定实施方案。按照本公开,本领域普通技术人员应认识到,在不脱离本公开的精神和范围的情况下可对本文所公开的特定实施方案做出许多改变并且仍获得相似或类似结果。
示例性实施方案:
1.一种修饰的光合微生物,其与没有修饰的相同物种的光合微生物相比具有增加的光合活性,其中所述增加的光合活性由所述修饰的光合微生物内功能性非天然后胆色素的存在引起。
2.如实施方案1所述的修饰的光合微生物,其中所述非天然后胆色素为藻蓝胆素(PCB)、藻红胆素(PEB)、藻尿胆素(PUB)或藻紫胆素(PVB)。
3.如实施方案2所述的修饰的光合微生物,其中所述非天然后胆色素为PEB或PUB。
4.如实施方案1、2或3所述的修饰的光合微生物,其中非天然后胆色素的存在部分地由至少一种非天然蛋白质的存在引起。
5.如实施方案4所述的修饰的光合微生物,其中所述至少一种非天然蛋白质是酶、裂解酶或藻胆蛋白。
6.如实施方案4所述的修饰的光合微生物,其中所述至少一种非天然蛋白质是胆绿素还原酶。
7.如实施方案6所述的修饰的光合微生物,其中所述胆绿素还原酶是PycA、PebA、PebB或PebS。
8.如实施方案7所述的修饰的光合微生物,其中所述胆绿素还原酶是由包含SEQ ID NO:4的序列编码的PebA,和/或由包含SEQ ID NO:5的序列编码的PebB,和/或由包含SEQ ID NO:6或SEQ ID NO:9的序列编码的PebA和PebB。
9.如实施方案4所述的修饰的光合微生物,其中所述至少一种非天然蛋白质是RpcG、CpcA、变体CpcA(例如,A86K或Y130C)、CpcB和/或CpeS(在特定实施方案中为RpcG以及(i)CpcB和变体CpcA或(ii)CpeS)。
10.如实施方案9所述的修饰的光合微生物,其中所述
(i)RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,
(ii)变体CpcA由包含SEQ ID NO:66或SEQ ID NO:67的序列编码,
(iii)CpcB由包含SEQ ID NO:68的序列编码;
(iv)变体CpcA和CpcB由包含SEQ ID NO:57或SEQ ID NO:58的序列编码;并且/或者
(v)CpeS由包含SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69或SEQ ID NO:70的序列编码。
11.如实施方案4所述的修饰的光合微生物,其中所述至少一种非天然蛋白质是选自CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE或PecF的裂解酶。
12.如实施方案4所述的修饰的光合微生物,其中所述至少一种非天然蛋白质是选自别藻蓝蛋白、藻蓝蛋白、CpcA、变体CpcA(例如,A86K或Y130C)、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RPcA的藻胆蛋白。
13.如实施方案12所述的修饰的光合微生物,其中所述非天然蛋白质是共有PEB附接位点被共有PUB附接位点替换的藻蓝蛋白。
14.如实施方案13所述的修饰的光合微生物,其中所述替换的共有PEB附接位点是SEQ ID NO:12,并且/或者所述共有PUB附接位点是SEQ ID NO:13。
15.如实施方案4所述的修饰的光合微生物,其中所述至少一种非天然蛋白质选自PycA、PebA、PebB、PebS、RpcG、CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE、PecF别藻蓝蛋白、藻蓝蛋白、CpcA、变体CpcA(例如,A86K或Y130C)、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RpcA中的一种或多种。
16.如实施方案1-15中任一项所述的修饰的光合微生物,其中功能性非天然后胆色素的存在部分地由所述修饰的光合微生物基因组的一部分的上调或下调引起。
17.如实施方案16所述的修饰的光合微生物,其中所述上调或下调包括上调或下调至少一种天然蛋白质的存在或活性。
18.如实施方案17所述的修饰的光合微生物,其中所述至少一种天然蛋白质是酶、裂解酶或藻胆蛋白。
19.如实施方案17所述的修饰的光合微生物,其中所述至少一种天然蛋白质是胆绿素还原酶。
20.如实施方案19所述的修饰的光合微生物,其中所述胆绿素还原酶是PycA、PebA、PebB或PebS。
21.如实施方案17所述的修饰的光合微生物,其中所述至少一种天然蛋白质是RpcG。
22.如实施方案17所述的修饰的光合微生物,其中所述至少一种天然蛋白质是选自CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE或PecF的裂解酶。
23.如实施方案17所述的修饰的光合微生物,其中所述至少一种天然蛋白质是将天然PCB缀合到天然藻蓝蛋白上的特异性位点的天然裂解酶。
24.如实施方案17所述的修饰的光合微生物,其中所述至少一种天然蛋白质是天然裂解酶,所述天然裂解酶然后被非天然裂解酶替换。
25.如实施方案24所述的修饰的光合微生物,其中所述天然裂解酶由包含SEQ ID NO:10的序列编码,并且所述非天然裂解酶由包含SEQ ID NO:7或SEQ ID NO:8、SEQ ID NO:11、SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69和/或SEQ ID NO:70的序列编码。
26.如实施方案17所述的修饰的光合微生物,其中所述至少一种天然蛋白质是选自别藻蓝蛋白、藻蓝蛋白、CpcA、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RpcA的藻胆蛋白。
27.如实施方案17所述的修饰的光合微生物,其中所述至少一种天然蛋白质选自PycA、PebA、PebB、PebS、RpcG、CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE、PecF别藻蓝蛋白、藻蓝蛋白、CpcA、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RpcA中的一种或多种。
28.如前述实施方案中任一项所述的修饰的光合微生物,其中所述遗传修饰的光合微生物是蓝细菌。
29.如前述实施方案中任一项所述的修饰的光合微生物,其中所述修饰的光合微生物是选自细长聚球藻、极大节旋藻、钝顶螺旋藻和Cyanobacterium aponinum的蓝细菌。
30.如前述实施方案中任一项所述的修饰的光合微生物,其中所述增加的光合活性由光吸收能力扩大和/或自遮蔽减少引起。
31.一种经修饰以利用参与后胆色素产生和/或功能的至少一种非天然蛋白质的蓝细菌。
32.如实施方案31所述的蓝细菌,其中所述至少一种非天然蛋白质是酶、裂解酶或藻胆蛋白。
33.如实施方案31所述的蓝细菌,其中所述至少一种非天然蛋白质是胆绿素还原酶。
34.如实施方案33所述的蓝细菌,其中所述胆绿素还原酶是PycA、PebA、PebB或PebS。
35.如实施方案33所述的蓝细菌,其中所述胆绿素还原酶是由包含SEQ ID NO:4的序列编码的PebA,和/或由包含SEQ ID NO:5的序列编码的PebB,和/或由包含SEQ ID NO:6或SEQ ID NO:9的序列编码的PebA和PebB。
36.如实施方案31所述的蓝细菌,其中所述至少一种非天然蛋白质是RpcG、CpcA、变体CpcA(例如,A86K或Y130C)、CpcB和/或CpeS(在特定实施方案中为RpcG、(i)CpcB和变体CpcA或(ii)CpeS)。
37.如实施方案36所述的蓝细菌,其中所述
(i)RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,
(ii)变体CpcA由包含SEQ ID NO:66或SEQ ID NO:67的序列编码,
(iii)CpcB由包含SEQ ID NO:68的序列编码;
(iv)变体CpcA和CpcB由包含SEQ ID NO:57或SEQ ID NO:58的序列编码;并且/或者
(v)CpeS由包含SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69或SEQ ID NO:70的序列编码。
38.如实施方案31所述的蓝细菌,其中所述至少一种非天然蛋白质是选自CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE或PecF的裂解酶。
39.如实施方案31所述的蓝细菌,其中所述至少一种非天然蛋白质是选自别藻蓝蛋白、藻蓝蛋白、CpcA、变体CpcA(例如,A86K或Y130C)、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RpcA的藻胆蛋白。
40.如实施方案39所述的蓝细菌,其中所述非天然蛋白质是共有PEB附接位点被共有PUB附接位点替换的藻蓝蛋白。
41.如实施方案40所述的蓝细菌,其中所述替换的共有PEB附接位点是SEQ ID NO:12,并且/或者所述共有PUB附接位点是SEQ ID NO:13。
42.如实施方案31所述的蓝细菌,其中所述至少一种非天然蛋白质选自PycA、PebA、PebB、PebS、RpcG、CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE、PecF别藻蓝蛋白、藻蓝蛋白、CpcA、变体CpcA(例如,A86K或Y130C)、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RpcA中的一种或多种。
43.如实施方案31所述的蓝细菌,其经进一步修饰以上调或下调编码参与后胆色素产生和/或功能的至少一种天然蛋白质的所述蓝细菌基因组的一部分。
44.如实施方案43所述的蓝细菌,其中所述至少一种天然蛋白质是胆绿素还原酶。
45.如实施方案44所述的蓝细菌,其中所述胆绿素还原酶是PycA、PebA、PebB或PebS。
46.如实施方案45所述的蓝细菌,其中所述胆绿素还原酶是由包含SEQ ID NO:4的序列编码的PebA,和/或由包含SEQ ID NO:5的序列编码的PebB,和/或由包含SEQ ID NO:6或SEQ ID NO:9的序列编码的PebA和PebB。
47.如实施方案43所述的蓝细菌,其中所述至少一种天然蛋白质是RpcG、CpcA、CpcB或CpeS。
48.如实施方案47所述的蓝细菌,其中
(i)RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,
(ii)变体CpcA由包含SEQ ID NO:66或SEQ ID NO:67的序列编码,
(iii)CpcB由包含SEQ ID NO:68的序列编码;
(iv)变体CpcA和CpcB由包含SEQ ID NO:57或SEQ ID NO:58的序列编码;并且/或者
(v)CpeS由包含SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69或SEQ ID NO:70的序列编码。
49.如实施方案43所述的蓝细菌,其中所述至少一种天然蛋白质是选自CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE或PecF的裂解酶。
50.如实施方案43所述的蓝细菌,其中所述至少一种天然蛋白质是将天然PCB缀合到天然藻蓝蛋白上的特异性位点的天然裂解酶。
51.如实施方案43所述的蓝细菌,其中所述至少一种天然蛋白质是天然裂解酶,所述天然裂解酶然后被非天然裂解酶替换。
52.如实施方案51所述的蓝细菌,其中所述天然裂解酶由包含SEQ ID NO:10的序列编码,并且所述非天然裂解酶由包含SEQ ID NO:7、SEQ ID NO:8、SEQ ID NO:11、SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69和/或SEQ ID NO:70的序列编码。
53.如实施方案43所述的蓝细菌,其中所述至少一种天然蛋白质是选自别藻蓝蛋白、藻蓝蛋白、CpcA、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RpcA的藻胆蛋白。
54.如实施方案43所述的蓝细菌,其中所述至少一种天然蛋白质选自PycA、PebA、PebB、PebS、RpcG、CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE、PecF别藻蓝蛋白、藻蓝蛋白、CpcA、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RpcA中的一种或多种。
55.如实施方案42-54中任一项所述的蓝细菌,其中所述蓝细菌选自细长聚球藻、极大节旋藻、钝顶螺旋藻和Cyanobacterium aponinum。
56.如实施方案42-55中任一项所述的蓝细菌,其中所述修饰扩大所述蓝细菌的光吸收能力。
57.如实施方案42-56中任一项所述的蓝细菌,其中所述修饰增加光合活性。
58.如实施方案57所述的蓝细菌,其中所述增加的光合活性由自遮蔽减少引起。
59.一种增加光合微生物中的光合活性的方法,其包括修饰所述光合微生物以利用功能性非天然后胆色素,从而增加光合活性。
60.如实施方案59所述的方法,其中所述修饰导致利用(例如,产生)选自藻蓝胆素(PCB)、藻红胆素(PEB)、藻尿胆素(PUB)、藻紫胆素(PVB)的功能性非天然后胆色素。
61.如实施方案60所述的方法,其中所述修饰导致利用(例如,产生)功能性非天然PEB或PUB。
62.如实施方案59、60或61所述的方法,其中所述修饰包括插入编码参与后胆色素合成和/或功能的至少一种非天然蛋白质的一种或多种外源核苷酸序列。
63.如实施方案62所述的方法,其中所述至少一种非天然蛋白质是酶、裂解酶或藻胆蛋白。
64.如实施方案62所述的方法,其中所述至少一种非天然蛋白质是胆绿素还原酶。
65.如实施方案64所述的方法,其中所述胆绿素还原酶是PycA、PebA、PebB或PebS。
66.如实施方案64所述的方法,其中所述胆绿素还原酶是由包含SEQ ID NO:4的序列编码的PebA,和/或由包含SEQ ID NO:5的序列编码的PebB,和/或由包含SEQ ID NO:6或SEQ ID NO:9的序列编码的PebA和PebB。
67.如实施方案62所述的方法,其中所述至少一种非天然蛋白质是RpcG、CpcA、变体CpcA(例如,A86K或Y130C)、CpcB和/或CpeS(在特定实施方案中为RpcG以及(i)CpcB和变体CpcA或(ii)CpeS)。
68.如实施方案67所述的方法,其中所述
(i)RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,
(ii)变体CpcA由包含SEQ ID NO:66或SEQ ID NO:67的序列编码,
(iii)CpcB由包含SEQ ID NO:68的序列编码;
(iv)变体CpcA和CpcB由包含SEQ ID NO:57或SEQ ID NO:58的序列编码;并且/或者
(v)CpeS由包含SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69或SEQ ID NO:70的序列编码。
69.如实施方案62所述的方法,其中所述至少一种非天然蛋白质是选自CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE或PecF的裂解酶。
70.如实施方案62所述的方法,其中所述至少一种非天然蛋白质是选自别藻蓝蛋白、藻蓝蛋白、CpcA、变体CpcA(例如,A86K或Y130C)、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RPCA的藻胆蛋白。
71.如实施方案62所述的方法,其中所述非天然蛋白质是共有PEB附接位点被共有PUB附接位点替换的藻蓝蛋白。
72.如实施方案71所述的方法,其中所述替换的共有PEB附接位点是SEQ ID NO:12,并且/或者所述共有PUB附接位点是SEQ ID NO:13。
73.如实施方案62所述的方法,其中所述至少一种非天然蛋白质选自PycA、PebA、PebB、PebS、RpcG、CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE、PecF别藻蓝蛋白、藻蓝蛋白、CpcA、变体CpcA(例如,A86K或Y130C)、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RpcA中的一种或多种。
74.如实施方案59、60、61或62所述的方法,其中所述修饰包括或还包括上调或下调编码参与后胆色素合成和/或功能的至少一种天然蛋白质的一种或多种内源核苷酸序列。
75.如实施方案74所述的方法,其中所述至少一种天然蛋白质是胆绿素还原酶。
76.如实施方案75所述的方法,其中所述胆绿素还原酶是PycA、PebA、PebB或PebS。
77.如实施方案75所述的方法,其中所述胆绿素还原酶是由包含SEQ ID NO:4的序列编码的PebA,和/或由包含SEQ ID NO:5的序列编码的PebB,和/或由包含SEQ ID NO:6或SEQ ID NO:9的序列编码的PebA和PebB。
78.如实施方案74所述的方法,其中所述至少一种天然蛋白质是RpcG、CpcA、CpcB和/或CpeS。
79.如实施方案78所述的方法,其中所述
(i)RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,
(ii)变体CpcA由包含SEQ ID NO:66或SEQ ID NO:67的序列编码,
(iii)CpcB由包含SEQ ID NO:68的序列编码;
(iv)变体CpcA和CpcB由包含SEQ ID NO:57或SEQ ID NO:58的序列编码;并且/或者
(v)CpeS由包含SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69或SEQ ID NO:70的序列编码。
80.如实施方案62所述的方法,其中所述至少一种天然蛋白质是选自CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE或PecF的裂解酶。
81.如实施方案74所述的方法,其中所述至少一种天然蛋白质是将天然PCB缀合到天然藻蓝蛋白上的特异性位点的天然裂解酶。
82.如实施方案74所述的方法,其中所述至少一种天然蛋白质是天然裂解酶,所述天然裂解酶然后被非天然裂解酶替换。
83.如实施方案82所述的方法,其中所述天然裂解酶由包含SEQ ID NO:10的序列编码,并且所述非天然裂解酶由包含SEQ ID NO:7、SEQ ID NO:8、SEQ ID NO:11、SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69和/或SEQ ID NO:70的序列编码。
84.如实施方案74所述的方法,其中所述至少一种天然蛋白质是选自别藻蓝蛋白、藻蓝蛋白、CpcA、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RpcA的藻胆蛋白。
85.如实施方案74所述的方法,其中所述至少一种天然蛋白质选自PycA、PebA、PebB、PebS、RpcG、CpcE、CpeS、CpcF、CpcS、CpcU、CpcT、PecE、PecF别藻蓝蛋白、藻蓝蛋白、CpcA、PecA、CpcB、ApcA、ApcB、ApcD、ApcF和RpcA中的一种或多种。
86.如实施方案59-85中任一项所述的方法,其中所述光合微生物是蓝细菌。
87.如实施方案86所述的方法,其中所述蓝细菌选自细长聚球藻、极大节旋藻、钝顶螺旋藻和Cyanobacterium aponinum。
88.如实施方案59-87中任一项所述的方法,其中所述修饰扩大所述蓝细菌的光吸收能力。
89.如实施方案59-88中任一项所述的方法,其中所述修饰增加光合活性。
90.如实施方案89所述的方法,其中所述增加的光合活性由自遮蔽减少引起。
91.一种减少自遮蔽的方法,其包括实践如实施方案59-90中任一项所述的方法。
92.一种具有红色色素或紫色色素的藻蓝蛋白。
93.一种蓝细菌,其产生具有红色色素的藻蓝蛋白和/或具有紫色色素的藻蓝蛋白。
94.如实施方案93所述的蓝细菌,其中所述蓝细菌选自细长聚球藻、极大节旋藻、钝顶螺旋藻和Cyanobacterium aponinum。
95.一种产生蓝细菌的方法,所述方法产生具有红色色素的藻蓝蛋白和/或具有紫色色素的藻蓝蛋白,所述方法包括选择产生天然PCB的蓝细菌,以及修饰所述蓝细菌以产生PEB和/或PUB。
96.如实施方案95所述的方法,其中所述修饰包括插入编码参与PEB和/或PUB合成和/或功能的至少一种非天然蛋白质的一种或多种外源核苷酸序列。
97.如实施方案96所述的方法,其中所述至少一种非天然蛋白质是酶、裂解酶或藻胆蛋白。
98.如实施方案96所述的方法,其中所述至少一种非天然蛋白质是胆绿素还原酶。
99.如实施方案98所述的方法,其中所述胆绿素还原酶是PebA或PebB。
100.如实施方案99所述的方法,其中所述PebA由包含SEQ ID NO:4的序列编码,并且/或者所述PebB由包含SEQ ID NO:5的序列编码,并且/或者PebA和PebB由包含SEQ ID NO:6或SEQ ID NO:9的序列编码。
101.如实施方案96所述的方法,其中所述至少一种非天然蛋白质是RpcG、CpcA、变体CpcA(例如,A86K或Y130C)、CpcB和/或CpeS(在特定实施方案中为RpcG以及(i)CpcB和变体CpcA或(ii)CpeS)。
102.如实施方案101所述的方法,其中所述
(i)RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,
(ii)变体CpcA由包含SEQ ID NO:66或SEQ ID NO:67的序列编码,
(iii)CpcB由包含SEQ ID NO:68的序列编码;
(iv)变体CpcA和CpcB由包含SEQ ID NO:57或SEQ ID NO:58的序列编码;并且/或者
(v)CpeS由包含SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69或SEQ ID NO:70的序列编码。
103.如实施方案96所述的方法,其中所述非天然蛋白质是共有PEB附接位点被共有PUB附接位点替换的藻蓝蛋白。
104.如实施方案103所述的方法,其中所述替换的共有PEB附接位点是SEQ ID NO:12,并且/或者所述共有PUB附接位点是SEQ ID NO:13。
105.如实施方案96所述的方法,其中所述至少一种非天然蛋白质是裂解酶。
106.如实施方案95或96所述的方法,其中所述修饰包括或还包括缺失或下调编码参与PCB、PEB和/或PUB合成和/或功能的至少一种天然蛋白质的一种或多种内源核苷酸序列。
107.如实施方案106所述的方法,其中所述至少一种天然蛋白质是胆绿素还原酶。
108.如实施方案107所述的方法,其中所述胆绿素还原酶是PycA、PebA或PebB。
109.如实施方案108所述的方法,其中所述胆绿素还原酶是由包含SEQ ID NO:4的序列编码的PebA,和/或由包含SEQ ID NO:5的序列编码的PebB,和/或由包含SEQ ID NO:6或SEQ ID NO:9的序列编码的PebA和PebB。
110.如实施方案106所述的方法,其中所述至少一种天然蛋白质是裂解酶。
111.如实施方案110所述的方法,其中所述裂解酶是CpcE。
112.如实施方案110所述的方法,其中所述裂解酶是将天然PCB缀合到天然藻蓝蛋白上的特异性位点的天然裂解酶。
113.如实施方案110所述的方法,其中所述裂解酶然后被非天然裂解酶替换。
114.如实施方案113所述的方法,其中替换的裂解酶由包含SEQ ID NO:10的序列编码,并且所述非天然裂解酶由包含SEQ ID NO:7、SEQ ID NO:8、SEQ ID NO:11、SEQ ID NO;59、SEQ ID NO:60、SEQ ID NO:69和/或SEQ ID NO:70的序列编码。
115.如实施方案95-114中任一项所述的方法,其中所述蓝细菌选自细长聚球藻、极大节旋藻、钝顶螺旋藻和Cyanobacterium aponinum。
116.如实施方案95-115中任一项所述的方法,其中所述修饰扩大所述蓝细菌的光吸收能力。
117.如实施方案95-116中任一项所述的方法,其中所述修饰增加光合活性。
118.如实施方案117所述的方法,其中所述增加的光合活性由自遮蔽减少引起。
119.一种修饰的蓝细菌,其与没有修饰的相同物种的蓝细菌相比具有增加的光合活性,其中所述修饰导致功能性非天然PEB的产生和所述增加的光合活性。
120.如实施方案119所述的修饰的蓝细菌,其中所述修饰包括插入编码PebA和PebB的一种或多种外源核苷酸序列。
121.如实施方案120所述的修饰的蓝细菌,其中所述PebA由包含SEQ ID NO:4的序列编码,并且/或者所述PebB由包含SEQ ID NO:5的序列编码,并且/或者PebA和PebB由包含SEQ ID NO:6或SEQ ID NO:9的序列编码。
122.如实施方案119或120所述的修饰的蓝细菌,其中进一步修饰导致功能性非天然PUB的产生和增加的光合活性。
123.如实施方案122所述的修饰的蓝细菌,其中所述修饰包括插入编码其中共有PEB附接位点被共有PUB附接位点替换的藻蓝蛋白的一种或多种外源核苷酸序列。
124.如实施方案123所述的方法,其中所述替换的共有PEB附接位点是SEQ ID NO:12,并且/或者所述共有PUB附接位点是SEQ ID NO:13。
125.如实施方案122所述的方法,其中所述修饰包括缺失或下调将天然PCB缀合到天然藻蓝蛋白上的特异性位点的天然裂解酶。
126.如实施方案125所述的方法,其中所述修饰还包括用非天然裂解酶替换缺失或下调的裂解酶。
127.如实施方案122-125所述的修饰的蓝细菌,其中所述进一步修饰包括(i)插入编码RpcG和/或CpeS的一种或多种外源核苷酸序列,以及(ii)缺失或下调编码CpcE的一种或多种内源核苷酸序列。
128.如实施方案127所述的修饰的蓝细菌,其中所述RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,并且所述CpeS由包含SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69或SEQ ID NO:70的序列编码。
129.如实施方案127所述的修饰的蓝细菌,其中编码CpcE的所述内源核苷酸序列包含SEQ ID NO:10。
130.如实施方案129所述的修饰的蓝细菌,其中缺失或下调的SEQ ID NO:10被包含SEQ ID NO:7、SEQ ID NO:8、SEQ ID NO:11、SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69和/或SEQ ID NO:70的序列替换。
131.如实施方案119或120所述的修饰的蓝细菌,其中所述进一步修饰包括插入编码RpcG、CpcB和变体CpcA的一种或多种外源核苷酸序列。
132.如实施方案131所述的修饰的蓝细菌,其中所述
(i)RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,
(ii)变体CpcA由包含SEQ ID NO:66或SEQ ID NO:67的序列编码,
(iii)CpcB由包含SEQ ID NO:68的序列编码;并且/或者
(iv)变体CpcA和CpcB由包含SEQ ID NO:57或SEQ ID NO:58的序列编码。
133.如实施方案119-132所述的修饰的蓝细菌,其中所述修饰的蓝细菌选自细长聚球藻、极大节旋藻、钝顶螺旋藻和Cyanobacterium aponinum。
134.一种增加蓝细菌中的光合活性的方法,其包括插入编码PebA和PebB的一种或多种外源核苷酸序列,其中所述PebA和PebB的表达导致功能性非天然PEB的产生和增加的光合活性。
135.如实施方案134所述的方法,其中所述PebA由包含SEQ ID NO:4的序列编码,并且/或者所述PebB由包含SEQ ID NO:5的序列编码,并且/或者PebA和PebB由包含SEQ ID NO:6或SEQ ID NO:9的序列编码。
136.如实施方案134或135所述的方法,其还包括插入编码RpcG和CpeS的一种或多种外源核苷酸序列,以及缺失或下调编码CpcE的内源核苷酸序列,并且其中另外的修饰导致功能性非天然PUB的产生和增加的光合活性。
137.如实施方案134所述的方法,其中所述RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,并且所述CpeS由包含SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69或SEQ ID NO:70的序列编码。
138.如实施方案136或137所述的方法,其中编码CpcE的所述内源核苷酸序列包含SEQ ID NO:10。
139.如实施方案138所述的方法,其中缺失或下调的SEQ ID NO:10被包含SEQ ID NO:7或SEQ ID NO:8、SEQ ID NO:11、SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69和/或SEQ ID NO:70的序列替换。
140.如实施方案134或135所述的方法,其还包括插入编码RpcG、CpcB和变体CpcA的一种或多种外源核苷酸序列。
141.如实施方案140所述的方法,其中所述
(i)RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,
(ii)变体CpcA由包含SEQ ID NO:66或SEQ ID NO:67的序列编码,
(iii)CpcB由包含SEQ ID NO:68的序列编码;并且/或者
(iv)变体CpcA和CpcB由包含SEQ ID NO:57或SEQ ID NO:58的序列编码。
142.如实施方案119-141中任一项所述的方法,其中所述修饰的蓝细菌选自细长聚球藻、极大节旋藻、钝顶螺旋藻和Cyanobacterium aponinum。
143.如权利要求119-142中任一项所述的方法,其中所述增加的光合活性由蓝细菌的光吸收能力的扩大和/或自遮蔽的减少引起。
144.一种天然产生藻蓝胆素(PCB)的蓝细菌,其中蓝细菌经修饰以产生功能性非天然PEB和功能性非天然PUB,并且其中所述蓝细菌产生具有红色色素的藻蓝蛋白和具有紫色色素的藻蓝蛋白。
145.如实施方案144所述的蓝细菌,其由于插入编码PebA和PebB的一种或多种外源核苷酸序列而产生功能性非天然PEB。
146.如实施方案145所述的蓝细菌,其由于插入编码PebA、PebB、CpeS和RpcG的一种或多种外源核苷酸序列以及缺失或下调编码CpcE的一种或多种内源核苷酸序列而产生功能性非天然PUB。
147.如实施方案145所述的蓝细菌,其由于插入编码PebA、PebB、RpcG、CpcB和变体CpcA的一种或多种外源核苷酸序列而产生功能性非天然PUB。
148.如实施方案145-147中任一项所述的方法,其中所述PebA由包含SEQ ID NO:4的序列编码,并且/或者所述PebB由包含SEQ ID NO:5的序列编码,并且/或者PebA和PebB由包含SEQ ID NO:6或SEQ ID NO:9的序列编码。
149.如实施方案146所述的方法,其中所述RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,并且所述CpeS由包含SEQ ID NO:59、SEQ ID NO:60、SEQ ID NO:69或SEQ ID NO:70的序列编码。
150.如实施方案146、148或149中任一项所述的方法,其中编码CpcE的所述内源核苷酸序列包含SEQ ID NO:10。
151.如实施方案147或148所述的方法,其中
(i)RpcG由包含SEQ ID NO:7或SEQ ID NO:8的序列编码,
(ii)变体CpcA由包含SEQ ID NO:66或SEQ ID NO:67的序列编码,
(iii)CpcB由包含SEQ ID NO:68的序列编码;并且/或者
(iv)变体CpcA和CpcB由包含SEQ ID NO:57或SEQ ID NO:58的序列编码。
152.一种具有红色或紫色色素的藻蓝蛋白,其来源于如权利要求144-151中任一项所述的蓝细菌。
153.如前述实施方案中任一项所述的实施方案,其中
PebA包含SEQ ID NO:2。
154.如前述实施方案中任一项所述的实施方案,其中
PebB包含SEQ ID NO:3。
155.如前述实施方案中任一项所述的实施方案,其中RpcG包含SEQ ID NO:1。
156.如前述实施方案中任一项所述的实施方案,其中变体CpcA包含SEQ ID NO:61。
157.如前述实施方案中任一项所述的实施方案,其中
变体CpcA包含SEQ ID NO:62。
158.如前述实施方案中任一项所述的实施方案,其中
CpcB包含SEQ ID NO:63。
159.如前述实施方案中任一项所述的实施方案,其中
CpeS包含SEQ ID NO:64。
160.如前述实施方案中任一项所述的实施方案,其中
CpeS包含SEQ ID NO:65。
161.一种修饰的蓝细菌,其与没有修饰的相同物种的蓝细菌相比具有增加的光合活性,其中所述修饰包括:(1)插入编码以下的一种或多种外源核苷酸序列:(i)用于合成外源后胆色素的一种或多种酶,(ii)一种或多种外源后胆色素裂解酶,和/或(iii)一种或多种野生型和/或变体藻蓝蛋白;和/或(2)(i)插入编码以下的一种或多种外源核苷酸序列:(a)用于合成外源后胆色素的一种或多种酶,和/或(b)一种或多种外源后胆色素裂解酶,和(ii)缺失或下调编码一种或多种内源后胆色素裂解酶的一种或多种内源核苷酸序列。
162.一种修饰的蓝细菌,其与没有修饰的相同物种的蓝细菌相比具有增加的光合活性,其中所述修饰包括:(1)插入编码以下的一种或多种外源核苷酸序列:用于合成外源后胆色素的酶、外源后胆色素裂解酶,和藻蓝蛋白的野生型和/或变体;或(2)(i)插入编码以下的一种或多种外源核苷酸序列:用于合成外源后胆色素的酶、2种或更多种外源后胆色素裂解酶,和(ii)缺失或下调编码内源后胆色素裂解酶的内源核苷酸序列。
163.如实施方案161或162所述的修饰的蓝细菌,其中所述酶、后胆色素、后胆色素裂解酶、藻蓝蛋白是本文公开的一种或多种酶、后胆色素、后胆色素裂解酶、藻蓝蛋白。
如先前所陈述,CpcE和CpcF经常形成异二聚体。因此,如本文所用,并且在特定实施方案中,CpcE的缺失或下调(包括编码CpcE的内源核苷酸序列的缺失或下调)可包括CpcE自身的缺失或下调和/或CpcF的缺失或下调,因为当活性是基于异二聚体形成时,任一者或两者的缺失或下调缺失或下调CpcE活性。
详细的实验方法。生物体和培养条件。在本公开的实验中使用的蓝细菌菌株是野生型Syn 7942或来源于Syn 7942的突变菌株。使所有菌株在荧光灯(120μE×m-2×s-1)下,在3×BG-11培养基、10mM磷酸钠(pH 7.2)中,在30℃、1%CO2下以及在轨道式震荡器(200rpm)上连续搅拌的情况下以光合自养方式生长。
后胆色素吸收光谱。因为后胆色素的检测在全细胞吸收光谱中可能被其他色素(诸如类胡萝卜素)的存在阻碍,所以制备水溶性无细胞裂解液。将培养物通过离心(3,000×g)收获,重悬于150μl缓冲液(由pH 7的20mM HEPES+10mM EDTA+100mM DTT+100mM Na2CO3组成)中,添加至玻璃珠(100μm直径)中,并且置于珠搅拌器(bead beater)中3min(运行30秒,休息30秒)。然后将液体转移至1.7ml离心管中并且以14,000rpm离心20min。用SpectraMax M5光谱仪采集所得细胞和无膜上清液的吸收光谱。
通过硫酸铵沉淀将无细胞裂解物进一步分级成别藻蓝蛋白或藻蓝蛋白。别藻蓝蛋白在大约40%硫酸铵下沉淀,并且藻蓝蛋白在50%硫酸铵下沉淀。在分级和沉淀后,将别藻蓝蛋白或藻蓝蛋白重悬于10mM HEPES(pH 7)中,并且用SpectraMax M5光谱仪采集吸收光谱。
作用光谱。使用克拉克式O2电极和LED光源采集作用光谱,其中测试不同波长的光在Syn 7942的全细胞中刺激氧气释放的能力。使用可变输出装置提供LED照明,所述可变输出装置可自20μE×m-2×s-1调整至1000μE×m-2×s-1,并且可供应作为405nm、450nm、505nm、520nm、590nm、650nm或710nm波长光的光。此外,还可通过光源供应白色LED光(4,000K)。LED光源由Reliance Laboratories LLC,1240West Sims Way#137,Port Townsend,WA98368生产。对于全细胞作用光谱,将培养物通过离心(14,000rpm)收获,并且重悬于具有20mM碳酸氢钠和10mM HEPES的新鲜BG-11培养基(pH 7.2)中。为了从重悬的培养物中去除一些氧气,用氩气或氮气冲洗培养物10秒。然后立即将重悬的细胞置于克拉克电极室中,使其在黑暗中在不断搅拌的情况下平衡2min,并且然后供应图中所指示的波长和强度的光。
SDS PAGE和醋酸锌染色。将3ml的OD750为1的培养物通过离心(3,000×g)收获,重悬于150μl缓冲液(由pH 7的20mM HEPES+10mM EDTA+100mM DTT+100mM Na2CO3组成)中。将样品添加至玻璃珠,在珠搅拌器中破碎,并且通过以14,000rpm离心20min从可溶性细胞裂解物中分离膜。将80μL水溶性上清液添加至54μL30%蔗糖和5%SDS的溶液中。然后用针在每个样品管中放置一个小孔,将样品煮沸60秒,在冰上冷却2分钟,并且装载到4%-20%梯度的聚丙烯酰胺凝胶上。在165伏下进行凝胶电泳1小时,并且可基于其颜色使藻胆蛋白可视化。然后将凝胶用20mM醋酸锌染色20min,并且用蓝色光源(470nm波长的光)和琥珀色滤光片可视化。然后可基于荧光鉴定后胆色素。
通过LC/MS检测藻胆素。将部分纯化的PCB溶解在1ml MeOH中。通过LC/MS分析蓝色/绿色溶液以使用具有Waters 2998光电二极管阵列(PDA)、Waters 2424ELSD和Micromass ZQ的Waters 2695分离模块鉴定PCB和降解产物的主要构象。电喷雾源(ESI+)150-2000,采用多次25ul注射以进行分析。将样品在Phenomenex Gemini C-18:250mm x 4.6mm;4.6um柱上在25C下分离。水、0.1%三氟乙酸(A)和乙腈、0.1%三氟乙酸(B)的线性梯度如下:T=0min(A=25%B=75%),T=30min(A=75%B=25%)。通过215nm-700nm的UV/Vis并且通过150至2000amu的质量来监测代谢物。
如本领域普通技术人员将理解的,本文所公开的每个实施方案可包括以下项、基本上由或由以下项组成:其特别陈述的元件、步骤、成分或组分。因此,术语“包括(include)”或“包括(including)”应理解为叙述:“包括(comprise)、由...组成(consist of)或基本上由...组成(consist essentially of)”。如本文所用的过渡术语“包括(comprise)”或“包括(comprises)”意指包括但不限于,并且允许包括未指定的元件、步骤、成分或组分,甚至在较大的量时。过渡短语“由...组成”排除未指定的任何元件、步骤、成分或组分。过渡短语“基本上由...组成”将实施方案的范围限制为指定的元件、步骤、成分或组分以及并不实质地影响所述实施方案的那些。如本文所用,材料效应将引起光吸收能力扩大的统计学上显著的降低。
除非另外指示,否则说明书和权利要求书中所用的表示成分的量,如分子量、反应条件的性质等的所有数字应当理解为在所有情况下都由术语“约”修饰。因此,除非有相反的指示,否则说明书和所附权利要求书中所陈述的数值参数都是近似值,该值可根据本发明要寻求获得的所需性质而变化。在最低限度并且不试图限制对权利要求书范围的等效范围的原则的应用,每个数值参数均应当至少根据报道的有效数字的数值和通过应用普通四舍五入技术来解读。当需要进一步清楚时,术语“约”在与陈述的数值或范围结合使用时具有本领域技术人员合理地归于它的含义,即将稍微大于或稍微小于陈述值或范围表示为在以下范围内:陈述值的±20%;陈述值±19%;陈述值±18%;陈述值±17%;陈述值±16%;陈述值±15%;陈述值±14%;陈述值±13%;陈述值±12%;陈述值±11%;陈述值±10%;陈述值±9%;陈述值±8%;陈述值±7%;陈述值±6%;陈述值±5%;陈述值±4%;陈述值±3%;陈述值±2%;或陈述值±1%。
尽管阐述本发明的宽泛范围的数值范围和参数是近似值,但在特定实例中所阐述的数值尽可能精确地加以报告。然而,任何数值固有地包含必然由它们各自测试测量值中发现的标准偏差所产生的某些误差。
除非本文另外指示或者与上下文含义明显相悖,否则在描述本发明的上下文中(尤其在所附权利要求书的上下文中)使用的术语“一个/种”、“所述”以及类似的指代语应解读为涵盖单数和复数指代语。本文中所列举数值的范围仅意在作为一种对落在所述范围内的每个单独数值单独提及的简便方法。除非本文另外指示,否则每个单独数值都并入到本说明书中,如同本文中单独列举了每个单独数值一样。可按任何适合的顺序执行本文所描述的所有方法,除非本文另外指示或明显与上下文矛盾。本文提供的任何和所有实施例或示例性语言(例如,“诸如”)仅意在更好地说明本发明,而不会对以其他方式要求保护的本发明的范围构成限制。本说明书中的语言不应解读为指示任何未要求保护的要素是实践本发明所必需的。
本文所公开的本发明的替代性元素或实施方案的分组不应被理解为限制。每个组成员可单独或者以与组中的其他成员或本文发现的其他要素的任何组合被提及和要求保护。出于便利和/或可专利性的原因,预期组中的一个或多个成员可包括在组中或从组中删除。当出现任何这种包括或删除时,本说明书被认为包含所修改的组,因此满足所附权利要求书中使用的所有马库什组的书面说明。
本文描述了本发明的某些实施方案,包括为本发明人所知用于实施本发明的最佳方式。当然,本领域技术人员在阅读前面的说明后将会明了这些所述实施方案的变型。本发明人预期本领域的熟练技术人员会适时采用此类变型,而且本发明人预期能以除了本文具体描述的方式之外的方式来实施。因此,本发明包括在随附的适用法律允许的权利要求中叙述的主题的所有修改和等效物。此外,除非本文另有指示或与上下文明显矛盾,否则在所有可行变型中的上述元素的任何组合都涵盖在本发明内。
此外,如果在本说明书(本文中的参考材料)通篇中对专利、印刷出版物、期刊文章和其他书面文本进行参考,则参考材料中的每个的参考教义均单独地以引用方式整体并入本文。
最后,应当理解,本文所公开的本发明的实施方案是对本发明原理的说明。可用的其他修改形式也在本发明的范围内。因此,例如,但不限于,可根据本文的教义利用本发明的替代性配置。因而,本发明不限于明确示出和描述的方案。
本文示出的细节是作为实施例并且仅出于对本发明的优选实施方案的说明性讨论的目的,并且之所以呈现这些细节,是为了提供认为是本发明的各种实施方案的原理和概念方面的最有用和易理解的描述的内容。在这点上,除对于本发明的基本理解所必需的之外,没有企图更详细地示出本发明的结构细节,借助于附图和/或实施例所作的描述使得本领域技术人员明了如何可以在实践中实施本发明的若干形式。
除非在以下实施例中明确并毫无疑问地修改或是当含义的应用使得任何构建无意义或基本上无意义,否则在本公开中使用的定义和解释意指并且旨在控制任何未来构建。在术语的构建将使它无意义或基本上无意义的情况下,定义应该从韦氏字典(Webster's Dictionary),第3版或本领域普通技术人员已知的字典诸如生物化学和分子生物学牛津字典(Anthony Smith编著,牛津大学出版社,Oxford,2004)中取得。
序列表
<110> 鲁门生物科学股份有限公司
<120> 具有扩大的光吸收能力和增加的光合活性的微生物
<130> M077-0021PCT
<150> US 62/191,171
<151> 2015-07-10
<160> 70
<170> PatentIn 3.5版
<210> 1
<211> 478
<212> PRT
<213> 蓝细菌
<400> 1
Met Phe Leu His Val Asp Arg Lys Gln Glu Leu His Met Pro Ile Asp
1 5 10 15
Ser Val Thr Ala Ala Leu Glu Ala Leu Asp His Gln Asp Ala Gly Val
20 25 30
Arg Tyr His Gly Ala Trp Trp Leu Gly Lys Asn Arg Ser Ala Glu Gly
35 40 45
Val Pro Arg Leu Val Glu Cys Leu Leu Asp Glu Arg Asp Lys Thr Cys
50 55 60
Thr Gly Gly Tyr Pro Leu Arg Arg Gln Ala Ala Arg Ser Leu Gly Met
65 70 75 80
Ile Lys Asp Ser Arg Cys Leu Pro Glu Leu Leu Lys Thr Leu Glu Thr
85 90 95
Asp Asp Val Gln Leu His Glu Ala Thr Leu Arg Ala Leu Ile Gln Ile
100 105 110
Lys Ser Asp Gln Cys Ser Ser Ser Leu Ile Asn Tyr Leu Asp Arg Asp
115 120 125
Ile Pro Asn Lys Pro Ile Glu Ala Leu Ile Glu Ala Leu Thr Glu Gln
130 135 140
Arg Met Trp Asp Val Ser Glu Lys Ile Gln Pro Phe Leu Asn Asp Lys
145 150 155 160
Ser Glu Arg Ile Ala Gly Ser Ala Ala Ala Phe Phe Tyr Ser Tyr Thr
165 170 175
Gly Glu Met Thr Tyr Leu Asn Lys Val Ile Ser Leu Leu Asp His Gln
180 185 190
Asn Arg Phe Ile Arg Gln Ser Ala Ala Phe Asp Leu Ala Arg Ile Gly
195 200 205
Thr Ile Lys Ala Thr Asp Pro Ile Leu Thr Ala Lys Ile Pro Asn Asn
210 215 220
Val Lys Met Phe Ala Ile Lys Ala Ile Leu Asn Lys Ser Leu Ser Arg
225 230 235 240
Ser Asn Gln Ala Asp Ser Ile Pro Asp Thr Asp Leu Ala Ser Ile His
245 250 255
Ser Ser Leu Phe Lys Ala Leu Asp Ser Leu Ala Arg Asp Asn Phe Ser
260 265 270
Gly Asn Leu Leu Ile Glu Gln Asp Asn Gln Ile Pro Glu Thr Tyr Pro
275 280 285
Gly Asp Gly Ser Thr Glu Ser Asp Leu Leu Ser Asn Ala Phe Asp Asn
290 295 300
Leu Arg Ser Pro Ser Leu Thr Ser Arg Lys Ser Gly Ile Lys Gln Leu
305 310 315 320
Val Arg Gly Ala Asn Arg Phe Lys Ile Asp Leu Leu Asp Leu Tyr Phe
325 330 335
Ser Glu Ser Asp Gln Asp Ile Thr Met Gly Leu Ile Lys Ala Met Ala
340 345 350
Glu Leu Lys Asn Pro His Tyr Ala Asn Ala Leu Ile Asp Ala Ile Gly
355 360 365
Val Glu Ile Gly Asn His Cys Gln Gly Asn Ile Arg Arg Val Ala Ala
370 375 380
Cys Ala Leu Gly Asp Ile Asn Trp Asn Ala Lys Ile Ser Ser Gln Ser
385 390 395 400
Leu His Ala Val Phe Asn Lys Leu Lys Trp Thr Leu His Ser Pro Glu
405 410 415
Asp Trp Gly Leu Arg Tyr Ser Ala Cys Leu Ala Leu Glu Gly Ile Gly
420 425 430
Asn Ala Asp Ser Ile Lys Leu Leu Asn Glu Ala Lys Ala Lys Glu Thr
435 440 445
Asp Pro Val Leu Ser Ala Arg Leu Asp Lys Ala Ile Leu Lys Ser Lys
450 455 460
Asn Lys Thr Ser Ile His His Ile Glu Asn Lys Lys Val Leu
465 470 475
<210> 2
<211> 235
<212> PRT
<213> 蓝细菌
<400> 2
Met Phe Asp Pro Phe Leu Glu Glu Leu Gln Thr Gly Ile Gln Ala Arg
1 5 10 15
Gly Gly Ile Ser Val Glu Val Pro Ala Gly Leu Glu His Asn Gln Ser
20 25 30
Gln Lys Gly Ser Ser Thr Ile Gln Ser Trp Leu Trp Gln Val Pro Gly
35 40 45
Phe Arg Arg Trp Arg Val Thr Arg Leu Asp Ala Gly Asp Ser Leu Gln
50 55 60
Val Leu Asn Ser Val Ala Tyr Pro Asp Phe Asp Leu Asp His Pro Leu
65 70 75 80
Met Gly Val Asp Leu Leu Trp Phe Gly Ala Arg Gln Lys Leu Val Ala
85 90 95
Val Leu Asp Phe Gln Pro Leu Val Gln Asp Lys Asp Tyr Leu Asp Arg
100 105 110
His Phe Asp Gly Leu Lys Asp Leu Asn Ala Arg Phe Pro Asp Leu Asn
115 120 125
Gly Glu Glu Thr Met Arg Ser Phe Asp Pro Asn Gln Tyr Phe Ser Ser
130 135 140
Trp Leu Leu Phe Cys Arg Gly Gly Ser Glu Glu Ala Asp Arg Ser Leu
145 150 155 160
Pro Lys Ala Phe Ser Ala Phe Leu Lys Ala Tyr Trp Gly Leu His Asp
165 170 175
Glu Ala Ser Lys Glu Pro Ser Ser Ile Ser Pro Gly Asp Val Glu Arg
180 185 190
Leu Gln Asn Ala Tyr Asp Val Tyr Ser Ala Glu Arg Asp Pro Ala His
195 200 205
Gly Leu Phe Thr Ser His Phe Gly Lys Glu Trp Ser Asp Arg Phe Leu
210 215 220
His Glu Phe Leu Phe Pro Ala Ser Gln Pro Ala
225 230 235
<210> 3
<211> 262
<212> PRT
<213> 蓝细菌
<400> 3
Met Ser Ile Asp Leu Arg Ala Ser Ser Leu Asp Pro Val Gln Ile Pro
1 5 10 15
Gly Trp Arg Trp Gln Pro Phe Leu Asp Glu Ala Ser Ala Ala Leu Lys
20 25 30
Pro Phe Asn Pro Ser Pro Tyr Pro Ile Ala Glu Thr Phe Leu Gln Lys
35 40 45
Glu Gly Ser Thr Gly Ser Lys Ala Lys Pro Val Pro Val Thr Thr Ala
50 55 60
Thr Trp Ala Cys Ser Thr Asp Lys Leu Arg Gln Val Arg Cys Ala Cys
65 70 75 80
Val Glu Ala Gly Met Ala Ala Ser Val Leu Asn Phe Val Ile Asn Pro
85 90 95
Ser Cys Arg Phe Asp Leu Pro Phe Phe Gly Ala Asp Leu Val Thr Leu
100 105 110
Pro Asn Gly His Leu Leu Ala Leu Asp Leu Gln Pro Val Asp Lys Ala
115 120 125
Asp Pro Asp His Thr Gln Pro Val Trp Glu Arg Leu Met Pro Leu Phe
130 135 140
Glu Arg Trp Gln Ala Glu Leu Pro Asp Gly Gly Pro Ile Pro Glu Glu
145 150 155 160
Ala Gln Pro Tyr Phe Ser Pro Ala Phe Leu Trp Thr Arg Ile Pro Leu
165 170 175
Gly Glu Glu Gly Asp Glu Leu Ile Glu Arg Val Ile Arg Pro Ala Phe
180 185 190
Ile Asp Tyr Leu Gln Leu Tyr Leu Asn Leu Val Ala Glu Ala Glu Pro
195 200 205
Val Ser Asp Asp Arg Ala Glu Leu Leu Leu Ser Gly Gln Lys Arg Tyr
210 215 220
Thr Ala Tyr Arg Ala Glu Lys Asp Pro Ala Arg Gly Met Leu Thr Arg
225 230 235 240
Phe Tyr Gly Ser Glu Trp Thr Glu Ser Tyr Ile His Gly Val Leu Phe
245 250 255
Asp Leu Glu Asp Ala Ala
260
<210> 4
<211> 708
<212> DNA
<213> 蓝细菌
<400> 4
atgtttgatc cgtttcttga ggaattacaa actggaattc aagcccgcgg tggcatatca 60
gttgaagttc cggccgggct ggaacacaat caatcccaga agggctcaag caccatccaa 120
agctggcttt ggcaggttcc aggttttcgt cgctggcgcg tcacccgact tgatgcaggt 180
gacagccttc aagttctgaa ttccgtcgca tatcccgatt tcgatttgga ccatcctttg 240
atgggtgttg atctgctctg gtttggcgca cgtcaaaagc tagttgcggt tcttgatttt 300
caaccactgg ttcaagacaa agactatctc gatcgtcatt ttgatggtct gaaagatctg 360
aatgctcgtt tcccggatct aaacggagaa gaaacgatgc gatctttcga tccgaatcaa 420
tacttctcat catggctact tttttgccgt ggaggttctg aagaggctga caggtcactg 480
ccaaaggcct tcagcgcctt tttgaaagcc tattggggtt tacacgatga ggcttccaag 540
gaaccatcct caatctcacc tggagatgtg gaacggcttc agaacgccta cgacgtgtac 600
agcgccgagc gtgatcctgc ccatggattg ttcaccagcc atttcggcaa ggagtggtct 660
gaccggttcc tgcacgaatt ccttttccct gccagtcagc ccgcatga 708
<210> 5
<211> 789
<212> DNA
<213> 蓝细菌
<400> 5
atgagcattg atctccgcgc gtcgagcctt gatcccgttc agattccggg gtggcgatgg 60
cagccctttc tcgatgaagc cagtgctgca ctcaagccgt tcaacccgtc tccctatccc 120
atagcggaaa cgtttctgca aaaggagggc agcaccggtt caaaagcgaa acccgttccg 180
gtgacaacgg cgacctgggc ctgttccaca gacaagttgc gtcaggtgcg ttgtgcctgc 240
gttgaagcgg gtatggctgc ttcggtgctc aattttgtga tcaacccgag ctgtcggttc 300
gacctgccgt tcttcggagc cgatctggtg acgcttccaa acggccattt gctcgctctg 360
gatcttcaac cggttgacaa ggctgatccc gatcacaccc aacccgtgtg ggagcgactg 420
atgccgttgt tcgagcgctg gcaagccgaa ctccccgatg gaggccccat cccagaagaa 480
gcccaaccct atttctcacc ggcgtttctc tggacccgca tcccgcttgg ggaggagggg 540
gatgaactga ttgaacgcgt gatccgcccg gccttcatcg actatctgca gctttacctc 600
aacctcgtgg ctgaagcgga acccgtgtct gacgaccgtg cggaattgct cctttcaggc 660
caaaagcgct acaccgcgta tcgcgccgag aaggatccag cccgcggcat gttgacgcgg 720
ttctacggga gcgagtggac agagtcgtac atccatggcg tgctgttcga tctcgaggac 780
gccgcttaa 789
<210> 6
<211> 4485
<212> DNA
<213> 人工序列
<220>
<223> 用于PEB生物合成的途径;包含或编码lacI、pebA、pebB和链霉素抗性盒
<400> 6
catcgcttgc aattcgcgct aacttacatt aattgcgttg cgctcattga ccactctcca 60
aacggctcac ttgccgtgcc agctgcatga gactatcggc cagagcacgc ggggaggccg 120
tttgcgtgtt tggcgccaag gtggttttgc gtttcaccag cgacacgggc agcagctgat 180
ttcctttaac tgcctgccct tggctcagtt gcaacagtcg atccacggac gtctgaccca 240
acaagcggaa atcttgcttg atcgtggtca gaggcgggat ataacaactc gaatcttcag 300
tatcatcata gcccacgaca ctaatatctg cgccgacgcg gaggccgctc tccgtgatcg 360
cacgcatcgc gcccaaagcc atctggtcat tcgcgaccag catggccgta ggcacgatgc 420
cttcattcag catttgcatt gtttgctgga aacccgacat agcgctccaa tcgccctcgc 480
gctcggcgat tggttggatc tgattgcggg tgaggtattt atgccagccc gcgagtcgca 540
ggcgagcact cacgctggag agcgggcctg ccaagagagc aatctgctga tggcccagcg 600
cgaccagatg ctccacaccc aagcgtgtac cgtcctcgtg cgagaagatg atgctattaa 660
tgggggtttg atcggagacg tccaggaaca acgcgggaac gttcgtgcag gccgcttcaa 720
cagcgatagc atcttggtca tccagcgggt agttgataat caggcccgac acacgctgag 780
ccaggaggtt gtggaccgca gctttgcaag cttccacgcc actccgttca accatggaga 840
cgaccacgct tgcccccagt tgatccgcac gcgatttaat cgcggcaaca atttgactcg 900
gggcgtgcag cgcgagagag ctcgtggcaa ccccgatcaa caagctctgt tttccggcca 960
gctgctgcgc gacgcggttg gggatataat tcagctcggc catcgcagcc tcgacttttt 1020
cacgcgtctt tgccgacaca tgggaggctt gattaaccac gcgactgaca gtttggtagc 1080
tcacacctgc gtattctgca acatcataca gcgtgactgg cttcacattg accatcctga 1140
attgactctc ttccgggcgc tatcatgcca taccgcgaaa ggttttgcac cattcgatgg 1200
tgtcaacgta cgactgcacg gtgcaccaat gcttctggcg tcaggcagcc atcggaagct 1260
gtggtatggc tgtgcaggtc gtaaatcact gcataattcg tgtcgctcaa ggcgcactcc 1320
cgttctggat aatgtttttt gcgccgacat cataacggtt ctggcaaata ttctgaaatg 1380
agctgttgac aattaatcat ccggctcgta taatgtgtgg aattgtgagc ggataacaat 1440
tgtcgacagg aaacaggaat tacatatgtt tgatccgttt cttgaggaat tacaaactgg 1500
aattcaagcc cgcggtggca tatcagttga agttccggcc gggctggaac acaatcaatc 1560
ccagaagggc tcaagcacca tccaaagctg gctttggcag gttccaggtt ttcgtcgctg 1620
gcgcgtcacc cgacttgatg caggtgacag ccttcaagtt ctgaattccg tcgcatatcc 1680
cgatttcgat ttggaccatc ctttgatggg tgttgatctg ctctggtttg gcgcacgtca 1740
aaagctagtt gcggttcttg attttcaacc actggttcaa gacaaagact atctcgatcg 1800
tcattttgat ggtctgaaag atctgaatgc tcgtttcccg gatctaaacg gagaagaaac 1860
gatgcgatct ttcgatccga atcaatactt ctcatcatgg ctactttttt gccgtggagg 1920
ttctgaagag gctgacaggt cactgccaaa ggccttcagc gcctttttga aagcctattg 1980
gggtttacac gatgaggctt ccaaggaacc atcctcaatc tcacctggag atgtggaacg 2040
gcttcagaac gcctacgacg tgtacagcgc cgagcgtgat cctgcccatg gattgttcac 2100
cagccatttc ggcaaggagt ggtctgaccg gttcctgcac gaattccttt tccctgccag 2160
tcagcccgca tgagcattga tctccgcgcg tcgagccttg atcccgttca gattccgggg 2220
tggcgatggc agccctttct cgatgaagcc agtgctgcac tcaagccgtt caacccgtct 2280
ccctatccca tagcggaaac gtttctgcaa aaggagggca gcaccggttc aaaagcgaaa 2340
cccgttccgg tgacaacggc gacctgggcc tgttccacag acaagttgcg tcaggtgcgt 2400
tgtgcctgcg ttgaagcggg tatggctgct tcggtgctca attttgtgat caacccgagc 2460
tgtcggttcg acctgccgtt cttcggagcc gatctggtga cgcttccaaa cggccatttg 2520
ctcgctctgg atcttcaacc ggttgacaag gctgatcccg atcacaccca acccgtgtgg 2580
gagcgactga tgccgttgtt cgagcgctgg caagccgaac tccccgatgg aggccccatc 2640
ccagaagaag cccaacccta tttctcaccg gcgtttctct ggacccgcat cccgcttggg 2700
gaggaggggg atgaactgat tgaacgcgtg atccgcccgg ccttcatcga ctatctgcag 2760
ctttacctca acctcgtggc tgaagcggaa cccgtgtctg acgaccgtgc ggaattgctc 2820
ctttcaggcc aaaagcgcta caccgcgtat cgcgccgaga aggatccagc ccgcggcatg 2880
ttgacgcggt tctacgggag cgagtggaca gagtcgtaca tccatggcgt gctgttcgat 2940
ctcgaggacg ccgcttaaac tagtcatcga gctagcaagc ttggccggat ccggccggat 3000
ccggagtttg tagaaacgca aaaaggccat ccgtcaggat ggccttctgc ttaatttgat 3060
gcctggcagt ttatggcggg cgtcctgccc gccaccctcc gggccgttgc ttcgcaacgt 3120
tcaaatccgc tcccggcgga tttgtcctac tcaggagagc gttcaccgac aaacaacaga 3180
taaaacgaaa ggcccagtct ttcgactgag cctttcgttt tatttgatgc ctggcagttc 3240
cctactctcg gtacccgtcg gcttgaacga attgttagac attatttgcc gactaccttg 3300
gtgatctcgc ctttcacgta gtggacaaat tcttccaact gatctgcgcg cgaggccaag 3360
cgatcttctt cttgtccaag ataagcctgt ctagcttcaa gtatgacggg ctgatactgg 3420
gccggcaggc gctccattgc ccagtcggca gcgacatcct tcggcgcgat tttgccggtt 3480
actgcgctgt accaaatgcg ggacaacgta agcactacat ttcgctcatc gccagcccag 3540
tcgggcggcg agttccatag cgttaaggtt tcatttagcg cctcaaatag atcctgttca 3600
ggaaccggat caaagagttc ctccgccgct ggacctacca aggcaacgct atgttctctt 3660
gcttttgtca gcaagatagc cagatcaatg tcgatcgtgg ctggctcgaa gatacctgca 3720
agaatgtcat tgcgctgcca ttctccaaat tgcagttcgc gcttagctgg ataacgccac 3780
ggaatgatgt cgtcgtgcac aacaatggtg acttctacag cgcggagaat ctcgctctct 3840
ccaggggaag ccgaagtttc caaaaggtcg ttgatcaaag ctcgccgcgt tgtttcatca 3900
agccttacgg tcaccgtaac cagcaaatca atatcactgt gtggcttcag gccgccatcc 3960
actgcggagc cgtacaaatg tacggccagc aacgtcggtt cgagatggcg ctcgatgacg 4020
ccaactacct ctgatagttg agtcgatact tcggcgatca ccgcttccct catgatgttt 4080
aactttgttt tagggcgact gccctgctgc gtaacatcgt tgctgctcca taacatcaaa 4140
catcgaccca cggcgtaacg cgcttgctgc ttggatgccc gaggcataga ctgtacccca 4200
aaaaaacagt cataacaagc catgaaaacc gccactgcgc cgttaccacc gctgcgttcg 4260
gtcaaggttc tggaccagtt gcgtgagcgc atacgctact tgcattacag cttacgaacc 4320
gaacaggctt atgtccactg ggttcgtgcc ttcatccgtt tccacggtgt gcgtcacccg 4380
gcaaccttgg gcagcagcga agtcgaggca tttctgtcct ggctggcgaa cgagcgcaag 4440
gtttcgaatt cacatacgcg gccgcctggg ccttgagctc gaatt 4485
<210> 7
<211> 1437
<212> DNA
<213> 人工序列
<220>
<223> 具有终止密码子的RpcG
<400> 7
atgtttctcc atgtcgacag gaaacaggaa ttacatatgc caattgactc agttacagcc 60
gctcttgaag ccctcgacca ccaagatgcg ggtgttcggt atcacggtgc ttggtggctc 120
gggaaaaaca ggtcggctga gggagtacca cgattggtgg aatgccttct agacgaaagg 180
gacaaaacat gtacaggcgg atacccccta agaaggcaag cagcaagatc actgggaatg 240
atcaaagact cacgctgttt accagagctt cttaaaacac tagaaacaga tgacgtgcaa 300
ttgcatgaag caacacttag agccctaatt caaatcaaga gtgatcaatg ctcaagctca 360
ctcattaact accttgaccg agatattccc aacaaaccaa tagaagcgct tatagaagcc 420
ttaacagagc aaagaatgtg ggatgtttca gaaaagatcc aaccctttct taacgacaaa 480
tcagaaagga tcgctggctc tgcagcagct tttttctaca gctacaccgg tgagatgacc 540
tatttaaaca aagttatctc acttcttgat caccagaatc gcttcatcag gcaatccgct 600
gcattcgacc tagcccgtat cggaacaatc aaagcaacag atccaatcct gactgccaag 660
atccccaaca acgtcaagat gtttgccata aaagccatac tcaataaatc gctcagccga 720
agcaatcaag cagattctat tccagatacc gacctcgcat caattcattc ctccctcttc 780
aaagcacttg acagtctcgc cagagacaac ttttcgggga acctattgat tgagcaagac 840
aaccaaattc cagaaaccta tccaggagac ggctcaacag agagcgatct actttcaaat 900
gcattcgaca acctaaggtc accatccttg acgagcagaa aatcaggcat aaaacaactc 960
gtccgtggtg ctaatcgttt caaaatcgat cttcttgatc tgtacttctc agaatcagat 1020
caagacatca caatggggct gatcaaggcc atggctgaac tcaaaaatcc ccattacgca 1080
aacgcactta ttgatgctat tggggtcgaa attggcaatc attgccaagg aaacattcga 1140
cgcgtcgcag cgtgcgccct tggtgacatc aattggaacg cgaagatttc gtcgcaatca 1200
ctgcatgccg ttttcaacaa actcaaatgg acacttcatt cacctgaaga ctggggtttg 1260
cgctacagcg catgcttggc gctggaagga attggcaatg ccgattcgat taaactctta 1320
aatgaagcta aagcaaaaga aacagatcca gtcctctctg cacgccttga caaggcaata 1380
ctaaaatcaa aaaataagac ttctatccat cacatcgaaa acaaaaaagt tctctaa 1437
<210> 8
<211> 4182
<212> DNA
<213> 人工序列
<220>
<223> 包含或编码lacI、rpcG和庆大霉素抗性盒
<400> 8
ttgaccactc tccaaacggc tcacttgccg tgccagctgc atgagactat cggccagagc 60
acgcggggag gccgtttgcg tgtttggcgc caaggtggtt ttgcgtttca ccagcgacac 120
gggcagcagc tgatttcctt taactgcctg cccttggctc agttgcaaca gtcgatccac 180
ggacgtctga cccaacaagc ggaaatcttg cttgatcgtg gtcagaggcg ggatataaca 240
actcgaatct tcagtatcat catagcccac gacactaata tctgcgccga cgcggaggcc 300
gctctccgtg atcgcacgca tcgcgcccaa agccatctgg tcattcgcga ccagcatggc 360
cgtaggcacg atgccttcat tcagcatttg cattgtttgc tggaaacccg acatagcgct 420
ccaatcgccc tcgcgctcgg cgattggttg gatctgattg cgggtgaggt atttatgcca 480
gcccgcgagt cgcaggcgag cactcacgct ggagagcggg cctgccaaga gagcaatctg 540
ctgatggccc agcgcgacca gatgctccac acccaagcgt gtaccgtcct cgtgcgagaa 600
gatgatgcta ttaatggggg tttgatcgga gacgtccagg aacaacgcgg gaacgttcgt 660
gcaggccgct tcaacagcga tagcatcttg gtcatccagc gggtagttga taatcaggcc 720
cgacacacgc tgagccagga ggttgtggac cgcagctttg caagcttcca cgccactccg 780
ttcaaccatg gagacgacca cgcttgcccc cagttgatcc gcacgcgatt taatcgcggc 840
aacaatttga ctcggggcgt gcagcgcgag agagctcgtg gcaaccccga tcaacaagct 900
ctgttttccg gccagctgct gcgcgacgcg gttggggata taattcagct cggccatcgc 960
agcctcgact ttttcacgcg tctttgccga cacatgggag gcttgattaa ccacgcgact 1020
gacagtttgg tagctcacac ctgcgtattc tgcaacatca tacagcgtga ctggcttcac 1080
attgaccatc ctgaattgac tctcttccgg gcgctatcat gccataccgc gaaaggtttt 1140
gcaccattcg atggtgtcaa cgtacgactg cacggtgcac caatgcttct ggcgtcaggc 1200
agccatcgga agctgtggta tggctgtgca ggtcgtaaat cactgcataa ttcgtgtcgc 1260
tcaaggcgca ctcccgttct ggataatgtt ttttgcgccg acatcataac ggttctggca 1320
aatattctga aatgagctgt tgacaattaa tcatccggct cgtataatgt gtggaattgt 1380
gagcggataa caattgtcga caggaaacag gaattacata tgccaattga ctcagttaca 1440
gccgctcttg aagccctcga ccaccaagat gcgggtgttc ggtatcacgg tgcttggtgg 1500
ctcgggaaaa acaggtcggc tgagggagta ccacgattgg tggaatgcct tctagacgaa 1560
agggacaaaa catgtacagg cggatacccc ctaagaaggc aagcagcaag atcactggga 1620
atgatcaaag actcacgctg tttaccagag cttcttaaaa cactagaaac agatgacgtg 1680
caattgcatg aagcaacact tagagcccta attcaaatca agagtgatca atgctcaagc 1740
tcactcatta actaccttga ccgagatatt cccaacaaac caatagaagc gcttatagaa 1800
gccttaacag agcaaagaat gtgggatgtt tcagaaaaga tccaaccctt tcttaacgac 1860
aaatcagaaa ggatcgctgg ctctgcagca gcttttttct acagctacac cggtgagatg 1920
acctatttaa acaaagttat ctcacttctt gatcaccaga atcgcttcat caggcaatcc 1980
gctgcattcg acctagcccg tatcggaaca atcaaagcaa cagatccaat cctgactgcc 2040
aagatcccca acaacgtcaa gatgtttgcc ataaaagcca tactcaataa atcgctcagc 2100
cgaagcaatc aagcagattc tattccagat accgacctcg catcaattca ttcctccctc 2160
ttcaaagcac ttgacagtct cgccagagac aacttttcgg ggaacctatt gattgagcaa 2220
gacaaccaaa ttccagaaac ctatccagga gacggctcaa cagagagcga tctactttca 2280
aatgcattcg acaacctaag gtcaccatcc ttgacgagca gaaaatcagg cataaaacaa 2340
ctcgtccgtg gtgctaatcg tttcaaaatc gatcttcttg atctgtactt ctcagaatca 2400
gatcaagaca tcacaatggg gctgatcaag gccatggctg aactcaaaaa tccccattac 2460
gcaaacgcac ttattgatgc tattggggtc gaaattggca atcattgcca aggaaacatt 2520
cgacgcgtcg cagcgtgcgc ccttggtgac atcaattgga acgcgaagat ttcgtcgcaa 2580
tcactgcatg ccgttttcaa caaactcaaa tggacacttc attcacctga agactggggt 2640
ttgcgctaca gcgcatgctt ggcgctggaa ggaattggca atgccgattc gattaaactc 2700
ttaaatgaag ctaaagcaaa agaaacagat ccagtcctct ctgcacgcct tgacaaggca 2760
atactaaaat caaaaaataa gacttctatc catcacatcg aaaacaaaaa agttctctaa 2820
aactaatgat aaaagtattg ttcgtctgcc tcggactagt catcgagcta gcaagcttgg 2880
ccggatccgg ccggatccgg agtttgtaga aacgcaaaaa ggccatccgt caggatggcc 2940
ttctgcttaa tttgatgcct ggcagtttat ggcgggcgtc ctgcccgcca ccctccgggc 3000
cgttgcttcg caacgttcaa atccgctccc ggcggatttg tcctactcag gagagcgttc 3060
accgacaaac aacagataaa acgaaaggcc cagtctttcg actgagcctt tcgttttatt 3120
tgatgcctgg cagttcccta ctctcggtac ccgtcggctt gaacgaattg tcgatctcgg 3180
cttgaacgaa ttgttaggtg gcggtacttg ggtcgatatc aaagtgcatc acttcttccc 3240
gtatgcccaa ctttgtatag agagccactg cgggatcgtc accgtaatct gcttgcacgt 3300
agatcacata agcaccaagc gcgttggcct catgcttgag gagattgatg agcgcggtgg 3360
caatgccctg cctccggtgc tcgccggaga ctgcgagatc atagatatag atttcactac 3420
gcggctgctc aaacttgggc agaacgtaag ccgcgagagc gccaacaacc gcttcttggt 3480
cgaaggcagc aagcgcgatg aatgtcttac tacggagcaa gttcccgagg taatcggagt 3540
ccggctgatg ttgggagtag gtggctacgt ctccgaactc acgaccgaaa agatcaagag 3600
cagcccgcat ggatttgact tggtcagggc cgagcctaca tgtgcgaatg atgcccatac 3660
ttgagccacc taactttgtt ttagggcgac tgccctgctg cgtaacatcg ttgctgctgc 3720
gtaacatcgt tgctgctcca taacatcaaa catcgaccca cggcgtaacg cgcttgctgc 3780
ttggatgccc gaggcataga ctgtacaaaa aaacagtcat aacaagccat gaaaaccgcc 3840
actgcgccgt taccaccgct gcgttcggtc aaggttctgg accagttgcg tgagcgcata 3900
cgctacttgc attacagttt acgaaccgaa caggcttatg tcaactgggt tcgtgccttc 3960
atccgtttcc acggtgtgcg tcacccggca accttgggca gcagcgaagt cgaggcattt 4020
ctgtcctggc tggcgaacga gcgcaaggtt tcggtctcca cgcatcgtca ggcattggcg 4080
gccttgctgt tcttctacgg caaggtgctg tgcacggatc tgccctggct tcaggagatc 4140
ggaattcaca tacgcggccg cctgggcctt gagctcgaat tt 4182
<210> 9
<211> 4499
<212> DNA
<213> 人工序列
<220>
<223> 包含或编码paraup1、pebA、pebB和潮霉素抗性盒
<400> 9
aagcttgcat gcctgcaggt ggcgcgccat cgctaggaca gcttaaccgc cacgtcattg 60
actttttctt cacaacctgc gcgaaattca gagggcgatg caccagtgca cttcttgaaa 120
actcgcgaga aatacaactg gtcatcaaac ccgacattgc ggccaacagt agcgatgggc 180
atccgcgtgg tggacaagag cagcttggcc tgcgaaatcc gttgatcttc gcgccaagac 240
aggaccgaga taccgagctg ttggcgaaac aggtggctca agcgcgacgg gctgagacaa 300
acatgttgcg cgacggacgc gatgtcgaaa ttgctgtcag ccagatgatc gctgatgtac 360
tggcatgctt cgcggactcg gttatccatc ggcgggtgga gcgactcgtt gatggcttcc 420
atccgccgca acaggagttg ttccaacaga ttgatagcga ggagttcaga gtaacggccc 480
tcgccttggc cggcgttaat aatctggcca aacagatcac taaagtgtgg ttgatgggct 540
tcatcggggc gaaaaaaacc cgtattggca aaaatagaag gccagttcaa ccattcgtgc 600
cagtaagccc gtggccgaaa gtaaacccac tggtgatacc attcgcgggc ctcgggatgg 660
cgaccgtagt gatgaatttc accaggtggg aacagcaaaa tgtcaccggg gcgacaaaca 720
aactcccgcc cttggttttt gacaacgccc tgaccacgaa tcgtcaagtt caaaatatag 780
cccttcatac ccaaaggtcg atcgatgaag aaatccaaat acccattggc ttcgataggt 840
gtcaggccgg ccacgagatg ggcattaaaa ctataacccg gcaacaaagg atcattctga 900
gcttcggcca tacttttcat actcccgcca ttcagagaag aaaccaattg tccatattgc 960
atcagacatt gccgtcactg cgtcttttac tggctcttct cgctaaccaa accggtaacc 1020
ccgcttatta aaagcattct gtaacaaagc gggaccaaag ccatgacaaa aacgcgtaac 1080
aaaagtgtct ataatcacgg cagaaaagtc cacattgatt atttgcacgg cgtcacactt 1140
tgctatgcca tagcattttt atccataaga ttagcggatc ctacctgacg ctttttatcg 1200
caactcgtat aatgtttctc catgtcgaca ggaaacagga attacatatg tttgatccgt 1260
ttcttgagga attacaaact ggaattcaag cccgcggtgg catatcagtt gaagttccgg 1320
ccgggctgga acacaatcaa tcccagaagg gctcaagcac catccaaagc tggctttggc 1380
aggttccagg ttttcgtcgc tggcgcgtca cccgacttga tgcaggtgac agccttcaag 1440
ttctgaattc cgtcgcatat cccgatttcg atttggacca tcctttgatg ggtgttgatc 1500
tgctctggtt tggcgcacgt caaaagctag ttgcggttct tgattttcaa ccactggttc 1560
aagacaaaga ctatctcgat cgtcattttg atggtctgaa agatctgaat gctcgtttcc 1620
cggatctaaa cggagaagaa acgatgcgat ctttcgatcc gaatcaatac ttctcatcat 1680
ggctactttt ttgccgtgga ggttctgaag aggctgacag gtcactgcca aaggccttca 1740
gcgccttttt gaaagcctat tggggtttac acgatgaggc ttccaaggaa ccatcctcaa 1800
tctcacctgg agatgtggaa cggcttcaga acgcctacga cgtgtacagc gccgagcgtg 1860
atcctgccca tggattgttc accagccatt tcggcaagga gtggtctgac cggttcctgc 1920
acgaattcct tttccctgcc agtcagcccg catgagcatt gatctccgcg cgtcgagcct 1980
tgatcccgtt cagattccgg ggtggcgatg gcagcccttt ctcgatgaag ccagtgctgc 2040
actcaagccg ttcaacccgt ctccctatcc catagcggaa acgtttctgc aaaaggaggg 2100
cagcaccggt tcaaaagcga aacccgttcc ggtgacaacg gcgacctggg cctgttccac 2160
agacaagttg cgtcaggtgc gttgtgcctg cgttgaagcg ggtatggctg cttcggtgct 2220
caattttgtg atcaacccga gctgtcggtt cgacctgccg ttcttcggag ccgatctggt 2280
gacgcttcca aacggccatt tgctcgctct ggatcttcaa ccggttgaca aggctgatcc 2340
cgatcacacc caacccgtgt gggagcgact gatgccgttg ttcgagcgct ggcaagccga 2400
actccccgat ggaggcccca tcccagaaga agcccaaccc tatttctcac cggcgtttct 2460
ctggacccgc atcccgcttg gggaggaggg ggatgaactg attgaacgcg tgatccgccc 2520
ggccttcatc gactatctgc agctttacct caacctcgtg gctgaagcgg aacccgtgtc 2580
tgacgaccgt gcggaattgc tcctttcagg ccaaaagcgc tacaccgcgt atcgcgccga 2640
gaaggatcca gcccgcggca tgttgacgcg gttctacggg agcgagtgga cagagtcgta 2700
catccatggc gtgctgttcg atctcgagga cgccgcttaa actagtcatc gagctagcaa 2760
gcttggccgg atccggccgg atccggagtt tgtagaaacg caaaaaggcc atccgtcagg 2820
atggccttct gcttaatttg atgcctggca gtttatggcg ggcgtcctgc ccgccaccct 2880
ccgggccgtt gcttcgcaac gttcaaatcc gctcccggcg gatttgtcct actcaggaga 2940
gcgttcaccg acaaacaaca gataaaacga aaggcccagt ctttcgactg agcctttcgt 3000
tttatttgat gcctggcagt tccctactct cggtacccgt cggcttgaac gaattgttag 3060
acactattcc tttgccctcg gacgagtgct ggggcgtcgg tttccactat cggcgagtac 3120
ttctacacag ccatcggtcc agacggccgc gcttctgcgg gcgatttgtg tacgcccgac 3180
agtcccggct ccggatcgga cgattgcgtc gcatcgaccc tgcgcccaag ctgcatcatc 3240
gaaattgccg tcaaccaagc tctgatagag ttggtcaaga ccaatgcgga gcatatacgc 3300
ccggagccgc ggcgatcctg caagctccgg atgcctccgc tcgaagtagc gcgtctgctg 3360
ctccatacaa gccaaccacg gcctccagaa gaagatgttg gcgacctcgt attgggaatc 3420
cccgaacatc gcctcgctcc agtcaatgac cgctgttatg cggccattgt ccgtcaggac 3480
attgttggag ccgaaatccg cgtgcacgag gtgccggact tcggggcagt cctcggccca 3540
aagcatcagc tcatcgagag cctgcgcgac ggacgcactg acggtgtcgt ccatcacagt 3600
ttgccagtga tacacatggg gatcagcaat cgcgcaaatg aaatcacgcc atgtagtgta 3660
ttgaccgatt ccttgcggtc cgaatgggcc gaacccgctc gtctggctaa gatcggccgc 3720
agcgatcgca tccatggcct ccgcgaccgg ctgcagaaca gcgggcagtt cggtttcagg 3780
caggtcttgc aacgtgacac cctgtgcacg gcgggagatg caataggtca ggctctcgct 3840
gaattcccca atgtcaagca cttccggaat cgggagcgcg gccgatgcaa agtgccgata 3900
aacataacga tctttgtaga aaccatcggc gcagctattt acccgcagga catatccacg 3960
ccctcctaca tcgaagctga aagcacgaga ttcttcgccc tccgagagct gcatcaggtc 4020
ggagacgctg tcgaactttt cgatcagaaa cttctcgaca gacgtcgcgg tgagttcagg 4080
ctttttcatt gtttaacttt gttttagggc gactgccctg ctgcgtaaca tcgttgctgc 4140
tccataacat caaacatcga cccacggcgt aacgcgcttg ctgcttggat gcccgaggca 4200
tagactgtac cccaaaaaaa cagtcataac aagccatgaa aaccgccact gcgccgttac 4260
caccgctgcg ttcggtcaag gttctggacc agttgcgtga gcgcatacgc tacttgcatt 4320
acagcttacg aaccgaacag gcttatgtcc actgggttcg tgccttcatc cgtttccacg 4380
gtgtgcgtca cccggcaacc ttgggcagca gcgaagtcga ggcatttctg tcctggctgg 4440
cgaacgagcg caaggtttcg aattcacata cgcggccgcc tgggccttga gctcgaatt 4499
<210> 10
<211> 822
<212> DNA
<213> 蓝细菌
<400> 10
atgagtgaag cggggacgcc agctgaggcg ctgacctacg aacaagcgat cgcgaatctc 60
cgacagacag cagatacggg cgatcgctac tacgcagctt ggtggctggg tcggtttcgg 120
atgaagcagc cagaggcgat cgcgctgttg attgaagcct tagatgatag cctcgatcgc 180
gcacctgatg gcggctatcc cctacggcgc aatgccgcac gcgcattggg aaaactggaa 240
agtcctgagg cgatcgcacc gttgattgcc tgcttgcagt gcgaggacta ctacgttcgc 300
gaggctgcaa cccagtcctt aggtgagttg caagccacag ttgcggttcc agcgttattg 360
aaactgttag agggcggacc tgaggcgatc gccgcgattc cgggtaaacc ccatctgact 420
cagccagcgg atgcggtgat ggaaaccctg ggacaactgc gagcaacggt tgctgtccct 480
gtggtgcaag cgtttctgga gcatccgatc gataaaattc gcctagcagc cgcacgatcg 540
ctctatcagc tcaccggcga cgatcactat gctgagcggg ttgttcaagg tttgagtgac 600
ccgaaattac agcgccggcg gtcggccctg atggatttag gggcgatcgg ctatttgccc 660
gctgcaccgc aaattgccca gacgcttgcc gagaatagtc tcaaactgat ctcgctcaaa 720
gggctgctcg atactcatct gcggcaacag acccccgagg cgatcgcaga gttggatgag 780
tcggcaatcg cgctgatgga tttgatggat ggtttgctgt ag 822
<210> 11
<211> 926
<212> DNA
<213> 人工序列
<220>
<223> 替换缺失的SEQ ID NO: 10的序列;包含卡那霉素抗性盒
<400> 11
ttagaaaaac tcatcgagca tcaaatgaaa ctgcaattta ttcatatcag gattatcaat 60
accatatttt tgaaaaagcc gtttctgtaa tgaaggagaa aactcaccga ggcagttcca 120
taggatggca agatcctggt atcggtctgc gattccgact cgtccaacat caatacaacc 180
tattaatttc ccctcgtcaa aaataaggtt atcaagtgag aaatcaccat gagtgacgac 240
tgaatccggt gagaatggca aaagcttatg catttctttc cagacttgtt caacaggcca 300
gccattacgc tcgtcatcaa aatcactcgc atcaaccaaa ccgttattca ttcgtgattg 360
cgcctgagcg agacgaaata cgcgatcgct gttaaaagga caattacaaa caggaatcga 420
atgcaaccgg cgcaggaaca ctgccagcgc atcaacaata ttttcacctg aatcaggata 480
ttcttctaat acctggaatg ctgttttccc ggggatcgca gtggtgagta accatgcatc 540
atcaggagta cggataaaat gcttgatggt cggaagaggc ataaattccg tcagccagtt 600
tagtctgacc atctcatctg taacatcatt ggcaacgcta cctttgccat gtttcagaaa 660
caactctggc gcatcgggct tcccatacaa tcgatagatt gtcgcacctg attgcccgac 720
attatcgcga gcccatttat acccatataa atcagcatcc atgttggaat ttaatcgcgg 780
cctcgagcaa gacgtttccc gttgaatatg gctcataaca ccccttgtat tactgtttat 840
gtaagcagac agttttattg ttcatgatga tatattttta tcttgtgcaa tgtaacatca 900
gagattttga gacacaacgt ggcttt 926
<210> 12
<211> 6
<212> PRT
<213> 人工序列
<220>
<223> 藻蓝蛋白中PCB附接位点的共有氨基酸序列
<220>
<221> X
<222> (1)..(1)
<223> Xaa是Ala或Ser
<220>
<221> X
<222> (2)..(2)
<223> Xaa是Lys或Ala
<220>
<221> X
<222> (4)..(4)
<223> Xaa是Ile或Leu或Ala
<400> 12
Xaa Xaa Cys Xaa Arg Asp
1 5
<210> 13
<211> 6
<212> PRT
<213> 人工序列
<220>
<223> 共有PUB附接位点
<220>
<221> X
<222> (1)..(1)
<223> Xaa是Ala或Ser
<220>
<221> X
<222> (2)..(2)
<223> Xaa是Lys或Ala
<400> 13
Xaa Xaa Cys Ser Arg Asp
1 5
<210> 14
<211> 327
<212> DNA
<213> 蓝细菌
<400> 14
tgagaaaaag tgtaaacaaa tattaagaaa aagatcagaa aaatttaaca acacgtaata 60
aaaaaatgcg tcactacggg ttataaattt acatgaaagg ttaaaacact tttctgagac 120
gattttgata aaaaagttgt caaaaaatta agtttcttta caaatgctta acaaaaactt 180
ggttttaagc acaaaataag agagactaat ttgcagaagt tttacaagga aatcttgaag 240
aaaaagatct aagtaaaacg actctgttta accaaaattt aacaaattta acaaaacaaa 300
ctaaatctat taggagatta actaagc 327
<210> 15
<211> 409
<212> DNA
<213> 蓝细菌
<400> 15
cttgaaaaag ttgaggtatt aatagagctt gataaatgat aataaaaaca gatttagctc 60
ttattttaag ggaaaaagaa ataaataaaa tattagtaaa tatcaaaaat atcagccttt 120
caaaaataat ttgactcttt tcaaaaaaaa atgttatctt taaggtatgc tttaaacctt 180
aaatacttct attggtaaca ctgttctcaa tcttatttca gattttccca ttgagcataa 240
ataaaatatt aagcagaagt agaaaaggtt gatattagca ataataaaaa ttaacaataa 300
aatgtgaaaa cagattacta ctgattattt attgccatga gctaattagt aataatttgt 360
cttttttgat cgaaaaatga aattttttaa gcggaggaac tgaaaatta 409
<210> 16
<211> 205
<212> DNA
<213> 蓝细菌
<400> 16
tagagtatga taaaatgaca aggaaaggat tattttctct tgtttaaatt ctcaagattc 60
ttatgcttat ttattttatg taagtgtctc ttttccttga aatagaaaga aaaaagtggc 120
taattttgag aaaagctaac aacgctttgg ttaactaaaa atcaaaagtg agattactga 180
tcgcttaaga aatggagtat tgatt 205
<210> 17
<211> 338
<212> DNA
<213> 蓝细菌
<400> 17
gcagttagat aaataagtaa tgagcgggag aaataggggc aaatggccat tcgcccctac 60
agggaggtgg caggtgttag ggtgtttagg ggatgaggtg atgagggtag agggagataa 120
ggtgtcgggt ttcagatttc aggttttaga agaaagtaac gagtaattat caactattca 180
ctattcacta ttgcctgttg cccttctctc cttgaaatat aaaaaaatgt aaaaatatca 240
ttaagaaaag taacaaaata aacagaaagg ttgacaaagt tgacgcttta atatccgtat 300
gttagcttta taacaacgaa atcaacggag gagtgaaa 338
<210> 18
<211> 411
<212> DNA
<213> 蓝细菌
<400> 18
tatttatata taaactcgaa taaaattatc aatataaagt caaactatat ctatcctatt 60
ttaactgcta ttggtaagtc ccttaattag tgttggggtg aatagatttt aaaagggcaa 120
accccccttt atcctccctc gagagggggg agggcaaaag gcaaggggca agggaaaaat 180
taagaattaa gaattaaaaa ctccgaacac ctgtaggggc gaatagccat tcgcttcccc 240
tcatcccccc atctccccaa caccctaagc ccctactcgt tactcattta tttacatcat 300
ttatttacat cattaagaaa agtaacaaat tttgacaagt agtcttttga caggaaaaag 360
caaattctcg aagatgaaaa caatagaaaa aaattcaatc ttacagtaac g 411
<210> 19
<211> 214
<212> DNA
<213> 蓝细菌
<400> 19
agagttatat ttacatagtg tgtgcgagta agggcaactt ttgtaggtag atgaataaac 60
ctcaaattac tcatcttaaa agacgatatt tttaatctat tcttctgtaa taaaatactt 120
ctttcgatag agatatttaa tacttttgag agatgaaaat aatttcaata attgtcatga 180
tagagagtaa gtgcaaataa gaaaaaattg attt 214
<210> 20
<211> 211
<212> DNA
<213> 蓝细菌
<400> 20
gtgatatttg gtttattcta tattttcctt aagtaaaaat tcagtcatga gggaaacttt 60
tgttaaaatt tgctttaaat taataggaag atcattaaga aaatcttaaa aagattgagt 120
ttttagatcg aaattattga agaaaaatta acaggggttc tgctcaaaat tttattaaat 180
tactctactg tagtaaagga gaaattttat t 211
<210> 21
<211> 380
<212> DNA
<213> 蓝细菌
<400> 21
ataaccaatg ggacttgaat tttagatcca tttatttaat tctatttttg ttacatttct 60
ttatattaat cagaattatg ttactttgtt ttgttttatg tcgttacctt attgaagaaa 120
gagtggatga gaaggtaaat gacggggcat aaatatcgat tcgttgtcag aataagctgt 180
tttattcact taactggttg tttgccaatt tctccctaat tcccataact tgtataacta 240
aatttaataa tcaattttag taaattaaga ataggttaaa agtagtattt agaattaagt 300
taactttaat aaatttcctg tattttttta tagaaaaaag tataaaataa aaacatatca 360
aaaaagtttg aaatgacaat 380
<210> 22
<211> 542
<212> DNA
<213> 蓝细菌
<400> 22
gaatagttga taattactcg ttactcatta ctcacttaaa cctgccacct gatacctgcc 60
acctctcccc ccatcacctc atcccctcaa cattccgaac cccttgacac tttgaactaa 120
aattgtatta aagtgcaaat ctggacgggg ttaaccagtg tgacttataa tagtaaacgc 180
tgttttttat aataaataag ctaaatattt aaaaactatg agtaaatata cactaaatgg 240
tactagacgt aagcagaaaa gaacctccgg tttccgcgcc cgtatgagaa ccaaaaatgg 300
tagaaaagta attcaagctc gtcgtaataa gggtagaaaa agattagcag tataaaatta 360
ctgttaaata aggaagctaa gtttagcatt ttaagtttga tattactaat cattaaattt 420
actgtgaaat ataggtggga ctaccatcaa agcatcgact gaaacggcgt ttaaatttcc 480
aatctgttta tcaacagggt attcgccgct ctagtcgtta ttttattgtc cgagggttac 540
gg 542
<210> 23
<211> 70
<212> DNA
<213> 人工序列
<220>
<223> 广义PnirA序列
<220>
<221> misc_feature
<222> (1)..(1)
<223> n是任何116个核苷酸
<220>
<221> misc_feature
<222> (17)..(17)
<223> n是任何7个核苷酸
<220>
<221> misc_feature
<222> (33)..(33)
<223> n是任何15个核苷酸
<220>
<221> misc_feature
<222> (48)..(48)
<223> n是任何22个核苷酸
<220>
<221> misc_feature
<222> (55)..(55)
<223> n是任何55个核苷酸
<220>
<221> misc_feature
<222> (67)..(67)
<223> n是任何2个核苷酸
<400> 23
natgcaaaaa acgaatnatg tgtaaaaaga aangtagtca aagttacnta atgtnccgag 60
gacaaanatg 70
<210> 24
<211> 71
<212> DNA
<213> 人工序列
<220>
<223> 具有RBS中的核苷酸变化的广义PnirA序列
<220>
<221> misc_feature
<222> (1)..(1)
<223> n是任何116个核苷酸
<220>
<221> misc_feature
<222> (17)..(17)
<223> n是任何7个核苷酸
<220>
<221> misc_feature
<222> (33)..(33)
<223> n是任何15个核苷酸
<220>
<221> misc_feature
<222> (48)..(48)
<223> n是任何22个核苷酸
<220>
<221> misc_feature
<222> (55)..(55)
<223> n是任何55个核苷酸
<220>
<221> misc_feature
<222> (68)..(68)
<223> n是任何2个核苷酸
<400> 24
natgcaaaaa acgaatnatg tgtaaaaaga aangtagtca aagttacnta atgtnggagg 60
atcagccnat g 71
<210> 25
<211> 70
<212> DNA
<213> 人工序列
<220>
<223> 具有操纵子区域和TATA盒中的核苷酸变化的广义PnirA序列
<220>
<221> misc_feature
<222> (1)..(1)
<223> n是任何116个核苷酸
<220>
<221> misc_feature
<222> (17)..(17)
<223> n是任何7个核苷酸
<220>
<221> misc_feature
<222> (33)..(33)
<223> n是任何15个核苷酸
<220>
<221> misc_feature
<222> (48)..(48)
<223> n是任何22个核苷酸
<220>
<221> misc_feature
<222> (55)..(55)
<223> n是任何55个核苷酸
<220>
<221> misc_feature
<222> (67)..(67)
<223> n是任何2个核苷酸
<400> 25
natgcaaaaa acgcatnatg cgtaaaaagc atngtaatca aagttacnta atatnccgag 60
gacaaanatg 70
<210> 26
<211> 71
<212> DNA
<213> 人工序列
<220>
<223> 具有RBS、操纵子区域和TATA盒中的核苷酸变化的广义PnirA序列
<220>
<221> misc_feature
<222> (1)..(1)
<223> n是任何116个核苷酸
<220>
<221> misc_feature
<222> (17)..(17)
<223> n是任何7个核苷酸
<220>
<221> misc_feature
<222> (33)..(33)
<223> n是任何15个核苷酸
<220>
<221> misc_feature
<222> (48)..(48)
<223> n是任何22个核苷酸
<220>
<221> misc_feature
<222> (55)..(55)
<223> n是任何22个核苷酸
<220>
<221> misc_feature
<222> (68)..(68)
<223> n是任何22个核苷酸
<400> 26
natgcaaaaa acgcatnatg cgtaaaaagc atngtaatca aagttacnta atatnggagg 60
atcagccnat g 71
<210> 27
<211> 67
<212> DNA
<213> 人工序列
<220>
<223> Co2+诱导型PcorT
<220>
<221> misc_feature
<222> (4)..(4)
<223> n是任何7个核苷酸
<220>
<221> misc_feature
<222> (51)..(51)
<223> n是任何15个核苷酸
<400> 27
catngtttac tcaaaacctt gacattgaca ctaatgttaa ggtttaggct ncaagttaaa 60
aagcatg 67
<210> 28
<211> 67
<212> DNA
<213> 人工序列
<220>
<223> 修饰的PcorT变体包含RBS中的变化
<220>
<221> misc_feature
<222> (4)..(4)
<223> n是任何7个核苷酸
<220>
<221> misc_feature
<222> (51)..(51)
<223> n是任何15个核苷酸
<400> 28
catngtttac tcaaaacctt gacattgaca ctaatgttaa ggtttaggct ngaggataaa 60
aagcatg 67
<210> 29
<211> 65
<212> DNA
<213> 人工序列
<220>
<223> 修饰的PcorT变体包含TATA盒中的变化
<220>
<221> misc_feature
<222> (4)..(4)
<223> n是任何7个核苷酸
<220>
<221> misc_feature
<222> (49)..(49)
<223> n是任何15个核苷酸
<400> 29
catngtttac tcaaaacctt gacattgact aatgttaagg tttagaatnc aagttaaaaa 60
gcatg 65
<210> 30
<211> 67
<212> DNA
<213> 人工序列
<220>
<223> 修饰的PcorT变体包含RBS和TATA盒中的变化
<220>
<221> misc_feature
<222> (4)..(4)
<223> n是任何7个核苷酸
<220>
<221> misc_feature
<222> (51)..(51)
<223> n是任何15个核苷酸
<400> 30
catngtttac tcaaaacctt gacattgaca ctaatgttaa ggtttagaat ngaggataaa 60
aaccatg 67
<210> 31
<211> 69
<212> DNA
<213> 人工序列
<220>
<223> Zn2+诱导型PsmtA
<220>
<221> misc_feature
<222> (1)..(1)
<223> n是任何8个核苷酸
<220>
<221> misc_feature
<222> (28)..(28)
<223> n是任何4个核苷酸
<220>
<221> misc_feature
<222> (59)..(59)
<223> n是任何12个核苷酸
<220>
<221> misc_feature
<222> (66)..(66)
<223> n是任何7个核苷酸
<400> 31
naatacctga ataattgttc atgtgttnta aaaatgtgaa caatcgttca actatttang 60
gaggtnatg 69
<210> 32
<211> 74
<212> DNA
<213> 人工序列
<220>
<223> 具有RBS中的变化的Zn2+诱导型PsmtA
<220>
<221> misc_feature
<222> (1)..(1)
<223> n是任何8个核苷酸
<220>
<221> misc_feature
<222> (28)..(28)
<223> n是任何4个核苷酸
<220>
<221> misc_feature
<222> (59)..(59)
<223> n是任何10个核苷酸
<220>
<221> misc_feature
<222> (71)..(71)
<223> n是任何4个核苷酸
<400> 32
naatacctga ataattgttc atgtgttnta aaaatgtgaa caatcgttca actatttana 60
aggaggtgat natg 74
<210> 33
<211> 73
<212> DNA
<213> 人工序列
<220>
<223> 具有RBS中的变化的Zn2+诱导型PsmtA
<220>
<221> misc_feature
<222> (1)..(1)
<223> n是任何8个核苷酸
<220>
<221> misc_feature
<222> (28)..(28)
<223> n是任何4个核苷酸
<220>
<221> misc_feature
<222> (59)..(59)
<223> n是任何10个核苷酸
<220>
<221> misc_feature
<222> (70)..(70)
<223> n是任何5个核苷酸
<400> 33
naatacctga ataattgttc atgtgttnta aaaatgtgaa caatcgttca actatttana 60
aggaggtatn atg 73
<210> 34
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> CpcA同系物-结合至PCB的后胆色素结合蛋白片段
<400> 34
Lys Ser Lys Cys Ala Arg Asp Val Gly His Tyr Leu Arg Ile Ile Thr
1 5 10 15
Tyr Ser Leu Val Ala Gly Gly Thr Gly Pro Leu Asp Glu Tyr Leu Ile
20 25 30
Ala Gly Leu Ala Glu Ile
35
<210> 35
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> CpcA同系物-结合至PCB的后胆色素结合蛋白片段
<400> 35
Lys Ala Lys Cys Ala Arg Asp Ile Gly Tyr Tyr Leu Arg Ile Val Thr
1 5 10 15
Tyr Ala Leu Val Ala Gly Gly Thr Gly Pro Ile Asp Glu Tyr Leu Leu
20 25 30
Ala Gly Leu Asp Glu Ile
35
<210> 36
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> RpcA同系物-结合至PUB的后胆色素结合蛋白片段
<400> 36
Lys Ser Lys Cys Ser Arg Asp Val Gly Tyr Tyr Leu Arg Met Ile Thr
1 5 10 15
Tyr Cys Leu Val Ala Gly Gly Thr Gly Pro Met Asp Asp Tyr Leu Ile
20 25 30
Ala Gly Leu Ala Glu Ile
35
<210> 37
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> RpcA同系物-结合至PUB的后胆色素结合蛋白片段
<400> 37
Lys Ala Lys Cys Ser Arg Asp Val Gly Tyr Tyr Leu Arg Met Ile Thr
1 5 10 15
Tyr Cys Leu Val Ala Gly Gly Thr Gly Pro Met Asp Asp Tyr Leu Ile
20 25 30
Ala Gly Leu Asp Glu Ile
35
<210> 38
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> CpeA同系物-结合至PEB的后胆色素结合蛋白片段
<400> 38
Val Asp Lys Cys Tyr Arg Asp Leu Gly His Tyr Leu Arg Leu Ile Asn
1 5 10 15
Tyr Cys Leu Val Val Gly Gly Thr Gly Pro Leu Asp Glu Trp Gly Ile
20 25 30
Ala Gly Ala Arg Glu Val
35
<210> 39
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> CpeA同系物-结合至PEB的后胆色素结合蛋白片段
<400> 39
Ile Asp Lys Cys Tyr Arg Asp Leu Gly His Tyr Leu Arg Leu Ile Asn
1 5 10 15
Tyr Cys Leu Ile Val Gly Gly Thr Gly Pro Leu Asp Glu Trp Gly Ile
20 25 30
Ala Gly Ala Arg Glu Val
35
<210> 40
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> MpeA同系物-结合至PUB的后胆色素结合蛋白片段
<400> 40
Lys Glu Lys Cys Lys Arg Asp Phe Val His Tyr Leu Arg Leu Ile Asn
1 5 10 15
Tyr Ser Leu Val Val Gly Gly Thr Gly Pro Leu Asp Glu Leu Ala Ile
20 25 30
Asn Gly Gln Arg Glu Val
35
<210> 41
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> MpeA同系物-结合至PUB的后胆色素结合蛋白片段
<400> 41
Lys Glu Lys Cys Lys Arg Asp Phe Val His Tyr Leu Arg Leu Ile Asn
1 5 10 15
Tyr Cys Leu Val Thr Gly Gly Thr Gly Pro Leu Asp Glu Leu Ala Ile
20 25 30
Asn Gly Gln Lys Glu Val
35
<210> 42
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> ApcA同系物-结合至PCB的后胆色素结合蛋白片段
<400> 42
Arg Ala Gln Cys Leu Glu Ser Tyr Phe Gln Tyr Leu Glu Leu Ile Arg
1 5 10 15
Tyr Gly Val Leu Ala Gly Asp Lys Glu Pro Ile Glu Ser Ile Gly Leu
20 25 30
Leu Gly Ala Arg Glu Met
35
<210> 43
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> ApcA同系物-结合至PCB的后胆色素结合蛋白片段
<400> 43
Thr Ala Thr Cys Leu Arg Asp Met Asp Tyr Tyr Leu Arg Leu Ile Thr
1 5 10 15
Tyr Ser Ile Val Ala Gly Asp Ser Thr Pro Ile Gln Glu Ile Gly Asx
20 25 30
Ile Gly Asx Arg Glu Met
35
<210> 44
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> ApcA同系物-结合至PCB的后胆色素结合蛋白片段
<400> 44
Thr Ala Thr Cys Leu Arg Asp Met Asp Tyr Tyr Leu Arg Leu Val Thr
1 5 10 15
Tyr Gly Ile Val Ala Gly Asp Val Thr Pro Ile Glu Glu Ile Gly Val
20 25 30
Ile Gly Ala Lys Glu Leu
35
<210> 45
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> ApcA同系物-结合至PCB的后胆色素结合蛋白片段
<400> 45
Thr Ala Thr Cys Leu Arg Asp Met Asp Tyr Tyr Leu Arg Leu Val Thr
1 5 10 15
Tyr Gly Val Val Ala Gly Asp Val Thr Pro Ile Glu Glu Ile Gly Val
20 25 30
Ile Gly Ala Arg Glu Leu
35
<210> 46
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> ApcA同系物-结合至PCB的后胆色素结合蛋白片段
<400> 46
Thr Ala Thr Cys Leu Arg Asp Leu Asp Tyr Tyr Leu Arg Leu Val Thr
1 5 10 15
Tyr Gly Ile Val Ala Gly Asp Val Thr Pro Ile Glu Glu Ile Gly Val
20 25 30
Ile Gly Val Arg Glu Met
35
<210> 47
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> ApcA同系物-结合至PCB的后胆色素结合蛋白片段
<400> 47
Thr Ala Thr Cys Leu Arg Asp Leu Asp Tyr Tyr Leu Arg Leu Val Thr
1 5 10 15
Tyr Gly Asx Asx Ser Gly Asp Ile Thr Pro Ile Glu Glu Ile Gly Ile
20 25 30
Val Gly Val Arg Glu Met
35
<210> 48
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> 藻蓝蛋白和别藻蓝蛋白的β亚基
<400> 48
Met Ala Ala Cys Leu Arg Asp Met Glu Ile Ile Leu Arg Tyr Val Thr
1 5 10 15
Tyr Ser Ala Phe Thr Gly Asp Ala Ser Val Met Glu Asp Arg Val Leu
20 25 30
Asn Gly Leu Arg Glu Thr
35
<210> 49
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> 藻蓝蛋白和别藻蓝蛋白的β亚基
<400> 49
Met Ala Ala Cys Leu Arg Asp Met Glu Ile Ile Leu Arg Tyr Val Thr
1 5 10 15
Tyr Ala Val Phe Thr Gly Asp Ala Ser Ile Leu Asp Asp Arg Cys Leu
20 25 30
Asn Gly Leu Arg Glu Thr
35
<210> 50
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> 藻蓝蛋白和别藻蓝蛋白的β亚基
<400> 50
Met Ala Ala Cys Leu Arg Asp Gly Glu Ile Ile Leu Arg Tyr Ile Ser
1 5 10 15
Tyr Ala Leu Leu Ala Gly Asp Ala Ser Val Leu Asp Asp Arg Cys Leu
20 25 30
Asn Gly Leu Lys Glu Thr
35
<210> 51
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> 藻蓝蛋白和别藻蓝蛋白的β亚基
<400> 51
Met Ala Ala Cys Leu Arg Asp Gly Glu Ile Val Leu Arg Tyr Ile Ser
1 5 10 15
Tyr Ala Leu Leu Ala Gly Asp Ala Ser Val Leu Asp Asp Arg Cys Leu
20 25 30
Asn Gly Leu Lys Glu Thr
35
<210> 52
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> 藻蓝蛋白和别藻蓝蛋白的β亚基
<400> 52
Met Ala Ala Cys Leu Arg Asp Ala Glu Ile Ile Met Arg Tyr Val Ser
1 5 10 15
Tyr Ala Leu Leu Ala Gly Asp Ala Ser Val Leu Gln Asp Arg Cys Leu
20 25 30
Asn Gly Leu Arg Glu Thr
35
<210> 53
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> 藻蓝蛋白和别藻蓝蛋白的β亚基
<400> 53
Met Ala Ala Cys Leu Arg Asp Gly Glu Ile Val Leu Arg Tyr Val Ser
1 5 10 15
Tyr Ala Leu Leu Ala Gly Asp Ala Ser Val Leu Gln Asp Arg Cys Leu
20 25 30
Asn Gly Leu Arg Glu Thr
35
<210> 54
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> 藻蓝蛋白和别藻蓝蛋白的β亚基
<400> 54
Tyr Ser Ala Cys Leu Arg Asp Leu Asp Tyr Tyr Leu Arg Tyr Ala Thr
1 5 10 15
Tyr Ala Leu Val Ala Gly Asn Thr Glu Val Leu Asp Glu Arg Val Leu
20 25 30
Gln Gly Leu Arg Glu Thr
35
<210> 55
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> 藻蓝蛋白和别藻蓝蛋白的β亚基
<400> 55
Tyr Ala Ala Cys Ile Arg Asp Leu Glu Tyr Tyr Leu Arg Tyr Ala Thr
1 5 10 15
Tyr Ala Met Leu Ala Gly Asp Thr Ser Ile Leu Asp Glu Arg Val Leu
20 25 30
Asn Gly Leu Lys Glu Thr
35
<210> 56
<211> 38
<212> PRT
<213> 人工序列
<220>
<223> 藻蓝蛋白和别藻蓝蛋白的β亚基
<400> 56
Tyr Ala Ala Cys Ile Arg Asp Leu Asp Tyr Tyr Leu Arg Tyr Ser Thr
1 5 10 15
Tyr Ala Met Leu Ala Gly Asp Thr Ser Ile Leu Asp Glu Arg Val Leu
20 25 30
Asn Gly Leu Lys Glu Thr
35
<210> 57
<211> 3887
<212> DNA
<213> 人工序列
<220>
<223> 包含或编码Ptrc、编码Y130C突变的cpcAB基因与cpcA基因和卡那霉素抗性盒(插入到Syn 7942菌株MX2479的中性位点2中)
<400> 57
cgcgccatcg cttgcaattc gcgctaactt acattaattg cgttgcgctc attgaccact 60
ctccaaacgg ctcacttgcc gtgccagctg catgagacta tcggccagag cacgcgggga 120
ggccgtttgc gtgtttggcg ccaaggtggt tttgcgtttc accagcgaca cgggcagcag 180
ctgatttcct ttaactgcct gcccttggct cagttgcaac agtcgatcca cggacgtctg 240
acccaacaag cggaaatctt gcttgatcgt ggtcagaggc gggatataac aactcgaatc 300
ttcagtatca tcatagccca cgacactaat atctgcgccg acgcggaggc cgctctccgt 360
gatcgcacgc atcgcgccca aagccatctg gtcattcgcg accagcatgg ccgtaggcac 420
gatgccttca ttcagcattt gcattgtttg ctggaaaccc gacatagcgc tccaatcgcc 480
ctcgcgctcg gcgattggtt ggatctgatt gcgggtgagg tatttatgcc agcccgcgag 540
tcgcaggcga gcactcacgc tggagagcgg gcctgccaag agagcaatct gctgatggcc 600
cagcgcgacc agatgctcca cacccaagcg tgtaccgtcc tcgtgcgaga agatgatgct 660
attaatgggg gtttgatcgg agacgtccag gaacaacgcg ggaacgttcg tgcaggccgc 720
ttcaacagcg atagcatctt ggtcatccag cgggtagttg ataatcaggc ccgacacacg 780
ctgagccagg aggttgtgga ccgcagcttt gcaagcttcc acgccactcc gttcaaccat 840
ggagacgacc acgcttgccc ccagttgatc cgcacgcgat ttaatcgcgg caacaatttg 900
actcggggcg tgcagcgcga gagagctcgt ggcaaccccg atcaacaagc tctgttttcc 960
ggccagctgc tgcgcgacgc ggttggggat ataattcagc tcggccatcg cagcctcgac 1020
tttttcacgc gtctttgccg acacatggga ggcttgatta accacgcgac tgacagtttg 1080
gtagctcaca cctgcgtatt ctgcaacatc atacagcgtg actggcttca cattgaccat 1140
cctgaattga ctctcttccg ggcgctatca tgccataccg cgaaaggttt tgcaccattc 1200
gatggtgtca acgtacgact gcacggtgca ccaatgcttc tggcgtcagg cagccatcgg 1260
aagctgtggt atggctgtgc aggtcgtaaa tcactgcata attcgtgtcg ctcaaggcgc 1320
actcccgttc tggataatgt tttttgcgcc gacatcataa cggttctggc aaatattctg 1380
aaatgagctg ttgacaatta atcatccggc tcgtataatg tgtggaattg tgagcggata 1440
acaattgtcg acaggaaaca ggaattacat atgacttttg atgctttcac caaggtggtg 1500
gcacaagccg atgcccgtgg cgaatttttg agcgacgccc aactggacgc gctgagccgc 1560
ttggttgcag aaggcaacaa acggattgat acggtcaacc gcatcaccgg taatgcttcg 1620
tcgatcgtcg ctaacgcagc gcgtgcattg tttgcagagc aaccttctct gattgctcct 1680
ggcggcaacg catacacgaa ccgtcggatg gcggcttgtc tgcgcgacat ggaaatcatt 1740
ctccgctacg tgacctacgc ggtcttcacc ggcgatgctt ccattctcga cgaccgctgt 1800
ttgaacggtc tgcgtgagac ctacttggct ctgggcgtgc ccggtgcatc ggtggcagaa 1860
ggcgttcgca agatgaaaga cgcagctgtg gcgattgtga gcgaccgcaa cggcatcacc 1920
caaggtgact gttcagcgat catttccgag ctgggcagct acttcgacaa agctgctgct 1980
gcagttgcct agtcatcgac tgggattgag ataacagacc ttttttcaga gaaataggga 2040
atcatgtcca agactcctct gaccgaagct gtcgctgctg ctgattcgca agggcgtttt 2100
ctgagcagca ctgaactgca agttgcattt ggtcgtttcc gtcaagctgc ttctggtttg 2160
gcagcggcta aggcgttggc aaacaatgct gacagcttgg tcaacggtgc agcgaacgct 2220
gtttacagca agttccccta caccaccagc acgcctggca acaactttgc atcgacgccg 2280
gaaggcaaag cgaagtgtgc gcgtgacatt ggttactatc tgcggattgt gacctatgca 2340
ttggttgcgg gtggcacggg tccgattgat gagtacctgt tggcaggtct tgatgagatc 2400
aacaagacct tcgacttggc gccgagctgg tgtgtggaag cgctgaagta catcaaagcg 2460
aatcatggct tgagtggtga ctctcgcgat gaagccaact cctacatcga ctacctcatc 2520
aatgccctca gctagactag tcatcgagct agcaagcttg gccggatccg gccggatccg 2580
gagtttgtag aaacgcaaaa aggccatccg tcaggatggc cttctgctta atttgatgcc 2640
tggcagttta tggcgggcgt cctgcccgcc accctccggg ccgttgcttc gcaacgttca 2700
aatccgctcc cggcggattt gtcctactca ggagagcgtt caccgacaaa caacagataa 2760
aacgaaaggc ccagtctttc gactgagcct ttcgttttat ttgatgcctg gcagttccct 2820
actctcggta cccgtcggct tgaacgaatt gtcaagtcag cgtaatgctc tgccagtgtt 2880
acaaccaatt aaccaattct gattagaaaa actcatcgag catcaaatga aactgcaatt 2940
tattcatatc aggattatca ataccatatt tttgaaaaag ccgtttctgt aatgaaggag 3000
aaaactcacc gaggcagttc cataggatgg caagatcctg gtatcggtct gcgattccga 3060
ctcgtccaac atcaatacaa cctattaatt tcccctcgtc aaaaataagg ttatcaagtg 3120
agaaatcacc atgagtgacg actgaatccg gtgagaatgg caaaagctta tgcatttctt 3180
tccagacttg ttcaacaggc cagccattac gctcgtcatc aaaatcactc gcatcaacca 3240
aaccgttatt cattcgtgat tgcgcctgag cgagacgaaa tacgcgatcg ctgttaaaag 3300
gacaattaca aacaggaatc gaatgcaacc ggcgcaggaa cactgccagc gcatcaacaa 3360
tattttcacc tgaatcagga tattcttcta atacctggaa tgctgttttc ccggggatcg 3420
cagtggtgag taaccatgca tcatcaggag tacggataaa atgcttgatg gtcggaagag 3480
gcataaattc cgtcagccag tttagtctga ccatctcatc tgtaacatca ttggcaacgc 3540
tacctttgcc atgtttcaga aacaactctg gcgcatcggg cttcccatac aatcgataga 3600
ttgtcgcacc tgattgcccg acattatcgc gagcccattt atacccatat aaatcagcat 3660
ccatgttgga atttaatcgc ggcctagagc aagacgtttc ccgttgaata tggctcataa 3720
caccccttgt attactgttt atgtaagcag acagttttat tgttcatgat gatatatttt 3780
tatcttgtgc aatgtaacat cagagatttt gagacacaac gtggctttcg aacgagcgca 3840
aggtttcgaa ttcacatacg cggccgcctg ggccttgagc tcgaatt 3887
<210> 58
<211> 3887
<212> DNA
<213> 人工序列
<220>
<223> 包含或编码Ptrc、编码A86K突变的cpcAB基因与cpcA基因和卡那霉素抗性盒(插入到Syn 7942菌株MX2507的中性位点2中)
<400> 58
cgcgccatcg cttgcaattc gcgctaactt acattaattg cgttgcgctc attgaccact 60
ctccaaacgg ctcacttgcc gtgccagctg catgagacta tcggccagag cacgcgggga 120
ggccgtttgc gtgtttggcg ccaaggtggt tttgcgtttc accagcgaca cgggcagcag 180
ctgatttcct ttaactgcct gcccttggct cagttgcaac agtcgatcca cggacgtctg 240
acccaacaag cggaaatctt gcttgatcgt ggtcagaggc gggatataac aactcgaatc 300
ttcagtatca tcatagccca cgacactaat atctgcgccg acgcggaggc cgctctccgt 360
gatcgcacgc atcgcgccca aagccatctg gtcattcgcg accagcatgg ccgtaggcac 420
gatgccttca ttcagcattt gcattgtttg ctggaaaccc gacatagcgc tccaatcgcc 480
ctcgcgctcg gcgattggtt ggatctgatt gcgggtgagg tatttatgcc agcccgcgag 540
tcgcaggcga gcactcacgc tggagagcgg gcctgccaag agagcaatct gctgatggcc 600
cagcgcgacc agatgctcca cacccaagcg tgtaccgtcc tcgtgcgaga agatgatgct 660
attaatgggg gtttgatcgg agacgtccag gaacaacgcg ggaacgttcg tgcaggccgc 720
ttcaacagcg atagcatctt ggtcatccag cgggtagttg ataatcaggc ccgacacacg 780
ctgagccagg aggttgtgga ccgcagcttt gcaagcttcc acgccactcc gttcaaccat 840
ggagacgacc acgcttgccc ccagttgatc cgcacgcgat ttaatcgcgg caacaatttg 900
actcggggcg tgcagcgcga gagagctcgt ggcaaccccg atcaacaagc tctgttttcc 960
ggccagctgc tgcgcgacgc ggttggggat ataattcagc tcggccatcg cagcctcgac 1020
tttttcacgc gtctttgccg acacatggga ggcttgatta accacgcgac tgacagtttg 1080
gtagctcaca cctgcgtatt ctgcaacatc atacagcgtg actggcttca cattgaccat 1140
cctgaattga ctctcttccg ggcgctatca tgccataccg cgaaaggttt tgcaccattc 1200
gatggtgtca acgtacgact gcacggtgca ccaatgcttc tggcgtcagg cagccatcgg 1260
aagctgtggt atggctgtgc aggtcgtaaa tcactgcata attcgtgtcg ctcaaggcgc 1320
actcccgttc tggataatgt tttttgcgcc gacatcataa cggttctggc aaatattctg 1380
aaatgagctg ttgacaatta atcatccggc tcgtataatg tgtggaattg tgagcggata 1440
acaattgtcg acaggaaaca ggaattacat atgacttttg atgctttcac caaggtggtg 1500
gcacaagccg atgcccgtgg cgaatttttg agcgacgccc aactggacgc gctgagccgc 1560
ttggttgcag aaggcaacaa acggattgat acggtcaacc gcatcaccgg taatgcttcg 1620
tcgatcgtcg ctaacgcagc gcgtgcattg tttgcagagc aaccttctct gattgctcct 1680
ggcggcaacg catacacgaa ccgtcggatg gcggcttgtc tgcgcgacat ggaaatcatt 1740
ctccgctacg tgacctacgc ggtcttcacc ggcgatgctt ccattctcga cgaccgctgt 1800
ttgaacggtc tgcgtgagac ctacttggct ctgggcgtgc ccggtgcatc ggtggcagaa 1860
ggcgttcgca agatgaaaga cgcagctgtg gcgattgtga gcgaccgcaa cggcatcacc 1920
caaggtgact gttcagcgat catttccgag ctgggcagct acttcgacaa agctgctgct 1980
gcagttgcct agtcatcgac tgggattgag ataacagacc ttttttcaga gaaataggga 2040
atcatgtcca agactcctct gaccgaagct gtcgctgctg ctgattcgca agggcgtttt 2100
ctgagcagca ctgaactgca agttgcattt ggtcgtttcc gtcaagctgc ttctggtttg 2160
gcagcggcta aggcgttggc aaacaatgct gacagcttgg tcaacggtgc agcgaacgct 2220
gtttacagca agttccccta caccaccagc acgcctggca acaactttgc atcgacgccg 2280
gaaggcaaag cgaagtgtgc gcgtgacatt ggttactatc tgcggattgt gacctatgca 2340
ttggttgcgg gtggcacggg tccgattgat gagtacctgt tggcaggtct tgatgagatc 2400
aacaagacct tcgacttggc gccgagctgg tgtgtggaag cgctgaagta catcaaagcg 2460
aatcatggct tgagtggtga ctctcgcgat gaagccaact cctacatcga ctacctcatc 2520
aatgccctca gctagactag tcatcgagct agcaagcttg gccggatccg gccggatccg 2580
gagtttgtag aaacgcaaaa aggccatccg tcaggatggc cttctgctta atttgatgcc 2640
tggcagttta tggcgggcgt cctgcccgcc accctccggg ccgttgcttc gcaacgttca 2700
aatccgctcc cggcggattt gtcctactca ggagagcgtt caccgacaaa caacagataa 2760
aacgaaaggc ccagtctttc gactgagcct ttcgttttat ttgatgcctg gcagttccct 2820
actctcggta cccgtcggct tgaacgaatt gtcaagtcag cgtaatgctc tgccagtgtt 2880
acaaccaatt aaccaattct gattagaaaa actcatcgag catcaaatga aactgcaatt 2940
tattcatatc aggattatca ataccatatt tttgaaaaag ccgtttctgt aatgaaggag 3000
aaaactcacc gaggcagttc cataggatgg caagatcctg gtatcggtct gcgattccga 3060
ctcgtccaac atcaatacaa cctattaatt tcccctcgtc aaaaataagg ttatcaagtg 3120
agaaatcacc atgagtgacg actgaatccg gtgagaatgg caaaagctta tgcatttctt 3180
tccagacttg ttcaacaggc cagccattac gctcgtcatc aaaatcactc gcatcaacca 3240
aaccgttatt cattcgtgat tgcgcctgag cgagacgaaa tacgcgatcg ctgttaaaag 3300
gacaattaca aacaggaatc gaatgcaacc ggcgcaggaa cactgccagc gcatcaacaa 3360
tattttcacc tgaatcagga tattcttcta atacctggaa tgctgttttc ccggggatcg 3420
cagtggtgag taaccatgca tcatcaggag tacggataaa atgcttgatg gtcggaagag 3480
gcataaattc cgtcagccag tttagtctga ccatctcatc tgtaacatca ttggcaacgc 3540
tacctttgcc atgtttcaga aacaactctg gcgcatcggg cttcccatac aatcgataga 3600
ttgtcgcacc tgattgcccg acattatcgc gagcccattt atacccatat aaatcagcat 3660
ccatgttgga atttaatcgc ggcctagagc aagacgtttc ccgttgaata tggctcataa 3720
caccccttgt attactgttt atgtaagcag acagttttat tgttcatgat gatatatttt 3780
tatcttgtgc aatgtaacat cagagatttt gagacacaac gtggctttcg aacgagcgca 3840
aggtttcgaa ttcacatacg cggccgcctg ggccttgagc tcgaatt 3887
<210> 59
<211> 2922
<212> DNA
<213> 人工序列
<220>
<223> 包含或编码Ptrc、CpeS1和杀稻瘟菌素抗性(插入到Syn 7942菌株MX2505的中性位点5中)
<400> 59
agcttgcatg cctgcaggtg gcgcgccatc gcttgcaatt cgcgctaact tacattaatt 60
gcgttgcgct cattgaccac tctccaaacg gctcacttgc cgtgccagct gcatgagact 120
atcggccaga gcacgcgggg aggccgtttg cgtgtttggc gccaaggtgg ttttgcgttt 180
caccagcgac acgggcagca gctgatttcc tttaactgcc tgcccttggc tcagttgcaa 240
cagtcgatcc acggacgtct gacccaacaa gcggaaatct tgcttgatcg tggtcagagg 300
cgggatataa caactcgaat cttcagtatc atcatagccc acgacactaa tatctgcgcc 360
gacgcggagg ccgctctccg tgatcgcacg catcgcgccc aaagccatct ggtcattcgc 420
gaccagcatg gccgtaggca cgatgccttc attcagcatt tgcattgttt gctggaaacc 480
cgacatagcg ctccaatcgc cctcgcgctc ggcgattggt tggatctgat tgcgggtgag 540
gtatttatgc cagcccgcga gtcgcaggcg agcactcacg ctggagagcg ggcctgccaa 600
gagagcaatc tgctgatggc ccagcgcgac cagatgctcc acacccaagc gtgtaccgtc 660
ctcgtgcgag aagatgatgc tattaatggg ggtttgatcg gagacgtcca ggaacaacgc 720
gggaacgttc gtgcaggccg cttcaacagc gatagcatct tggtcatcca gcgggtagtt 780
gataatcagg cccgacacac gctgagccag gaggttgtgg accgcagctt tgcaagcttc 840
cacgccactc cgttcaacca tggagacgac cacgcttgcc cccagttgat ccgcacgcga 900
tttaatcgcg gcaacaattt gactcggggc gtgcagcgcg agagagctcg tggcaacccc 960
gatcaacaag ctctgttttc cggccagctg ctgcgcgacg cggttgggga tataattcag 1020
ctcggccatc gcagcctcga ctttttcacg cgtctttgcc gacacatggg aggcttgatt 1080
aaccacgcga ctgacagttt ggtagctcac acctgcgtat tctgcaacat catacagcgt 1140
gactggcttc acattgacca tcctgaattg actctcttcc gggcgctatc atgccatacc 1200
gcgaaaggtt ttgcaccatt cgatggtgtc aacgtacgac tgcacggtgc accaatgctt 1260
ctggcgtcag gcagccatcg gaagctgtgg tatggctgtg caggtcgtaa atcactgcat 1320
aattcgtgtc gctcaaggcg cactcccgtt ctggataatg ttttttgcgc cgacatcata 1380
acggttctgg caaatattct gaaatgagct gttgacaatt aatcatccgg ctcgtataat 1440
gtgtggaatt gtgagcggat aacaattgtc gacaggaaac aggaattaca tatgtttccc 1500
ctccagtcat atccaccaat gacgatggtg gattttttcg aagcaagccg tgggacctgg 1560
ttgaaccgac gtgctgttca tcatttggat caccaggatg atgaagcagc agattctaat 1620
cttgttatcg aaccatttaa aaatgatgat ccggcagttc gcagcatttg cgaagcccta 1680
aacatcaaca tgatcgacag tactggtgga gctagatttt ggtgggaaag taatattaaa 1740
aaaggagtcc gcaacgaaga ttatgctgct gttgtcatcg atgtacccaa ccgagataat 1800
gctcgaaagg gtttcttact acgagatgta ggatatgttg aaaagcaggc ggtattgagc 1860
acttacgttt ttgccgaaga tggcgtgttg acgatcacta caagatatga cacgaatatt 1920
ggaattgaac gatgctggtt tgtgactgat cagatccgaa tgcgtgtcag ttctgtccaa 1980
tgcttggatg gtgtcgcaat gactacctac tgcactgaat ttcgctgtcc aacagatgct 2040
gatatcaatg ccatatctga gcatgccagg cagatcgctc gttcgactgc atctattgga 2100
gcttaaacta gtcatcgagc tagcaagctt ggccggatcc ggccggatcc ggagtttgta 2160
gaaacgcaaa aaggccatcc gtcaggatgg ccttctgctt aatttgatgc ctggcagttt 2220
atggcgggcg tcctgcccgc caccctccgg gccgttgctt cgcaacgttc aaatccgctc 2280
ccggcggatt tgtcctactc aggagagcgt tcaccgacaa acaacagata aaacgaaagg 2340
cccagtcttt cgactgagcc tttcgtttta tttgatgcct ggcagttccc tactctcggt 2400
acccgtcggc ttgaacgaat tgttagacac taaccttccc aaacatagcc actagggagc 2460
agctcccgga tgccaacagc cgtgggctgg ccatcgctat ctttcacaat tgctttgatg 2520
ccggggtgca gatccaggag cacctgccgg caacggccac aggggctcaa gatcccgcgg 2580
ttttcgttgc cgatggcaac gatgcacgtc aggttgccgg ctgcagccgc tgctgcagta 2640
cccaggacga ccagttccgc acaagggcca ccggtaaaat ggtagacatt cacaccggta 2700
aagatgcggc catcgctcga caatgcagcc gacgccacgc tataatcttc gctaatagga 2760
atcgagttaa tagtcgcggt cgcgcgttcg atcagtgtag actcctcttg actgaggggt 2820
ttcgccatgg tttagttcct caccttgtcg tattatacta tgccgatata ctatgccgat 2880
gattaattgt caacgcggcc gcctgggcct tgagctcgaa tt 2922
<210> 60
<211> 2844
<212> DNA
<213> 人工序列
<220>
<223> 包含或编码Ptrc、CpeS2和杀稻瘟菌素抗性盒(插入到Syn 7942菌株MX2506的中性位点5中)
<400> 60
atcgcttgca attcgcgcta acttacatta attgcgttgc gctcattgac cactctccaa 60
acggctcact tgccgtgcca gctgcatgag actatcggcc agagcacgcg gggaggccgt 120
ttgcgtgttt ggcgccaagg tggttttgcg tttcaccagc gacacgggca gcagctgatt 180
tcctttaact gcctgccctt ggctcagttg caacagtcga tccacggacg tctgacccaa 240
caagcggaaa tcttgcttga tcgtggtcag aggcgggata taacaactcg aatcttcagt 300
atcatcatag cccacgacac taatatctgc gccgacgcgg aggccgctct ccgtgatcgc 360
acgcatcgcg cccaaagcca tctggtcatt cgcgaccagc atggccgtag gcacgatgcc 420
ttcattcagc atttgcattg tttgctggaa acccgacata gcgctccaat cgccctcgcg 480
ctcggcgatt ggttggatct gattgcgggt gaggtattta tgccagcccg cgagtcgcag 540
gcgagcactc acgctggaga gcgggcctgc caagagagca atctgctgat ggcccagcgc 600
gaccagatgc tccacaccca agcgtgtacc gtcctcgtgc gagaagatga tgctattaat 660
gggggtttga tcggagacgt ccaggaacaa cgcgggaacg ttcgtgcagg ccgcttcaac 720
agcgatagca tcttggtcat ccagcgggta gttgataatc aggcccgaca cacgctgagc 780
caggaggttg tggaccgcag ctttgcaagc ttccacgcca ctccgttcaa ccatggagac 840
gaccacgctt gcccccagtt gatccgcacg cgatttaatc gcggcaacaa tttgactcgg 900
ggcgtgcagc gcgagagagc tcgtggcaac cccgatcaac aagctctgtt ttccggccag 960
ctgctgcgcg acgcggttgg ggatataatt cagctcggcc atcgcagcct cgactttttc 1020
acgcgtcttt gccgacacat gggaggcttg attaaccacg cgactgacag tttggtagct 1080
cacacctgcg tattctgcaa catcatacag cgtgactggc ttcacattga ccatcctgaa 1140
ttgactctct tccgggcgct atcatgccat accgcgaaag gttttgcacc attcgatggt 1200
gtcaacgtac gactgcacgg tgcaccaatg cttctggcgt caggcagcca tcggaagctg 1260
tggtatggct gtgcaggtcg taaatcactg cataattcgt gtcgctcaag gcgcactccc 1320
gttctggata atgttttttg cgccgacatc ataacggttc tggcaaatat tctgaaatga 1380
gctgttgaca attaatcatc cggctcgtat aatgtgtgga attgtgagcg gataacaatt 1440
gtcgacagga aacaggaatt acatatgagc acaatattaa aaagtatgac gattgagcaa 1500
tttgttgctc aaagtgtggg taaatggcgc tccatgagat caggccattc tctcgctttt 1560
caacaatttg aagacgttct tagcgaagta attattgaat ccatcgagaa agacgattct 1620
gctgttcaag atttactctc aaccgcaact tctaaccaag gacatagctc cgacatcgtc 1680
gcgccattca ggatggaatg gtcagctgaa agtgactggg agcccgaaga tccatctcaa 1740
gtttcatcag gctcgtgcct gatcatccca ctgaaaaaaa atgattattc tggcatcttg 1800
atcagaagtg tggggtatgc tgaatccgaa ttagcagagt cgacatacca gtttttagac 1860
gatggcacat tcttgcttac aacgcattat gagcaatcaa tggcagagga aagaatctgg 1920
tttgtttcag acaatgttcg gtgcagatca tctgtattga agacatctgc aggctcagga 1980
gttctacaaa cttcatttgc ctctgaagtc agacgagtca aggcctagac tagtcatcga 2040
gctagcaagc ttggccggat ccggccggat ccggagtttg tagaaacgca aaaaggccat 2100
ccgtcaggat ggccttctgc ttaatttgat gcctggcagt ttatggcggg cgtcctgccc 2160
gccaccctcc gggccgttgc ttcgcaacgt tcaaatccgc tcccggcgga tttgtcctac 2220
tcaggagagc gttcaccgac aaacaacaga taaaacgaaa ggcccagtct ttcgactgag 2280
cctttcgttt tatttgatgc ctggcagttc cctactctcg gtacccgtcg gcttgaacga 2340
attgttagac actaaccttc ccaaacatag ccactaggga gcagctcccg gatgccaaca 2400
gccgtgggct ggccatcgct atctttcaca attgctttga tgccggggtg cagatccagg 2460
agcacctgcc ggcaacggcc acaggggctc aagatcccgc ggttttcgtt gccgatggca 2520
acgatgcacg tcaggttgcc ggctgcagcc gctgctgcag tacccaggac gaccagttcc 2580
gcacaagggc caccggtaaa atggtagaca ttcacaccgg taaagatgcg gccatcgctc 2640
gacaatgcag ccgacgccac gctataatct tcgctaatag gaatcgagtt aatagtcgcg 2700
gtcgcgcgtt cgatcagtgt agactcctct tgactgaggg gtttcgccat ggtttagttc 2760
ctcaccttgt cgtattatac tatgccgata tactatgccg atgattaatt gtcaacgcgg 2820
ccgcctgggc cttgagctcg aatt 2844
<210> 61
<211> 163
<212> PRT
<213> 人工序列
<220>
<223> 变体CpcA (A86K)蛋白
<400> 61
Met Ser Lys Thr Pro Leu Thr Glu Ala Val Ala Ala Ala Asp Ser Gln
1 5 10 15
Gly Arg Phe Leu Ser Ser Thr Glu Leu Gln Val Ala Phe Gly Arg Phe
20 25 30
Arg Gln Ala Ala Ser Gly Leu Ala Ala Ala Lys Ala Leu Ala Asn Asn
35 40 45
Ala Asp Ser Leu Val Asn Gly Ala Ala Asn Ala Val Tyr Ser Lys Phe
50 55 60
Pro Tyr Thr Thr Ser Thr Pro Gly Asn Asn Phe Ala Ser Thr Pro Glu
65 70 75 80
Gly Lys Ala Lys Cys Lys Arg Asp Ile Gly Tyr Tyr Leu Arg Ile Val
85 90 95
Thr Tyr Ala Leu Val Ala Gly Gly Thr Gly Pro Ile Asp Glu Tyr Leu
100 105 110
Leu Ala Gly Leu Asp Glu Ile Asn Lys Thr Phe Asp Leu Ala Pro Ser
115 120 125
Trp Thr Val Glu Ala Leu Lys Tyr Ile Lys Ala Asn His Gly Leu Ser
130 135 140
Gly Asp Ser Arg Asp Glu Ala Asn Ser Tyr Ile Asp Tyr Leu Ile Asn
145 150 155 160
Ala Leu Ser
<210> 62
<211> 163
<212> PRT
<213> 人工序列
<220>
<223> 变体CpcA (Y130C)蛋白
<400> 62
Met Ser Lys Thr Pro Leu Thr Glu Ala Val Ala Ala Ala Asp Ser Gln
1 5 10 15
Gly Arg Phe Leu Ser Ser Thr Glu Leu Gln Val Ala Phe Gly Arg Phe
20 25 30
Arg Gln Ala Ala Ser Gly Leu Ala Ala Ala Lys Ala Leu Ala Asn Asn
35 40 45
Ala Asp Ser Leu Val Asn Gly Ala Ala Asn Ala Val Tyr Ser Lys Phe
50 55 60
Pro Tyr Thr Thr Ser Thr Pro Gly Asn Asn Phe Ala Ser Thr Pro Glu
65 70 75 80
Gly Lys Ala Lys Cys Ala Arg Asp Ile Gly Tyr Tyr Leu Arg Ile Val
85 90 95
Thr Tyr Ala Leu Val Ala Gly Gly Thr Gly Pro Ile Asp Glu Tyr Leu
100 105 110
Leu Ala Gly Leu Asp Glu Ile Asn Lys Thr Phe Asp Leu Ala Pro Ser
115 120 125
Trp Cys Val Glu Ala Leu Lys Tyr Ile Lys Ala Asn His Gly Leu Ser
130 135 140
Gly Asp Ser Arg Asp Glu Ala Asn Ser Tyr Ile Asp Tyr Leu Ile Asn
145 150 155 160
Ala Leu Ser
<210> 63
<211> 173
<212> PRT
<213> 蓝细菌
<400> 63
Met Thr Phe Asp Ala Phe Thr Lys Val Val Ala Gln Ala Asp Ala Arg
1 5 10 15
Gly Glu Phe Leu Ser Asp Ala Gln Leu Asp Ala Leu Ser Arg Leu Val
20 25 30
Ala Glu Gly Asn Lys Arg Ile Asp Thr Val Asn Arg Ile Thr Gly Asn
35 40 45
Ala Ser Ser Ile Val Ala Asn Ala Ala Arg Ala Leu Phe Ala Glu Gln
50 55 60
Pro Ser Leu Ile Ala Pro Gly Gly Asn Ala Tyr Thr Asn Arg Arg Met
65 70 75 80
Ala Ala Cys Leu Arg Asp Met Glu Ile Ile Leu Arg Tyr Val Thr Tyr
85 90 95
Ala Val Phe Thr Gly Asp Ala Ser Ile Leu Asp Asp Arg Cys Leu Asn
100 105 110
Gly Leu Arg Glu Thr Tyr Leu Ala Leu Gly Val Pro Gly Ala Ser Val
115 120 125
Ala Glu Gly Val Arg Lys Met Lys Asp Ala Ala Val Ala Ile Val Ser
130 135 140
Asp Arg Asn Gly Ile Thr Gln Gly Asp Cys Ser Ala Ile Ile Ser Glu
145 150 155 160
Leu Gly Ser Tyr Phe Asp Lys Ala Ala Ala Ala Val Ala
165 170
<210> 64
<211> 204
<212> PRT
<213> 蓝细菌
<400> 64
Met Phe Pro Leu Gln Ser Tyr Pro Pro Met Thr Met Val Asp Phe Phe
1 5 10 15
Glu Ala Ser Arg Gly Thr Trp Leu Asn Arg Arg Ala Val His His Leu
20 25 30
Asp His Gln Asp Asp Glu Ala Ala Asp Ser Asn Leu Val Ile Glu Pro
35 40 45
Phe Lys Asn Asp Asp Pro Ala Val Arg Ser Ile Cys Glu Ala Leu Asn
50 55 60
Ile Asn Met Ile Asp Ser Thr Gly Gly Ala Arg Phe Trp Trp Glu Ser
65 70 75 80
Asn Ile Lys Lys Gly Val Arg Asn Glu Asp Tyr Ala Ala Val Val Ile
85 90 95
Asp Val Pro Asn Arg Asp Asn Ala Arg Lys Gly Phe Leu Leu Arg Asp
100 105 110
Val Gly Tyr Val Glu Lys Gln Ala Val Leu Ser Thr Tyr Val Phe Ala
115 120 125
Glu Asp Gly Val Leu Thr Ile Thr Thr Arg Tyr Asp Thr Asn Ile Gly
130 135 140
Ile Glu Arg Cys Trp Phe Val Thr Asp Gln Ile Arg Met Arg Val Ser
145 150 155 160
Ser Val Gln Cys Leu Asp Gly Val Ala Met Thr Thr Tyr Cys Thr Glu
165 170 175
Phe Arg Cys Pro Thr Asp Ala Asp Ile Asn Ala Ile Ser Glu His Ala
180 185 190
Arg Gln Ile Ala Arg Ser Thr Ala Ser Ile Gly Ala
195 200
<210> 65
<211> 187
<212> PRT
<213> 蓝细菌
<400> 65
Met Ser Thr Ile Leu Lys Ser Met Thr Ile Glu Gln Phe Val Ala Gln
1 5 10 15
Ser Val Gly Lys Trp Arg Ser Met Arg Ser Gly His Ser Leu Ala Phe
20 25 30
Gln Gln Phe Glu Asp Val Leu Ser Glu Val Ile Ile Glu Ser Ile Glu
35 40 45
Lys Asp Asp Ser Ala Val Gln Asp Leu Leu Ser Thr Ala Thr Ser Asn
50 55 60
Gln Gly His Ser Ser Asp Ile Val Ala Pro Phe Arg Met Glu Trp Ser
65 70 75 80
Ala Glu Ser Asp Trp Glu Pro Glu Asp Pro Ser Gln Val Ser Ser Gly
85 90 95
Ser Cys Leu Ile Ile Pro Leu Lys Lys Asn Asp Tyr Ser Gly Ile Leu
100 105 110
Ile Arg Ser Val Gly Tyr Ala Glu Ser Glu Leu Ala Glu Ser Thr Tyr
115 120 125
Gln Phe Leu Asp Asp Gly Thr Phe Leu Leu Thr Thr His Tyr Glu Gln
130 135 140
Ser Met Ala Glu Glu Arg Ile Trp Phe Val Ser Asp Asn Val Arg Cys
145 150 155 160
Arg Ser Ser Val Leu Lys Thr Ser Ala Gly Ser Gly Val Leu Gln Thr
165 170 175
Ser Phe Ala Ser Glu Val Arg Arg Val Lys Ala
180 185
<210> 66
<211> 492
<212> DNA
<213> 人工序列
<220>
<223> 具有终止密码子的变体CpcA (A86K)基因
<400> 66
atgtccaaga ctcctctgac cgaagctgtc gctgctgctg attcgcaagg gcgttttctg 60
agcagcactg aactgcaagt tgcatttggt cgtttccgtc aagctgcttc tggtttggca 120
gcggctaagg cgttggcaaa caatgctgac agcttggtca acggtgcagc gaacgctgtt 180
tacagcaagt tcccctacac caccagcacg cctggcaaca actttgcatc gacgccggaa 240
ggcaaagcga agtgtaagcg tgacattggt tactatctgc ggattgtgac ctatgcattg 300
gttgcgggtg gcacgggtcc gattgatgag tacctgttgg caggtcttga tgagatcaac 360
aagaccttcg acttggcgcc gagctggtgt gtggaagcgc tgaagtacat caaagcgaat 420
catggcttga gtggtgactc tcgcgatgaa gccaactcct acatcgacta cctcatcaat 480
gccctcagct ag 492
<210> 67
<211> 492
<212> DNA
<213> 人工序列
<220>
<223> 具有终止密码子的变体CpcA (Y130C)基因
<400> 67
atgtccaaga ctcctctgac cgaagctgtc gctgctgctg attcgcaagg gcgttttctg 60
agcagcactg aactgcaagt tgcatttggt cgtttccgtc aagctgcttc tggtttggca 120
gcggctaagg cgttggcaaa caatgctgac agcttggtca acggtgcagc gaacgctgtt 180
tacagcaagt tcccctacac caccagcacg cctggcaaca actttgcatc gacgccggaa 240
ggcaaagcga agtgtgcgcg tgacattggt tactatctgc ggattgtgac ctatgcattg 300
gttgcgggtg gcacgggtcc gattgatgag tacctgttgg caggtcttga tgagatcaac 360
aagaccttcg acttggcgcc gagctggtgt gtggaagcgc tgaagtacat caaagcgaat 420
catggcttga gtggtgactc tcgcgatgaa gccaactcct acatcgacta cctcatcaat 480
gccctcagct ag 492
<210> 68
<211> 522
<212> DNA
<213> 蓝细菌
<400> 68
atgacttttg atgctttcac caaggtggtg gcacaagccg atgcccgtgg cgaatttttg 60
agcgacgccc aactggacgc gctgagccgc ttggttgcag aaggcaacaa acggattgat 120
acggtcaacc gcatcaccgg taatgcttcg tcgatcgtcg ctaacgcagc gcgtgcattg 180
tttgcagagc aaccttctct gattgctcct ggcggcaacg catacacgaa ccgtcggatg 240
gcggcttgtc tgcgcgacat ggaaatcatt ctccgctacg tgacctacgc ggtcttcacc 300
ggcgatgctt ccattctcga cgaccgctgt ttgaacggtc tgcgtgagac ctacttggct 360
ctgggcgtgc ccggtgcatc ggtggcagaa ggcgttcgca agatgaaaga cgcagctgtg 420
gcgattgtga gcgaccgcaa cggcatcacc caaggtgact gttcagcgat catttccgag 480
ctgggcagct acttcgacaa agctgctgct gcagttgcct ag 522
<210> 69
<211> 615
<212> DNA
<213> 蓝细菌
<400> 69
atgtttcccc tccagtcata tccaccaatg acgatggtgg attttttcga agcaagccgt 60
gggacctggt tgaaccgacg tgctgttcat catttggatc accaggatga tgaagcagca 120
gattctaatc ttgttatcga accatttaaa aatgatgatc cggcagttcg cagcatttgc 180
gaagccctaa acatcaacat gatcgacagt actggtggag ctagattttg gtgggaaagt 240
aatattaaaa aaggagtccg caacgaagat tatgctgctg ttgtcatcga tgtacccaac 300
cgagataatg ctcgaaaggg tttcttacta cgagatgtag gatatgttga aaagcaggcg 360
gtattgagca cttacgtttt tgccgaagat ggcgtgttga cgatcactac aagatatgac 420
acgaatattg gaattgaacg atgctggttt gtgactgatc agatccgaat gcgtgtcagt 480
tctgtccaat gcttggatgg tgtcgcaatg actacctact gcactgaatt tcgctgtcca 540
acagatgctg atatcaatgc catatctgag catgccaggc agatcgctcg ttcgactgca 600
tctattggag cttaa 615
<210> 70
<211> 564
<212> DNA
<213> 蓝细菌
<400> 70
atgagcacaa tattaaaaag tatgacgatt gagcaatttg ttgctcaaag tgtgggtaaa 60
tggcgctcca tgagatcagg ccattctctc gcttttcaac aatttgaaga cgttcttagc 120
gaagtaatta ttgaatccat cgagaaagac gattctgctg ttcaagattt actctcaacc 180
gcaacttcta accaaggaca tagctccgac atcgtcgcgc cattcaggat ggaatggtca 240
gctgaaagtg actgggagcc cgaagatcca tctcaagttt catcaggctc gtgcctgatc 300
atcccactga aaaaaaatga ttattctggc atcttgatca gaagtgtggg gtatgctgaa 360
tccgaattag cagagtcgac ataccagttt ttagacgatg gcacattctt gcttacaacg 420
cattatgagc aatcaatggc agaggaaaga atctggtttg tttcagacaa tgttcggtgc 480
agatcatctg tattgaagac atctgcaggc tcaggagttc tacaaacttc atttgcctct 540
gaagtcagac gagtcaaggc ctag 564
- 还没有人留言评论。精彩留言会获得点赞!