一种深海来源超氧化物歧化酶及其制备方法与流程
本发明涉及Rimicaris exoculata超氧化物歧化酶,尤其是涉及一种深海来源超氧化物歧化酶及其制备方法。
背景技术:
氧化还原反应是生命体最重要的代谢途径,它不仅为生物提供能量,同时还决定着生命体的衰老和死亡。氧对于生命活动极其重要,但氧参与的代谢经常产生一些对细胞有毒害作用的副产物——氧自由基,即通常所述的活性氧(reactiveoxygen species,ROS)(1、方允中,李文杰.自由基与酶:基础理论及其在生物学和医学中的应用[M].科学出版社,1989.)。细胞产生的活性氧包括:超氧根阴离子(O2·-)、氢氧根离子(OH-)、羟自由基(-OH)、过氧化氢(H2O2)、单线态氧(1O2)和过氧化物自由基(ROO-)等。通常产生的氧自由基能够被机体清除,但当机体氧化应激反应发生障碍,或者外界的氧压力过大,产生过量的氧自由基时,会引起一系列有害的生化反应,造成蛋白质损伤、脂质过氧化、DNA突变和酶失活等(2、陈鸿鹏,谭晓风.超氧化物歧化酶(SOD)研究综述.经济林研究,2007,25(1):59-65)。为了防止氧自由基对细胞体的破坏,几乎所有细胞都有一套完整的保护体系,来清除细胞新陈代谢产生的各种活性氧。其中,超氧化物歧化酶(surperoxide dismutase,EC.1.5.1.1,SOD)在保护细胞免受氧自由基的毒害中发挥着重要作用。
超氧化物歧化酶的底物为超氧根阴离子,其同时含有一个负电荷,又带有一个未成对的电子,所以其既可被氧化成氧气,也可被还原为过氧化氢。超氧化物歧化酶所催化的反应如下:
SOD+O2·-→SOD-+O2 第一步
SOD-+O2·-+2H+→SOD+H2O2 第二步
2O2·-+2H++SOD→O2+H2O2 总反应
紧接着,产生的过氧化氢,会在过氧化氢酶的作用下被消除,产生氧气和水。
不但机体会产生超氧阴离子自由基,环境中多种物理及化学因素也能产生该自由基,如电离辐射、紫外线、光敏剂及金属离子等。因此SOD在防止这些环境因素对生物体造成的损伤方面也发挥着重要作用。
现如今,在多个领域超氧化物歧化酶都扮演着至关重要的作用。
SOD在化妆品领域中的应用:研究表明,化妆品中添加SOD可起到四方面的作用(3、马振华,杨红强,杨琼.超氧化物歧化酶(SOD)的功能及应用[J].新学术,2008(4):322-326):①有明显的防晒效果;②SOD为抗氧酶,能有效防止皮肤衰老、祛斑、抗皱;③有明显的抗炎效果;④对防治皮肤病有一定效果。(4、何献君,梁晓冬,吕晓峰,等.超氧化物歧化酶应用研究状态[J].中国医药指南,2010,8(15):37-39.)
SOD在农业上的应用:研究表明,在植物生长过程中,氧自由基的含量与植物受到的生物及非生物环境胁迫紧密相关。在干旱、强光、霜冻、高盐碱、病害、有毒物质等因素影响下,都会激发体内的ROS,对植物体造成损伤,影响植物的正常生长。许多学者开始研究SOD与植物抗逆性的关系。姜岩岩等研究得出低温敏感性番茄通过提高SOD的活性来适应低温(5、姜岩岩,徐晓飞,郝桂杰,等.夜间低温对栽培和野生番茄果实抗逆性的影响[J].华北农学报,2012,27(6):97-101.),姚冉等在烟草中过表达Cu/Zn-SOD,发现能提高对盐的胁迫性(6、姚冉,李轶女,张志芳,等.Cu/Zn-SOD基因植物表达载体的构建及其在烟草中的表达[J].生物技术通报,2012(11):78-82.)。以及一系列的报道都证明了,SOD的表达量高低与活性的强弱都直接决定了植物的抗逆性。(7、覃鹏,刘飞虎,梁雪妮.超氧化物歧化酶与植物抗逆性[J].黑龙江农业科学,2002(1):31-34.)
SOD在食品工业中的应用:基于SOD优质的保健作用,人们将SOD添加应用于食品中,让人食用后吸收利用,发挥其作用。如SOD草莓(8、魏勇,谷志金,尹逊堂,等.SOD草莓栽培要点[J].农业知识,2004(11).)SOD桃子(9、张洪河.河北乐亭开发出SOD鲜桃[J].山东农业:农村经济版,2002(9):47.)等的发展都很顺利,其营养丰富,具有很好的经济价值。
SOD与医学研究:将5例系统性红斑狼疮和5例系统性硬化症患者的淋巴细胞进行培养,发现其细胞中染色体断裂现象增加,其血清中存在一种基因诱变剂(Clastogenic),在体外试验中证实,后者引起正常人细胞染色体的断裂,但加入SOD后,这种现象即被抑制,连同一些临床观察表明,以SOD治疗红斑狼疮,皮肌炎等免疫性疾病可获得一定疗效。(10、冯高闳.超氧化物歧化酶临床药理研究进展[J].南昌大学学报(医学版),1993(4):102-106.)
艾滋病的辅助治疗:研究表明,艾滋病的发生与一种Clastogenic因子(CFs)有重要关系,该因子在HIV-1基因表达调控中起了重要作用。SOD对艾滋病有一定的治疗作用,SOD能抑制CFs因子从而减除了病毒诱导因素。在感染早期如果能将SOD和其他药物联合使用,将大大延长病毒的潜伏期(11、周赞虎,张海艳,刘仁海,等.人铜锌超氧化物歧化酶基因改良及在聚球藻中表达[J]微生物学报,2006,46(1):147-149.)。AZT(三叠氮二脱氧胸苷)是目前治疗艾滋病的首选药物,但其毒性较强,治疗中往往伴随着氧损伤,从而引起很多组织的细胞功能紊乱,应用上受到较大的限制。若将SOD作为AZT的辅助药应用于艾滋病的治疗,将为艾滋病的治疗提供更广阔的前景。(2、陈鸿鹏,谭晓风.超氧化物歧化酶(SOD)研究综述.经济林研究,2007,25(1):59-65.)。
在病毒性疾病、心肌缺血和缺血再灌流综合症、老年性白内障、心血管疾病、辐射病、癌和癌的放射治疗预防以及人类长寿等领域的研究中也己有突破性进展(12、盛良全,郑晓云,闫向阳,等.生命体中的超氧化物歧化酶(综述)[J].安徽卫生职业技术学院学报,2002,1(2):48-53.13、顾洪雁.猪血铜锌超氧化物歧化酶的纯化及其性质研究[D].华中师范大学,2003.14、魏文树,曾昭全,刘锡钧.超氧化物歧化酶模拟物研究进展[J].药物生物技术,2003,10(4):256-260)
技术实现要素:
本发明的第一个目的是提供Rimicaris exoculata超氧化物歧化酶基因。
本发明的第二个目的是提供Rimicaris exoculata超氧化物歧化酶蛋白序列。
本发明的第三个目的是提供Rimicaris exoculata超氧化物歧化酶的制备方法。
本发明的第四个目的是提供Rimicaris exoculata超氧化物歧化酶的应用。
所述Rimicaris exoculata超氧化物歧化酶基因,其编码Rimicaris exoculata超氧化物歧化酶的核苷酸序列,记作resod。
所述Rimicaris exoculata超氧化物歧化酶基因resod的分子类型为DNA,序列特征:长度为684bp,类型为核酸,链性为双链,拓扑结构为线性,所述Rimicaris exoculata超氧化物歧化酶基因resod的序列如下所示,记为SEQ ID No.1:
所述Rimicaris exoculata超氧化物歧化酶基因,其核苷酸序列采用以下方法获得:提取Rimicaris exoculata的总RNA,反转成cDNA,以合成序列SEQ ID No.3,SEQ ID No.4作为引物,经PCR扩增获得resod基因核苷酸序列SEQ ID No.1。resod基因相关的核苷酸序列SEQ ID No.1通过基因预测获得基因编码的多肽SEQ ID No.2序列。然后通过原核重组表达resod基因,纯化resod基因的表达产物,并通过生物学方法证明resod基因所编码的多肽SEQ ID No.2(不含信号肽)具有超氧化物歧化酶活性;
所述Rimicaris exoculata超氧化物歧化酶包括具有消除超氧根阴离子活性的Rimicaris exoculata超氧化物歧化酶RESOD。
所述合成序列SEQ ID No.3:CGGGATCCCAGCCAAGGGAACGCGGT;(不含信号肽,下划线为核酸内切酶,酶切位点BamHⅠ。)
所述合成序列SEQ ID No.4:CGGAATTCCCTATGCGTAGCCAATAACT;(下划线为核酸内切酶,酶切位点EcoRⅠ。)
所述Rimicaris exoculata超氧化物歧化酶RESOD的分子类型为蛋白质,序列特征:长度为227aa,类型为氨基酸,所述Rimicaris exoculata超氧化物歧化酶RESOD的序列如下所示,记为SEQ ID No.2:
所述Rimicaris exoculata超氧化物歧化酶RESOD,其蛋白具有SEQ ID No.2所示氨基酸序列的多肽;或将SEQ ID No.2氨基酸序列经过一个或多个氨基酸残基的取代、缺失或添加而形成的,且具有与SEQ ID No.2所示的氨基酸序列相似功能的多肽。
所述Rimicaris exoculata超氧化物歧化酶的制备方法,包括以下步骤:
1)Rimicaris exoculata总RNA提取和反转录;
2)Rimicaris exoculata超氧化物歧化酶基因resod的PCR扩增和序列分析;
3)Rimicaris exoculata超氧化物歧化酶RESOD的重组表达与纯化。
本发明通过提取Rimicaris exoculata总RNA,cDNA反转录,引物扩增,克隆和鉴定了超氧化物歧化酶基因resod。通过构建原核重组表达质粒,在大肠杆菌中进行重组蛋白表达,纯化后的蛋白纯度高、活力好,为该蛋白能广泛应用于生物、食品、医药等研究领域奠定基础。
附图说明
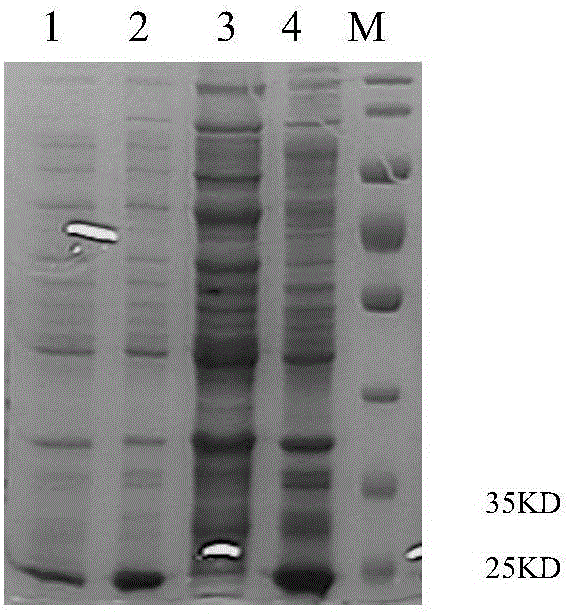
图1为resod基因重组表达的SDS-PAGE电泳图谱。在图1中,M:蛋白Marker;1:重组载体pET-HIS-RESOD在菌株BL21(DE3)的未诱导表达;2:重组载体pET-HIS-RESOD在菌株BL21(DE3)的诱导表达;3:重组质粒pET-HIS-RESOD在菌株BL21(DE3)中诱导表达后超声破碎后上清;4:重组载体pET-HIS-RESOD在菌株BL21(DE3)中诱导表达超声破碎后包涵体。
图2为pET-HIS-RESOD纯化的SDS-PAGE电泳图谱。在图2中,M:蛋白Marker;1:红色箭头所示经镍柱介质纯化所得的目的蛋白RESOD。
图3为温度对重组超氧化物歧化酶RESOD的影响图谱。在图3中,横坐标表示不同温度;纵坐标表示超氧化物歧化酶的相对活性。
图4为pH对重组超氧化物歧化酶RESOD的影响图谱。在图4中,横坐标表示不同pH;纵坐标表示超氧化物歧化酶的相对活性。
具体实施方式
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。
下列实施例中未注明具体条件的实验方法,通常按照常规条件如分子克隆实验室手册(15、萨姆布鲁克,拉塞尔(著),黄培堂(译),《分子克隆实验指南》,科学出版社,2002年,第三版)中所述的实验条件,或按照试剂或仪器生产厂商所建议的条件。
为实现上述目的,本发明采用以下技术措施,其具体步骤是:
1.Rimicaris exoculata总RNA的提取以及反转录合成cDNA:
盲虾Rimicaris exoculata取自于大西洋中脊新发现的热液口(15°9′S,13°21′W),3059m的深海,样品采集后,马上用液氮保存。回实验室后,将盲虾样品(50~100mg),放入装有500μL Trizol(MRC)印管中。在超净台中,研磨均匀,再加入500μLTrizol。4℃,12000g,离心10min,去除沉淀,转移上清至干净RNA-free EP管。加入200μL氯仿,剧烈震荡混匀30s(液体呈乳白色),室温静置10min。4℃,12000g,离心15min。转移上清至干净RNA-FREE EP管。加入500μL异丙醇,上下颠倒混匀,室温放置8min。4℃,12000g离心10min,小心去除上清,保留沉淀。加入75%乙醇700μL,上下颠倒。4℃7500g,离心5min,去除上清。重复步骤2次。在超净台中,开盖放置,使乙醇挥发干净。
加入42.5μL DEPC-H2O,轻弹管壁是溶解彻底。再加入10×DNase I buffer 5μL,DNase I 2μL,RNAase inhibitor 0.5μL。混匀后37℃培养箱反应30min。加入5μL 3M RNA-FREE醋酸钠。加入130μL无水乙醇,-20℃放置1h。4℃,12000g,离心10min。回收沉淀,用75%无水乙醇洗涤,吹干。取20μLDEPC-H20轻弹管壁,溶解,并测RNA浓度。
经电泳检测无降解后进行反转录合成cDNA。取RNA 2μg,加入Oligo(dT)12-18primer 1μL,以DEPC-H20配置到13.5μL。70℃保温10min后,迅速在冰上放置2min。稍离心数秒使混合物聚于管底,加5×M-MLV buffer 4μL,10mM dNTP mixture 1μL,Reverse Transcriptase M-MLV 1μL,RNAase inhibitor 0.5μL。混匀后42℃保温1h。70℃保温15min后,冰上冷却,得到的cDNA即可用于PCR扩增。
2.Rimicaris exoculata超氧化物歧化酶resod基因的PCR扩增和序列分析
以Rimicaris exoculata cDNA为模板,用引物F和R扩增超氧化物歧化酶resod基因(不含信号肽)。
F:CGGGATCCCAGCCAAGGGAACGCGGT(SEQ ID No.3);
R:CGGAATTCCCTATGCGTAGCCAATAACT(SEQ ID No.4);
划线部分GGATCC代表Bam H I酶切位点,GAATTC代表Eco R I酶切位点。
在50μL的反应体系中含0.5μL模板,1μL引物F(10μM),1μL引物R(10μM),4μL dNTP(各2.5mM),10μL 5×PrimerSTARTM Buffer,0.5μL PrimerSTARTM HS DNA Polymerase(2.5U/μL)(TaKARa公司),用H2O将总体积补至50μL。PCR反应条件为:98℃1min,30cycles(98℃10s、55℃30s、72℃50s),72℃5min;4℃保存。PCR产物经纯化后与T载体连接,转化到大肠杆菌感受态细胞Top10中,菌落PCR后选取有DNA片段的阳性克隆测序。
3.Rimicaris exoculata超氧化物歧化酶基因resod的表达与纯化
将含有resod基因的质粒用Bam H I和Eco R I酶切,胶回收resod基因片段,同时将质粒pET-HIS也用相同的酶进行酶切。将两者连接过夜(12~16h)。连接产物转化到大肠杆菌感受态细胞Top10中,提取质粒,测序验证。
将测序正确的质粒pET-HIS-RESOD转化BL21(DE3),37℃生长15h后菌落PCR验证质粒转化的正确性。挑选转化正确的菌落,接种到500mL含有100mg/L氨苄氯霉素的LB培养基中,37℃摇培至A600=0.6时,加入异丙基硫代-β-D-半乳糖苷(IPTG)至终浓度0.4mM,37℃诱导4~5h;将菌液收集至200mL的离心管中,8000g离心10min沉淀细菌细胞;将细菌细胞重新悬浮在20mL Binding Buffer缓冲液(50mM磷酸钠缓冲液,300mM氯化钠,pH=7.4)中,超声波处理至菌液成半透明,再14000rpm离心20min,弃上清;将包涵体沉淀用含8M尿素的Binding Buffer缓冲液混匀溶解;待溶解完全后4000g离心5min,所得上清与平衡后的GE Healthcare Ni sepharose(GE公司)室温结合过夜。纯化过程按照纯化试剂盒说明进行。纯化得到的融合蛋白his-RESOD经10%的SDS-PAGE电泳分析,其分子量约为25kDa,纯度达90%以上(图1和2)。
4.重组超氧化物歧化酶RESOD的酶学性质
4.1、NBT(16、Stewart R R,Bewley J D.Lipid peroxidation associated with accelerated aging of soybean axes[J].Plant Physiology,1980,65(2):245-8.)
法检测超氧化物歧化酶活
用蒸馏水分别配制反应母液:130mM蛋氨酸,7.5μM NBT(氮蓝四唑),100μM EDTA,2mM核黄素母液。核黄素和NBT严格避光。
做3个样品管,实验组为XμL的重组酶纯化产物RESOD与300μL蛋氨酸母液,30μL NBT母液,3μLEDTA母液混合,用缓冲液(默认为50mM磷酸缓冲液pH=7.8)加至2.997mL,最后加入3μL核黄素,混匀,转移至样品试管中,对照组1和2,不加入酶,加入相同体积与酶相同的缓冲液,其余如上。将实验组与对照组1,放在试管架上,距离两个18W荧光灯管约15.5cm位置,照度4000勒克斯反应15~20min,温度低则时间可延迟。对照组2避光处理。取对照组和实验组各200μL,于96孔板上。用酶标仪在吸光值为560nm测定。根据酶活定义,抑制率为50%,即为1U
酶活计算公式为:
4.2 WST-1法(17、Ukeda H,Ashok K S,Kawana D,et al.Flow-Injection Assay of Superoxide Dismutase Based on the Reduction of Highly Water-Soluble Tetrazolium[J].Analytical Sciences,1999,15(4):353-357.)测定超氧化物歧化酶活
采用南京建成公司的总超氧化物歧化酶(SOD)测定试剂盒(WST-1法),按表1分别配制实验组、对照组反应液,于37℃反应20min,用酶标仪在450nm处测定各孔吸光值,酶活计算公式为:
表1 WST-1法测定SOD酶活各组分组成
4.3超氧化物歧化酶RESOD的反应温度
采用WST-1法,将对照、对照空白、测定、测定空白样品按上述方案配制,并作为一个组,共8个组,分别置于4℃、10℃、20℃、30℃、40℃、50℃、60℃和70℃反应30min,用酶标仪检测450nm处吸光值,并换算成酶活,以最高酶活为100%,其余温度下的酶活与最高酶活的比值为比酶活。结果显示,超氧化物歧化酶RESOD的最适温度为10℃(图3)。
4.4超氧化物歧化酶RESOD的最适反应pH
分别配制不同pH的缓冲液NaAc-HAc buffer(pH4,5)、KH2PO4-K2HPO4 buffer(pH 6,7,8)、Glycine–NaOH buffer(pH 9,10),用NBT法测定RESOD在不同pH缓冲液中的酶活。结果表明,超氧化物歧化酶RESOD的最适pH为8.0(图4)。
序列表
<110> 国家海洋局第三海洋研究所
<120> 一种深海来源超氧化物歧化酶及其制备方法
<130> 2017
<160> 4
<170> PatentIn version 3.3
<210> 1
<211> 684
<212> DNA
<213> Rimicaris exoculata
<400> 1
atgatgttgg tattgaggta cacattgtgg gcggcgttgg ctgtcagtgt gggaatgtgt 60
cagccaaggg aacgcggtcg gcacatcgtg tacattaacc acaataatta cccgtcattg 120
ctgtacatca attctgccag tggtgggagt accagtatga acaccagggc agatacttcc 180
gacatcatcc aactcatcct ttacccagct gaacaagatt caaagaggtc tcgtagtgct 240
gtggctgtat taggtggggg tgtcgagggg acgatctact tcactcaaga aaaccctccc 300
ataggaacca ctcgcattga gggtgaaata aataatttga ctcctggtct acatggcttc 360
catatccaca tgtttggcga tctctccgat gggtgtattt cagctggagg tcattacaac 420
ccatacatga gaaaccacgg cggtccagaa cacagagatc gccatgtggg agatttgggc 480
aatattgaag ctgaccaatt tgggcgagcc cttgtcaaca tgaccgatcc cttggtgagt 540
ttggttggtc cgagatcaat cgtgggtcgt tcagtggtcg tgcatgtggg cgaggacgac 600
ttaggaatgg gtggcgatgc cggcagtctc aaaactggga acgctggagg acgagctggt 660
tgtggagtta ttggctacgc atag 684
<210> 2
<211> 227
<212> PRT
<213> Rimicaris exoculata
<220>
<221> SIGNAL
<222> (1)..(20)
<400> 2
Met Met Leu Val Leu Arg Tyr Thr Leu Trp Ala Ala Leu Ala Val Ser
1 5 10 15
Val Gly Met Cys Gln Pro Arg Glu Arg Gly Arg His Ile Val Tyr Ile
20 25 30
Asn His Asn Asn Tyr Pro Ser Leu Leu Tyr Ile Asn Ser Ala Ser Gly
35 40 45
Gly Ser Thr Ser Met Asn Thr Arg Ala Asp Thr Ser Asp Ile Ile Gln
50 55 60
Leu Ile Leu Tyr Pro Ala Glu Gln Asp Ser Lys Arg Ser Arg Ser Ala
65 70 75 80
Val Ala Val Leu Gly Gly Gly Val Glu Gly Thr Ile Tyr Phe Thr Gln
85 90 95
Glu Asn Pro Pro Ile Gly Thr Thr Arg Ile Glu Gly Glu Ile Asn Asn
100 105 110
Leu Thr Pro Gly Leu His Gly Phe His Ile His Met Phe Gly Asp Leu
115 120 125
Ser Asp Gly Cys Ile Ser Ala Gly Gly His Tyr Asn Pro Tyr Met Arg
130 135 140
Asn His Gly Gly Pro Glu His Arg Asp Arg His Val Gly Asp Leu Gly
145 150 155 160
Asn Ile Glu Ala Asp Gln Phe Gly Arg Ala Leu Val Asn Met Thr Asp
165 170 175
Pro Leu Val Ser Leu Val Gly Pro Arg Ser Ile Val Gly Arg Ser Val
180 185 190
Val Val His Val Gly Glu Asp Asp Leu Gly Met Gly Gly Asp Ala Gly
195 200 205
Ser Leu Lys Thr Gly Asn Ala Gly Gly Arg Ala Gly Cys Gly Val Ile
210 215 220
Gly Tyr Ala
225
<210> 3
<211> 28
<212> DNA
<213> unknown
<220>
<223> resod基因扩增正向引物,带有Bam HI酶切位点
<400> 3
CG GGATCC GCATCAACATCTAGTCAACT 28
<210> 4
<211> 27
<212> DNA
<213> Unknown
<220>
<223> resod基因扩增反向引物,带有Eco RI酶切位点
<400> 4
CG GAATTC CTATGCGTAGCCAATAACT 27
- 还没有人留言评论。精彩留言会获得点赞!
- 桑树过氧化物酶MmPOD.12及其应用
- Marinamoeba thermophila生产耐热超氧化物歧化酶(SOD)的方法
- Corynascus thermophilus 生产耐热超氧化物歧化酶(SOD)的方法
- Thermoanaerobacter thermocopriae 生产耐热超氧化物歧化酶(SOD)的方法
- Stilbella thermophila生产耐热超氧化物歧化酶(SOD)的方法
- Thermoflavimicrobium dichotomicum生产耐热超氧化物歧化酶(SOD)的方法
- 普通嗜热放线菌生产耐热超氧化物歧化酶(sod)的方法
- 中间型嗜热放线菌生产耐热超氧化物歧化酶(sod)的方法
- Spirochaeta thermophila生产耐热超氧化物歧化酶(SOD)的方法
- Platyamoeba thermophila生产耐热超氧化物歧化酶(SOD)的方法
- 一种储片盒换气歧管的制作方法
- 一种能增加氧化反应速度的化工试剂反应釜结构的制作方法
- 标志物及其应用、试剂盒、该标志物的检测方法
- Marinamoeba thermophila生产耐热超氧化物歧化酶(SOD)的方法
- Corynascus thermophilus 生产耐热超氧化物歧化酶(SOD)的方法
- Thermoanaerobacter thermocopriae 生产耐热超氧化物歧化酶(SOD)的方法
- Stilbella thermophila生产耐热超氧化物歧化酶(SOD)的方法
- 普通嗜热放线菌生产耐热超氧化物歧化酶(sod)的方法
- 中间型嗜热放线菌生产耐热超氧化物歧化酶(sod)的方法
- Spirochaeta thermophila生产耐热超氧化物歧化酶(SOD)的方法
- 月桂酸修饰超氧化物歧化酶的制备方法
- Marinamoeba thermophila生产耐热超氧化物歧化酶(SOD)的方法
- Corynascus thermophilus 生产耐热超氧化物歧化酶(SOD)的方法
- Thermoanaerobacter thermocopriae 生产耐热超氧化物歧化酶(SOD)的方法
- Stilbella thermophila生产耐热超氧化物歧化酶(SOD)的方法
- Thermoflavimicrobium dichotomicum生产耐热超氧化物歧化酶(SOD)的方法
- 普通嗜热放线菌生产耐热超氧化物歧化酶(sod)的方法
- 中间型嗜热放线菌生产耐热超氧化物歧化酶(sod)的方法
- Spirochaeta thermophila生产耐热超氧化物歧化酶(SOD)的方法
- Platyamoeba thermophila生产耐热超氧化物歧化酶(SOD)的方法
- Thermoleophilum minutum生产耐热超氧化物歧化酶(SOD)的方法
- Cryptococcus thermophilus生产耐热超氧化物歧化酶(SOD)的方法
- Thermobispora bispora生产耐热超氧化物歧化酶(SOD)的方法
- Thermoleophilum album生产耐热超氧化物歧化酶(SOD)的方法
- Talaromyces thermo-citrinum 生产耐热超氧化物歧化酶(SOD)的方法
- Moorella thermoautotrophica 生产耐热超氧化物歧化酶(SOD)的方法
- Lagenidium thermophilum生产耐热超氧化物歧化酶(SOD)的方法
- Thermoanaerobacterium thermosulfurigenes生产耐热超氧化物歧化酶(SOD)的方法
- Calcarisporiella thermophila生产耐热超氧化物歧化酶(SOD)的方法
- Crithidia luciliae thermophila生产耐热超氧化物歧化酶(SOD)的方法