CRISPR/Cas9技术介导miR167前体序列编辑体系的建立方法与流程
本发明涉及到利用crispr/cas9技术对番茄、柑橘等植物mir167前体序列进行编辑的方法的建立。
背景技术:
crispr/cas9技术作为一门新兴的基因编辑技术,可以实现在基因组水平上对目标基因定点修饰,产生的突变可以遗传,近些年来逐渐成为分子生物学研究的热点。crispr/cas9技术的基本原理是设计一段grna序列,与cas9蛋白形成核酸蛋白复合体,该复合体可以在体内或者体外对靶标基因的特定位点进行切割,造成双链dna断裂,在dna自我修复过程中引入碱基的缺失或者插入。crispr/cas9技术因其载体组装简便、通用性强以及使用门槛低等特点已经在动植物中得到广泛应用。目前用于研究的crispr/cas9载体有很多,其基本结构单元是grna和cas9,还有一些标记筛选位点。grna设计的严谨性是该技术成功的关键点之一,不同的靶位点之间突变效率有很大差异,因此选择合适的靶位点尤为重要。目前已经有多个在线网站提供植物grna的设计,但网站设计有一定的缺陷,不能准确得到合适的grna。关于crispr/cas9基因组编辑突变的检测方法也比较多,大多数是应用限制性内切酶和pcr测序相结合的方法检测突变效率,其工作量大且成本高。
现有技术中,grna的设计方法是直接采用在线软件设计,输入一段长度为23-5000bp的序列,软件自动找出序列上所有能够设计grna的靶标位点并按照打分高低排列,根据软件给出的评分,选择评分较高的grna序列用于构建crispr/cas9载体。载体的结构:一般包含grna和cas9,grna的启动子有常用的u6或u3启动子以及物种特异性启动子,cas9大多数使用camv35s启动子驱动。除此之外载体上还含有抗性筛选标记。这两张方法存在的问题是:1、直接使用在线软件设计grna,不同软件的评分标准不同,评分排名会有变化,依据此方法得到的评分较高的grna也会不一样,设计出的grna在体内可能与靶位点的结合能力较弱,这对后续靶位点的编辑存在很大影响。因此仅用软件设计grna后随即选择得分较高的grna构建载体,后续的基因编辑成功率得不到保障。载体在转染植株后,有可能不会发生编辑,这主要是grna在设计后缺乏有效验证。2、载体结构中若只含有抗性筛选标记,后续的检测工作较麻烦,不能更有效的鉴定载体是否成功转入到植株中。
技术实现要素:
本发明基于上述情况,改进相关关键点,利用crispr/cas9技术成功编辑了番茄的mir167前体序列。其中的grna设计方面采用在线软件设计和体外酶切检测相结合的方法,同时在载体上插入一个cfp荧光蛋白位点,便于后期对突变体植株的检测和筛选。
为实现上述目的,本发明采用的技术方案是:一种crispr/cas9技术介导mir167前体序列编辑体系的建立方法,其特征在于,步骤包括:
a、grna设计和体外验证:利用在线软件设计后选择评分较高的多个靶标位点,序列合成后使用体外酶切检测的方法来筛选合适的grna,通过t7rna聚合酶将软件设计出的靶标位点序列在体外转录生成grna,再利用cas9核酸酶体外切割的方法来验证grna-cas9的体外切割效率,从而筛选出目的grna;
b、载体构建:构建crispr/cas9表达系统的两个载体pp1c.5和pp1c.1
通过puc19载体改造pp1c.5载体,使其含有atu6启动子驱动的grna插入位点和sgrnascaffold;通过pcambia1302载体改造pp1c.1载体,使其含有camv35s启动子驱动的cas9,同时含有hptⅱ潮霉素筛选位点,并将潮霉素筛选位点替换成卡那霉素筛选位点,并加上cfp荧光蛋白的基因,重新命名为pp1c.1c。
进一步的,步骤(1)中,grna体外验证中,所用的扩增引物,上游引物t7-grna-fpg:taatacgactcactatagnnnnnnnnnnnnnnnnnnnngttttagagctagaaatagc,下游引物grna-rp:agcaccgactcggtgccactt;其中nnnnnnnnnnnnnnnnnnnn是grna靶点,不带pam序列。
本发明的有益技术效果是:根据先前所述,我们应用两种不同的grna设计方法,筛选出grna后构建同一类型的crispr/cas9敲除载体,再进行番茄的遗传转化试验,对两者产生的突变效果进行对比。其中直接使用在线软件设计grna后构建载体转化植株,共转化外植体材料(子叶)数238个,产生愈伤组织数121个,未得到突变植株。而改进技术后利用在线软件设计多个靶标位点,再使用体外酶切检测的方法来筛选到合适的grna,进行载体构建和遗传转化,共转化子叶数202个,产生愈伤组织数104个,鉴定到3个突变体材料。说明使用软件设计结合体外检测的方法更容易得到合适的grna,且明显提高转基因植株发生突变的成功率。
与此同时我们也进行了不同类型的crispr/cas9载体敲除效果的比较。其中一种类型的载体为pkse401载体,该载体含有atu6启动子驱动的grna和camv35s启动子驱动的cas9,同时含卡那霉素抗性筛选标记,但无荧光蛋白和gus位点,在转基因植株筛选阶段只能依靠抗性筛选,且灵敏度不高,突变效果也不明显,假阳性居多。另一种类型的载体是pp1c.1c,构建好的载体上含有atu6启动子驱动的grna与camv35s启动子驱动的cas9,且含有卡那霉素抗性筛选位点和cfp荧光蛋白位点,在转基因植株的筛选阶段比较方便,只需用蓝光激发光照射转基因再生芽,即可初步筛选到转化成功的植株,后续再对含有荧光标记的植株进行dna测序,大大降低了转基因植株检测的工作量,提高获得突变植株的效率。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动性的前提下,还可以根据这些附图获得其他的附图。
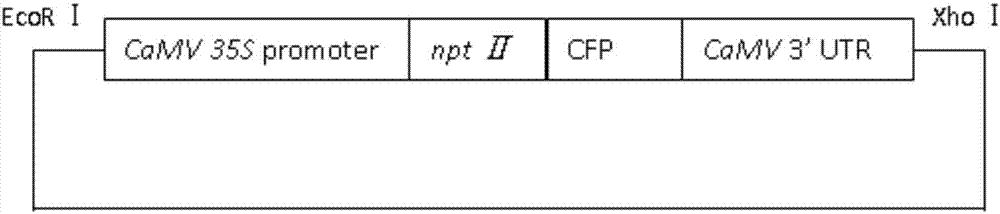
图1是本发明中puc19载体改造pp1c.1载体结构示意图;
图2是本发明中pcambia1302载体改造d130载体结构示意图;
图3是本发明中构建好的crispr/cas9重组载体结构示意图;
图4本发明的实施例,应用本发明构建的载体遗传转化番茄,得到再生植株的荧光标记检测(g1-1、g1-2、g1-3分别表示三个不同的再生愈伤组织);
图5转基因再生愈伤组织g1-1的基因型检测结果(图中可看出靶标区域发生了碱基缺失);
图6转基因再生愈伤组织g1-2的基因型检测结果(图中可看出靶标区域分别发生了碱基缺失和碱基插入);
图7转基因再生愈伤组织g1-3的基因型检测结果(图中可看出靶标区域分别发生了碱基缺失和碱基插入)。
具体实施方式
下面将结合本发明实施例,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
实施例1
一种crispr/cas9技术介导mir167前体序列编辑体系的建立方法,步骤包括:
a、grna设计和体外验证:利用在线软件设计后选择评分较高的多个靶标位点,序列合成后使用体外酶切检测的方法来筛选合适的grna,通过t7rna聚合酶将软件设计出的靶标位点序列在体外转录生成grna,再利用cas9核酸酶体外切割的方法来验证grna-cas9的体外切割效率,从而筛选出目的grna;
具体操作如下:
1、在线软件设计grna
将拟敲除靶标基因的dna片段输入在线软件,按照软件运行结果,根据得分高低给出的grna参考序列,选择评分较高的几个grna,进行体外酶切验证试验,筛选更合适的grna。
2、grna体外检测与筛选
2.1体外转录grna
(1)引物合成,如表1所示
表1grna体外扩增引物
(注:其中nnnnnnnnnnnnnnnnnnnn是grna靶点,不带pam序列)
(2)pcr扩增
使用t7-grna-fpg和grna-rp引物对,以标准grna模板为模板进行pcr扩增(pcr产物120bp),反应体系参照表2.4,每个样品需扩增3管。
表2grna体外扩增的pcr反应体系
pcr反应程序如下:98℃10s,58℃15s,72℃2s,35个循环。
(3)pcr产物的检测和纯化回收
①同一样品的3管pcr产物混合到一个管中,取混合后的3μl在1.3%琼脂糖凝胶上电泳检测pcr结果。
②检测出目的条带后使用pcr产物纯化试剂盒(takaraminibestdnafragmentpurificationkitver.4.0)进行纯化回收,回收的pcr产物使用40μldepc水溶解,并使用nanodrop2000检测浓度(最好为70ng/μl以上),将其作为后续体外转录的dna模板。
(4)grna的转录
表3grna转录反应体系
将以上混合液置于37℃金属浴中反应2h,反应结束后,加入2μldnasei,继续37℃反应30min。
(6)grna的转录产物纯化回收
①向转录反应结束后的混合液中加入115μldepc水、15μlstopsolution混匀后,再加入2倍体积的无水乙醇(注意无rnase污染),充分混匀后,置于-20℃冻存过夜。
②以上液体在4℃条件下13000rpm离心20min,去除上清保留沉淀。
③加入300μl的70%冷乙醇清洗(用depc水配制),4℃条件下13000rpm离心15min,去除上清保留沉淀,继续4℃条件下13000rpm离心1min,小心地吸净离心管中残留的液体,室温晾置1-2min后,加入40μldepc水溶解离心管的rna沉淀。
④检测rna浓度,并取1μl进行1.2%琼脂糖凝胶电泳检测,来判断条带亮度是否和所测浓度相符,确定浓度正确后可直接用于后续的spcas9/grna酶切实验,或者-80℃保存待用。
2.2扩增目标基因的基因组dna片段
①以植物基因组dna为模板,pcr扩增出带有靶标位点的dsdna片段。
②pcr产物纯化回收后检测浓度。
2.3体外酶切反应
①按照表4反应体系中的次序加样,准备酶切反应。
表4酶切反应体系
以上液体充分混合后,置于37℃反应30min,反应结束后加入3μldnaloadingbuffer,混合后置于65℃,5min。
②用2%琼脂糖凝胶电泳检测分析酶切结果。
b、载体构建:构建crispr/cas9表达系统的两个载体pp1c.5和pp1c.1
通过puc19载体改造pp1c.5载体,使其含有atu6启动子驱动的grna插入位点和sgrnascaffold;通过pcambia1302载体改造pp1c.1载体,使其含有camv35s启动子驱动的cas9,同时含有hptⅱ潮霉素筛选位点,并将潮霉素筛选位点替换成卡那霉素筛选位点,并加上cfp荧光蛋白的基因,重新命名为pp1c.1c。
pp1c.1载体改造具体操作如下:
pp1c.1载体是根据pcambia1302改造而来,含有潮霉素抗性hptⅱ基因,简单示意如图1下所示。
d130载体(本实验室保存)也是根据pcambia1302改造而来,在相同的位置上d130载体含有卡那霉素抗性位点nptⅱ基因和cfp荧光蛋白基因。简单示意如图2下所示。
(1)双酶切后回收片段
分别用ecorⅰ和xhoⅰ双酶切pp1c.1质粒和d130质粒,酶切后分别回收目的片段,pp1c.1回收大片段(>10kb),d130回收小片段(2.6kb)。
(2)连接
用takara公司的t4dna连接酶将回收到的两个目的片段连接起来。
(3)连接产物转化大肠杆菌
①取已灭菌的1.5ml离心管置于冰上,大肠杆菌感受态细胞从-80℃拿出后同样置于冰上融化。
②向离心管中加入已融化的大肠杆菌感受态细胞33μl,再加10μl的连接产物,轻轻混匀,冰浴30min;
③冰浴结束后快速取出离心管,放入42℃水浴中热激45s;
④热激结束后立即取出置于冰上冰浴2min(注意不要剧烈晃动离心管);
⑤在无菌条件下,向离心管中加入800μl的lb液体培养基,37℃条件下220r/min振荡培养45min;
⑥培养结束后的菌液5000rpm离心5min收集菌体沉淀,用移液枪将上清液吸出一部分后预留100μl左右,轻轻吹打重悬菌体;
⑦将重悬液加到含kana抗性的lb固体培养基平板上,用无菌涂布器将细胞均匀涂开,直至液体完全吸收,将平板倒置于37℃过夜培养。
(2)单克隆检测
①取无菌的1.5ml离心管,向其中加入400μl含kan和rif抗性的lb液体培养基,挑取边缘清晰光滑的圆形单克隆菌落,接种于上述lb液体培养基中,28℃220r/min振荡培养过夜,取部分菌液做pcr检测。检测结果为阳性的菌液加入60%甘油后置于-80℃保存,命名为pp1c.1c。
构建好的crispr/cas9重组载体如图3所示。
实施例2
使用本技术中的方法对番茄mir167a的前体序列loc101259485基因进行编辑,通过软件设计结合体外检测筛选到一个合适的grna-g1,并且将其连接到含有荧光标记的pp1c.1c载体中,进行载体的构建和番茄的遗传转化实验。共转化番茄子叶数202个,产生愈伤组织数104个,其中有荧光标记的愈伤组织5个,经过筛选后得到3个有较明显愈伤组织的材料,分别将他们命名为g1-1、g1-2和g1-3(图4)。
提取三个材料的基因组dna后用pcr的方法进行检测,检测结果表明载体已成功转入植物愈伤组织后,再使用植物基因组dna引物和高保真酶扩增包含靶标位点的区域,将pcr产物进行直接测序,通过与野生型植株(wt)和转对照载体的转基因材料(ck)进行对比,发现g1-1、g1-2和g1-3的测序结果中靶标区域均出现多峰现象。将出现多峰的样品重新用植物基因组dna引物和高保真酶扩增包含靶标位点的区域,回收目的片段并连接到pmd19-t载体上,转化大肠杆菌后挑取单克隆检测,对阳性菌液进行测序分析。通过与野生型植株(wt)靶标区域dna比对分析后发现,g1-1、g1-2和g1-3均为突变体材料,且发生不同程度的碱基突变,测序结果显示g1-1、g1-2和g1-3中分别有不同类型的碱基突变,主要是单碱基插入和碱基缺失。
其中g1-1突变体共检测了30个单克隆,其中有5个发生碱基突变(m-8、m-15、m-23、m-26和m-30),主要的突变类型是碱基缺失,缺失碱基数4-6bp不等,如图5所示。
g1-2突变体共检测了26个单克隆,其中有9个发生碱基突变(m-1、m-2、m-3、m-9、m-15、m-17、m-19、m-21和m-22),主要的突变类型是碱基插入和碱基缺失,缺失碱基数1-33bp不等,并且产生了大片段缺失现象(m-17缺失33bp),而插入的碱基数目为1bp,即单碱基插入,如图6所示。
g1-3突变体共检测了30个单克隆,其中有8个发生碱基突变(m-9、m-12、m-14、m-16、m-17、m-23、m-26和m-28),主要的突变类型是碱基插入和碱基缺失,缺失碱基数为6bp,插入碱基数为1bp,如图7所示。
以上结果表明,应用此方法构建crispr/cas9敲除载体并转化番茄,可以有效的筛选到发生碱基突变的株系,且方法简单,突变效率高,试验周期短,可快速获得突变型植株,为未知基因功能的研究以及作物新品种的创制提供基础。
例仅用以说明而非限制本发明的技术方案,尽管参照上述实施例对本发明进行了详细说明,本领域的普通技术人员应该理解:依然可以对本发明进行修改或者等同替换,而不脱离本发明的精神和范围的任何修改或局部替换,其均应涵盖在本发明的权利要求范围当中。
- 还没有人留言评论。精彩留言会获得点赞!