多重扩增子测序用引物系统、其应用以及测序文库的构建方法与流程
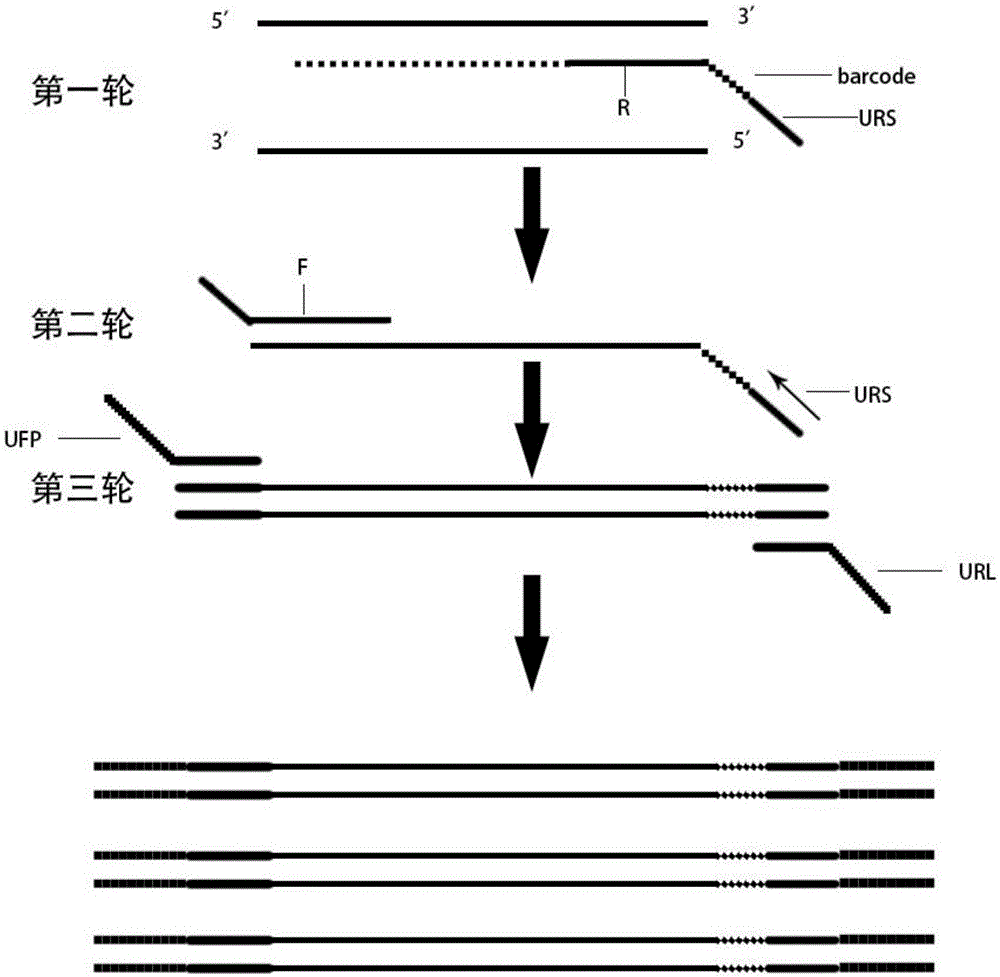
本发明涉及分子生物学领域,尤其是涉及一种多重扩增子测序用引物系统、其应用以及测序文库的构建方法。
背景技术:
高通量测序(又称下一代测序,ngs)技术在发现和转化研究的诸多方面已逐渐成为一种被广泛采用的技术,目前其主要应用于基因组dna的变异分析和rna的表达分析。这些分析的范围可以是整个基因组和转录组,也可以是特定的区域或多个基因组合的靶向测序。靶向测序尤其有利于实现对感兴趣区域的高覆盖,同时可以控制测序成本和数据判读的复杂性。测序覆盖度高对于癌症等低频突变尤为重要,例如,对于检测单个核苷酸变异(snvs)的平均序列深度超过1000次,有比较好的可信度,但是对于小于5%的核苷酸变异,则需要更高的测序深度来检测。
富集目标区域的方法有很多种,最常用的方法是:1)利用目标特异的探针从样本dna中提取出杂交捕获产物;2)利用特定的引物扩增富集样本dna。虽然在引物设计和pcr反应优化方面还需要付出更多的努力,但因pcr过程更容易处理、需要总体时间较少、目标富集更为具体和明确、并且能够适应更低的起始dna浓度等,目前多数仍然采用基于pcr扩增来富集扩增子。
随着高多重pcr的出现,现在成千上万的扩增子可以同时扩增。然而,现有目标富集、文库准备和测序步骤等所有利用dna聚合酶的扩增过程,都会引入非均一扩增和错配(聚合酶错误生成序列变化)。pcr非均一性扩增对精确定量有很大的影响,因为最后的测序读数可能不会准确地代表原始dna或者rna片段的相对丰度。聚合酶在pcr扩增循环中将最有可能导致许多失误(“false”),这些低水平的变异易导致难以识别真正的低频率的突变。这些问题的根源是在pcr过程中相同的引物分子对不同的初始原始分子的多次采样。这些问题在需要更多的pcr循环处理低起始量的dna或质量差的dna时会加剧。
技术实现要素:
基于此,有必要提供一种有利于提高结果精确度的多重扩增子测序用引物系统、其应用以及测序文库的构建方法。
本发明解决上述技术问题的技术方案如下。
一种多重扩增子测序用引物系统,包括第一轮反向特异性引物组和第二轮正向特异性引物组;其中,
所述第一轮反向特异性引物组包括多个不同的第一轮反向特异性引物,每个所述反向特异性引物从5’端至3’端分别为反向接头序列、条形码序列和反向特异性序列,所述第一轮反向特异性引物组中的多个不同的第一轮反向特异性引物的条形码序列不同,且所述第一轮反向特异性引物组中的多个不同的第一轮反向特异性引物的反向特异性序列分别与待测目标区域的不同区段互补;
所述第二轮正向特异性引物组包括多个不同的第二轮正向特异性引物,每个所述第二轮正向特异性引物从5’端至3’端分别为正向接头序列和正向特异性序列,所述第二轮正向特异性引物组中的多个不同的第二轮正向特异性引物的正向特异性序列分别与待测目标区域的不同区段互补。
在其中一个实施例中,所述反向接头序列和/或所述正向接头序列的长度为18~24mer,优选为21mer。
在其中一个实施例中,所述条形码序列的长度为6~12mer,优选为10mer。
在其中一个实施例中,所述多重扩增子测序用引物系统还包括第二轮反向通用引物,所述第二轮反向通用引物的3’端含有所述反向接头序列。
在其中一个实施例中,所述多重扩增子测序用引物系统还包括第三轮正向测序接头引物和第三轮反向测序接头引物,所述第三轮正向测序接头引物的3’端含有所述正向接头序列,所述第三轮反向测序接头引物的3’端含有所述第二轮反向通用引物的序列。
在其中一个实施例中,所述第三轮反向测序接头引物的序列中含有索引序列,所述第三轮反向测序接头引物也有多个,各个所述第三轮反向测序接头引物中的所述索引序列不同。
上述任一实施例所述的多重扩增子测序用引物系统在制备高通量测序的引物试剂中的应用。
一种多重扩增子测序文库的构建方法,使用上述任一实施例所述的多重扩增子测序用引物系统,所述多重扩增子测序方法包括如下步骤:
用所述第一轮反向特异性引物组对含待测目标区域的模板dna进行第一轮单引物pcr扩增,第一轮单引物pcr扩增只进行一个循环,之后进行纯化处理,得到多个第一轮产物片段,各个所述第一轮产物片段带有不同的所述条形码序列;
以所述第二轮正向特异性引物组以及3’端含有所述反向接头序列的第二轮反向通用引物对纯化后的第一轮单引物pcr扩增的产物进行第二轮pcr扩增,并对所述第二轮pcr扩增的产物进行纯化处理,得到多重扩增子;
对纯化后的第二轮pcr扩增的产物以3’端含有所述正向接头序列的第三轮正向测序接头引物和3’端含有所述第二轮反向通用引物的序列的第三轮反向测序接头引物进行第三轮pcr扩增,纯化后得到用所述条形码序列标记的目标区域的测序文库。
在其中一个实施例中,所述多重扩增子测序文库的构建方法还包括在所述第一轮单引物pcr扩增之前以及在所述第二轮pcr扩增之前,分别将所述第一轮反向特异性引物组和所述第二轮正向特异性引物组分成多个小组的步骤,每个第一轮反向特异性引物小组含多种不同的第一轮反向特异性引物,且每个第二轮正向特异性引物小组含多种不同的第二轮正向特异性引物组,后续所述第一轮单引物pcr扩增和所述第二轮pcr扩增均分别使用不同小组的引物进行独立扩增,并在进行第三轮pcr扩增之前,将多个小组的第二轮pcr扩增的产物进行混合后再进行第三轮pcr扩增。
在其中一个实施例中,在对第一轮单引物pcr扩增的产物进行纯化处理时是进行至少两次磁珠纯化;
在对第二轮pcr扩增的产物以及第三轮pcr扩增的产物进行纯化是使用磁珠纯化和筛选。
在其中一个实施例中,用于各pcr扩增反应的酶试剂可以是2×pcrbufferforkod-multi&epi-、kod-multi&epi-(1.0u/μl)。含条形码序列的第一轮反向特异性引物组的各引物混合浓度为25~250pmoles/μl,优选50pmoles/μl,作为第一轮单引物pcr扩增反应的引物。
在其中一个实施例中,第二轮正向特异性引物组中各引物的混合浓度为25~250pmoles/μl,优选50pmoles/μl;纯化用磁珠:vahtstmdnacleanbeadsillumina。
在其中一个实施例中,第一轮单引物pcr扩增的条件为:98℃2分钟、58℃15分钟、68℃15分钟、68℃7分钟;pcr扩增体系:2×pcrbufferforkod-multi&epi-5μl、kod-multi&epi-(1.0u/μl)0.2μl、第一轮反向特异性引物组混合液1μl、dna模板10-200ng,双蒸水补至10μl;用1.0x磁珠进行两次纯化,纯化后的洗脱体积为10μl。
在其中一个实施例中,第二轮pcr扩增条件为:94℃5分钟,(98℃10秒、58℃15分钟、68℃15分钟)共3个循环,68℃5分钟;pcr扩增体系:2×pcrbufferforkod-multi&epi-12.5μl、kod-multi&epi-(1.0u/μl)0.5μl、第二轮正向特异性引物组混合液0.5μl,第二轮通用反向引物(urs)1.5μl、磁珠纯化两次后的第一轮单引物pcr产物10μl。
在其中一个实施例中,第三轮pcr扩增的条件为:94℃3分钟,(98℃10秒、60℃15秒、68℃30秒)共22个循环、68℃5分钟;pcr扩增体系:2×pcrbufferforkod-multi&epi-25μl、kod-multi&epi-(1.0u/μl)1μl、illumina用的第三轮正向测序接头引物(ufp)1μl、illumina用的第三轮反向测序接头引物(url)1μl、磁珠纯化筛选后的第二轮pcr扩增产物22μl;
在其中一个实施例中,还包括将所构建的测序文库经agilentbioanalyzer2100和qubit3.0检测合格后采用illuminanextseq500测序仪进行测序分析的步骤。
本发明的多重扩增子测序用引物系统、其应用以及测序文库的构建方法通过在多重扩增子测序过程中引入分子层面的条形码序列(以下也简称为分子条形码),用带含条形码序列的第一轮反向特异性引物和含目标区域的目标dna作为模板进行第一轮单引物pcr扩增,含条形码序列的第一轮反向特异性引物经过退火与延伸分配到目标dna分子上,再对第一轮单引物pcr扩增产物进行第二轮pcr扩增,引物为不含条形码序列的第二轮正向特异性引物和第二轮反向通用引物,此时每个扩增子都已经引入了条形码序列,最后用第三轮正向测序接头引物和第三轮反向测序接头引物对第二轮pcr扩增的产物进行第三轮pcr扩增,得到用分子条形码序列标记的目标区域的测序文库。
因为只是在第一轮单引物pcr扩增反应时引入了条形码序列,也即对原始dna分子只进行一次标记,通过计算读数中唯一的条形码序列数量,而不是计算总读数的数量,可以更好地对目标量进行准确定量。分子条形码序列提供了一种很好的方法来跟踪错配,利用下游分析,聚合酶在pcr过程中产生的错配可以与原始分子中出现的序列变异区分开来,最后通过去除低水平的假阳性来提高1%或更低的突变的检测准确率。通过计算读数中唯一的分子条形码序列数量,而不是计算总读数的数量,也可以更好地实现目标量化,因为总读数更有可能被非均一性扩增的所误读。
此外,本发明的测序文库的构建方法免去了一般建库需要加a、加接头等步骤,只需要一种聚合酶,三步pcr反应就可以完成文库构建,具有低成本、方便快捷等优点。
附图说明
图1为多重扩增子测序文库的构建流程示意图;
图2为所构建的测序文库经agilentbioanalyzer2100的检测结果。
具体实施方式
为了便于理解本发明,下面将参照相关附图对本发明进行更全面的描述。附图中给出了本发明的较佳实施例。但是,本发明可以以许多不同的形式来实现,并不限于本文所描述的实施例。相反地,提供这些实施例的目的是使对本发明的公开内容的理解更加透彻全面。
除非另有定义,本文所使用的所有的技术和科学术语与属于本发明的技术领域的技术人员通常理解的含义相同。本文中在本发明的说明书中所使用的术语只是为了描述具体的实施例的目的,不是旨在于限制本发明。本文所使用的术语“和/或”包括一个或多个相关的所列项目的任意的和所有的组合。
下面通过具体实施例结合附图对本发明作进一步详细说明。
本实施例所使用的kod-multi&epi-酶体系和对应的扩增反应体系只是作为一个具体的可选的实施例来进行,不能认定本发明的具体实施只局限于这些酶体系及其对应的扩增反应体系。本实施例适用于各种目标区域的扩增,所使用的h1975细胞系也只是作为一个具体的可选实施例来详细描述本发明的多重扩增子测序用引物系统、其应用以及测序文库的构建方法的详细技术方案。
1、提取人全血dna和肺癌h1975细胞系dna,人全血dna作为阴性样本(n),h1975为阳性样本(p)。用qubit3.0荧光定量仪准确定量dna样本浓度。
2、按照表1中的比例进行混合,最后将浓度稀释为25ng/μl~80ng/μl之间。
表1
3、引物稀释与混合
含条形码序列(以下简称为barcode)的第一轮反向特异性引物组一(分a/b管):ra/b-barcode-mix,混合浓度50pmoles/μl,相对应的,第二轮正向特异性引物组二(分a/b管):fa/b–nonbarcode-mix,混合浓度为50pmoles/μl。具体见下表2。
表2
第二轮通用引物urs:10pmoles/μl。第三轮正向测序接头引物ufp:10pmoles/μl;第三轮反向测序接头引物url:10pmoles/μl。具体见下表3。
表3
4、文库构建
4.1第一轮单引物pcr扩增
第一轮单引物pcr扩增反应体系配制,将同一样本分成a、b两管进行反应。
1)a管反应体系如下:
2)b管反应体系如下:
a管与b管的反应条件如下:
磁珠纯化第一轮反应pcr产物两次,具体如下:
1)将vahtstmdnacleanbeads提前30min从4~8℃取出,静置使其温度平衡至室温。
2)分别将a管、b管的第一轮单引物pcr反应的产物,补40μlnuclease-freewater至体积50μl。
3)涡旋振荡vahtstmdnacleanbeads充分混匀,向1.5ml低吸附ep管中50μl(1.0x)vahtstmdnacleanbeads,分别转移a管、b管第一轮单引物pcr反应的产物至ep管中,盖上盖子,低速涡旋振荡混合均匀。
4)室温静置5min。
5)将管置于磁力架上,直至液体澄清(约5min),小心吸去上清,不要吸到磁珠。
6)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠。
7)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠,盖上盖子,短暂离心,用10μl枪头将残留液体吸取,不要吸到磁珠。
8)室温晾干3min(不要让磁珠晾干至出现裂痕),管保持在磁力架上。
9)从磁力架上取下ep管,加入52μlnuclease-freewater。盖上盖子,低速涡旋振荡混合均匀,室温静置2min,将管置于磁力架上直至液体澄清。
10)小心转移50μl上清至1.5ml低吸附ep管中,不要吸到磁珠,丢弃磁珠。
11)向上清中加入50μl(1.0x)涡旋振荡后的vahtstmdnacleanbeads,盖上盖子,低速涡旋振荡混合均匀。
12)室温静置5min。
13)将管置于磁力架上,直至液体澄清(约5min),小心吸去上清,不要吸到磁珠。
14)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠。
15)加入200μll80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠。盖上盖子,短暂离心,用10μl枪头将残留液体吸取,不要吸到磁珠。
16)室温晾干3min(不要让磁珠晾干至出现裂痕),管保持在磁力架上。
17)从磁力架上取下ep管,加入11μlnuclease-freewater。盖上盖子,低速涡旋振荡混合均匀,室温静置2min,将管置于磁力架上直至液体澄清。
18)小心转移10μl上清至0.2mlpcr反应管中,不要吸到磁珠,丢弃磁珠。
4.2第二轮pcr扩增
第二轮pcr扩增反应体系配制,反应体系如下:
将第一轮pcr扩增反应的a、b分别进行反应。
1)a管反应体系如下:
2)b管反应体系如下:
反应程序如下(pcr仪热盖设置温度为105℃):
磁珠纯化第二轮pcr扩增的产物及片段大小的筛选,具体如下:
1)将vahtstmdnacleanbeads提前30min从4~8℃取出,静置使其温度平衡至室温。
2)将第二轮pcr扩增反应同一样本的a、b两管pcr产物混合总体积50μl(25μl第二轮反应a管pcr产物+25μl第二轮反应b管pcr产物)。
3)涡旋振荡vahtstmdnacleanbeads充分混匀,向1.5ml低吸附ep管中50μl(1.0x)vahtstmdnacleanbeads,转移50μla、b两管混合pcr产物至ep管中,盖上盖子,低速涡旋振荡混合均匀。
4)室温静置5min。
5)将管置于磁力架上,直至液体澄清(约5min),小心吸去上清,不要吸到磁珠。
6)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠。
7)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠,盖上盖子,短暂离心,用10μl枪头将残留液体吸取,不要吸到磁珠。
8)室温晾干3min(不要让磁珠晾干至出现裂痕),管保持在磁力架上。
9)从磁力架上取下ep管,加入102μlnuclease-freewater。盖上盖子,低速涡旋振荡混合均匀,室温静置2min,将管置于磁力架上直至液体澄清。
10)小心转移100μl上清至1.5ml低吸附ep管中,不要吸到磁珠,丢弃磁珠。
11)向上清中加入82μl(0.82x)涡旋振荡后的vahtstmdnacleanbeads,盖上盖子,低速涡旋振荡混合均匀。
12)室温静置5min。
13)将管置于磁力架上,直至液体澄清(约5min)。小心吸取178μl上清至1.5ml低吸附ep管中(不要吸到磁珠)。
14)向上清中加入20μl(0.2x)涡旋振荡后的vahtstmdnacleanbeads,盖上盖子,低速涡旋振荡混合均匀。
15)室温静置5min。
16)将管置于磁力架上,直至液体澄清(约5min),小心吸去上清,不要吸到磁珠。
17)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠。
18)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠。盖上盖子,短暂离心,用10μl枪头将残留液体吸取,不要吸到磁珠。
19)室温晾干3min(不要让磁珠晾干至出现裂痕),管保持在磁力架上。
20)从磁力架上取下ep管,加入22μlnuclease-freewater。盖上盖子,低速涡旋振荡混合均匀,室温静置2min,将管置于磁力架上直至液体澄清。
21)小心转移20μl上清至0.2mlpcr反应管中,不要吸到磁珠,丢弃磁珠。
4.3第三轮pcr扩增反应
第三轮反应体系配制如下:
注:url*为index,每个样本对应1个url。具体见下表4。
表4
反应程序如下(pcr仪热盖设置温度为105℃):
磁珠纯化文库及文库片段大小的筛选,具体如下:
1)将vahtstmdnacleanbeads提前30min从4~8℃取出,静置使其温度平衡至室温。
2)涡旋振荡vahtstmdnacleanbeads充分混匀,向1.5ml低吸附ep管中50μl(1.0x)vahtstmdnacleanbeads,转移50μl第三轮反应pcr产物至ep管中,盖上盖子,低速涡旋振荡混合均匀。
3)室温静置5min。
4)将管置于磁力架上,直至液体澄清(约5min),小心吸去上清,不要吸到磁珠。
5)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠。
6)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠,盖上盖子,短暂离心,用10μl枪头将残留液体吸取,不要吸到磁珠。
7)室温晾干3min(不要让磁珠晾干至出现裂痕),管保持在磁力架上。
8)从磁力架上取下ep管,加入102μlnuclease-freewater,盖上盖子,低速涡旋振荡混合均匀,室温静置2min,将管置于磁力架上直至液体澄清。
9)小心转移100μl上清至1.5ml低吸附ep管中,不要吸到磁珠,丢弃磁珠。
10)向上清中加入82μl(0.82x)涡旋振荡后的vahtstmdnacleanbeads,盖上盖子,低速涡旋振荡混合均匀。
11)室温静置5min。
12)将管置于磁力架上,直至液体澄清(约5min)。小心吸取178μl上清至1.5ml低吸附ep管中(不要吸到磁珠)。
13)向上清中加入20μl(0.2x)涡旋振荡后的vahtstmdnacleanbeads,盖上盖子,低速涡旋振荡混合均匀。
14)室温静置5min。
15)将管置于磁力架上,直至液体澄清(约5min),小心吸去上清,不要吸到磁珠。
16)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠。
17)加入200μl80%乙醇,静置30sec,小心吸去上清,不要吸到磁珠。盖上盖子,短暂离心,用10μl枪头将残留液体吸取,不要吸到磁珠。
18)室温晾干3min(不要让磁珠晾干至出现裂痕),管保持在磁力架上。
19)从磁力架上取下ep管,加入20μlnuclease-freewater。盖上盖子,低速涡旋振荡混合均匀,室温静置2min,将管置于磁力架上直至液体澄清。
20)小心转移18μl上清至1.5mlep管中。
将所构建的测序文库经agilentbioanalyzer2100(图2)和qubit3.0检测合格后采用illuminanextseq500测序仪进行测序分析;
数据分析如表5和表6所示:
表5
表6
从表5和表6的测序数据统计中看到,比对率达到70%以上,准确度达到99.9%以上,5%阳性样本的敏感度达到85%以上,而特异性也达到99.9%以上。这充分说明,本发明的测序文库的构建方法是一种高效的、有效的高通量测序方法。
并且由以上实施例可以看出,本发明的测序文库的构建方法并不需要通常建库所做的末端修复、加a尾、加接头等繁琐的步骤,只需要一种酶,三步pcr反应就可以完成建库方面快捷,成本很低。不仅如此,通过引入分子条形码,通过对barcode的准确定量而达到初始dna的准确定量,可以最大程度的排除在pcr过程中产生的错配。
以上所述实施例的各技术特征可以进行任意的组合,为使描述简洁,未对上述实施例中的各个技术特征所有可能的组合都进行描述,然而,只要这些技术特征的组合不存在矛盾,都应当认为是本说明书记载的范围。
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
序列表
<110>广州永诺生物科技有限公司
广州永诺健康科技有限公司
<120>多重扩增子测序用引物系统、其应用以及测序文库的构建方法
<160>53
<170>siposequencelisting1.0
<210>1
<211>52
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>1
agacgtgtgctcttccgatctnnnnnnnnnntccggactatgaccacttgac52
<210>2
<211>53
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>2
agacgtgtgctcttccgatctnnnnnnnnnnttctacctgaagagcaagtccc53
<210>3
<211>50
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>3
agacgtgtgctcttccgatctnnnnnnnnnncaggttaccccaaaggcca50
<210>4
<211>52
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>4
agacgtgtgctcttccgatctnnnnnnnnnngcaacagcttctggactcact52
<210>5
<211>51
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>5
agacgtgtgctcttccgatctnnnnnnnnnnaccgttgacatcctcttcct51
<210>6
<211>51
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>6
agacgtgtgctcttccgatctnnnnnnnnnnttaagagagccacctgcagc51
<210>7
<211>51
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>7
agacgtgtgctcttccgatctnnnnnnnnnncctctccctgtgctgcattt51
<210>8
<211>51
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>8
agacgtgtgctcttccgatctnnnnnnnnnntggttttcccaccacatcct51
<210>9
<211>51
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>9
agacgtgtgctcttccgatctnnnnnnnnnncctgcactcactccttgtgt51
<210>10
<211>53
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>10
agacgtgtgctcttccgatctnnnnnnnnnnctatgaggtgaattgaggtccc53
<210>11
<211>51
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>11
agacgtgtgctcttccgatctnnnnnnnnnncatccacagagacctggctg51
<210>12
<211>57
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>12
agacgtgtgctcttccgatctnnnnnnnnnngaaatgttctgttctccttcactttc57
<210>13
<211>52
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>13
agacgtgtgctcttccgatctnnnnnnnnnngtgtcaggaaaatgctggctg52
<210>14
<211>50
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>14
agacgtgtgctcttccgatctnnnnnnnnnnaacatcaccaacgccacca50
<210>15
<211>59
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>15
agacgtgtgctcttccgatctnnnnnnnnnnatatgtttgaggatgtaagctagtttca59
<210>16
<211>52
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>16
agacgtgtgctcttccgatctnnnnnnnnnnccctctccctttcctctgttc52
<210>17
<211>56
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>17
agacgtgtgctcttccgatctnnnnnnnnnnaggtaaacataattgttccttctga56
<210>18
<211>59
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>18
agacgtgtgctcttccgatctnnnnnnnnnntctaaaactctctgaaaatgaagtttct59
<210>19
<211>51
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>19
agacgtgtgctcttccgatctnnnnnnnnnnacagacacgtgaaggcatga51
<210>20
<211>49
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,ort
<400>20
agacgtgtgctcttccgatctnnnnnnnnnntccaggaggcagccgaag49
<210>21
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>21
tacacgacgctcttccgatctgttccaggagagcgcagg39
<210>22
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>22
tacacgacgctcttccgatctccactccagcatcactcact41
<210>23
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>23
tacacgacgctcttccgatctcatgtcggtgcatgccttc40
<210>24
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>24
tacacgacgctcttccgatcttgctgggatttcctgatcttcc43
<210>25
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>25
tacacgacgctcttccgatctcagaggggcatgtaacaggc41
<210>26
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>26
tacacgacgctcttccgatcttccacactgcggggtggg39
<210>27
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>27
tacacgacgctcttccgatctgaaggagttgccaagggtgt41
<210>28
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>28
tacacgacgctcttccgatcttctcctcccaaatttgtagaccc44
<210>29
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>29
tacacgacgctcttccgatctgaggtccgtgcagagtgc39
<210>30
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>30
tacacgacgctcttccgatctaaacatgttcatgctgtgtatgta45
<210>31
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>31
tacacgacgctcttccgatctccacgttccctctcctcaat41
<210>32
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>32
tacacgacgctcttccgatctctacattgacggcccccac40
<210>33
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>33
tacacgacgctcttccgatctggcagccaggaacgtactg40
<210>34
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>34
tacacgacgctcttccgatctggttggttgttaataaggaagaaggg47
<210>35
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>35
tacacgacgctcttccgatctgcccagcaccaaaaagagc40
<210>36
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>36
tacacgacgctcttccgatctaggaaccgtttatggcccc40
<210>37
<211>49
<212>dna
<213>人工序列(artificialsequence)
<400>37
tacacgacgctcttccgatctaggagtcatttatatactttgatgaaga49
<210>38
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>38
tacacgacgctcttccgatctagttgggattgcgaacaact41
<210>39
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>39
tacacgacgctcttccgatcttaatgatggcagcgtgtccc41
<210>40
<211>0
<212>dna
<213>人工序列(artificialsequence)
<400>40
<210>41
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>41
agacgtgtgctcttccgatct21
<210>42
<211>58
<212>dna
<213>人工序列(artificialsequence)
<400>42
aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatct58
<210>43
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>43
caagcagaagacggcatacgagatcgtgatgtgactggagttcagacgtgtgctcttccg60
atct64
<210>44
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>44
caagcagaagacggcatacgagatacatcggtgactggagttcagacgtgtgctcttccg60
atct64
<210>45
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>45
caagcagaagacggcatacgagatgcctaagtgactggagttcagacgtgtgctcttccg60
atct64
<210>46
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>46
caagcagaagacggcatacgagattggtcagtgactggagttcagacgtgtgctcttccg60
atct64
<210>47
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>47
caagcagaagacggcatacgagatcactgtgtgactggagttcagacgtgtgctcttccg60
atct64
<210>48
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>48
caagcagaagacggcatacgagatattggcgtgactggagttcagacgtgtgctcttccg60
atct64
<210>49
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>49
caagcagaagacggcatacgagatgatctggtgactggagttcagacgtgtgctcttccg60
atct64
<210>50
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>50
caagcagaagacggcatacgagattcaagtgtgactggagttcagacgtgtgctcttccg60
atct64
<210>51
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>51
caagcagaagacggcatacgagatctgatcgtgactggagttcagacgtgtgctcttccg60
atct64
<210>52
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>52
caagcagaagacggcatacgagataagctagtgactggagttcagacgtgtgctcttccg60
atct64
<210>53
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>53
caagcagaagacggcatacgagatgtagccgtgactggagttcagacgtgtgctcttccg60
atct64
- 还没有人留言评论。精彩留言会获得点赞!