一种控制水稻谷粒大小和粒重的基因SNG1及其应用的制作方法
本发明涉及植物基因工程技术领域。具体涉及到一种控制水稻谷粒性状特别是控制水稻谷粒粒长、粒宽和粒重性状的基因sng1及其应用。所述的sng1基因位于水稻第一染色体长臂末端,该基因控制水稻谷粒粒长、粒宽和粒重相关性状。
背景技术:
水稻谷粒的大小由粒长、粒宽、粒厚三个子性状控制,它直接决定粒重,而粒重是单株产量的三大构成因子(单株有效穗数、每穗实粒数和千粒重)之一,因此,谷粒大小是影响产量的重要因素;水稻谷粒大小也是一项重要的外观品质性状,以籼稻为例,大部分中国消费者比较喜欢粒形瘦长的,在国家标准中(优质稻谷gb/t17891-1999),要求优质米长宽比超过2.8。
水稻谷粒大小是受多个数量性状位点控制的复杂性状。近年来,随着遗传作图、基因组测序和功能基因组研究的发展,大量控制水稻谷粒大小的数量性状位点(quantitativetraitloci,qtls)被定位,其中有多个被克隆:gs3(fanetal.,2006)、gw2(songetal.,2007)、gw5/qsw5(shomuraetal.,2008;wengetal.,2008)、gs5(lietal.,2011)、gw8(wangetal.,2012)、qgl3/qgl3.1(qietal.,2012;zhangetal.,2012)、tgw6(ishimaruetal.,2013)、gs2/gl2(cheetal.,2015;duanetal.,2015;huetal.,2015)、gl7/gw7(wangetal.,2015a;wangetal.,2015b)、gw6a(songetal.,2015)和osspl13(sietal.,2016)。这些基因调控水稻种子大小分子机制多种多样,如g蛋白信号传导、蛋白酶体降解途径、植物激素和转录调控等(liandli,2016;zuoandli,2014)。我们还需要继续克隆更多的粒形基因,并研究粒形基因的相互关系,构建粒形的分子调控网络,这样才能更好的理解水稻籽粒发育的生物学基础,并指导水稻产量和外观品质的遗传改良。
本发明的申请人分离鉴定了一个水稻小粒突变体sng1(shortandnarrowgrain1),利用图位克隆的方法克隆了一个新的正调控种子大小的基因sng1,同时利用遗传转化方法验证了该基因的功能,丰富了粒形的分子调控网络,也为水稻粒形的遗传改良提供了新的基因资源。
技术实现要素:
本发明的目的在于克服现有技术的不足,利用一个小粒突变体sng1分离克隆得到一个位于第一染色体上控制粒长、粒宽和粒重的相关基因,申请人将这个基因命名为sng1基因。
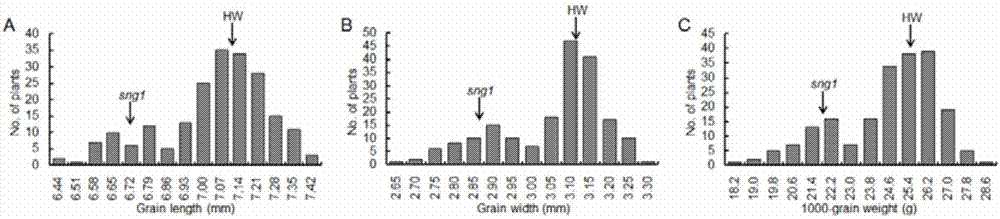
本发明分离鉴定了一个组织培养产生的小粒突变体sng1,其背景是是粳稻品种hwayoung(简称hw,下同,常规来源品种,已在韩国大面积种植)。与野生型水稻相比,突变体种子粒长、粒宽和千粒重显著降低(见图1),利用hw基因和sng1基因构建的f2代分离群体的粒长、粒宽和千粒重均呈现双峰分布,且大粒材料与小粒材料的分离比符合经典遗传学3:1的分离比例(见图1),表明突变体种子变小是由单位点引起的。
申请人利用图位克隆的方法克隆变异位点,将sng1和种子较大的籼稻品种珍汕97b(简称zs97,中国大面积应用)杂交构建f2代分离群体,进行初定位。并通过回交的方法构建了以珍汕97b(zs97b)为背景的近等基因系,本发明克隆基因的来源材料是水稻突变体种子sng1,oryzasatival.sng1,于2017年11月17日送交中国.武汉.武汉大学的中国典型培养物保藏中心保藏,保藏编号为cctccno:p201723(构建近等基因系的技术路线见图2),利用近等基因系分离群体验证sng1的效应,表明群体的粒长、粒宽和千粒重均呈双峰分布(图3)。利用sng1近等基因系的遗传大群体和图位克隆的方法,将sng1精细定位到5.5kb的染色体区段内(图4)。目标区段内包含两个不完整的候选基因,测序并进行序列比对发现,与hw野生型相比,sng1突变体整个区段内只有一个单核苷酸突变(即t-a的突变),该变异位于候选基因(登陆号loc_os01g71310)的最后一个外显子上,且该变异使编码精氨酸的密码子(aga)变成终止密码子(tga),预测sng1的候选基因编码一个缺少c端103个氨基酸的截短的蛋白(图4)。生物信息学分析表明loc_os01g71310包含9个外显子,共编码500个氨基酸,该基因编码一个己糖激酶(oshxk3)。通过农杆菌介导的遗传转化,在hwayoung中超量表达(oe)sng1,转基因t0代阳性单株表现出更大的种子(图5),其t1代家系共分离检测发现粒长、粒宽和千粒重与基因型共分离(图6),且t2代转基因纯合家系的植株形态和抽穗期等性状与野生型差异不大(图7),表明sng1的超表达具有一定的应用前景。
本发明的优点在于:
(1)本发明在水稻中克隆了正调控水稻谷粒长、粒宽和粒重性状的基因sng1,为水稻的高产优质育种提供了新的基因资源;
(2)本发明中克隆的基因具有很强的进化上的保守性,本发明也可以为其他作物的遗传改良提供借鉴。
附图说明
序列表seqidno:1是本发明分离克隆的来源于野生型水稻品种hwayoung(hw)的sng1等位基因的核苷酸序列。
序列表seqidno:2是野生型hwayoung(hw)的sng1等位基因编码的蛋白质序列。
序列表seqidno:3是本发明分离克隆的来源突变体的sng1等位基因的核苷酸序列。
序列表seqidno:4是本发明的突变体的sng1等位基因编码的蛋白质序列。
图1:sng1/hwf2分离群体粒长、粒宽和千粒重的分布。附图标记说明:图1中的a图是粒长的频数分布图;图1中的b图是粒宽的频数分布图;图1中的c图是千粒重的频数分布图。
图2.:是本发明的近等基因系构建流程图(回交时,以珍汕97作为母本)。
图3:bc3f2随机群体中谷粒粒长、粒宽和千粒重的频率分布。附图标记说明:图3中的a图是粒长的频数分布图;图3中的b图是粒宽的频数分布图;图3中的c图是千粒重的频数分布图。其中,黑色、条纹和白色的棒分别表示sng1位点突变体sng1纯合基因型(a)、杂合基因型(h)和珍汕97纯合基因型(b),sng1的三种基因型是通过分子标记检测得到的。
图4:本发明sng1基因的图位克隆。附图标记说明:图4中的a图是初步定位的结果,sng1基因被定位到480kb的区段内;图4中的b图是精细定位的结果,sng1基因被定位到5.5kb的区段内;图4中的c图是展示关键重组单株目的区段基因型和后代测验的结果。图4中标记间的数字代表各标记与sng1基因位点间发生的重组次数。
图5:本发明的sng1基因超量表达t0代转基因单株的粒形和粒重的表现和表型统计分析。附图标记说明:图5中的a图为转基因植株粒形,在该图的左边是转基因阴性材料(oe(-)),右边是转基因阳性材料(oe(+));图5中的b图、c图和d图分别表示转基因阴性与转基因阳性植株的粒长、粒宽和千粒重统计结果,统计方法为双尾t测验,p值在上方显示,转基因阴性植株共12株(n=12),转基因阳性植株共20株(n=20)。
图6:本发明的sng1基因超量表达t1代转基因家系表型与基因型共分离分析的结果。附图标记说明:图6中的a图是粒长与基因型共分离的结果;图6中的b图是粒宽与基因型共分离的结果;图6中的c图是千粒重与基因型共分离的结果。白色空心柱子和蓝色实心柱子分别表示转基因阴性与转基因阳性植株,基因型是通过用pcr的方法检测报告基因gus得到的。
图7:本发明的sng1基因超量表达t2代转基因家系灌浆期植株表型和sng1基因表达量的检测结果。附图标记说明:图7中的a图是野生型(hw)和两个sng1基因超量表达t2代转基因纯合家系(oe-4和oe-35)的灌浆期植株表型;图7中的b图是用real-timepcr方法检测的抽穗期剑叶中sng1基因的相对表达量。
图8:本发明用到的超量表达载体pu1301的图谱。
具体实施方式
本实施例鉴定到一个种子变小的水稻突变体,来自韩国t-dna插入突变体库(jeonetal.,2000),编号为pfg_1c-12303,转化受体是粳稻品种hwayoung(简称hw)。与野生型hw相比,突变体种子粒长和粒宽明显变小,千粒重显著降低(图1),而株形、株高、抽穗期等其他性状变化不大。申请人将突变体命名为sng1(本发明克隆基因的来源材料是水稻突变体种子sng1,oryzasatival.sng1,于2017年11月17日送交中国.武汉.武汉大学的中国典型培养物保藏中心保藏,保藏编号为cctccno:p201723)。hw和突变体构建的f2代分离群体的粒长、粒宽和千粒重均呈现双峰分布,大粒材料的粒形与hw接近,小粒材料的粒形与突变体接近,且其分离比符合经典遗传学3:1分离比例(图1),表明突变体种子变小是由单位点隐性突变引起的。但申请人分离不到突变体的t-dna侧翼序列,认为变异可能是组织培养导致的,利用图位克隆的方法克隆变异位点。
实施例2:sng1基因的定位和图位克隆
1.回交与选择
本发明利用图位克隆的方法克隆变异位点,将sng1和种子较宽的籼稻品种珍汕97b(来源于江西省农业科学院)杂交构建得到f2代分离群体(图2),用分离群体分组分析法(bulkedsegregantanalysis,bsa)进行初定位。发现第1染色体末端存在一个qtl同时控制粒长和粒宽,珍汕97基因型增加表型值,将该qtl命名为sng1。
从f2代分离群体中挑选小粒单株,与母本珍汕97杂交一次,在初定位的基础上,结合分子标记辅助选择(mas),以珍汕97为轮回亲本,连续回交3次,之后自交1次,得到sng1的近等基因系,用于sng1的精细定位(图2)。利用192株bc3f2分离群体验证sng1的效应,表明群体的粒长、粒宽和千粒重均呈双峰分布(图3)。表明在此bc3f2群体中,籽粒大小是由一个主效基因控制的。
2.基因型和表型鉴定方法
基因型测定采用ssr方法:pcr标准程序参见参见j.萨姆布鲁克等,2002,分子克隆实验指南,第三版,金冬雁等(译),科学出版社介绍的方法。pcr采用20μl的反应体系,包含:20-50ngdna模板,10mmtris-hcl,50mmkcl,0.1%tritonx-100,1.8mmmgcl2,0.1mmdntp,0.2μm引物和1utaqdnapolymerase。pcr扩增的条件为:94℃预变性4min;94℃1min,55℃1min,72℃1min,34个循环;72℃延伸10min。pcr产物在6%的聚丙烯酰胺胶上分离后进行银染(bassam等,1991,anal.biochem.196:80-83)。
本发明中所用到的ssr标记信息全部来自于gramene网站数据库(http://www.gramene.org/).此外,还根据网上公布的粳稻品种nipponbare的基因组序列(http://rgp.dna.affrc.go.jp)以及籼稻品种93-11的基因组序列(http://rise.genomics.org.cn/)设计在珍汕97b和sng1间具有多态性的indel(insert/deletion)标记,用于sng1的精细定位分析。这些标记的序列信息见表1。
表型考察方法:
粒长、粒宽和千粒重的测量:种子收获后晒干,室温至少放置3个月,以保证种子的干燥和各株系间种子含水量的相对一致。从每单株中随机选取30粒饱满种子,10粒为一组,按照同一个方向肩并肩、不重叠、不留隙的方式摆成一行,用游标卡尺读出宽度,3次重复取平均值即为粒宽;10粒种子首尾相连排列,用游标卡尺读出长度,3次重复取平均值即为粒长。称量随机挑选的200粒饱满重量的重量来计算千粒重。3.sng1基因的精细定位及候选基因确定
在效应验证的基础上,以粒宽作为目标性状对sng1基因进行精细定位。用来验证sng1基因效应的192株bc3f2分离群体中,sng1基因所在区段两端indel标记y11和y91之间有13株重组单株(图4),对这13株重组单株进行后代测验(progenytesting),根据24株后代的粒宽表型来推断该重组单株sng1的基因型:若后代粒宽没有分离且均为大粒则重组单株当代的sng1是珍汕97纯合型;若后代粒宽没有分离且均为小粒则重组单株当代的sng1是sng1纯合型;若后代粒宽和种子大小发生分离则重组单株当代的sng1的基因型是杂合型。在目标区段加密分子标记,并检测标记的基因型,进行连锁分析,将sng1精细定位到标记y6和y54之间480kb的区段内(图4)。
以此为基础,申请人种植zs97/sng1bc3f3分离群体7680株,用标记y6和y54筛选到125株重组单株,对重组单株进行后代测验,推断重组单株sng1的基因型,进一步加密标记,最终将sng1定位到indel标记y1和y7之间5.5kb的区段内,与标记snp3共分离(图4)。
参考粳稻“日本晴”基因组(http://rice.plantbiology.msu.edu/index.shtml),标记y1和y7之间5.5kb的区段内包含两个不完整的orf,且两个orf以尾对尾的方式排列。以“日本晴”基因组为参考,设计引物对(件表1所示)hw、sng1和zs97的5.5kb定位区段进行测序,序列比对发现,与水稻品种hw野生型相比,sng1突变体整个区段内只有一个snp(t-a),该snp位于候选基因loc_os01g71310的最后一个外显子上,且该变异使编码精氨酸的密码子(aga)变成终止密码子(tga),预测sng1的候选基因编码一个缺少c端103个氨基酸的截短的蛋白;zs97该snp基因型为t(与hw相同),5.5kb的区段内zs97和sng1之间还有14个snp变异,其中12个位于非编码区,2个位于外显子且为同义突变。因此,将loc_os01g71310定为sng1的候选基因,且用图位克隆的方法定位到的sng1qtl是由sng1的突变产生的(可能来自于组织培养),而不是hw和zs97之间的自然变异。
表1用于本发明图位克隆和基因功能分析的引物序列
实施例3:sng1的转基因互补试验
1.sng1超量表达载体pu1301的构建
根据sng1“日本晴”全长cdna序列(登陆号ay884171,dq116385),设计引物扩增野生型hwsng1的编码区。其中oef和oer引物分别带有kpni和bamhi限制性内切酶识别位点,pcr反应体系为:hw第一链cdna4μl+2×kodfxpcrbuffer25μl+2mmdntps10μl+oef/oer各1.5μl+kodfx(1.0u/μl)1μl+dh2o7μl;pcr扩增条件为:94℃4min,98℃10sec,68℃2min,32cycles,72℃10min,25℃1min;扩增长度约为1.6kb。扩增产物先进行3’端加da反应(高保真kod酶扩增产物为平末端,不能直接用来做ta克隆),再经挖胶回收和纯化后连接到pgem-t(购自promega公司,图8)克隆载体上,连接反应体系为:回收的扩增产物3μl+pgem-t载体(50ng/μl)1μl+2×t4ligasebuffer5μl+t4ligase(400u/μl)1μl。16℃反应过夜,转化大肠杆菌,挑单克隆摇菌抽质粒,用kpni和bamhi双酶切以及测序验证挑选无突变的阳性克隆,阳性克隆用kpni和bamhi双酶切,经挖胶回收纯化后连接到双元超表达载体pu1301(图8),连接反应体系为:回收产物4μl+kpni和bamhi双酶切后回收的pu1301载体4μl+10×t4ligasebuffer1μl+t4ligase(400u/μl)1μl。16℃反应过夜,转化大肠杆菌,挑单克隆摇菌抽质粒,kpni和bamhi双酶切验证,该正确的克隆即为构建好的sng1超量表达载体pu1301(sng1oe)。2.sng1超量表达载体pu1301的转化
本实施例的遗传转化方法参考了下列文献:
(1)专利号zl2004100133457;华中农业大学,一种籼稻的离体培养方法;或
(2)专利号zl2013104541628,华中农业大学,组蛋白甲基转移酶sdg723调控水稻抽穗期中的应用,报道的方法。
本发明的遗传转化的具体的主要步骤、培养基及其配制的方法:
1)试剂和溶液缩写
本实验中培养基所用到的植物激素的缩写表示如下:6-ba(6-benzylaminopurine,6-苄基腺嘌呤);cn(carbenicillin,羧苄青霉素);kt(kinetin,激动素);naa(napthaleneaceticacid,萘乙酸);iaa(indole-3-aceticacid,吲哚乙酸);2,4-d(2,4-dichlorophenoxyaceticacid,2,4-二氯苯氧乙酸);as(acetosringone,乙酰丁香酮);ch(caseinenzymatichydrolysate,水解酪蛋白);hn(hygromycinb,潮霉素);dmso(dimethylsμlfoxide,二甲基亚砜);n6max(n6大量元素成分溶液);n6mix(n6微量元素成分溶液);msmax(ms大量元素成分溶液);msmix(ms微量元素成分溶液)
2)主要溶液配方
a)n6max培养基大量元素母液(按照10倍浓缩液配制):
将上述试剂逐一溶解,然后室温下用蒸馏水定容至1000ml,室温储存。
b)n6min培养基微量元素母液(按照100倍浓缩液配制):
将上述试剂在室温下溶解并用蒸馏水定容至1000ml,室温储存。
c)铁盐(fe2+edta)贮存液(按照100x浓缩液配制):
将3.73g乙二铵四乙酸二钠(na2edta·2h2o)和2.78gfeso4·7h2o分别溶解,混合并用蒸馏水定容至1000ml,至70℃温浴2小时,4℃保存备用。
d)维生素贮存液(按照100x浓缩液配制):
烟酸(nicotinicacid)0.1g
维生素b1(thiaminehcl)0.1g
维生素b6(pyridoxinehcl)0.1g
甘氨酸(glycine)0.2g
肌醇(inositol)10g
加蒸馏水定容至1000ml,4℃保存备用。
e)ms培养基大量元素母液(msmax母液)(按照10x浓缩液配制):
硝酸铵(nh4no3)16.5g
硝酸钾(kno3)19.0g
磷酸二氢钾(kh2po4)1.7g
硫酸镁(mgso4·7h2o)3.7g
氯化钙(cacl2·2h2o)4.4g
将上述试剂在室温下溶解,并用蒸馏水定容至1000ml,室温储存。
f)ms培养基微量元素母液(msmin母液)(按照100x浓缩液配制):
将上述试剂在室温下溶解,并用蒸馏水定容至1000ml,室温储存。
g)2,4-d贮存液(1mg/ml)的配制:
秤取2,4-d100mg,用1ml1n氢氧化钾溶解5分钟,然后加10ml蒸馏水溶解完全后定容至100ml,于室温下保存。
h)6-ba贮存液(1mg/ml)的配制:
秤取6-ba100mg,用1ml1n氢氧化钾溶解5分钟,然后加10ml蒸馏水溶解完全后定容至100ml,室温保存。
i)萘乙酸(naa)贮存液(1mg/ml)的配制:
秤取naa100mg,用1ml1n氢氧化钾溶解5分钟,然后加10ml蒸馏水溶解完全后定容至100ml,4℃避光保存。
j)吲哚乙酸(iaa)贮存液(1mg/ml)的配制:
秤取iaa100mg,用1ml1n氢氧化钾溶解5分钟,然后加10ml蒸馏水溶解完全后定容至100ml,4℃避光保存。
k)葡萄糖贮存液(0.5g/ml)的配制:
秤取葡萄糖125g,然后用蒸馏水溶解定容至250ml,灭菌后4℃保存备用。
l)as贮存液的配制:
秤取as0.392g,加入dmso10ml溶解,分装至1.5ml离心管内,-20℃保存备用。
m)1n氢氧化钾贮存液配制:
秤取氢氧化钾5.6g,用蒸馏水溶解定容至100ml,室温保存备用。
3)用于粳稻遗传转化的培养基配方
a)诱导培养基
加蒸馏水至900ml,1n氢氧化钾调节ph值到5.9,煮沸并定容至1000ml,分装到50ml三角瓶(30ml/瓶),封口后按常规方法灭菌(例如121℃下灭菌15分钟,下述的培养基灭菌方法与本培养基的灭菌方法相同)。
b)继代培养基:
加蒸馏水至900ml,1n氢氧化钾调节ph值到5.9,煮沸并定容至1000ml,分装到50ml三角瓶(30ml/瓶),封口,按上述方法灭菌。
c)悬浮培养基:
加蒸馏水至100ml,调节ph值到5.4,分装到两个100ml的三角瓶中,封口,按上述方法灭菌。
使用前加入1ml无菌葡萄糖贮存液和100μlas贮存液。
d)共培养基:
加蒸馏水至250ml,1n氢氧化钾调节ph值到5.6,封口,按上述方法灭菌。
使用前加热溶解培养基并加入5ml葡萄糖贮存液和250μlas贮存液,分装倒入培养皿中(25ml/每皿)。
e)筛选培养基:
加蒸馏水至250ml,调节ph值到6.0,封口,按上述方法灭菌。
使用前溶解培养基,加入250μlhn(50mg/ml)和400μlcn(10gcn/36ml水)分装倒入培养皿中(25ml/皿)。(注:第一次选择培养基羧苄青霉素浓度为400mg/升,第二次及以后选择培养基羧苄青霉素浓度为250mg/升)。
f)分化培养基:
加蒸馏水至900ml,1n氢氧化钾调节ph值到6.0。
煮沸并用蒸馏水定容至1000ml,分装到100ml三角瓶(50ml/瓶),封口,按上述方法灭菌。
g)生根培养基
加蒸馏水至900ml,用1n氢氧化钾调节ph值到5.8。
煮沸并用蒸馏水定容至1000ml,分装到生根管中(25ml/管),封口,按上述方法灭菌。
4)农杆菌介导的遗传转化步骤
a)愈伤诱导
将成熟的水稻种子去壳,然后依次用70%的乙醇处理1分钟,0.15%氯化汞(hgcl2)种子表面消毒15-25分钟;用灭菌水洗种子4-5次;将8-10粒种子放在诱导培养基上;将接种后的培养基置于黑暗处培养4-5周,温度26±1℃。
b)愈伤继代:
挑选亮黄色、紧实且相对干燥的胚性愈伤,放于继代培养基上黑暗下培养2周,温度25±1℃。
c)预培养:
挑选紧实且相对干燥的胚性愈伤,放于预培养基上黑暗下培养2周,温度26±1℃。
d)农杆菌培养:
在带有对应抗性选择的la培养基(la培养基的配制参照j.萨姆布鲁克等,分子克隆实验指南,第三版,金冬雁等(译),科学出版社,2002,北京)上划线预培养农杆菌eha105(该菌株来自cambia公司公开使用的农杆菌菌株)两天,温度28℃;将农杆菌转移至悬浮培养基里,28℃摇床上培养2-3小时。
e)农杆菌侵染:
将预培养的愈伤转移至灭好菌的瓶子内;调节农杆菌的悬浮液至od6000.8-1.0;将愈伤在农杆菌悬浮液中浸泡30分钟;转移愈伤至灭菌好的滤纸上吸干;然后放置在共培养基上培养3天,温度19-20℃。
f)愈伤洗涤和选择培养:
灭菌水洗涤愈伤至看不见农杆菌;浸泡在含400mg/l羧苄青霉素(cn)的灭菌水中摇30分钟;转移愈伤至灭菌好的滤纸上吸干;转移愈伤至选择培养基上选择培养2-3次,每次2周至长出好的抗性愈伤。
g)分化:
将抗性愈伤转移至预分化培养基上于黑暗处培养5-7天;转移预分化培养的愈伤至分化培养基上,每瓶平均分布三个独立的愈伤,光照下培养5周-6周至长出大的苗子,温度26℃。
h)生根与炼苗:
剪掉分化时产生的老根;然后将其转移至生根培养基中光照下培养2-3周至长出大的苗子后去掉封口膜加部分自来水炼苗一周再移栽,温度26℃。
i)移栽:
洗掉根上的残留培养基,将具有良好根系的幼苗转入温室,同时在最初的几天保持水分湿润,等长势很壮再移栽至大田。
2.转基因植株的性状考察
本发明共获得32株独立的sng1超表达(oe)t0代转基因植株,通过pcr扩增报告基因gus来确定转基因植株的基因型,其中20株为转基因阳性单株,12株为转基因阴性单株。通过real-timepcr检测sng1的表达量,发现转基因阳性单株sng1的表达量有不同程度的上升。考查上述32个转基因单株种子的粒长、粒宽和千粒重,发现超表达的阳性单株的粒长、粒宽和千粒重极显著大于阴性植株(p<0.01)(图5)。
为了进一步验证超表转化的结果,从20个t0代阳性植株中随机选择了3个单株,种植t1代对基因型和表型做共分离检测,每个家系为36株,利用gus报告基因(常规标记基因)pcr扩增结果来确定各单株的基因型。结果表明,基因型与粒长、粒宽和千粒重完全共分离(图6),进一步证明sng1基因为正控制粒长、粒宽和千粒重的候选基因。
另外,超表达sng1的转基因材料对植株的形态和抽穗期等农艺性状影响不大(图7),这表明sng1的超表达可能具有一定的应用前景,可以通过这个基因转化来改良水稻品种。
参考文献
1.cherh,tonghn,shibh,liuyq,fangsr,liudp,xiaoyh,hub,liulc,wanghr,zhaomf,chucc.controlofgrainsizeandriceyieldbygl2-mediatedbrassinosteroidresponses.natureplants,2015,2:15195
2.duanpg,nis,wangjm,zhangbl,xur,wangyx,chenhq,zhuxd,liyh.regulationofosgrf4byosmir396controlsgrainsizeandyieldinrice.natureplants,2015,2:15203
3.fanc,xingy,maoh,lut,hanb,xuc,lix,zhangq.gs3,amajorqtlforgrainlengthandweightandminorqtlforgrainwidthandthicknessinrice,encodesaputativetransmembraneprotein.theoreticalandappliedgenetics,2006,112:1164-1171
4.huj,wangy,fangy,zengl,xuj,yuh,shiz,panj,zhangd,kangs,zhul,dongg,guol,zengd,zhangg,xiel,xiongg,lij,qianq.ararealleleofgs2enhancesgrainsizeandgrainyieldinrice.molecularplant,2015,8:1455-1465
5.ishimaruk,hirotsun,madokay,murakamin,haran,onoderah,kashiwagit,ujiiek,shimizub,onishia,miyagawah,katohe.lossoffunctionoftheiaa-glucosehydrolasegenetgw6enhancesricegrainweightandincreasesyield.naturegenetics,2013,45:707-711
6.jeonjs,lees,jungkh,junsh,jeongdh,leej,kimc,jangs,yangk,namj,ank,hanmj,sungrj,choihs,yujh,choijh,chosy,chass,kimsi,ang.t-dnainsertionalmutagenesisforfunctionalgenomicsinrice.plantj,2000,22:561-570
7.li,n.,andli,y.signalingpathwaysofseedsizecontrolinplants.currentopinioninplantbiology,2016,33:23-32
8.liy,fanc,xingy,jiangy,luol,sunl,shaod,xuc,lix,xiaoj,hey,zhangq.naturalvariationings5playsanimportantroleinregulatinggrainsizeandyieldinrice.naturegenetics,2011c,43:1266-1269
9.qip,linys,songxj,shenjb,huangw,shanjx,zhumz,jiangl,gaojp,linhx.thenovelquantitativetraitlocusgl3.1controlsricegrainsizeandyieldbyregulatingcyclin-t1;3.cellresearch,2012,22:1666-1680
10.shomuraa,izawat,ebanak,ebitanit,kanegaeh,konishis,yanom.deletioninageneassociatedwithgrainsizeincreasedyieldsduringricedomestication.naturegenetics,2008,40:1023-1028
11.sil,chenj,huangx,gongh,luoj,houq,zhout,lut,zhuj,shangguany,chene,gongc,zhaoq,jingy,zhaoy,liy,cuil,fand,luy,wengq,wangy,zhanq,liuk,weix,ank,ang,hanb.osspl13controlsgrainsizeincultivatedrice.naturegenetics,2016,48:447-456
12.songxj,huangw,shim,zhumz,linhx.aqtlforricegrainwidthandweightencodesapreviouslyunknownring-typee3ubiquitinligase.naturegenetics,2007,39:623-630
13.songxj,kurohat,ayanom,furutat,nagaik,komedan,segamis,miurak,ogawad,kamurat,suzukit,higashiyamat,yamasakim,morih,inukaiy,wuj,kitanoh,sakakibarah,jacobsense,ashikarim.rarealleleofapreviouslyunidentifiedhistoneh4acetyltransferaseenhancesgrainweight,yield,andplantbiomassinrice.procnatlacadsciusa,2015,112:76-81
14.wangs,lis,liuq,wuk,zhangj,wangs,wangy,chenx,zhangy,gaoc,wangf,huangh,fux.theosspl16-gw7regulatorymoduledeterminesgrainshapeandsimultaneouslyimprovesriceyieldandgrainquality.naturegenetics,2015a,47:949-954.
15.wangs,wuk,yuanq,liux,liuz,linx,zengr,zhuh,dongg,qianq,zhangg,fux.controlofgrainsize,shapeandqualitybyosspl16inrice.naturegenetics,2012,44:950-954
16.wangy,xiongg,huj,jiangl,yuh,xuj,fangy,zengl,xue,xuj,yew,mengx,liur,chenh,jingy,wangy,zhux,lij,qianq.copynumbervariationatthegl7locuscontributestograinsizediversityinrice.naturegenetics,2015b,47:944-948
17.wengj,gus,wanx,gaoh,guot,sun,leic,zhangx,chengz,guox,wangj,jiangl,zhaih,wanj.isolationandinitialcharacterizationofgw5,amajorqtlassociatedwithricegrainwidthandweight.cellresearch,2008,18:1199-1209
18.zhangx,wangj,huangj,lanh,wangc,yinc,wuy,tangh,qianq,lij,zhangh.rarealleleofosppkl1associatedwithgrainlengthcausesextra-largegrainandasignificantyieldincreaseinrice.procnatlacadsciusa,2012,109:21534-21539
19.zuo,j.,andli,j.moleculargeneticdissectionofquantitativetraitlociregulatingricegrainsize.annualreviewofgenetics.2014,48:99-118。
序列表
<110>华中农业大学
<120>一种控制水稻谷粒大小和粒重的基因sng1及其应用
<141>2017-11-20
<160>4
<170>siposequencelisting1.0
<210>1
<211>1503
<212>dna
<213>水稻(oryzasativa)
<220>
<221>gene
<222>(1)..(1503)
<220>
<221>cds
<222>(1)..(1503)
<400>1
atggggagggtggggctcggcgtggcggtggggtgcgcggcggtgacc48
metglyargvalglyleuglyvalalavalglycysalaalavalthr
151015
tgcgcgatcgccgcggcgctcgtggcgcgcagggcgtcggcgcgggcg96
cysalailealaalaalaleuvalalaargargalaseralaargala
202530
cggtggcggcgggcggtggcgctgctgcgggagttcgaggaggggtgt144
argtrpargargalavalalaleuleuargglupheglugluglycys
354045
gccacgccgcccgcgaggctgcgccaggtcgtggacgccatggtcgtc192
alathrproproalaargleuargglnvalvalaspalametvalval
505560
gagatgcacgccggcctcgcgtccgatggcggcagcaagctcaagatg240
glumethisalaglyleualaseraspglyglyserlysleulysmet
65707580
ctgctcaccttcgtcgacgcgctccccagcgggagtgaagaaggtgta288
leuleuthrphevalaspalaleuproserglyserglugluglyval
859095
tattattctattgatcttggaggaacaaacttcagagtcttgagggta336
tyrtyrserileaspleuglyglythrasnpheargvalleuargval
100105110
caagttggtgcgggatctgtgatcgtcaaccaaaaggttgaacagcaa384
glnvalglyalaglyservalilevalasnglnlysvalgluglngln
115120125
cccatccctgaggaactgaccaaaggcactactgagggtttattcaac432
proileproglugluleuthrlysglythrthrgluglyleupheasn
130135140
tttgttgccctggcactaaagaattttcttgaaggagaagatgaccaa480
phevalalaleualaleulysasnpheleugluglygluaspaspgln
145150155160
gatggaaaaatggcacttggttttacattttctttccctgttagacaa528
aspglylysmetalaleuglyphethrpheserpheprovalarggln
165170175
atttcagtgtcttcagggtcattaattaggtggacaaaaggattttcc576
ileservalserserglyserleuileargtrpthrlysglypheser
180185190
atcagagacacggttggcagagatgttgctcagtgcttaaatgaagcg624
ileargaspthrvalglyargaspvalalaglncysleuasngluala
195200205
cttgccaattgtgggctaaatgtgcgggtcactgcattggtgaatgat672
leualaasncysglyleuasnvalargvalthralaleuvalasnasp
210215220
acagtggggacattagctctagggcattactatgatgaagacacagtg720
thrvalglythrleualaleuglyhistyrtyraspgluaspthrval
225230235240
gctgctgtgataattgggtctggcaccaacgcttgctacattgaacgc768
alaalavalileileglyserglythrasnalacystyrilegluarg
245250255
actgatgcaattatcaagtgccagggtcttctaacgaactctggaggc816
thraspalaileilelyscysglnglyleuleuthrasnserglygly
260265270
atggtagtaaacatggagtgggggaatttctggtcatcacatttgcca864
metvalvalasnmetglutrpglyasnphetrpserserhisleupro
275280285
aggacgccatatgacatcttgctggatgatgaaacacacaatcgcaat912
argthrprotyraspileleuleuaspaspgluthrhisasnargasn
290295300
gatcagggctttgagaaaatgatatcaggaatgtatcttggggaaatt960
aspglnglypheglulysmetileserglymettyrleuglygluile
305310315320
gcaagattggtatttcatagaatggcccaggaatcagatgtttttggt1008
alaargleuvalphehisargmetalaglngluseraspvalphegly
325330335
gatgctgctgatagtctatccaaccctttcattttgagcacaccgttt1056
aspalaalaaspserleuserasnpropheileleuserthrprophe
340345350
ctggccgcaattcgcgaggacgattcaccagatctgagcgaagtcaga1104
leualaalailearggluaspaspserproaspleusergluvalarg
355360365
aggatacttcgagaacatctgaagattcccgatgctcctctgaaaact1152
argileleuarggluhisleulysileproaspalaproleulysthr
370375380
cgacggctggtcgtgaaagtctgcgacattgtgactcgcagagccgcc1200
argargleuvalvallysvalcysaspilevalthrargargalaala
385390395400
cgtctagccgctgcaggcatcgtggggatactgaagaagctggggagg1248
argleualaalaalaglyilevalglyileleulyslysleuglyarg
405410415
gatgggagcggcgcggcgtcgagcgggagaggtagagggcagccgagg1296
aspglyserglyalaalaserserglyargglyargglyglnproarg
420425430
aggacggtggtggcgatcgagggcgggctgtaccagggttacccggtg1344
argthrvalvalalailegluglyglyleutyrglnglytyrproval
435440445
ttcagggagtacctggacgaggccctggtggagatcctgggggaggag1392
pheargglutyrleuaspglualaleuvalgluileleuglygluglu
450455460
gtggcgcggaacgtgacgctgagggtgacggaggatgggtcgggggtc1440
valalaargasnvalthrleuargvalthrgluaspglyserglyval
465470475480
ggggctgccctcctcgccgccgtacattcgtcgaatagacagcaacaa1488
glyalaalaleuleualaalavalhisserserasnargglnglngln
485490495
ggaggtcccatatag1503
glyglyproile
500
<210>2
<211>500
<212>prt
<213>水稻(oryzasativa)
<400>2
metglyargvalglyleuglyvalalavalglycysalaalavalthr
151015
cysalailealaalaalaleuvalalaargargalaseralaargala
202530
argtrpargargalavalalaleuleuargglupheglugluglycys
354045
alathrproproalaargleuargglnvalvalaspalametvalval
505560
glumethisalaglyleualaseraspglyglyserlysleulysmet
65707580
leuleuthrphevalaspalaleuproserglyserglugluglyval
859095
tyrtyrserileaspleuglyglythrasnpheargvalleuargval
100105110
glnvalglyalaglyservalilevalasnglnlysvalgluglngln
115120125
proileproglugluleuthrlysglythrthrgluglyleupheasn
130135140
phevalalaleualaleulysasnpheleugluglygluaspaspgln
145150155160
aspglylysmetalaleuglyphethrpheserpheprovalarggln
165170175
ileservalserserglyserleuileargtrpthrlysglypheser
180185190
ileargaspthrvalglyargaspvalalaglncysleuasngluala
195200205
leualaasncysglyleuasnvalargvalthralaleuvalasnasp
210215220
thrvalglythrleualaleuglyhistyrtyraspgluaspthrval
225230235240
alaalavalileileglyserglythrasnalacystyrilegluarg
245250255
thraspalaileilelyscysglnglyleuleuthrasnserglygly
260265270
metvalvalasnmetglutrpglyasnphetrpserserhisleupro
275280285
argthrprotyraspileleuleuaspaspgluthrhisasnargasn
290295300
aspglnglypheglulysmetileserglymettyrleuglygluile
305310315320
alaargleuvalphehisargmetalaglngluseraspvalphegly
325330335
aspalaalaaspserleuserasnpropheileleuserthrprophe
340345350
leualaalailearggluaspaspserproaspleusergluvalarg
355360365
argileleuarggluhisleulysileproaspalaproleulysthr
370375380
argargleuvalvallysvalcysaspilevalthrargargalaala
385390395400
argleualaalaalaglyilevalglyileleulyslysleuglyarg
405410415
aspglyserglyalaalaserserglyargglyargglyglnproarg
420425430
argthrvalvalalailegluglyglyleutyrglnglytyrproval
435440445
pheargglutyrleuaspglualaleuvalgluileleuglygluglu
450455460
valalaargasnvalthrleuargvalthrgluaspglyserglyval
465470475480
glyalaalaleuleualaalavalhisserserasnargglnglngln
485490495
glyglyproile
500
<210>3
<211>1503
<212>dna
<213>水稻(oryzasativa)
<220>
<221>gene
<222>(1)..(1503)
<220>
<221>cds
<222>(1)..(1503)
<400>3
atggggagggtggggctcggcgtggcggtggggtgcgcggcggtgacc48
metglyargvalglyleuglyvalalavalglycysalaalavalthr
151015
tgcgcgatcgccgcggcgctcgtggcgcgcagggcgtcggcgcgggcg96
cysalailealaalaalaleuvalalaargargalaseralaargala
202530
cggtggcggcgggcggtggcgctgctgcgggagttcgaggaggggtgt144
argtrpargargalavalalaleuleuargglupheglugluglycys
354045
gccacgccgcccgcgaggctgcgccaggtcgtggacgccatggtcgtc192
alathrproproalaargleuargglnvalvalaspalametvalval
505560
gagatgcacgccggcctcgcgtccgatggcggcagcaagctcaagatg240
glumethisalaglyleualaseraspglyglyserlysleulysmet
65707580
ctgctcaccttcgtcgacgcgctccccagcgggagtgaagaaggtgta288
leuleuthrphevalaspalaleuproserglyserglugluglyval
859095
tattattctattgatcttggaggaacaaacttcagagtcttgagggta336
tyrtyrserileaspleuglyglythrasnpheargvalleuargval
100105110
caagttggtgcgggatctgtgatcgtcaaccaaaaggttgaacagcaa384
glnvalglyalaglyservalilevalasnglnlysvalgluglngln
115120125
cccatccctgaggaactgaccaaaggcactactgagggtttattcaac432
proileproglugluleuthrlysglythrthrgluglyleupheasn
130135140
tttgttgccctggcactaaagaattttcttgaaggagaagatgaccaa480
phevalalaleualaleulysasnpheleugluglygluaspaspgln
145150155160
gatggaaaaatggcacttggttttacattttctttccctgttagacaa528
aspglylysmetalaleuglyphethrpheserpheprovalarggln
165170175
atttcagtgtcttcagggtcattaattaggtggacaaaaggattttcc576
ileservalserserglyserleuileargtrpthrlysglypheser
180185190
atcagagacacggttggcagagatgttgctcagtgcttaaatgaagcg624
ileargaspthrvalglyargaspvalalaglncysleuasngluala
195200205
cttgccaattgtgggctaaatgtgcgggtcactgcattggtgaatgat672
leualaasncysglyleuasnvalargvalthralaleuvalasnasp
210215220
acagtggggacattagctctagggcattactatgatgaagacacagtg720
thrvalglythrleualaleuglyhistyrtyraspgluaspthrval
225230235240
gctgctgtgataattgggtctggcaccaacgcttgctacattgaacgc768
alaalavalileileglyserglythrasnalacystyrilegluarg
245250255
actgatgcaattatcaagtgccagggtcttctaacgaactctggaggc816
thraspalaileilelyscysglnglyleuleuthrasnserglygly
260265270
atggtagtaaacatggagtgggggaatttctggtcatcacatttgcca864
metvalvalasnmetglutrpglyasnphetrpserserhisleupro
275280285
aggacgccatatgacatcttgctggatgatgaaacacacaatcgcaat912
argthrprotyraspileleuleuaspaspgluthrhisasnargasn
290295300
gatcagggctttgagaaaatgatatcaggaatgtatcttggggaaatt960
aspglnglypheglulysmetileserglymettyrleuglygluile
305310315320
gcaagattggtatttcatagaatggcccaggaatcagatgtttttggt1008
alaargleuvalphehisargmetalaglngluseraspvalphegly
325330335
gatgctgctgatagtctatccaaccctttcattttgagcacaccgttt1056
aspalaalaaspserleuserasnpropheileleuserthrprophe
340345350
ctggccgcaattcgcgaggacgattcaccagatctgagcgaagtcaga1104
leualaalailearggluaspaspserproaspleusergluvalarg
355360365
aggatacttcgagaacatctgaagattcccgatgctcctctgaaaact1152
argileleuarggluhisleulysileproaspalaproleulysthr
370375380
cgacggctggtcgtgaaagtctgcgacattgtgactcgcagagccgcc1200
argargleuvalvallysvalcysaspilevalthrargargalaala
385390395400
cgtctagccgctgcaggcatcgtggggatactgaagaagctggggagg1248
argleualaalaalaglyilevalglyileleulyslysleuglyarg
405410415
gatgggagcggcgcggcgtcgagcgggagaggtagagggcagccgagg1296
aspglyserglyalaalaserserglyargglyargglyglnproarg
420425430
aggacggtggtggcgatcgagggcgggctgtaccagggttacccggtg1344
argthrvalvalalailegluglyglyleutyrglnglytyrproval
435440445
ttcagggagtacctggacgaggccctggtggagatcctgggggaggag1392
pheargglutyrleuaspglualaleuvalgluileleuglygluglu
450455460
gtggcgcggaacgtgacgctgagggtgacggaggatgggtcgggggtc1440
valalaargasnvalthrleuargvalthrgluaspglyserglyval
465470475480
ggggctgccctcctcgccgccgtacattcgtcgaatagacagcaacaa1488
glyalaalaleuleualaalavalhisserserasnargglnglngln
485490495
ggaggtcccatatag1503
glyglyproile
500
<210>4
<211>500
<212>prt
<213>水稻(oryzasativa)
<400>4
metglyargvalglyleuglyvalalavalglycysalaalavalthr
151015
cysalailealaalaalaleuvalalaargargalaseralaargala
202530
argtrpargargalavalalaleuleuargglupheglugluglycys
354045
alathrproproalaargleuargglnvalvalaspalametvalval
505560
glumethisalaglyleualaseraspglyglyserlysleulysmet
65707580
leuleuthrphevalaspalaleuproserglyserglugluglyval
859095
tyrtyrserileaspleuglyglythrasnpheargvalleuargval
100105110
glnvalglyalaglyservalilevalasnglnlysvalgluglngln
115120125
proileproglugluleuthrlysglythrthrgluglyleupheasn
130135140
phevalalaleualaleulysasnpheleugluglygluaspaspgln
145150155160
aspglylysmetalaleuglyphethrpheserpheprovalarggln
165170175
ileservalserserglyserleuileargtrpthrlysglypheser
180185190
ileargaspthrvalglyargaspvalalaglncysleuasngluala
195200205
leualaasncysglyleuasnvalargvalthralaleuvalasnasp
210215220
thrvalglythrleualaleuglyhistyrtyraspgluaspthrval
225230235240
alaalavalileileglyserglythrasnalacystyrilegluarg
245250255
thraspalaileilelyscysglnglyleuleuthrasnserglygly
260265270
metvalvalasnmetglutrpglyasnphetrpserserhisleupro
275280285
argthrprotyraspileleuleuaspaspgluthrhisasnargasn
290295300
aspglnglypheglulysmetileserglymettyrleuglygluile
305310315320
alaargleuvalphehisargmetalaglngluseraspvalphegly
325330335
aspalaalaaspserleuserasnpropheileleuserthrprophe
340345350
leualaalailearggluaspaspserproaspleusergluvalarg
355360365
argileleuarggluhisleulysileproaspalaproleulysthr
370375380
argargleuvalvallysvalcysaspilevalthrargargalaala
385390395400
argleualaalaalaglyilevalglyileleulyslysleuglyarg
405410415
aspglyserglyalaalaserserglyargglyargglyglnproarg
420425430
argthrvalvalalailegluglyglyleutyrglnglytyrproval
435440445
pheargglutyrleuaspglualaleuvalgluileleuglygluglu
450455460
valalaargasnvalthrleuargvalthrgluaspglyserglyval
465470475480
glyalaalaleuleualaalavalhisserserasnargglnglngln
485490495
glyglyproile
500
- 还没有人留言评论。精彩留言会获得点赞!