一种适用于单细胞真核藻类基因组大小的测定方法与流程
本发明涉及一种适用于单细胞真核藻类基因组大小的测定方法,通过流式细胞分析和二代测序技术联合分析藻类基因组大小,特别涉及微藻物种。
背景技术:
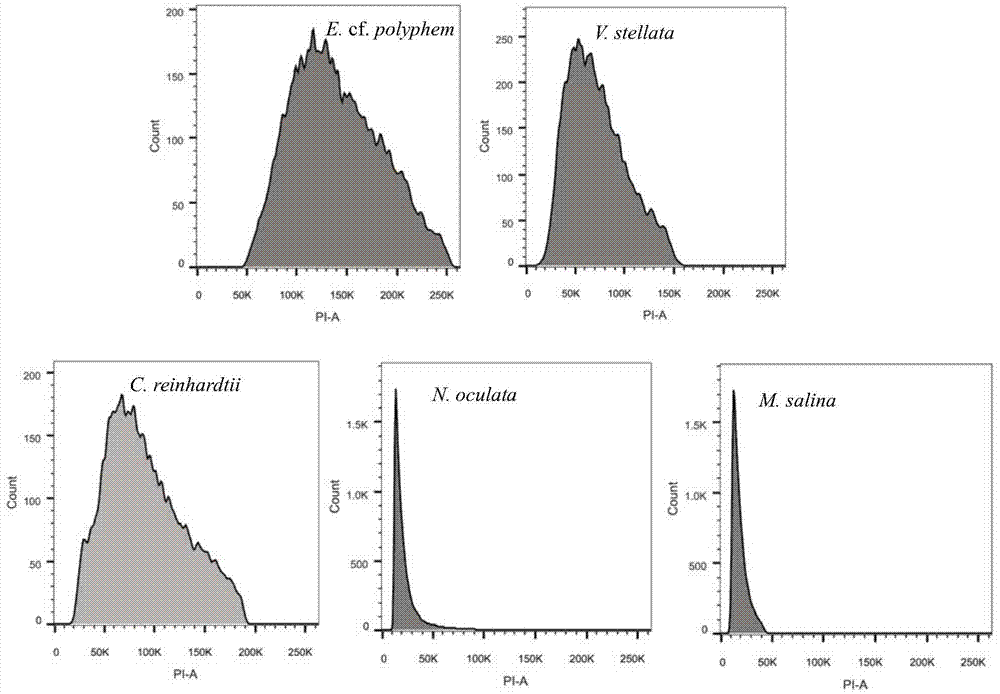
:为了确定一个未知物种复杂性,首先需要从基因组水平了解基因组的特征,快速获得未知的物种基因组大小。通常使用流式细胞术(flowcytometry,fcm),利用外标法或内标法,利用碘化丙啶(pi)对细胞核进行荧光染色后,利用流式细胞仪进行测定。fcm是目前最普遍使用的估测物种基因组大小的方法之一,相比较早的脉冲场凝胶电泳等手段,具有操作简单,样品制备流程短等显著优点,在动物、植物以及部分真菌中有较好应用。但对于不同物种,特别是不同类型的物种需要优化fcm的测定方法,如细胞壁破碎、细胞核提取和染色条件优化,提高fcm技术的准确性。微藻(单细胞真核藻类)是一类系统发生各异、个体较小、通常为单细胞或群体的、能进行光合作用的水生低等生物。目前,对于单细胞真核微藻物种的基因组评估的研究较少,由于其细胞较小,且通常含有较厚的细胞壁,加之细胞内存在大量的色素和次生代谢产物,会降低pi的荧光强度。此外,由于不同藻类物种之间的进化尺度较大,同一科的物种基因组大小差异可能会很大,因此,对于内参物种的选择严重地影响了流式细胞法测定基因组大小的准确性,需要我们进一步明确适合于单细胞真核藻类基因组测定方法。技术实现要素:为了解决目前单细胞真核微藻基因组大小准确测定的技术困难,本发明的目的在于提供一种适用于单细胞真核藻类基因组大小的测定方法。目前基于二代测序技术的基因组小片段文库的低深度测序数据,通过k-mer分析,能够有效地评估基因组大小、gc含量、杂合度以及重复序列的含量等相关信息,也是全面了解某一物种基因组特征的有效方法。因此,我们优化流式细胞分析方法,并与高通量测序数据相结合,将准确全面地评估待测藻类物种基因组的大小,对后续藻类资源的系统学和基因组学研究具有重要理论意义。准确的获得基因组大小估测后,可为后续的遗传与进化分析及全基因组测序的组装策略的制定提供帮助。本发明的目的通过下述技术方案实现:一种适用于单细胞真核藻类基因组大小的测定方法,包括如下步骤:(1)材料准备:单细胞真核藻类无菌培养物,离心去上清,再用无菌水漂洗2~3次,去除培养基成分,获得藻细胞;(2)内参选择:仪器调试后进行样品荧光强度及基因组大小的预测,根据预测基因组大小选择莱茵衣藻(chlamydomonasreinhardtii)为主要内参,眼点拟纳绿球藻(nannochlropsisoculata)和/或盐生拟微绿球藻(microchloropsissalina)为第二内参,并依据内参基因组大小进行相互校正;(3)细胞壁破坏及色素的去除:待测藻类和内参藻类,按照每105个藻细胞加入1~2ml甲醇的量;将甲醇分别加入到待测藻类和内参藻类的藻细胞中,常温下磁力搅拌破坏细胞壁及叶绿体结构去除叶绿素的干扰,每次1~2h,离心去上清,重复提取3~5次,直到细胞变为白色;然后按照每105个藻细胞加入0.1~0.2mllysisbufferlb01重悬细胞,于冰上裂解处理,获得细胞核悬浮液;(4)藻细胞核染色:按照1ml细胞核悬浮液加入50μgpi、50μgrnase和5μlβ-巯基乙醇的量,混匀后避光染色,之后取细胞核悬浮液进行流式细胞仪分析测定待测藻类和内参藻类的荧光值,计算待测藻类基因组大小。为了更好的实现本发明的目的,还包括:(5)高通量测序验证待测藻类的基因组,综合评估待测藻类的基因组大小与复杂性。优选的,步骤(1)中所述的单细胞真核藻类无菌培养物中藻细胞密度为1×103~1×105个/ml;更优选为1×105个/ml。优选的,步骤(1)中所述的离心的速度为3000~5000rpm;更优选为3000rpm。优选的,步骤(3)中所述的待测藻类为真眼点藻纲藻类,更优选为类波氏真眼点藻(eustigmatoscf.polyphem)或星形魏氏藻(vischeriastellata)。优选的,步骤(3)中所述的离心的速度为3000~5000rpm;更优选为3000rpm。步骤(3)中所述的lysisbufferlb01的配方为:以200ml计,363.4mgtris、148.9mgna2edta、34.8mgsperminetetrahydrochloride(四盐酸精胺)、1.193gkcl、233.8mgnacl和200μl0.1%(v/v)tritonx-100。优选的,步骤(3)中所述的裂解处理的时间为0.5~1h;更优选为0.5h。优选的,步骤(4)中所述的避光染色的条件为于4℃,避光染色30~60min。优选的,步骤(4)中所述的流式细胞仪分析的条件如下:激发波长488nm,流速20μl/min,每次至少收集104个细胞,重复3次以上,变异系数<5%,利用flowjoxv10软件进行图像和数据的处理分析,获得待测藻类和内参藻类的pi荧光强度(pi-a);单细胞真核藻类基因组大小可由下列计算公式给出:待测藻类基因组大小=待测藻类荧光强度/(内参藻类荧光强度×内参藻类基因组大小)。优选的,步骤(5)所述的高通量测序的步骤如下:(a)材料准备:单细胞真核藻类无菌培养液,离心去上清,收集藻泥100~200mg,提取样品dna;(b)dna测序:构建一个170bp~350bp的小片段dna文库,利用illumina或者bgi500平台对进行双末端pe150bp测序,测序数据量为5~10g,测序深度不低于30×,测序由深圳华大基因科技服务有限公司完成;(c)数据分析:利用fastqc软件检测碱基质量,gc分离、gc峰图检测是否污染、重复序列水平;然后利用ngsqctoolkit软件去除接头序列和过滤低质量reads,进一步利用bless软件对reads中错误碱基进行修正;获得高质量的cleanreads后,利用gce(genomecharacteristicsestimation)软件,基于kmer-17评估基因组的大小、重复序列含量和杂合率等。(d)利用soapdenovo软件对目的基因组进行初步组装,然后利用samtools软件提取gc含量与测序深度,计算藻细胞的gc含量分布图,分析基因组是否受到其他外源物种的污染。优选的,步骤(a)中所述的离心的速度为3000~5000rpm;更优选为3000rpm。优选的,步骤(b)中所述的小片段dna文库为270bp的小片段dna文库;优选的,步骤(b)中所述的测序数据量为10g。优选的,步骤(b)中所述的测序深度为30×~100×。本发明根据单细胞藻类的性质,从流式细胞仪分析和高通量测序分析两个方法入手,综合准确地评估基因组大小,该方法具有较高的可操作性和准确性,完全能满足后续的相关研究。本发明相对于现有技术具有如下的优点及效果:本发明基于单细胞真核藻类的特性,优化了细胞核的提取与染色方法,获得了较为准确的流式细胞分析结果,并结合高通量二代illumina测序技术,综合评估藻细胞的基因组大小,明确基因组的特征。该技术方法优化了传统植物流式细胞分析,利用甲醇去除了藻细胞色素和细胞壁的干扰,大大的提高了pi的染色效果,提供了流式细胞分析的准确性,同时结合目前较为前沿的高通量测序技术,综合分析了待测物种基因组结构特征,可为后续物种的遗传结构研究和全基因组测序的组装策略的制定提供理论依据。附图说明图1是5种单细胞真核藻类荧光峰图。图2是5种藻类峰图及荧光值的比较。图3是dna样品琼脂糖凝胶电泳检测结果;其中,1,2:类波氏真眼点藻;3,4:星形魏氏藻;m1:λ-hindⅲdigest(takara);m2:d2000(tiangen)。图4是fastqc分析碱基质量分布图。图5是类波氏真眼点藻kmer-17频率分布图图6是星形魏氏藻kmer-17频率分布图。图7是类波氏真眼点藻gc含量与scaffolds测序深度分布图。图8是星形魏氏藻gc含量与scaffolds测序深度分布图。具体实施方式下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。下列实施例中未注明具体实验条件的试验方法,通常按照常规实验条件或按照制造厂所建议的实验条件。所使用的材料、试剂等,如无特殊说明,为从商业途径得到的试剂和材料。所述的类波氏真眼点藻(eustigmatoscf.polyphem)h4302购买于捷克布拉格查尔斯大学。星形魏氏藻(vischeriastellata)sag33.83、莱茵衣藻(chlamydomonasreinhardtii)sag54.72、眼点拟纳绿球藻(nannochlropsisoculata)sag38.85和盐生拟微绿球藻(microchloropsissalina)sag40.85均购买于德国哥根廷大学藻种保藏中心。实施例1以两株未知基因组的单细胞真核微藻类波氏真眼点藻(e.cf.polyphem)与星形魏氏藻(v.stellata)为例,分别以莱茵衣藻(c.reinhardtii)为第一内参,眼点拟纳绿球藻(n.oculata)和盐生拟微绿球藻(m.salina)为第二内参,3个已知物种基因组相互校正。5种微藻培养物(约50ml,1×105个/ml)分别离心后,加入50ml甲醇,磁力搅拌,每次1~2h,3000rpm离心去上清,重复提取3~5次,直到细胞变为白色;离心去上清后加入5mllysisbufferlb01重悬细胞,处理裂解细胞核0.5h,获得细胞核悬浮液,于冰上备用;取1ml细胞核悬浮液,加入50μlpi(1mg/ml)、50μlrnase(1mg/ml)和5μlβ-巯基乙醇,混匀后于4℃,避光染色0.5h;取染色后的细胞悬浮液300μl,加入到流式专用圆底试管(fluorescenceactivatedcellsorter,facs)中,于bdfacscantoii流式细胞仪中进行分析,激发波长488nm,流速20μl/min,每次收集不低于104个细胞,重复3次,变异系数<5%,利用flowjoxv10软件进行图像和数据的处理分析,得到荧光峰图,如图1和图2。由此可获得,类波氏真眼点藻与星形魏氏藻的荧光值分别为140109和91119,如表1,获得待测样品和已知基因组大小样品的pi荧光强度(表1),根据基因组大小的计算公式计算:待测藻类基因组大小=待测藻类荧光强度/(内参藻类荧光强度×内参藻类基因组大小)。最终,估测的星形魏氏藻基因组约为90~110mb,类波氏真眼点藻基因组约为180~220mb。表1流式细胞估测基因组大小流式细胞获得了一个初步的估计结果,为了进一步确证结果,利用takara植物dna提取试剂盒(minibestplantgenomicdnaextractionkit)分别提取待测的类波氏真眼点藻与星形魏氏藻的全基因组dna,分别提取2管dna,利用qubitfluorometer测定dna浓度和纯度检测,见表2,然后利用琼脂糖凝胶电泳检测样品的完整性,图3。表2dna提取检测结果检测结果均显示,4管dna质量好,浓度>30ng/μl,每种藻dna总量>10μg,凝胶电泳图显示无蛋白,rna/盐离子等污染,样本未发生降解,主峰>20kb完整度高,满足文库构建的要求。构建一个270bp的小片段dna文库,利用illuminahiseq4000平台进行双末端pe150bp测序,测序数据量为10g,测序由深圳华大基因科技服务有限公司完成,如表3。测序质量经过fastqc质控,去除低质量的的序列(图4),进一步利用bless软件对reads中错误碱基进行修正,获得高质量的cleanread。表3测序原始数据统计获得cleanreads后,利用gce软件,获得两个物种的kmer-17频率分布图,如图5、图6。类波氏真眼点藻存在两个明显的峰,主要的深度峰值大约在27,根据公式genomesize=k-mernum/peakdepth,可以估算出该物种的基因组大小约为267mb。第二个深度峰值在61,表明基因组存在大量的重复序列。星形魏氏藻kmer-17频率分布图存在三个明显的峰,深度峰值分别为14,58和117(图6),主深度峰为58,根据公式genomesize=k-mernum/peakdepth,基因组大小约为103mb,另外两个峰值分别表示基因组存在一定比例的杂合和重复序列,如表4。表417-kmer分析统计利用测序获得的cleanread进行初步的组装,如表5,类波氏真眼点藻与星形魏氏藻的scaffolds总长度分别为220mb和114mb,n50分别为6328bp与19101bp。同时,选取2kbp以上的scaffolds,统计了scaffolds的覆盖度与gc含量的关系,计算两个藻gc含量分布图,如图7,类波氏真眼点藻的gc深度主要在30左右,对应的gc含量在50%左右。而星形魏氏藻,如图8,gc分布呈现出两部分的区域分布,两部分的重心分别在深度20左右和100左右,深度在20左右的中心区域是杂合scaffolds的gc分布区域,同时,两个深度对应的gc含量也在50%的位置,这和我们计算得到的基因组组装的gc含量基本一致,说明该基因组没有受到其他外源物种的污染。表5两株真眼点藻初步组装结果类波氏真眼点藻星形魏氏藻genomescaffoldssize(bp)230490304119222094genomecontigsize(bp)223863295116367984scaffoldsnumber27665896294contigsnumber400415153276gcrate(%)53.4%53.5%scaffoldn50632819101contign5020244378longestlength(bp)519339211848totallength>=1kb184992717104796597totallength>=2kb167673725100696179totallength>=3kb15308704197238189上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1