杀虫蛋白的用途的制作方法
本发明涉及一种杀虫蛋白的用途,特别是涉及一种cry1a蛋白质通过在植物中表达来控制草地螟为害植物的用途。
背景技术:
草地螟loxostegesticticalis,属于鳞翅目螟蛾科锥额野螟属,主要分布于东北、西北、华北一带,即吉林、内蒙古、黑龙江、宁夏、甘肃、青海、河北、山西、陕西、江苏等省。草地螟为多食性大害虫,可取食35科,200余种植物,主要为甜菜、大豆、向日葵、马铃薯、麻类、蔬菜、药材等多种作物;大发生时禾谷类作物、林木等均受其害。草地螟一般10年左右发生一次大暴发,其为害特点为:幼虫多集中在枝梢上结网躲藏,取食叶肉,留下表皮与叶脉,重者只剩下叶脉或被吃光,严重影响产量。
栽培大豆(glycinemax(l.)merri),是一种全球种植的作为植物油和植物蛋白主要来源的重要经济作物,是中国重要的粮食作物。大豆是草地螟最喜取食的植物之一,每年因草地螟会造成不同程度的粮食损失,轻者减产1-2成,重者减产3-4成。为了防治草地螟,人们通常采用的主要方法有农业防治、化学防治、物理防治和生物防治。
农业防治是把整个农田生态系统多因素的综合协调管理,调控作物、害虫、环境因素、创造一个有利于作物生长而不利于草地螟发生的农田生态环境。如对为害严重的田块实行轮作倒茬,使草地螟失去寄主;可在秋、冬多次灌水或开花期灌水,杀死幼虫;还可在草地螟集中越冬场所,采取秋翻、春耕方法,通过机械杀伤和土块压伤越冬幼虫,增加越冬幼虫死亡率;在草荒较重的田块,在幼虫为害迁移之前可以挖防虫沟,在沟内喷上粉剂农药或在中间立一道地膜,防止幼虫迁入田块扩散为害作物;在卵已产下而大部分未孵化时,结合中耕除草灭卵,将除掉的杂草带出田外沤肥或挖坑埋掉;同时要除净田边地埂的杂草,以免幼虫迁入农田危害;在幼虫已孵化的田块,一定要先打药后除草,以免加快幼虫向农作物转移而加重危害。
化学防治即药剂防治,是利用化学杀虫剂来杀灭害虫,是草地螟综合治理的重要组成部分,它具有快速、方便、简便和高经济效益的特点,特别是草地螟大发生的情况下,是必不可少的应急措施。目前化学防治方法主要是药液喷雾,在草地螟幼虫1-3龄期间有较好的防治效果,随着虫体越大,抗药性越强,药剂防治效果越差,将很难达到防治的目的。在龄期较大的幼虫集中危害的田块,当药剂防治效果不好时,可在该田块四周挖沟或打药带封锁,防治扩散危害。同时化学防治也有其局限性,如使用不当往往会导致农作物发生药害、害虫产生抗药性,以及杀伤天敌、污染环境,使农田生态系统遭到破坏和农药残留对人、畜的安全构成威胁等不良后果。
物理防治主要根据害虫对环境条件中各种物理因素的反应,利用各种物理因素如光、电、色、温湿度等以及机械设备进行诱杀、辐射不育等方法来防治害虫。成虫盛发时用网扑或灯光诱杀。利用草地螟迁飞、趋光的习性,设置高压汞灯、频振式杀虫灯等对其成虫进行诱杀;但是频振式杀虫灯需要每天及时清理高压电网上的污垢,否则会影响杀虫效果;并且在雷雨天不能开灯,在操作上还有电击伤人的危险;此外安装灯的一次性投入较大。
生物防治是利用某些有益生物或生物代谢产物来控制害虫种群数量,以达到降低或消灭害虫的目的,如用赤眼蜂或白僵菌防治草地螟。其特点是对人、畜安全,对环境污染少,对某些害虫可达到长期控制的目的;但是效果常不稳定,并且不论草地螟发生轻重均需同样投资进行。
为了解决农业防治、化学防治、物理防治和生物防治在实际应用中的局限性,科学家们经过研究发现将编码杀虫蛋白的抗虫基因转入植物中,可获得一些抗虫转基因植物以防治植物虫害。
cry1a杀虫蛋白是众多杀虫蛋白中的一种,是由苏云金芽孢杆菌产生的不溶性伴孢结晶蛋白。cry1a蛋白被昆虫摄入进入中肠,毒蛋白原毒素被溶解在昆虫中肠的碱性ph环境下。蛋白n-和c-末端被碱性蛋白酶消化,将原毒素转变成活性片段;活性片段和昆虫中肠上皮细胞膜上表面上受体结合,插入肠膜,导致细胞膜出现穿孔病征,破坏细胞膜内外的渗透压变化及ph平衡等,扰乱昆虫的消化过程,最终导致其死亡。
已证明转cry1a基因的植株可以抵抗玉米螟、棉铃虫、草地贪夜蛾(秋黏虫)等鳞翅目(lepidoptera)害虫的侵害,然而,至今尚无关于通过产生表达cry1a蛋白的转基因植株来控制草地螟对植物为害的报道。
技术实现要素:
本发明的目的是提供一种杀虫蛋白的用途,首次提供了通过产生表达cry1a蛋白的转基因植株来控制草地螟对植物危害的方法,且有效克服现有技术农业防治、化学防治、物理防治和生物防治等技术缺陷。
为实现上述目的,本发明提供了一种控制草地螟害虫的方法,包括将草地螟害虫至少与cry1a蛋白接触。
进一步地,所述cry1a蛋白存在于至少产生所述cry1a蛋白的宿主细胞中,所述草地螟害虫通过摄食所述宿主细胞至少与所述cry1a蛋白接触。
更进一步地,所述cry1a蛋白存在于至少产生所述cry1a蛋白的细菌或转基因植物中,所述草地螟害虫通过摄食所述细菌或转基因植物的组织至少与所述cry1a蛋白接触,接触后所述草地螟害虫生长受到抑制和/或导致死亡,以实现对草地螟危害植物的控制。
所述转基因植物的组织为根、叶片、茎秆、果实、雄穗、雌穗、花药或花丝。
所述植物为大豆、灰菜、甜菜、向日葵、马铃薯、麻类和玉米。
所述转基因植物可以处于任意生育期。
所述对草地螟危害植物的控制不因种植地点和/或种植时间的改变而改变。
所述接触步骤之前的步骤为种植含有编码所述cry1a蛋白的多核苷酸的植物。
优选地,所述cry1a蛋白为cry1ab蛋白、cry1ac蛋白或cry1a.105蛋白。
更优选地,所述cry1a蛋白具有seqidno:1、seqidno:3、seqidno:5、seqidno:7和seqidno:9所示的氨基酸序列。所述cry1a蛋白具有seqidno:2、seqidno:4、seqidno:6、seqidno:8和seqidno:10所示的核苷酸序列。
在上述技术方案的基础上,所述植物还可以包括至少一种不同于编码所述cry1a蛋白的核苷酸的第二种核苷酸。
进一步地,所述第二种核苷酸编码cry类杀虫蛋白质、vip类杀虫蛋白质、蛋白酶抑制剂、凝集素、α-淀粉酶或过氧化物酶。
在本发明中,cry1a蛋白在一种转基因植物中的表达可以伴随着一个或多个cry类杀虫蛋白质和/或vip类杀虫蛋白质的表达。这种超过一种的杀虫毒素在同一株转基因植物中共同表达可以通过遗传工程使植物包含并表达所需的基因来实现。另外,一种植物(第1亲本)可以通过遗传工程操作表达cry1a蛋白质,第二种植物(第2亲本)可以通过遗传工程操作表达cry类杀虫蛋白质和/或vip类杀虫蛋白质。通过第1亲本和第2亲本杂交获得表达引入第1亲本和第2亲本的所有基因的后代植物。
优选地,所述第二种核苷酸编码vip3a蛋白、cry2ab蛋白或cry1fa蛋白。
更优选地,所述第二种核苷酸编码具有seqidno:11、seqidno:13或seqidno:15所示的氨基酸序列。所述第二种核苷酸具有seqidno:12、seqidno:14或seqidno:16所示的核苷酸序列。
可选择地,所述第二种核苷酸为抑制目标昆虫害虫中重要基因的dsrna。
为实现上述目的,本发明还提供了一种cry1a蛋白质控制草地螟害虫的用途。
本发明还提供了一种产生控制草地螟害虫的植物的方法,包括向所述植物的基因组中引入编码cry1a蛋白的多核苷酸序列。
本发明还提供了一种产生控制草地螟害虫的植物繁殖体的方法,包括将由所述方法获得的第一植株与第二植株杂交,和/或取下由所述方法获得的植株上具有繁殖能力的组织进行培养,从而产生含有编码cry1a蛋白的多核苷酸序列的植物繁殖体。
本发明还提供了一种培养控制草地螟害虫的植物的方法,包括:
种植至少一个植物繁殖体,所述植物繁殖体的基因组中包括编码cry1a蛋白的多核苷酸序列;
使所述植物繁殖体长成植株;
使所述植株在人工接种草地螟害虫和/或草地螟害虫自然发生危害的条件下生长,收获与其他不具有编码cry1a蛋白的多核苷酸序列的植株相比具有减弱的植物损伤和/或具有增加的植物产量的植株。
本发明中所述的“植物繁殖体”包括但不限于植物有性繁殖体和植物无性繁殖体。所述植物有性繁殖体包括但不限于植物种子;所述植物无性繁殖体是指植物体的营养器官或某种特殊组织,其可以在离体条件下产生新植株;所述营养器官或某种特殊组织包括但不限于根、茎和叶,例如:以根为无性繁殖体的植物包括草莓和甘薯等;以茎为无性繁殖体的植物包括甘蔗和马铃薯(块茎)等;以叶为无性繁殖体的植物包括芦荟和秋海棠等。
本发明中所述的“接触”是指碰触、停留和/或摄食,具体为昆虫和/或害虫触碰、停留和/或摄食植物、植物器官、植物组织或植物细胞,所述植物、植物器官、植物组织或植物细胞既可以是其体内表达杀虫蛋白,还可以是所述植物、植物器官、植物组织或植物细胞的表面具有杀虫蛋白和/或具有产生杀虫蛋白的微生物。
本发明所述的“控制”和/或“防治”是指草地螟害虫至少与cry1a蛋白接触,接触后草地螟害虫生长受到抑制和/或导致死亡。进一步地,草地螟害虫通过摄食植物组织至少与cry1a蛋白接触,接触后全部或部分草地螟害虫生长受到抑制和/或导致死亡。抑制是指亚致死,即尚未致死但能引起生长发育、行为、生理、生化和组织等方面的某种效应,如生长发育缓慢和/或停止。同时,植物在形态上应是正常的,且可在常规方法下培养以用于产物的消耗和/或生成。此外,含有编码cry1a蛋白的多核苷酸序列的控制草地螟害虫的植物和/或植物繁殖体,在人工接种草地螟害虫和/或草地螟害虫自然发生危害的条件下,与非转基因的野生型植株相比具有减弱的植物损伤,具体表现包括但不限于改善的叶片抗性、和/或提高的籽粒重量、和/或增产等。cry1a蛋白对草地螟的“控制”和/或“防治”作用是可以独立存在的,具体地,转基因植物(含有编码cry1a蛋白的多核苷酸序列)的任何组织同时和/或不同步地,存在和/或产生,cry1a蛋白和/或可控制草地螟害虫的另一种物质,则所述另一种物质的存在既不影响cry1a对草地螟的“控制”和/或“防治”作用,也不能导致所述“控制”和/或“防治”作用完全和/或部分由所述另一种物质实现,而与cry1a蛋白无关。通常情况下,在大田,草地螟害虫摄食植物组织的过程短暂且很难用肉眼观察到,因此,在人工接种草地螟害虫和/或草地螟害虫自然发生危害的条件下,如转基因植物(含有编码cry1a蛋白的多核苷酸序列)的任何组织存在死亡的草地螟害虫、和/或在其上停留生长受到抑制的草地螟害虫、和/或与非转基因的野生型植株相比具有减弱的植物损伤,即为实现了本发明的方法和/或用途,即通过草地螟害虫至少与cry1a蛋白接触以实现控制草地螟害虫的方法和/或用途。
rna干扰(rnainterference,rnai)是指在进化过程中高度保守的、由双链rna(double-strandedrna,dsrna)诱发的、同源mrna高效特异性降解的现象。因此在本发明中可以使用rnai技术特异性剔除或关闭目标昆虫害虫中特定基因的表达,特别是与目标昆虫害虫生长发育的相关的基因。
草地螟的成虫淡褐色,体长8-10mm,前翅灰褐色,外缘有淡黄色条纹,翅中央近前缘有一深黄色斑,顶角内侧前缘有不明显的三角形浅黄色小斑,后翅浅灰黄色,有两条与外缘平行的波状纹。卵呈椭圆形,长0.8-1.2mm,为3、5粒或7、8粒串状粘成复瓦状的卵块。幼虫共5龄,老熟幼虫16-25mm,1龄淡绿色,体背有许多暗褐色纹;3龄灰绿色,体侧有淡色纵带,周身有毛瘤;5龄多为灰黑色,两侧有鲜黄色线条。蛹长14-20mm,背部各节有14个赤褐色小点,排列于两侧,尾刺8根。
草地螟分布于我国北方地区,一年发生2-4代,成虫飞翔力弱,喜食花蜜,卵散产于叶背主脉两侧,常3-4粒在一起,以距地面2-8cm的茎叶上最多。初孵幼虫多集中在枝梢上结网躲藏,取食叶肉,3龄后食量剧增,幼虫共5龄。草地螟以老熟幼虫在土内吐丝作茧越冬。越冬幼虫在翌春,随着日照增长和气温回升,开始化蛹,一般在5月下至6月上旬进入羽化盛期。成虫羽化后,从越冬地迁往发生地,在发生地繁殖1-2代后,再迁往越冬地,产卵繁殖到老熟幼虫入土越冬。
在分类系统上,一般主要根据成虫翅的脉序、连锁方式和触角的类型等形态特征,将鳞翅目分为亚目、总科、科等。而螟蛾科是鳞翅目中种类最多的科之一,全世界已发现1万种以上,仅中国记录就有几千条。大部分螟蛾科昆虫是农作物的害虫,多数以蛀茎形式为害,如二化螟和玉米螟。尽管玉米螟和草地螟同属于鳞翅目螟蛾科,除了在分类标准上存在相似性,在其它形态结构上则存在极大差异;就好比植物中的草莓与苹果一样(同属于蔷薇目蔷薇科),它们都有花两性,辐射对称,花瓣5片等特征,但是其果实以及植株形态却是千差万别。但因人们较少接触昆虫,尤其是较少接触农业害虫,对于昆虫形态上的差异较少关注,而使得人们以为昆虫的形态大同小异。而事实上,草地螟不管是从幼虫形态还是成虫形态上来看,都具有其独特的特征。
同属螟蛾科的昆虫不仅在形态特征上存在较大差异,同时在取食习性上,也存在差异。例如同为螟蛾科的玉米螟主要为害禾本科的玉米,极少为害其它阔叶类作物。而草地螟喜取食灰菜、甜菜和大豆等植物,大发生时才为害禾谷类作物、林木等。取食习性的不同,也暗示着体内消化系统所产生的酶和受体蛋白不同。而消化道中产生的酶是bt基因起作用的关键点,只有能够与特异性bt基因相结合的酶或受体蛋白,才有可能使得某个bt基因对该害虫具有抗虫效果。越来越多的研究表明,同目不同科、甚至同科不同种的昆虫对同种bt蛋白的敏感性表现不同。例如vip3aa基因对螟蛾科的二化螟chilosuppressalis和亚洲玉米螟ostriniafurnacalis都表现出了抗虫活性,但是vip3aa基因对于同属螟蛾科的印度谷螟plodiainterpunctella和欧洲玉米螟ostrinianubilalis却没有抗虫效果。上述几种害虫均属于鳞翅目螟蛾科,但同种bt蛋白对这几种螟蛾科害虫表现出不同的抗性效果。尤其是欧洲玉米螟和亚洲玉米螟在分类上甚至同属于螟蛾科ostrinia属(同目同科同属),但是其对同种bt蛋白的反应却是截然不同的,更加充分说明了bt蛋白与昆虫体内酶和受体的相互作用方式是复杂且难以预料的。
本发明中所述的植物、植物组织或植物细胞的基因组,是指植物、植物组织或植物细胞内的任何遗传物质,且包括细胞核和质体和线粒体基因组。
本发明中所述的多核苷酸和/或核苷酸形成完整“基因”,在所需宿主细胞中编码蛋白质或多肽。本领域技术人员很容易认识到,可以将本发明的多核苷酸和/或核苷酸置于目的宿主中的调控序列控制下。
本领域技术人员所熟知的,dna典型的以双链形式存在。在这种排列中,一条链与另一条链互补,反之亦然。由于dna在植物中复制产生了dna的其它互补链。这样,本发明包括对序列表中示例的多核苷酸及其互补链的使用。本领域常使用的“编码链”指与反义链结合的链。为了在体内表达蛋白质,典型将dna的一条链转录为一条mrna的互补链,它作为模板翻译出蛋白质。mrna实际上是从dna的“反义”链转录的。“有义”或“编码”链有一系列密码子(密码子是三个核苷酸,一次读三个可以产生特定氨基酸),其可作为开放阅读框(orf)阅读来形成目的蛋白质或肽。本发明还包括与示例的dna有相当功能的rna。
本发明中核酸分子或其片段在严格条件下与本发明cry1a基因杂交。任何常规的核酸杂交或扩增方法都可以用于鉴定本发明cry1a基因的存在。核酸分子或其片段在一定情况下能够与其他核酸分子进行特异性杂交。本发明中,如果两个核酸分子能形成反平行的双链核酸结构,就可以说这两个核酸分子彼此间能够进行特异性杂交。如果两个核酸分子显示出完全的互补性,则称其中一个核酸分子是另一个核酸分子的“互补物”。本发明中,当一个核酸分子的每一个核苷酸都与另一个核酸分子的对应核苷酸互补时,则称这两个核酸分子显示出“完全互补性”。如果两个核酸分子能够以足够的稳定性相互杂交从而使它们在至少常规的“低度严格”条件下退火且彼此结合,则称这两个核酸分子为“最低程度互补”。类似地,如果两个核酸分子能够以足够的稳定性相互杂交从而使它们在常规的“高度严格”条件下退火且彼此结合,则称这两个核酸分子具有“互补性”。从完全互补性中偏离是可以允许的,只要这种偏离不完全阻止两个分子形成双链结构。为了使一个核酸分子能够作为引物或探针,仅需保证其在序列上具有充分的互补性,以使得在所采用的特定溶剂和盐浓度下能形成稳定的双链结构。
本发明中,基本同源的序列是一段核酸分子,该核酸分子在高度严格条件下能够和相匹配的另一段核酸分子的互补链发生特异性杂交。促进dna杂交的适合的严格条件,例如,大约在45℃条件下用6.0×氯化钠/柠檬酸钠(ssc)处理,然后在50℃条件下用2.0×ssc洗涤,这些条件对本领域技术人员是公知的。例如,在洗涤步骤中的盐浓度可以选自低度严格条件的约2.0×ssc、50℃到高度严格条件的约0.2×ssc、50℃。此外,洗涤步骤中的温度条件可以从低度严格条件的室温约22℃,升高到高度严格条件的约65℃。温度条件和盐浓度可以都发生改变,也可以其中一个保持不变而另一个变量发生改变。优选地,本发明所述严格条件可为在6×ssc、0.5%sds溶液中,在65℃下与seqidno:2、seqidno:4、seqidno:6、seqidno:8、seqidno:10、seqidno:12、seqidno:14和seqidno:16发生特异性杂交,然后用2×ssc、0.1%sds和1×ssc、0.1%sds各洗膜1次。
因此,具有抗虫活性并在严格条件下与本发明seqidno:2、seqidno:4、seqidno:6、seqidno:8、seqidno:10、seqidno:12、seqidno:14和seqidno:16杂交的序列包括在本发明中。这些序列与本发明序列至少大约40%-50%同源,大约60%、65%或70%同源,甚至至少大约75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更大的序列同源性。
本发明中所述的基因和蛋白质不但包括特定的示例序列,还包括保存了所述特定示例的蛋白质的杀虫活性特征的部分和/片段(包括与全长蛋白质相比在内和/或末端缺失)、变体、突变体、取代物(有替代氨基酸的蛋白质)、嵌合体和融合蛋白。所述“变体”或“变异”是指编码同一蛋白或编码有杀虫活性的等价蛋白的核苷酸序列。所述“等价蛋白”是指与权利要求的蛋白具有相同或基本相同的抗草地螟害虫的生物活性的蛋白。
本发明中所述的dna分子或蛋白序列的“片段”或“截短”是指涉及的原始dna或蛋白序列(核苷酸或氨基酸)的一部分或其人工改造形式(例如适合植物表达的序列),前述序列的长度可存在变化,但长度足以确保(编码)蛋白质为昆虫毒素。
使用标准技术可以修饰基因和容易的构建基因变异体。例如,本领域熟知制造点突变的技术。又例如美国专利号5605793描述了在随机断裂后使用dna重装配产生其它分子多样性的方法。可以使用商业化核酸内切酶制造全长基因的片段,并且可以按照标准程序使用核酸外切酶。例如,可以使用酶诸如bal31或定点诱变从这些基因的末端系统地切除核苷酸。还可以使用多种限制性内切酶获取编码活性片段的基因。可以使用蛋白酶直接获得这些毒素的活性片段。
本发明可以从bt分离物和/或dna文库衍生出等价蛋白和/或编码这些等价蛋白的基因。有多种方法获取本发明的杀虫蛋白。例如,可以使用本发明公开和要求保护的杀虫蛋白的抗体从蛋白质混合物鉴定和分离其它蛋白。特别地,抗体可能是由蛋白最恒定和与其它bt蛋白最不同的蛋白部分引起的。然后可以通过免疫沉淀、酶联免疫吸附测定(elisa)或western印迹方法使用这些抗体专一地鉴定有特征活性的等价蛋白。可使用本领域标准程序容易的制备本发明中公开的蛋白或等价蛋白或这类蛋白的片段的抗体。然后可以从微生物中获得编码这些蛋白的基因。
由于遗传密码子的丰余性,多种不同的dna序列可以编码相同的氨基酸序列。产生这些编码相同或基本相同的蛋白的可替代dna序列正在本领域技术人员的技术水平内。这些不同的dna序列包括在本发明的范围内。所述“基本上相同的”序列是指有氨基酸取代、缺失、添加或插入但实质上不影响杀虫活性的序列,亦包括保留杀虫活性的片段。
本发明中氨基酸序列的取代、缺失或添加是本领域的常规技术,优选这种氨基酸变化为:小的特性改变,即不显著影响蛋白的折叠和/或活性的保守氨基酸取代;小的缺失,通常约1-30个氨基酸的缺失;小的氨基或羧基端延伸,例如氨基端延伸一个甲硫氨酸残基;小的连接肽,例如约20-25个残基长。
保守取代的实例是在下列氨基酸组内发生的取代:碱性氨基酸(如精氨酸、赖氨酸和组氨酸)、酸性氨基酸(如谷氨酸和天冬氨酸)、极性氨基酸(如谷氨酰胺、天冬酰胺)、疏水性氨基酸(如亮氨酸、异亮氨酸和缬氨酸)、芳香氨基酸(如苯丙氨酸、色氨酸和酪氨酸),以及小分子氨基酸(如甘氨酸、丙氨酸、丝氨酸、苏氨酸和甲硫氨酸)。通常不改变特定活性的那些氨基酸取代在本领域内是众所周知的,并且已由,例如,n.neurath和r.l.hill在1979年纽约学术出版社(academicpress)出版的《protein》中进行了描述。最常见的互换有ala/ser,val/ile,asp/glu,thu/ser,ala/thr,ser/asn,ala/val,ser/gly,tyr/phe,ala/pro,lys/arg,asp/asn,leu/ile,leu/val,ala/glu和asp/gly,以及它们相反的互换。
对于本领域的技术人员而言显而易见地,这种取代可以在对分子功能起重要作用的区域之外发生,而且仍产生活性多肽。对于由本发明的多肽,其活性必需的并因此选择不被取代的氨基酸残基,可以根据本领域已知的方法,如定点诱变或丙氨酸扫描诱变进行鉴定(如参见,cunningham和wells,1989,science244:1081-1085)。后一技术是在分子中每一个带正电荷的残基处引入突变,检测所得突变分子的抗虫活性,从而确定对该分子活性而言重要的氨基酸残基。底物-酶相互作用位点也可以通过其三维结构的分析来测定,这种三维结构可由核磁共振分析、结晶学或光亲和标记等技术测定(参见,如devos等,1992,science255:306-312;smith等,1992,j.mol.biol224:899-904;wlodaver等,1992,febsletters309:59-64)。
在本发明中,cry1a蛋白包括但不限于cry1ab、cry1ac蛋白或cry1a.105、,或者与seqidno:1、seqidno:3、seqidno:5、seqidno:7、seqidno:9、seqidno:11、seqidno:13或seqidno:15所示的氨基酸序列具有一定同源性。这些序列与本发明序列类似性/相同性典型的大于78%,优选的大于85%,更优选的大于90%,甚至更优选的大于95%,并且可以大于99%。也可以根据更特定的相同性和/或类似性范围定义本发明的优选的多核苷酸和蛋白质。例如与本发明示例的序列有78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的相同性和/或类似性。
在本发明中,产生所述cry1a蛋白的转基因植物包括但不限于mon87701转基因大豆事件和/或包含mon87701转基因大豆事件的植物材料(如在cn101861392b所描述的)、mon87751转基因大豆事件和/或包含mon87751转基因大豆事件的植物材料(如在cn105531376a所描述的)、das81419(9582.814.19.1)转基因大豆事件和/或包含das81419(9582.814.19.1)转基因大豆事件的植物材料(如在cn103826444a、cn103826445a和/或cn103827132b所描述的)、9582.816.15.1转基因大豆事件和/或包含9582.816.15.1转基因大豆事件的植物材料(如在cn104583404a和/或cn104718293a所描述的)、mon810转基因玉米事件和/或包含mon810转基因玉米事件的植物材料(如在us6713259b2所描述的)、bt11转基因玉米事件和/或包含bt11转基因玉米事件的植物材料(如在usdaaphis非管制状态申请95-195-01p所描述的,其所包含的cry1ab蛋白的氨基酸序列如本发明seqidno:3所示)、bt176转基因玉米事件和/或包含bt176转基因玉米事件的植物材料(如在usdaaphis非管制状态申请94-319-01p所描述的,其所包含的cry1ab蛋白的氨基酸序列如us5625136b2所描述的)、tt51转基因水稻事件和/或包含tt51转基因水稻事件的植物材料(如在cn100582223c和cn101302520b所描述的)、223f-s21转基因水稻品系和/或包含223f-s21转基因水稻品系的植物材料(如在cn103773759a所描述的)、mon15985转基因棉花事件和/或包含mon15985转基因棉花事件的植物材料(如在cn101413028b所描述的)、mon531转基因棉花事件和/或包含mon531转基因棉花事件的植物材料(如在usdaaphis非管制状态申请00-342-01p所描述的)、cot67b转基因棉花事件和/或包含cot67b转基因棉花事件的植物材料(如在usdaaphis非管制状态申请07-108-01p所描述的)或者3006-210-23转基因棉花事件和/或包含3006-210-23转基因棉花事件的植物材料(如在usdaaphis非管制状态申请03-036-02p所描述的),其均可以实现本发明的方法和/或用途,即通过草地螟害虫至少与cry1a蛋白接触以实现控制草地螟害虫的方法和/或用途。本领域技术人员所理解的,使上述转基因事件中的cry1a蛋白在不同植物中表达亦能实现本发明的方法和/或用途。更具体地,所述cry1a蛋白存在于至少产生所述cry1a蛋白的转基因植物中,所述草地螟害虫通过摄食所述转基因植物的组织至少与所述cry1a蛋白接触,接触后所述草地螟害虫生长受到抑制和/或导致死亡,以实现对草地螟危害植物的控制。
本发明中所述调控序列包括但不限于启动子、转运肽、终止子、增强子、前导序列、内含子以及其它可操作地连接到所述cry1a蛋白的调节序列。
所述启动子为植物中可表达的启动子,所述的“植物中可表达的启动子”是指确保与其连接的编码序列在植物细胞内进行表达的启动子。植物中可表达的启动子可为组成型启动子。指导植物内组成型表达的启动子的示例包括但不限于,来源于花椰菜花叶病毒的35s启动子、拟南芥ubi10启动子、玉米ubi启动子、水稻gos2基因的启动子等。备选地,植物中可表达的启动子可为组织特异的启动子,即该启动子在植物的一些组织内如在绿色组织中指导编码序列的表达水平高于植物的其他组织(可通过常规rna试验进行测定),如pep羧化酶启动子。备选地,植物中可表达的启动子可为创伤诱导启动子。创伤诱导启动子或指导创伤诱导的表达模式的启动子是指当植物经受机械或由昆虫啃食引起的创伤时,启动子调控下的编码序列的表达较正常生长条件下有显著提高。创伤诱导启动子的示例包括但不限于,马铃薯和西红柿的蛋白酶抑制基因(pinⅰ和pinⅱ)和玉米蛋白酶抑制基因(mpi)的启动子。
所述转运肽(又称分泌信号序列或导向序列)是指导转基因产物到特定的细胞器或细胞区室,对受体蛋白质来说,所述转运肽可以是异源的,例如,利用编码叶绿体转运肽序列靶向叶绿体,或者利用‘kdel’保留序列靶向内质网,或者利用大麦植物凝集素基因的ctpp靶向液泡。
所述前导序列包含但不限于,小rna病毒前导序列,如emcv前导序列(脑心肌炎病毒5’非编码区);马铃薯y病毒组前导序列,如mdmv(玉米矮缩花叶病毒)前导序列;人类免疫球蛋白质重链结合蛋白质(bip);苜蓿花叶病毒的外壳蛋白质mrna的不翻译前导序列(amvrna4);烟草花叶病毒(tmv)前导序列。
所述增强子包含但不限于,花椰菜花叶病毒(camv)增强子、玄参花叶病毒(fmv)增强子、康乃馨风化环病毒(cerv)增强子、木薯脉花叶病毒(csvmv)增强子、紫茉莉花叶病毒(mmv)增强子、夜香树黄化曲叶病毒(cmylcv)增强子、木尔坦棉花曲叶病毒(clcumv)、鸭跖草黄斑驳病毒(coymv)和花生褪绿线条花叶病毒(pclsv)增强子。
对于单子叶植物应用而言,所述内含子包含但不限于,玉米hsp70内含子、玉米泛素内含子、adh内含子1、蔗糖合酶内含子或水稻act1内含子。对于双子叶植物应用而言,所述内含子包含但不限于,cat-1内含子、pkannibal内含子、piv2内含子和“超级泛素”内含子。
所述终止子可以为在植物中起作用的适合多聚腺苷酸化信号序列,包括但不限于,来源于农杆菌(agrobacteriumtumefaciens)胭脂碱合成酶(nos)基因的多聚腺苷酸化信号序列、来源于蛋白酶抑制剂ⅱ(pinⅱ)基因的多聚腺苷酸化信号序列、来源于豌豆ssrubiscoe9基因的多聚腺苷酸化信号序列和来源于α-微管蛋白(α-tubulin)基因的多聚腺苷酸化信号序列。
本发明中所述“有效连接”表示核酸序列的联结,所述联结使得一条序列可提供对相连序列来说需要的功能。在本发明中所述“有效连接”可以为将启动子与感兴趣的序列相连,使得该感兴趣的序列的转录受到该启动子控制和调控。当感兴趣的序列编码蛋白并且想要获得该蛋白的表达时“有效连接”表示:启动子与所述序列相连,相连的方式使得得到的转录物高效翻译。如果启动子与编码序列的连接是转录物融合并且想要实现编码的蛋白的表达时,制造这样的连接,使得得到的转录物中第一翻译起始密码子是编码序列的起始密码子。备选地,如果启动子与编码序列的连接是翻译融合并且想要实现编码的蛋白的表达时,制造这样的连接,使得5’非翻译序列中含有的第一翻译起始密码子与启动子相连结,并且连接方式使得得到的翻译产物与编码想要的蛋白的翻译开放读码框的关系是符合读码框的。可以“有效连接”的核酸序列包括但不限于:提供基因表达功能的序列(即基因表达元件,例如启动子、5’非翻译区域、内含子、蛋白编码区域、3’非翻译区域、聚腺苷化位点和/或转录终止子)、提供dna转移和/或整合功能的序列(即t-dna边界序列、位点特异性重组酶识别位点、整合酶识别位点)、提供选择性功能的序列(即抗生素抗性标记物、生物合成基因)、提供可计分标记物功能的序列、体外或体内协助序列操作的序列(即多接头序列、位点特异性重组序列)和提供复制功能的序列(即细菌的复制起点、自主复制序列、着丝粒序列)。
本发明中所述的“杀虫”或“抗虫”是指对农作物害虫是有毒的,从而实现“控制”和/或“防治”农作物害虫。优选地,所述“杀虫”或“抗虫”是指杀死农作物害虫。更具体地,目标昆虫是草地螟害虫。
本发明中cry1a蛋白对草地螟害虫具有毒性。本发明中的植物,特别是大豆,在其基因组中含有外源dna,所述外源dna包含编码cry1a蛋白的核苷酸序列,草地螟害虫通过摄食植物组织与该蛋白接触,接触后草地螟害虫生长受到抑制和/或导致死亡。抑制是指致死或亚致死。同时,植物在形态上应是正常的,且可在常规方法下培养以用于产物的消耗和/或生成。此外,该植物可基本消除对化学或生物杀虫剂的需要(所述化学或生物杀虫剂为针对cry1a蛋白所靶向的草地螟害虫的杀虫剂)。
植物材料中杀虫晶体蛋白(icp)的表达水平可通过本领域内所描述的多种方法进行检测,例如通过应用特异引物对组织内产生的编码杀虫蛋白质的mrna进行定量,或直接特异性检测产生的杀虫蛋白质的量。
可以应用不同的试验测定植物中icp的杀虫效果。本发明中目标昆虫主要为草地螟。
本发明中,所述cry1a蛋白可以具有seqidno:1、seqidno:3、seqidno:5、seqidno:7或seqidno:9所示的氨基酸序列。除了包含cry1a蛋白的编码区外,也可包含其他元件,例如编码选择性标记的蛋白质。
此外,包含编码本发明cry1a蛋白的核苷酸序列的表达盒在植物中还可以与至少一种编码除草剂抗性基因的蛋白质一起表达,所述除草剂抗性基因包括但不限于,草铵膦抗性基因(如bar基因、pat基因)、苯敌草抗性基因(如pmph基因)、草甘膦抗性基因(如epsps基因)、溴苯腈(bromoxynil)抗性基因、磺酰脲抗性基因、对除草剂茅草枯的抗性基因、对氨腈的抗性基因或谷氨酰胺合成酶抑制剂(如ppt)的抗性基因,从而获得既具有高杀虫活性、又具有除草剂抗性的转基因植物。
本发明中,将外源dna导入植物,如将编码所述cry1a蛋白的基因或表达盒或重组载体导入植物细胞,常规的转化方法包括但不限于,农杆菌介导的转化、微量发射轰击、直接将dna摄入原生质体、电穿孔或晶须硅介导的dna导入。
本发明提供了一种杀虫蛋白的用途,具有以下优点:
1、内因防治。现有技术主要是通过外部作用即外因来控制草地螟害虫的危害,如农业防治、化学防治、物理防治和生物防治;而本发明是通过植物体内产生能够杀死草地螟的cry1a蛋白来控制草地螟害虫的,即通过内因来防治。
2、无污染、无残留。现有技术使用的化学防治方法虽然对控制草地螟害虫的危害起到了一定作用,但同时也对人、畜和农田生态系统带来了污染、破坏和残留;使用本发明控制草地螟害虫的方法,可以消除上述不良后果。
3、全生育期防治。现有技术使用的控制草地螟害虫的方法都是阶段性的,而本发明是对植物进行全生育期的保护,转基因植物(cry1a蛋白)从发芽、生长,一直到开花、结果,都可以避免遭受草地螟的侵害。
4、全植株防治。现有技术使用的控制草地螟害虫的方法大多是局部性的,如叶面喷施;而本发明是对整个植株进行保护,如转基因植物(cry1a蛋白)的根、叶片、茎秆、果实、雄穗、雌穗、花药或花丝等都是可以抵抗草地螟侵害的。
5、效果稳定。现有技术使用的无论是农业防治方法还是物理防治方法都需要利用环境条件对害虫进行防治,可变因素较多;本发明是使所述cry1a蛋白在植物体内进行表达,有效地克服了环境条件不稳定的缺陷,且本发明转基因植物(cry1a蛋白)的防治效果在不同地点、不同时间、不同遗传背景也都是稳定一致的。
6、简单、方便、经济。现有技术使用的频振式杀虫灯的一次性投入较大,且操作不当还有电击伤人的危险;本发明只需种植能够表达cry1a蛋白的转基因植物即可,而不需要采用其它措施,从而节省了大量人力、物力和财力。
7、效果彻底。现有技术使用的控制草地螟害虫的方法,其效果是不彻底的,只起到减轻作用;而本发明转基因植物(cry1a蛋白)对草地螟初孵幼虫的防治效果几乎为百分之百,极个别存活幼虫也基本上停止发育,3天后幼虫基本仍处于初孵状态,都是明显的发育不良,且已停止发育,在田间自然环境中无法存活,而转基因植物大体上只受到轻微损伤。
下面通过附图和实施例,对本发明的技术方案做进一步的详细描述。
附图说明
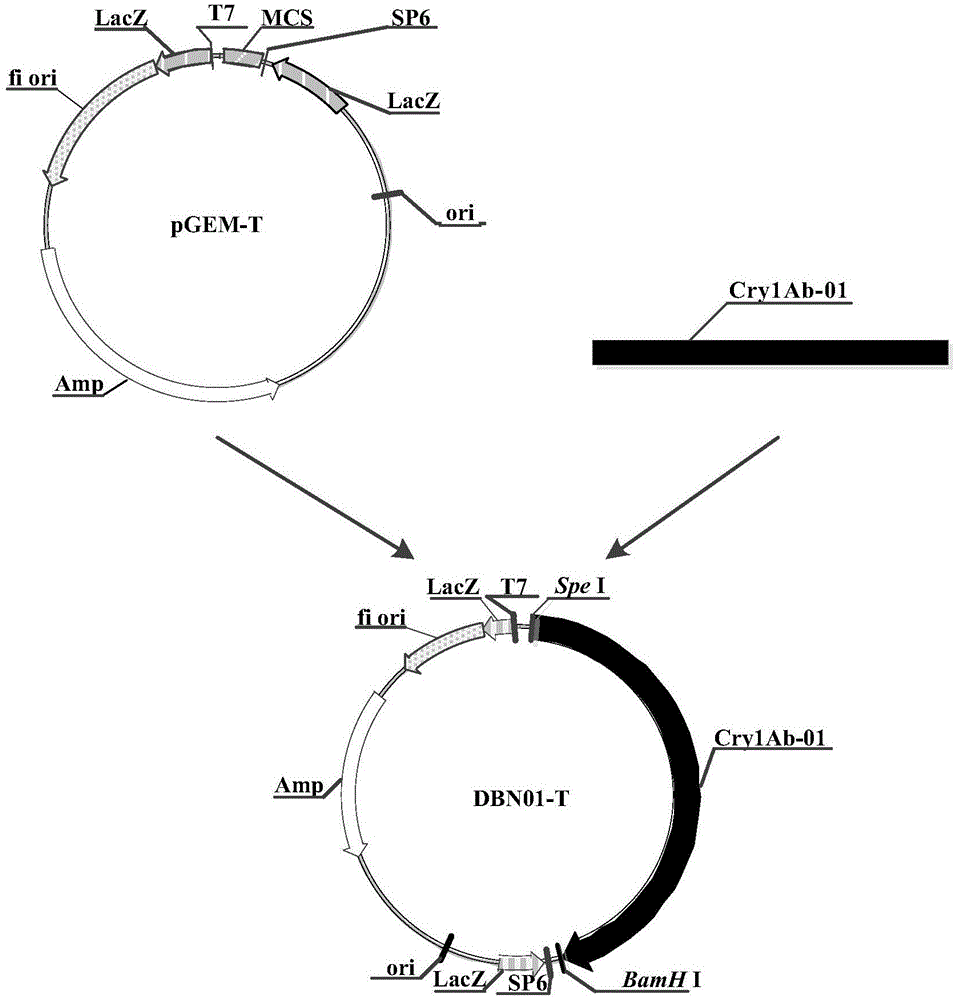
图1为本发明杀虫蛋白的用途的含有cry1a核苷酸序列的重组克隆载体dbn01-t构建流程图;
图2为本发明杀虫蛋白的用途的含有cry1a核苷酸序列的大豆重组表达载体dbn100125构建流程图;
图3为本发明杀虫蛋白的用途的含有cry1a核苷酸序列的玉米重组表达载体dbn100738构建流程图。
具体实施方式
下面通过具体实施例进一步说明本发明杀虫蛋白的用途的技术方案。
第一实施例、基因的获得和合成
1、获得核苷酸序列
cry1ab-01杀虫蛋白质的氨基酸序列(818个氨基酸),如序列表中seqidno:1所示;编码相应于所述cry1ab-01杀虫蛋白质的氨基酸序列的cry1ab-01核苷酸序列(2457个核苷酸),如序列表中seqidno:2所示。cry1ab-02杀虫蛋白质的氨基酸序列(615个氨基酸),如序列表中seqidno:3所示;编码相应于所述cry1ab-02杀虫蛋白质的氨基酸序列的cry1ab-02核苷酸序列(1848个核苷酸),如序列表中seqidno:4所示。
cry1ac-01杀虫蛋白质的氨基酸序列(1178个氨基酸),如序列表中seqidno:5所示;编码相应于所述cry1ac-01杀虫蛋白质的氨基酸序列的cry1ac-01核苷酸序列(3537个核苷酸),如序列表中seqidno:6所示。cry1ac-02杀虫蛋白质的氨基酸序列(1156个氨基酸),如序列表中seqidno:7所示;编码相应于所述cry1ac-02杀虫蛋白质的氨基酸序列的cry1ac-02核苷酸序列(3471个核苷酸),如序列表中seqidno:8所示。
cry1a.105杀虫蛋白质的氨基酸序列(1177个氨基酸),如序列表中seqidno:9所示;编码相应于所述cry1a.105杀虫蛋白质的氨基酸序列的cry1a.105核苷酸序列(3534个核苷酸),如序列表中seqidno:10所示。
cry2ab杀虫蛋白质的氨基酸序列(634个氨基酸),如序列表中seqidno:11所示;编码相应于所述cry2ab杀虫蛋白质的氨基酸序列的cry2ab核苷酸序列(1905个核苷酸),如序列表中seqidno:12所示。
cry1fa杀虫蛋白质的氨基酸序列(1148个氨基酸),如序列表中seqidno:13所示;编码相应于所述cry1fa杀虫蛋白质的氨基酸序列的cry1fa核苷酸序列(3447个核苷酸),如序列表中seqidno:14所示。
vip3a杀虫蛋白质的氨基酸序列(789个氨基酸),如序列表中seqidno:15所示;编码相应于所述vip3a杀虫蛋白质的氨基酸序列的vip3a核苷酸序列(2370个核苷酸),如序列表中seqidno:16所示。
2、合成上述核苷酸序列
合成所述cry1ab-01核苷酸序列(如序列表中seqidno:2所示)、所述cry1ab-02核苷酸序列(如序列表中seqidno:4所示)、所述cry1ac-01核苷酸序列(如序列表中seqidno:6所示)、所述cry1ac-02核苷酸序列(如序列表中seqidno:8所示)、所述cry1a.105核苷酸序列(如序列表中seqidno:10所示)、所述cry2ab核苷酸序列(如序列表中seqidno:12所示)、所述cry1fa核苷酸序列(如序列表中seqidno:14所示)和所述vip3a核苷酸序列(如序列表中seqidno:16所示)。合成的所述cry1ab-01核苷酸序列(seqidno:2)的5’端还连接有spei酶切位点,所述cry1ab-01核苷酸序列(seqidno:2)的3’端还连接有bamhi酶切位点;合成的所述cry1ab-02核苷酸序列(seqidno:4)的5’端还连接有spei酶切位点,所述cry1ab-02核苷酸序列(seqidno:4)的3’端还连接有bamhi酶切位点;合成的所述cry1ac-01核苷酸序列(seqidno:6)的5’端还连接有saci酶切位点,所述cry1ac-01核苷酸序列(seqidno:6)的3’端还连接有kasi酶切位点;合成的所述cry1ac-02核苷酸序列(seqidno:8)的5’端还连接有saci酶切位点,所述cry1ac-02核苷酸序列(seqidno:8)的3’端还连接有kasi酶切位点;合成的所述cry1a.105核苷酸序列(seqidno:10)的5’端还连接有ncoi酶切位点,所述cry1a.105核苷酸序列(seqidno:10)的3’端还连接有hindiii酶切位点;合成的所述cry2ab核苷酸序列(seqidno:12)的5’端还连接有ncoi酶切位点,所述cry2ab核苷酸序列(seqidno:12)的3’端还连接有spei酶切位点;合成的所述cry1fa核苷酸序列(seqidno:14)的5’端还连接有asci酶切位点,所述cry1fa核苷酸序列(seqidno:14)的3’端还连接有bamhi酶切位点;合成的所述vip3a核苷酸序列(seqidno:16)的5’端还连接有asci酶切位点,所述vip3a核苷酸序列(seqidno:16)的3’端还连接有bamhi酶切位点。
第二实施例、重组表达载体的构建及重组表达载体转化农杆菌
1、构建含有cry1a基因的重组克隆载体
将合成的cry1ab-01核苷酸序列连入克隆载体pgem-t(promega,madison,usa,cat:a3600)上,操作步骤按promega公司产品pgem-t载体说明书进行,得到重组克隆载体dbn01-t,其构建流程如图1所示(其中,amp表示氨苄青霉素抗性基因;f1表示噬菌体f1的复制起点;lacz为lacz起始密码子;sp6为sp6rna聚合酶启动子;t7为t7rna聚合酶启动子;cry1ab-01为cry1ab-01核苷酸序列(seqidno:2);mcs为多克隆位点)。
然后将重组克隆载体dbn01-t用热激方法转化大肠杆菌t1感受态细胞(transgen,beijing,china,cat:cd501),其热激条件为:50μl大肠杆菌t1感受态细胞、10μl质粒dna(重组克隆载体dbn01-t),42℃水浴30s;37℃振荡培养1h(100rpm转速下摇床摇动),在表面涂有iptg(异丙基硫代-β-d-半乳糖苷)和x-gal(5-溴-4-氯-3-吲哚-β-d-半乳糖苷)的氨苄青霉素(100mg/l)的lb平板(胰蛋白胨10g/l、酵母提取物5g/l、nacl10g/l、琼脂15g/l,用naoh调ph至7.5)上生长过夜。挑取白色菌落,在lb液体培养基(胰蛋白胨10g/l、酵母提取物5g/l、nacl10g/l、氨苄青霉素100mg/l,用naoh调ph至7.5)中于温度37℃条件下培养过夜。碱法提取其质粒:将菌液在12000rpm转速下离心1min,去上清液,沉淀菌体用100μl冰预冷的溶液i(25mmtris-hcl、10mmedta(乙二胺四乙酸)、50mm葡萄糖,ph8.0)悬浮;加入200μl新配制的溶液ii(0.2mnaoh、1%sds(十二烷基硫酸钠)),将管子颠倒4次,混合,置冰上3-5min;加入150μl冰冷的溶液iii(3m醋酸钾、5m醋酸),立即充分混匀,冰上放置5-10min;于温度4℃、转速12000rpm条件下离心5min,在上清液中加入2倍体积无水乙醇,混匀后室温放置5min;于温度4℃、转速12000rpm条件下离心5min,弃上清液,沉淀用浓度(v/v)为70%的乙醇洗涤后晾干;加入30μl含rnase(20μg/ml)的te(10mmtris-hcl、1mmedta,ph8.0)溶解沉淀;于温度37℃下水浴30min,消化rna;于温度-20℃保存备用。
提取的质粒经spei和bamhi酶切鉴定后,对阳性克隆进行测序验证,结果表明重组克隆载体dbn01-t中插入的所述cry1ab-01核苷酸序列为序列表中seqidno:2所示的核苷酸序列,即cry1ab-01核苷酸序列正确插入。
按照上述构建重组克隆载体dbn01-t的方法,将合成的所述cry1ab-02核苷酸序列连入克隆载体pgem-t上,得到重组克隆载体dbn02-t,其中,cry1ab-02为cry1ab-02核苷酸序列(seqidno:4)。酶切和测序验证重组克隆载体dbn02-t中所述cry1ab-02核苷酸序列正确插入。
按照上述构建重组克隆载体dbn01-t的方法,将合成的所述cry1ac-01核苷酸序列连入克隆载体pgem-t上,得到重组克隆载体dbn03-t,其中,cry1ac-01为cry1ac-01核苷酸序列(seqidno:6)。酶切和测序验证重组克隆载体dbn03-t中所述cry1ac-01核苷酸序列正确插入。
按照上述构建重组克隆载体dbn01-t的方法,将合成的所述cry1ac-02核苷酸序列连入克隆载体pgem-t上,得到重组克隆载体dbn04-t,其中,cry1ac-02为cry1ac-02核苷酸序列(seqidno:8)。酶切和测序验证重组克隆载体dbn04-t中所述cry1ac-02核苷酸序列正确插入。
按照上述构建重组克隆载体dbn01-t的方法,将合成的所述cry1a.105核苷酸序列连入克隆载体pgem-t上,得到重组克隆载体dbn05-t,其中,cry1a.105为cry1a.105核苷酸序列(seqidno:10)。酶切和测序验证重组克隆载体dbn05-t中所述cry1a.105核苷酸序列正确插入。
按照上述构建重组克隆载体dbn01-t的方法,将合成的所述cry2ab核苷酸序列连入克隆载体pgem-t上,得到重组克隆载体dbn06-t,其中,cry2ab为cry2ab核苷酸序列(seqidno:12)。酶切和测序验证重组克隆载体dbn06-t中所述cry2ab核苷酸序列正确插入。
按照上述构建重组克隆载体dbn01-t的方法,将合成的所述cry1fa核苷酸序列连入克隆载体pgem-t上,得到重组克隆载体dbn07-t,其中,cry1fa为cry1fa核苷酸序列(seqidno:14)。酶切和测序验证重组克隆载体dbn07-t中所述cry1fa核苷酸序列正确插入。
按照上述构建重组克隆载体dbn01-t的方法,将合成的所述vip3a核苷酸序列连入克隆载体pgem-t上,得到重组克隆载体dbn08-t,其中,vip3a为vip3a核苷酸序列(seqidno:16)。酶切和测序验证重组克隆载体dbn08-t中所述vip3a核苷酸序列正确插入。
2、构建含有cry1a基因的大豆重组表达载体
用限制性内切酶spei和bamhi分别酶切重组克隆载体dbn01-t和表达载体dbnbc-01(载体骨架:pcambia2301(cambia机构可以提供)),将切下的cry1ab-01核苷酸序列片段插到表达载体dbnbc-01的spei和bamhi位点之间,利用常规的酶切方法构建载体是本领域技术人员所熟知的,构建成重组表达载体dbn100125,其构建流程如图2所示(kan:卡那霉素基因;rb:右边界;pratubi10:拟南芥泛素(ubiquitin)基因启动子(seqidno:17);cry1ab-01:cry1ab-01核苷酸序列(seqidno:2);tnos:胭脂碱合成酶基因的终止子(seqidno:18);pr35s:花椰菜花叶病毒35s启动子(seqidno:19);pat:草丁膦乙酰转移酶基因(seqidno:20);t35s:花椰菜花叶病毒35s终止子(seqidno:21);lb:左边界)。
将重组表达载体dbn100125用热激方法转化大肠杆菌t1感受态细胞,其热激条件为:50μl大肠杆菌t1感受态细胞、10μl质粒dna(重组表达载体dbn100125),42℃水浴30s;37℃振荡培养1h(100rpm转速下摇床摇动);然后在含50mg/l卡那霉素(kanamycin)的lb固体平板(胰蛋白胨10g/l、酵母提取物5g/l、nacl10g/l、琼脂15g/l,用naoh调ph至7.5)上于温度37℃条件下培养12h,挑取白色菌落,在lb液体培养基(胰蛋白胨10g/l、酵母提取物5g/l、nacl10g/l、卡那霉素50mg/l,用naoh调ph至7.5)中于温度37℃条件下培养过夜。碱法提取其质粒。将提取的质粒用限制性内切酶spei和bamhi酶切后鉴定,并将阳性克隆进行测序鉴定,结果表明重组表达载体dbn100125在spei和bamhi位点间的核苷酸序列为序列表中seqidno:2所示核苷酸序列,即cry1ab-01核苷酸序列。
按照上述构建重组表达载体dbn100125的方法,将spei和bamhi酶切重组克隆载体dbn02-t切下的所述cry1ab-02核苷酸序列插入表达载体dbnbc-01,得到重组表达载体dbn100744。酶切和测序验证重组表达载体dbn100744中的核苷酸序列含有为序列表中seqidno:4所示核苷酸序列,即cry1ab-02核苷酸序列,所述cry1ab-02核苷酸序列可以连接所述pratubi10启动子和tnos终止子。
按照上述构建重组表达载体dbn100125的方法,将saci和kasi酶切重组克隆载体dbn03-t切下的所述cry1ac-01核苷酸序列插入表达载体dbnbc-01,得到重组表达载体dbn100645(kan:卡那霉素基因;rb:右边界;pratrbcs4:拟南芥核酮糖1,5-二磷酸羧化酶小亚基基因启动子(seqidno:22);cry1ac-01:cry1ac-01核苷酸序列(seqidno:6);tnos:胭脂碱合成酶基因的终止子(seqidno:18);pr35s:花椰菜花叶病毒35s启动子(seqidno:19);pat:草丁膦乙酰转移酶基因(seqidno:20);t35s:花椰菜花叶病毒35s终止子(seqidno:21);lb:左边界)。酶切和测序验证重组表达载体dbn100645中的核苷酸序列含有为序列表中seqidno:6所示核苷酸序列,即cry1ac-01核苷酸序列。
按照上述构建重组表达载体dbn100125的方法,将saci和kasi、ncoi和spei分别酶切重组克隆载体dbn03-t和dbn06-t切下的所述cry1ac-01核苷酸序列和cry2ab核苷酸序列插入表达载体dbnbc-01,得到重组表达载体dbn100175(kan:卡那霉素基因;rb:右边界;pratubi10:拟南芥泛素(ubiquitin)基因启动子(seqidno:17);cry2ab:cry2ab核苷酸序列(seqidno:12);tnos:胭脂碱合成酶基因的终止子(seqidno:18);pratrbcs4:拟南芥核酮糖1,5-二磷酸羧化酶小亚基基因启动子(seqidno:22);cry1ac-01:cry1ac-01核苷酸序列(seqidno:6);tnos:胭脂碱合成酶基因的终止子(seqidno:18);pr35s:花椰菜花叶病毒35s启动子(seqidno:19);pat:草丁膦乙酰转移酶基因(seqidno:20);t35s:花椰菜花叶病毒35s终止子(seqidno:21);lb:左边界)。酶切和测序验证重组表达载体dbn100175中的核苷酸序列含有为序列表中seqidno:6和seqidno:12所示核苷酸序列,即cry1ac-01核苷酸序列和cry2ab核苷酸序列。
按照上述构建重组表达载体dbn100125的方法,将saci和kasi、asci和bamhi分别酶切重组克隆载体dbn04-t和dbn07-t切下的所述cry1ac-02核苷酸序列和cry1fa核苷酸序列插入表达载体dbnbc-01,得到重组表达载体dbn100656(kan:卡那霉素基因;rb:右边界;pratubi10:拟南芥泛素(ubiquitin)基因启动子(seqidno:17);cry1fa:cry1fa核苷酸序列(seqidno:14);tnos:胭脂碱合成酶基因的终止子(seqidno:18);prcsvmv:木薯叶脉花叶病毒启动子(seqidno:23);cry1ac-02:cry1ac-02核苷酸序列(seqidno:8);tnos:胭脂碱合成酶基因的终止子(seqidno:18);pr35s:花椰菜花叶病毒35s启动子(seqidno:19);pat:草丁膦乙酰转移酶基因(seqidno:20);t35s:花椰菜花叶病毒35s终止子(seqidno:21);lb:左边界)。酶切和测序验证重组表达载体dbn100656中的核苷酸序列含有为序列表中seqidno:8和seqidno:14所示核苷酸序列,即cry1ac-02核苷酸序列和cry1fa核苷酸序列。
按照上述构建重组表达载体dbn100125的方法,将ncoi和hindiii酶切重组克隆载体dbn05-t切下的所述cry1a.105核苷酸序列插入表达载体dbnbc-01,得到重组表达载体dbn100757。酶切和测序验证重组表达载体dbn100757中的核苷酸序列含有为序列表中seqidno:10所示核苷酸序列,即cry1a.105核苷酸序列,所述cry1a.105核苷酸序列可以连接所述pratubi10启动子和tnos终止子。
按照上述构建重组表达载体dbn100125的方法,将ncoi和hindiii、ncoi和spei分别酶切重组克隆载体dbn05-t和dbn06-t切下的所述cry1a.105核苷酸序列和cry2ab核苷酸序列插入表达载体dbnbc-01,得到重组表达载体dbn100176(kan:卡那霉素基因;rb:右边界;pratubi10:拟南芥泛素(ubiquitin)基因启动子(seqidno:17);cry2ab:cry2ab核苷酸序列(seqidno:12);tnos:胭脂碱合成酶基因的终止子(seqidno:18);pratrbcs4:拟南芥核酮糖1,5-二磷酸羧化酶小亚基基因启动子(seqidno:22);cry1a.105:cry1a.105核苷酸序列(seqidno:10);tnos:胭脂碱合成酶基因的终止子(seqidno:18);pr35s:花椰菜花叶病毒35s启动子(seqidno:19);pat:草丁膦乙酰转移酶基因(seqidno:20);t35s:花椰菜花叶病毒35s终止子(seqidno:21);lb:左边界)。酶切和测序验证重组表达载体dbn100176中的核苷酸序列含有为序列表中seqidno:10和seqidno:12所示核苷酸序列,即cry1a.105核苷酸序列和cry2ab核苷酸序列。
按照上述构建重组表达载体dbn100125的方法,将asci和bamhi酶切重组克隆载体dbn08-t切下的所述vip3a核苷酸序列插入表达载体dbnbc-01,得到重组表达载体dbn100003。酶切和测序验证重组表达载体dbn100003中的核苷酸序列含有为序列表中seqidno:16所示核苷酸序列,即vip3a核苷酸序列。所述vip3a核苷酸序列可以分别连接所述pratubi10启动子和tnos终止子。
3、构建含有cry1a基因的玉米重组表达载体
用限制性内切酶spei和bamhi分别酶切重组克隆载体dbn01-t和表达载体dbnbc-02(载体骨架:pcambia2301(cambia机构可以提供)),将切下的cry1ab-01核苷酸序列片段插到表达载体dbnbc-02的spei和bamhi位点之间,利用常规的酶切方法构建载体是本领域技术人员所熟知的,构建成重组表达载体dbn100738,其构建流程如图3所示(kan:卡那霉素基因;rb:右边界;prubi:玉米泛素(ubiquitin)基因启动子(seqidno:24);cry1ab-01:cry1ab-01核苷酸序列(seqidno:2);tnos:胭脂碱合成酶基因的终止子(seqidno:18);pmi:磷酸甘露糖异构酶基因(seqidno:25)lb:左边界)。
将重组表达载体dbn100738用热激方法转化大肠杆菌t1感受态细胞,其热激条件为:50μl大肠杆菌t1感受态细胞、10μl质粒dna(重组表达载体dbn100125),42℃水浴30s;37℃振荡培养1h(100rpm转速下摇床摇动);然后在含50mg/l卡那霉素(kanamycin)的lb固体平板(胰蛋白胨10g/l、酵母提取物5g/l、nacl10g/l、琼脂15g/l,用naoh调ph至7.5)上于温度37℃条件下培养12h,挑取白色菌落,在lb液体培养基(胰蛋白胨10g/l、酵母提取物5g/l、nacl10g/l、卡那霉素50mg/l,用naoh调ph至7.5)中于温度37℃条件下培养过夜。碱法提取其质粒。将提取的质粒用限制性内切酶spei和bamhi酶切后鉴定,并将阳性克隆进行测序鉴定,结果表明重组表达载体dbn100738在spei和bamhi位点间的核苷酸序列为序列表中seqidno:2所示核苷酸序列,即cry1ab-01核苷酸序列。
按照上述构建重组表达载体dbn100738的方法,将spei和bamhi、asci和bamhi分别酶切重组克隆载体dbn01-t和dbn08-t切下的所述cry1ab-01核苷酸序列和vip3a核苷酸序列插入表达载体dbnbc-02,得到重组表达载体dbn100053。酶切和测序验证重组表达载体dbn100053中的核苷酸序列含有为序列表中seqidno:2和seqidno:16所示核苷酸序列,即cry1ab-01核苷酸序列和vip3a核苷酸序列。所述cry1ab-01核苷酸序列和所述vip3a核苷酸序列可以分别连接所述ubi启动子和nos终止子。
按照上述构建重组表达载体dbn100738的方法,将spei和bamhi酶切重组克隆载体dbn02-t切下的所述cry1ab-02核苷酸序列插入表达载体dbnbc-02,得到重组表达载体dbn100106。酶切和测序验证重组表达载体dbn100106中的核苷酸序列含有为序列表中seqidno:4所示核苷酸序列,即cry1ab-02核苷酸序列,所述cry1ab-02核苷酸序列可以连接所述ubi启动子和nos终止子。
按照上述构建重组表达载体dbn100738的方法,将saci和kasi酶切重组克隆载体dbn03-t切下的所述cry1ac-01核苷酸序列插入表达载体dbnbc-02,得到重组表达载体dbn100734。酶切和测序验证重组表达载体dbn100734中的核苷酸序列含有为序列表中seqidno:6所示核苷酸序列,即cry1ac-01核苷酸序列。所述cry1ac-01核苷酸序列可以连接所述ubi启动子和nos终止子。
按照上述构建重组表达载体dbn100738的方法,将ncoi和hindiii酶切重组克隆载体dbn05-t切下的所述cry1a.105核苷酸序列插入表达载体dbnbc-02,得到重组表达载体dbn100029。酶切和测序验证重组表达载体dbn100029中的核苷酸序列含有为序列表中seqidno:10所示核苷酸序列,即cry1a.105核苷酸序列,所述cry1a.105核苷酸序列可以连接所述ubi启动子和nos终止子。
按照上述构建重组表达载体dbn100738的方法,将ncoi和hindiii、ncoi和spei分别酶切重组克隆载体dbn05-t和dbn06-t切下的所述cry1a.105核苷酸序列和cry2ab核苷酸序列插入表达载体dbnbc-02,得到重组表达载体dbn100076。酶切和测序验证重组表达载体dbn100076中的核苷酸序列含有为序列表中seqidno:10和seqidno:12所示核苷酸序列,即cry1a.105核苷酸序列和cry2ab核苷酸序列。所述cry1a.105核苷酸序列和所述cry2ab核苷酸序列可以分别连接所述ubi启动子和nos终止子。
按照上述构建重组表达载体dbn100738的方法,将asci和bamhi酶切重组克隆载体dbn08-t切下的所述vip3a核苷酸序列插入表达载体dbnbc-02,得到重组表达载体dbn100004。酶切和测序验证重组表达载体dbn100004中的核苷酸序列含有为序列表中seqidno:16所示核苷酸序列,即vip3a核苷酸序列。所述vip3a核苷酸序列可以分别连接所述ubi启动子和nos终止子。
4、重组表达载体转化农杆菌
对己经构建正确的大豆重组表达载体dbn100125、dbn100744、dbn100645、dbn100175、dbn100656、dbn100757、dbn100176和dbn100003,和玉米重组表达载体dbn100738、dbn100053、dbn100106、dbn100734、dbn100029、dbn100076和dbn100004用液氮法转化到农杆菌lba4404(invitrgen,chicago,usa,cat:18313-015)中,其转化条件为:100μl农杆菌lba4404、3μl质粒dna(重组表达载体);置于液氮中10min,37℃温水浴10min;将转化后的农杆菌lba4404接种于lb试管中于温度28℃、转速为200rpm条件下培养2h,涂于含50mg/l的利福平(rifampicin)和100mg/l的卡那霉素的lb平板上直至长出阳性单克隆,挑取单克隆培养并提取其质粒,用限制性内切酶对大豆重组表达载体dbn100125、dbn100744、dbn100645、dbn100175、dbn100656、dbn100757、dbn100176和dbn100003,和玉米重组表达载体dbn100738、dbn100053、dbn100106、dbn100734、dbn100029、dbn100076和dbn100004酶切后进行酶切验证,结果表明大豆重组表达载体dbn100125、dbn100744、dbn100645、dbn100175、dbn100656、dbn100757、dbn100176和dbn100003,和玉米重组表达载体dbn100738、dbn100053、dbn100106、dbn100734、dbn100029、dbn100076和dbn100004结构完全正确。
第三实施例、转基因植株的获得
1、获得转基因大豆植株
按照常规采用的农杆菌侵染法,将无菌培养的大豆品种中黄13的子叶节组织与第二实施例中4所述的农杆菌共培养,以将第二实施例中2构建的大豆重组表达载体dbn100125、dbn100744、dbn100645、dbn100175、dbn100656、dbn100757、dbn100176和dbn100003的t-dna(包括cry1ab-01核苷酸序列、cry1ab-02核苷酸序列、cry1ac-01核苷酸序列、cry1ac-02核苷酸序列、cry1a.105核苷酸序列、cry2ab核苷酸序列、cry1fa核苷酸序列、vip3a核苷酸序列和pat基因)转入到大豆染色体组中,获得了转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry1fa核苷酸序列的大豆植株、转入cry1ac-02-cry2ab核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株、转入cry1a.105-cry2ab核苷酸序列的大豆植株和转入vip3a核苷酸序列的大豆植株,同时以野生型大豆植株作为对照。
对于农杆菌介导的大豆转化,简要地,将成熟的大豆种子在大豆萌发培养基(b5盐3.1g/l、b5维他命、蔗糖20g/l、琼脂8g/l,ph5.6)中进行萌发,将种子接种于萌发培养基上,按以下条件培养:温度25±1℃;光周期(光/暗)为16/8h。萌发4-6天后取鲜绿的子叶节处膨大的大豆无菌苗,在子叶节下3-4mm处切去下胚轴,纵向切开子叶,去顶芽、侧芽和种子根。用解剖刀的刀背在子叶节处进行创伤,用农杆菌悬浮液接触创伤过的子叶节组织,其中农杆菌能够将cry1a核苷酸序列传递至创伤过的子叶节组织(步骤1:侵染步骤)在此步骤中,子叶节组织优选地浸入农杆菌悬浮液(od660=0.5-0.8,侵染培养基(ms盐2.15g/l、b5维他命、蔗糖20g/l、葡萄糖10g/l、乙酰丁香酮(as)40mg/l、2-吗啉乙磺酸(mes)4g/l、玉米素(zt)2mg/l,ph5.3)中以启动接种。子叶节组织与农杆菌共培养一段时期(3天)(步骤2:共培养步骤)。优选地,子叶节组织在侵染步骤后在固体培养基(ms盐4.3g/l、b5维他命、蔗糖20g/l、葡萄糖10g/l、mes4g/l、zt2mg/l、琼脂8g/l,ph5.6)上培养。在此共培养阶段后,可以有一个选择性的“恢复”步骤。在“恢复”步骤中,恢复培养基(b5盐3.1g/l、b5维他命、mes1g/l、蔗糖30g/l、zt2mg/l、琼脂8g/l、头孢霉素150mg/l、谷氨酸100mg/l、天冬氨酸100mg/l,ph5.6)中至少存在一种己知抑制农杆菌生长的抗生素(头孢霉素),不添加植物转化体的选择剂(步骤3:恢复步骤)。优选地,子叶节再生的组织块在有抗生素但没有选择剂的固体培养基上培养,以消除农杆菌并为侵染细胞提供恢复期。接着,子叶节再生的组织块在含选择剂(草丁膦)的培养基上培养并选择生长着的转化愈伤组织(步骤4:选择步骤)。优选地,子叶节再生的组织块在有选择剂的筛选固体培养基(b5盐3.1g/l、b5维他命、mes1g/l、蔗糖30g/l、6-苄基腺嘌呤(6-bap)1mg/l、琼脂8g/l、头孢霉素150mg/l、谷氨酸100mg/l、天冬氨酸100mg/l、草丁膦6mg/l,ph5.6)上培养,导致转化的细胞选择性生长。然后,转化的细胞再生成植物(步骤5:再生步骤),优选地,在含选择剂的培养基上生长的子叶节再生的组织块在固体培养基(b5分化培养基和b5生根培养基)上培养以再生植物。
筛选得到的抗性组织块转移到所述b5分化培养基(b5盐3.1g/l、b5维他命、mes1g/l、蔗糖30g/l、zt1mg/l、琼脂8g/l、头孢霉素150mg/l、谷氨酸50mg/l、天冬氨酸50mg/l、赤霉素1mg/l、生长素1mg/l、草丁膦6mg/l,ph5.6)上,25℃下培养分化。分化出来的小苗转移到所述b5生根培养基(b5盐3.1g/l、b5维他命、mes1g/l、蔗糖30g/l、琼脂8g/l、头孢霉素150mg/l、吲哚-3-丁酸(iba)1mg/l),在生根培养上,25℃下培养至约10cm高,移至温室培养至结实。在温室中,每天于26℃下培养16h,再于20℃下培养8h。
2、获得转基因玉米植株
按照常规采用的农杆菌侵染法,将无菌培养的玉米品种综31(z31)的幼胚与第二实施例中4所述的农杆菌共培养,以将第二实施例中3构建的玉米重组表达载体dbn100738、dbn100053、dbn100106、dbn100734、dbn100029、dbn100076和dbn100004的t-dna(包括cry1ab-01核苷酸序列、cry1ab-02核苷酸序列、cry1ac-01核苷酸序列、cry1a.105核苷酸序列、cry2ab核苷酸序列、vip3a核苷酸序列和pmi基因)转入到玉米染色体组中,获得了转入cry1ab-01核苷酸序列的玉米植株、转入cry1ab-01-vip3a核苷酸序列的玉米植株、转入cry1ab-02核苷酸序列的玉米植株、转入cry1ac-01核苷酸序列的玉米植株、转入cry1a.105核苷酸序列的玉米植株、转入cry1a.105-cry2ab核苷酸序列的玉米植株和转入vip3a核苷酸序列的玉米植株;同时以野生型玉米植株作为对照。
对于农杆菌介导的玉米转化,简要地,从玉米中分离未成熟的幼胚,用农杆菌悬浮液接触幼胚,其中农杆菌能够将cry1a核苷酸序列传递至幼胚之一的至少一个细胞(步骤1:侵染步骤),在此步骤中,幼胚优选地浸入农杆菌悬浮液(od660=0.4-0.6,侵染培养基(ms盐4.3g/l、ms维他命、干酪素300mg/l、蔗糖68.5g/l、葡萄糖36g/l、as40mg/l、2,4-二氯苯氧乙酸(2,4-d)1mg/l,ph5.3))中以启动接种。幼胚与农杆菌共培养一段时期(3天)(步骤2:共培养步骤)。优选地,幼胚在侵染步骤后在固体培养基(ms盐4.3g/l、ms维他命、干酪素300mg/l、蔗糖20g/l、葡萄糖10g/l、as100mg/l、2,4-d1mg/l、琼脂8g/l,ph5.8)上培养。在此共培养阶段后,可以有一个选择性的“恢复”步骤。在“恢复”步骤中,恢复培养基(ms盐4.3g/l、ms维他命、干酪素300mg/l、蔗糖30g/l、2,4-d1mg/l、植物凝胶3g/l,ph5.8)中至少存在一种己知抑制农杆菌生长的抗生素(头孢霉素),不添加植物转化体的选择剂(步骤3:恢复步骤)。优选地,幼胚在有抗生素但没有选择剂的固体培养基上培养,以消除农杆菌并为侵染细胞提供恢复期。接着,接种的幼胚在含选择剂(甘露糖)的培养基上培养并选择生长着的转化愈伤组织(步骤4:选择步骤)。优选地,幼胚在有选择剂的筛选固体培养基(ms盐4.3g/l、ms维他命、干酪素300mg/l、蔗糖30g/l、甘露糖12.5g/l、2,4-d1mg/l、植物凝胶3g/l,ph5.8)上培养,导致转化的细胞选择性生长。然后,愈伤组织再生成植物(步骤5:再生步骤),优选地,在含选择剂的培养基上生长的愈伤组织在固体培养基(ms分化培养基和ms生根培养基)上培养以再生植物。
筛选得到的抗性愈伤组织转移到所述ms分化培养基(ms盐4.3g/l、ms维他命、干酪素300mg/l、蔗糖30g/l、6-苄基腺嘌呤2mg/l、甘露糖5g/l、植物凝胶3g/l,ph5.8)上,25℃下培养分化。分化出来的小苗转移到所述ms生根培养基(ms盐2.15g/l、ms维他命、干酪素300mg/l、蔗糖30g/l、吲哚-3-乙酸1mg/l、植物凝胶3g/l,ph5.8)上,25℃下培养至约10cm高,移至温室培养至结实。在温室中,每天于28℃下培养16h,再于20℃下培养8h。
第四实施例、用taqman验证转基因植株
分别取转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry1fa核苷酸序列的大豆植株、转入cry1ac-02-cry2ab核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株、转入cry1a.105-cry2ab核苷酸序列的大豆植株和转入vip3a核苷酸序列的大豆植株的叶片各约100mg作为样品,用qiagen的dneasyplantmaxikit提取其基因组dna,通过taqman探针荧光定量pcr方法检测pat基因的拷贝数以确定cry1a基因的拷贝数。同时以野生型大豆植株作为对照,按照上述方法进行检测分析。实验设3次重复,取平均值。
检测pat基因拷贝数的具体方法如下:
步骤11、分别取转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry1fa核苷酸序列的大豆植株、转入cry1ac-02-cry2ab核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株、转入cry1a.105-cry2ab核苷酸序列的大豆植株、转入vip3a核苷酸序列的大豆植株和野生型大豆植株的叶片各100mg,分别在研钵中用液氮研成匀浆,每个样品取3个重复;
步骤12、使用qiagen的dneasyplantminikit提取上述样品的基因组dna,具体方法参考其产品说明书;
步骤13、用nanodrop2000(thermoscientific)测定上述样品的基因组dna浓度;
步骤14、调整上述样品的基因组dna浓度至同一浓度值,所述浓度值的范围为80-100ng/μl;
步骤15、采用taqman探针荧光定量pcr方法鉴定样品的拷贝数,以经过鉴定已知拷贝数的样品作为标准品,以野生型大豆植株的样品作为对照,每个样品3个重复,取其平均值;荧光定量pcr引物和探针序列分别是:
以下引物和探针用来检测pat基因:
引物1:gagggtgttgtggctggtattg如序列表中seqidno:26所示;
引物2:tctcaactgtccaatcgtaagcg如序列表中seqidno:27所示;
探针1:cttacgctgggccctggaaggctag如序列表中seqidno:28所示;
pcr反应体系为:
所述50×引物/探针混合物包含1mm浓度的每种引物各45μl,100μm浓度的探针50μl和860μl1×te缓冲液,并且在4℃,贮藏在琥珀试管中。
pcr反应条件为:
利用sds2.3软件(appliedbiosystems)分析数据。
实验结果表明,cry1ab-01核苷酸序列、cry1ab-02核苷酸序列、cry1ac-01核苷酸序列、cry1ac-01-cry2ab核苷酸序列、cry1ac-02-cry1fa核苷酸序列、cry1a.105核苷酸序列、cry1a.105-cry2ab核苷酸序列和vip3a核苷酸序列均己整合到所检测的大豆植株的染色体组中,而且转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry2ab核苷酸序列的大豆植株、转入cry1ac-02-cry1fa核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株、转入cry1a.105-cry2ab核苷酸序列的大豆植株和转入vip3a核苷酸序列的大豆植株均获得了单拷贝的转基因大豆植株。
按照上述用taqman验证转基因大豆植株的方法,对转基因玉米植株进行检测分析(pmi基因)。实验结果表明,cry1ab-01核苷酸序列、cry1ab-01-vip3a核苷酸序列、cry1ab-02核苷酸序列、cry1ac-01核苷酸序列、cry1a.105核苷酸序列、cry1a.105-cry2ab核苷酸序列和vip3a核苷酸序列均己分别整合到所检测的玉米植株的染色体组中,而且转入cry1ab-01核苷酸序列的玉米植株、转入cry1ab-01-vip3a核苷酸序列的玉米植株、转入cry1ab-02核苷酸序列的玉米植株、转入cry1ac-01核苷酸序列的玉米植株、转入cry1a.105核苷酸序列的玉米植株、转入cry1a.105-cry2ab核苷酸序列的玉米植株和转入vip3a核苷酸序列的玉米植株均获得了单拷贝的转基因植株。
第五实施例、转基因大豆植株的抗虫效果检测
将转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry2ab核苷酸序列的大豆植株、转入cry1ac-02-cry1fa核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株、转入cry1a.105-cry2ab核苷酸序列的大豆植株、转入vip3a核苷酸序列的大豆植株、野生型大豆植株和经taqman鉴定为非转基因的大豆植株对草地螟进行抗虫效果检测。
分别取转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry2ab核苷酸序列的大豆植株、转入cry1ac-02-cry1fa核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株、转入cry1a.105-cry2ab核苷酸序列的大豆植株、转入vip3a核苷酸序列的大豆植株、野生型大豆植株和经taqman鉴定为非转基因的大豆植株(三叶期)的新鲜叶片,用无菌水冲洗干净并用纱布将叶片上的水吸干,然后将大豆叶片去除叶脉,同时剪成约2cm×3.5cm的长条状,取1片剪后的长条状叶片放入圆形塑料培养皿底部的保湿滤纸上,每个培养皿中放10头草地螟(初孵幼虫)后加盖,在温度25-28℃、相对湿度70%-80%、光周期(光/暗)16:8的条件下放置3天后,根据草地螟幼虫发育进度、死亡率和叶片损伤率三项指标,获得抗性总分(满分300分):抗性总分=100×死亡率+[100×死亡率+90×(初孵虫数/接虫总数)+60×(初孵-阴性对照虫数/接虫总数)+10×(阴性对照虫数/接虫总数)]+100×(1-叶片损伤率)。转入cry1ab-01核苷酸序列的共3个转化事件株系(s1、s2、s3),转入cry1ab-02核苷酸序列的共3个转化事件株系(s4、s5、s6),转入cry1ac-01核苷酸序列的共3个转化事件株系(s7、s8、s9),转入cry1ac-01-cry2ab核苷酸序列的共3个转化事件株系(s10、s11、s12),转入cry1ac-02-cry1fa核苷酸序列的共3个转化事件株系(s13、s14、s15),转入cry1a.105核苷酸序列的共3个转化事件株系(s16、s17、s18),转入cry1a.105-cry2ab核苷酸序列的共3个转化事件株系(s19、s20、s21),转入vip3a核苷酸序列的共3个转化事件株系(s22、s23、s24),经taqman鉴定为非转基因的(ngm1)共1个株系,野生型的(ck1)共1个株系;从每个株系选3株进行测试,每株重复1次。结果如表1所示。
表1、转基因大豆植株接种草地螟的抗虫实验结果
表1的结果表明:转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry2ab核苷酸序列的大豆植株、转入cry1ac-02-cry1fa核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株和转入cry1a.105-cry2ab核苷酸序列的大豆植株对草地螟具有较好的杀虫效果,草地螟的平均死亡率几乎为100%,其抗性总分基本上接近满分300分;而转入vip3a核苷酸序列的大豆植株虽然对草地螟具有杀虫活性,但致死率仅为20%以下,经taqman鉴定为非转基因的大豆植株和野生型大豆植株对草地螟均无致死或抑制作用,抗性总分一般在80左右。
与野生型大豆植株相比,转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry2ab核苷酸序列的大豆植株、转入cry1ac-02-cry1fa核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株和转入cry1a.105-cry2ab核苷酸序列的大豆植株对草地螟初孵幼虫的防治效果几乎为百分之百,极个别存活幼虫也基本上停止发育,3天后幼虫基本仍处于初孵状态,都是明显的发育不良,且已停止发育,在田间自然环境中无法存活;且转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry2ab核苷酸序列的大豆植株、转入cry1ac-02-cry1fa核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株和转入cry1a.105-cry2ab核苷酸序列的大豆植株大体上只受到轻微损伤,其叶片损伤率均在1%左右;而转入vip3a核苷酸序列的大豆植株虽然对草地螟具有杀虫活性,但防治效果不如cry1a蛋白明显,大部分幼虫可以存活,且其对叶片的损伤明显。
由此证明转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry2ab核苷酸序列的大豆植株、转入cry1ac-02-cry1fa核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株和转入cry1a.105-cry2ab核苷酸序列的大豆植株显示出高抗草地螟的活性,这种活性足以对草地螟的生长产生不良效应从而使其在田间得以控制。
第六实施例、转基因玉米植株的抗虫效果检测
将转入cry1ab-01核苷酸序列的玉米植株、转入cry1ab-01-vip3a核苷酸序列的玉米植株、转入cry1ab-02核苷酸序列的玉米植株、转入cry1ac-01核苷酸序列的玉米植株、转入cry1a.105核苷酸序列的玉米植株、转入cry1a.105-cry2ab核苷酸序列的玉米植株、转入vip3a核苷酸序列的玉米植株、野生型玉米植株和经taqman鉴定为非转基因的玉米植株对草地螟进行抗虫效果检测。
分别取转入cry1ab-01核苷酸序列的玉米植株、转入cry1ab-01-vip3a核苷酸序列的玉米植株、转入cry1ab-02核苷酸序列的玉米植株、转入cry1ac-01核苷酸序列的玉米植株、转入cry1a.105核苷酸序列的玉米植株、转入cry1a.105-cry2ab核苷酸序列的玉米植株、转入vip3a核苷酸序列的玉米植株、野生型玉米植株和经taqman鉴定为非转基因的玉米植株(v3-v4期)的新鲜叶片(心叶),用无菌水冲洗干净并用纱布将叶片上的水吸干,然后将玉米叶片去除叶脉,同时剪成约1cm×4cm的长条状,取1片剪后的长条状叶片放入圆形塑料培养皿底部的保湿滤纸上,每个培养皿中放1头草地螟(三龄幼虫)后加盖,在温度25-28℃、相对湿度70%-80%、光周期(光/暗)16:8的条件下放置3天后,根据草地螟幼虫发育进度、死亡率和叶片损伤率三项指标,获得抗性总分(满分300分):抗性总分=100×死亡率+[100×死亡率+90×(初孵虫数/接虫总数)+60×(初孵-阴性对照虫数/接虫总数)+10×(阴性对照虫数/接虫总数)]+100×(1-叶片损伤率)。转入cry1ab-01核苷酸序列的共3个转化事件株系(s25、s26、s27),转入cry1ab-01-vip3a核苷酸序列的共3个转化事件株系(s28、s29、s30),转入cry1ab-02核苷酸序列的共3个转化事件株系(s31、s32、s33),转入cry1ac-01核苷酸序列的共3个转化事件株系(s34、s35、s36),转入cry1a.105核苷酸序列的共3个转化事件株系(s37、s38、s39),转入cry1a.105-cry2ab核苷酸序列的共3个转化事件株系(s40、s41、s42),转入vip3a核苷酸序列的共3个转化事件株系(s43、s44、s45),经taqman鉴定为非转基因的(ngm2)共1个株系,野生型的(ck2)共1个株系;从每个株系选3株进行测试,每株重复1次。结果如表2所示。
表2、转基因玉米植株接种草地螟的抗虫实验结果
表2的结果表明:转入cry1ab-01核苷酸序列的玉米植株、转入cry1ab-01-vip3a核苷酸序列的玉米植株、转入cry1ab-02核苷酸序列的玉米植株、转入cry1ac-01核苷酸序列的玉米植株、转入cry1a.105核苷酸序列的玉米植株和转入cry1a.105-cry2ab核苷酸序列的玉米植株对草地螟具有较好的杀虫效果,草地螟的平均死亡率几乎为100%,其抗性总分基本上接近满分300分;而转入vip3a核苷酸序列的玉米植株虽然对草地螟具有杀虫活性,但致死率仅为10%以下,经taqman鉴定为非转基因的玉米植株和野生型玉米植株对草地螟均无致死或抑制作用,抗性总分一般在50左右。
同时,与野生型玉米植株相比,转入cry1ab-01核苷酸序列的玉米植株、转入cry1ab-01-vip3a核苷酸序列的玉米植株、转入cry1ab-02核苷酸序列的玉米植株、转入cry1ac-01核苷酸序列的玉米植株、转入cry1a.105核苷酸序列的玉米植株和转入cry1a.105-cry2ab核苷酸序列的玉米植株对草地螟三龄幼虫的防治效果几乎为百分之百,极个别存活幼虫也基本上停止发育,在田间自然环境中无法存活;且转入cry1ab-01核苷酸序列的玉米植株、转入cry1ab-01-vip3a核苷酸序列的玉米植株、转入cry1ab-02核苷酸序列的玉米植株、转入cry1ac-01核苷酸序列的玉米植株、转入cry1a.105核苷酸序列的玉米植株和转入cry1a.105-cry2ab核苷酸序列的玉米植株大体上只受到轻微损伤,其叶片损伤率均在5%左右;而转入vip3a核苷酸序列的玉米植株虽然对草地螟具有杀虫活性,但防治效果不如cry1a蛋白明显,大部分幼虫可以存活,且其对叶片的损伤明显。
由此证明转入cry1ab-01核苷酸序列的玉米植株、转入cry1ab-01-vip3a核苷酸序列的玉米植株、转入cry1ab-02核苷酸序列的玉米植株、转入cry1ac-01核苷酸序列的玉米植株、转入cry1a.105核苷酸序列的玉米植株和转入cry1a.105-cry2ab核苷酸序列的玉米植株显示出高抗草地螟的活性,这种活性足以对草地螟的生长产生不良效应从而使其在田间得以控制。
上述实验结果还表明:转入cry1ab-01核苷酸序列的大豆植株、转入cry1ab-02核苷酸序列的大豆植株、转入cry1ac-01核苷酸序列的大豆植株、转入cry1ac-01-cry2ab核苷酸序列的大豆植株、转入cry1ac-02-cry1fa核苷酸序列的大豆植株、转入cry1a.105核苷酸序列的大豆植株和转入cry1a.105-cry2ab核苷酸序列的大豆植株;转入cry1ab-01核苷酸序列的玉米植株、转入cry1ab-01-vip3a核苷酸序列的玉米植株、转入cry1ab-02核苷酸序列的玉米植株、转入cry1ac-01核苷酸序列的玉米植株、转入cry1a.105核苷酸序列的玉米植株和转入cry1a.105-cry2ab核苷酸序列的玉米植株对草地螟的控制/防治显然是因为植物本身可产生cry1a类蛋白,如cry1ab蛋白、cry1ac蛋白或cry1a.105蛋白,所以,本领域技术人员熟知的,根据cry1a蛋白对草地螟的毒杀作用,本发明中转入cry1a蛋白的植株还可以产生至少一种不同于cry1a蛋白的第二种杀虫蛋白质,如vip类蛋白或cry类蛋白等。
综上所述,本发明杀虫蛋白的用途通过植物体内产生能够杀死草地螟的cry1a蛋白来控制草地螟害虫;与现有技术使用的农业防治方法、化学防治方法、物理防治方法和生物防治方法相比,本发明对植物进行全生育期、全植株的保护以防治草地螟害虫的侵害,且无污染、无残留,效果稳定、彻底,简单、方便、经济。
最后所应说明的是,以上实施例仅用以说明本发明的技术方案而非限制,尽管参照较佳实施例对本发明进行了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的精神和范围。
序列表
<110>北京大北农生物技术有限公司
<120>杀虫蛋白的用途
<130>dbnbc135
<160>28
<170>siposequencelisting1.0
<210>1
<211>818
<212>prt
<213>人工序列-cry1ab-01氨基酸序列(artificialsequence)
<400>1
metaspasnasnproasnileasnglucysileprotyrasncysleu
151015
serasnprogluvalgluvalleuglyglygluargilegluthrgly
202530
tyrthrproileaspileserleuserleuthrglnpheleuleuser
354045
gluphevalproglyalaglyphevalleuglyleuvalaspileile
505560
trpglyilepheglyproserglntrpaspalapheleuvalglnile
65707580
gluglnleuileasnglnargileglugluphealaargasnglnala
859095
ileserargleugluglyleuserasnleutyrglniletyralaglu
100105110
serpheargglutrpglualaaspprothrasnproalaleuargglu
115120125
glumetargileglnpheasnaspmetasnseralaleuthrthrala
130135140
ileproleuphealavalglnasntyrglnvalproleuleuserval
145150155160
tyrvalglnalaalaasnleuhisleuservalleuargaspvalser
165170175
valpheglyglnargtrpglypheaspalaalathrileasnserarg
180185190
tyrasnaspleuthrargleuileglyasntyrthrasphisalaval
195200205
argtrptyrasnthrglyleugluargvaltrpglyproaspserarg
210215220
asptrpileargtyrasnglnpheargarggluleuthrleuthrval
225230235240
leuaspilevalserleupheproasntyraspserargthrtyrpro
245250255
ileargthrvalserglnleuthrarggluiletyrthrasnproval
260265270
leugluasnpheaspglyserpheargglyseralaglnglyileglu
275280285
glyserileargserprohisleumetaspileleuasnserilethr
290295300
iletyrthraspalahisargglyglutyrtyrtrpserglyhisgln
305310315320
ilemetalaserprovalglypheserglyprogluphethrphepro
325330335
leutyrglythrmetglyasnalaalaproglnglnargilevalala
340345350
glnleuglyglnglyvaltyrargthrleuserserthrleutyrarg
355360365
argpropheasnileglyileasnasnglnglnleuservalleuasp
370375380
glythrgluphealatyrglythrserserasnleuproseralaval
385390395400
tyrarglysserglythrvalaspserleuaspgluileproprogln
405410415
asnasnasnvalproproargglnglypheserhisargleuserhis
420425430
valsermetpheargserglypheserasnserservalserileile
435440445
argalaprometphesertrpilehisargseralaglupheasnasn
450455460
ileileproserserglnilethrglnileproleuthrlysserthr
465470475480
asnleuglyserglythrservalvallysglyproglyphethrgly
485490495
glyaspileleuargargthrserproglyglnileserthrleuarg
500505510
valasnilethralaproleuserglnargtyrargvalargilearg
515520525
tyralaserthrthrasnleuglnphehisthrserileaspglyarg
530535540
proileasnglnglyasnpheseralathrmetserserglyserasn
545550555560
leuglnserglyserpheargthrvalglyphethrthrpropheasn
565570575
pheserasnglyserservalphethrleuseralahisvalpheasn
580585590
serglyasngluvaltyrileaspargilegluphevalproalaglu
595600605
valthrpheglualaglutyraspleugluargalaglnlysalaval
610615620
asngluleuphethrserserasnglnileglyleulysthraspval
625630635640
thrasptyrhisileaspglnvalserasnleuvalglucysleuser
645650655
aspgluphecysleuaspglulyslysgluleuserglulysvallys
660665670
hisalalysargleuseraspgluargasnleuleuglnaspproasn
675680685
pheargglyileasnargglnleuaspargglytrpargglyserthr
690695700
aspilethrileglnglyglyaspaspvalphelysgluasntyrval
705710715720
thrleuleuglythrpheaspglucystyrprothrtyrleutyrgln
725730735
lysileaspgluserlysleulysalatyrthrargtyrglnleuarg
740745750
glytyrilegluaspserglnaspleugluiletyrleuileargtyr
755760765
asnalalyshisgluthrvalasnvalproglythrglyserleutrp
770775780
proleuseralaproserproileglylyscysalahishisserhis
785790795800
hispheserleuaspileaspvalglycysthraspleuasngluasp
805810815
phearg
<210>2
<211>2457
<212>dna
<213>人工序列-cry1ab-01核苷酸序列(artificialsequence)
<400>2
atggacaacaacccaaacatcaacgagtgcatcccgtacaactgcctcagcaaccctgag60
gtcgaggtgctcggcggtgagcgcatcgagaccggttacacccccatcgacatctccctc120
tccctcacgcagttcctgctcagcgagttcgtgccaggcgctggcttcgtcctgggcctc180
gtggacatcatctggggcatctttggcccctcccagtgggacgccttcctggtgcaaatc240
gagcagctcatcaaccagaggatcgaggagttcgccaggaaccaggccatcagccgcctg300
gagggcctcagcaacctctaccaaatctacgctgagagcttccgcgagtgggaggccgac360
cccactaacccagctctccgcgaggagatgcgcatccagttcaacgacatgaacagcgcc420
ctgaccaccgccatcccactcttcgccgtccagaactaccaagtcccgctcctgtccgtg480
tacgtccaggccgccaacctgcacctcagcgtgctgagggacgtcagcgtgtttggccag540
aggtggggcttcgacgccgccaccatcaacagccgctacaacgacctcaccaggctgatc600
ggcaactacaccgaccacgctgtccgctggtacaacactggcctggagcgcgtctggggc660
cctgattctagagactggattcgctacaaccagttcaggcgcgagctgaccctcaccgtc720
ctggacattgtgtccctcttcccgaactacgactcccgcacctacccgatccgcaccgtg780
tcccaactgacccgcgaaatctacaccaaccccgtcctggagaacttcgacggtagcttc840
aggggcagcgcccagggcatcgagggctccatcaggagcccacacctgatggacatcctc900
aacagcatcactatctacaccgatgcccaccgcggcgagtactactggtccggccaccag960
atcatggcctccccggtcggcttcagcggccccgagtttacctttcctctctacggcacg1020
atgggcaacgccgctccacaacaacgcatcgtcgctcagctgggccagggcgtctaccgc1080
accctgagctccaccctgtaccgcaggcccttcaacatcggtatcaacaaccagcagctg1140
tccgtcctggatggcactgagttcgcctacggcacctcctccaacctgccctccgctgtc1200
taccgcaagagcggcacggtggattccctggacgagatcccaccacagaacaacaatgtg1260
ccccccaggcagggtttttcccacaggctcagccacgtgtccatgttccgctccggcttc1320
agcaactcgtccgtgagcatcatcagagctcctatgttctcctggattcatcgcagcgcg1380
gagttcaacaatatcattccgtcctcccaaatcacccaaatccccctcaccaagtccacc1440
aacctgggcagcggcacctccgtggtgaagggcccaggcttcacgggcggcgacatcctg1500
cgcaggacctccccgggccagatcagcaccctccgcgtcaacatcaccgctcccctgtcc1560
cagaggtaccgcgtcaggattcgctacgctagcaccaccaacctgcaattccacacctcc1620
atcgacggcaggccgatcaatcagggtaacttctccgccaccatgtccagcggcagcaac1680
ctccaatccggcagcttccgcaccgtgggtttcaccacccccttcaacttctccaacggc1740
tccagcgttttcaccctgagcgcccacgtgttcaattccggcaatgaggtgtacattgac1800
cgcattgagttcgtgccagccgaggtcaccttcgaagccgagtacgacctggagagagcc1860
cagaaggctgtcaatgagctcttcacgtccagcaatcagatcggcctgaagaccgacgtc1920
actgactaccacatcgaccaagtctccaacctcgtggagtgcctctccgatgagttctgc1980
ctcgacgagaagaaggagctgtccgagaaggtgaagcatgccaagcgtctcagcgacgag2040
aggaatctcctccaggaccccaatttccgcggcatcaacaggcagctcgaccgcggctgg2100
cgcggcagcaccgacatcacgatccagggcggcgacgatgtgttcaaggagaactacgtg2160
actctcctgggcactttcgacgagtgctaccctacctacttgtaccagaagatcgatgag2220
tccaagctcaaggcttacactcgctaccagctccgcggctacatcgaagacagccaagac2280
ctcgagatttacctgatccgctacaacgccaagcacgagaccgtcaacgtgcccggtact2340
ggttccctctggccgctgagcgcccccagcccgatcggcaagtgtgcccaccacagccac2400
cacttctccttggacatcgatgtgggctgcaccgacctgaacgaggactttcggtag2457
<210>3
<211>615
<212>prt
<213>人工序列-cry1ab-02氨基酸序列(artificialsequence)
<400>3
metaspasnasnproasnileasnglucysileprotyrasncysleu
151015
serasnprogluvalgluvalleuglyglygluargilegluthrgly
202530
tyrthrproileaspileserleuserleuthrglnpheleuleuser
354045
gluphevalproglyalaglyphevalleuglyleuvalaspileile
505560
trpglyilepheglyproserglntrpaspalapheleuvalglnile
65707580
gluglnleuileasnglnargileglugluphealaargasnglnala
859095
ileserargleugluglyleuserasnleutyrglniletyralaglu
100105110
serpheargglutrpglualaaspprothrasnproalaleuargglu
115120125
glumetargileglnpheasnaspmetasnseralaleuthrthrala
130135140
ileproleuphealavalglnasntyrglnvalproleuleuserval
145150155160
tyrvalglnalaalaasnleuhisleuservalleuargaspvalser
165170175
valpheglyglnargtrpglypheaspalaalathrileasnserarg
180185190
tyrasnaspleuthrargleuileglyasntyrthrasphisalaval
195200205
argtrptyrasnthrglyleugluargvaltrpglyproaspserarg
210215220
asptrpileargtyrasnglnpheargarggluleuthrleuthrval
225230235240
leuaspilevalserleupheproasntyraspserargthrtyrpro
245250255
ileargthrvalserglnleuthrarggluiletyrthrasnproval
260265270
leugluasnpheaspglyserpheargglyseralaglnglyileglu
275280285
glyserileargserprohisleumetaspileleuasnserilethr
290295300
iletyrthraspalahisargglyglutyrtyrtrpserglyhisgln
305310315320
ilemetalaserprovalglypheserglyprogluphethrphepro
325330335
leutyrglythrmetglyasnalaalaproglnglnargilevalala
340345350
glnleuglyglnglyvaltyrargthrleuserserthrleutyrarg
355360365
argpropheasnileglyileasnasnglnglnleuservalleuasp
370375380
glythrgluphealatyrglythrserserasnleuproseralaval
385390395400
tyrarglysserglythrvalaspserleuaspgluileproprogln
405410415
asnasnasnvalproproargglnglypheserhisargleuserhis
420425430
valsermetpheargserglypheserasnserservalserileile
435440445
argalaprometphesertrpilehisargseralaglupheasnasn
450455460
ileileproserserglnilethrglnileproleuthrlysserthr
465470475480
asnleuglyserglythrservalvallysglyproglyphethrgly
485490495
glyaspileleuargargthrserproglyglnileserthrleuarg
500505510
valasnilethralaproleuserglnargtyrargvalargilearg
515520525
tyralaserthrthrasnleuglnphehisthrserileaspglyarg
530535540
proileasnglnglyasnpheseralathrmetserserglyserasn
545550555560
leuglnserglyserpheargthrvalglyphethrthrpropheasn
565570575
pheserasnglyserservalphethrleuseralahisvalpheasn
580585590
serglyasngluvaltyrileaspargilegluphevalproalaglu
595600605
valthrpheglualaglutyr
610615
<210>4
<211>1848
<212>dna
<213>人工序列-cry1ab-02核苷酸序列(artificialsequence)
<400>4
atggacaacaacccaaacatcaacgaatgcattccatacaactgcttgagtaacccagaa60
gttgaagtacttggtggagaacgcattgaaaccggttacactcccatcgacatctccttg120
tccttgacacagtttctgctcagcgagttcgtgccaggtgctgggttcgttctcggacta180
gttgacatcatctggggtatctttggtccatctcaatgggatgcattcctggtgcaaatt240
gagcagttgatcaaccagaggatcgaagagttcgccaggaaccaggccatctctaggttg300
gaaggattgagcaatctctaccaaatctatgcagagagcttcagagagtgggaagccgat360
cctactaacccagctctccgcgaggaaatgcgtattcaattcaacgacatgaacagcgcc420
ttgaccacagctatcccattgttcgcagtccagaactaccaagttcctctcttgtccgtg480
tacgttcaagcagctaatcttcacctcagcgtgcttcgagacgttagcgtgtttgggcaa540
aggtggggattcgatgctgcaaccatcaatagccgttacaacgaccttactaggctgatt600
ggaaactacaccgaccacgctgttcgttggtacaacactggcttggagcgtgtctggggt660
cctgattctagagattggattagatacaaccagttcaggagagaattgaccctcacagtt720
ttggacattgtgtctctcttcccgaactatgactccagaacctaccctatccgtacagtg780
tcccaacttaccagagaaatctatactaacccagttcttgagaacttcgacggtagcttc840
cgtggttctgcccaaggtatcgaaggctccatcaggagcccacacttgatggacatcttg900
aacagcataactatctacaccgatgctcacagaggagagtattactggtctggacaccag960
atcatggcctctccagttggattcagcgggcccgagtttacctttcctctctatggaact1020
atgggaaacgccgctccacaacaacgtatcgttgctcaactaggtcagggtgtctacaga1080
accttgtcttccaccttgtacagaagacccttcaatatcggtatcaacaaccagcaactt1140
tccgttcttgacggaacagagttcgcctatggaacctcttctaacttgccatccgctgtt1200
tacagaaagagcggaaccgttgattccttggacgaaatcccaccacagaacaacaatgtg1260
ccacccaggcaaggattctcccacaggttgagccacgtgtccatgttccgttccggattc1320
agcaacagttccgtgagcatcatcagagctcctatgttctcatggattcatcgtagtgct1380
gagttcaacaatatcattccttcctctcaaatcacccaaatcccattgaccaagtctact1440
aaccttggatctggaacttctgtcgtgaaaggaccaggcttcacaggaggtgatattctt1500
agaagaacttctcctggccagattagcaccctcagagttaacatcactgcaccactttct1560
caaagatatcgtgtcaggattcgttacgcatctaccactaacttgcaattccacacctcc1620
atcgacggaaggcctatcaatcagggtaacttctccgcaaccatgtcaagcggcagcaac1680
ttgcaatccggcagcttcagaaccgtcggtttcactactcctttcaacttctctaacgga1740
tcaagcgttttcacccttagcgctcatgtgttcaattctggcaatgaagtgtacattgac1800
cgtattgagtttgtgcctgccgaagttaccttcgaggctgagtactga1848
<210>5
<211>1178
<212>prt
<213>人工序列-cry1ac-01氨基酸序列(artificialsequence)
<400>5
metaspasnasnproasnileasnglucysileprotyrasncysleu
151015
serasnprogluvalgluvalleuglyglygluargilegluthrgly
202530
tyrthrproileaspileserleuserleuthrglnpheleuleuser
354045
gluphevalproglyalaglyphevalleuglyleuvalaspileile
505560
trpglyilepheglyproserglntrpaspalapheleuvalglnile
65707580
gluglnleuileasnglnargileglugluphealaargasnglnala
859095
ileserargleugluglyleuserasnleutyrglniletyralaglu
100105110
serpheargglutrpglualaaspprothrasnproalaleuargglu
115120125
glumetargileglnpheasnaspmetasnseralaleuthrthrala
130135140
ileproleuphealavalglnasntyrglnvalproleuleuserval
145150155160
tyrvalglnalaalaasnleuhisleuservalleuargaspvalser
165170175
valpheglyglnargtrpglypheaspalaalathrileasnserarg
180185190
tyrasnaspleuthrargleuileglyasntyrthrasphisalaval
195200205
argtrptyrasnthrglyleugluargvaltrpglyproaspserarg
210215220
asptrpileargtyrasnglnpheargarggluleuthrleuthrval
225230235240
leuaspilevalserleupheproasntyraspserargthrtyrpro
245250255
ileargthrvalserglnleuthrarggluiletyrthrasnproval
260265270
leugluasnpheaspglyserpheargglyseralaglnglyileglu
275280285
glyserileargserprohisleumetaspileleuasnserilethr
290295300
iletyrthraspalahisargglyglutyrtyrtrpserglyhisgln
305310315320
ilemetalaserprovalglypheserglyprogluphethrphepro
325330335
leutyrglythrmetglyasnalaalaproglnglnargilevalala
340345350
glnleuglyglnglyvaltyrargthrleuserserthrleutyrarg
355360365
argpropheasnileglyileasnasnglnglnleuservalleuasp
370375380
glythrgluphealatyrglythrserserasnleuproseralaval
385390395400
tyrarglysserglythrvalaspserleuaspgluileproprogln
405410415
asnasnasnvalproproargglnglypheserhisargleuserhis
420425430
valsermetpheargserglypheserasnserservalserileile
435440445
argalaprometphesertrpilehisargseralaglupheasnasn
450455460
ileilealaseraspserilethrglnileproalavallysglyasn
465470475480
pheleupheasnglyservalileserglyproglyphethrglygly
485490495
aspleuvalargleuasnserserglyasnasnileglnasnarggly
500505510
tyrilegluvalproilehispheproserthrserthrargtyrarg
515520525
valargvalargtyralaservalthrproilehisleuasnvalasn
530535540
trpglyasnserserilepheserasnthrvalproalathralathr
545550555560
serleuaspasnleuglnserserasppheglytyrphegluserala
565570575
asnalaphethrserserleuglyasnilevalglyvalargasnphe
580585590
serglythralaglyvalileileaspargpheglupheileproval
595600605
thralathrleuglualaglutyrasnleugluargalaglnlysala
610615620
valasnalaleuphethrserthrasnglnleuglyleulysthrasn
625630635640
valthrasptyrhisileaspglnvalserasnleuvalthrtyrleu
645650655
seraspgluphecysleuaspglulysarggluleuserglulysval
660665670
lyshisalalysargleuseraspgluargasnleuleuglnaspser
675680685
asnphelysaspileasnargglnprogluargglytrpglyglyser
690695700
thrglyilethrileglnglyglyaspaspvalphelysgluasntyr
705710715720
valthrleuserglythrpheaspglucystyrprothrtyrleutyr
725730735
glnlysileaspgluserlysleulysalaphethrargtyrglnleu
740745750
argglytyrilegluaspserglnaspleugluiletyrserilearg
755760765
tyrasnalalyshisgluthrvalasnvalproglythrglyserleu
770775780
trpproleuseralaglnserproileglylyscysglygluproasn
785790795800
argcysalaprohisleuglutrpasnproaspleuaspcyssercys
805810815
argaspglyglulyscysalahishisserhishispheserleuasp
820825830
ileaspvalglycysthraspleuasngluaspleuglyvaltrpval
835840845
ilephelysilelysthrglnaspglyhisalaargleuglyasnleu
850855860
glupheleugluglulysproleuvalglyglualaleualaargval
865870875880
lysargalaglulyslystrpargasplysargglulysleuglutrp
885890895
gluthrasnilevaltyrlysglualalysgluservalaspalaleu
900905910
phevalasnserglntyraspglnleuglnalaaspthrasnileala
915920925
metilehisalaalaasplysargvalhisserileargglualatyr
930935940
leuprogluleuservalileproglyvalasnalaalailepheglu
945950955960
gluleugluglyargilephethralapheserleutyraspalaarg
965970975
asnvalilelysasnglyasppheasnasnglyleusercystrpasn
980985990
vallysglyhisvalaspvalglugluglnasnasnglnargserval
99510001005
leuvalvalproglutrpglualagluvalserglngluvalargval
101010151020
cysproglyargglytyrileleuargvalthralatyrlysglugly
1025103010351040
tyrglygluglycysvalthrilehisgluilegluasnasnthrasp
104510501055
gluleulyspheserasncysvalgluglugluiletyrproasnasn
106010651070
thrvalthrcysasnasptyrthrvalasnglngluglutyrglygly
107510801085
alatyrthrserargasnargglytyrasnglualaproservalpro
109010951100
alaasptyralaservaltyrgluglulyssertyrthraspglyarg
1105111011151120
arggluasnprocysglupheasnargglytyrargasptyrthrpro
112511301135
leuprovalglytyrvalthrlysgluleuglutyrpheprogluthr
114011451150
asplysvaltrpilegluileglygluthrgluglythrpheileval
115511601165
aspservalgluleuleuleumetgluglu
11701175
<210>6
<211>3537
<212>dna
<213>人工序列-cry1ac-01核苷酸序列(artificialsequence)
<400>6
atggacaacaacccaaacatcaacgaatgcattccatacaactgcttgagtaacccagaa60
gttgaagtacttggtggagaacgcattgaaaccggttacactcccatcgacatctccttg120
tccttgacacagtttctgctcagcgagttcgtgccaggtgctgggttcgttctcggacta180
gttgacatcatctggggtatctttggtccatctcaatgggatgcattcctggtgcaaatt240
gagcagttgatcaaccagaggatcgaagagttcgccaggaaccaggccatctctaggttg300
gaaggattgagcaatctctaccaaatctatgcagagagcttcagagagtgggaagccgat360
cctactaacccagctctccgcgaggaaatgcgtattcaattcaacgacatgaacagcgcc420
ttgaccacagctatcccattgttcgcagtccagaactaccaagttcctctcttgtccgtg480
tacgttcaagcagctaatcttcacctcagcgtgcttcgagacgttagcgtgtttgggcaa540
aggtggggattcgatgctgcaaccatcaatagccgttacaacgaccttactaggctgatt600
ggaaactacaccgaccacgctgttcgttggtacaacactggcttggagcgtgtctggggt660
cctgattctagagattggattagatacaaccagttcaggagagaattgaccctcacagtt720
ttggacattgtgtctctcttcccgaactatgactccagaacctaccctatccgtacagtg780
tcccaacttaccagagaaatctatactaacccagttcttgagaacttcgacggtagcttc840
cgtggttctgcccaaggtatcgaaggctccatcaggagcccacacttgatggacatcttg900
aacagcataactatctacaccgatgctcacagaggagagtattactggtctggacaccag960
atcatggcctctccagttggattcagcgggcccgagtttacctttcctctctatggaact1020
atgggaaacgccgctccacaacaacgtatcgttgctcaactaggtcagggtgtctacaga1080
accttgtcttccaccttgtacagaagacccttcaatatcggtatcaacaaccagcaactt1140
tccgttcttgacggaacagagttcgcctatggaacctcttctaacttgccatccgctgtt1200
tacagaaagagcggaaccgttgattccttggacgaaatcccaccacagaacaacaatgtg1260
ccacccaggcaaggattctcccacaggttgagccacgtgtccatgttccgttccggattc1320
agcaacagttccgtgagcatcatcagagctcctatgttctcttggatacatcgtagtgct1380
gagttcaacaacatcatcgcatccgatagtattactcaaatccctgcagtgaagggaaac1440
tttctcttcaacggttctgtcatttcaggaccaggattcactggtggagacctcgttaga1500
ctcaacagcagtggaaataacattcagaatagagggtatattgaagttccaattcacttc1560
ccatccacatctaccagatatagagttcgtgtgaggtatgcttctgtgacccctattcac1620
ctcaacgttaattggggtaattcatccatcttctccaatacagttccagctacagctacc1680
tccttggataatctccaatccagcgatttcggttactttgaaagtgccaatgcttttaca1740
tcttcactcggtaacatcgtgggtgttagaaactttagtgggactgcaggagtgattatc1800
gacagattcgagttcattccagttactgcaacactcgaggctgagtacaaccttgagaga1860
gcccagaaggctgtgaacgccctctttacctccaccaatcagcttggcttgaaaactaac1920
gttactgactatcacattgaccaagtgtccaacttggtcacctaccttagcgatgagttc1980
tgcctcgacgagaagcgtgaactctccgagaaagttaaacacgccaagcgtctcagcgac2040
gagaggaatctcttgcaagactccaacttcaaagacatcaacaggcagccagaacgtggt2100
tggggtggaagcaccgggatcaccatccaaggaggcgacgatgtgttcaaggagaactac2160
gtcaccctctccggaactttcgacgagtgctaccctacctacttgtaccagaagatcgat2220
gagtccaaactcaaagccttcaccaggtatcaacttagaggctacatcgaagacagccaa2280
gaccttgaaatctactcgatcaggtacaatgccaagcacgagaccgtgaatgtcccaggt2340
actggttccctctggccactttctgcccaatctcccattgggaagtgtggagagcctaac2400
agatgcgctccacaccttgagtggaatcctgacttggactgctcctgcagggatggcgag2460
aagtgtgcccaccattctcatcacttctccttggacatcgatgtgggatgtactgacctg2520
aatgaggacctcggagtctgggtcatcttcaagatcaagacccaagacggacacgcaaga2580
cttggcaaccttgagtttctcgaagagaaaccattggtcggtgaagctctcgctcgtgtg2640
aagagagcagagaagaagtggagggacaaacgtgagaaactcgaatgggaaactaacatc2700
gtttacaaggaggccaaagagtccgtggatgctttgttcgtgaactcccaatatgatcag2760
ttgcaagccgacaccaacatcgccatgatccacgccgcagacaaacgtgtgcacagcatt2820
cgtgaggcttacttgcctgagttgtccgtgatccctggtgtgaacgctgccatcttcgag2880
gaacttgagggacgtatctttaccgcattctccttgtacgatgccagaaacgtcatcaag2940
aacggtgacttcaacaatggcctcagctgctggaatgtgaaaggtcatgtggacgtggag3000
gaacagaacaatcagcgttccgtcctggttgtgcctgagtgggaagctgaagtgtcccaa3060
gaggttagagtctgtccaggtagaggctacattctccgtgtgaccgcttacaaggaggga3120
tacggtgagggttgcgtgaccatccacgagatcgagaacaacaccgacgagcttaagttc3180
tccaactgcgtcgaggaagaaatctatcccaacaacaccgttacttgcaacgactacact3240
gtgaatcaggaagagtacggaggtgcctacactagccgtaacagaggttacaacgaagct3300
ccttccgttcctgctgactatgcctccgtgtacgaggagaaatcctacacagatggcaga3360
cgtgagaacccttgcgagttcaacagaggttacagggactacacaccacttccagttggc3420
tatgttaccaaggagcttgagtactttcctgagaccgacaaagtgtggatcgagatcggt3480
gaaaccgagggaaccttcatcgtggacagcgtggagcttctcttgatggaggaataa3537
<210>7
<211>1156
<212>prt
<213>人工序列-cry1ac-02氨基酸序列(artificialsequence)
<400>7
metaspasnasnproasnileasnglucysileprotyrasncysleu
151015
serasnprogluvalgluvalleuglyglygluargilegluthrgly
202530
tyrthrproileaspileserleuserleuthrglnpheleuleuser
354045
gluphevalproglyalaglyphevalleuglyleuvalaspileile
505560
trpglyilepheglyproserglntrpaspalapheleuvalglnile
65707580
gluglnleuileasnglnargileglugluphealaargasnglnala
859095
ileserargleugluglyleuserasnleutyrglniletyralaglu
100105110
serpheargglutrpglualaaspprothrasnproalaleuargglu
115120125
glumetargileglnpheasnaspmetasnseralaleuthrthrala
130135140
ileproleuphealavalglnasntyrglnvalproleuleuserval
145150155160
tyrvalglnalaalaasnleuhisleuservalleuargaspvalser
165170175
valpheglyglnargtrpglypheaspalaalathrileasnserarg
180185190
tyrasnaspleuthrargleuileglyasntyrthrasptyralaval
195200205
argtrptyrasnthrglyleugluargvaltrpglyproaspserarg
210215220
asptrpvalargtyrasnglnpheargarggluleuthrleuthrval
225230235240
leuaspilevalalaleupheproasntyraspserargargtyrpro
245250255
ileargthrvalserglnleuthrarggluiletyrthrasnproval
260265270
leugluasnpheaspglyserpheargglyseralaglnglyileglu
275280285
argserileargserprohisleumetaspileleuasnserilethr
290295300
iletyrthraspalahisargglytyrtyrtyrtrpserglyhisgln
305310315320
ilemetalaserprovalglypheserglyprogluphethrphepro
325330335
leutyrglythrmetglyasnalaalaproglnglnargilevalala
340345350
glnleuglyglnglyvaltyrargthrleuserserthrleutyrarg
355360365
argpropheasnileglyileasnasnglnglnleuservalleuasp
370375380
glythrgluphealatyrglythrserserasnleuproseralaval
385390395400
tyrarglysserglythrvalaspserleuaspgluileproprogln
405410415
asnasnasnvalproproargglnglypheserhisargleuserhis
420425430
valsermetpheargserglypheserasnserservalserileile
435440445
argalaprometphesertrpilehisargseralaglupheasnasn
450455460
ileilealaseraspserilethrglnileproalavallysglyasn
465470475480
pheleupheasnglyservalileserglyproglyphethrglygly
485490495
aspleuvalargleuasnserserglyasnasnileglnasnarggly
500505510
tyrilegluvalproilehispheproserthrserthrargtyrarg
515520525
valargvalargtyralaservalthrproilehisleuasnvalasn
530535540
trpglyasnserserilepheserasnthrvalproalathralathr
545550555560
serleuaspasnleuglnserserasppheglytyrphegluserala
565570575
asnalaphethrserserleuglyasnilevalglyvalargasnphe
580585590
serglythralaglyvalileileaspargpheglupheileproval
595600605
thralathrleuglualagluseraspleugluargalaglnlysala
610615620
valasnalaleuphethrserserasnglnileglyleulysthrasp
625630635640
valthrasptyrhisileaspargvalserasnleuvalglucysleu
645650655
seraspgluphecysleuaspglulyslysgluleuserglulysval
660665670
lyshisalalysargleuseraspgluargasnleuleuglnasppro
675680685
asnpheargglyileasnargglnleuaspargglytrpargglyser
690695700
thraspilethrileglnglyglyaspaspvalphelysgluasntyr
705710715720
valthrleuleuglythrpheaspglucystyrprothrtyrleutyr
725730735
glnlysileaspgluserlysleulysalatyrthrargtyrglnleu
740745750
argglytyrilegluaspserglnaspleugluiletyrleuilearg
755760765
tyrasnalalyshisgluthrvalasnvalproglythrglyserleu
770775780
trpproleuseralaproserproileglylyscysalahishisser
785790795800
hishispheserleuaspileaspvalglycysthraspleuasnglu
805810815
aspleuglyvaltrpvalilephelysilelysthrglnaspglyhis
820825830
alaargleuglyasnleuglupheleugluglulysproleuvalgly
835840845
glualaleualaargvallysargalaglulyslystrpargasplys
850855860
argglulysleuglutrpgluthrasnilevaltyrlysglualalys
865870875880
gluservalaspalaleuphevalasnserglntyraspargleugln
885890895
alaaspthrasnilealametilehisalaalaasplysargvalhis
900905910
serileargglualatyrleuprogluleuservalileproglyval
915920925
asnalaalailepheglugluleugluglyargilephethralaphe
930935940
serleutyraspalaargasnvalilelysasnglyasppheasnasn
945950955960
glyleusercystrpasnvallysglyhisvalaspvalgluglugln
965970975
asnasnhisargservalleuvalvalproglutrpglualagluval
980985990
serglngluvalargvalcysproglyargglytyrileleuargval
99510001005
thralatyrlysgluglytyrglygluglycysvalthrilehisglu
101010151020
ilegluasnasnthraspgluleulyspheserasncysvalgluglu
1025103010351040
gluvaltyrproasnasnthrvalthrcysasnasptyrthralathr
104510501055
glngluglutyrgluglythrtyrthrserargasnargglytyrasp
106010651070
glyalatyrgluserasnserservalproalaasptyralaserala
107510801085
tyrgluglulysalatyrthraspglyargargaspasnprocysglu
109010951100
serasnargglytyrglyasptyrthrproleuproalaglytyrval
1105111011151120
thrlysgluleuglutyrpheprogluthrasplysvaltrpileglu
112511301135
ileglygluthrgluglythrpheilevalaspservalgluleuleu
114011451150
leumetgluglu
1155
<210>8
<211>3471
<212>dna
<213>人工序列-cry1ac-02核苷酸序列(artificialsequence)
<400>8
atggacaacaatcccaacatcaacgagtgcattccttacaactgcctgagcaaccctgag60
gttgaggtgctgggtggagaacggattgagactggttacacacctatcgacatctcgttg120
tcacttacccaattccttttgtcagagttcgtgcccggtgctggattcgtgcttggactt180
gtcgatatcatttggggaatctttggtccctctcaatgggacgcctttcttgtacagata240
gagcagttaattaaccaaagaatagaagaattcgctaggaaccaagccatctcaaggtta300
gaaggcctcagcaacctttaccagatttacgcagaatcttttcgagagtgggaagcagac360
ccgaccaatcctgccttaagagaggagatgcgcattcaattcaatgacatgaacagcgcg420
ctgacgaccgcaattccgctcttcgccgttcagaattaccaagttcctcttttatccgtg480
tacgtgcaggctgccaacctgcacttgtcggtgctccgcgatgtctccgtgttcggacaa540
cggtggggctttgatgccgcaactatcaatagtcgttataatgatctgactaggcttatt600
ggcaactataccgattatgctgttcgctggtacaacacgggtctcgaacgtgtctgggga660
ccggattctagagattgggtcaggtacaaccagttcaggcgagagttgacactaactgtc720
ctagacattgtcgctctctttcccaactacgactctaggcgctacccaatccgtactgtg780
tcacaattgacccgggaaatctacacaaacccagtcctcgagaacttcgacggtagcttt840
cgaggctcggctcagggcatagagagaagcatcaggtctccacacctgatggacatattg900
aacagtatcacgatctacaccgatgcgcaccgcggttattactactggtcagggcatcag960
atcatggcatcacccgttgggttctctggaccagaattcactttcccactttacgggact1020
atgggcaatgcagctccacaacaacgtattgttgctcaactcggtcagggcgtgtataga1080
accttgtccagcactctatataggagacctttcaacatcggcatcaacaatcaacaattg1140
tctgtgcttgacgggacagaatttgcctatggaacctcctcaaatctgccatccgctgtc1200
tacagaaagagcggaacagttgatagcttggatgagatccctccacagaacaacaacgtt1260
ccacctaggcaagggtttagccatcgccttagccatgtgtccatgttccgttcaggcttt1320
agtaatagcagcgttagtatcatcagagctccgatgttctcttggatacatcgtagtgct1380
gagtttaacaacataattgcatccgatagcattactcagatcccagctgtcaaggggaac1440
tttctctttaatggttctgtcatttcaggaccaggattcactggaggcgacttggttagg1500
ctgaattcttccggcaacaacatccagaatagagggtatattgaagtgcccattcacttc1560
ccatcgacatctaccagatatcgtgttcgtgtaaggtatgcctctgttacccctattcac1620
ctcaacgtcaattggggtaattcctccatcttttccaatacagtaccagcgacagctaca1680
tccttggataatctccaatctagcgatttcggttacttcgaaagtgccaatgccttcacc1740
tcttccctaggtaacatagtaggtgttagaaatttctccggaaccgccggagtgataatc1800
gaccgcttcgaattcattcccgttactgcaacgctcgaggcagagtctgacttggaaaga1860
gcacagaaggcggtgaatgctctgttcacttcgtccaatcagattgggctcaagacagat1920
gtgactgactatcacatcgatcgcgtttccaaccttgttgagtgcctctctgatgagttc1980
tgtttggatgagaagaaggagttgtccgagaaggtcaaacatgctaagcgacttagtgat2040
gagcggaacttgcttcaagatcccaactttcgcgggatcaacaggcaactagatcgtgga2100
tggaggggaagtacggacatcaccattcaaggaggtgatgatgtgttcaaggagaactat2160
gttacgctcttgggtacctttgatgagtgctatccaacatacctgtaccagaagatagat2220
gaatcgaaactcaaagcctacacaagataccagttgagaggttacatcgaggacagtcaa2280
gaccttgagatctacctcatcagatacaacgccaaacatgagacagtcaatgtgcctggg2340
acgggttcactctggccactttcagccccaagtcccatcggcaagtgtgcccatcactca2400
caccacttctccttggacatagacgttggctgtaccgacctgaacgaagacctcggtgtg2460
tgggtgatcttcaagatcaagactcaagatggccatgccaggctaggcaatctggagttt2520
ctagaagagaaaccacttgttggagaagccctcgctagagtgaagagggctgagaagaag2580
tggagggacaagagagagaagttggaatgggaaacaaacattgtgtacaaagaagccaaa2640
gaaagcgttgacgctctgtttgtgaactctcagtatgataggctccaagctgataccaac2700
atagctatgattcatgctgcagacaaacgcgttcatagcattcgggaagcttaccttcct2760
gaacttagcgtgattccgggtgtcaatgctgctatctttgaagagttagaagggcgcatc2820
ttcactgcattctccttgtatgatgcgaggaatgtcatcaagaatggtgacttcaacaat2880
ggcctatcctgctggaatgtgaaagggcacgtagatgtagaagaacagaacaatcaccgc2940
tctgtccttgttgttcctgagtgggaagcagaagtttcacaagaagttcgtgtctgtcct3000
ggtcgtggctacattcttcgtgttaccgcgtacaaagaaggatacggagaaggttgcgtc3060
accatacacgagattgagaacaacaccgacgagctgaagttcagcaactgcgtcgaggag3120
gaagtctacccaaacaacaccgtaacttgcaatgactacactgcgactcaagaggagtat3180
gagggtacttacacttctcgcaatcgaggatacgatggagcctatgagagcaactcttct3240
gtacccgctgactatgcatcagcctatgaggagaaggcttacaccgatggacgtagggac3300
aatccttgcgaatctaacagaggctatggggactacacaccgttaccagccggctatgtc3360
accaaagagttagagtactttccagaaaccgacaaggtttggattgagattggagaaacg3420
gaaggaacattcattgttgatagcgtggagttacttctgatggaggaatga3471
<210>9
<211>1177
<212>prt
<213>人工序列-cry1a.105氨基酸序列(artificialsequence)
<400>9
metaspasnasnproasnileasnglucysileprotyrasncysleu
151015
serasnprogluvalgluvalleuglyglygluargilegluthrgly
202530
tyrthrproileaspileserleuserleuthrglnpheleuleuser
354045
gluphevalproglyalaglyphevalleuglyleuvalaspileile
505560
trpglyilepheglyproserglntrpaspalapheleuvalglnile
65707580
gluglnleuileasnglnargileglugluphealaargasnglnala
859095
ileserargleugluglyleuserasnleutyrglniletyralaglu
100105110
serpheargglutrpglualaaspprothrasnproalaleuargglu
115120125
glumetargileglnpheasnaspmetasnseralaleuthrthrala
130135140
ileproleuphealavalglnasntyrglnvalproleuleuserval
145150155160
tyrvalglnalaalaasnleuhisleuservalleuargaspvalser
165170175
valpheglyglnargtrpglypheaspalaalathrileasnserarg
180185190
tyrasnaspleuthrargleuileglyasntyrthrasphisalaval
195200205
argtrptyrasnthrglyleugluargvaltrpglyproaspserarg
210215220
asptrpileargtyrasnglnpheargarggluleuthrleuthrval
225230235240
leuaspilevalserleupheproasntyraspserargthrtyrpro
245250255
ileargthrvalserglnleuthrarggluiletyrthrasnproval
260265270
leugluasnpheaspglyserpheargglyseralaglnglyileglu
275280285
glyserileargserprohisleumetaspileleuasnserilethr
290295300
iletyrthraspalahisargglyglutyrtyrtrpserglyhisgln
305310315320
ilemetalaserprovalglypheserglyprogluphethrphepro
325330335
leutyrglythrmetglyasnalaalaproglnglnargilevalala
340345350
glnleuglyglnglyvaltyrargthrleuserserthrleutyrarg
355360365
argpropheasnileglyileasnasnglnglnleuservalleuasp
370375380
glythrgluphealatyrglythrserserasnleuproseralaval
385390395400
tyrarglysserglythrvalaspserleuaspgluileproprogln
405410415
asnasnasnvalproproargglnglypheserhisargleuserhis
420425430
valsermetpheargserglypheserasnserservalserileile
435440445
argalaprometphesertrpilehisargseralaglupheasnasn
450455460
ileilealaseraspserilethrglnileproleuvallysalahis
465470475480
thrleuglnserglythrthrvalvalargglyproglyphethrgly
485490495
glyaspileleuargargthrserglyglyprophealatyrthrile
500505510
valasnileasnglyglnleuproglnargtyrargalaargilearg
515520525
tyralaserthrthrasnleuargiletyrvalthrvalalaglyglu
530535540
argilephealaglyglnpheasnlysthrmetaspthrglyasppro
545550555560
leuthrpheglnserphesertyralathrileasnthralaphethr
565570575
pheprometserglnserserphethrvalglyalaaspthrpheser
580585590
serglyasngluvaltyrileaspargphegluleuileprovalthr
595600605
alathrleuglualaglutyrasnleugluargalaglnlysalaval
610615620
asnalaleuphethrserthrasnglnleuglyleulysthrasnval
625630635640
thrasptyrhisileaspglnvalserasnleuvalthrtyrleuser
645650655
aspgluphecysleuaspglulysarggluleuserglulysvallys
660665670
hisalalysargleuseraspgluargasnleuleuglnaspserasn
675680685
phelysaspileasnargglnprogluargglytrpglyglyserthr
690695700
glyilethrileglnglyglyaspaspvalphelysgluasntyrval
705710715720
thrleuserglythrpheaspglucystyrprothrtyrleutyrgln
725730735
lysileaspgluserlysleulysalaphethrargtyrglnleuarg
740745750
glytyrilegluaspserglnaspleugluiletyrserileargtyr
755760765
asnalalyshisgluthrvalasnvalproglythrglyserleutrp
770775780
proleuseralaglnserproileglylyscysglygluproasnarg
785790795800
cysalaprohisleuglutrpasnproaspleuaspcyssercysarg
805810815
aspglyglulyscysalahishisserhishispheserleuaspile
820825830
aspvalglycysthraspleuasngluaspleuglyvaltrpvalile
835840845
phelysilelysthrglnaspglyhisalaargleuglyasnleuglu
850855860
pheleugluglulysproleuvalglyglualaleualaargvallys
865870875880
argalaglulyslystrpargasplysargglulysleuglutrpglu
885890895
thrasnilevaltyrlysglualalysgluservalaspalaleuphe
900905910
valasnserglntyraspglnleuglnalaaspthrasnilealamet
915920925
ilehisalaalaasplysargvalhisserileargglualatyrleu
930935940
progluleuservalileproglyvalasnalaalailephegluglu
945950955960
leugluglyargilephethralapheserleutyraspalaargasn
965970975
valilelysasnglyasppheasnasnglyleusercystrpasnval
980985990
lysglyhisvalaspvalglugluglnasnasnglnargservalleu
99510001005
valvalproglutrpglualagluvalserglngluvalargvalcys
101010151020
proglyargglytyrileleuargvalthralatyrlysgluglytyr
1025103010351040
glygluglycysvalthrilehisgluilegluasnasnthraspglu
104510501055
leulyspheserasncysvalgluglugluiletyrproasnasnthr
106010651070
valthrcysasnasptyrthrvalasnglngluglutyrglyglyala
107510801085
tyrthrserargasnargglytyrasnglualaproservalproala
109010951100
asptyralaservaltyrgluglulyssertyrthraspglyargarg
1105111011151120
gluasnprocysglupheasnargglytyrargasptyrthrproleu
112511301135
provalglytyrvalthrlysgluleuglutyrpheprogluthrasp
114011451150
lysvaltrpilegluileglygluthrgluglythrpheilevalasp
115511601165
servalgluleuleuleumetgluglu
11701175
<210>10
<211>3534
<212>dna
<213>人工序列-cry1a.105核苷酸序列(artificialsequence)
<400>10
atggacaacaacccaaacatcaacgagtgcatcccgtacaactgcctcagcaaccctgag60
gtcgaggtgctcggcggtgagcgcatcgagaccggttacacccccatcgacatctccctc120
tccctcacgcagttcctgctcagcgagttcgtgccaggcgctggcttcgtcctgggcctc180
gtggacatcatctggggcatctttggcccctcccagtgggacgccttcctggtgcaaatc240
gagcagctcatcaaccagaggatcgaggagttcgccaggaaccaggccatcagccgcctg300
gagggcctcagcaacctctaccaaatctacgctgagagcttccgcgagtgggaggccgac360
cccactaacccagctctccgcgaggagatgcgcatccagttcaacgacatgaacagcgcc420
ctgaccaccgccatcccactcttcgccgtccagaactaccaagtcccgctcctgtccgtg480
tacgtccaggccgccaacctgcacctcagcgtgctgagggacgtcagcgtgtttggccag540
aggtggggcttcgacgccgccaccatcaacagccgctacaacgacctcaccaggctgatc600
ggcaactacaccgaccacgctgtccgctggtacaacactggcctggagcgcgtctggggc660
cctgattctagagactggattcgctacaaccagttcaggcgcgagctgaccctcaccgtc720
ctggacattgtgtccctcttcccgaactacgactcccgcacctacccgatccgcaccgtg780
tcccaactgacccgcgaaatctacaccaaccccgtcctggagaacttcgacggtagcttc840
aggggcagcgcccagggcatcgagggctccatcaggagcccacacctgatggacatcctc900
aacagcatcactatctacaccgatgcccaccgcggcgagtactactggtccggccaccag960
atcatggcctccccggtcggcttcagcggccccgagtttacctttcctctctacggcacg1020
atgggcaacgccgctccacaacaacgcatcgtcgctcagctgggccagggcgtctaccgc1080
accctgagctccaccctgtaccgcaggcccttcaacatcggtatcaacaaccagcagctg1140
tccgtcctggatggcactgagttcgcctacggcacctcctccaacctgccctccgctgtc1200
taccgcaagagcggcacggtggattccctggacgagatcccaccacagaacaacaatgtg1260
ccccccaggcagggtttttcccacaggctcagccacgtgtccatgttccgctccggcttc1320
agcaactcgtccgtgagcatcatcagagctcctatgttctcttggatacaccgtagtgct1380
gagttcaacaacatcattgcatccgacagcattactcaaatacccttggtgaaagcacat1440
acacttcagtcaggtactactgttgtcagaggtccagggtttacaggaggagacattctt1500
cgtcgcacaagtggaggaccctttgcttacactattgttaacatcaatggccaattgccc1560
caaaggtatcgtgcaagaatccgctatgcctctactacaaatctcaggatctacgtgact1620
gttgcaggtgaaaggatctttgctggtcagttcaacaagactatggataccggtgaccct1680
ttgacattccaatcttttagctacgcaactatcaacacagcttttacattcccaatgagc1740
cagagtagcttcacagtaggtgctgacactttcagctcagggaatgaagtttacatcgac1800
aggtttgaattgattccagttactgcaaccctcgaggctgagtacaaccttgagagagcc1860
cagaaggctgtgaacgccctctttacctccaccaatcagcttggcttgaaaactaacgtt1920
actgactatcacattgaccaagtgtccaacttggtcacctaccttagcgatgagttctgc1980
ctcgacgagaagcgtgaactctccgagaaagttaaacacgccaagcgtctcagcgacgag2040
aggaatctcttgcaagactccaacttcaaagacatcaacaggcagccagaacgtggttgg2100
ggtggaagcaccgggatcaccatccaaggaggcgacgatgtgttcaaggagaactacgtc2160
accctctccggaactttcgacgagtgctaccctacctacttgtaccagaagatcgatgag2220
tccaaactcaaagccttcaccaggtatcaacttagaggctacatcgaagacagccaagac2280
cttgaaatctactcgatcaggtacaatgccaagcacgagaccgtgaatgtcccaggtact2340
ggttccctctggccactttctgcccaatctcccattgggaagtgtggagagcctaacaga2400
tgcgctccacaccttgagtggaatcctgacttggactgctcctgcagggatggcgagaag2460
tgtgcccaccattctcatcacttctccttggacatcgatgtgggatgtactgacctgaat2520
gaggacctcggagtctgggtcatcttcaagatcaagacccaagacggacacgcaagactt2580
ggcaaccttgagtttctcgaagagaaaccattggtcggtgaagctctcgctcgtgtgaag2640
agagcagagaagaagtggagggacaaacgtgagaaactcgaatgggaaactaacatcgtt2700
tacaaggaggccaaagagtccgtggatgctttgttcgtgaactcccaatatgatcagttg2760
caagccgacaccaacatcgccatgatccacgccgcagacaaacgtgtgcacagcattcgt2820
gaggcttacttgcctgagttgtccgtgatccctggtgtgaacgctgccatcttcgaggaa2880
cttgagggacgtatctttaccgcattctccttgtacgatgccagaaacgtcatcaagaac2940
ggtgacttcaacaatggcctcagctgctggaatgtgaaaggtcatgtggacgtggaggaa3000
cagaacaatcagcgttccgtcctggttgtgcctgagtgggaagctgaagtgtcccaagag3060
gttagagtctgtccaggtagaggctacattctccgtgtgaccgcttacaaggagggatac3120
ggtgagggttgcgtgaccatccacgagatcgagaacaacaccgacgagcttaagttctcc3180
aactgcgtcgaggaagaaatctatcccaacaacaccgttacttgcaacgactacactgtg3240
aatcaggaagagtacggaggtgcctacactagccgtaacagaggttacaacgaagctcct3300
tccgttcctgctgactatgcctccgtgtacgaggagaaatcctacacagatggcagacgt3360
gagaacccttgcgagttcaacagaggttacagggactacacaccacttccagttggctat3420
gttaccaaggagcttgagtactttcctgagaccgacaaagtgtggatcgagatcggtgaa3480
accgagggaaccttcatcgtggacagcgtggagcttctcttgatggaggaataa3534
<210>11
<211>634
<212>prt
<213>人工序列-cry2ab氨基酸序列(artificialsequence)
<400>11
metaspasnservalleuasnserglyargthrthrilecysaspala
151015
tyrasnvalalaalahisasppropheserpheglnhislysserleu
202530
aspthrvalglnlysglutrpthrglutrplyslysasnasnhisser
354045
leutyrleuaspproilevalglythrvalalaserpheleuleulys
505560
lysvalglyserleuvalglylysargileleusergluleuargasn
65707580
leuilepheproserglyserthrasnleumetglnaspileleuarg
859095
gluthrglulyspheleuasnglnargleuasnthraspthrleuala
100105110
argvalasnalagluleuthrglyleuglnalaasnvalglugluphe
115120125
asnargglnvalaspasnpheleuasnproasnargasnalavalpro
130135140
leuserilethrserservalasnthrmetglnglnleupheleuasn
145150155160
argleuproglnpheglnmetglnglytyrglnleuleuleuleupro
165170175
leuphealaglnalaalaasnleuhisleuserpheileargaspval
180185190
ileleuasnalaaspglutrpglyileseralaalathrleuargthr
195200205
tyrargasptyrleulysasntyrthrargasptyrserasntyrcys
210215220
ileasnthrtyrglnseralaphelysglyleuasnthrargleuhis
225230235240
aspmetleuglupheargthrtyrmetpheleuasnvalpheglutyr
245250255
valseriletrpserleuphelystyrglnserleuleuvalserser
260265270
glyalaasnleutyralaserglyserglyproglnglnthrglnser
275280285
phethrserglnasptrppropheleutyrserleupheglnvalasn
290295300
serasntyrvalleuasnglypheserglyalaargleuserasnthr
305310315320
pheproasnilevalglyleuproglyserthrthrthrhisalaleu
325330335
leualaalaargvalasntyrserglyglyileserserglyaspile
340345350
glyalaserpropheasnglnasnpheasncysserthrpheleupro
355360365
proleuleuthrprophevalargsertrpleuaspserglyserasp
370375380
arggluglyvalalathrvalthrasntrpglnthrgluserpheglu
385390395400
thrthrleuglyleuargserglyalaphethralaargglyasnser
405410415
asntyrpheproasptyrpheileargasnileserglyvalproleu
420425430
valvalargasngluaspleuargargproleuhistyrasngluile
435440445
argasnilealaserproserglythrproglyglyalaargalatyr
450455460
metvalservalhisasnarglysasnasnilehisalavalhisglu
465470475480
asnglysermetilehisleualaproasnasptyrthrglyphethr
485490495
ileserproilehisalathrglnvalasnasnglnthrargthrphe
500505510
ileserglulyspheglyasnglnglyaspserleuargpheglugln
515520525
asnasnthrthralaargtyrthrleuargglyasnglyasnsertyr
530535540
asnleutyrleuargvalserserileglyasnserthrileargval
545550555560
thrileasnglyargvaltyrthralathrasnvalasnthrthrthr
565570575
asnasnaspglyvalasnaspasnglyalaargpheseraspileasn
580585590
ileglyasnvalvalalaserserasnseraspvalproleuaspile
595600605
asnvalthrleuasnserglythrglnpheaspleumetasnilemet
610615620
leuvalprothrasnileserproleutyr
625630
<210>12
<211>1905
<212>dna
<213>人工序列-cry2ab核苷酸序列(artificialsequence)
<400>12
atggacaactccgtcctgaactctggtcgcaccaccatctgcgacgcctacaacgtcgcg60
gcgcatgatccattcagcttccagcacaagagcctcgacactgttcagaaggagtggacg120
gagtggaagaagaacaaccacagcctgtacctggaccccatcgtcggcacggtggccagc180
ttccttctcaagaaggtcggctctctcgtcgggaagcgcatcctctcggaactccgcaac240
ctgatctttccatctggctccaccaacctcatgcaagacatcctcagggagaccgagaag300
tttctcaaccagcgcctcaacactgatacccttgctcgcgtcaacgctgagctgacgggt360
ctgcaagcaaacgtggaggagttcaaccgccaagtggacaacttcctcaaccccaaccgc420
aatgcggtgcctctgtccatcacttcttccgtgaacaccatgcaacaactgttcctcaac480
cgcttgcctcagttccagatgcaaggctaccagctgctcctgctgccactctttgctcag540
gctgccaacctgcacctctccttcattcgtgacgtgatcctcaacgctgacgagtggggc600
atctctgcagccacgctgaggacctaccgcgactacctgaagaactacaccagggactac660
tccaactattgcatcaacacctaccagtcggccttcaagggcctcaatacgaggcttcac720
gacatgctggagttcaggacctacatgttcctgaacgtgttcgagtacgtcagcatctgg780
tcgctcttcaagtaccagagcctgctggtgtccagcggcgccaacctctacgccagcggc840
tctggtccccaacaaactcagagcttcaccagccaggactggccattcctgtattcgttg900
ttccaagtcaactccaactacgtcctcaacggcttctctggtgctcgcctctccaacacc960
ttccccaacattgttggcctccccggctccaccacaactcatgctctgcttgctgccaga1020
gtgaactactccggcggcatctcgagcggcgacattggtgcatcgccgttcaaccagaac1080
ttcaactgctccaccttcctgccgccgctgctcaccccgttcgtgaggtcctggctcgac1140
agcggctccgaccgcgagggcgtggccaccgtcaccaactggcaaaccgagtccttcgag1200
accacccttggcctccggagcggcgccttcacggcgcgtggaaattctaactacttcccc1260
gactacttcatcaggaacatctctggtgttcctctcgtcgtccgcaacgaggacctccgc1320
cgtccactgcactacaacgagatcaggaacatcgcctctccgtccgggacgcccggaggt1380
gcaagggcgtacatggtgagcgtccataacaggaagaacaacatccacgctgtgcatgag1440
aacggctccatgatccacctggcgcccaatgattacaccggcttcaccatctctccaatc1500
cacgccacccaagtgaacaaccagacacgcaccttcatctccgagaagttcggcaaccag1560
ggcgactccctgaggttcgagcagaacaacaccaccgccaggtacaccctgcgcggcaac1620
ggcaacagctacaacctgtacctgcgcgtcagctccattggcaactccaccatcagggtc1680
accatcaacgggagggtgtacacagccaccaatgtgaacacgacgaccaacaatgatggc1740
gtcaacgacaacggcgcccgcttcagcgacatcaacattggcaacgtggtggccagcagc1800
aactccgacgtcccgctggacatcaacgtgaccctgaactctggcacccagttcgacctc1860
atgaacatcatgctggtgccaactaacatctcgccgctgtactga1905
<210>13
<211>1148
<212>prt
<213>人工序列-cry1fa氨基酸序列(artificialsequence)
<400>13
metgluasnasnileglnasnglncysvalprotyrasncysleuasn
151015
asnprogluvalgluileleuasnglugluargserthrglyargleu
202530
proleuaspileserleuserleuthrargleuleuleusergluphe
354045
valproglyvalglyvalalapheglyleupheaspleuiletrpgly
505560
pheilethrproserasptrpserleupheleuleuglnileglugln
65707580
leuilegluglnargilegluthrleugluargasnargalailethr
859095
thrleuargglyleualaaspsertyrgluiletyrileglualaleu
100105110
argglutrpglualaasnproasnasnalaglnleuarggluaspval
115120125
argileargphealaasnthraspaspalaleuilethralaileasn
130135140
asnphethrleuthrserphegluileproleuleuservaltyrval
145150155160
glnalaalaasnleuhisleuserleuleuargaspalavalserphe
165170175
glyglnglytrpglyleuaspilealathrvalasnasnhistyrasn
180185190
argleuileasnleuilehisargtyrthrlyshiscysleuaspthr
195200205
tyrasnglnglyleugluasnleuargglythrasnthrargglntrp
210215220
alaargpheasnglnpheargargaspleuthrleuthrvalleuasp
225230235240
ilevalalaleupheproasntyraspvalargthrtyrproilegln
245250255
thrserserglnleuthrarggluiletyrthrserservalileglu
260265270
aspserprovalseralaasnileproasnglypheasnargalaglu
275280285
pheglyvalargproprohisleumetaspphemetasnserleuphe
290295300
valthralagluthrvalargserglnthrvaltrpglyglyhisleu
305310315320
valserserargasnthralaglyasnargileasnpheprosertyr
325330335
glyvalpheasnproglyglyalailetrpilealaaspgluasppro
340345350
argprophetyrargthrleuseraspprovalphevalargglygly
355360365
pheglyasnprohistyrvalleuglyleuargglyvalalaphegln
370375380
glnthrglythrasnhisthrargthrpheargasnserglythrile
385390395400
aspserleuaspgluileproproglnaspasnserglyalaprotrp
405410415
asnasptyrserhisvalleuasnhisvalthrphevalargtrppro
420425430
glygluileserglyseraspsertrpargalaprometphesertrp
435440445
thrhisargseralathrprothrasnthrileaspprogluargile
450455460
thrglnileproleuvallysalahisthrleuglnserglythrthr
465470475480
valvalargglyproglyphethrglyglyaspileleuargargthr
485490495
serglyglyprophealatyrthrilevalasnileasnglyglnleu
500505510
proglnargtyrargalaargileargtyralaserthrthrasnleu
515520525
argiletyrvalthrvalalaglygluargilephealaglyglnphe
530535540
asnlysthrmetaspthrglyaspproleuthrpheglnserpheser
545550555560
tyralathrileasnthralaphethrpheprometserglnserser
565570575
phethrvalglyalaaspthrpheserserglyasngluvaltyrile
580585590
aspargphegluleuileprovalthralathrleuglualagluser
595600605
aspleugluargalaglnlysalavalasnalaleuphethrserser
610615620
asnglnileglyleulysthraspvalthrasptyrhisileasparg
625630635640
valserasnleuvalglucysleuseraspgluphecysleuaspglu
645650655
lyslysgluleuserglulysvallyshisalalysargleuserasp
660665670
gluargasnleuleuglnaspproasnpheargglyileasnarggln
675680685
leuaspargglytrpargglyserthraspilethrileglnglygly
690695700
aspaspvalphelysgluasntyrvalthrleuleuglythrpheasp
705710715720
glucystyrprothrtyrleutyrglnlysileaspgluserlysleu
725730735
lysalatyrthrargtyrglnleuargglytyrilegluaspsergln
740745750
aspleugluiletyrleuileargtyrasnalalyshisgluthrval
755760765
asnvalproglythrglyserleutrpproleuseralaproserpro
770775780
ileglylyscysalahishisserhishispheserleuaspileasp
785790795800
valglycysthraspleuasngluaspleuglyvaltrpvalilephe
805810815
lysilelysthrglnaspglyhisalaargleuglyasnleugluphe
820825830
leugluglulysproleuvalglyglualaleualaargvallysarg
835840845
alaglulyslystrpargasplysargglulysleuglutrpgluthr
850855860
asnilevaltyrlysglualalysgluservalaspalaleupheval
865870875880
asnserglntyraspargleuglnalaaspthrasnilealametile
885890895
hisalaalaasplysargvalhisserileargglualatyrleupro
900905910
gluleuservalileproglyvalasnalaalailepheglugluleu
915920925
gluglyargilephethralapheserleutyraspalaargasnval
930935940
ilelysasnglyasppheasnasnglyleusercystrpasnvallys
945950955960
glyhisvalaspvalglugluglnasnasnhisargservalleuval
965970975
valproglutrpglualagluvalserglngluvalargvalcyspro
980985990
glyargglytyrileleuargvalthralatyrlysgluglytyrgly
99510001005
gluglycysvalthrilehisgluilegluasnasnthraspgluleu
101010151020
lyspheserasncysvalgluglugluvaltyrproasnasnthrval
1025103010351040
thrcysasnasptyrthralathrglngluglutyrgluglythrtyr
104510501055
thrserargasnargglytyraspglyalatyrgluserasnserser
106010651070
valproalaasptyralaseralatyrgluglulysalatyrthrasp
107510801085
glyargargaspasnprocysgluserasnargglytyrglyasptyr
109010951100
thrproleuproalaglytyrvalthrlysgluleuglutyrphepro
1105111011151120
gluthrasplysvaltrpilegluileglygluthrgluglythrphe
112511301135
ilevalaspservalgluleuleuleumetgluglu
11401145
<210>14
<211>3447
<212>dna
<213>人工序列-cry1fa核苷酸序列(artificialsequence)
<400>14
atggagaacaacatacagaatcagtgcgtcccctacaactgcctcaacaatcctgaagta60
gagattctcaacgaagagaggtcgactggcagattgccgttagacatctccctgtccctt120
acacgtctcctgttgtctgagtttgttccaggtgtgggagttgcgtttggcctcttcgac180
ctcatctggggcttcatcactccatctgattggagcctctttcttctccagattgaacag240
ttgattgaacaaaggattgagaccttggaaaggaatcgggccatcactacccttcgtggc300
ttagcagacagctatgagatctacattgaagcactaagagagtgggaagccaatcctaac360
aatgcccaactgagagaagatgtgcgtatacgctttgctaacacagatgatgctttgatc420
acagccatcaacaacttcacccttaccagcttcgagatccctcttctctcggtctatgtt480
caagctgctaacctgcacttgtcactactgcgcgacgctgtgtcgtttgggcaaggttgg540
ggactggacatagctactgtcaacaatcactacaacagactcatcaatctgattcatcga600
tacacgaaacattgtttggatacctacaatcagggattggagaacctgagaggtactaac660
actcgccaatgggccaggttcaatcagttcaggagagaccttacacttactgtgttagac720
atagttgctctctttccgaactacgatgttcgtacctatccgattcaaacgtcatcccaa780
cttacaagggagatctacaccagttcagtcattgaagactctccagtttctgcgaacata840
cccaatggtttcaacagggctgagtttggagtcagaccaccccatctcatggacttcatg900
aactctttgtttgtgactgcagagactgttagatcccaaactgtgtggggaggacactta960
gttagctcacgcaacacggctggcaatcgtatcaactttcctagttacggggtcttcaat1020
cccgggggcgccatctggattgcagatgaagatccacgtcctttctatcggaccttgtca1080
gatcctgtcttcgtccgaggaggctttggcaatcctcactatgtactcggtcttagggga1140
gtggcctttcaacaaactggtacgaatcacacccgcacattcaggaactccgggaccatt1200
gactctctagatgagataccacctcaagacaacagcggcgcaccttggaatgactactcc1260
catgtgctgaatcatgttacctttgtgcgctggccaggtgagatctcaggttccgactca1320
tggagagcaccaatgttctcttggacgcatcgtagcgctacccccacaaacaccattgat1380
ccagagagaatcactcagattcccttggtgaaggcacacacacttcagtcaggaactaca1440
gttgtaagagggccggggttcacgggaggagacattcttcgacgcactagtggaggacca1500
ttcgcgtacaccattgtcaacatcaatgggcaacttccccaaaggtatcgtgccaggata1560
cgctatgcctctactaccaatctaagaatctacgttacggttgcaggtgaacggatcttt1620
gctggtcagttcaacaagacaatggataccggtgatccacttacattccaatctttctcc1680
tacgccactatcaacaccgcgttcacctttccaatgagccagagcagtttcacagtaggt1740
gctgataccttcagttcaggcaacgaagtgtacattgacaggtttgagttgattccagtt1800
actgccacactcgaggcagagtctgacttggaaagagcacagaaggcggtgaatgctctg1860
ttcacttcgtccaatcagattgggctcaagacagatgtgactgactatcacatcgatcgc1920
gtttccaaccttgttgagtgcctctctgatgagttctgtttggatgagaagaaggagttg1980
tccgagaaggtcaaacatgctaagcgacttagtgatgagcggaacttgcttcaagatccc2040
aactttcgcgggatcaacaggcaactagatcgtggatggaggggaagtacggacatcacc2100
attcaaggaggtgatgatgtgttcaaggagaactatgttacgctcttgggtacctttgat2160
gagtgctatccaacatacctgtaccagaagatagatgaatcgaaactcaaagcctacaca2220
agataccagttgagaggttacatcgaggacagtcaagaccttgagatctacctcatcaga2280
tacaacgccaaacatgagacagtcaatgtgcctgggacgggttcactctggccactttca2340
gccccaagtcccatcggcaagtgtgcccatcactcacaccacttctccttggacatagac2400
gttggctgtaccgacctgaacgaagacctcggtgtgtgggtgatcttcaagatcaagact2460
caagatggccatgccaggctaggcaatctggagtttctagaagagaaaccacttgttgga2520
gaagccctcgctagagtgaagagggctgagaagaagtggagggacaagagagagaagttg2580
gaatgggaaacaaacattgtgtacaaagaagccaaagaaagcgttgacgctctgtttgtg2640
aactctcagtatgataggctccaagctgataccaacatagctatgattcatgctgcagac2700
aaacgcgttcatagcattcgggaagcttaccttcctgaacttagcgtgattccgggtgtc2760
aatgctgctatctttgaagagttagaagggcgcatcttcactgcattctccttgtatgat2820
gcgaggaatgtcatcaagaatggtgacttcaacaatggcctatcctgctggaatgtgaaa2880
gggcacgtagatgtagaagaacagaacaatcaccgctctgtccttgttgttcctgagtgg2940
gaagcagaagtttcacaagaagttcgtgtctgtcctggtcgtggctacattcttcgtgtt3000
accgcgtacaaagaaggatacggagaaggttgcgtcaccatacacgagattgagaacaac3060
accgacgagctgaagttcagcaactgcgtcgaggaggaagtctacccaaacaacaccgta3120
acttgcaatgactacactgcgactcaagaggagtatgagggtacttacacttctcgcaat3180
cgaggatacgatggagcctatgagagcaactcttctgtacccgctgactatgcatcagcc3240
tatgaggagaaggcttacaccgatggacgtagggacaatccttgcgaatctaacagaggc3300
tatggggactacacaccgttaccagccggctatgtcaccaaagagttagagtactttcca3360
gaaaccgacaaggtttggattgagattggagaaacggaaggaacattcattgttgatagc3420
gtggagttacttctgatggaggaatga3447
<210>15
<211>789
<212>prt
<213>人工序列-vip3a氨基酸序列(artificialsequence)
<400>15
metasnlysasnasnthrlysleuserthrargalaleuproserphe
151015
ileasptyrpheasnglyiletyrglyphealathrglyilelysasp
202530
ilemetasnmetilephelysthraspthrglyglyaspleuthrleu
354045
aspgluileleulysasnglnglnleuleuasnaspileserglylys
505560
leuaspglyvalasnglyserleuasnaspleuilealaglnglyasn
65707580
leuasnthrgluleuserlysgluileleulysilealaasnglugln
859095
asnglnvalleuasnaspvalasnasnlysleuaspalaileasnthr
100105110
metleuargvaltyrleuprolysilethrsermetleuseraspval
115120125
metlysglnasntyralaleuserleuglnileglutyrleuserlys
130135140
glnleuglngluileserasplysleuaspileileasnvalasnval
145150155160
leuileasnserthrleuthrgluilethrproalatyrglnargile
165170175
lystyrvalasnglulyspheglugluleuthrphealathrgluthr
180185190
serserlysvallyslysaspglyserproalaaspileleuaspglu
195200205
leuthrgluleuthrgluleualalysservalthrlysasnaspval
210215220
aspglyphegluphetyrleuasnthrphehisaspvalmetvalgly
225230235240
asnasnleupheglyargseralaleulysthralasergluleuile
245250255
thrlysgluasnvallysthrserglysergluvalglyasnvaltyr
260265270
asnpheleuilevalleuthralaleuglnalaglnalapheleuthr
275280285
leuthrthrcysarglysleuleuglyleualaaspileasptyrthr
290295300
serilemetasngluhisleuasnlysglulysgluglupheargval
305310315320
asnileleuprothrleuserasnthrpheserasnproasntyrala
325330335
lysvallysglyseraspgluaspalalysmetilevalglualalys
340345350
proglyhisalaleuileglyphegluileserasnaspserilethr
355360365
valleulysvaltyrglualalysleulysglnasntyrglnvalasp
370375380
lysaspserleusergluvaliletyrglyaspmetasplysleuleu
385390395400
cysproaspglnsergluglniletyrtyrthrasnasnilevalphe
405410415
proasnglutyrvalilethrlysileaspphethrlyslysmetlys
420425430
thrleuargtyrgluvalthralaasnphetyraspserserthrgly
435440445
gluileaspleuasnlyslyslysvalgluserserglualaglutyr
450455460
argthrleuseralaasnaspaspglyvaltyrmetproleuglyval
465470475480
ilesergluthrpheleuthrproileasnglypheglyleuglnala
485490495
aspgluasnserargleuilethrleuthrcyslyssertyrleuarg
500505510
gluleuleuleualathraspleuserasnlysgluthrlysleuile
515520525
valproproserglypheileserasnilevalgluasnglyserile
530535540
glugluaspasnleugluprotrplysalaasnasnlysasnalatyr
545550555560
valasphisthrglyglyvalasnglythrlysalaleutyrvalhis
565570575
lysaspglyglyileserglnpheileglyasplysleulysprolys
580585590
thrglutyrvalileglntyrthrvallysglylysproserilehis
595600605
leulysaspgluasnthrglytyrilehistyrgluaspthrasnasn
610615620
asnleugluasptyrglnthrileasnlysargphethrthrglythr
625630635640
aspleulysglyvaltyrleuileleulysserglnasnglyaspglu
645650655
alatrpglyaspasnpheileileleugluileserproserglulys
660665670
leuleuserprogluleuileasnthrasnasntrpthrserthrgly
675680685
serthrasnileserglyasnthrleuthrleutyrglnglyglyarg
690695700
glyileleulysglnasnleuglnleuaspserpheserthrtyrarg
705710715720
valtyrpheservalserglyaspalaasnvalargileargasnser
725730735
arggluvalleupheglulysargtyrmetserglyalalysaspval
740745750
serglumetphethrthrlyspheglulysaspasnphetyrileglu
755760765
leuserglnglyasnasnleutyrglyglyproilevalhisphetyr
770775780
aspvalserilelys
785
<210>16
<211>2370
<212>dna
<213>人工序列-vip3a核苷酸序列(artificialsequence)
<400>16
atgaacaagaacaacaccaagctctccacacgggcacttccctcctttattgactacttt60
aatggcatctatgggtttgctacggggatcaaggacattatgaacatgatcttcaagaca120
gacactggcggggatcttacgctcgacgagattcttaagaatcagcaactcctgaacgat180
atctctggcaagctggacggcgtgaatgggtcacttaacgacctcatcgctcaggggaat240
ctcaacacagaactgtctaaggagatcctcaagattgcaaatgagcagaaccaagttctt300
aatgatgtgaacaataagctcgacgccatcaacacaatgcttcgcgtgtacctcccaaag360
attactagcatgctctcggacgtcatgaagcagaactacgcgctgtcccttcaaattgag420
tatctgagcaagcagcttcaagaaatctcggacaagctggatatcattaatgtgaacgtc480
ctcatcaacagcaccctgacggagattacaccggcgtaccagaggatcaagtatgtgaat540
gagaagttcgaggaactcacttttgctacagaaacttccagcaaggtcaagaaggatggc600
tcaccagccgacatcctggatgagcttacagaactcactgagctggcgaagtccgtgacc660
aagaatgacgtcgatggcttcgagttttacctgaacacgttccacgacgttatggtgggc720
aacaatctttttgggcggagcgctctcaagactgcatcggaactgatcaccaaggagaac780
gttaagacgagcggctcggaggtcgggaatgtttacaacttccttatcgtcctcaccgca840
ctccaggcccaagcgtttctcacgctgaccacctgccgcaagctcctcggcctcgcagac900
atcgattacacctccatcatgaacgagcacctgaacaaggagaaggaggagttccgcgtg960
aatatccttccgacactctcgaacactttttctaatccaaactacgctaaggtcaagggc1020
tccgacgaagatgcaaagatgatcgttgaggccaagcctggccatgcgctcatcgggttc1080
gagatttctaacgactcaattaccgtgctgaaggtctacgaggcgaagctcaagcagaat1140
tatcaagtggacaaggattctctgtcagaggttatctacggcgacatggataagctgctt1200
tgccctgatcagtccgagcaaatctactatacgaacaatattgtcttccccaacgaatac1260
gtgatcaccaagattgactttacgaagaagatgaagacactccggtacgaggtgacggct1320
aacttctatgattcgtctacgggcgagatcgacctcaacaagaagaaggtcgaatcatcc1380
gaggccgaatacagaaccctgtcggcgaacgacgatggcgtgtatatgcctcttggggtc1440
atttctgagaccttcctcacgcccatcaatggctttgggctccaggcagatgagaactcc1500
cgcctgatcacccttacgtgcaagagctacctcagggagctgctgcttgccaccgacctc1560
tctaacaaggaaacgaagctgatcgttccgccatcaggcttcatctccaatattgtggag1620
aacgggtcaattgaggaagataatctggaaccgtggaaggctaacaataagaacgcatac1680
gttgaccacacaggcggggtgaatggcactaaggcgctctatgtgcataaggatggtggc1740
atctcccagttcattggcgacaagctgaagccgaagacagaatacgtgattcaatatact1800
gtgaagggcaagccaagcatccacctcaaggatgagaacacagggtacatccattacgaa1860
gatactaacaacaacctggaggactaccagacaatcaataagaggttcacaactggcact1920
gacctgaagggggtctatcttattctcaagtcccagaatggcgatgaggcctggggcgac1980
aacttcatcattctcgaaatctcccctagcgagaagctcctgagccccgagctgattaac2040
accaataactggacatccactggcagcacgaatatctcggggaacaccctgacgctttac2100
cagggcgggagaggcattctgaagcagaacctccaactggattcgttctctacctacaga2160
gtctatttttcagtttccggcgacgcgaatgtgcgcatcaggaactcgcgggaagtcctc2220
ttcgagaagagatacatgtctggcgctaaggatgtgtcagaaatgttcaccacgaagttt2280
gagaaggacaacttttatatcgaactgtcccaagggaataacctctacggcggccccatt2340
gttcatttttacgacgtgagcatcaagtga2370
<210>17
<211>1322
<212>dna
<213>拟南芥(arabidopsisthaliana)
<400>17
gtcgacctgcaggtcaacggatcaggatattcttgtttaagatgttgaactctatggagg60
tttgtatgaactgatgatctaggaccggataagttcccttcttcatagcgaacttattca120
aagaatgttttgtgtatcattcttgttacattgttattaatgaaaaaatattattggtca180
ttggactgaacacgagtgttaaatatggaccaggccccaaataagatccattgatatatg240
aattaaataacaagaataaatcgagtcaccaaaccacttgccttttttaacgagacttgt300
tcaccaacttgatacaaaagtcattatcctatgcaaatcaataatcatacaaaaatatcc360
aataacactaaaaaattaaaagaaatggataatttcacaatatgttatacgataaagaag420
ttacttttccaagaaattcactgattttataagcccacttgcattagataaatggcaaaa480
aaaaacaaaaaggaaaagaaataaagcacgaagaattctagaaaatacgaaatacgcttc540
aatgcagtgggacccacggttcaattattgccaattttcagctccaccgtatatttaaaa600
aataaaacgataatgctaaaaaaatataaatcgtaacgatcgttaaatctcaacggctgg660
atcttatgacgaccgttagaaattgtggttgtcgacgagtcagtaataaacggcgtcaaa720
gtggttgcagccggcacacacgagtcgtgtttatcaactcaaagcacaaatacttttcct780
caacctaaaaataaggcaattagccaaaaacaactttgcgtgtaaacaacgctcaataca840
cgtgtcattttattattagctattgcttcaccgccttagctttctcgtgacctagtcgtc900
ctcgtcttttcttcttcttcttctataaaacaatacccaaagcttcttcttcacaattca960
gatttcaatttctcaaaatcttaaaaactttctctcaattctctctaccgtgatcaaggt1020
aaatttctgtgttccttattctctcaaaatcttcgattttgttttcgttcgatcccaatt1080
tcgtatatgttctttggtttagattctgttaatcttagatcgaagacgattttctgggtt1140
tgatcgttagatatcatcttaattctcgattagggtttcataaatatcatccgatttgtt1200
caaataatttgagttttgtcgaataattactcttcgatttgtgatttctatctagatctg1260
gtgttagtttctagtttgtgcgatcgaatttgtcgattaatctgagtttttctgattaac1320
ag1322
<210>18
<211>530
<212>dna
<213>agrobacteriumtumefaciens
<400>18
ccatggagtcaaagattcaaatagaggacctaacagaactcgccgtaaagactggcgaac60
agttcatacagagtctcttacgactcaatgacaagaagaaaatcttcgtcaacatggtgg120
agcacgacacgcttgtctactccaaaaatatcaaagatacagtctcagaagaccaaaggg180
caattgagacttttcaacaaagggtaatatccggaaacctcctcggattccattgcccag240
ctatctgtcactttattgtgaagatagtggaaaaggaaggtggctcctacaaatgccatc300
attgcgataaaggaaaggccatcgttgaagatgcctctgccgacagtggtcccaaagatg360
gacccccacccacgaggagcatcgtggaaaaagaagacgttccaaccacgtcttcaaagc420
aagtggattgatgtgatatctccactgacgtaagggatgacgcacaatcccactatcctt480
cgcaagacccttcctctatataaggaagttcatttcatttggagaggaca530
<210>19
<211>529
<212>dna
<213>启动子(cauliflowermosaicvirus)
<400>19
gtcctctccaaatgaaatgaacttccttatatagaggaagggtcttgcgaaggatagtgg60
gattgtgcgtcatcccttacgtcagtggagatatcacatcaatccacttgctttgaagac120
gtggttggaacgtcttctttttccacgatgctcctcgtgggtgggggtccatctttggga180
ccactgtcggcagaggcatcttcaacgatggcctttcctttatcgcaatgatggcatttg240
taggagccaccttccttttccactatcttcacaataaagtgacagatagctgggcaatgg300
aatccgaggaggtttccggatattaccctttgttgaaaagtctcaattgccctttggtct360
tctgagactgtatctttgatatttttggagtagacaagcgtgtcgtgctccaccatgttg420
acgaagattttcttcttgtcattgagtcgtaagagactctgtatgaactgttcgccagtc480
tttacggcgagttctgttaggtcctctatttgaatctttgactccatgg529
<210>20
<211>552
<212>dna
<213>streptomycesviridochromogenes
<400>20
atgtctccggagaggagaccagttgagattaggccagctacagcagctgatatggccgcg60
gtttgtgatatcgttaaccattacattgagacgtctacagtgaactttaggacagagcca120
caaacaccacaagagtggattgatgatctagagaggttgcaagatagatacccttggttg180
gttgctgaggttgagggtgttgtggctggtattgcttacgctgggccctggaaggctagg240
aacgcttacgattggacagttgagagtactgtttacgtgtcacataggcatcaaaggttg300
ggcctaggatccacattgtacacacatttgcttaagtctatggaggcgcaaggttttaag360
tctgtggttgctgttataggccttccaaacgatccatctgttaggttgcatgaggctttg420
ggatacacagcccggggtacattgcgcgcagctggatacaagcatggtggatggcatgat480
gttggtttttggcaaagggattttgagttgccagctcctccaaggccagttaggccagtt540
acccagatctga552
<210>21
<211>195
<212>dna
<213>终止子(cauliflowermosaicvirus)
<400>21
ctgaaatcaccagtctctctctacaaatctatctctctctataataatgtgtgagtagtt60
cccagataagggaattagggttcttatagggtttcgctcatgtgttgagcatataagaaa120
cccttagtatgtatttgtatttgtaaaatacttctatcaataaaatttctaattcctaaa180
accaaaatccagtgg195
<210>22
<211>1744
<212>dna
<213>拟南芥启动子(arabidopsisthaliana)
<400>22
aattcaaatttattatgtgttttttttccgtggtcgagattgtgtattattctttagtta60
ttacaagacttttagctaaaatttgaaagaatttactttaagaaaatcttaacatctgag120
ataatttcagcaatagattatatttttcattactctagcagtatttttgcagatcaatcg180
caacatatatggttgttagaaaaaatgcactatatatatatatattattttttcaattaa240
aagtgcatgatatataatatatatatatatatatatgtgtgtgtgtatatggtcaaagaa300
attcttatacaaatatacacgaacacatatatttgacaaaatcaaagtattacactaaac360
aatgagttggtgcatggccaaaacaaatatgtagattaaaaattccagcctccaaaaaaa420
aatccaagtgttgtaaagcattatatatatatagtagatcccaaatttttgtacaattcc480
acactgatcgaatttttaaagttgaatatctgacgtaggatttttttaatgtcttacctg540
accatttactaataacattcatacgttttcatttgaaatatcctctataattatattgaa600
tttggcacataataagaaacctaattggtgatttattttactagtaaatttctggtgatg660
ggctttctactagaaagctctcggaaaatcttggaccaaatccatattccatgacttcga720
ttgttaaccctattagttttcacaaacatactatcaatatcattgcaacggaaaaggtac780
aagtaaaacattcaatccgatagggaagtgatgtaggaggttgggaagacaggcccagaa840
agagatttatctgacttgttttgtgtatagttttcaatgttcataaaggaagatggagac900
ttgagaagttttttttggactttgtttagctttgttgggcgtttttttttttgatcaata960
actttgttgggcttatgatttgtaatattttcgtggactctttagtttatttagacgtgc1020
taactttgttgggcttatgacttgttgtaacatattgtaacagatgacttgatgtgcgac1080
taatctttacacattaaacatagttctgttttttgaaagttcttattttcatttttattt1140
gaatgttatatatttttctatatttataattctagtaaaaggcaaattttgcttttaaat1200
gaaaaaaatatatattccacagtttcacctaatcttatgcatttagcagtacaaattcaa1260
aaatttcccatttttattcatgaatcataccattatatattaactaaatccaaggtaaaa1320
aaaaggtatgaaagctctatagtaagtaaaatataaattccccataaggaaagggccaag1380
tccaccaggcaagtaaaatgagcaagcaccactccaccatcacacaatttcactcataga1440
taacgataagattcatggaattatcttccacgtggcattattccagcggttcaagccgat1500
aagggtctcaacacctctccttaggcctttgtggccgttaccaagtaaaattaacctcac1560
acatatccacactcaaaatccaacggtgtagatcctagtccacttgaatctcatgtatcc1620
tagaccctccgatcactccaaagcttgttctcattgttgttatcattatatatagatgac1680
caaagcactagaccaaacctcagtcacacaaagagtaaagaagaacaatggcttcctcta1740
tgct1744
<210>23
<211>515
<212>dna
<213>木薯叶脉花叶病毒启动子(manihotesculenta)
<400>23
ccagaaggtaattatccaagatgtagcatcaagaatccaatgtttacgggaaaaactatg60
gaagtattatgtgagctcagcaagaagcagatcaatatgcggcacatatgcaacctatgt120
tcaaaaatgaagaatgtacagatacaagatcctatactgccagaatacgaagaagaatac180
gtagaaattgaaaaagaagaaccaggcgaagaaaagaatcttgaagacgtaagcactgac240
gacaacaatgaaaagaagaagataaggtcggtgattgtgaaagagacatagaggacacat300
gtaaggtggaaaatgtaagggcggaaagtaaccttatcacaaaggaatcttatcccccac360
tacttatccttttatatttttccgtgtcatttttgcccttgagttttcctatataaggaa420
ccaagttcggcatttgtgaaaacaagaaaaaatttggtgtaagctattttctttgaagta480
ctgaggatacaagttcagagaaatttgtaagtttg515
<210>24
<211>1992
<212>dna
<213>启动子(zeamays)
<400>24
ctgcagtgcagcgtgacccggtcgtgcccctctctagagataatgagcattgcatgtcta60
agttataaaaaattaccacatattttttttgtcacacttgtttgaagtgcagtttatcta120
tctttatacatatatttaaactttactctacgaataatataatctatagtactacaataa180
tatcagtgttttagagaatcatataaatgaacagttagacatggtctaaaggacaattga240
gtattttgacaacaggactctacagttttatctttttagtgtgcatgtgttctccttttt300
ttttgcaaatagcttcacctatataatacttcatccattttattagtacatccatttagg360
gtttagggttaatggtttttatagactaatttttttagtacatctattttattctatttt420
agcctctaaattaagaaaactaaaactctattttagtttttttatttaataatttagata480
taaaatagaataaaataaagtgactaaaaattaaacaaataccctttaagaaattaaaaa540
aactaaggaaacatttttcttgtttcgagtagataatgccagcctgttaaacgccgtcga600
cgagtctaacggacaccaaccagcgaaccagcagcgtcgcgtcgggccaagcgaagcaga660
cggcacggcatctctgtcgctgcctctggacccctctcgagagttccgctccaccgttgg720
acttgctccgctgtcggcatccagaaattgcgtggcggagcggcagacgtgagccggcac780
ggcaggcggcctcctcctcctctcacggcacggcagctacgggggattcctttcccaccg840
ctccttcgctttcccttcctcgcccgccgtaataaatagacaccccctccacaccctctt900
tccccaacctcgtgttgttcggagcgcacacacacacaaccagatctcccccaaatccac960
ccgtcggcacctccgcttcaaggtacgccgctcgtcctccccccccccccctctctacct1020
tctctagatcggcgttccggtccatggttagggcccggtagttctacttctgttcatgtt1080
tgtgttagatccgtgtttgtgttagatccgtgctgctagcgttcgtacacggatgcgacc1140
tgtacgtcagacacgttctgattgctaacttgccagtgtttctctttggggaatcctggg1200
atggctctagccgttccgcagacgggatcgatttcatgattttttttgtttcgttgcata1260
gggtttggtttgcccttttcctttatttcaatatatgccgtgcacttgtttgtcgggtca1320
tcttttcatgcttttttttgtcttggttgtgatgatgtggtctggttgggcggtcgttct1380
agatcggagtagaattctgtttcaaactacctggtggatttattaattttggatctgtat1440
gtgtgtgccatacatattcatagttacgaattgaagatgatggatggaaatatcgatcta1500
ggataggtatacatgttgatgcgggttttactgatgcatatacagagatgctttttgttc1560
gcttggttgtgatgatgtggtgtggttgggcggtcgttcattcgttctagatcggagtag1620
aatactgtttcaaactacctggtgtatttattaattttggaactgtatgtgtgtgtcata1680
catcttcatagttacgagtttaagatggatggaaatatcgatctaggataggtatacatg1740
ttgatgtgggttttactgatgcatatacatgatggcatatgcagcatctattcatatgct1800
ctaaccttgagtacctatctattataataaacaagtatgttttataattattttgatctt1860
gatatacttggatgatggcatatgcagcagctatatgtggatttttttagccctgccttc1920
atacgctatttatttgcttggtactgtttcttttgtcgatgctcaccctgttgtttggtg1980
ttacttctgcag1992
<210>25
<211>1176
<212>dna
<213>escherichiacoli
<400>25
atgcaaaaactcattaactcagtgcaaaactatgcctggggcagcaaaacggcgttgact60
gaactttatggtatggaaaatccgtccagccagccgatggccgagctgtggatgggcgca120
catccgaaaagcagttcacgagtgcagaatgccgccggagatatcgtttcactgcgtgat180
gtgattgagagtgataaatcgactctgctcggagaggccgttgccaaacgctttggcgaa240
ctgcctttcctgttcaaagtattatgcgcagcacagccactctccattcaggttcatcca300
aacaaacacaattctgaaatcggttttgccaaagaaaatgccgcaggtatcccgatggat360
gccgccgagcgtaactataaagatcctaaccacaagccggagctggtttttgcgctgacg420
cctttccttgcgatgaacgcgtttcgtgaattttccgagattgtctccctactccagccg480
gtcgcaggtgcacatccggcgattgctcactttttacaacagcctgatgccgaacgttta540
agcgaactgttcgccagcctgttgaatatgcagggtgaagaaaaatcccgcgcgctggcg600
attttaaaatcggccctcgatagccagcagggtgaaccgtggcaaacgattcgtttaatt660
tctgaattttacccggaagacagcggtctgttctccccgctattgctgaatgtggtgaaa720
ttgaaccctggcgaagcgatgttcctgttcgctgaaacaccgcacgcttacctgcaaggc780
gtggcgctggaagtgatggcaaactccgataacgtgctgcgtgcgggtctgacgcctaaa840
tacattgatattccggaactggttgccaatgtgaaattcgaagccaaaccggctaaccag900
ttgttgacccagccggtgaaacaaggtgcagaactggacttcccgattccagtggatgat960
tttgccttctcgctgcatgaccttagtgataaagaaaccaccattagccagcagagtgcc1020
gccattttgttctgcgtcgaaggcgatgcaacgttgtggaaaggttctcagcagttacag1080
cttaaaccgggtgaatcagcgtttattgccgccaacgaatcaccggtgactgtcaaaggc1140
cacggccgtttagcgcgtgtttacaacaagctgtaa1176
<210>26
<211>22
<212>dna
<213>人工序列-引物1(artificialsequence)
<400>26
gagggtgttgtggctggtattg22
<210>27
<211>23
<212>dna
<213>人工序列-引物2(artificialsequence)
<400>27
tctcaactgtccaatcgtaagcg23
<210>28
<211>25
<212>dna
<213>人工序列-探针1(artificialsequence)
<400>28
cttacgctgggccctggaaggctag25
- 还没有人留言评论。精彩留言会获得点赞!