短肽重组克隆构建筛选的载体及其应用的制作方法
本发明涉及基因工程技术领域,更具体地涉及用于短肽重组克隆构建筛选的载体及其非构象型抗体表位鉴定的应用。
背景技术:
众所周知,表位肽疫苗设计和病毒抗体检测用的肽抗原试剂研制,都基于靶蛋白上特异性非构象型抗体线性表位肽的研究,因此鉴定那些功能性和保守性/特异性表位始终是是非常重要的研究内容。
从上世纪七八十年代起,相继发明建立了多种靶抗原线性表位扫描作图或鉴定的方法[沈关心等译.抗体技术实验指南.科学出版社,2002;王洪林等.b细胞表位定位的研究进展.生命科学,21:317-319,2009]。基本上可以分类为四大类:1)化学合成肽(chemicallysyntheticpeptide,csp)法和肽芯片法;2)通过基因工程短肽融合表达的生物合成肽(biosyntheticpeptide,bsp)法、噬菌体随机肽或抗原片段展示文库法和表达pcr技术等;3)通过依据特定靶抗原的物理化学参数的计算机表位预测法;和4)应用酶和化学试剂的抗原水解法。但是这四大类方法,包括其大量改良或衍生方法,都存在这样那样的限制,在实际应用中效果始终不理想,都无法达到自然科学和应用研究所期望实现的目标和要求。以应用csp法为例,在使用多克隆抗体(多抗)的场合,始终不能鉴定每一个非构象型抗体所能识别的表位最小基序肽(最短3个最长8个aa残基),报道的往往是含特异性表位的较长甚至是37个aa残基的抗原性肽),导致常常只能鉴定一或少数几个抗原性肽,因此不能揭示是发现更多功能性表位基础的任一靶蛋白抗原的完整非构象型igg-表位组;尽管用单克隆抗体(单抗)能鉴定它们所识别表位的最小基序,但csp法存在业界公知的缺陷,如短肽化学合成费用高和用elisa法的抗原抗体反应的结果不易判定或不方便被他人重复等,所以用csp法鉴定表位最小基序的研究报道至今屈指可数,难得一见。
最近,许多研究者试图使用类似于csp法的bsp技术开展感兴趣抗原或特定功能域的抗体鉴定和表位作图研究,其中作为短肽(融合蛋白)表达的载体,分别使用了由118aa组成的核心链亲和素(corestreptavidin,stv118)、带肽酶切位点的全长gst蛋白和由590aa组成的截短β-半乳糖苷酶(β-galactosidase,gal590)蛋白。这些研究,包括编码四肽基因片段与stv118基因连接的克隆成功、使用由188个aa组成的截短gst188蛋白作为载体,真正能像csp法一样通过基因工程任意表达8/18肽(gst188-8/18肽融合蛋白)的最初bsp法建立,其特征是使用了短肽和对照蛋白热诱导原核表达质粒,以及随后能进一步体现这一bsp法突出优点的改良,即相对elisa方法,在表位作图和精细表位识别基序鉴定的蛋白印迹鉴定中,第一次实现了可清晰明确显示抗原抗体反应条带的ecl化学发光显色。但是,bsp法尚存一些待克服的明显缺陷,如难以排除不完全酶切产生的自连克隆,严重干扰重组克隆构建和筛选的效率,目的融合蛋白与自连克隆表达的蛋白难以区分,容易将自连克隆误判为重组克隆,耗费不必要的测序费用和时间,影响耽搁研究进程。
综上所述,本领域迫切需要寻找更佳的质粒表达系统和融合蛋白表达载体,以更方便、准确和高效地构建和筛选短肽重组克隆,包括使用短肽融合蛋白鉴定精细抗原表位的应用。
技术实现要素:
本发明的目的是提供一种能更方便、准确和高效地构建和筛选短肽重组克隆的表达盒及热诱导表达质粒,以进一步完善专门用于表位作图鉴定的生物合成肽法。
本发明的第一方面,提供了一种表达盒,所述表达盒从5’-3’具有式i结构:
z1-s1-z2-s2-z3-z4式(i)
式中,
z1为第一核酸元件,所述第一核酸元件编码截短gst188多肽;
s1为第一酶切位点;
z2为第二核酸元件,所述第二核酸元件编码抗干扰多肽;
s2为第二酶切位点;
z3为无或密码子;
z4为无或polya;
其中,所述z2具有以下特征:
(a)长度为3m个核苷酸,其中m为70-140的正整数(较佳地80-130,更佳地90-120,最佳地100-110);
(b)不含终止密码子;和
(c)不含s1和s2。
在另一优选例中,所述s1和s2分别为单酶切位点或多克隆酶切位点。
在另一优选例中,所述s1为bamhi酶切位点。
在另一优选例中,所述s2为sali酶切位点。
在另一优选例中,所述z1的序列如seqidno.:1中第16-579位所示。
在另一优选例中,所述z2的序列如seqidno.:1中第586-900位所示。
在另一优选例中,所述表达盒的序列如seqidno.:1中第16-906位所示。
本发明的第二方面,提供了一种载体,所述载体包含本发明第一方面所述的表达盒。
在另一优选例中,所述载体依次含有以下元件:复制起点(ori)、clt857元件、prpl元件、本发明第一方面所述的表达盒和抗性筛选基因(如ampr、氨苄青霉素抗性基因)。
在另一优选例中,所述载体为质粒。
在另一优选例中,所述载体的序列如seqidno.:1所示。
本发明的第三方面,提供了一种短肽融合蛋白表达载体的构建方法,所述构建方法包括步骤:
(1)提供第一核酸分子和第二核酸分子,其中,所述第一核酸分子为本发明第二方面所述的载体经针对s1和s2酶切位点的双酶切消化后得到的大片段,所述第二核酸分子为两端带有s1和s2酶切位点的短肽编码序列;
(2)连接所述第一核酸分子和第二核酸分子,得到短肽融合蛋白表达载体。
在另一优选例中,所述短肽融合蛋白具有式ii结构:
gst188-a式(ii)
式中,gst188为截短gst蛋白的188个氨基酸多肽,a元件为长度为6-20个氨基酸的短肽;
在另一优选例中,所述gst188的氨基酸序列如seqidno.:3中第1-188位所示。
在另一优选例中,a元件是长度为8-18个氨基酸的短肽。
本发明的第四方面,提供了一种靶抗原表位鉴定方法,所述方法包括步骤:
(a)提供第一核酸分子和第二核酸分子,其中,所述第一核酸分子为本发明第二方面所述的载体经针对s1和s2酶切位点的酶消化后得到的大片段,所述第二核酸分子为两端带有s1和s2酶切位点的短肽编码序列;
(b)连接所述第一核酸分子和第二核酸分子,得到短肽融合蛋白表达载体;
(c)培养含所述短肽融合蛋白表达载体的宿主细胞,并诱导所述宿主细胞表达式ii所示的融合蛋白
gst188-a式(ii)
式中,gst188为截短gst蛋白的188个氨基酸多肽,a元件为长度为6-20个氨基酸的短肽;
(d)将表达的式ii所示的融合蛋白与抗体混合,观察抗体是否与式ii所述的融合蛋白发生抗体-抗原反应;
其中,如果抗体与式ii所述的融合蛋白发生抗体-抗原反应,那么就表示所述融合蛋白中的a元件是一靶蛋白的抗原性多肽。
在另一优选例中,所述步骤(c)中表达2-100个氨基酸(更佳地4-50个,最佳地5-20个)式ii所示的融合蛋白。
在另一优选例中,所述步骤(c)中的短肽来自同一个蛋白。
在另一优选例中,所述宿主细胞是大肠杆菌。
本发明的第五方面,提供了一种本发明第一方面所述的表达盒或本发明第二方面所述的载体的用途,其特征在于,用于短肽生物合成和/或靶抗原或抗原性肽的精细表位鉴定。
应理解,在本发明范围内中,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。
附图说明
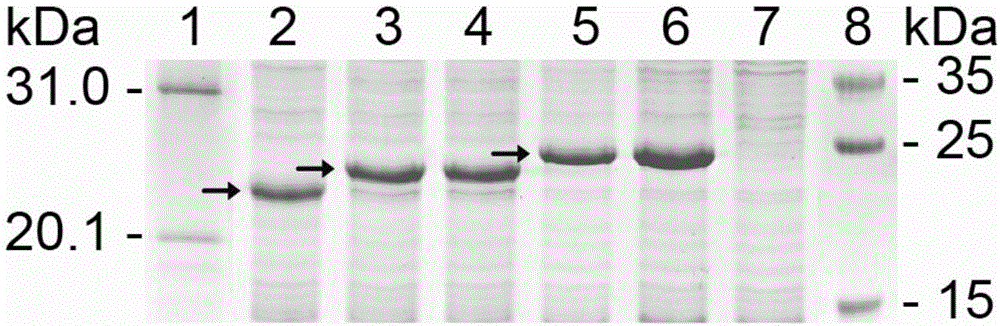
图1显示了目的短肽融合蛋白特异表达的sds-page图。泳道1/8,蛋白分子量标准;泳道2,对照gst188蛋白;泳道3-4,质粒pxxgst-3表达的8肽融合蛋白;泳道5-6,表达的18肽融合蛋白;泳道7,自连pxxgst-3克隆诱导总蛋白阴性对照。箭头分别指示对照蛋白、8和18肽融合蛋白。
图2为热诱导gst188-短肽表达质粒pxxgst-3示意图。
图3为短肽融合表达质粒pxxgst-1示意图。
图4为双酶切pxxgst-3质粒的0.7%琼脂糖凝胶电泳图。泳道1,dna分子量标准;泳道2,用bamhi单酶切的4511bp产物对照;泳道3,用bamhi和sali双酶切产生的4190bp质粒载体。
图5用兔抗血清的系列8肽免疫印迹鉴定(a)和反应性8肽的表位最小基序分析(b)。图5a:泳道1-11,以gst188蛋白为载体表达的系列8肽(p1-p11)融合蛋白。图5b:阴影指示印迹阳性的两个8肽(p6和p7)融合蛋白和它们被识别的共有氨基酸序列。
具体实施方式
本发明人经过广泛而深入的研究,首次开发了一种用于构建和筛选短肽重组克隆的表达盒及含有该表达盒的载体,所述表达盒具有优化的式i所示结构。实验结果表明,本发明所提供的含所述表达盒的载体(如pxxgst-3载体)可以无限接近100%地避免由不完全酶切导致自连克隆的生成,通过聚丙烯酰胺凝胶电泳的重组克隆筛选不再需要设置对照蛋白,同时不像pxxgst-1自连克隆表达与8肽融合蛋白分子量接近的gst融合蛋白(如gst192蛋白),本发明载体不表达出现在细菌总蛋白弱抗原区(图1泳道1蛋白分子量标准指示的约21-30kda特定范围)内类似gst192蛋白(介于泳道2的gst188对照蛋白和泳道3gst188-8肽融合表达的gst196蛋白之间)的判断干扰性蛋白,再无将之误判为重组克隆的可能性,为此可大大减少工作量,避免浪费测序费用和时间,从而能高效构建和更方便筛选短肽重组克隆,并使短肽生物合成、靶蛋白抗原表位组扫描作图和表位鉴定方面的应用操作更简便、经济、高效和准确。在此符合设计理念实现预想目标的基础上形成了本发明。
在大多数双酶切反应中,或多或少会存在少量由不完全酶切(单酶切)导致的线性质粒载体(造成其自连克隆的原因),因其与目的双酶切载体分子量十分接近(两者大小仅是6bp差异),而不可能通过琼脂糖电泳将它们分离开。因此,用pxxgst-1载体构建和筛选8/18肽重组克隆存在的第一个缺陷是无法很好避免pxxgst-1载体自连克隆的生成。同时,又因为其自连克隆表达的非重组gst192蛋白位于细菌蛋白弱抗原区,且与对照gst188蛋白的分子量也存在0.5kda的差异,所以带来第二个缺陷是很容易将自连克隆误判为8肽重组克隆,乃至造成不必要的dna测序验证花费和时间。解决这些问题的方向显而易见,即:第一,作为构建用质粒载体要设法消除不完全双酶切形成的4196bp产物,以尽可能避免其自连克隆生成;第二,针对无法回避极少量’漏网’分子存在的事实,必须使自连克隆表达的类似gst192非重组蛋白不在细菌蛋白弱抗原区范围内,以使重组克隆的筛选不再有设置任何对照蛋白如gst188蛋白的必要。
基于以上认知和思考,最终开发了本发明的表达盒及含有该表达盒的载体。通过本发明载体构建的短肽重组克隆,仍能恒定高表达位于弱抗原区的8/18肽融合蛋白,无需纯化目的短肽就能进行免疫印迹鉴定。同时由于本发明载体中第二核酸元件的存在,1)可使双酶切完全和不完全酶切产物(4190bp和4511bp)能在琼脂糖凝胶电泳中彻底分开,进而方便纯化回收前者4190bp目的载体;2)使可能表达的gst188-抗干扰多肽融合蛋白完全脱离细菌蛋白弱抗原区(在上限为30kda外),因该区域内不再存在由pxxgst-3自连克隆表达的非特异蛋白,也即不再存在由非特异蛋白带来对判别8肽或18肽融合表达克隆的判定干扰,所以它们的凝胶电泳重组克隆判定不再需要任何蛋白对照。
术语
第二核酸元件
如本文所用,术语“z2”、“第二核酸元件”可互换使用,均指本发明第一方面所述表达盒中位于s1和s2之间的核酸序列,可用于通过琼脂糖电泳区分单酶切产物和双酶切产物,乃至能纯化后者载体并进而提高目的短肽克隆构建的成功率。又,设计其表达的抗干扰多肽分子量在10kda以上,可确保即便表达的融合蛋白(gst188-抗干扰多肽)的蛋白条带不在细菌蛋白弱抗原区,因此不存在非特异性融合蛋白(gst188-抗干扰多肽)的干扰,因而凝胶电泳无需设置对照蛋白,从而便于筛选短肽重组克隆和认定由极个别‘漏网’不完全酶切产物导致的自连克隆(弱抗原区内无特异蛋白表达即为判定标准)。
一个优选例中,所述z2为e105基因,序列如seqidno.:1中第586-900位所示。
抗干扰多肽
如本文所用,“抗干扰多肽”或“抗干扰多肽e105”指由z2第二核酸元件编码的多肽,可被目的短肽替代,以表达gst188-短肽融合蛋白,其真正用途是因为其编码基因长度足以能分开双酶切和不完全酶切质粒载体,进而通过琼脂糖凝胶电泳回收前者目的酶切载体而排除自连克隆生成。同时其多肽分子量能将表达的gst188-多肽融合蛋白排除在细菌蛋白弱抗原区外,进而方便通过聚丙烯酰胺凝胶电泳筛选短肽重组克隆。一个优选例中,所述抗干扰多肽为e105蛋白,序列如seqidno.:3中第191-295位所示。
gst192蛋白
如本文所用,“gst192蛋白”、“gst192非重组蛋白”或“gst192多肽”,为长188个氨基酸的截短gst蛋白和由taa终止密码子前bamhi和sali酶切位点(12个bp)编码的4个氨基酸短肽共同构成的蛋白,总计含192个氨基酸残基。
gst196蛋白
如本文所用,“gst196蛋白”特指8肽与截短gst188蛋白载体共表达的融合蛋白。
表达盒
如本文所用,术语“表达盒”、“本发明表达盒”均指本发明第一方面所述的表达盒,其从5’到3’端具有式i所示的结构:
z1-s1-z2-s2-z3-z4式(i)
式中,z1为第一核酸元件,所述第一核酸元件编码截短gst188多肽;
s1为第一酶切位点;
z2为第二核酸元件,所述第二核酸元件编码抗干扰多肽;
s2为第二酶切位点;
z3为无或密码子;
z4为无或polya;
其中,所述z2具有以下特征:(a)长度为3m个核苷酸,其中m为70-140的正整数(较佳地80-130,更佳地90-120,最佳地100-110);(b)不含终止密码子;和(c)不含s1和s2。在一优选例中,所述表达盒的序列如seqidno.:1中第16-906位所示。
载体
载体具体分蛋白载体(如前述的gst188蛋白融合表达载体)和质粒载体(如pxxgst-1和pxxgst-3)。如本文所用,术语“本发明质粒”、“本发明载体”均指本发明第二方面所述的载体。本发明载体能高效构建和更方便筛选短肽重组克隆,从而准确而高效地用于短肽生物合成和/或靶抗原表位鉴定。利用本发明载体构建的短肽重组克隆仍能恒定高表达位于弱抗原区的8/18肽融合蛋白,无需纯化目的短肽就能进行免疫印迹鉴定;同时其双酶切完全和不完全酶切产物能在琼脂糖凝胶电泳中彻底分开,通过割胶回收可避免其自连克隆出现,进而提高短肽重组克隆的效率;另一突出优点是编码的蛋白分子量使可能表达的抗干扰多肽与gst188的融合蛋白脱离细菌蛋白弱抗原区(上限为30kda)以外,不需要设置蛋白对照。
在一优选例中,所述载体为如图2所示的质粒pxxgst-3。在一优选例中,所述载体的序列如seqidno.:1所示。
在本发明的一个具体实施方式中,提供了具有gst188-bamhi-e105-sali表达盒的pxxgst-3质粒载体(图2)。该质粒通过双酶切去除e105基因片段并将编码短肽dna片段重组插入载体,能恒定高表达位于细菌蛋白谱弱抗原区的系列8/18肽,无需纯化目的短肽就能进行免疫印迹鉴定。e105基因长度为315bp,连同两个限制酶之间的6bp(合计321bp)。酶切反应后可产生大小明显不同的4190bp双酶切片段、切除的321bpe105基因片段(包括了切除的两个限制酶之间的6bp)和少量不完全酶切产生的4511bp片段,足以使双酶切完全的载体和不完全酶切的少量产物在0.7%琼脂糖凝胶电泳(50v90min或100v60min)中被分离开来。因而可通过割胶回收试剂盒纯化回收双酶切产物,显然就可排除4511bp载体,避免pxxgst-3自连克隆出现,从而大大提高构建不同短肽重组克隆的成功率。又因为它编码105aa的e105蛋白,理论分子量约为11.5kda,所以即便pxxgst-3自连质粒表达gst188-e105蛋白(22kda+11.5kda),其蛋白条带也在弱抗原区上限的30kda外。如图1泳道7所示,在弱抗原区不存在有碍判别8肽融合蛋白gst196的条带(如类似pxxgst-1自连克隆所表达gst192蛋白),所以筛选确定8/18肽重组克隆不再需要在凝胶电泳中设置包括gst188在内的任何蛋白对照,也完全排除了误将其自连克隆判断为’可能是’重组克隆的可能性。换言之,只要看到如图1泳道3-6中所示特异8/18肽融合蛋白条带的存在,就可确定它(们)的各自诱导菌都是短肽重组克隆,反之未见特异表达条带的,即是偶尔会出现的质粒自连克隆(尽管其出现的概率已非常小)。
pxxgst-3质粒的构建,具体步骤如下:
(1)委托上海捷瑞生物技术服务公司化学合成5’-和3’-端带bamhi和sali酶切位点粘性末端的e105基因,并重组插入发明人提供的热诱导psy621质粒;
(2)用bamhi和sali限制性酶双酶切psy621质粒后,再用qia快速凝胶抽提试剂盒回收两头带bamhi和sali粘性末端的e105基因片段;
(3)利用dna重组技术将回收的e105基因插入用bamhi和sali双酶切的pxxgst-1质粒,继而将获得的pxxgst-1/e105重组质粒转化大肠杆菌宿主菌,在加氨苄青霉素(amp)的肉汤培养基(lb)平板上挑取重组克隆,进行插入基因片段的dna测序。经测序验证的新改良质粒命名为pxxgst-3。
在另一优选例中,所述载体(自连克隆)表达的多肽氨基酸序列如seqidno.:3所示。该多肽长297aa,其中seqidno.:3中的第1-188位为gst188氨基酸序列,第189-190位为bamhi酶切位点编码的氨基酸序列,第191-295位为e105基因编码的抗干扰多肽的氨基酸序列,第296-297位为sali酶切位点编码的氨基酸序列。
m-s-p-i-l-g-y-w-k-i-k-g-l-v-q-p-t-r-l-l-l-e-v-l-e-e-k-y-e-e-h-l-y-e-r-d-e-g-d-k-w-r-n-k-k-f-e-l-g-l-e-f-p-n-l-p-y-y-i-d-g-d-v-k-l-t-q-s-m-a-i-i-r-y-i-a-d-k-h-n-m-l-g-g-c-p-k-e-r-a-e-i-s-m-l-e-g-a-v-l-d-i-r-y-g-v-s-r-i-a-y-s-k-d-f-e-t-l-k-v-d-f-l-s-k-l-p-e-m-l-k-m-f-e-d-r-l-c-h-k-t-y-l-n-g-d-h-v-t-h-p-d-f-m-l-y-d-a-l-d-v-v-l-y-m-d-p-m-c-l-d-a-f-p-k-l-v-c-f-k-k-r-i-e-a-i-p-q-g-s-m-h-g-p-k-a-t-l-q-d-i-v-l-h-l-e-p-q-n-e-i-p-v-d-l-l-c-h-e-q-l-s-d-s-e-e-e-n-d-e-i-d-g-v-n-h-q-h-l-p-a-r-r-a-e-p-q-r-h-t-m-l-c-m-c-c-k-c-e-a-r-i-e-l-v-v-e-s-s-a-d-d-l-r-a-f-q-q-l-f-l-n-t-l-s-f-v-c-p-w-c-a-s-q-q-v-d(seqidno.:3)
短肽融合蛋白及其表达载体
如本文所用,术语“短肽融合蛋白”、“8/18肽融合蛋白”可互换使用,指具有式ii所示的融合蛋白
gst188-a式(ii)
式中,gst188为截短由188残基组成的gst188多肽载体,a元件为长度为希望表达的6-20个氨基酸短肽。
利用本发明提供的载体,在bamh1和sal1间切除z2元件并重组插入编码目的蛋白的外源基因,可热诱导直接表达与gst载体融合的任一目的短肽。
如本文所用,术语“短肽融合蛋白表达载体”、“短肽重组克隆”、“短肽生物合成质粒”指能够表达式ii所示的短肽融合蛋白的载体。
本发明还提供一种构建短肽融合蛋白表达载体的方法,包括以下步骤:
(1)提供第一核酸分子和第二核酸分子,其中,所述第一核酸分子为本发明第二方面所述的载体经针对s1和s2酶切位点的酶消化后得到的大片段,所述第二核酸分子为两端带有s1和s2酶切位点的短肽编码序列;
(2)连接所述第一核酸分子和第二核酸分子,得到表达短肽融合蛋白的载体。
在一优选例中,用目的短肽的编码核酸替换z2元件。其中,所述“替换”包括利用将所述z2切除,并将短肽的编码核酸插入s1和s2之间。
抗原表位扫描
本发明提供了一种基于本发明所提供的载体,可以方便地进行抗原表位扫描鉴定的方法。如本发明第四方面所述的靶抗原表位鉴定方法,包括以下步骤:
(a)提供第一核酸分子和第二核酸分子,其中,所述第一核酸分子为本发明第二方面所述的载体经针对s1和s2酶切位点的酶消化后得到的大片段,所述第二核酸分子为两端带有s1和s2酶切位点的短肽编码序列;
(b)连接所述第一核酸分子和第二核酸分子,得到表达短肽融合蛋白的载体;
(c)培养含所述短肽融合蛋白表达载体的宿主细胞,并诱导所述宿主细胞表达式ii所示的融合蛋白
gst188-a式(ii)
式中,gst188为截短gst蛋白的长188个氨基酸多肽,a元件为长度为6-20个氨基酸的短肽;
(d)将表达的式ii所示的融合蛋白与抗体混合,观察抗体是否与式ii所述的融合蛋白发生抗体-抗原反应;其中,如果抗体与式ii所述的融合蛋白发生抗体-抗原反应,那么就表示所述融合蛋白中的a元件是一靶蛋白的抗原性肽。在另一优选例中,所述a元件来自同一个蛋白。在另一优选例中,a元件为来自同一抗原性肽。在另一优选例中,所述a元件为相互重叠7个残基的系列8肽。
在一优选例中,本发明也提供了pxxgst-3质粒的应用实例,即针对一已知huzp4蛋白18聚抗原性肽,再次构建一组覆盖其全长序列相互重叠7个残基的系列8肽重组克隆,然后用兔抗重组huzp4蛋白抗血清进行抗原抗体印迹反应,进而鉴定出其抗原性肽中一精细线性表位基序。
在另一优选例中,利用pxxgst-3质粒表达序列部分重叠的系列gst188-8肽融合蛋白的具体步骤如下:
(1)用bamhi和sali限制性酶双酶切1μg量pxxgst-3质粒,继而以bamhi或sali单酶切的pxxgst-3质粒作为参照物(双酶切反应中经由不完全酶切(单酶切)形成的4511bp产物量少,所以需要另外设置作为割胶回收4190bp质粒载体的平行参照物),将100μl酶切反应液跑0.7%琼脂糖凝胶电泳(稳压50v90min),割胶后用快速凝胶抽提试剂盒回收目的双酶切4190bp载体dna片段,放-20℃备用;
(2)基于先前用兔抗天然猪zp蛋白(包含zp4蛋白组份)抗血清鉴定过精细表位的已知huzp4抗原性肽(aa308-325),设计覆盖其18肽全长序列相互重叠7个残基的一组11个系列8肽融合蛋白;然后外送化学合成它们的系列8肽编码正负链dna片段;在将它们分别退火复性后,分别重组插入第四步纯化获得的双酶切完全pxxgst-3质粒载体;
(3)将上述连接液分别转化大肠杆菌bl21宿主菌,在过夜培养的lb平板上挑取重组克隆转接3mllb培养液中;过夜培养后转接3ml新鲜lb培养液继续37℃培养;在菌体浓度od600值达到0.6左右时,进行42℃热诱导表达4h;
(4)4℃离心收集诱导菌总蛋白,进行sds-page分析,确定凝胶上细菌总蛋白弱抗原区内显示特异表达条带的诱导菌为重组克隆,并挑取它们的诱导菌克隆外送进行dna测序验证(确定各8肽编码dna片段有无核苷酸碱基序列的合成错误);
(5)最后用兔抗重组huzp4c(aa227-463)抗血清进行8肽免疫印迹鉴定,完成目的抗原性肽的精细表位鉴定,同时确认pxxgst-3用于短肽生物合成和抗原表位鉴定的适用性。
本发明的主要优点
本发明的载体具有以下优点:
1)由于第二核酸元件的存在,能通过割胶高纯度回收其双酶切完全质粒载体,进而通过消除未完全酶切的载体,避免其自连克隆出现而提高短肽重组克隆效率;
2)由于抗干扰多肽与gst188融合的蛋白分子量大于30kda,因此不再需要gst188对照蛋白,而能更方便通过sds-page电泳方法筛选18肽尤其是8肽重组克隆。总之,筛选鉴别目的短肽重组克隆更简便经济、结果更准确可靠;
3)不再存在将极少数‘漏网’不完全酶切载体形成的自连克隆误判为重组克隆的可能性,因为在其细菌蛋白弱抗原区内无任何其他非特异表达的明显蛋白条带存在。
4)用热诱导替代价高的异丙基-d-硫代半乳糖苷(iptg)诱导表达,既避免了易干扰特异目的表达短肽判断的阴性对照菌“泄露表达”现象,也更方便操作且经济;
5)能恒定高表达位于细菌蛋白谱弱抗原区的系列8/18肽,无需纯化目的短肽就能直接使用诱导菌总蛋白进行免疫印迹鉴定。
本发明的质粒载体能高效构建和更方便筛选短肽重组克隆,从而使短肽生物合成、靶蛋白抗原表位组扫描作图和抗体识别表位鉴定方面的应用操作更简便、经济和高效率。
以上专门用于抗原表位鉴定的短肽生物合成质粒构建,及其用之以物种不同但同源的蛋白抗血清鉴定它们之间100%保守的抗原性肽精细表位的应用,显然为今后蛋白抗原表位鉴定及其表位组解码研究提供了以使用本发明质粒为载体的更方便、经济和高效的gst188-短肽生物方法,为其更广泛的应用奠定了坚实基础。
尽管本发明描述了专门用于抗原表位鉴定和表位组作图的pxxgst-3质粒构建策略或要点,然而有一点对于相关领域研究技术人员来说是显而易见的,即在不脱离本发明的精神和范围的前提下,可对本发明制品作各种变化改动,如改变特定z1元件或z2元件的基因核苷酸片段长短或双酶切位点等。因此,所附权利要求覆盖了所有这些本发明范围内的变动。
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件,例如sambrook等人编著的分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,2001)中所述的条件,或按照制造厂商所建议的条件。
材料和方法
1.热诱导融合表达pxxgst-1质粒和psy621克隆载体质粒由发明人之前构建[1.xuwx,heyp,tanghp,jiaxf,jicn,gush,sunxx,xiey.minimalmotifmappingofaknownepitopeonhumanzonapellucidaprotein-1usingpeptidebiosynthesisstrategy.jreprodimmunol,2009,81:9–16;2.沈芸等.大肠杆菌高效表达载体psy621的构建.复旦学报(自然科学版),2000;39(3):313–317],大肠杆菌tg1和bl21(de3)菌株购自上海生工有限公司。
2.兔抗重组huzp4c234-463蛋白抗血清,其制备方法参考发明人之前huzp3b抗血清制备研究(宋力雯等.人卵透明带蛋白zp3a和zp3b肽段的免疫原性及其抗血清体外抑制人精子-半透明带结合.生理学报,57(6):682-688,2005)。
3.限制性内切酶bamhi和sali以及t4dna连接酶购自大连takara生物技术公司,预染蛋白质分子量标准购自华美生物工程公司,氨苄青霉素(amp)、辣根过氧化酶标记羊抗兔二抗(igg/hrp)等购自上海生工公司,ecl化学发光试剂盒购自ge公司。
4.质粒抽提试剂盒和酶切目的基因片段凝胶回收试剂盒购自德国qiagen公司。
5.第二核酸元件e105基因由上海捷瑞生物技术服务公司合成,并通过其两端的bamhi和sali位点重组插入psy621质粒的克隆位点,转化形成psy621/e105克隆菌。其中,第二核酸元件e105的长度为315bp,序列如seqidno.:1中第586-900位所示。
实施例1pxxgst-3质粒的制备
1.获得第二核酸元件e105基因片段
将捷瑞公司技术服务提供的psy621/e105克隆菌接种于3mllb肉汤培养基中,于37℃培养过夜后离心收集菌体,用专门试剂盒抽提质粒,再用bamhi和sali限制性酶进行质粒双酶切产生e105基因片段。将酶切后的反应液进行1.2%琼脂糖凝胶电泳,40分钟后割取带两端限制性酶粘性末端的e105基因小片段(321bp)凝胶,用快速胶回收试剂盒纯化提取e105基因片段。
2.pxxgst-3质粒构建
在0.5ml管的100μl酶连接反应体系中,按照大约1:10等分子量比例将回收的约0.1μg的e105基因片段与用bamhi和sali限制酶双酶切的1μgpxxgst-1质粒混合,加入t4dna连接酶后于4℃连接过夜,翌日将连接反应液转化感受态tg1宿主菌,并涂布加入amp的lb固体平板上,于37℃恒温培养过夜。在lb平板上挑取若干菌落分别转接3mllb液体培养基中培养过夜;用质粒纯化试剂盒提取质粒。随后进行bamhi和sali限制酶双酶切反应验证gst188-e105重组克隆,凝胶电泳后凝胶上显示321bp小片段的质粒,即为插入e105基因的pxxgst-3质粒。(注:热诱导的pxxgst-3克隆菌总蛋白在sds-page凝胶上未显示gst188-e105融合蛋白条带,因此只能通过此步重组质粒双酶切反应予以验证gst188-e105重组克隆。)
pxxgst-3质粒的示意图如图2所示,序列如seqidno.1所示。
实施例2利用pxxgst-3质粒对一个huzp4蛋白18聚抗原性肽的表位鉴定
选择已被重新命名为huzp4的一已知huzp1蛋白18聚(petqpgpltlelqiakdk)抗原性肽308-325(xuwx,etal.minimalmotifmappingofaknownepitopeonhumanzonapellucidaprotein-1usingpeptidebiosynthesisstrategy.jreprodimmunol,2009,81:9-16)作为靶抗原,用pxxgst-3质粒构建覆盖其全长序列相互重叠7个残基的11个系列8肽融合蛋白重组克隆,然后用兔抗重组huzp4c227-463抗血清对表达的系列8肽进行免疫印迹鉴定,揭示其抗体能识别结合的表位最小基序。
第一步:用bamhi和sali双酶切pxxgst-3质粒;将酶切后的反应液进行0.7%琼脂糖凝胶(适合分离800-12,000bp核苷酸片段)电泳50v90分钟,使酶切所产生的双酶切4190bp载体、321bp的e105基因片段和少量4511bp单酶切产物分离开;在紫外光灯下切割目的4190bp条带凝胶(可在凝胶电泳时另外设置单酶切pxxgst-3产物参照物),再用凝胶回收试剂盒提取pxxgst-3质粒载体,放置4℃备用。
结果如图4所示,单酶切(泳道2)和双酶切(泳道3)后的载体大小分别为4511bp和4190bp,差异明显,条带完全分开,可明显区分完全双酶切和未完全双酶切(单酶切)的载体。因此本发明pxxgst-3载体可以简单地通过凝胶电泳将未完全双酶切的载体分离,回收得到完全双酶切的载体,因此可排除不完全酶切质粒载体而大幅度减少其自连克隆的生成。
第二步:设计含bamhi和sali酶粘性末端的上述目的抗原性肽(正链为5’-gatcc——taag-3’;负链为5’-tcgactta———g-3’,正负链中也分别包含了sali位点前设置的以下划线显示的taa终止密码子碱基)11个系列8肽编码基因片段,合成它们的22条正负链片段。
第三步:按生物技术公司提供的dna片段合成报告单数据,用ddh2o将1或2od值的互补正负链片段溶解成20μmol/μl储存液;各取10μl配对储存液和20μlddh2o于1.5ml管中,进行94℃水浴加热5min,然后自然降温至25℃室温的两两退火复性;在15μl反应体系中加入2μl退火片段、1μl约200ng/μl双酶切pxxgst-3载体、1μlt4dna连接酶和其1.5μl连接酶缓冲液,于16℃保温瓶中过夜,完成短肽编码片段与载体的连接反应。翌日将连接液转化大肠杆菌感受态bl21(de3)宿主菌,并涂布于含amp的lb平板上,37℃恒温箱培养过夜;第二天挑取lb平板上的单克隆转接加amp的3ml新鲜lb液体培养基管中,30℃振荡培养过夜;翌日晨按1:50比例再次分别转接加amp的3ml新鲜lb培养液中振荡培养,待菌体浓度od值达到0.5-0.6时将培养温度上调至42℃,进行热诱导表达短肽融合蛋白4h,;最后离心收集的菌体加入凝胶电泳上样裂解液,煮沸5min后于4℃储存备用。
第四步:用上述诱导菌总蛋白裂解液用进行不加gst188对照蛋白的15%sds-page分析,待溴酚蓝走到接近凝胶底部时,结束电泳,取出凝胶先后进行考马斯亮蓝染色和脱色。如图1泳道3-6所示,在对应21-30kda范围的细菌蛋白谱弱抗原区内发现明显表达条带,就可确定它(们)的母体菌为重组8/18肽重组克隆(因为从泳道7中可知晓pxxgst-3自连克隆总蛋白在该区域不存在明显表达条带);将确定的各重组克隆外送进行dna测序,验证所委托化学合成的8肽编码片段是否存在碱基突变和/或合成片段编号错误等。
第五步:重新诱导经sds-page分析和dna测序确定的8肽重组克隆菌,分别各取10-15μl诱导菌总蛋白上样裂解液同时走15%sds-page,于电泳结束后一块凝胶如图1那样用于考马斯亮蓝染色,一块用于将凝胶上蛋白电转移(100ma1.5-2h)至0.22μm硝酸纤维素膜,转印膜用丽春红染色10min,在染色上的短肽融合蛋白条带处打孔作标记,最后用水冲洗掉膜上丽春红着色。转印膜用生理盐水(pbs)缓冲液反复洗涤四次,用5%脱脂奶粉于4℃封闭过夜,再用pbs缓冲液洗涤四次,在8-10ml印迹反应液中加入30μl兔抗重组huzp4c抗血清作为(一抗),室温反应2h和用pbs缓冲液洗膜后,加入10μl羊抗兔igg/hrp二抗,继续室温反应1h,最后用ecl发光试剂盒显色(图5a)。
结果如图5所示。图5a为用兔抗血清的8肽免疫印迹鉴定,在各目的8肽融合蛋白免疫印迹中,仅泳道6和7中有印迹条带,表明这两个8肽(p6和p7)印迹阳性,含有靶抗原的表位。反应性8肽的表位最小基序分析如图5b中阴影指示,即为它们被识别的共有氨基酸序列(pltlelq)。就使用短肽生物合成法的精细表位鉴定而言,与之前用兔抗天然猪zp(含zp4)蛋白抗血清确定的精细表位pltlel相比,用兔抗重组人zp4抗血清确定的表位基序(pltlelq)仅是在c端多了1个q残基差异。本实例表明本发明的pxxgst-3载体用于短肽生物合成的可靠性,和体现在避免质粒载体自连和筛选目的重组克隆更便利等优点。同时也表明同一物种(兔)免疫系统有可能识别分别来自猪和人同源抗原的相同抗原性位点,无论是来自天然蛋白,还是重组蛋白或其大片段抗原。
本实验通过在双酶切位点之间插入一抗干扰多肽,构建形成gst188-bamhi-e105融合基因单元结构的pxxgst-3载体。该载体具有以下显著特点:
1)通过双酶切去除e105基因并将短肽编码dna片段重组插入载体,可恒定任意表达且位于细菌蛋白弱抗原区的系列或任一8肽(gst188-8肽)融合蛋白;
2)可基本上排除pxxgst-3非重组的自连克隆出现。因为在其bamhi和sali位点之间插入一段315bp的e105基因片段,足以排除由不完全酶切导致的4511bp产物,使得双酶切产生的4190bp的pxxgst-3载体能通过琼脂糖凝胶电泳予以回收,从而大幅度减少其pxxgst-3自连克隆的生成。
3)筛选短肽重组克隆更方便,也就是省去了在凝胶电泳中设置gst188对照蛋白步骤。因为即便存在极少数漏网不完全酶切产物分子,它们的自连克隆也不表达gst188-e105融合蛋白,即便表达其融合蛋白也不在弱抗原区,因而在凝胶上的弱抗原区不会出现除细菌自身固有少量蛋白外的其他类似gst192蛋白条带。也就是说,只要在凝胶上的特定区域(21-30kda)内观察到特异蛋白条带,就可确定其诱导菌为8/18肽重组克隆,反之认定为自连克隆。
4)e105蛋白分子量约为11.5kda,可使表达的gst188(约22kda)-e105融合蛋白位于细菌蛋白弱抗原区的30kda上限之外,因而如图1所示,可因此无需用对照蛋白比对,只要该区域内出现类似泳道3-6中的8/18肽条带,就可判定它们各自的诱导菌为重组克隆,反之无明显条带(泳道7)出现,即为自连克隆。
对比例
bamhi和sali酶切位点之间不含第二核酸元件e105,其余序列与pxxgst-3质粒完全相同的热诱导融合表达pxxgst-1质粒(图3),序列如seqidno.2所示,共4196bp。
利用pxxgst-1载体构建短肽融合蛋白表达载体时,用bamhi和sali两个限制性酶双酶切,仅切除它们之间6bp,切除的长度太短而无法通过割胶回收4190bp载体。因无法进行分离纯化,由不完全酶切带来的少量4196bp载体自连,导致假阳性克隆产生几率高。因此利用pxxgst-1载体构建短肽融合蛋白表达载体时无法避免自连克隆的生成,干扰影响重组克隆构建,效率低,工作量增加,耗费不必要的测序费用和时间。
同时,其自连克隆表达的gst192蛋白与8肽重组克隆表达的gst196融合蛋白(gst188+8aa)仅少4aa,即两者分子量仅相差0.5kda(此估算依据20个氨基酸残基平均分子量110kda),因此为方便确定8肽的特异表达而需要设置更容易辨别两者的分子量相差1kda的对照gst188蛋白(图1中泳道2),并且也存在将自连克隆误判为重组克隆的可能性,因为gst192蛋白比对照蛋白gst188大0.5kda。
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
序列表
<110>上海市计划生育科学研究所
<120>短肽重组克隆构建筛选的载体及其应用
<130>p2017-1657
<160>3
<170>patentinversion3.5
<210>1
<211>4511
<212>dna
<213>人工序列(artificialsequence)
<400>1
ttaggagaagaattcatgtcccctatactaggttattggaaaattaagggccttgtgcaa60
cccactcgacttcttttggaatatcttgaagaaaaatatgaagagcatttgtatgagcgc120
gatgaaggtgataaatggcgaaacaaaaagtttgaattgggtttggagtttcccaatctt180
ccttattatattgatggtgatgttaaattaacacagtctatggccatcatacgttatata240
gctgacaagcacaacatgttgggtggttgtccaaaagagcgtgcagagatttcaatgctt300
gaaggagcggttttggatattagatacggtgtttcgagaattgcatatagtaaagacttt360
gaaactctcaaagttgattttcttagcaagctacctgaaatgctgaaaatgttcgaagat420
cgtttatgtcataaaacatatttaaatggtgatcatgtaacccatcctgacttcatgttg480
tatgacgctcttgatgttgttttatacatggacccaatgtgcctggatgcgttcccaaaa540
ttagtttgttttaaaaaacgtattgaagctatcccacaaggatccatgcatggacctaag600
gcaacattgcaagacattgtattgcatttagagccccaaaatgaaattccggttgacctt660
ctatgtcacgagcaattaagcgactcagaggaagaaaacgatgaaatagatggagttaat720
catcaacatttaccagcccgacgagccgaaccacaacgtcacacaatgttgtgtatgtgt780
tgtaagtgtgaagccagaattgagctagtagtagaaagctcagcagacgaccttcgagca840
ttccagcagctgtttctgaacaccctgtcctttgtgtgtccgtggtgtgcatcccagcag900
gtcgactaatagctgcagccaagcttggctgttttggcggatgagagaagattttcagcc960
tgatacagattaaatcagaacgcagaagcggtctgataaaacagaatttgcctggcggca1020
gtagcgcggtggtcccacctgaccccatgccgaactcagaagtgaaacgccgtagcgccg1080
atggtagtgtggggtctccccatgcgagagtagggaactgccaggcatcaaataaaacga1140
aaggctcagtcgaaagactgggcctttcgttttatctgttgtttgtcggtgaacgctctc1200
ctgagtaggacaaatccgccgggagcggatttgaacgttgcgaagcaacggcccggaggg1260
tggcgggcaggacgcccgccataaactgccaggcatcaaattaagcagaaggccatcctg1320
acggatggcctttttgcgtttctacaaactcttttgtttatttttctaaatacattcaaa1380
tatgtatccgctcatgagacaataaccctgataaatgcttcaataatattgaaaaaggaa1440
gagtatgagtattcaacatttccgtgtcgcccttattcccttttttgcggcattttgcct1500
tcctgtttttgctcacccagaaacgctggtgaaagtaaaagatgctgaagatcagttggg1560
tgcacgagtgggttacatcgaactggatctcaacagcggtaagatccttgagagttttcg1620
ccccgaagaacgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtatt1680
atcccgtgttgacgccgggcaagagcaactcggtcgccgcatacactattctcagaatga1740
cttggttgagtactcaccagtcacagaaaagcatcttacggatggcatgacagtaagaga1800
attatgcagtgctgccataaccatgagtgataacactgcggccaacttacttctgacaac1860
gatcggaggaccgaaggagctaaccgcttttttgcacaacatgggggatcatgtaactcg1920
ccttgatcgttgggaaccggagctgaatgaagccataccaaacgacgagcgtgacaccac1980
gatgcctgtagcaatggcaacaacgttgcgcaaactattaactggcgaactacttactct2040
agcttcccggcaacaattaatagactggatggaggcggataaagttgcaggaccacttct2100
gcgctcggcccttccggctggctggtttattgctgataaatctggagccggtgagcgtgg2160
gtctcgcggtatcattgcagcactggggccagatggtaagccctcccgtatcgtagttat2220
ctacacgacggggagtcaggcaactatggatgaacgaaatagacagatcgctgagatagg2280
tgcctcactgattaagcattggtaactgtcagaccaagtttactcatatatactttagat2340
tgatttaaaacttcatttttaatttaaaaggatctaggtgaagatcctttttgataatct2400
catgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaa2460
gatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaa2520
aaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttcc2580
gaaggtaactggcttcagcagagcgcagataccaaatactgttcttctagtgtagccgta2640
gttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcct2700
gttaccagtggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacg2760
atagttaccggataaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccag2820
cttggagcgaacgacctacaccgaactgagatacctacagcgtgagctatgagaaagcgc2880
cacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacagg2940
agagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtt3000
tcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatg3060
gaaaaacgccagcaacgcggcctttttacggttcctggccttttgctggcccttttgctc3120
acatgttctttcctgcgttatcccctgattctgtggataaccgtattaccgcctttgagt3180
gagctgataccgctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaag3240
cggaagagcgcttatctttccctttatttttgctgcggtaagtcgcataaaaaccattct3300
tcataattcaatccatttactatgttatgttctgaggggagtgaaaattcccctaattcg3360
atgaagattcttgctcaattgttatcagctatgcgccgaccagaacaccttgccgatcag3420
ccaaacgtctcttcaggccactgactagcgataactttccccacaacggaacaactctca3480
ttgcatgggatcattgggtactgtgggtttagtggttgtaaaaacacctgaccgctatcc3540
ctgatcagtttcttgaaggtaaactcatcacccccaagtctggctatgcagaaatcacct3600
ggctcaacagcctgctcagggtcaacgagaattaacattccgtcaggaaagcttggcttg3660
gagcctgttggtgcggtcatggaattaccttcaacctcaagccagaatgcagaatcactg3720
gcttttttggttgtgcttacccatctctccgcatcacctttggtaaaggttctaagctta3780
ggtgagaacatccctgcctgaacatgagaaaaaacagggtactcatactcacttctaagt3840
gacggctgcatactaaccgcttcatacatctcgtagatttctctggcgattgaagggcta3900
aattcttcaacgctaactttgagaatttttgtaagcaatgcggcgttataagcatttaat3960
gcattgatgccattaaataaagcaccaacgcctgactgccccatccccatcttgtctgcg4020
acagattcctgggataagccaagttcatttttctttttttcataaattgctttaaggcga4080
cgtgcgtcctcaagctgctcttgtgttaatggtttcttttttgtgctcatacgttaaatc4140
tatcaccgcaagggataaatatctaacaccgtgcgtgttgactattttacctctggcggt4200
gataatggttgcatgtactaaggaggttgtatggaacaacgcataaccctgaaagattat4260
gcaatgcgctttgggcaaaccaagacagctaaagatctctcacctaccaaacaatgcccc4320
cctgcaaaaaataaattcatataaaaaacatacagataaccatctgcggtgataaattat4380
ctctggcggtgttgacataaataccactggcggtgatactgagcacatcagtgacgtcac4440
gctcttaaaaaaagaagggcagcattcaaagcagaaggctttggggtgagagaaaaaaaa4500
aaaaaaaaaaa4511
<210>2
<211>4196
<212>dna
<213>人工序列(artificialsequence)
<400>2
ttaggagaagaattcatgtcccctatactaggttattggaaaattaagggccttgtgcaa60
cccactcgacttcttttggaatatcttgaagaaaaatatgaagagcatttgtatgagcgc120
gatgaaggtgataaatggcgaaacaaaaagtttgaattgggtttggagtttcccaatctt180
ccttattatattgatggtgatgttaaattaacacagtctatggccatcatacgttatata240
gctgacaagcacaacatgttgggtggttgtccaaaagagcgtgcagagatttcaatgctt300
gaaggagcggttttggatattagatacggtgtttcgagaattgcatatagtaaagacttt360
gaaactctcaaagttgattttcttagcaagctacctgaaatgctgaaaatgttcgaagat420
cgtttatgtcataaaacatatttaaatggtgatcatgtaacccatcctgacttcatgttg480
tatgacgctcttgatgttgttttatacatggacccaatgtgcctggatgcgttcccaaaa540
ttagtttgttttaaaaaacgtattgaagctatcccacaaggatccgtcgactaatagctg600
cagccaagcttggctgttttggcggatgagagaagattttcagcctgatacagattaaat660
cagaacgcagaagcggtctgataaaacagaatttgcctggcggcagtagcgcggtggtcc720
cacctgaccccatgccgaactcagaagtgaaacgccgtagcgccgatggtagtgtggggt780
ctccccatgcgagagtagggaactgccaggcatcaaataaaacgaaaggctcagtcgaaa840
gactgggcctttcgttttatctgttgtttgtcggtgaacgctctcctgagtaggacaaat900
ccgccgggagcggatttgaacgttgcgaagcaacggcccggagggtggcgggcaggacgc960
ccgccataaactgccaggcatcaaattaagcagaaggccatcctgacggatggccttttt1020
gcgtttctacaaactcttttgtttatttttctaaatacattcaaatatgtatccgctcat1080
gagacaataaccctgataaatgcttcaataatattgaaaaaggaagagtatgagtattca1140
acatttccgtgtcgcccttattcccttttttgcggcattttgccttcctgtttttgctca1200
cccagaaacgctggtgaaagtaaaagatgctgaagatcagttgggtgcacgagtgggtta1260
catcgaactggatctcaacagcggtaagatccttgagagttttcgccccgaagaacgttt1320
tccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgtgttgacgc1380
cgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggttgagtactc1440
accagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgc1500
cataaccatgagtgataacactgcggccaacttacttctgacaacgatcggaggaccgaa1560
ggagctaaccgcttttttgcacaacatgggggatcatgtaactcgccttgatcgttggga1620
accggagctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgtagcaat1680
ggcaacaacgttgcgcaaactattaactggcgaactacttactctagcttcccggcaaca1740
attaatagactggatggaggcggataaagttgcaggaccacttctgcgctcggcccttcc1800
ggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgcggtatcat1860
tgcagcactggggccagatggtaagccctcccgtatcgtagttatctacacgacggggag1920
tcaggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactgattaa1980
gcattggtaactgtcagaccaagtttactcatatatactttagattgatttaaaacttca2040
tttttaatttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaaatccc2100
ttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttc2160
ttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctacc2220
agcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactggctt2280
cagcagagcgcagataccaaatactgttcttctagtgtagccgtagttaggccaccactt2340
caagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgc2400
tgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataa2460
ggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgac2520
ctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacgcttcccgaagg2580
gagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgaggga2640
gcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgact2700
tgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaa2760
cgcggcctttttacggttcctggccttttgctggcccttttgctcacatgttctttcctg2820
cgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgataccgctc2880
gccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgcttat2940
ctttccctttatttttgctgcggtaagtcgcataaaaaccattcttcataattcaatcca3000
tttactatgttatgttctgaggggagtgaaaattcccctaattcgatgaagattcttgct3060
caattgttatcagctatgcgccgaccagaacaccttgccgatcagccaaacgtctcttca3120
ggccactgactagcgataactttccccacaacggaacaactctcattgcatgggatcatt3180
gggtactgtgggtttagtggttgtaaaaacacctgaccgctatccctgatcagtttcttg3240
aaggtaaactcatcacccccaagtctggctatgcagaaatcacctggctcaacagcctgc3300
tcagggtcaacgagaattaacattccgtcaggaaagcttggcttggagcctgttggtgcg3360
gtcatggaattaccttcaacctcaagccagaatgcagaatcactggcttttttggttgtg3420
cttacccatctctccgcatcacctttggtaaaggttctaagcttaggtgagaacatccct3480
gcctgaacatgagaaaaaacagggtactcatactcacttctaagtgacggctgcatacta3540
accgcttcatacatctcgtagatttctctggcgattgaagggctaaattcttcaacgcta3600
actttgagaatttttgtaagcaatgcggcgttataagcatttaatgcattgatgccatta3660
aataaagcaccaacgcctgactgccccatccccatcttgtctgcgacagattcctgggat3720
aagccaagttcatttttctttttttcataaattgctttaaggcgacgtgcgtcctcaagc3780
tgctcttgtgttaatggtttcttttttgtgctcatacgttaaatctatcaccgcaaggga3840
taaatatctaacaccgtgcgtgttgactattttacctctggcggtgataatggttgcatg3900
tactaaggaggttgtatggaacaacgcataaccctgaaagattatgcaatgcgctttggg3960
caaaccaagacagctaaagatctctcacctaccaaacaatgcccccctgcaaaaaataaa4020
ttcatataaaaaacatacagataaccatctgcggtgataaattatctctggcggtgttga4080
cataaataccactggcggtgatactgagcacatcagtgacgtcacgctcttaaaaaaaga4140
agggcagcattcaaagcagaaggctttggggtgagagaaaaaaaaaaaaaaaaaaa4196
<210>3
<211>297
<212>prt
<213>人工序列(artificialsequence)
<400>3
metserproileleuglytyrtrplysilelysglyleuvalglnpro
151015
thrargleuleuleugluvalleugluglulystyrglugluhisleu
202530
tyrgluargaspgluglyasplystrpargasnlyslysphegluleu
354045
glyleuglupheproasnleuprotyrtyrileaspglyaspvallys
505560
leuthrglnsermetalaileileargtyrilealaasplyshisasn
65707580
metleuglyglycysprolysgluargalagluilesermetleuglu
859095
glyalavalleuaspileargtyrglyvalserargilealatyrser
100105110
lysaspphegluthrleulysvalasppheleuserlysleuproglu
115120125
metleulysmetphegluaspargleucyshislysthrtyrleuasn
130135140
glyasphisvalthrhisproaspphemetleutyraspalaleuasp
145150155160
valvalleutyrmetaspprometcysleuaspalapheprolysleu
165170175
valcysphelyslysargileglualaileproglnglysermethis
180185190
glyprolysalathrleuglnaspilevalleuhisleugluprogln
195200205
asngluileprovalaspleuleucyshisgluglnleuseraspser
210215220
gluglugluasnaspgluileaspglyvalasnhisglnhisleupro
225230235240
alaargargalagluproglnarghisthrmetleucysmetcyscys
245250255
lyscysglualaargilegluleuvalvalgluserseralaaspasp
260265270
leuargalapheglnglnleupheleuasnthrleuserphevalcys
275280285
protrpcysalaserglnglnvalasp
290295
- 还没有人留言评论。精彩留言会获得点赞!