一种绵羊羊睾丸细胞永生化细胞系的构建方法及其应用与流程
本发明属于分子生物学技术领域,具体涉及一种绵羊羊睾丸细胞永生化细胞系的构建方法及其应用。
背景技术:
细胞永生化(immortalization)是指细胞自发或受外界因素影响逃离增殖危机,从而获得无限增殖的能力。自发性的细胞永生化概率很小,对于啮齿类动物为10-5~10-6,人类细胞概率更低,小于10-12。正常动物细胞在体外增殖培养是有限的,该自然极限称为海弗里克极限(hayflicklimit),细胞生长代谢的过程需要复杂的调节网络,一旦该网络发生错误,细胞会跨越自然极限,形成永生化。细胞的分裂增殖过程会要经过衰老期m1期和致死期m2期,跨越这两期的细胞可实现永生化。真核细胞内存在多种抑癌基因如p53、prb、dpc4、brca1等,抑癌基因表达下调与端粒酶的相互作用可能对细胞癌变及永生化产生影响。有学者从人乳腺癌细胞核中分离得到p53蛋白,进一步的试验证明p53蛋白可以体外抑制端粒酶的活性。同时期,人们发现p53基因突变可以导致端粒酶激活,从而促使细胞的永生化。通过不同放射性元素如x射线、核辐射、电离辐射等刺激细胞也可实现细胞永生化,有研究中发现核弹辐射后的人皮细胞具有无限增殖的能力。通过基因转染等技术,可将病毒的基因、原癌基因、端粒酶等导入目的细胞内,或者可以延长细胞传代次数或者实现完全细胞永生化。
端粒是位于真核细胞染色体两端末端,由长度为5~15bp的重复dna序列和周围蛋白质组成的帽状结构。端粒可以保证染色体的完整性,同时调节细胞生命周期,端粒的功能障碍会直接导致细胞增殖停止和凋亡。端粒酶由rna和蛋白质组成,是一种逆转录酶。端粒酶利用自身的rna为模板,可以延伸端粒3’末端或者合成新的端粒dna。因此端粒酶可以维持端粒结构和长度稳定,还可以提高细胞对应急预案和毒力因子的耐受力,在细胞永生化过程发挥重要作用。人端粒酶逆转录酶亚基(humantelomerasereversetranscriptase,htert)首次在人类宫颈癌细胞中发现,同时还发现了端粒酶rna(telomeraserna,tr)和端粒酶相关蛋白(telomerase-associatedprotein,tep)。目前的研究表明,端粒(telomere)及端粒酶(telomerase)与细胞永生化有关。约80%的肿瘤细胞和永生化细胞系端粒酶活性检测均为阳性,其余肿瘤细胞和永生化细胞系可检测出足够的端粒长度。因此,恶性肿瘤的治疗方案中使用降低端粒酶的方法曾一度受到广泛关注。但是,恶性肿瘤前期细胞端粒很短,而且端粒短的人更能抵抗黑色素瘤。这与前期的研究结果是相互矛盾的。hockemeyer等发现,永生化分两个过程,首先是端粒酶的突变引起的,端粒酶基因上游的启动子(tert)发生突变,但是该突变水平很低,还不足以使癌细胞永生化,但可以延缓细胞衰老,且端粒酶水平会逐渐上调;第二步,含有短端粒的染色体可能发生功能异常,促使基因组的改变,从而细胞实现永生化。这也解释了,端粒较短,但端粒酶水平高的细胞更容易演变成癌细胞的原因。因此,运用质粒转导及病毒感染等方法,将端粒酶基因导入细胞,端粒酶的表达及激活是实现细胞永生化的重要步骤。
由于目前还没有建立绵羊睾丸永生化细胞系的先例,因此本研究以建立永生化羊睾丸细胞系为目的,通过向体外羊睾丸细胞导入外源性基因,成功建立了绵羊羊睾丸细胞永生化的方法。
技术实现要素:
本发明的目的正是为了解决上述问题,而提出一种绵羊羊睾丸细胞永生化细胞系的构建方法,有利于改善目前细胞系匮乏的研究现状,有利于资源的推广使用。绵羊羊睾丸永生化细胞系的建立减少了实验动物的使用量,提供了一种更为稳定的实验研究对象。
本发明提供了一种绵羊羊睾丸细胞永生化细胞系的构建方法,具体步骤如下:
1)重组质粒构建:根据人的端粒酶的全基因序列设计一对扩增htert基因pcr特异性引物,将扩增的htert基因重组转载到重组质粒plov-cmv上,获得重组质粒plov-cmv-htert;
2)慢病毒样颗粒的制备:对步骤1)重组质粒plov-cmv-htert用试剂盒进行纯化得无内毒素的重组质粒plov-cmv-htert,将无内毒素的重组质粒plov-cmv-htert、包装质粒pspax2以及外膜质粒pmd2.g质粒用转染试剂共转染到293t细胞中,转染30h收集细胞上清液,并置于-80℃保存;
3)表达htert基因的细胞系构建和筛选:用步骤2)中慢病毒样颗粒的细胞培养上清液感染绵羊睾丸细胞,用嘌呤霉素培养基进行筛选;
4)htert基因表达及其mrna转录水平检测:提取步骤3)筛选后细胞系的总dna和总rna,用pcr检测htert基因表达,用rt-pcr方法检测htert基因的mrna转录水平;
5)hetrt蛋白表达检测:取步骤3)细胞系的细胞进行westernblotting检测和间接免疫荧光检测,以鉴定htert蛋白的表达。
作为优选手段,所述htert基因pcr特异性引物的序列如下:
htert-nheⅰ-f:5’-ctagctagctagatgccgcgcgctccccgct-3’,
htert-notⅰ-r:5’-ttgcggccgctcagtccaggatggtcttgaa-3’。
作为进一步地优选手段,所述步骤3)中嘌呤霉素培养基的嘌呤霉素浓度为0.75μg/ml。
作为进一步地优选手段,所述步骤5)中westernblot检测中,一抗为flag标签单抗,β-actin为内参蛋白,二抗为辣根酶标记的抗鼠二抗;间接免疫荧光检测中,一抗为htert多克隆抗体,二抗为alexafluor594goatanti-rabbitigg。
作为进一步地优选手段,所述htert基因的基因序列如seqidno.1所示。
作为进一步地优选手段,所述htert基因的氨基酸序列如seqidno.2所示。
一种采用上述绵羊羊睾丸细胞永生化细胞系构建方法构建的细胞系在病毒感染实验中的应用。
本发明有益效果:1、ov-st(绵羊羊睾丸细胞永生化细胞系)的建立成功,也为其他细胞系的建立,提供了很好的借鉴,有利于改善目前细胞系匮乏的研究现状,有利于资源的推广使用。绵羊羊睾丸永生化细胞系的建立减少了实验动物的使用量,提供了一种更为稳定的实验研究对象。2、由于htert基因的片段相对较长,不易扩增以及基因片段不易转入质粒中,本发明设计的引物,其扩增效果好,且慢病毒系统能够携带较大的片段的外源基因,对细胞毒性小,由慢病毒介导并整合到靶细胞基因组中的靶基因不仅对转录沉默有较强的抗性,比使用脂质体瞬转穿梭质粒的方法更易于获得稳定表达目的基因的细胞系。3、绵羊羊睾丸细胞的分化度低,易于细胞培养,再分化,通过慢病毒样颗粒感染绵羊睾丸细胞,易于生成永生化细胞,且生成的永生化细胞稳定。
附图说明
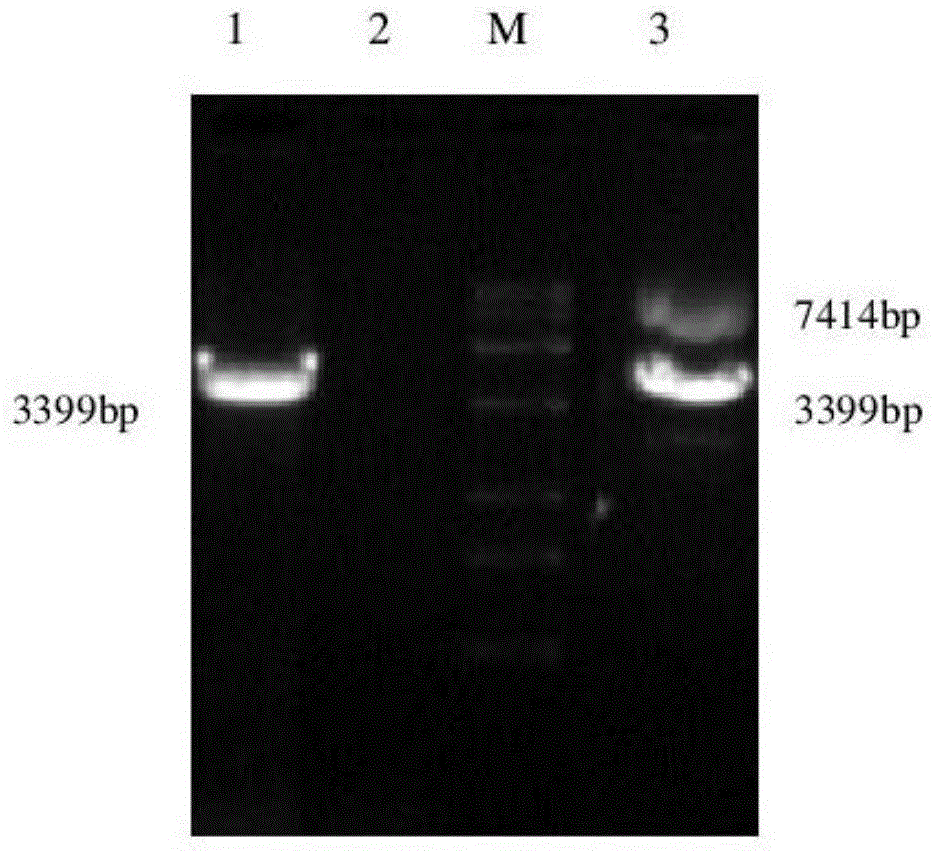
图1为本发明的plov-cmv-htert的构建与鉴定结果图(m:dna分子质量标准;1:htert基因的pcr扩增;2:阴性对照;3:plov-cmv-htert经nheⅰ和notⅰ酶双酶切产物)。
图2为本发明的ov-st细胞的htert基因检测结果图(m:dna分子质量标准;1:第10代ov-st细胞的htert基因;2:第20代ov-st细胞的htert基因;3:rt-pcr检测第10代ov-st细胞;4:rt-pcr检测第20代ov-st细胞;5:阴性对照绵羊睾丸细胞)。
图3为本发明的western-blot检测htert蛋白的结果图。
图4为本发明的ifa检测htert蛋白的表达的结果图(a:白光视野下的ov-st第5代细胞;b:绿色荧光视野下的ov-st第5代细胞;c:ifa检测第10代ov-st细胞的htert蛋白的表达;d:ifa检测第20代ov-st细胞的htert蛋白的表达;e:阴性对照)。
图5为羊口疮病毒ah-gy13株在ov-st细胞系中应用的pcr的检测结果图(m:dna分子质量标准;1-3:细胞毒第3、4、5代pcr检测;4:阳性对照;5:阴性对照)。
图6为羊口疮病毒ah-gy13株在ov-st细胞系中应用的ifa检测结果图(a:感染组;b:阴性对照)。
保藏证明:
羊口疮病毒ah-gy13株已于2015年2月4号被中国武汉的武汉大学,中国典型培养物保藏中心保藏,保藏编号:cctccno:v201511。该病毒株存活性本保藏中心于2015年3月12日检测完毕,检测存活。
具体实施方式
下面结合附图对本发明作进一步详细描述:
具体实施方案如下:
1)重组质粒plov-cmv-htert的构建:
根据端粒酶(genbank:kt923469.1)的全基因序列设计一对扩增htert基因pcr特异性引物,序列如下:
htert-nheⅰ-f:5'-ctagctagctagatgccgcgcgctccccgct-3',
htert-notⅰ-r:5'-ttgcggccgctcagtccaggatggtcttgaa-3'。
pcr体系为:2×pfumixture25μl,上下游引物各1μl(浓度为10umol/l),pbabe-neo-htert质粒0.5μl(100ng),ddh2o补至50μl。pcr扩增程序:94℃预变性3min,94℃变性30s,63℃退火45s,72℃延伸4min,31个循环,72℃后延伸5min。pcr产物切胶进行胶回收,纯化产物及plov-cmv-3flag载体均用nheⅰ和notⅰ限制性内切酶进行双酶切,酶切产物经跑胶,切胶及胶回收后,利用t4连接酶,16℃连接过夜,转化及对单克隆克隆菌进行测序。测序正确的质粒,连同辅助质粒pspax2、pmd2.g经转化并扩大培养,利用qiagenmaxiprependo-freeplasmidkit提取高浓度无内毒素的质粒,用于下一步的转染。
2)慢病毒样颗粒的制备:对步骤1)获得的无内毒素的重组质粒plov-cmv-htert、包装质粒pspax2以及外膜质粒pmd2.g质粒用转染试剂共转染到293t细胞中,转染步骤如下:
将293t细胞从液氮罐中取出复苏,调整细胞状态,并铺板于10cm的培养皿,调整细胞密度在24h前为80%以上。转染剂量如下表,采用lipofectaminetm2000进行转染。
重组慢病毒样颗粒的质粒转染
(1)转染前1h更换新鲜培养液,opti-mem培养基37℃预热;
(2)脂质体88μl加入2ml的opti-mem培养基室温孵育5min;
(3)pspax2、pmd2.g、plov-cmv-htert、plov-gfp-3flag分别加入2ml的opti-mem培养基;
(4)将(2)和(3)混合并补加6mlpti-mem培养基,室温孵育30min;
(5)将混合均匀的转染液均匀加入293t细胞培养皿中,37℃,5%co2孵育6h;
(6)更换含5%胎牛血清的dmem高糖培养基,继续培养48h,收集细胞上清,并置于-80℃保存。
3)表达htert基因的细胞系构建和筛选:将步骤2)中慢病毒样颗粒的细胞培养上清液感染绵羊睾丸细胞,感染时间为72h,其中,每隔24h更换含嘌呤霉素的新培养基,嘌呤霉素含0.75μg/ml,4d后将阳性细胞株继续以含嘌呤霉素的培养基进行传代培养,对传代10、20代的细胞进行鉴定。
4)htert基因表达及mrna转录水平检测:提取步骤3)第10代、第20代细胞的基因组总dna及总rna,总dna进行pcr扩增,总rna经反转录、pcr扩增,并用1%琼脂糖凝胶电泳检测pcr产物。
反转录具体步骤如下:分别提取ov-st细胞第10代、20代细胞的总dna和总rna。提取dna试剂盒均为omegae.z.n.a.tmtissuednakit,提取rna试剂盒为上海生工的柱式trizol总rna抽提试剂盒。rna的反转录体系为:2μgrna、1μl随机引物、12μldepc水,置于70℃孵育10min,-20℃冷却3min。再加入5μlm-mlv5×buffer、1μldntpmix、1μlrna酶抑制剂(20u/μl)、1μlm-mlv酶,补加depc至50μl。反转录程序为:37℃孵育60min,72℃孵育10min。
pcr的体系为:2×taqpcrmastermix25μl,上下游引物各1μl(浓度为10umol/l),上述提取的dna2μl,ddh2o补至50μl。pcr扩增程序:94℃预变性3min,94℃变性30s,63℃退火45s,72℃延伸4min,31个循环,72℃后延伸5min。
rt-pcr的体系为:2×taqpcrmastermix25μl,上下游引物各1μl(浓度为10umol/l),上述提取的反转录的cdna2μl,ddh2o补至50μl。pcr扩增程序:94℃预变性3min,94℃变性30s,63℃退火45s,72℃延伸4min,31个循环,72℃后延伸5min。
pcr产物及rt-pcr产物用1%琼脂糖凝胶电泳检测。
5)htert蛋白表达水平的检测:取第10和20代次细胞进行westernblotting检测和间接免疫荧光(ifa),以鉴定htert蛋白的表达;具体步骤如下:
westernblotting试验步骤:
选取培养的第10、20代细胞进行westernblot鉴定,一抗为flag标签单抗,1:2500稀释,β-actin为内参蛋白,二抗为辣根酶(hrp)标记的抗鼠二抗,1:5000倍稀释。
主要步骤如下:
(1)蛋白样品的制备:用1×pbs清洗待检细胞,2次,使用细胞裂解液裂解细胞,冰上放置10min,6000g离心5min,取上清,加入5×sds-page蛋白上样缓冲液,水浴100℃10min;
(2)sds-page凝胶电泳:根据蛋白分子量大小,配制合适的sds-聚丙烯酰胺凝胶,电泳时间为80v,30min,120v,100min,直至溴酚蓝电泳至凝胶最底部;
(3)转印:根据凝胶大小,剪裁出适当大小的硝酸纤维素膜和滤纸,浸泡至转膜液中,叠放次序为滤纸-硝酸纤维素膜-凝胶-滤纸,转印时间根据蛋白大小和膜大小进行适当调整;
(4)封闭:使用pbs配制5%的脱脂奶粉,37℃封闭2h,封闭结束后,tbst清洗3次,10min/次;
(5)一抗孵育:使用pbs按照1:2500稀释相应的一抗,4℃过夜孵育,tbst清洗3次,10min/次;
(6)二抗孵育:使用pbs按照1:5000稀释相应的二抗,37℃孵育1h,tbst清洗3次,10min/次;
(7)显色:在避光条件下,dab显色1min。
间接免疫荧光试验步骤:
将bhk-21细胞铺于24孔板中,待细胞融合度达80%时,用dtmuvah-f10株感染细胞,37℃、5%co2细胞培养箱中培养36h后收取细胞样品。
按照下列步骤进行间接免疫荧光(ifa)实验:
(1)用pbs清洗细胞3次,每孔加入4%的多聚甲醛500μl固定、4℃固定15min,pbs清洗3次,5min/次;
(2)使用pbs配制5%的脱脂奶粉,37℃封闭2h,pbst清洗4次,5min/次;
(4)一抗孵育:使用tbst按照1:1000稀释相应的一抗(htert多克隆抗体),4℃过夜孵育,tbst清洗3次,10min/次;
(5)二抗孵育:使用tbst按照1:1000稀释相应的二抗(alexafluor594goatanti-rabbitigg),37℃孵育1h,tbst清洗3次,10min/次;
(6)避光加入1:1000倍稀释的dapi,室温孵育10min,pbst清洗4次,5min/次;使用荧光显微镜观察实验结果,分析荧光强度。
6)羊口疮病毒ah-gy13株在ov-st中的稳定传代,具体方法如下:将ov-st细胞消化铺板于6孔板,待细胞密度达80%时,每孔加入200μl羊口疮病毒ah-gy13株病毒组织液,37℃孵育5h后弃去病毒液,并继续以含1%山羊血清的培养液继续培养,培养7天后,利用pcr及ifa检测病毒是否增值。
pcr检测具体方法如下:收集贴壁细胞,加入pbs反复冻融3次,用上海生工的dna提取试剂盒提取病毒dna,进行pcr检测。
具体方法如下:根据ah-gy13株(保藏编号cctccno:v201511)vir(orf020)基因序列设计一对pcr扩增特异性引物,020-p1:5'-ggaattcatggcctgcgagtgcgc-3',020-p2:5'-cccaagcttttagaagctgatgccgcag-3',扩增片段大小569bp,引物由生工生物工程(上海)股份有限公司合成。
取上述培养的细胞(经羊口疮病毒ah-gy13株感染的细胞),用上海生工的dna抽提试剂盒提取,步骤按照试剂盒说明书进行。
pcr体系为:2×taqpcrmastermix25μl,上下游引物各1μl(浓度为10umol/l),上述提取的dna2μl,ddh2o补至50μl。pcr扩增程序:94℃预变性3min,94℃变性30s,57℃退火45s,72℃延伸45s,共32个循环,72℃后延伸10min。
pcr产物用2%琼脂糖凝胶电泳检测。
间接免疫荧光(ifa)检测具体方法:将ov-st细胞消化铺板于6孔板且内置爬片,取盲传第4代的羊口疮病毒ah-gy13株的细胞毒感染细胞,并于感染4d后,弃去细胞上清,用pbs液清洗3次,向每孔加入预冷的4%多聚甲醛组织固定液,4℃固定15min,弃去固定液,用pbs液轻洗3次,再加入500μl5%脱脂奶粉,室温封闭1h,用pbs轻洗3次。按照1:200的比例加入制备的小鼠b2l抗血清,37℃孵育1h,弃抗体,用pbst液洗3次,每次5min,避光加入1ml1000倍稀释的alexafluor594goatanti-mouseigg,37℃避光孵育45min,弃去孔内液体,用pbst洗涤3次,每次5min,置于共聚焦显微镜下观察。同时设置pbs组作为阴性对照。
绵羊羊睾丸原代细胞的分离。
无菌手术刀摘取绵羊睾丸,置于含青、链霉素各1000u/ml的pbs溶液中,低温运输且时间短于4h。与实验室操作台中,除去结蹄组织、脂肪组织、鞘膜、白膜,并用无菌含青链霉素的pbs液冲洗3次。避开精曲小管,剪碎睾丸组织,加入pbs液,出去残留结蹄组织和脂肪及其他杂质。将剪碎的组织1000r/min离心5min去除上清,并加入组织量3倍的0.25%胰蛋白酶液,37℃消化15min至睾丸组织呈蓬松状,加入等体积含胎牛血清的dmem/f12培养液终止消化,经450目的细胞筛过滤得到单细胞悬液,1000r/min离心5min,用含10%胎牛血清的dmem/f12培养液重悬细胞密度至1.0×106细胞/ml,置于细胞培养瓶中,在37℃5%co2的培养箱中进行培养。绵羊睾丸细胞生长密度达90%以上,胰酶37℃消化2min,并以1:3继续传代,直至细胞生长停止。
绵羊睾丸细胞经胰酶消化传代培养,以1/3分瓶传代培养至第10代,细胞形态差异不明显,均呈纤维状或者梭形,生长速度仍是2.5d~3d长满。细胞传代一般6h开始贴壁,36h至60h是生长高峰期。
对于本领域技术人员而言,显然本发明不限于上述示范性实施例的细节,而且在不背离本发明的精神或基本特征的情况下,能够以其他的具体形式实现本发明。因此,无论从哪一点来看,均应将实施例看作是示范性的,而且是非限制性的,本发明的范围由所附权利要求而不是上述说明限定,因此旨在将落在权利要求的等同要件的含义和范围内的所有变化囊括在本发明内。
此外,应当理解,虽然本说明书按照实施方式加以描述,但并非每个实施方式仅包含一个独立的技术方案,说明书的这种叙述方式仅仅是为清楚起见,本领域技术人员应当将说明书作为一个整体,各实施例中的技术方案也可以经适当组合,形成本领域技术人员可以理解的其他实施方式。
本发明不限于以上对实施例的描述,本领域技术人员根据本发明揭示的内容,在本发明基础上不必经过创造性劳动所进行的改进和修改,都应该在本发明的保护范围之内。
sequencelisting
<110>安徽农业大学
<120>一种绵羊羊睾丸细胞永生化细胞系的构建方法及其应用
<130>2018/11/16
<160>2
<170>patentinversion3.5
<210>1
<211>3399
<212>dna
<213>pabelloniaincrassata
<400>1
atgccgcgcgctccccgctgccgagccgtgcgctccctgctgcgcagccactaccgcgag60
gtgctgccgctggccacgttcgtgcggcgcctggggccccagggctggcggctggtgcag120
cgcggggacccggcggctttccgcgcgctggtggcccagtgcctggtgtgcgtgccctgg180
gacgcacggccgccccccgccgccccctccttccgccaggtgtcctgcctgaaggagctg240
gtggcccgagtgctgcagaggctgtgcgagcgcggcgcgaagaacgtgctggccttcggc300
ttcgcgctgctggacggggcccgcgggggcccccccgaggccttcaccaccagcgtgcgc360
agctacctgcccaacacggtgaccgacgcactgcgggggagcggggcgtgggggctgctg420
ctgcgccgcgtgggcgacgacgtgctggttcacctgctggcacgctgcgcgctctttgtg480
ctggtggctcccagctgcgcctaccaggtgtgcgggccgccgctgtaccagctcggcgct540
gccactcaggcccggcccccgccacacgctagtggaccccgaaggcgtctgggatgcgaa600
cgggcctggaaccatagcgtcagggaggccggggtccccctgggcctgccagccccgggt660
gcgaggaggcgcgggggcagtgccagccgaagtctgccgttgcccaagaggcccaggcgt720
ggcgctgcccctgagccggagcggacgcccgttgggcaggggtcctgggcccacccgggc780
aggacgcgtggaccgagtgaccgtggtttctgtgtggtgtcacctgccagacccgccgaa840
gaagccacctctttggagggtgcgctctctggcacgcgccactcccacccatccgtgggc900
cgccagcaccacgcgggccccccatccacatcgcggccaccacgtccctgggacacgcct960
tgtcccccggtgtacgccgagaccaagcacttcctctactcctcaggcgacaaggagcag1020
ctgcggccctccttcctactcagctctctgaggcccagcctgactggcgctcggaggctc1080
gtggagaccatctttctgggttccaggccctggatgccagggactccccgcaggttgccc1140
cgcctgccccagcgctactggcaaatgcggcccctgtttctggagctgcttgggaaccac1200
gcgcagtgcccctacggggtgctcctcaagacgcactgcccgctgcgagctgcggtcacc1260
ccagcagccggtgtctgtgcccgggagaagccccagggctctgtggcggcccccgaggag1320
gaggacacagacccccgtcgcctggtgcagctgctccgccagcacagcagcccctggcag1380
gtgtacggcttcgtgcgggcctgcctgcgccggctggtgcccccaggcctctggggctcc1440
aggcacaacgaacgccgcttcctcaggaacaccaagaagttcatctccctggggaagcat1500
gccaagctctcgctgcaggagctgacgtggaagatgagcgtgcggggctgcgcttggctg1560
cgcaggagcccaggggttggctgtgttccggccgcagagcaccgtctgcgtgaggagatc1620
ctggccaagttcctgcactggctgatgagtgtgtacgtcgtcgagctgctcaggtctttc1680
ttttatgtcacggagaccacgtttcaaaagaacaggctctttttctaccggaagagtgtc1740
tggagcaagttgcaaagcattggaatcagacagcacttgaagagggtgcagctgcgggag1800
ctgtcggaagcagaggtcaggcagcatcgggaagccaggcccgccctgctgacgtccaga1860
ctccgcttcatccccaagcctgacgggctgcggccgattgtgaacatggactacgtcgtg1920
ggagccagaacgttccgcagagaaaagagggccgagcgtctcacctcgagggtgaaggca1980
ctgttcagcgtgctcaactacgagcgggcgcggcgccccggcctcctgggcgcctctgtg2040
ctgggcctggacgatatccacagggcctggcgcaccttcgtgctgcgtgtgcgggcccag2100
gacccgccgcctgagctgtactttgtcaaggtggatgtgacgggcgcgtacgacaccatc2160
ccccaggacaggctcacggaggtcatcgccagcatcatcaaaccccagaacacgtactgc2220
gtgcgtcggtatgccgtggtccagaaggccgcccatgggcacgtccgcaaggccttcaag2280
agccacgtctctaccttgacagacctccagccgtacatgcgacagttcgtggctcacctg2340
caggagaccagcccgctgagggatgccgtcgtcatcgagcagagctcctccctgaatgag2400
gccagcagtggcctcttcgacgtcttcctacgcttcatgtgccaccacgccgtgcgcatc2460
aggggcaagtcctacgtccagtgccaggggatcccgcagggctccatcctctccacgctg2520
ctctgcagcctgtgctacggcgacatggagaacaagctgtttgcggggattcggcgggac2580
gggctgctcctgcgtttggtggatgatttcttgttggtgacacctcacctcacccacgcg2640
aaaaccttcctcaggaccctggtccgaggtgtccctgagtatggctgcgtggtgaacttg2700
cggaagacagtggtgaacttccctgtagaagacgaggccctgggtggcacggcttttgtt2760
cagatgccggcccacggcctattcccctggtgcggcctgctgctggatacccggaccctg2820
gaggtgcagagcgactactccagctatgcccggacctccatcagagccagtctcaccttc2880
aaccgcggcttcaaggctgggaggaacatgcgtcgcaaactctttggggtcttgcggctg2940
aagtgtcacagcctgtttctggatttgcaggtgaacagcctccagacggtgtgcaccaac3000
atctacaagatcctcctgctgcaggcgtacaggtttcacgcatgtgtgctgcagctccca3060
tttcatcagcaagtttggaagaaccccacatttttcctgcgcgtcatctctgacacggcc3120
tccctctgctactccatcctgaaagccaagaacgcagggatgtcgctgggggccaagggc3180
gccgccggccctctgccctccgaggccgtgcagtggctgtgccaccaagcattcctgctc3240
aagctgactcgacaccgtgtcacctacgtgccactcctggggtcactcaggacagcccag3300
acgcagctgagtcggaagctcccggggacgacgctgactgccctggaggccgcagccaac3360
ccggcactgccctcagacttcaagaccatcctggactga3399
<210>2
<211>1132
<212>prt
<213>pabelloniaincrassata
<400>2
metproargalaproargcysargalavalargserleuleuargser
151015
histyrarggluvalleuproleualathrphevalargargleugly
202530
proglnglytrpargleuvalglnargglyaspproalaalaphearg
354045
alaleuvalalaglncysleuvalcysvalprotrpaspalaargpro
505560
proproalaalaproserpheargglnvalsercysleulysgluleu
65707580
valalaargvalleuglnargleucysgluargglyalalysasnval
859095
leualapheglyphealaleuleuaspglyalaargglyglypropro
100105110
glualaphethrthrservalargsertyrleuproasnthrvalthr
115120125
aspalaleuargglyserglyalatrpglyleuleuleuargargval
130135140
glyaspaspvalleuvalhisleuleualaargcysalaleupheval
145150155160
leuvalalaprosercysalatyrglnvalcysglyproproleutyr
165170175
glnleuglyalaalathrglnalaargproproprohisalasergly
180185190
proargargargleuglycysgluargalatrpasnhisservalarg
195200205
glualaglyvalproleuglyleuproalaproglyalaargargarg
210215220
glyglyseralaserargserleuproleuprolysargproargarg
225230235240
glyalaalaprogluprogluargthrprovalglyglnglysertrp
245250255
alahisproglyargthrargglyproseraspargglyphecysval
260265270
valserproalaargproalagluglualathrserleugluglyala
275280285
leuserglythrarghisserhisproservalglyargglnhishis
290295300
alaglyproproserthrserargproproargprotrpaspthrpro
305310315320
cysproprovaltyralagluthrlyshispheleutyrsersergly
325330335
asplysgluglnleuargproserpheleuleuserserleuargpro
340345350
serleuthrglyalaargargleuvalgluthrilepheleuglyser
355360365
argprotrpmetproglythrproargargleuproargleuprogln
370375380
argtyrtrpglnmetargproleupheleugluleuleuglyasnhis
385390395400
alaglncysprotyrglyvalleuleulysthrhiscysproleuarg
405410415
alaalavalthrproalaalaglyvalcysalaargglulysprogln
420425430
glyservalalaalaprogluglugluaspthraspproargargleu
435440445
valglnleuleuargglnhisserserprotrpglnvaltyrglyphe
450455460
valargalacysleuargargleuvalproproglyleutrpglyser
465470475480
arghisasngluargargpheleuargasnthrlyslyspheileser
485490495
leuglylyshisalalysleuserleuglngluleuthrtrplysmet
500505510
servalargglycysalatrpleuargargserproglyvalglycys
515520525
valproalaalagluhisargleuargglugluileleualalysphe
530535540
leuhistrpleumetservaltyrvalvalgluleuleuargserphe
545550555560
phetyrvalthrgluthrthrpheglnlysasnargleuphephetyr
565570575
arglysservaltrpserlysleuglnserileglyileargglnhis
580585590
leulysargvalglnleuarggluleuserglualagluvalarggln
595600605
hisargglualaargproalaleuleuthrserargleuargpheile
610615620
prolysproaspglyleuargproilevalasnmetasptyrvalval
625630635640
glyalaargthrpheargargglulysargalagluargleuthrser
645650655
argvallysalaleupheservalleuasntyrgluargalaargarg
660665670
proglyleuleuglyalaservalleuglyleuaspaspilehisarg
675680685
alatrpargthrphevalleuargvalargalaglnasppropropro
690695700
gluleutyrphevallysvalaspvalthrglyalatyraspthrile
705710715720
proglnaspargleuthrgluvalilealaserileilelysprogln
725730735
asnthrtyrcysvalargargtyralavalvalglnlysalaalahis
740745750
glyhisvalarglysalaphelysserhisvalserthrleuthrasp
755760765
leuglnprotyrmetargglnphevalalahisleuglngluthrser
770775780
proleuargaspalavalvalilegluglnserserserleuasnglu
785790795800
alaserserglyleupheaspvalpheleuargphemetcyshishis
805810815
alavalargileargglylyssertyrvalglncysglnglyilepro
820825830
glnglyserileleuserthrleuleucysserleucystyrglyasp
835840845
metgluasnlysleuphealaglyileargargaspglyleuleuleu
850855860
argleuvalaspasppheleuleuvalthrprohisleuthrhisala
865870875880
lysthrpheleuargthrleuvalargglyvalproglutyrglycys
885890895
valvalasnleuarglysthrvalvalasnpheprovalgluaspglu
900905910
alaleuglyglythralaphevalglnmetproalahisglyleuphe
915920925
protrpcysglyleuleuleuaspthrargthrleugluvalglnser
930935940
asptyrsersertyralaargthrserileargalaserleuthrphe
945950955960
asnargglyphelysalaglyargasnmetargarglysleuphegly
965970975
valleuargleulyscyshisserleupheleuaspleuglnvalasn
980985990
serleuglnthrvalcysthrasniletyrlysileleuleuleugln
99510001005
alatyrargphehisalacysvalleuglnleuprophehisgln
101010151020
glnvaltrplysasnprothrphepheleuargvalileserasp
102510301035
thralaserleucystyrserileleulysalalysasnalagly
104010451050
metserleuglyalalysglyalaalaglyproleuproserglu
105510601065
alavalglntrpleucyshisglnalapheleuleulysleuthr
107010751080
arghisargvalthrtyrvalproleuleuglyserleuargthr
108510901095
alaglnthrglnleuserarglysleuproglythrthrleuthr
110011051110
alaleuglualaalaalaasnproalaleuproseraspphelys
111511201125
thrileleuasp
1130
- 还没有人留言评论。精彩留言会获得点赞!