一株假交替单胞菌属多糖降解菌及其培养方法与应用与流程
本发明涉及一株假交替单胞菌属多糖降解菌及其培养方法与应用,属于生物技术领域。
背景技术:
多糖,是由重复的单一或不同糖单元通过糖苷键连接而成的聚合物。根据生物来源的不同,常见微生物多糖有肽聚糖、黄原胶等,海藻多糖如琼脂糖、褐藻胶、卡拉胶等,高等植物多糖如纤维素、甘露聚糖、木聚糖和淀粉等,以及动物多糖如糖胺聚糖、几丁质等[1]。这些多糖是生物体的重要成分,有些还是能源物质,部分是参与细胞识别的信息分子[2]。其中,包括透明质酸、硫酸软骨素/硫酸皮肤素、肝素/硫酸乙酰肝素、硫酸角质素等在内的糖胺聚糖或寡糖,广泛分布于海洋动物和陆生动物组织的细胞表面或细胞外基质,参与了中枢神经系统发育[3-4],组织形态发生[5],信号传导[6-7],细胞增殖分化[8-10],伤口修复[11-12],病毒侵染[13-15]等多种生理学和病理学行为,因此糖胺聚糖的研究具有重要意义。研究还表明,其它多糖及其降解产物也具有多种重要生理活性如:新琼寡糖具有抗氧化作用[16]、黄原胶具有优良的稳定性[17]、褐藻胶具有吸水成胶能力[18]等。综上,多糖及其寡糖产物具有重要的应用价值,因此多糖降解菌及其多糖降解酶资源的开发一直是酶法制备寡糖的研究热点之一,其中新型多糖降解菌的发现是研制新型工具酶的源头保障。
多能型多糖降解菌是指能以多种多糖为唯一碳源进行生长的菌株[19-20]。一株多能型多糖降解菌可产生多种不同类型的多糖降解酶,催化底物分子中糖单元间糖苷键的断裂,产生一系列寡糖乃至单糖,从而实现菌株对不同多糖碳源的降解和利用,其突出特征即是一菌多能。因而,对新型多糖降解菌及其酶资源的开发具有重要价值和意义。海洋蕴含着大量的微生物资源,因此一直是生物资源开发领域的研究重点。目前,海洋来源的多能型多糖降解菌仅见国内flammeovirgasp.my04[21]、persicobactersp.jzb09[22]、pseudoalteromonassp.a601[23]、vibriosp.fc509[24]及国外如saccharophagusdegrada-ns[25-28]、zobelliagalactanivorans[29]等少数菌株的报道。
假交替单胞菌和弧菌被认为是海洋环境中细菌的代表性物种,它们数量大、种类多、分布广,其资源学和生态学的研究意义十分重要。
2014年,发明人曾经筛选到一株弧菌vibriosp.fc509[24],它可产生多种多糖降解酶,因而既能降解海藻多糖又能降解动物多糖尤其能高效降解糖胺聚糖,如透明质酸、硫酸软骨素、硫酸皮肤素、肝素等。所产糖胺聚糖内切酶hclase能高效降解透明质酸和多种类型硫酸软骨素[30],硫酸酯酶可以通过内切模式脱去糖链上4位硫酸根[31],糖胺聚糖外切酶hcdlase可以从寡糖还原端向非还原端逐渐产生二糖单元[32],内切型软骨素裂解酶hclaseer是第一个被鉴定的不能作用于e单元的透明质酸/硫酸软骨素裂解酶[33]。在此基础上,综合利用来自同一菌株的这些多糖降解酶,可对复杂的cs寡糖进行测序,进而进行糖胺聚糖构效关系的研究,因此应用价值极其重要[34]。
2017年,nicoleking课题组发现费氏弧菌能够促进领鞭毛虫的交配繁殖,并证实费氏弧菌vibriofischeriatcc700601分泌的蛋白-eros(透明质酸/糖胺聚糖裂解酶)能发挥“催情剂”的作用,从而导致了这一现象的产生,这表明弧菌可以诱导真核生物的有性生殖[35]。我们分析了ncbi网站公开的该费氏弧菌菌株的基因组,发现富含多糖降解酶基因,因此推测其是多能型多糖降解菌。这提示:多能型多糖降解菌有可能参与了众多跨物种的生殖过程,它们所产多糖降解酶具有重要的生理功能和生态学效应。
lee.s等[36]认为假交替单胞菌属细菌能够抑制藻类孢子萌发,或溶解藻类细胞,在维持生态环境的平衡过程中扮演了重要角色。假交替单胞菌属(pseudoalteromonas)是gauthier等根据16srrna序列建立起来的区别于假单胞菌属(pseudomonas)和交替单胞菌属(alteromonas)的新属[37],国内外学者已分离并鉴定出了多株假交替单胞菌[38-42]。相比之下,假交替单胞菌属来源的多能型多糖降解菌较少,仅见如pseudoalteromonassp.a601的研究[23]。该菌株只能降解淀粉、琼脂糖等植物或海藻多糖,但是不能降解糖胺聚糖等动物多糖。那么,假交替单胞菌属是否存在能降解如透明质酸、硫酸软骨素等糖胺聚糖的菌株?这样的菌株是否也能参与真核生物的生理过程?目前未见相关菌株的发现与深入研究。
已有研究表明,假交替单胞菌能产生胞外酶、胞外多糖、毒素等多种与致病性有关的物质,是一类条件致病菌。因此,对于假交替单胞菌的研究主要集中于病害防治和菌株药敏特性等方面,关于多糖降解酶的研究相对较少且集中于菌株所产单一多糖降解酶,如琼胶酶、β-半乳糖苷酶,α-淀粉酶和果胶裂解酶等[43-46]。然而,少见假交替单胞菌属的具有糖胺聚糖降解能力的多能型多糖降解菌。因此,对于这一重要海洋微生物物种的资源开发必然有利于相关分子致病机制的认知和功能寡糖药物的研制。
技术实现要素:
本发明针对现有技术的不足,提供一株来源于海岸沉积物的能降解、利用多种不同类型多糖的假交替单胞菌及其培养方法与应用。
本发明是通过以下技术方案实现的:
一株假交替单胞菌(pseudoalteromonassp.)q02,于2018年11月8日保藏于中国微生物菌种保藏管理委员会普通微生物中心,地址:北京市朝阳区北辰西路1号院3号中国科学院微生物研究所,保藏编号:cgmccno.16720。
假交替单胞菌(pseudoalteromonassp.)q02菌株,革兰氏染色阴性,该菌株在平板上形成圆形菌落,表面光滑,整体呈乳白色,同时可以发现到在菌落周围有较为明显的凹陷。用于菌体形态观察的固体培养基为初筛固体培养基,组分如下:蛋白胨10g,酵母提取物5g,固体平板加1.5%琼脂糖,余量人工海水(nacl30g,kcl7.5g,cacl21.1g,mgso47.2g,nh4cl1.5g,ddh2o1000ml,ph7.2),ph7.2。
假交替单胞菌(pseudoalteromonassp.)q02菌株,经测定,其16srrna的基因序列长度为1464bp,如seqidno.1所示。通过使用美国生物工程信息中心(nationalcenterforbiotechnologyinformation,ncbi)blastn程序搜索比对,证明本发明的菌株的16srrna的基因序列与ncbi注册的假交替单胞菌标准菌株(pseudoalteromonas)16srrna的基因序列具有较高的同源性,其中与标准菌株pseudoalteromonasmariniglutinosadsm15203的最近,序列相似度为98.8%。
假交替单胞菌(pseudoalteromonassp.)q02菌株,采用16srrna基因构建系统发育树,结果如图3所示。结果表明,q02菌株与假交替单胞菌属的模式菌株聚类,且位于该分支内部。因此,将q02菌株鉴定至假交替单胞菌属。
上述假交替单胞菌(pseudoalteromonassp.)q02菌株的培养方法,包括如下步骤:
(1)将冷冻保存的假交替单胞菌(pseudoalteromonassp.)q02菌株样品,划线至初筛固体培养基上,25~30℃倒置培养18~48h,制得活化菌株;
(2)挑取步骤(1)制得的活化菌株,接种至初筛液体培养基中,在温度为25~30℃、转速为180~220转/分钟的条件下,摇床培养16~24h,制得种子液;
(3)将步骤(2)制得的种子液,按1~5%的体积百分比接种于扩大培养基中,在温度为25~30℃、转速为180~220转/分钟的条件下,扩大培养4~6天,制得假交替单胞菌(pseudoalteromonassp.)q02菌液。
根据本发明优选的,所述步骤(1)中的初筛固体培养基,每升组分如下:
酵母提取物10g,胰蛋白胨20g,葡萄糖20g,琼脂15g,余量人工海水,ph7.2;
上述人工海水组份如下:
nacl30g,kcl7.5g,cacl21.1g,mgso47.2g,nh4cl1.5g,ddh2o定容至1000ml。
根据本发明优选的,所述步骤(2)中的初筛液体培养基,每升组分如下:
酵母提取物10g,胰蛋白胨20g,葡萄糖20g,余量人工海水,ph7.2;
上述人工海水组份如下:
nacl30g,kcl7.5g,cacl21.1g,mgso47.2g,nh4cl1.5g,ddh2o定容至1000ml。
根据本发明优选的,所述步骤(3)中的扩大培养基,每升组分如下:
蛋白胨4g,酵母提取物2.5g,余量人工海水;
上述人工海水组份如下:
nacl30g,kcl7.5g,cacl21.1g,mgso47.2g,nh4cl1.5g,ddh2o定容至1000ml。
上述假交替单胞菌(pseudoalteromonassp.)q02菌株在制备降解海藻多糖酶制剂中的应用。
根据本发明优选的,所述应用,步骤如下:
(i)取上述假交替单胞菌(pseudoalteromonassp.)q02菌液,按1~5%的体积百分比接种于发酵培养基中,在温度为25~30℃、转速为180~220转/分钟的条件下,扩大培养3~7天,制得假交替单胞菌q02菌株发酵液;
(ii)取步骤(i)制得的假交替单胞菌q02菌株发酵液,固液分离,取液体,加入硫酸铵,使浓度达到饱和度的80%,离心,收集沉淀,用20~50ml的tge缓冲液重悬沉淀,透析去除硫酸铵,制得胞外酶制剂。
根据本发明优选的,所述步骤(i)中的发酵培养基,每升组分如下:
蛋白胨4g,酵母提取物2.5g,余量人工海水,ph7.2;
上述人工海水组份如下:
nacl30g,kcl7.5g,cacl21.1g,mgso47.2g,nh4cl1.5g,ddh2o定容至1000ml。
根据本发明优选的,所述步骤(ii)中,固液分离的条件为:12000×g,4℃离心5~20min。
根据本发明优选的,所述步骤(ii)中,离心为:15000×g,4℃离心15~30min。
根据本发明优选的,所述步骤(ii)中,tge缓冲液组分如下:
50mmtris,50mmnacl,0.5mmedta,5mm二硫苏糖醇,5.0%(v/v)甘油,ddh2o定容至1000ml,ph7.9。
根据本发明优选的,所述步骤(ii)中,透析为采用分子截留量10,000da的透析袋,进行搅拌透析。
上述降解海藻多糖酶制剂在降解微生物多糖、海藻多糖、高等植物多糖和/或动物多糖中的应用。
根据本发明优选的,所述微生物多糖为黄原胶;海藻多糖为褐藻胶、琼脂糖、卡拉胶、κ型卡拉胶、ι型卡拉胶、λ型卡拉胶;高等植物多糖为纤维素、微晶纤维素、羧甲基纤维素、淀粉、木聚糖、甘露聚糖、果胶、葡萄糖;动物多糖为透明质酸、硫酸软骨素a、硫酸软骨素c、硫酸软骨素e、硫酸乙酰肝素、硫酸皮肤素、几丁质、壳聚糖。
上述假交替单胞菌(pseudoalteromonassp.)q02菌株在降解微生物多糖、海藻多糖、高等植物多糖和/或动物多糖中的应用。
根据本发明优选的,所述微生物多糖为黄原胶;海藻多糖为褐藻胶、琼脂糖、卡拉胶、κ型卡拉胶、ι型卡拉胶、λ型卡拉胶;高等植物多糖为纤维素、微晶纤维素、羧甲基纤维素、淀粉、木聚糖、甘露聚糖、果胶;动物多糖为透明质酸、硫酸软骨素a、硫酸软骨素c、硫酸软骨素e、硫酸乙酰肝素、硫酸皮肤素、几丁质、壳聚糖。
有益效果
本发明从海岸分离获得一株假交替单胞菌(pseudoalteromonassp.)q02菌株,该菌株能以微生物、海藻、陆地植物以及动物来源的多种类型的多糖为唯一碳源生长,所产多糖降解酶的种类丰富,是一株多能型多糖降解菌,用该菌株制备的胞外酶制剂可降解微生物、海藻、植物和动物多糖,尤其对来源于海藻的褐藻胶、琼脂糖以及来源于动物的ha、csc、cse降解活性显著,具有潜在的应用价值,这与现有已知的假交替单胞菌(pseudoalteromonassp.)功能完全不相同。
附图说明
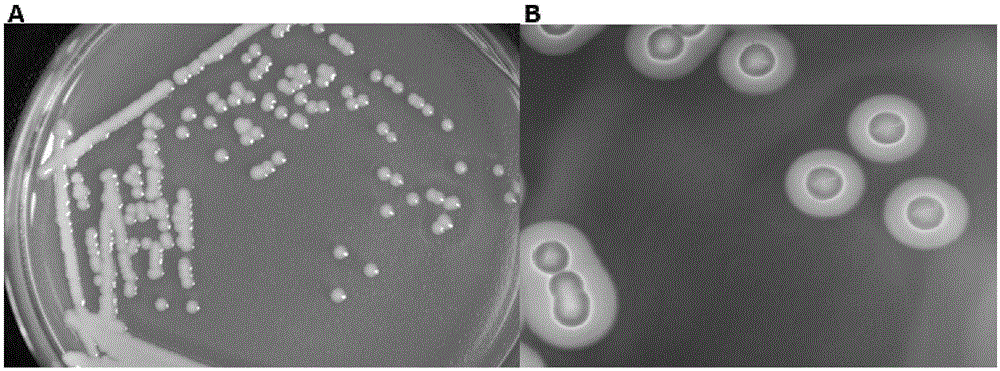
图1假交替单胞菌(pseudoalteromonassp.)q02菌株的基本形态学特征照片;
其中,a、培养2天后菌株在初筛培养基上的菌落形态;b、卢戈氏碘液染色图;
图2、假交替单胞菌q02菌株在唯一碳源培养基中生长的最高菌体浓度柱状图;
图3、假交替单胞菌q02菌株基于16srrna基因序列绘制的进化树;
图4、假交替单胞菌q02菌株胞外酶制剂的sds-变性凝胶电泳分析照片;
图中:泳道m、10μl蛋白质分子量标准物sm0431;泳道1、5μlq02胞外酶制剂;
图5、假交替单胞菌q02菌株胞外酶系酶谱分析照片;
图中:泳道m、10μl蛋白质分子量标准物sm0431;泳道1、2.5μlq02胞外酶制剂;泳道2、10μlq02胞外酶琼胶酶系;
图6、假交替单胞菌q02菌株胞外酶制剂对多糖降解能力的分析柱状图(dns-还原糖法测定);
图7、胞外酶制剂降解琼脂糖所得产物的tlc分析图;
图中:na2、新琼二糖;na4、新琼四糖;na6、新琼六糖;na8、新琼八糖;
图8、胞外酶制剂降解褐藻胶所得产物的hplc分析图;
图中:udp2:不饱和二糖;udp3:不饱和三糖;udp4:不饱和四糖;udp5:不饱和五糖;udp6:不饱和六糖;udp7:不饱和七糖;udp8:不饱和八糖;udp9:不饱和九糖;udp10:不饱和十糖;
图9、胞外酶制剂降解ha、csc、cse、hs/ds所得产物的hplc分析图;
图中:udp4:不饱和四糖。
具体实施方式
下面结合实施例对本发明的技术方案做进一步说明,但本发明所保护范围不限于此。
生物材料来源
假交替单胞菌(pseudoalteromonassp.)q02,于2018年11月8日保藏于中国微生物菌种保藏管理委员会普通微生物中心,地址:北京市朝阳区北辰西路1号院3号中国科学院微生物研究所,保藏编号:cgmccno.16720。
实施例1、海岸沉积物微生物的分离与纯化
取青岛栈桥附近海岸沉积物,取1g样品,置于体积为100ml的琼脂糖、褐藻胶或卡拉胶唯一碳源培养基中,在温度为28℃、转数为200转/分钟的条件下,培养至溶液浑浊;取溶液1ml加入到9ml无菌水中,分别稀释至浓度为10-1、10-2、10-3、10-4、10-5的5个浓度梯度。将稀释后的菌悬液与涂布于初筛培养基,每个浓度做两个平行,28℃培养7天。挑出能使平板凹陷的单菌落,经过3次平板划线分离纯化后,挑单菌落于初筛液体培养基中,28℃、200转/分钟培养3天,取培养物1.6ml加入400μl甘油,混匀后于-80℃冰箱长期保存。
上述初筛培养基,每升组分如下:
酵母提取物10g,胰蛋白胨20g,葡萄糖20g,余量人工海水,ph7.2。
固体培养基中加入质量浓度为1.5%(w/v)的琼脂。
人工海水,每升组分如下:
nacl30g,kcl7.5g,cacl21.1g,mgso47.2g,nh4cl1.5g,ddh2o1000ml,ph7.2。
实施例2、多糖降解菌的筛选
把实施例1中所得到的菌株单克隆,分别接种到唯一碳源液体培养基中,200转/分钟、30℃培养72h,观察菌液浑浊情况及培养液粘稠度的变化情况。依据上述两个指标选择产酶菌株。将在各唯一碳源培养基中菌体浑浊度以及粘稠度变化较大的菌株,挑取到初筛固体培养基上划线并培养,分别保种并编号记录如q01、q02、q03,…等;将单菌落培养24h后,用改良卢戈氏碘液染色;
上述唯一碳源培养基的配制方法是:
向人工海水中分别添加多糖底物至终浓度为0.10%(w/v);
上述多糖底物选自:纤维素,微晶纤维素,羧甲基纤维素,木聚糖,甘露聚糖,淀粉,果胶,几丁质,壳聚糖,琼脂糖,海藻酸钠,透明质酸,硫酸软骨素a,硫酸软骨素c,硫酸软骨素e,硫酸乙酰肝素,硫酸皮肤素,黄原胶;在115℃高压灭菌20min。
上述人工海水,每升组分如下:
nacl30g,kcl7.5g,cacl21.1g,mgso47.2g,nh4cl1.5g,ddh2o1000ml,ph7.2。
上述改良卢戈氏碘液,每升组分如下:
ki2.0g,i21.0g,水定容至1l。
按照如上所述方法,分离得到假交替单胞菌(pseudoalteromonassp.)q02菌株,如图1a所示。该菌株在固体培养基上,菌落中间乳白色、凸起、较湿润;在琼脂糖唯一碳源平板上,菌落四周有显著的凹陷坑;卢戈氏碘液染色后,菌株周围出现透明圈,如图1b所示。这表明,q02菌株能分泌胞外琼胶酶且降解琼脂糖后产生了水溶性还原糖。
又如图2所示,上述假交替单胞菌(pseudoalteromonassp.)q02菌株,能利用多种多糖为唯一碳源进行生长,但是在褐藻胶、甘露聚糖中生长较为旺盛,在糖胺聚糖(透明质酸、硫酸软骨素)等中生长较弱,在羧甲基纤维素和τ-卡拉胶中未见显著生长。这一分析表明,q02菌株至少能够降解19种多糖,是一株多能型多糖降解菌。
将假交替单胞菌(pseudoalteromonassp.)q02于2018年11月8日保藏于中国微生物菌种保藏管理委员会普通微生物中心,地址:北京市朝阳区北辰西路1号院3号中国科学院微生物研究所,保藏编号:cgmccno.16720。
实施例3、基于16srrna基因克隆的pseudoalteromonassp.q02菌株的分子鉴定
用细菌基因组提取试剂盒(天根生化)制备菌株q02的基因组dna,用作模板,用细菌16srrna基因的通用引物(27f和1492r)进行扩增,并进行琼脂糖凝胶电泳,然后用胶回收试剂盒(天根)纯化pcr扩增产物,电泳验证后,连接到peasy-bluntsimplecloningvector,转化到e.colitrans1t1感受态细胞。经氨苄抗性筛选,获得阳性克隆。所得16srrna基因测序由生工生物工程(上海)股份有限公司完成,将序列与美国国家生物信息中心(ncbi)收录的标准菌株的16srrna基因序列进行比对检索,应用mega6.0构建系统发育树。
上述用于菌株16srrna基因扩增的通用引物为:
正向引物为27f:5’-agagtttgatcctggctcag-3’;
反向引物为1492r:5’-ggttaccttgttacgactt-3’。
上述用于菌株16srrna基因扩增的反应体系如下,总体积30μl:
所用基因扩增试剂primestarhsdnapolymerase、dntp混合物、缓冲液购自大连宝生物技术有限公司。
上述用于菌株16srrna基因扩增的程序为:
94℃预变性5min;94℃变性30s,55℃退火30s,72℃延伸90s,共35个循环;72℃延伸15min;降温至4℃并保温15min。
上述基于16srrna基因系统发育树构建方法:
应用mega6.0软件包中的clustalw对测得的16srrna基因序列以及自ncbi基因数据库中获得的标准菌株的相似序列,一起进行多序列比对,用neighbor-joining法构建系统发育树,并进行1000次bootstraps检验,获得统计树。
结果如图3所示,本发明筛选到的q02与数据库中pseudoalteromonasariniglutinosadsm15203的亲缘关系最近,且16srrna基因之间的相似度为98.8%。基于16srrna基因构建所得的统计树表明,q02菌株与假交替单胞菌的多个标准菌株聚类,并位于该分支内部。因此,q02菌株被鉴定至假交替单胞菌属。
实施例4、假交替单胞菌(pseudoalteromonassp.)q02菌株胞外酶制剂的制备
(1)从-80℃冰箱划线假交替单胞菌q02菌株于初筛固体培养基上,28℃倒置培养18–48h;
(2)挑取q02落至初筛液体培养基中,在温度为28℃、转速为200转/分钟的条件下,摇床培养16-24h,制得种子液100ml;
(3)将步骤(2)制得的种子液,按1%的体积百分比接种于发酵培养基100ml/瓶中,在温度为28℃、转速为200转/分钟的条件下,扩大培养5天,制得假交替单胞菌q02菌株发酵液共1000ml;
(4)取步骤(3)制得的假交替单胞菌q02菌株的发酵液,12,000×g,4℃,离心10min,取上清,加入硫酸铵,使浓度达到饱和度的80%,15000×g、4℃离心30min后收集80%硫酸铵饱和度下所得沉淀,用20倍体积的tge缓冲液重悬沉淀,并使用分子截留量为10,000da的透析袋透析,以去除硫酸铵,制得胞外酶制剂。
所述步骤(1)、(2)中的初筛培养基,每升组分如下:
酵母提取物10g,胰蛋白胨20g,葡萄糖20g,余量人工海水(nacl30g,kcl7.5g,cacl21.1g,mgso47.2g,nh4cl1.5g,ddh2o1000ml,ph7.2),ph7.2,固体培养基中加入质量浓度为1.5%(w/v)的琼脂。
上述用于菌株发酵的培养基为产酶培养基,每升组分如下:
蛋白胨4g,酵母提取物2.5g,余量人工海水;
上述tge缓冲液的组分如下:
50mmtris,50mmnacl,0.5mmedta,5mmdtt(二硫苏糖醇),5.0%(v/v)glycerol(甘油),ddh2o1000ml,ph7.9。
所述的透析,使用分子截留量为10,000da的透析袋,在低温环境中对20~50倍体积的tge缓冲液进行搅拌透析,然后用sds变性凝胶电泳对假交替单胞菌q02菌株的胞外酶制剂进行检测。结果显示假交替单胞菌q02菌株胞外酶制剂含有多条蛋白条带,如图4所示。
实施例5、假交替单胞菌q02菌株胞外酶系酶谱分析;
(1)在冰水浴低温环境下,用sds变性凝胶电泳对假交替单胞菌q02菌株8胞外酶制剂进行分离,其中分离胶浓度为13.2%、层积胶浓度为5%;
(2)在电泳结束后,将胶块转入50ml缓冲液b中,室温下低速振荡,并更换缓冲液b至少3次,总时间不少于90min;
(3)将所得到的胶块贴在含1.0%琼脂糖的平皿上,固定后置于37℃,温育过夜;
(4)按照胶块中预染的分子量标准物,在平板的相应位置进行逐个标注,并用改良卢戈氏碘液对琼脂糖平皿染色,观察,并测定透明条带的相对位置。结果如图5所示,碘液染色后的平皿中对应分子量标准物约45kda处存在1条显著透明带,这表明:在无琼脂糖诱导物的培养条件下,菌株q02的胞外琼胶酶系含有至少含有1条琼胶酶,且分子量大小约45kda。
上述缓冲液b的组分如下:
50mmoltris,150mmolnacl,ddh2o1000ml,ph8.0。
上述改良卢戈氏碘液,组分如下:
ki2.0g,i21.0g,ddh2o1000ml。
实施例6、q02胞外酶制剂对不同多糖降解能力的分析
人工海水配制浓度为0.1%(w/v)的多糖底物、胞外酶制剂以及pbs缓冲液按1:1:1(体积比)的比例混合后,在30℃、ph7.4下反应24h,沸水浴中温育10min使酶失活,12,000×g,4℃离心10min,取上清,作为胞外酶制剂的酶解产物,用dns法检测生成的还原糖。
结果如图6所示,还原糖产物的分析表明,假交替单胞菌q02菌株制备的胞外酶制剂不仅能降解微生物多糖如肽聚糖、黄原胶等,还能够降解高等植物多糖如纤维素、淀粉等,尤其对来源于海藻的褐藻胶、琼脂糖以及来源于动物的ha、csc、cse降解活性显著。因此,利用该菌株制备的胞外酶制剂富含多种不同类型的多糖降解酶,具有潜在的开发价值。
实施例7、胞外酶制剂降解琼脂糖所得产物的tlc分析;
将浓度3mg/ml的琼脂糖、胞外酶制剂、pbs缓冲液按体积比1:1:1的比例混合后,在30℃、ph7.4的条件下反应0h、10min、30min、1h、4h、8h、16h、24h、48h、72h、96h,沸水浴中温育10min,12,000×g,4℃离心10min,取上清,作为胞外酶制剂的酶解产物。用沸水浴预先灭活的酶,进行对照组实验。取上述酶解产物4μl进行薄层色谱(tlc)分析,所用薄板为tlcsilicagel60f254,展开剂为正丁醇∶乙醇∶水=2∶1∶1,显色剂(二苯胺∶苯胺∶磷酸∶丙酮=1g∶1ml∶5ml∶50ml)染色后;110℃加热10min显色。
结果如图7所示,产物中含有与新琼寡糖na2、na4、na6及na8的rf值一致的系列寡糖。这表明:q02胞外酶制剂降解琼脂糖后的主产物是新琼二糖、四糖、六糖,其中na2是最小产物。
实施例8、胞外酶制剂降解褐藻胶所得产物的hplc分析
将浓度3mg/ml的多糖(褐藻胶、ha、csc、cse)、胞外酶制剂、pbs缓冲液按1:1:1(体积比)的比例混合后,在30℃、ph7.4的条件下反应72h,沸水浴中温育10min使酶失活,12,000×g,4℃离心15min,取上清,作为胞外酶制剂的酶解产物。用沸水浴预先灭活的酶,进行对照组实验。取上述酶解产物20μl进行高效液相(hplc)分析,所用凝胶柱为superdextm30increase10/300gl,流动相为0.2m碳酸氢铵,流速为0.4ml/min;检测条件为uv232nm或uv235nm。
上述pbs缓冲液,每升组分如下:
nacl8g,kcl0.2g,na2hpo41.44g,kh2po40.24g,ddh2o1000ml,ph7.4。
结果如图8所示,在胞外酶制剂与褐藻胶反应后的主产物中,具有最高吸收值的寡糖片段的出峰时间约为33.7min,提示是不饱和五糖。相比之下,对照组的产物中检测不到任何具有特征吸收的寡糖成分。因此,由假交替单胞菌(pseudoalteromonassp.)q02菌株制备的胞外酶制剂,含有褐藻胶裂解酶,可用于生产系列不饱和褐藻胶寡糖。
结果如图9所示,胞外酶制剂与ha的反应产物中,寡糖主产物的出峰时间约为34.5min,提示是不饱和四糖。胞外酶制剂与csa、csc、cse、hs/ds的反应产物中,均能检测到吸收峰,但在对照组的产物中未检测到具有特征吸收的寡糖成分。因此,由假交替单胞菌(pseudoalteromonassp.)q02菌株制备的胞外酶制剂,存在糖胺聚糖降解酶,为后期糖胺聚糖结构功能的研究提供了潜在的工具。
参考文献:
[1]生物化学.(上册)/王镜岩朱圣庚徐长法等著.-3版.-北京:高等教育出版社,2002.
[2]韩文君.海洋溶胶细菌flammeovirgasp.my04琼胶酶(系)的研究[d].青岛:中国海洋大学博士学位论文,2012.
[3]faissnera.,clementa.,lochtera.,streita.,mandlc.,schachnerm.(1994)isolationofaneuralchondroitinsulfateproteoglycanwithneuriteoutgrowthpromotingproperties.j.cellbiol.126,783–799.
[4]clementa.m.,nadanakas.,masayamak.,mandlc.,sugaharak.,faissnera.(1998)thedsd-1carbohydrateepitopedependsonsulfation,correlateswithchondroitinsulfatedmotifs,andissufficienttopromoteneuriteoutgrowth.j.biol.chem.273,28444–28453
[5]klüppelm.,wightt.n.,chanc.,hineka.,wranaj.l.(2005)maintenanceofchondroitinsulfationbalancebychondroitin-4-sulfotransferase1isrequiredforchondrocytedevelopmentandgrowthfactorsignalingduringcartilagemorphogenesis.development132,3989–4003
[6]nandis.,akhterm.p.,seifertm.f.,daix.m.,stanleye.r.(2006)developmentalandfunctionalsignificanceofthecsf-1proteoglycanchondroitinsulfatechain.blood107,786–795
[7]taylork.r.,gallor.l.(2006)glycosaminoglycansandtheirproteoglycans:host-associatedmolecularpatternsforinitiationandmodulationofinflammation.fasebj.20,9–22
[8]hwangh.y.,olsons.k.,eskoj.d.,horvitzh.r.(2003)caenorhabditiselegansearlyembryogenesisandvulvalmorphogenesisrequirechondroitinbiosynthesis.nature423,439–443
[9]mizuguchis.,uyamat.,kitagawah.,nomurak.h.,dejimak.,gengyo-andok.,mitanis.,sugaharak.,nomurak.(2003)chondroitinproteoglycansareinvolvedincelldivisionofcaenorhabditiselegans.nature423,443–448
[10]izumikawat.,kitagawah.,mizuguchis.,nomurak.h.,nomurak.,tamuraj.,gengyo-andok.,mitanis.,sugaharak.(2004)nematodechondroitinpolymerizingfactorshowingcell-/organ-specificexpressionisindispensableforchondroitinsynthesisandembryoniccelldivision.j.biol.chem.279,53755–53761
[11]pencs.f.,pomahacb.,winklert.,dorschnerr.a.,erikssone.,herndonm.,gallor.l.(1998)dermatansulfatereleasedafterinjuryisapotentpromoteroffibroblastgrowthfactor-2function.j.biol.chem.273,28116–28121
[12]trowbridgej.m.,rudisillj.a.,rond.,gallor.l.(2002)dermatansulfatebindsandpotentiatesactivityofkeratinocytegrowthfactor(fgf-7).j.biol.chem.277,42815–42820
[13]hsiaoj.c.,chungc.s.,changw.(1999)vacciniavirusenveloped8lproteinbindstocellsurfacechondroitinsulfateandmediatestheadsorptionofintracellularmaturevirionstocells.j.virol.73,8750–8761
[14]williamsr.k.,strauss.e.(1997)specificityandaffinityofbindingofherpessimplexvirustype2glycoproteinbtoglycosaminoglycans.j.virol.71,1375–1380
[15]bergefallk.,trybalae.,johanssonm.,uyamat.,naitos.,yamadas.,kitagawah.,sugaharak.,
[16]hataday,ohtay,horikoshik.hyperproductionandapplicationofα-agarasetoenzymaticenhancementofantioxidantactivityofporphyran.journalofagriculturalfoodandchemistry,2006,54(26):9895-9900.
[17]詹晓北,王卫平,朱莉.食用胶的生产、性能与应用[m].北京:中国轻工业出版社,2003:11-18.
[18]kloaregb,quatranors.1988.structureofthecellwallsofmarinealgaeandecophysiologicalfunctionsofthematrixpolysaccharides.oceanogrmarbiolannurev26:259-315.
[19]andrykovitchg,marxi.isolationofanewpolysaccharide-digestingbacteriumfromasaltmarsh.appliedandenvironmentalmicrobiology,1988,54(4):1061-1062.
[20]ekborgna,gonzalezjm,howardmb,etal.saccharophagusdegradansgen.novsp.nov.,aversatilemarinedegraderofcomplexpolysaccharides.internationaljournalofsystematicandevolutionarymicrobiology,2005,55(4):1545-1549.
[21]wenjunhan,jingyangu,qiujieyan,etal.apolysaccharide-degradingmarinebacteriu-m,flammeovirgasp.my04anditsextracellularagarosesystem.journalofoceanuniversityofchina.2012,11(3):375-382.
[22]韩文君,赵帅,刘会会等.一株多糖降解菌的分离、鉴定与琼脂糖降解能力[j].微生物学报,2012,52(6):776-783.
[23]jungangli,yuanyuancheng,dandanwang,jungeli,yanhuiwang,wenjunhan,fuchuanli.draftgenomesequenceofthepolysaccharide-degradingmarinebacteriumpseudoalteromonassp.straina601.genomeannouncements.2017,5(29):e00590-17.
[24]李福川,韩文君,王文爽,赵梅.一株弧菌fc509菌株及其培养方法与应用.专利公开号cn103923857a.
[25]taylorle,henrissatb,coutinhopm,ekborgna,hutchesonsw,weinerrm.compl-etecellulasesysteminthemarinebacteriumsaccharophagusdegradans2-40.journalofbacteriology,2006,188(11):3849-3861.
[26]ekborgna,taylorle,longmireag,henrissatb,weinerrm,hutchesonsw.genomicandproteomicanalysesoftheagarolyticsystemexpressedbysaccharophagusdegradans2-40.appliedenvironmentalmicrobiology,2006,72(5):3396-3405.
[27]howardmb,ekborgna,taylorle,weinerrm,hutchesonsw.genomicanalysisandinitialcharacterizationofthechitinolyticsystemofmicrobulbiferdegradansstrain2-40.journalofbacteriology,2003,185(11):3352-3360.
[28]weinerrm,taylorle,henrissatb,hauserl,landm,coutinhopm,rancurelc,saunderseh,longmireag,zhanght,bayerea,gilberthj,larimerf,zhulinib,ekborgna,lamedr,richardsonpm,borovoki,hutchesons.completegenomesequenceofthecomplexcarbohydrate-degradingmarinebacterium,saccharophagusdegradansstrain2-40t.plosgenetics,4(5):e1000087.doi:10.1371/journal.pgen.1000087.
[29]barbeyron,t,l’haridon,s,corre,e,etal.zobelliagalactanovoransgen.nov.,sp.nov.,amarinespeciesofflavobacteriaceaeisolatedfromaredalga,andclassificationof[cytophaga]uliginosa(zobellandupham1944)reichenbach1989aszobelliauliginosagen.nov.,comb.nov.internationaljournalofsystematicandevolutionarymicrobiology,51,985-997.
[30]han,w.,wang,w.,zhao,m.,sugahara,k.,andli,f.(2014)anoveleliminasefromamarinebacteriumthatdegradeshyaluronanandchondroitinsulfate.j.biol.chem.289,27886-27898.
[31]wang,w.,han,w.,cai,x.,zheng,x.,sugahara,k.,andli,f.(2015)cloningandcharacterizationofanovelchondroitinsulfate/dermatansulfate4-o-endosulfatasefromamarinebacterium.j.biol.chem.290,7823-7832.
[32]wang,w.,cai,x.,han,n.,han,w.,sugahara,k.,andli,f.(2017)sequencingofchondroitinsulfateoligosaccharidesusinganovelexolyasefromamarinebacteriumthatdegradeshyaluronanandchondroitinsulfate/dermatansulfate.biochem.j.474,3831-3848
[33]peng,c.,wang,q.,wang,s.,wang,w.,jiao,r.,han,w.,andli,f.(2018)achondroitinsulfateandhyaluronicacidlyasewithpooractivitytoglucuronyl4,6-o-disulfatedn-acetylgalactosamine(e-type)–containingstructures.j.biol.chem.293(12):4230-4243.
[34]王文爽.海洋细菌来源的新型糖胺聚糖降解酶的筛选、鉴定与应用[d].济南:山东大学博士学位论文,2017.
[35]ariellewoznica,josephp.gerdt,ryane.hulett,jonclardy,andnicoleking.matingintheclosestlivingrelativesofanimalsisinducedbyabacterialchondroitinase.cell170,1175–1183,september7,2017.
[36]lees,katoj,takiguchin,etal.applenvironmicrobiol,2000,66(1):4334-4339.
[37]gauthierg,gauthierm,christenr.phylogeneticanalysisofthegeneraalteromonas,shewanella,andmoritellausinggenescodingforsmall-subunitrrnasequencesanddivisionofthegenusalteromonasintotwogenera,alteromonas(emended)andpseudoalteromonasgen.nov.,andproposaloftwelvenewspeciescombinations[j].internationaljournalofsystematicbacteriology,1995,45(4):755-761.
[38]holmstromc,kjcllcbergs.marinepseudoalteromonasspeciesareassociatedwithhigherorganismsandproducebiologicallyactiveextracellularagents[j].femsmicrobiologyecology,1999,30(4):285-293.
[39]holmstromc,egans,franksa,etal.antifoulingactivitiesexpressedbymarinesurfaceassociatedpseudoalteromonasspecies[j].femsmicrobiologyecology,2002,41(1):47-58.
[40]isnansetyoa,kameiys.pseudoalteromonasphenolicasp.nov.anovelmarinebacteriumthatproducesphenolicanti-methicillin-resistantstaphylococcusaureussubstancees[j].internationaljournalofsystematicandevolutionarymicrobiology,2003,53:583-588.
[41]carolinacr,rowleyaf.effectofextracellularproductsofpseudoalteromonasatlanticaontheediblecrabcancerpagurus[j].appliedandenvironmentmicrobiology,2004,70(2):729-735.
[42]parkyd,baikks,yih,etal.pseudoalteromonasbyunsanensissp.nov.,isolatedfromtidalflatsedimentinkorea[j].internationaljournalofsystematicandevolutionarymicrobiology,2005,55(pt6):2519-2523.
[43]veraj,alvarezr,muranoe,etal.applenvironmicrobiol,1998,64(11):4378-4383.
[44]fernandess,geuekeb,delgadoo,etal.applmicrobiolbiotechnol,2002,58:313-321.
[45]tutinoml,parrillie,giaquintol,etal.journalofbacteriology,2002,5814-5817.
[46]truonglv,tuyenh,helmke,etal.extremophiles,2001,5:35-44.
sequencelisting
<110>山东大学
<120>一株假交替单胞菌属多糖降解菌及其培养方法与应用
<160>1
<170>patentinversion3.5
<210>1
<211>1464
<212>dna
<213>假交替单胞菌(pseudoalteromonassp.)
<400>1
attgaacgctggcggcaggcctaacacatgcaagtcgagcggtaacagagaggtgcttgc60
acctttgctgacgagcggcggacgggtgagtaatgcttgggaatatgccttttggtgggg120
gacaacagttggaaacgactgctaataccgcatgatgtctacggaccaaagtgggggacc180
ttcgggcctcacgccaaaagattagcccaagtgggattagctagttggtgaggtaatggc240
tcaccaaggcgacgatccctagctggtttgagaggatgatcagccacactgggactgaga300
cacggcccagactcctacgggaggcagcagtggggaatattgcacaatgggcgcaagcct360
gatgcagccatgccgcgtgtgtgaagaaggccttcgggttgtaaagcactttcagtaagg420
aggaaaggttaagtgttaatagcacttagctgtgacgttacttacagaagaagcaccggc480
taactccgtgccagcagccgcggtaatacggagggtgcgagcgttaatcggaattactgg540
gcgtaaagcgtacgcaggcggtttgttaagcgagatgtgaaagccccgggctcaacctgg600
gaactgcatttcgaactggcaaactagagtgtgatagagggtggtagaatttcaggtgta660
gcggtgaaatgcgtagagatctgaaggaataccgatggcgaaggcagccacctgggtcaa720
cactgacgctcatgtacgaaagcgtggggagcaaacaggattagataccctggtagtcca780
cgccgtaaacgatgtctactagaagctcggttcctcggaactgtttttcaaagctaacgc840
attaagtagaccgcctggggagtacggccgcaaggttaaaactcaaatgaattgacgggg900
gcccgcacaagcggtggagcatgtggtttaattcgatgcaacgcgaagaaccttacctac960
acttgacatacagagaacttactagagatagtttggtgccttcgggaactctgatacagg1020
tgctgcatggctgtcgtcagctcgtgttgtgagatgttgggttaagtcccgcaacgagcg1080
caacccctatccttagttgctagcaggtaatgctgagaactctaaggagactgccggtga1140
taaaccggaggaaggtggggacgacgtcaagtcatcatggcccttacgtgtagggctaca1200
cacgtgctacaatggcgcatacagagtgctgcgaactcgcgagagtaagcgaatcactta1260
aagtgcgtcgtagtccggattggagtctgcaactcgactccatgaagtcggaatcgctag1320
taatcgcgtatcagaatgacgcggtgaatacgttcccgggccttgtacacaccgcccgtc1380
acaccatgggagtgggttgctccagaagtagatagtctaaccctcgggaggacgtttacc1440
acggagtgattcatgactggggtg1464
- 还没有人留言评论。精彩留言会获得点赞!