一种耐盐碱反硝化微生物菌株的分离方法与流程
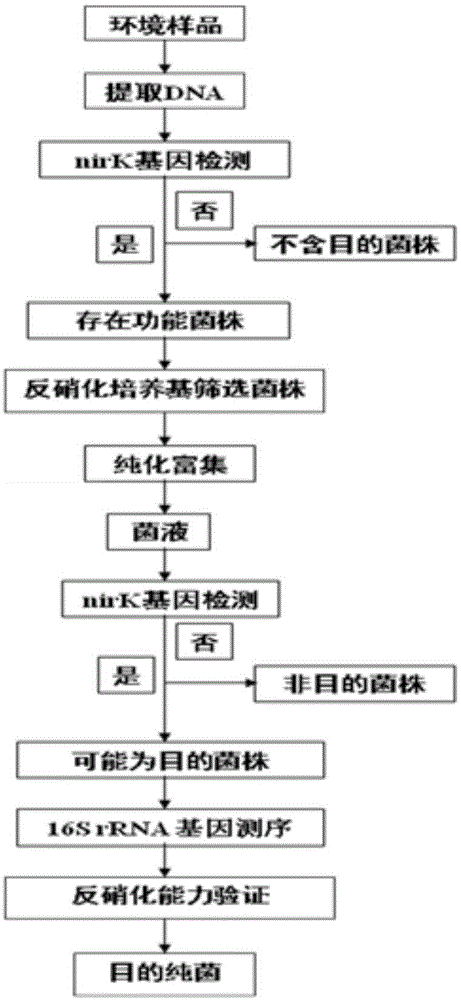
本发明涉及生物
技术领域:
,具体涉及一种耐盐碱反硝化微生物菌株的分离方法。
背景技术:
:近年来,由化肥流失产生的废水、养殖废水、生活污水及工业废水等都会引起水体的氮污染造成的水体氮污染日趋严重,水体中溶解态氮的主要存在方式是主硝态氮和铵态氮,现今许多行业排放的废水中含有高浓度的硝酸盐和亚硝酸盐,如化肥、钢铁、制药和饲料生产等,这些含氮污水有些具有高盐高碱的特点,由于污水自身代谢周期长,当氮污染物积累到一定程度时,会出现水体富营养化,对生态环境造成了巨大污染。自然生态系统中,微生物进行的硝化和反硝化作用是将化合态氨转化为气态氮,从而去除氮污染物的主要途径。硝化作用是将铵态氮转化为硝态氮的过程,反硝化作用可将硝态氮还原为n2o或n2回到大气中,在去氮过程中硝化作用只改变了氮素的存在形式,而反硝化作用是真正的脱氮过程,其中反硝化微生物起到至关重要的作用,可为水体中氮的去除提供有效手段,可以降低水体中的含氮污染物浓度,使水体富营养化的发生机率降低,且能够减轻因硝酸盐或亚硝酸盐积累对生物产生的毒害作用,有利于水质保护。反硝化作用是由反硝化微生物驱动完成的,其重要步骤是亚硝酸盐还原为一氧化氮,nir是该步骤的限速酶,因此,nir基因是研究反硝化微生物关键的功能基因,其中nirk成为研究反硝化微生物的重要的分子标记。关于反硝化微生物菌株分离的研究很多,但现有技术中披露的反硝化菌株不具有耐盐碱的特性,高盐碱的极端环境会降低非嗜盐碱微生物的活性,现有处理高盐高碱污水的过程中,现有的反硝化微生物菌株去除氮污染物的效果并不理想;因此,高效分离耐盐碱反硝化微生物菌株重要性日趋凸显,分离培养高盐高碱这一特殊环境中的反硝化微生物,并研究其特性,能为高盐碱废水的生物脱氮提供参考,需要对其进行深入研究。技术实现要素:有鉴于此,本发明的目的在于提供一种快速高效的分离耐盐碱反硝化微生物菌株的方法,所述方法为在较短周期内快速筛选盐碱土壤样品,根据筛选出的样品制备特异性反硝化分离培养基来获得反硝化菌株,并验证其反硝化能力,从而获得具有高效反硝化能力的耐盐碱菌株。为实现上述目的,本发明提供一种耐盐碱反硝化微生物菌株的分离方法,包括以下步骤:(1)样品筛选:采用dna抽提试剂盒提取环境样品中微生物的总dna,然后针对反硝化的特异基因nirk基因的序列,通过特异性引物对dna进行pcr扩增,筛选出含有反硝化微生物菌株的样品;(2)菌落分离培养:将步骤(1)中筛选出的样品在反硝化固体培养基中进行分离培养后得到单菌落,挑取纯化后的单菌落接种于液体培养基中进行富集培养得到纯菌株;(3)反硝化菌的验证:对纯菌株的dna进行pcr扩增以及电泳检测,扩增出nirk基因条带的菌株即为反硝化菌株。进一步地,本发明步骤(1)中,所述的pcr扩增特异性引物为nirkc1、nirkc2、nirkc3和nirkc4;其引物序列如序列表中seq.id.no:1-8所示所述引物nirkc1的扩增程序为95℃预热5min、95℃变性30s、54℃退火30s、72℃延伸40s、循环35次和72℃延伸10min;所述引物nirkc2的扩增程序为:95℃预热5min、95℃变性30s、56℃退火30s、72℃延伸40s、循环35次和72℃延伸10min;所述引物nirkc3的扩增程序为:95℃预热5min、95℃变性30s、58℃退火30s、72℃延伸40s、循环35次和72℃延伸10min;所述引物nirkc4的扩增程序为:95℃预热5min、95℃变性30s、60℃退火30s、72℃延伸40s、循环35次和72℃延伸10min。步骤(1)所述的pcr扩增的反应体系均为:缓冲液(buffer)2.5μl、mg2+(25mmol/l)2μl、dntp2μl、primerf0.5μl、primerr0.5μl、bsa(5mg/ml)2.5μl、taq酶0.1μl、dna模板1μl,并用ddh2o定容至25μl。进一步地,本发明步骤(2)中所述的反硝化固体培养基,以1l计,组成为:氯化钠5g,硝酸钠0.425g,亚硝酸钠0.345g,七水硫酸镁0.2g,磷酸氢二钾0.5g,琥珀酸钠0.54g,乙酸纳0.164g,草酸钠0.268g,丙酮酸钠0.22g,琼脂15g和余量的蒸馏水;培养基ph为9;步骤(2)中所述的液体培养基组分与反硝化固体培养基组分区别为液体培养基中不含琼脂。进一步地,步骤(2)中,所述的分离培养或富集培养的温度均为27℃。本发明步骤(3)中所述的特异性引物与步骤(1)中所述的特异性引物相同。进一步地,在上述技术方案中,所述步骤还包括对步骤(3)中得到的反硝化菌株的种属进行测序鉴定或/和对步骤(3)中得到的反硝化菌株进行反硝化能力测定,进一步筛选出具有反硝化能力的菌株。所述的测序鉴定为对反硝化菌株的dna进行pcr扩增,扩增后进行16s测序。所述的pcr扩增体系为(25μl):pcr缓冲液2.5μl,dntps2μl,模板dna1μl,正反向引物各0.5μl,taqpolymerase0.1μl,mgcl23μl,bsa(5mg/ml)2.5μl,ddh2o12.9μl;所述的pcr扩增条件为:95℃预变性5min;95℃变性30s,55℃退火30s,72℃延伸40s,25个循环;72℃延伸10min;所述的pcr扩增特异性引物为:27f和1541r,27f引物序列如序列表中seq.id.no:9所示,1541r引物序列如序列表中seq.id.no:10所示。进一步地,所述的反硝化能力测定为用离子色谱法和/或气相色谱法分别测定反硝化菌株的硝酸盐氮还原能力和/或产气的情况。所述离子色谱法条件包括:色谱柱:pursuit5c18尺寸为4.0mm×250mm9μm;流动相为5%硫酸、1.7mmol/l碳酸氢钠与1.8mmol/l碳酸钠混合液和超纯水的混合液,流速为1ml/min,样品过膜孔径为0.22μm。所述的气相色谱法中色谱柱为hp-plot/q,尺寸为30.0mm×0.530mm×40.0μm,进样量为1μl,载气为氦气,助燃气为空气,流速为5ml·min-1,ecd温度300℃,tcd温度200℃。与现有技术相比,本发明的有益效果是:与现有分离方法相比,本发明所述的耐盐碱反硝化微生物菌株的分离方法通过四组特异性引物的pcr扩增,有针对性的分离获得具有反硝化nirk基因的纯菌株,对其进行测序鉴定,并确定了种属,通过测定在盐碱条件下筛选出的具有反硝化nirk基因的菌株对硝酸盐氮的还原能力及产气的情况,验证了反硝化能力,确保获得的目的菌株在盐碱环境中具有反硝化能力;解决了现有方法中获得的反硝化菌株在盐碱条件下不具有高效反硝化能力,不能适应于盐碱环境的问题,填补了耐盐碱反硝化菌株的空白。附图说明图1为分离方法的工艺流程图;图2为离子色谱测定的硝酸盐降解率;图3为经气相色谱测定的n2o与n2o+n2的比值。具体实施方式下面结合附图和具体实施方式对技术方案作进一步详细说明。本发明所选的目标样品为盐碱土壤,对非本发明所述样品中的反硝化微生物的分离具有通用性,可参考本实施方式进行。以下实施方式仅用于说明本发明,而不用于限制本发明所要求保护的范围。实施例1一种耐盐碱反硝化微生物菌株的分离方法,分离方法流程如图1所示,包括以下步骤:(1)盐碱土壤样品的筛选:采用dna抽提试剂盒按说明书进行操作,提取东北黑龙江安达市苏打盐碱土壤环境样品中微生物的总dna,然后针对反硝化的特异基因nirk基因的序列,采用四套特异性引物对dna进行pcr扩增;引物、程序如表1所示,表1.反硝化特异基因nirk基因的pcr扩增引物、序列及扩增程序表引物序列扩增程序1nirkc1f/nirkc1r5’-atggcgccatcatggtnytncc-3’/5’-tcgaaggcctcgatnarrttrtg-3’95℃预热5min、95℃变性30s、54℃退火30s、72℃延伸40s、循环35次和72℃延伸10min2nirkc2f/nirkc2r5’-tgcacatcgccaacggnatgtwygg-3’/5’-ggcgcggaagatgshrtgrtcnac-3’95℃预热5min、95℃变性30s、56℃退火30s、72℃延伸40s、循环35次和72℃延伸10min3nirkc3f/nirkc3r5’-catcggcaacggcatgyayggngc-3’/5’-cgaccatggcgtggswnacraangg-3’95℃预热5min、95℃变性30s、58℃退火30s、72℃延伸40s、循环35次和72℃延伸10min4nirkc4f/nirkc4r5’-tacggtgtgatcatcrtsgatcc-3’/5’-gcatcacgcatggaatgatysac-3’95℃预热5min、95℃变性30s、60℃退火30s、72℃延伸40s、循环35次和72℃延伸10min上述引物pcr扩增的反应体系均为:buffer2.5μl、mg2+(25mmol/l)2μl、dntp2μl、primerf0.5μl、primerr0.5μl、bsa(5mg/ml)2.5μl、taq酶0.1μl、dna模板1μl,并用ddh2o定容至25μl,若没有nirk基因的序列,则将该样品淘汰,筛选出含有反硝化微生物菌株的盐碱土壤样品。(2)称取上述筛选出的含有反硝化微生物菌株的盐碱土样1g,用9ml无菌水梯度稀释,取200ul涂布于反硝化固体培养基平板上,放置在含有2个耗氧袋的密封培养箱中,在27℃培养箱内培养,其中,反硝化固体培养基由nacl5g,nano30.425g,琥珀酸钠0.54g,乙酸纳0.164g,草酸钠0.268g,丙酮酸钠0.22g,琼脂15g和1000ml蒸馏水组成,121℃灭菌20min,灭菌后用na2co3将ph调至9。待培养一段时间后,将平板上生长的菌体进行纯化富集培养,挑取单菌落在反硝化固体培养基上纯化后,将单菌落转接至反硝化液体培养基(反硝化液体培养基与反硝化固体培养基组分区别为反硝化液体培养基组分中不含琼脂),富集得到纯化菌株菌液。(3)反硝化菌的验证:重复步骤(1)中所述的pcr扩增方法对步骤(2)中得到的纯化菌株的dna进行pcr扩增,然后进行电泳检测,扩增出nirk基因条带的即为反硝化菌株。实施例2:反硝化菌株的种属测序鉴定利用通用引物27f和通用引物1541r对具有nirk基因的菌株进行16srrna基因的pcr扩增,27f引物序列如序列表中seq.id.no:9所示,即:5′-agagtttgatcctggctcag-3′;1541r引物序列如序列表中seq.id.no:10所示,即:5′-aaggaggtgatccagccgca-3′,试剂均产自takara试剂公司,扩增体系为(25μl):pcr缓冲液2.5μl,dntps2μl,模板dna1μl,正反向引物各0.5μl,taqpolymerase0.1μl,mgcl23μl,bsa(5mg/ml)2.5μl,ddh2o12.9μl;扩增条件为95℃预变性5min;95℃变性30s,55℃退火30s,72℃延伸40s,25个循环;72℃延伸10min。pcr产物送至上海生工测序鉴定。测序结果利用sepman软件拼接,确定反硝化菌株的种属。对东北黑龙江安达市苏打盐碱土壤环境样品中微生物的总dna进行分离筛选后获得13株具有反硝化能力的菌株,对其16srrna基因测序结果如表2所示。表2.反硝化菌株的16srrna基因测序结果对照表菌株编号最近似菌株相似度(%)1ensifersp.ccbau6572999.622bradyrhizobiumsp.ccbau5336399.373ralstoniasp.nfacc0198.964ralstoniasp.nfacc0199.515ensifersp.ccbau6572999.356boseasp.lmg2638398.997pseudoduganellasp.10r5-2199.238rhodoferaxsp.jcm781998.789rhodoferaxsp.jcm781998.4710rhodoferaxsp.jcm781998.211alcaligenessp.lmg2299610012pseudomonassp.jcm596299.8613halomonassp.althf1100序列比对结果表明这13株菌分属于rhodoferax、ensifer、bradyrhizobium、ralstonia、bosea、massilia、alcaligenes、pseudomonas以及halomonas。实施例3:反硝化能力的测定将步骤(3)中获得的反硝化菌株转接在反硝化液体培养基中富集培养,在小瓶中加入5ml反硝化液体培养基,接入5ul菌液后抽真空,充氦气,27℃密封培养一周,抽取气体进行气相色谱检测,色谱柱hp-plot/q(30.0m×0.530mm×40.0μm),进样量1μl,载气(he),助燃气空气,流速5ml·min-1,ecd温度300℃,tcd温度200℃。取菌液进行离子色谱仪的检测,色谱柱pursuit5c18(9μm,4.0mm×250mm),流动相为5%硫酸、1.7mmol/l碳酸氢钠与1.8mmol/l碳酸钠混合液、超纯水三者的混合物,流速1ml/min。所获得的13株反硝化菌在盐碱环境中对硝酸盐的降解率如图2所示,从图2中可以看出在ph=9.0时,菌株编号6~13对硝酸盐有较好的降解效果,硝酸盐降解率为55%~100%,其中编号11、13的硝酸盐降解率达到100%。进一步验证菌株编号6~13的产气情况,经气相色谱法测定n2o与n2o+n2的比值,如图3所示,在厌氧条件下都可以产生n2和n2o,表明这8株菌在盐碱环境中都具有较强的反硝化能力。尽管已经示出和描述了本发明的实施例,本领域的普通技术人员可以理解:在不脱离本发明的原理和宗旨的情况下可以对这些实施例进行多种变化、修改、替换和变型,本发明的范围由权利要求及其等同物限定。序列表<110>江苏大学<120>一种耐盐碱反硝化微生物菌株的分离方法<160>10<170>siposequencelisting1.0<210>1<211>22<212>dna<213>人工序列(artificialsequence)<400>1atggcgccatcatggtnytncc22<210>2<211>23<212>dna<213>人工序列(artificialsequence)<400>2tcgaaggcctcgatnarrttrtg23<210>3<211>25<212>dna<213>人工序列(artificialsequence)<400>3tgcacatcgccaacggnatgtwygg25<210>4<211>24<212>dna<213>人工序列(artificialsequence)<400>4ggcgcggaagatgshrtgrtcnac24<210>5<211>24<212>dna<213>人工序列(artificialsequence)<400>5catcggcaacggcatgyayggngc24<210>6<211>25<212>dna<213>人工序列(artificialsequence)<400>6cgaccatggcgtggswnacraangg25<210>7<211>23<212>dna<213>人工序列(artificialsequence)<400>7tacggtgtgatcatcrtsgatcc23<210>8<211>23<212>dna<213>人工序列(artificialsequence)<400>8gcatcacgcatggaatgatysac23<210>9<211>20<212>dna<213>人工序列(artificialsequence)<400>9agagtttgatcctggctcag20<210>10<211>20<212>dna<213>人工序列(artificialsequence)<400>10aaggaggtgatccagccgca20序列表<110>江苏大学<120>一种耐盐碱反硝化微生物菌株的分离方法<160>10<170>siposequencelisting1.0<210>1<211>22<212>dna<213>人工序列(artificialsequence)<400>1atggcgccatcatggtnytncc22<210>2<211>23<212>dna<213>人工序列(artificialsequence)<400>2tcgaaggcctcgatnarrttrtg23<210>3<211>25<212>dna<213>人工序列(artificialsequence)<400>3tgcacatcgccaacggnatgtwygg25<210>4<211>24<212>dna<213>人工序列(artificialsequence)<400>4ggcgcggaagatgshrtgrtcnac24<210>5<211>24<212>dna<213>人工序列(artificialsequence)<400>5catcggcaacggcatgyayggngc24<210>6<211>25<212>dna<213>人工序列(artificialsequence)<400>6cgaccatggcgtggswnacraangg25<210>7<211>23<212>dna<213>人工序列(artificialsequence)<400>7tacggtgtgatcatcrtsgatcc23<210>8<211>23<212>dna<213>人工序列(artificialsequence)<400>8gcatcacgcatggaatgatysac23<210>9<211>20<212>dna<213>人工序列(artificialsequence)<400>9agagtttgatcctggctcag20<210>10<211>20<212>dna<213>人工序列(artificialsequence)<400>10aaggaggtgatccagccgca20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1