一种中国鲎SSR引物组及其应用的制作方法
【
技术领域:
】本发明涉及分子标记
技术领域:
,特别涉及一种中国鲎ssr引物组及其应用。
背景技术:
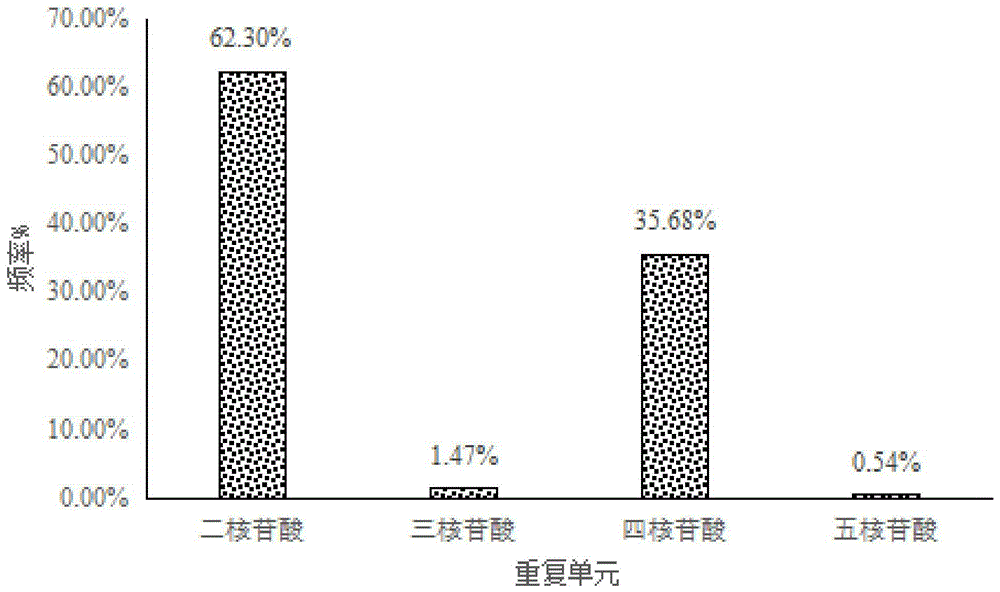
:中国鲎具有很高的经济及医药价值,以其血液为原料制作的鲎试剂可用于检测细菌内毒素,其肉、壳、尾皆可入药,在我国广西、海南、广东、香港、福建、台湾、浙江等沿海省份和地区均有分布。近年来,由于过度捕捞和环境恶化等人为或非人为因素导致中国鲎数量明显下降,频临灭绝。对于一个物种而言,其遗传多样性越高表明其对生存环境变化的潜在适应能力越强,中国鲎物种保护的实质在于保护其遗传的多样性,开展中国鲎遗传多样性研究对于保护我国这一物种资源具有十分重要的意义。现存的中国鲎主要分布于北部湾海域,对该海域中国鲎多样性的调查和物种资源的保护最为迫切。本研究利用ssr分子标记揭示北部湾中国鲎遗传多样性,掌握北部湾海域中国鲎的遗传多样性及其遗传结构,为中国鲎的保护提供策略和可靠的科学依据。中国鲎物种保护的内在实质就是对其遗传多样性的保护,目前,国内对中国鲎的研究多见于资源分布及保育和鲎试剂及重组鲎素抗菌肽方面。研究中国鲎的技术方法相对较少,从微卫星标记(microsatellitedna)分离方面对中国鲎进行研究的报道较为少见,由于中国鲎品种保护的缺陷,使得适合中国鲎生长的环境资源日益衰退,因此,目前用于中国鲎类研究的样本很少,难以很准确的反映中国鲎遗传多样性的真实情况;同时也表明目前对中国鲎的遗传多样性处于起步阶段,这些结果尚不能为中国鲎的保护提供有力的科学依据,为了能全面有效的对中国鲎进行研究,有必要开展本申请的研究工作,为中国鲎的ssr标记开展新的研究思路。技术实现要素:鉴于上述内容,有必要提供一种中国鲎ssr引物组及其应用,该引物能为今后开展遗传育种、基因组作图、基因定位和物种亲缘关系鉴别等研究提供新的思路。为达到上述目的,本发明所采用的技术方案是:一种中国鲎ssr分子标记引物组,所述ssr引物组的序列如seqidno.1~328所示。本申请还提供一种应用所述ssr分子标记引物组进行遗传分析的应用,所述应用可用于中国鲎的聚类分析。本申请还提供一种应用所述ssr分子标记引物组进行遗传分析的应用,所述应用可用于不同中国鲎品种遗传多样性。本申请还提供一种应用所述ssr分子标记引物组进行中国鲎聚类分析的方法,所述方法包括如下步骤:(1)中国鲎基因组的提取:对鲎血和鲎肌肉的基因组dna进行提取,并稀释成20ng·μl-1,置于-20℃冰箱保存备用;(2)设计样品中国鲎的ssr引物:对步骤(1)的dna进行测序,测序完成后进行质量控制,构建中国鲎的unigene序列;对ssr位点检索,获得并设计一系列ssr位点的引物;(3)对步骤(2)的ssr引物进行检测,得出164对ssr引物组;(4)中国鲎群体扩增:用步骤(3)的引物组中20对多态引物对样本鲎群体204个个体进行pcr扩增分析,将读取的条带信息应用genalex6.502软件进行统计分析;估算不同地域品种来源的中国鲎其等位基因数(na)、有效等位基因数(ne)、观测杂合度(ho)、期望杂合度(he)和pic值,同时,计算出遗传距离;以upgma算法对中国鲎进行聚类分析。进一步的,其特征在于,所述步骤(4)的pcr扩增反应体系为10μl,其组分为:1.0μl的模板dna、各0.4μl的上下游引物(10mmol/l)、1.0μl的dntps(2.5mmol/l)、1.0μl的10×buffer、1.5u的rtaq、最后加入6.2μl的ddh2o补足10μl。进一步的,其特征在于,所述步骤(4)的pcr扩增反应程序为:预变性95℃4min,变性95℃45sec,退火55℃45sec,延伸72℃45sec,33个循环,72℃延伸7min,4℃保存;或:预变性95℃4min,变性95℃45sec,退火60℃45sec,延伸72℃45sec,33个循环,72℃延伸7min,4℃保存。进一步的,其特征在于:所述步骤(4)的不同地域为钦州、北海和防城港。本发明具有如下有益效果:本发明以鲎血和鲎肌肉为材料,提取中国鲎的dna经过检测、分析得到相应的ssr引物,找到适合应用于中国鲎聚类分析的ssr引物组,本引物组具有较高的pic值,利用ssr引物组进行pcr分析并绘制聚类分析图,为今后开展遗传育种、基因定位、物种亲缘关系鉴别和中国鲎遗传资源保护等研究提供新的引物和思路。【附图说明】图1是本发明实施例ssr标记开发中四种核苷酸重复单元出现的频率;图2是本发明实施例ssr标记开发中重复单元的组成成分;图3是本发明实施例参试鲎聚类分析图。【具体实施方式】为使本发明的上述目的、特征和优点能够更加明显易懂,下面结合附图对本发明的具体实施方式做详细的说明。在下面的描述中阐述了很多具体细节以便于充分理解本发明。但是本发明能够以很多不同于在此描述的其它方式来实施,本领域技术人员可以在不违背本发明内涵的情况下做类似改进,因此本发明不受下面公开的具体实施的限制。实施例:一、实验方法:1、鲎基因组dna的提取方法:分别采用天根生化科技(北京)有限公司生产的血液组织基因组dna提取试剂盒(dp318-02)和海洋动物组织基因组dna提取试剂盒(dp324-03)对鲎血和鲎肌肉的基因组dna进行提取,并稀释成20ng·μl-1,置于-20℃冰箱保存备用。2、中国鲎ssr引物组的设计:以步骤1的中国鲎dna为材料构建300bp和500bp两个基因组文库。首先利用illuminahiseq4000平台对文库进行测序,测序完成后进行序列组装得到的序列总大小为318451264bp,序列总数为216448条,每个序列平均长度为1471bp,利用trimmomatic对测序结果进行质量检测,去除污染序列、接头和低质量序列得到cleandata;再利用soapdenovo对cleandata进行组装得到unigenes;接下来利用misa脚本在unigenes中搜索ssr位点,识别出含有ssr位点的序列总数为2581条,其中每个序列中包含多个ssr位点的序列为35条,每个序列中仅含1个ssr位点的有2546条,平均每123382bp或每84条序列中就有一个ssr位点,设计相应ssr位点的引物,从已开发出的只含有一个ssr位点的2546条序列中,筛选出了km为55℃的共62对引物和km为60℃的共142对引物,总共设计了204对引物。3、ssr多态引物筛选设置二核苷酸、三核苷酸、四核苷酸、五核苷酸、六核苷酸五个重复单元。从2581条包含有ssr位点的序列中筛选出的重复单元为二核苷酸的有1608条序列,占比62.30%、三核苷酸的有38条,占比1.47%、四核苷酸的有921条,占比35.68%、五核苷酸14条,占比0.54%图1;具体的重复单元类型和数量见图2。从只含有一个ssr位点的2546条序列中筛选出了204对引物,合成的204对引物经电泳检验有40对引物无产物,占20%,164对引物有产物,其中23对引物无多态,141对引物有多态,分别占11%和69%。引物中从合成引物中筛选出了20对多态性最好的引物作为群体扩增的引物,占合成引物数量的9.8%。经pcr扩增和凝胶电泳筛选出多态引物,从204对引物中筛选出了20对多态性为稳定且条带信息明显的引物,本研究最终选用于群体扩增的引物序列信息见表1:表1本研究中筛选出用于群体扩增的引物序列信息(seqidno.1~40)5、群体扩增:用20对引物对样本鲎群体204个个体进行pcr扩增分析,将读取的条带信息应用genalex6.502软件进行统计分析。上述pcr扩增的反应体系为:pcr扩增反应体系规格为10μl,其组分为:1.0μl的模板dna、各0.4μl的上下游引物(10mmol/l)、1.0μl的dntps(2.5mmol/l)、1.0μl的10×buffer、1.5u的rtaq、最后加入6.2μl的ddh2o补足10μl。引物送生工生物工程(上海)股份有限公司合成,第一批合成tm值为55℃的引物62个,第二批合成tm值为60℃的引物142个。根据引物tm值的不同,分别设置两个pcr反应程序:①tm值为55℃的pcr程序设置为:预变性95℃4min,变性95℃45sec,退火55℃45sec,延伸72℃45sec,33个循环,72℃延伸7min,4℃保存;①tm值为60℃的pcr程序设置为:预变性95℃4min,变性95℃45sec,退火60℃45sec,延伸72℃45sec,33个循环,72℃延伸7min,4℃保存。二、结果与分析:1、遗传多样性参数:实验结果如表2所示,经表1的20对引物检测后得出了208个等位基因,如表2所示,每个ssr位点的等位基因数(na)在6~19个之间,平均每个位点有10.4个等位基因,引物tt-req-43扩增出的等位基因数最多为19个,引物tt-req-40扩增出的等位基因数最少为6个;每个位点有效等位基因在3~9范围之间,平均有效等位基因为6个;观测杂合度位于0.0735~0.8777范围之内,平均值为0.5801;期望杂合度位于0.6708~0.8912范围之内,平均值为0.8203,观测杂合度明显低于期望杂合度,说明存在杂合度缺失。通过powermarker3.25软件计算得出多态信息含量(pic),从参试样本来看,多态信息含量均值为0.7974,其中pic最大的位点是tt-req-137,为0.8810;最小的是tt-req-40,为0.6316。有10个位点的pic值大于0.8;2个在0.6~0.7之间。20个ssr位点在3个地理群体鲎中的多样性信息分别见表3、4、5,在钦州群体中的平均观测杂合度(ho)为0.5803,平均期望杂合度(he)为0.7893;北海群体的平均观测杂合度为0.6828,平均期望杂合度为0.7556;防城港群体的平均观测杂合度为0.5018,平均期望杂合度为0.6711。表23个地理鲎群体在20个微卫星位点中的遗传多样性参数表3钦州鲎群体的遗传多样性参数表4北海鲎群体的遗传多样性参数位点等位基因数(na)有效等位基因数(ne)观测杂合度(ho)期望杂合度(he)tt-req-4254.36630.66670.7710tt-req-2963.29100.38100.6961tt-req-4053.09470.33330.6769tt-req-6696.48530.95240.8458tt-req-7275.09830.71430.8039tt-req-8142.60950.52380.6168tt-req-8697.35000.80950.8639tt-req-148105.97010.80000.8325tt-req-15052.47750.57140.5964tt-req-13884.76760.95240.7902tt-req-14553.50000.66670.7143tt-req-137107.36360.55560.8642tt-req-20075.08450.73680.8033tt-req-16476.04110.80950.8345tt-req-180106.39130.85710.8435tt-req-18665.51250.85710.8186tt-req-1752.92050.71430.6576tt-req-1143.57090.66670.7200tt-req-43145.94500.61110.8318tt-req-1072.13040.47620.5306mean7.154.69850.68280.7556表5防城港鲎群体在20个微卫星位点中的遗传多样性参数位点等位基因数(na)有效等位基因(ne)观测杂合度(ho)期望杂合度(he)tt-req-4242.74580.48150.6358tt-req-2963.10210.14810.6776tt-req-4052.86440.03700.6509tt-req-6695.08010.88890.8032tt-req-7251.64930.18520.3937tt-req-8142.28530.33330.5624tt-req-8673.08250.51850.6756tt-req-14894.97060.46150.7988tt-req-15042.43410.70370.5892tt-req-13863.79690.96300.7366tt-req-14553.24000.94440.6914tt-req-13782.50520.37040.6008tt-req-20063.53930.50000.7175tt-req-16472.96340.37040.6626tt-req-18052.89860.44440.6550tt-req-18675.70460.69230.8247tt-req-1764.13030.81480.7579tt-req-1164.31950.26920.7685tt-req-43127.46960.57690.8661tt-req-1061.54780.33330.3539mean6.353.51650.50180.67112、鲎群体遗传距离与遗传分化:参试个体之间最大遗传距离为0.9750,包括qz-t-3和fcg-c-13、qz-t-4和fcg-c-12、qz-t-4和fcg-c-13、qz-t-7和fcg-c-13、qz-t-35和fcg-c-12、qz-t-35和fcg-c-13、qz-t-38和fcg-c-13、qz-t-56和fcg-c-12共8对鲎;最小遗传距离的是fcg-c-12和fcg-c-14,为0.0789;平均遗传距离为0.7528。3个地理群体之间,防城港与北海之间鲎群体的遗传距离最小,为0.3656,最大的是钦州与防城港之间,为0.4896(见表6)。20个微卫星位点中遗传分化指数在0.022~0.216之间,最大位点的为tt-req-150,平均遗传分化指数为0.0923,大于0.05,表明3个鲎群体在各多态位点遗传分化较强。两两群体相比较,遗传分化指数以钦州和防城港鲎群体之间最大,为0.088;最小的是钦州与防城港之间,为0.054(表7),均大于0.05,表明3个鲎群体之间的遗传分化较强。基因流最大的位点是tt-req-210,为11.3173,最小的tt-req-150,为0.9057,平均基因流为3.2680。基因流最小的tt-req-150同时也是分化指数最大的。两两群体相比较,基因流最大的是钦州与防城港之间的鲎群体,为0.088;最小的是钦州与北海之间,为0.054。固定指数最大的是tt-req-40位点,为0.8936,只有位点tt-req-66和tt-req-138为负值,分别为-0.0117和-0.06其余均为正数,表明3个地理群体之间近交比较严重。表63个地理群体遗传距离表73个地理群体的基因流与遗传分化指数表820个微卫星位点中3个鲎群体固定指数、遗传分化指数与基因流3、聚类分析:应用软件mega6.0中的upgma方法根据遗传距离生成聚类图(见图3),通过观察聚类图发现钦州的中国鲎qz-t-53和圆尾鲎qz-c-2聚成一分支;北海中国鲎bh-t-17和防城港圆尾鲎fcg-c-10聚成一个分支,再与防城港中国鲎fcg-t-5聚成一支,最后与北海中国鲎bh-t-16聚在一起;北海中国鲎bh-t-13与防城港中国鲎fcg-t-11聚成一个分支,再与北海中国鲎bh-t-14聚在一起;钦州中国鲎qz-t-70和qz-t-73聚成一个分支,再与防城港中国鲎fcg-t-8聚在一起。这表明钦州中国鲎与圆尾鲎之间;北海中国鲎与防城港圆尾鲎和中国鲎之间;北海中国鲎与防城港中国鲎之间;钦州中国鲎与防城港中国鲎之间存在基因渐渗现象。4、遗传结构分析:运用软件structure2.3.4计算每一个个体源于某个特定群体的概率值q,以此分析鲎群体的遗传结构,具体操作过程为:假定输入的所有位点互为独立位点,先将不作数迭代的mcmc设置为10,000,再将不作数迭代后的mcmc设置为100,000,设定亚群数目参数k为2~10,从这9个不同的k值参数依据似然数最大原则选取合适的k值,即最为可能的亚群数。本研究中根据似然数最大原则取得k=9,将样本144只鲎划分为9个种群(表9)。钦州、北海、防城港各96、21、27只鲎分别被划分在了7、2、3个不同的种群中。其中个体数目最多的是种群2,包含了42只均来自钦州的个体,其中有41只中国鲎和1只圆尾鲎;个体数目最少的为种群8,包含了1只来自钦州的中国鲎和来自4只北海的圆尾鲎共5只个体;种群3中北海的中国鲎与防城港的中国鲎和圆尾鲎并存,反映了北海和防城港两地的鲎可能存在一定的基因交流,这与聚类分析的结果相一致;种群5中来自钦州的3只中国鲎与18只圆尾鲎被划分为同一个亚群,反映了钦州大风江沿海的中国鲎与圆尾鲎可能存在一定的基因交流。钦州的中国鲎和圆尾鲎;北海的中国鲎和防城港的中国鲎与圆尾鲎分别被划归为同一个亚群,同一地区的中国鲎和圆尾鲎各自划归为不同的亚群,同一个亚群中又有来自不同地区和不同种类的鲎,参试鲎群体呈现一个复杂的遗传关系。表9参试鲎亚群的划分三、分析:本申请的20个ssr标记在3个地理群体个体中检测出平均每个位点有10.4个等位基因;在钦州群体中平均等位基因数为9.1;在北海群体中平均等位基因数为7.15;在防城港群体中平均等位基因数为6.35。当等位基因数大于4,群体遗传多样性水平能被ssr标记较好的反映出来。多态信息含量(pic)是用于衡量分子遗传标记和群体遗传特征的重要参数,可衡量某一位点的基因变异程度。当某一位点的pic<0.25时,该位点为低度多态性位点;当0.25<pic<0.5时,为中度多态位点,当pic>0.5时,为高度多态性位点。3个地理群体在20个ssr位点中的pic均值为0.7974,pic值最大的为0.8810,最小的为0.6316,均大于0.5,表明本研究开发的ss位点均为高度多态位点,多态信息含量高,遗传变异较大。近亲繁殖会降低群体基因组中杂合子出现的频率,纯合子出现的频率增加,不利于维持较高水平的群体遗传变异和遗传多样性。本研究中北海鲎群体的平均观测杂合度最大,为0.6828,既北海鲎群体的遗传多样性较高;防城港鲎群体平均观测杂合度最小,为0.5018。钦州鲎群体的平均期待杂合度和平均观测杂合度相差最大,分别为0.7893、0.5803,表明钦州鲎群体之间遗传变异程度较高,受近交、杂交因素影响较大,存在较大的杂合度缺失,处于遗传不平衡状态。将三个地理群体视为一个大群体来看,平均观测杂合度为0.5801,平均期待杂合度为0.8203,平均观测杂合度明显小于期待杂合度,表明三个地理群体个体之间遗传变异程度较高,处于遗传不平衡状态。种群间的遗传距离越大,杂交后其子代的基因型杂合度越高,遗传多样性越丰富。通过3个地理鲎群体两两相比较发现,钦州与防城港鲎群体之间的遗传距离最大,为0.4896,表明钦州与防城港两群体之间基因交流较少,亲缘关系较远;防城港与北海鲎群体的遗传距离最小,为0.3656,表明防城港鲎群体与北海鲎群体之间的基因流比与钦州鲎群体之间的基因流大。这与防城港和北海两群体之间的基因流3.592大于防城港和钦州的基因流2.591的结果相一致。有趣的是钦州位于防城港与北海之间,反而防城港与北海鲎群体遗传距离最小,亲缘关系最近,基因交流更为频繁。结合遗传距离与以遗传距离为基础构建的聚类图进行分析发现,钦州中国鲎qz-t-53和圆尾鲎qz-c-2间的遗传距离为0.4000,在聚类图中聚成一个小分支;北海中国鲎bh-t-17和防城港圆尾鲎fcg-c-10间的遗传距离为0.5526,聚成一个分支;北海中国鲎bh-t-13与防城港中国鲎fcg-t-11之间的遗传距离为0.5500,聚成一个分支;钦州中国鲎qz-t-70和qz-t-73间遗传距离为0.8750,单独聚成一个分支。这表明钦州中国鲎与圆尾鲎之间;北海中国鲎与防城港圆尾鲎和中国鲎之间存在基因渐渗现象。遗传分化指数fst作为衡量群体遗传分化程度的重要参数。对fst值的范围划分,平均fst值在0.05~0.15范围之内时群体间遗传分化程度位于中等。本研究中20个微卫星位点参试样本的fst值分布范围为0.022~0.216,平均值为0.0923,这表明9.23%的遗传变异来自于样本群体间的分化,90.77%的遗传变异来自群体内,群体间随机交配较低。影响种群内遗传多样性的两大因素为近交衰退和遗传漂变,而基因流nm越大,由遗传漂变引起的群体遗传分化越容易被抑制;当nm>1时,群体分化被抑制;当nm<1时,群体分化明显。本研究中,除位点tt-req-150的nm值小于1外,其余19个微卫星位点的nm值均大于1,其中位点tt-req-210的nm值最大,达到了11.317,参试鲎样本的nm平均值为3.268,表明3个地理群体个体之间存在一定的基因交流,其基因交流的程度处于较高水平,一定程度上抵制了遗传漂变对群体分化的影响。通过利用structure2.3.4软件的显性等位基因模型对参试的204份鲎样本进行亚群划分,发现有6个亚群的个体数目在10个以上,有3个亚群的在20个个体以上,个体数量最少的为种群8,仅有5个个体。亚群较多,除种群2外,其他种群个体较少,群体正在碎片化,碎片化可能会趋向灭绝旋涡的边缘,最终导致物种的灭绝。幼鲎到成年栖息地由滩涂到浅海再到深海,这一过程需要花费11年左右的成长时间,成年后的鲎每年夏季再返回海滩进行繁殖产卵。对于成年个体而言,每年回归海滩的具体时间点和方位具有一定的随机性,遇到的配偶也不一定都来自同一个种群,因此同一区域的海滩上同时存在着不同种群的鲎的可能。我们选取的采样地点为广西钦州、北海、防城港三个沿海城市,其中钦州三娘湾大风江入海口沿线海滩、北海金海湾红树林生态旅游区西南沿线海滩、防城港港口区海韵名都东南沿海海滩均有中国鲎的踪迹记录。采样过程中,这三个海滩均发现有中国鲎和圆尾鲎共生,中国鲎较圆尾鲎数量少,防城港采样点红树林滩涂附近由于渔民围海养螺,渔网的围堵阻碍了鲎回游的去路,对当地中国鲎的栖息环境造成了严重的破坏。综上所述,本申请利用ssr分子标记对采自广西北部湾三大沿海城市的鲎群体开展了遗传多样性分析,从204对ssr引物中筛选出164对多态引物,引物多态率相对较好。选用的20对引物的多态信息含量均值为0.7974,参试样本间平均遗传距离为0.7528,遗传多样性较高,亲缘性较低。参试样本平均基因流为3.268,个体间基因交流的程度处于较高水平。利用structure2.3.4软件将群体划分得到9个亚群,发现亚群2、3、5、8中均有中国鲎和圆尾鲎组成,说明圆尾鲎与中国鲎存在基因交流。其中有一个比较有意思的现象是,北海的中国鲎与防城港的中国鲎和圆尾鲎同时被划归在亚群3之中,这可能是北海、防城港两地的鲎之间跨越了钦州海域相遇后产生基因交流的结果。种群越小遗传漂变越显著,划分的亚群中,有的种群很大有的种群很小,呈碎片化,小亚群应对环境变化的能力较小,有走向灭绝边缘的危险。人类活动会对中国鲎之间的基因交流产生影响,其遗传多样性会受到栖息地环境变化的深刻影响。在可以预见的未来由于人类活动对鲎栖息地破坏将使鲎面临着更加艰难的生存困境,对中国鲎资源及其遗传多样性的保护刻不容缓。以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明的保护范围应以所附权利要求为准。序列表<110>北部湾大学<120>一种中国鲎ssr引物组及其应用<141>2019-09-27<160>40<170>siposequencelisting1.0<210>1<211>22<212>dna<213>人工序列(artificialsequence)<400>1tcgccagactgtatttgtatta22<210>2<211>22<212>dna<213>人工序列(artificialsequence)<400>2acgaacttttctgaaccataca22<210>3<211>22<212>dna<213>人工序列(artificialsequence)<400>3agcttgccaatattagaactga22<210>4<211>22<212>dna<213>人工序列(artificialsequence)<400>4acttttggtattgtacagtggt22<210>5<211>22<212>dna<213>人工序列(artificialsequence)<400>5gaatggctctttctgggtataa22<210>6<211>22<212>dna<213>人工序列(artificialsequence)<400>6ggtactcatccacctgtctata22<210>7<211>22<212>dna<213>人工序列(artificialsequence)<400>7acattgatccactgattgacaa22<210>8<211>22<212>dna<213>人工序列(artificialsequence)<400>8agtcctgcctcttcaataatga22<210>9<211>22<212>dna<213>人工序列(artificialsequence)<400>9ttaaataagccaaacagtgcaa22<210>10<211>22<212>dna<213>人工序列(artificialsequence)<400>10ttatgcgaaaacagccaataat22<210>11<211>22<212>dna<213>人工序列(artificialsequence)<400>11ctaccacagtccagagttaaaa22<210>12<211>22<212>dna<213>人工序列(artificialsequence)<400>12tgaggactaaatcacacatctg22<210>13<211>22<212>dna<213>人工序列(artificialsequence)<400>13ttccatttgagtcacccataaa22<210>14<211>22<212>dna<213>人工序列(artificialsequence)<400>14tgttggaaaatgcaacagtaat22<210>15<211>22<212>dna<213>人工序列(artificialsequence)<400>15accaccagttttccacccttta22<210>16<211>22<212>dna<213>人工序列(artificialsequence)<400>16aggttctcagggtctcttacac22<210>17<211>22<212>dna<213>人工序列(artificialsequence)<400>17ccagaccaggagaacgagttac22<210>18<211>22<212>dna<213>人工序列(artificialsequence)<400>18attaggcctgcaatccgtgata22<210>19<211>22<212>dna<213>人工序列(artificialsequence)<400>19gtgtgaagaccgttgtgtttca22<210>20<211>22<212>dna<213>人工序列(artificialsequence)<400>20gcaggaataagtgacggcttta22<210>21<211>22<212>dna<213>人工序列(artificialsequence)<400>21caagtgtggatagcagctttgt22<210>22<211>22<212>dna<213>人工序列(artificialsequence)<400>22acccgtgtttgtattagagggt22<210>23<211>22<212>dna<213>人工序列(artificialsequence)<400>23aatgcagctacagggcctttta22<210>24<211>22<212>dna<213>人工序列(artificialsequence)<400>24cctcactggtcttggcagatag22<210>25<211>22<212>dna<213>人工序列(artificialsequence)<400>25tcctggagatatggcggttaca22<210>26<211>22<212>dna<213>人工序列(artificialsequence)<400>26cgctggaccgagaactgatatc22<210>27<211>22<212>dna<213>人工序列(artificialsequence)<400>27tggtgcaatgtcctcctgatta22<210>28<211>22<212>dna<213>人工序列(artificialsequence)<400>28catcgaatcaatggccctgaaa22<210>29<211>22<212>dna<213>人工序列(artificialsequence)<400>29tgggcaaagctgtaaccttgaa22<210>30<211>22<212>dna<213>人工序列(artificialsequence)<400>30tccaggacattgctctctgaca22<210>31<211>22<212>dna<213>人工序列(artificialsequence)<400>31tcaacagcatttccctcctaca22<210>32<211>22<212>dna<213>人工序列(artificialsequence)<400>32ccgacgtggctttgttttaaga22<210>33<211>22<212>dna<213>人工序列(artificialsequence)<400>33agctactgtgaagtgcaaaagg22<210>34<211>22<212>dna<213>人工序列(artificialsequence)<400>34ttgtgtagtcgggattggtaca22<210>35<211>22<212>dna<213>人工序列(artificialsequence)<400>35cccacgaatctaatgcggagtt22<210>36<211>22<212>dna<213>人工序列(artificialsequence)<400>36ccaccgtaagttcacccctact22<210>37<211>22<212>dna<213>人工序列(artificialsequence)<400>37ttcctttatcttgctcgcacac22<210>38<211>22<212>dna<213>人工序列(artificialsequence)<400>38aggagccaacccttctttgtta22<210>39<211>22<212>dna<213>人工序列(artificialsequence)<400>39accaagaatcggcactgactat22<210>40<211>22<212>dna<213>人工序列(artificialsequence)<400>40agcagttgaagttcggaattgt22当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1