耐低氧团头鲂生长性状基因及其定位方法和应用与流程
本发明涉及数量性状定位(quantitativetraitloci,qtl)方法,具体地说,涉及耐低氧团头鲂生长性状基因及其定位方法和应用。
背景技术:
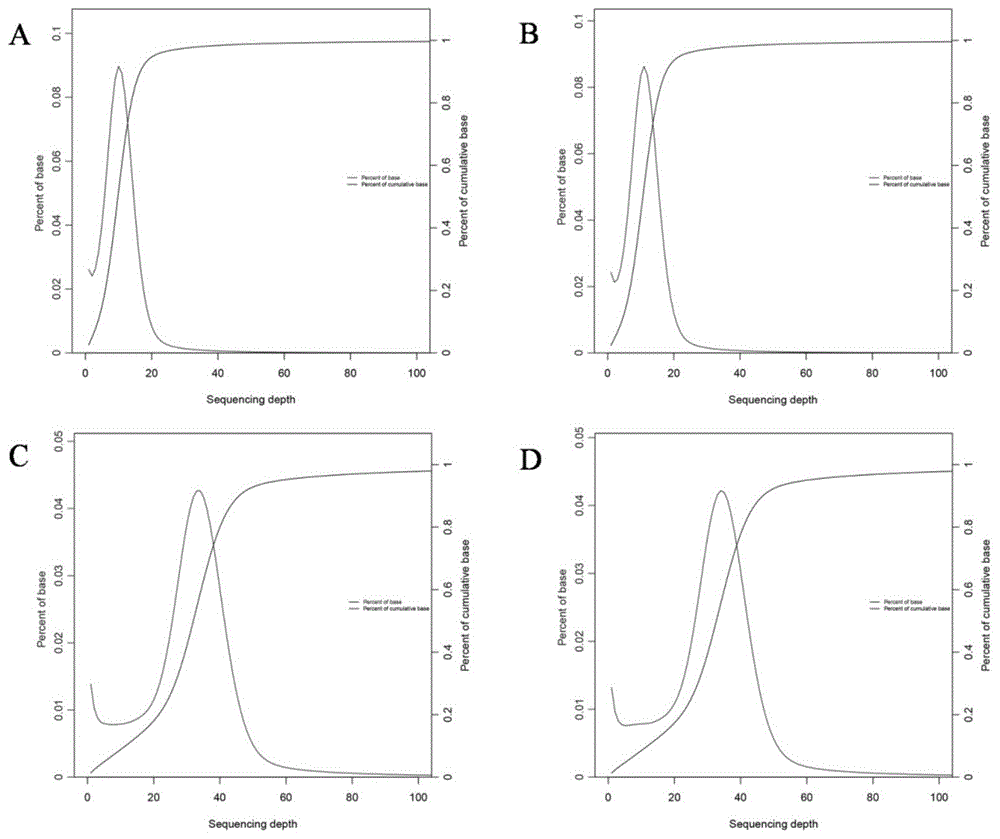
:目前,鱼类对特定性状进行选育获得控制性状发育的基因或qtl是进行分子育种的前提。数量性状是一个多世纪以来遗传学研究的主要对象之一,因为它是许多重要作物、畜牧和人类的重要性状,但开展数量性状研究长期以来主要借助于统计学手段。关于鱼类qtl定位研究主要集中在一些形态性状(如体高、体重、体长、体宽等)和一些适应性性状(如抗病、抗逆等)上,它们比较容易测得相关表型数据,但对于像肉色、口感等性状来说难以获得表型数据,限制此类性状的qtl定位研究,而且鱼类qtl定位研究常见于几种养殖品种如虹鳟、罗非鱼、大西洋鲑、斑点叉尾鮰、鲤鱼等,在团头鲂中鲜有报道。近年来,随着新一代测序技术与生物信息学的不断发展,分子生物学与基因组学的研究方法也随之改变。目前,应用最多的是开发大量的snp标记构建高密度的遗传图谱,遗传图谱是指基因或dna标志在染色体上的相对位置与遗传距离,遗传距离通常由基因或片段在染色体交换过程中分离的频率厘摩来表示,这种方法可以同时定位多个数量性状,但劳动强度大,耗时长,成本高。技术实现要素:针对现有技术中的不足,本发明提供耐低氧团头鲂生长性状基因,包括mdh(malatedehydrogenase)基因和pdxk(pyridoxalkinase)基因;其中:所述mdh基因的碱基序列如seqidno:1所示;所述pdxk基因的碱基序列如seqidno:2所示。所述mdh基因的编码蛋白具有如seqidno:3所示的氨基酸序列。所述pdxk基因的编码蛋白具有如seqidno:4所示的氨基酸序列。本发明还提供上述耐低氧团头鲂生长性状基因的定位方法,包括以下步骤:(ⅰ)团头鲂的新品种f4代和“浦江1号”杂交,获得家系群体;和(ⅱ)杂交后代自交,获得分离群体;和(ⅲ)分离群体中各取若干个极端表型个体,提取dna等量混和,获得dna混池;和(ⅳ)上述dna混池与亲本进行基因组重测序,检测与生长性状相关的基因候选区域,并进行数量性状定位。步骤(ⅳ)中,所述与生长性状相关的基因候选区域位于7号染色体上,区间为30.88~33.57mb,且其总长度为2.69mb。在某一/些实施方案中,上述耐低氧团头鲂生长性状基因的定位方法为:先将团头鲂的新品种f4代和“浦江1号”杂交获得家系f1代;2龄的f1代自交获得家系f2代,孵化后每天喂食两次;90天后,对家系f2代群体进行生长性状测量,分别选取生长最快和生长最慢的30个个体,获得dna混池;对两个亲本和上述60个f2代个体取样并提取基因组dna,等量混和构建快速池(f池)和慢池(s池)两个dna混池,并对其和两个亲本进行基因组重测序和数量性状定位。上述技术方案中,所述基因组重测序的步骤为:超声波将亲本和dna混池的dna序列片段化形成随机片段,对片段化的dna依次进行末端修复、3′端加a和连接测序接头后,用磁珠吸附富集基因组长度为400bp的片段,pcr扩增形成测序文库,经质检合格后经测序系统测序。上述技术方案中,所述数量性状定位的步骤为:将样品与团头鲂基因组序列比对、snp和smallindel标记检测和变异功能注释,得到snp和indel的功能注释信息;和基于亲本和dna混池的数据计算snp-index值并进行bsa关联分析,定位目的突变位点,并对突变位点周围的基因进行功能注释。优选技术方案中,基因组重测序结果根据gatk推荐的参数过滤,用bwa软件以mem方法将样品与团头鲂基因组序列比对;用gatk的bestpractices流程对bam文件校正,并进行snp和smallindel标记检测;用snpeff软件和参考基因组的基因预测信息进行变异功能注释,得到snp和indel的功能注释信息;基于亲本和突变混池的数据计算snp-index值并进行bsa关联分析,定位目的突变位点,并对突变位点周围的基因进行功能注释。在某一优选实施方案中,测序系统采用illuminahiseqtm平台,测序策略为illuminape150。本发明还提供上述耐低氧团头鲂生长性状基因在耐低氧团头鲂新品种分子育种中的应用。与现有技术相比,本发明的有益效果是:本发明针对目标性状,选择表型极端差异的亲本构建家系,对该家系目标性状表型极端的子代分别混合得到的两个样本池进行全基因组重测序,检测到的两池间dna差异片段即为候选区域,可进一步定位到目标性状相关的基因或标记,快速有效定位团头鲂耐低氧新品种生长性状基因,既节省时间又实用,为其分子育种提供新的路径。附图说明图1是测试样品的测序深度;其中,a:父本,b:母本,c:f池,d:s池。图2是测试样品的基因组覆盖度;其中,a:父本,b:母本,c:f池,d:s池。图3是测试样品的插入缺失片段长度;其中,a:父本,b:母本,c:f池,d:s池。图4是s池和f池的snp-index图(a,b)以及δ(snp-index)图(c);其中,a是s池的snp-index,b是f池的snp-index,c是s池与f池的snp-index之差δ(snp-index),横坐标为各染色体的物理位置,纵坐标为snp-index。具体实施方式下面结合附图和实施例对本发明进一步说明。1.材料与方法以下实施例中所用鱼类均取自上海海洋大学农业部团头鲂遗传育种中心。先用团头鲂“浦江1号”和新品种f4代构建家系,杂交获得f1代;再用2龄的f1代进行自交获得f2代用于qtl分析。f2代(约500尾)在孵化桶中孵育14天后转移到孵化池中继续孵化,每天喂食两次,待子代长到90天时进行实验。2.团头鲂耐低氧新品种生长性状基因的定位方法步骤一:生长数据测量与取样孵化90天后,对f2子代群体进行生长相关性状(全长、体长、体高、体宽和体重)的测量。根据测量数据,分别选取生长最快和生长最慢的30个个体,构建dna混池,对两个亲本及60个f2代个体进行取样,将所取鱼鳍保存于无水乙醇中用于后续基因组dna的提取。步骤二:基因组dna的提取、文库构建、测序及分析分别对亲本及60个f2代个体提取基因组dna,然后分别将两组子代个体基因组dna进行等量混和,构建快速池(f池)和慢池(s池)两个dna混池,将这两个dna混池外加两个亲本共四个样品进行基因组测序;超声波将亲本和混池dna序列片段化形成随机片段,对片段化的dna依次进行末端修复、3′端加a和连接测序接头后,再用磁珠吸附富集基因组长度为400bp的片段,经过pcr扩增形成测序文库。建好的文库先进行文库质检,质检合格的文库用illuminahiseqtm平台进行测序,测序策略为illuminape150。测序结果经过过滤后,利用bwa(http://bio-bwa.sourceforge.net/)软件以mem方法将4个样品分别与团头鲂基因组序列进行比对。利用gatk的bestpractices流程对bam文件进行校正,并进行snp和smallindel标记的检测。过滤条件是根据gatk推荐的参数进行的,(https://software.broadinstitute.org/gatk/documentation/article.php?id=3225)。利用snpeff软件和参考基因组的基因预测信息进行变异功能注释,得到snp和indel的功能注释信息。基于亲本和突变混池的数据特点,计算snp-index值进行bsa关联分析,定位目的突变位点,并对突变位点周围的基因进行功能注释。3.结果与分析对f2子代群体约500尾个体进行生长相关性状(全长、体长、体高、体宽和体重)的测量,体重从2.6g到17.3g,表现出巨大的表型差异,证明该家系中具有较高程度的遗传变异,可用于qtl分析。从每一组极端个体中各选取约10%来构建测序文库(30尾快速生长的个体和30尾生长缓慢的个体);其中,f池的平均体重为12.5±2g(10.1g~17.3g),s池的平均体重为3±0.3g(2.6g~3.9g),如表1所示。表1:两组极端个体生长相关性状测量数据<测序质量统计>在illuminape150平台上,利用两个亲本和两个极端性状混池构建四个文库。原始数据过滤后,获得490,700,237个高质量的数据(cleanreads)(约147.83gb),包括58,182,510个父本的cleanreads(gc%=38.09),63,256,683个母本的cleanreads(gc%=38.05),182,639,261个f池的cleanreads(gc%=38.07)和186,621,783个s池的cleanreads(gc%=38.02),如表2所示。表2:测序质量统计样本cleanreadscleanbasegc(%)q30(%)父本58,182,51017,525,850,99938.0993.96母本63,256,68319,057,845,39238.0594.09f池182,639,26155,020,964,37938.0793.75s池186,621,78356,222,079,31338.0293.99<基因组比对效率>以团头鲂的基因组序列作为参考基因组,参考基因组大小为1.1gb,gc含量36.18%。利用bwa软件将质控后的测序片段(cleanreads)比对参考基因组,得到4个文库的比对效率分别为99.37%、99.42%、99.32%、99.38%,如表3所示。表3:比对结果数据统计样本mappedratio(%)properlymapped(%)duplicationratio(%)父本99.3783.3929.18母本99.4283.9430.13f池99.3282.9229.74s池99.3883.2229.85<深度分布统计>reads锚定到参考基因组后,可以统计其对参考基因组的覆盖情况,所有样品的碱基覆盖度和平均覆盖深度如表4所示。表4:覆盖深度和覆盖度统计样本coveragebase(bp)coverage1x(%)coverage5x(%)averagedepth父本997,674,47289.2879.6111.05母本995,537,37489.0980.7511.86f池1,037,164,22792.8280.7534.42s池1,036,007,49592.7189.135.13碱基上覆盖的reads数为覆盖深度,基因组的覆盖深度会影响变异检测的准确性,在覆盖深度较高的区域(非重复序列区),变异检测的准确性也越高。如图1所示,亲本平均测序深度为11.46x,混池平均测序深度为34.78x。参考基因组上被reads覆盖到的碱基数占基因组总长度的百分比称为基因组覆盖度,基因组覆盖度可以反映变异检测的完整性,覆盖到参考基因组的区域越多,可以检测到的变异位点也越多,图2表明基因组被覆盖地较均匀,说明测序随机性较好。<snp及indel检测和注释>与对照基因组相比,检测到的snp和indel都被分为两类,snp包含转换和颠换,indel包含插入和缺失。根据变异检测结果进行相关分析,过滤后得到相应染色体上的snp和indel数,如表5所示。另外,还对4个样品的插入缺失片段长度进行统计,长度分布如图3所示。表5:各染色体上snp和indel数<关联分析>根据所得亲本间snp和indel位点,分别计算f池和s池中每个位点的snp-index和indel-index值。index值指在突变池和野生池中突变基因型占所有基因型中的深度比例,由于突变位点与周围标记具有连锁效应,在突变位点附近,突变型混池中的index值更接近于1,而野生型混池中的index值更接近于0;由于连锁效应较弱或不连锁位点,呈现出随机分布,应符合孟德尔分离比例,正常位点index值为0.5,两混池间的index差值关联的区域即是目标性状关联的候选区域。采用滑动窗分析方法,以2mb为单位窗口,10kb为滑动窗口绘制s池,f池的snp-index图(图4a,b)。根据f池和s池的snp-index,计算两个池的snp-index的差值,同样以染色体的位置为横坐标,2mb为单位窗口,10kb为滑动窗口绘制δ(snp-index)图(图4c)。经过对f池和s池的snp-index的分析发现,在7号染色体上的30.88mb到33.57mb之间出现snp不平衡的状况,此区域的δ(snp-index)在0.999的置信水平下大于阈值,因此7号染色体的30.88mb到33.57mb是控制生长性状的一个qtl位点,并发现本发明中两个与团头鲂生长性状相关的基因mdh和pdxk;其中:mdh基因的碱基序列如seqidno:1所示,编码蛋白具有如seqidno:3所示的氨基酸序列;pdxk基因的碱基序列如seqidno:2所示,其编码蛋白具有如seqidno:4所示的氨基酸序列。本发明中,利用qtl-seq技术鉴定团头鲂基因组发现,qtl上的两个基因mdh和pdxk与团头鲂生长性状显著关联。序列表<110>上海海洋大学<120>耐低氧团头鲂生长性状基因及其定位方法和应用<141>2019-11-21<160>4<170>siposequencelisting1.0<210>1<211>1002<212>dna<213>unknown<400>1atggccgaaccgatccgtgtcctggtgactggcgcagccggacagatcgcctattctctg60ctctacagcattgctaaaggagatgtgttcggcaaggatcagccaatcatcttggtgctt120ctggacatcactcccatgctgcccgtgctggatggggtcgtcatggaactgcaggattgt180gctcttcctcttctgagggaggtgattcctaccgataaggttgaggtgggcttcaaggac240cttgatgctgccatcttggtgggctctatgccaaggaaagagggcatggagagaaaggac300ctcctgaaggccaatgtggccatttttaaaacccaaggtgaagcactggagaagtatgcc360aagaagaccgtcaaggtgctagttgtcgggaacccagccaacaccaactgtttgatcgcc420tccaaatctgctccgtccattcctaaggagaacttctcctgcctgacccgtctggaccat480aacagggcccgctctcaggtggcgatgcgtgttggtgtgtcctctgacagtgtgaagaat540gtgattatctggggaaatcactcctcaactcagtacccagatgtgcaccatgctatcgtg600aaccaccatgggaaggagttggcagcctttgacgctgtgaatgacgaaagctggctgaag660ggtgacttcatctccacggtgcagcagagaggtgcagctgtcatcaaggccaggaagctc720tccagcgcaatgtctgctgccaaagccatctgtgaccacatgagggacatctggttcggc780actcctgatggcgagtgggtgtctatgggcgtctactcctctggtaattcctatggagtt840cctgatgacctcatgtactccttccctgttaagattaagaacaagacctggaaggtggtt900gacggactccccatcaacgatttctctcgcgctaagatggacgccaccgccgctgagctg960gttgaggagagagacacggcagtcaccttccttggagcgtga1002<210>2<211>957<212>dna<213>unknown<400>2tctggagcgattaaaacaactttaactgcgcgcgacatggaatgccgagtgctgtcgatt60cagagtcatgtcgtgagaggatacgtggggaataaatctgcatcctttccgttacaggtc120atggggtttgaggtggactcgatcaactctgttcagttctctaatcacacaggttactct180cactggaagggacaagtcttgactgcagatgagcttcacgtgctgtatgaagggatcaaa240ctgaacaacgtcaaccactacgactatgtcctgacaggttacactagagatacgtcattt300ctggagatggtggttgatattgtacaagagctaaagagggccaatcctaaccttgtatat360gtttgtgaccctgtcttaggtgaccatggttcaatgtatgttcctcagaatctccatcct420gtgtacaagaataaagtggtacctgtggccgatatcatcacacccaaccagtttgaggca480gagttattgacagggaagaatatcagcacagagaaagatgctgttgaggtgatggatctt540ctgcataagatgggtccagacaccgtggtcatcaccagctctgatctgccccctcgtctc600ggagacagatttctggtgtctcttggcagtcagcgcattttgatgccagacggtacaagg660aagacccagcggatccgaatagaggtgccgaaagtggatgcggtgtttgtggggactggt720gatctatttgctgctatgctgttggcctggactcaccattacccaacagacctgaagacg780gcatgtgagaagactttctccgtcatgcaccatgtcattcagaggactatatcttatgcc840catgaaatggcaggtcctggtagaagacctagcccggctcagctggagttacggatggtc900cagagtaaagcggacatagaggatccagccatagtcatggaggcgaccgttctatag957<210>3<211>333<212>prt<213>unknown<400>3metalagluproileargvalleuvalthrglyalaalaglyglnile151015alatyrserleuleutyrserilealalysglyaspvalpheglylys202530aspglnproileileleuvalleuleuaspilethrprometleupro354045valleuaspglyvalvalmetgluleuglnaspcysalaleuproleu505560leuarggluvalileprothrasplysvalgluvalglyphelysasp65707580leuaspalaalaileleuvalglysermetproarglysgluglymet859095gluarglysaspleuleulysalaasnvalalailephelysthrgln100105110glyglualaleuglulystyralalyslysthrvallysvalleuval115120125valglyasnproalaasnthrasncysleuilealaserlysserala130135140proserileprolysgluasnphesercysleuthrargleuasphis145150155160asnargalaargserglnvalalametargvalglyvalserserasp165170175servallysasnvalileiletrpglyasnhisserserthrglntyr180185190proaspvalhishisalailevalasnhishisglylysgluleuala195200205alapheaspalavalasnaspglusertrpleulysglyasppheile210215220serthrvalglnglnargglyalaalavalilelysalaarglysleu225230235240serseralametseralaalalysalailecysasphismetargasp245250255iletrppheglythrproaspglyglutrpvalsermetglyvaltyr260265270serserglyasnsertyrglyvalproaspaspleumettyrserphe275280285provallysilelysasnlysthrtrplysvalvalaspglyleupro290295300ileasnasppheserargalalysmetaspalathralaalagluleu305310315320valglugluargaspthralavalthrpheleuglyala325330<210>4<211>318<212>prt<213>unknown<400>4serglyalailelysthrthrleuthralaargaspmetglucysarg151015valleuserileglnserhisvalvalargglytyrvalglyasnlys202530seralaserpheproleuglnvalmetglyphegluvalaspserile354045asnservalglnpheserasnhisthrglytyrserhistrplysgly505560glnvalleuthralaaspgluleuhisvalleutyrgluglyilelys65707580leuasnasnvalasnhistyrasptyrvalleuthrglytyrthrarg859095aspthrserpheleuglumetvalvalaspilevalglngluleulys100105110argalaasnproasnleuvaltyrvalcysaspprovalleuglyasp115120125hisglysermettyrvalproglnasnleuhisprovaltyrlysasn130135140lysvalvalprovalalaaspileilethrproasnglnphegluala145150155160gluleuleuthrglylysasnileserthrglulysaspalavalglu165170175valmetaspleuleuhislysmetglyproaspthrvalvalilethr180185190serseraspleuproproargleuglyaspargpheleuvalserleu195200205glyserglnargileleumetproaspglythrarglysthrglnarg210215220ileargilegluvalprolysvalaspalavalphevalglythrgly225230235240aspleuphealaalametleuleualatrpthrhishistyrprothr245250255aspleulysthralacysglulysthrpheservalmethishisval260265270ileglnargthrilesertyralahisglumetalaglyproglyarg275280285argproserproalaglnleugluleuargmetvalglnserlysala290295300aspilegluaspproalailevalmetglualathrvalleu305310315当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1