山羊ZBP1基因第一内含子内与产羔数关联的单倍型标记及其应用的制作方法
本发明涉及山羊分子标记筛选技术领域,尤其涉及山羊zbp1基因第一内含子内与产羔数关联的单倍型标记及其应用。
背景技术:
产羔性状是山羊的最重要的经济性状之一。提高每胎的产羔数对于山羊而言是提升生产经济效益的重要措施。应用基因组学方法的研究能在整个基因组的水平去筛选出与山羊重要经济性状相关的候选基因和分子标记,使研究人员能够更深入的理解山羊性状的遗传机理。由于山羊产羔数是低遗传力性状,通过常规的育种方法很难在短期内获得较大遗传进展,随着应用基因组学、分子生物学和分子遗传学理论技术的不断发展,遗传标记技术可以较好的解决这一难题,并能有效的加快山羊产羔数性状的选择进展,可对我国山羊产业的可持续发展将带来巨大的经济效益。
单核苷酸多态性(singlenucleotidepolymorphism,snp)标记指由基因组单核苷酸变异引起的dna序列多态性,包括碱基转换、颠换、单碱基插入或缺失等,被公认为是最新的第三代dna分子标记。根据snp在基因中的位置可分为基因编码区snp(coding-regionsnpcsnp)、基因周边snp(perigenicsnp,psnp)以及基因间snp(intergenicsnp,isnp)三类。其中基因编码区的snp可能导致基因所编码的氨基酸序列发生改变,进而改变蛋白的高级形态,影响基因所编码的蛋白类激素的功能。
snapshot技术是一种基于荧光标记单碱基延伸原理的分型技术,也称小测序,主要针对中等通量的snp分型项目。在一个含有测序酶,四种荧光标记的ddntp,紧挨多态位点5’端的不同长度延伸引物和pcr产物模板的反应体系中,引物延伸一个碱基即终止,经abi测序仪电泳后,根据峰的颜色可知掺入的碱基种类,从而确定该样本的基因型,根据峰移动的胶位置确定该延伸产物对应的snp位点,通常用于5-30个snp位点分析。
z-dna结合蛋白1(z-dnabindingprotein1,zbp1)基因是一种细胞质传感器,当前研究重要集中在疾病和炎症反应等方面。迄今为止,尚无有关山羊zbp1基因做为山羊产羔数性状的分子标记的研究的相关报道。
技术实现要素:
本发明的目的在于克服现有技术之缺陷,提供了山羊zbp1基因第一内含子内与产羔数关联的单倍型标记及其应用,本发明发掘了山羊zbp1基因第一内含子内与产羔数关联的单倍型标记,为山羊产羔数性状的分子标记辅助育种提供了一种新的单倍型标记。
本发明的目的之一在于提供一种山羊zbp1基因第一内含子内与产羔数关联的单倍型标记,所述的单倍型标记位于山羊zbp1基因第一内含子的部分dna序列中,所述单倍型标记的核苷酸序列如序列表seqidno.1所示,该序列长度为2463bp;所述单倍型标记包括:
zbp1g.352c>t位点的分子标记:即在seqidno.1所示序列中第249bp处存在一处c>t碱基突变;
zbp1g.955c>t位点的分子标记:即在seqidno.1所示序列中第852bp处存在一处c>t碱基突变;
zbp1g.1880g>a位点的分子标记:即在seqidno.1所示序列中第1777bp处存在一处g>a碱基突变;
zbp1g.2566t>c位点的分子标记:即在seqidno.1所示序列中第2463bp处存在一处t>c碱基突变。
本发明的目的之二在于提供所述的单倍型标记在山羊产羔数性状标记辅助选择中的应用。
本发明的目的之三在于提供一种山羊zbp1基因第一内含子内与山羊产羔数性状相关snp的检测试剂盒,包括:
用于扩增包含zbp1g.352c>t位点的seqidno.2所示序列的引物对:上游引物的核苷酸序列如seqidno.6所示,下游引物的核苷酸序列如seqidno.7所示;
用于扩增包含zbp1g.955c>t位点的seqidno.3所示序列的引物对:上游引物的核苷酸序列如seqidno.8所示,下游引物的核苷酸序列如seqidno.9所示;
用于扩增包含zbp1g.1880g>a位点的seqidno.4所示序列的引物对:上游引物的核苷酸序列如seqidno.10所示,下游引物的核苷酸序列如seqidno.11所示;
用于扩增包含zbp1g.2566t>c位点的seqidno.5所示序列的引物对:上游引物的核苷酸序列如seqidno.12所示,下游引物的核苷酸序列如seqidno.13所示;
以及四个snapshot单碱基延伸引物,包括:
用于检测zbp1g.352c>t位点的单碱基延伸引物如seqidno.14所示;
用于检测zbp1g.955c>t位点的单碱基延伸引物如seqidno.15所示;
用于检测zbp1g.1880g>a位点的单碱基延伸引物如seqidno.16所示;
用于检测zbp1g.2566t>c位点的单碱基延伸引物如seqidno.17所示。
本发明的目的之四在于提供使用所述检测试剂盒山羊zbp1基因第一内含子内与山羊产羔数性状相关snp的方法,包括以下步骤:
步骤1、以从待测山羊血液样本中提取的基因组dna为模板,通过选自所述seqidno.6~13所示pcr引物对构建多重pcr扩增体系进行pcr扩增,并对pcr产物进行纯化处理;
步骤2、以所述纯化的pcr产物为模版,通过选自所述seqidno.14~17所示snapshot单碱基延伸引物构建snapshot反应体系进行单碱基延伸反应,并对反应制备的单碱基延伸产物进行纯化处理;
步骤3、使用遗传学分析仪检测所述纯化后的单碱基延伸产物,并使用基因分析软件对结果进行分析。
本发明的目的之五在于提供所述的检测试剂盒在山羊产羔数性状标记辅助选择中的应用。
本发明的有益效果:
本发明发掘了山羊zbp1基因第一内含子内与产羔数关联的单倍型标记所述的单倍型标记位于山羊zbp1基因第一内含子的部分dna序列中,含有zbp1g.352c>t、zbp1g.955c>t、zbp1g.1880g>a和zbp1g.2566t>c四个snp位点;由上述4个snp位点组成的单倍型可以作为山羊产羔数性状的单倍型标记,本发明为山羊产羔数性状的分子标记辅助育种提供了一种新的单倍型标记,实现了对山羊产羔数性状进行早期选择,检测方法快速、准确,且不受养殖环境条件因素影响。
附图说明
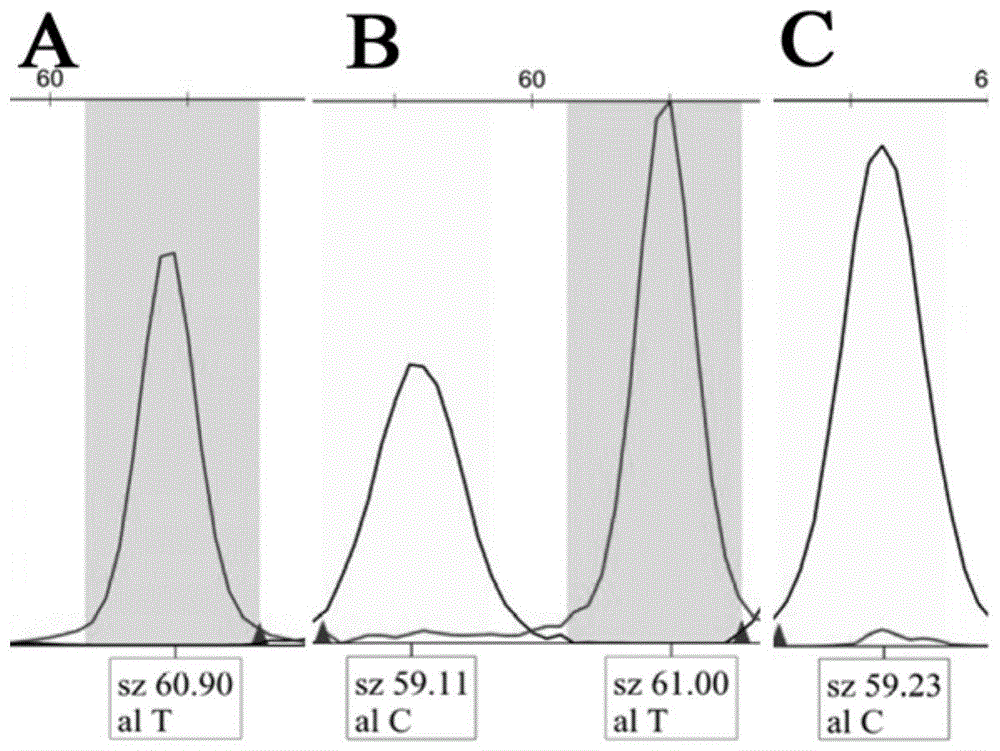
图1是本发明中山羊zbp1基因第一内含子中zbp1g.352c>t位点的genemapperv4.0软件读取结果图;a图:tt基因型;b图:ct基因型;c图:cc基因型;
图2是本发明中山羊zbp1基因第一内含子中zbp1g.955c>t位点的genemapperv4.0软件读取结果图;a图:cc基因型;b图:ct基因型;c图:tt基因型;
图3是本发明中山羊zbp1基因第一内含子中zbp1g.1880g>a位点的genemapperv4.0软件读取结果图;a图:gg基因型;b图:ga基因型;c图:aa基因型;
图4是本发明中山羊zbp1基因第一内含子中zbp1g.2566t>c位点的genemapperv4.0软件读取结果图;a图:tt基因型;b图:ct基因型;c图:cc基因型;
图5是山羊zbp1基因seqidno.2、seqidno.3、seqidno.4和seqidno.5序列片段琼脂糖凝胶电泳图谱;1:如seqidno.2所示序列,2:如seqidno.3所示序列,3:如seqidno.4所示序列,4:如seqidno.5所示序列;
图6是zbp1g.352c>t、zbp1g.955c>t、zbp1g.1880g>a和zbp1g.2566t>c位点在波尔山羊、黑头羊和麻城黑山羊群体中均为强连锁不平衡结果。
具体实施方式
实施例1山羊zbp1基因第一内含子片段的获得及多态性位点检测方法的建立
1、山羊基因组dna的提取
本发明的试验山羊品种为波尔山羊、麻城黑山羊和黑头羊(波尔山羊与麻城黑山羊杂交品种),样本来源于湖北省农业科学院畜牧兽医研究所种羊场。山羊基因组dna采用北京天根生化科技有限公司生产的血液基因组dna提取试剂盒(货号:dp348)进行提取,具体步骤参照试剂盒说明书。对提取出的dna进行浓度和质量检测,置于-40℃下保存备用。
2、山羊zbp1基因第一内含子片段的获得
(1)pcr扩增
根据山羊zbp1基因的基因组序列(genbank登陆号:nc_030820.1)中的第一内含子序列(如seqidno.1所示)设计四对引物,分别扩增包含4个多态性位点的片段(图5)。引物如表1所示。
表1
利用上述引物在波尔山羊、麻城黑山羊和黑头羊基因组dna中进行pcr扩增,pcr反应体系如表2所示,总体积为50μl;pcr反应程序如表3所示。
表2
表3
(2)pcr产物纯化
上述pcr产物用上海生工生物工程有限公司的gelextractionkit试剂盒(货号:b610353)进行纯化,具体步骤如下:首先从琼脂糖凝胶上切下含目的片段的凝胶,放入1.5ml离心管,加入400μl溶胶液,50-60℃水浴至胶彻底融化,加热融胶时,每2min混匀一次,冷却至室温;将离心柱放入收集管中,把混合液移至离心柱,室温放置2min;12000r/min离心1min,此时dna被吸附到柱上;倒掉收集管中废液,将离心柱放入同一个收集管中,加入700μl洗脱液,12000r/min离心1min;倒掉收集管中的废液,12000r/min离心1min;将离心柱放入一预先准备好的灭菌1.5ml离心管中,加入40μl洗脱液或双蒸水(ph>7.0),室温或37℃放置2-3min;12000r/min离心1min,离心管中的液体即为回收的dna片段。
3、将纯化后的pcr产物纯化后通过snapshot方法检测分子标记
根据山羊zbp1基因的基因组序列(genbank登陆号:nc_030820.1)设计zbp1g.352c>t、zbp1g.955c>t、zbp1g.1880g>a和zbp1g.2566t>c位点的snapshot延伸引物如表4所示:
表4
在15μl纯化后的pcr产物中加入5usap和2uexoi,震荡混匀,37℃保温1h,然后75℃保温15min以灭活sap(newenglandbiolabs)和exoi酶(newenglandbiolabs);使用appliedbiosystems公司的snapshotmultiplexkit将处理后的15μlpcr产物吸出3μl进行snapshot检测,pcr反应体系10μl,reactionmix试剂5μl,sap和exoi酶处理后pcr产物3μl,延伸引物各0.5μl,去离子水1μl,pcr扩增程序为96℃变性10s,50℃退火5s,60℃延伸30s,25个循环,4℃保存;将snapshot产物稀释20倍,稀释体系为hi-diformamide9.25μl,gs-120liz0.25μl,snapshot产物0.5μl,反应体系为95℃变性5min,冰浴4min;配制含有350μlhi-di甲酰胺和50μlmatrix标准品的混合液,95℃变性5min,迅速冰冷5min,平分2管,分装至上机板后对3730xldnaanalyzer仪器进行光谱校正;使用3730xldnaanalyzer对制备好的样品进行毛细管电泳并搜集信号;最后使用genemapperv4.0软件对实验结果进行分析(如图1、图2、图3和图4所示)。
实施例2本发明制备的分子标记在杜洛克猪中的多态性分布检测
本实施例分别在波尔山羊、黑头羊和麻城黑山羊群体中检测山羊zbp1基因第一内含子中zbp1g.352c>t、zbp1g.955c>t、zbp1g.1880g>a和zbp1g.2566t>c位点的多态性,检测结果如表5所示。
表5-山羊zbp1基因四个位点在山羊群体中的基因型频率和等位基因频率
由表5结果可知:zbp1g.352c>t、zbp1g.955c>t、zbp1g.1880g>a和zbp1g.2566t>c位点在波尔山羊、黑头羊和麻城黑山羊群体中均表现为三种基因型,其中基因型以纯合型占据优势。在波尔山羊、黑头羊和麻城黑山羊群体中优势等位基因保持一致,zbp1g.352c>t和zbp1g.955c>t位点在三个山羊群体中等位基因c均为优势等位基因,zbp1g.1880g>a位点中等位基因g为优势等位基因,zbp1g.2566t>c位点中等位基因t为优势等位基因。
由图6结果表明:zbp1g.352c>t、zbp1g.955c>t、zbp1g.1880g>a和zbp1g.2566t>c位点在波尔山羊、黑头羊和麻城黑山羊群体中均为连锁不平衡,其中zbp1g.955c>t和zbp1g.1880g>a在波尔山羊和麻城黑山羊群体中表现为完全连锁不平衡(d’=1)。
实施例3本发明制备的分子标记与山羊产羔数性状的关联分析及应用
为了确定山羊zbp1基因第一内含子中zbp1g.352c>t、zbp1g.955c>t、zbp1g.1880g>a和zbp1g.2566t>c位点与波尔山羊、黑头羊和麻城黑山羊产羔数性状差异是否相关,采用实施例1建立的方法进行多态性检测,并分析山羊zbp1基因中zbp1g.352c>t、zbp1g.955c>t、zbp1g.1880g>a和zbp1g.2566t>c位点的三种基因型与山羊产羔数性状的相关性。采用sas统计软件(sasinstituteinc,version9.1)glm程序进行不同snp基因型组合的方差分析,并进行显著性检验,所采用模型为:
yijklm=μ+pi+sj+fk+gl+mm+eijklm;
yijklm为性状表型值,μ为平均值,pi为第i个胎次的影响(i=1、2、3、4),sj为基因型效应(包括基因加性效应和显性效应;加性效应用1,0和-1分别代表cc、ct和tt基因型或aa、ga和gg基因型,显性效应用1,-1和1分别代表cc、ct和tt基因型或aa、ga和gg基因型),fk为第k个羊场的影响(k=1、2),gl为影响第l个基因型(l=1-3),mm为母畜效应,eijklm为残差效应。分别在波尔山羊、黑头羊和麻城黑山羊群体中进行三种基因型与产羔数性状间的关联分析,统计分析结果如表6、表7、表8和表9所示:
表6-山羊zbp1基因zbp1g.352c>t位点与产羔数性状的关联分析
注:a和b表示差异显著(p<0.05),a和b表示差异极显著(p<0.01);*表示差异极显著(p<0.05),**表示差异极显著(p<0.01)。
由表6可知,在波尔山羊群体中,zbp1g.352c>t位点的cc基因型基因型的总产羔数显著高于ct基因型(p<0.05);在黑头羊群体中,cc基因型和tt基因型的总产羔数极显著高于ct基因型(p<0.01);由此可见,即纯合基因型的总产羔数显著高于杂合基因型。
表7-山羊zbp1基因zbp1g.955c>t位点与产羔数性状的关联分析
注:a和b表示差异显著(p<0.05),a和b表示差异极显著(p<0.01);*表示差异极显著(p<0.05),**表示差异极显著(p<0.01)。
由表7可知,在波尔山羊群体中,zbp1g.955c>t位点的tt基因型的总产羔数显著高于cc基因型(p<0.05);zbp1g.955c>t位点在黑头羊和麻城黑山羊群体中的头胎产羔数和总体产羔数均无显著差异。
表8-山羊zbp1基因zbp1g.1880g>a位点与产羔数性状的关联分析
注:a和b表示差异显著(p<0.05),a和b表示差异极显著(p<0.01);*表示差异极显著(p<0.05),**表示差异极显著(p<0.01)。
由表8可知,在波尔山羊群体中,zbp1g.1880g>a位点的aa基因型和ga基因型的总产羔数显著高于gg基因型(p<0.05);zbp1g.1880g>a位点在黑头羊和麻城黑山羊群体中的头胎产羔数和总体产羔数均无显著差异。
表9-山羊zbp1基因zbp1g.2566t>c位点与产羔数性状的关联分析
注:a和b表示差异显著(p<0.05),a和b表示差异极显著(p<0.01);*表示差异极显著(p<0.05),**表示差异极显著(p<0.01)。
由表9可知,在波尔山羊群体中,zbp1g.2566t>c位点的tt基因型的总产羔数显著高于ct基因型(p<0.05);在麻城黑山羊群体中,cc基因型的总产羔数显著高于tt基因型(p<0.05);zbp1g.2566t>c位点在黑头羊群体中的头胎产羔数和总体产羔数均无显著差异。上述四个多态性位点在波尔山羊、黑头羊和麻城黑山羊群体中加性效应和显性效应均未达到显著水平(p>0.05)。以上结果表明,在波尔山羊和黑头羊群体中山羊zbp1基因的zbp1g.352c>t位点与总产羔数显著相关;zbp1g.955c>t位点和zbp1g.1880g>a在波尔山羊群体中与总产羔数显著相关;zbp1g.2566t>c在波尔山羊和麻城黑山羊群体中与总产羔数显著相关。上述四个snp位点所组成的单倍型可作为山羊产羔数性状遗传改良的新的单倍型分子标记。
所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包括在本发明的保护范围之内。
序列表
<110>湖北省农业科学院畜牧兽医研究所
<120>山羊zbp1基因第一内含子内与产羔数关联的单倍型标记及其应用
<160>17
<170>siposequencelisting1.0
<210>1
<211>2516
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>allele
<222>(249)..(249)
<223>y=c或t
<220>
<221>allele
<222>(852)..(852)
<223>y=c或t
<220>
<221>allele
<222>(1777)..(1777)
<223>r=g或a
<220>
<221>allele
<222>(2463)..(2463)
<223>y=c或t
<400>1
gtacttttttccttctaaggacccgcgggagtgcccgggcgggacttaacttttcttctg60
gttccttttcttggttcagatgacaaccacgataggctaatacctgaaggcaccaccgta120
acaagagagggagagagcattgcatcgggttcaatccgtgtgcgttttgcaggaactctc180
aggggaagcccgctgaggttcccacctcactcccgagatcacaggcaacacaggtgaaat240
gactcaggyggctgagcgtggggcctgaacacctgccttcactttcgttttgaggaagga300
gggcagttgctttgcttgtatgtggctttcctcctgctgctgttcttttattgaatggcg360
cacacgaaacttaactggcactgaacatagctgattcctagggtccccaccgaaagggtg420
tgggctgggggctgaggtgctttttgtctgtctttgttctgtgatcagtaactatctgaa480
cctcaccatgatctgaagtagtcatgaatctcaccatttcaaagatgggtaaatggaggt540
cacacagctggtaggtggcagagttttggctcaaacccaggaaaatatgctgtgtcaggg600
aggggggttaactgggagtggcccagaatcccagggtgggttcccaggttcctgttgtat660
tttcccaaggagacttgtcaggggcactgggtaactgaatttaactttcccagcaggttt720
tccttttaactgatgtatcgtttacacctcatcccattttgcaatttctggttcagcagc780
cagaagactcagtgggggaggcaagcccagccttcactgcatggaccccatttcctcttt840
ggggtyaggggactgcaggaggaggaagagagataggctcacccaggagcaggtttttca900
ggaatcaaaactcttttcaaggctaggcaaagcatcagtgggtgggacgggtatgacagt960
ctcgggggtagtgtttagtgagtgaagttcaggctgggtgaccccaggggttgtaatgca1020
catcacctgcccccagaagaaagcaaacattccacccacaagagcaagccacagtgccca1080
gcgcagagctggcaaacaggtcatcccctagtgctgcccactcagaatccagcaagaacc1140
cagaagtcaaagggttgggctgctgccctgggagggtaacttggtcttcccaagaaaggc1200
cctgctttgcattcccatggtgttcccaggggcagacactttctctcccagcgccatgca1260
ctgttggctctgcgcccaccagttctctcactccccacagggcagccggcttgggtttat1320
gggagcctgcctgggaggagtagaggctatttggaaagttcctctttgagcctagaggca1380
tcctgcttaggaccattaactctggcagaggggaggcggaaacagaggccttggaggaga1440
gcccatcccctgcccaggccatctgagcacacatctacccagtagtggacactcactctt1500
tcatccagtccctcctccatccaaatgctgttaatagttcgtgagaccctactgcatgcc1560
tggcccatggctagacacagaaccactacagtgaacctggcagtcacagcagcggatccc1620
tggggacactctgcaggggacccacaggaccagcgtcccacctgctcagggcactcagga1680
cagccttccagcattggagtctgagtgagaactcacactggacaggtggagaggggggga1740
agagcatgcctggcaaagggaatggcatgcrcaaaggcccaggggtgagtatggctgaaa1800
ggaattcaaacaggttggcacttgacgggcagtagagggtgggtaaagatgaggctggtt1860
ctgccagccaagaggagggtggactggagggaacctggacttggtggaactgctgtcatc1920
gaggggatgctgacaaggagtggggagtggaggcagaggaaaggagaaggggttgagaga1980
gtctcgagcggcaccaccaggactccatggttgggcagcagggtgaaagctctgtttctg2040
gatgcctgctgggtttctggggcggggggggatgaacagtgacatcgcccctgtggatgg2100
tgagcccgaggagcagattccgcagggcggggtggatgggggatggagggcaccagggac2160
aggcctcacttccccggctcctctgctccgcccttaccctcctgcatcctgccgcctcca2220
ctctcctctgctctgaggacacctgagggctgactgttctcatctgtcctccagtgctca2280
cacaggcaggggacactcggaaggcctggcagctcctctggccaccagactgtccggccc2340
aggtcacttctgagcagcccacgcgcccctccctgaggacaaggagacactgatgggtgt2400
ctgcttcaacctctgttcctccgtctgccctagacccctctctggggggcgcctgayggc2460
ccaggctgggtcgtggggacctcagttctgtctctgcccctgtctccctctcccag2516
<210>2
<211>270
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>allele
<222>(141)..(141)
<223>y=c或t
<400>2
ggcaccaccgtaacaagagagggagagagcattgcatcgggttcaatccgtgtgcgtttt60
gcaggaactctcaggggaagcccgctgaggttcccacctcactcccgagatcacaggcaa120
cacaggtgaaatgactcaggygaccacggctgagcgtggggcctgaacacctgccttcac180
tttcgttttgaggaaggagggcagttgctttgcttgtatgtggctttcctcctgctgctg240
ttcttttattgaatggcgcacacgaaactt270
<210>3
<211>234
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>allele
<222>(80)..(80)
<223>y=c或t
<400>3
tctggttcagcagccagaagactcagtgggggaggcaagcccagccttcactgcatggac60
cccatttcctctttggggtyaggggactgcaggaggaggaagagagataggctcacccag120
gagcaggtttttcaggaatcaaaactcttttcaaggctaggcaaagcatcagtgggtgga180
acgggtatgacagtctcgggggtagtgtttagtgagtgaagttcaggctgggtg234
<210>4
<211>267
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>allele
<222>(136)..(136)
<223>r=g或a
<400>4
ggacccacacaggaccagcgtcccacctgctcagggcactcaggacagccttccagcatt60
ggagtctgagtgagaactcacactggacaggtggagagggggggaagagcatgcctggca120
aagggaatggcatgcrcaaaggcccaggggtgagtatggctgaaaggaattcaaacaggt180
tggcacttgacgggcagtagagggtgggtaaagatgaggctggttctgccagccaagagg240
agggtggactggagggaacctggactt267
<210>5
<211>302
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>allele
<222>(231)..(231)
<223>y=c或t
<400>5
tctgctctgaggacacctgagggctgactgttctcatctgtcctccagtgctcacacagg60
caggggacactcggaaggcctggcagctcctctggccaccagactgtccggcccaggtca120
cttctgagcagcccacgcgcccctccctgaggacaaggagacactgatgggtgtctgctt180
caacctctgtccctccgtctgccctagacccctctctggggggcgcctgayggcccaggc240
tgggtcgtggggacctcagttctgtctctgcccctgtctccctctcccagaccttgagca300
ga302
<210>6
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>6
ggcaccaccgtaacaagaga20
<210>7
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>7
aagtttcgtgtgcgccattc20
<210>8
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>8
tctggttcagcagccagaag20
<210>9
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>9
cacccagcctgaacttcact20
<210>10
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>10
ggacccacacaggaccag18
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
aagtccaggttccctccagt20
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
tctgctctgaggacacctga20
<210>13
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>13
tctgctcaaggtctgggaga20
<210>14
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>14
tttttttttacacaggtgaaatgactcagg30
<210>15
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>15
ttttttttttttttttttttcctcctgcagtcccct36
<210>16
<211>57
<212>dna
<213>人工序列(artificialsequence)
<400>16
ttttttttttttttttttttttttttttttttttttttttcaaagggaatggcatgc57
<210>17
<211>52
<212>dna
<213>人工序列(artificialsequence)
<400>17
tttttttttttttttttttttttttttttttttttttttggggggcgcctga52
- 还没有人留言评论。精彩留言会获得点赞!