用于RNA-染色质相互作用分析的组合物及其用途的制作方法
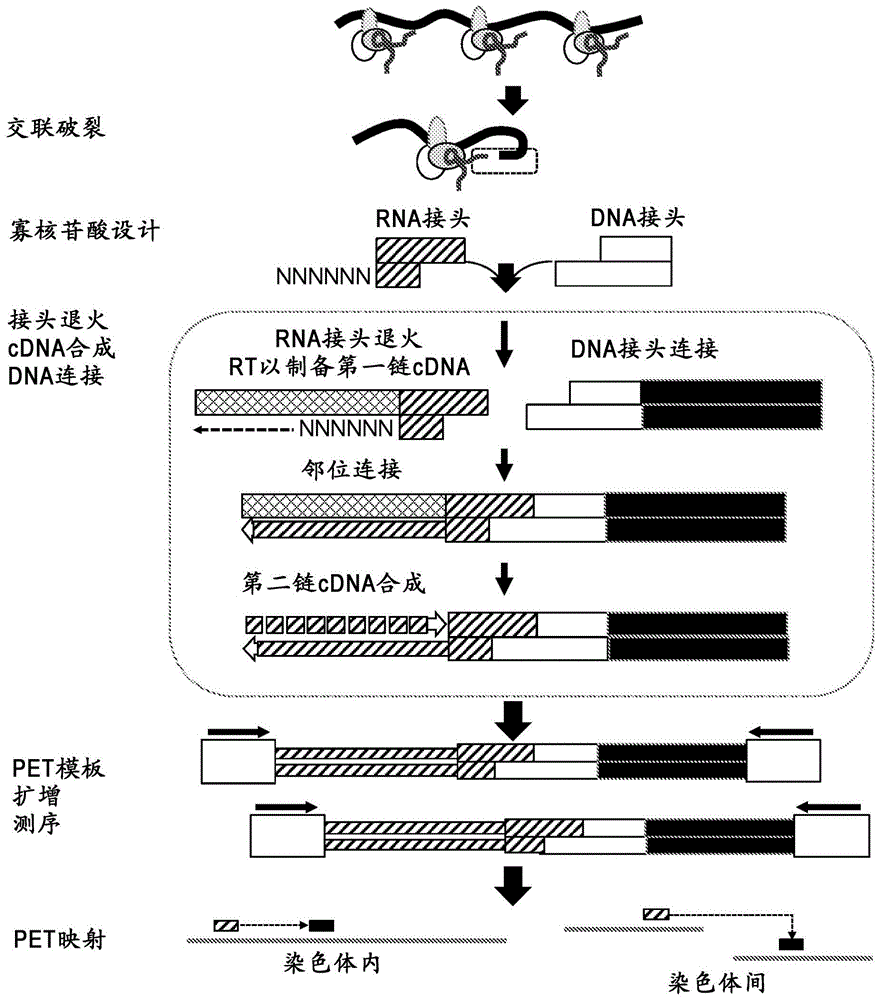
本申请是申请日为2014年9月5日、申请号为201480049110.5、发明名称为“用于rna-染色质相互作用分析的组合物及其用途”的发明专利申请的分案申请。相关申请的交叉引用本申请要求2013年9月5日提交的美国临时申请no.61/873,928的优先权和申请日权益,其全部内容通过引用并入本文。
背景技术:
:目前,非编码rna(ncrna)据信在基因组中普遍地转录,且已经鉴别了大量的ncrna。但是,与此不相称地,关于其功能性作用仍然知道的非常少。许多已知的ncrna功能通过扰动实验(perturbationexperiment)推导,其缺乏ncrna与哪种特异性靶标相互作用的细节。如clip/rip-seq和chirp-seq的技术提供了一些ncrna与哪些蛋白质因子和染色质位点相互作用的大量认识。但是,当前的方法限于一次一个地检验ncrna或相互作用靶标。因此希望的是具有无偏的全基因组策略以鉴定所有ncrna的功能性靶标。技术实现要素:本发明的一个方面提供试剂盒,包含(1)rna接头,其包含:(i)第一多核苷酸和(ii)第二多核苷酸,其中第一和第二多核苷酸形成侧接第一连接相容末端和第一多核苷酸3’-末端的3’-悬端的第一双链区域,其中3’-悬端包含随机序列引物;和(2)dna接头,其包含(iii)第三多核苷酸和(iv)第四多核苷酸,其中第三和第四多核苷酸形成侧接平端和第二连接相容末端的第二双链区域,其中第一和第二连接相容末端彼此连接或可适应于彼此连接。在某些实施方式中,第一连接相容末端是第二多核苷酸3’-末端的3’-悬端,且第二连接相容末端是第三多核苷酸3’-末端的3’-悬端,其中两个3’-悬端彼此退火而连接。在某些实施方式中,第一双链区域包含用于切割随机序列引物3’侧的第一限制性内切酶(re)的第一识别位点。在某些实施方式中,第二双链区域包含用于切割第三多核苷酸5’侧的第二限制性内切酶(re)的第二识别位点。在某些实施方式中,所述第一、第二、第三和第四多核苷酸中的一个或多个是dna。在某些实施方式中,所述第一、第二、第三和第四多核苷酸的一个或多个包含修饰的核苷酸。在某些实施方式中,修饰的核苷酸是生物素化的t(胸苷)。在某些实施方式中,第一多核苷酸包含多个多核苷酸,其中各多核苷酸仅在随机序列引物区域不同。在某些实施方式中,第一多核苷酸包含具有相同随机序列引物的多核苷酸的同质群体。在某些实施方式中,随机序列引物包含4、5、6、7、8或更多个核苷酸。在某些实施方式中,第一双链区域包含区分rna接头和dna接头的独特序列。在某些实施方式中,第二双链区域包含区分rna接头和dna接头的独特序。在某些实施方式中,第一识别位点的最后一个核苷酸是随机序列引物5’侧的最后一个碱基配对的核苷酸。在某些实施方式中,第二识别位点的最后一个核苷酸是平端处的碱基配对的核苷酸。在某些实施方式中,第一和第二限制性内切酶是相同的。在某些实施方式中,第一或第二限制性内切酶独立地选自:aari、aceiii、aloi、baei、bbr7i、bbvi、bbvii、bcci、bce83i、bceai、bcefi、bcgi、bcivi、bfii、bini、bpli、bsaxi、bscai、bsemii、bseri、bsgi、bsmi、bsmai、bsmfi、bsp24i、bspcni、bspmi、bsri、bsrdi、bstf5i、btgzi、btsi、cjei、cjepi、ecii、eco31i、eco57i、eco57mi、ecop15i、esp3i、fali、faui、foki、gsui、haeiv、hgai、hin4i、hphi、hpyav、ksp632i、mboii、mlyi、mmei、mnli、plei、ppii、psri、rleai、sapi、sfani、sspd5i、sth132i、stsi、taqii、tspdti、tspgwi、tspri或tth111ii。在某些实施方式中,第一或第二限制性内切酶的切割位点是识别位点的最后一个核苷酸3’侧的至少约10、12、14、16、18、20、22、24、26、28、30或更多个核苷酸。在某些实施方式中,第一和第四多核苷酸是去磷酸化的。在某些实施方式中,试剂盒还包含交联蛋白质和多核苷酸的试剂。在某些实施方式中,该试剂包含甲醛。在某些实施方式中,试剂盒还包含特异性或选择性结合染色质组分(例如,组蛋白)的亲和试剂(例如,抗体或单克隆抗体)。在某些实施方式中,试剂盒还包含将含有损伤的或不相容的5’-和/或3’-突出末端的dna转化为5’-磷酸化的平端dna的末端修复混合物。在某些实施方式中,试剂盒还包含dna连接酶(例如,t4连接酶)。在某些实施方式中,试剂盒还包含逆转蛋白质和多核苷酸(例如,蛋白酶k)的交联的试剂。在某些实施方式中,试剂盒还包含第一和/或第二限制性内切酶。在某些实施方式中,试剂盒还包含用于平端双链dna的pcr扩增的一对串联的连接体(concatenatingadapter)。在某些实施方式中,试剂盒还包含taqdna聚合酶。在某些实施方式中,试剂盒还包含逆转录酶。本发明的另一方面提供末端配对标签(paired-endtag)(pet)多核苷酸,包含含有所述rna和dna接头的第一和第二双链区域的中心区域,所述中心区域:(1)在接近所述第一双链区域的位点处侧接非编码rna(ncrna)的序列标签;和(2)在接近所述第二双链区域的位点处侧接基因组dna的序列标签。在某些实施方式中,非编码rna(ncrna)的序列标签具有由所述第一限制性内切酶的消化产生的自由端。在某些实施方式中,非编码rna(ncrna)的序列标签唯一地鉴别转录ncrna的基因组区域。在某些实施方式中,非编码rna(ncrna)的序列标签长度为约8-30个碱基对。在某些实施方式中,基因组dna的序列标签具有由所述第二限制性内切酶的消化产生的自由端。在某些实施方式中,基因组dna的序列标签唯一地鉴别基因组dna所在的基因组区域。在某些实施方式中,基因组dna的序列标签长度为约8-30个碱基对。本发明的另一方面提供末端配对标签(pet)文库,包含两个或更多个所述pet多核苷酸的成员,其中pet文库的各成员包含相同的所述中心区域,及不同的所述非编码rna(ncrna)的序列标签或不同的所述基因组dna的序列标签或两者。本发明的另一方面提供包含所述pet多核苷酸的载体。在某些实施方式中,所述载体包含多个串联的所述pet多核苷酸。本发明的另一方面提供两个或更多个所述pet多核苷酸的串联体。本发明的另一方面提供鉴别基因组内基因组的非编码rna(ncrna)的功能性相互作用位点的方法,该方法包括:(1)提供包含交联的基因组dna片段和交联的ncrna的染色质;(2)使用权利要求1的rna接头和dna接头,在用于邻位连接(proximityligation)的条件下,将交联的基因组dna片段的末端与交联的ncrna的cdna的末端连接,其中所述交联的基因组dna片段的末端连接于dna接头,且所述交联的ncrna的cdna的末端包含rna接头;(3)分离权利要求29的pet多核苷酸用于测序分析;和(4)将各所述pet多核苷酸内的基因组dna的序列标签和ncrna的序列标签映射到参照基因组,从而鉴别参照基因组内参照基因组的所述非编码rna(ncrna)的功能性相互作用位点。在某些实施方式中,ncrna和基因组dna在活细胞中通过甲醛介导的交联进行交联。在某些实施方式中,染色质片段通过超声处理生成。在某些实施方式中,交联的ncrna的cdna包含从rna接头的随机序列引物和ncrna模板逆转录的第一链cdna。在某些实施方式中,第二链cdna合成在邻位连接之后但在步骤(3)之前进行。在某些实施方式中,该方法还包括在步骤(2)之前将交联的基因组dna片段的末端修复为5’-磷酸化的平端dna。在某些实施方式中,dna接头的第三多核苷酸是去磷酸化的且dna接头不自身连接。在某些实施方式中,该方法还包括鉴别具有重叠的基因组dna的序列标签和重叠的ncrna的序列标签的两个或更多个pet多核苷酸的簇。在某些实施方式中,该方法还包括排除包含rrna的序列标签的pet多核苷酸。在某些实施方式中,该方法还包括在步骤(2)之前分离或富集染色质片段的亚集。在某些实施方式中,染色质片段的亚集使用对于染色质片段亚集的蛋白质组分特异性的抗体通过免疫沉淀进行分离或富集。在某些实施方式中,蛋白质组分是组蛋白、转录因子、多梳(pcg)家族蛋白、重组参与因子(recombinationinvolvedfactor)、染色质隔离子或染色质waver、甲基-cpg-结合蛋白或rna结合蛋白。应理解,为实施本发明的一个实施方式(如仅在实施例部分中描述的实施方式)而公开的任何说明,包括但不限于任何技术、试剂、实验条件、限制性位点、酶、载体、引物等等,也可以与本发明的其它实施方式结合使用,包括仅在本发明的一个(而非另外的任何)实施方式中详细描述的那些实施方式。本领域技术人员很清楚如何使对于其它实施方式公开的技术和材料适应于本发明的当前实施方式。1.一种试剂盒,包含:(1)rna接头,其包含:(i)第一多核苷酸,和(ii)第二多核苷酸,其中所述第一和第二多核苷酸形成侧接第一连接相容末端和在所述第一多核苷酸3’-末端的3’-悬端的第一双链区域,其中所述3’-悬端包含随机序列引物;和(2)dna接头,其包含:(iii)第三多核苷酸,和(iv)第四多核苷酸,其中所述第三和第四多核苷酸形成侧接平端和第二连接相容末端的第二双链区域,其中所述第一和第二连接相容末端彼此连接或适应于彼此连接。2.项目1的试剂盒,其中所述第一连接相容末端是所述第二多核苷酸3’-端的3’-悬端,和所述第二连接相容末端是所述第三多核苷酸3’-端的3’-悬端,其中两个3’-悬端彼此退火以连接。3.项目1的试剂盒,其中所述第一双链区域包含切割所述随机序列引物3’侧的第一限制性内切酶(re)的第一识别位点。4.项目1的试剂盒,其中所述第二双链区域包含切割所述第三多核苷酸5’侧的第二限制性内切酶(re)的第二识别位点。5.项目1的试剂盒,其中所述第一、第二、第三和第四多核苷酸中的一个或多个是dna。6.项目1的试剂盒,其中所述第一、第二、第三和第四多核苷酸中的一个或多个包含修饰的核苷酸。7.项目6的试剂盒,其中所述修饰的核苷酸是生物素化的t(胸苷)。8.项目1的试剂盒,其中所述第一多核苷酸包含多个多核苷酸,其中各多核苷酸仅在随机序列引物区域处不同。9.项目1的试剂盒,其中所述第一多核苷酸包含具有相同随机序列引物的多核苷酸的同质群体。10.项目1的试剂盒,其中所述随机序列引物包含4、5、6、7、8或更多个核苷酸。11.项目1的试剂盒,其中所述第一双链区域包含区分所述rna接头与所述dna接头的独特序列。12.项目1的试剂盒,其中所述第二双链区域包含区分所述rna接头与所述dna接头的独特序列。13.项目1的试剂盒,其中所述第一识别位点的最后一个核苷酸是所述随机序列引物5’侧的最后一个碱基配对的核苷酸。14.项目1的试剂盒,其中所述第二识别位点的最后一个核苷酸是所述平端处的碱基配对的核苷酸。15.项目1的试剂盒,其中所述第一和第二限制性内切酶是相同的。16.项目1的试剂盒,其中所述第一和第二限制性内切酶独立地选自:aari、aceiii、aloi、baei、bbr7i、bbvi、bbvii、bcci、bce83i、bceai、bcefi、bcgi、bcivi、bfii、bini、bpli、bsaxi、bscai、bsemii、bseri、bsgi、bsmi、bsmai、bsmfi、bsp24i、bspcni、bspmi、bsri、bsrdi、bstf5i、btgzi、btsi、cjei、cjepi、ecii、eco31i、eco57i、eco57mi、ecop15i、esp3i、fali、faui、foki、gsui、haeiv、hgai、hin4i、hphi、hpyav、ksp632i、mboii、mlyi、mmei、mnli、plei、ppii、psri、rleai、sapi、sfani、sspd5i、sth132i、stsi、taqii、tspdti、tspgwi、tspri或tth111ii。17.项目1的试剂盒,其中所述第一或第二限制性内切酶的切割位点是所述识别位点的最后一个核苷酸3’侧的至少约10、12、14、16、18、20或更多个核苷酸。18.项目1的试剂盒,其中所述第一和第四多核苷酸是去磷酸化的。19.项目1的试剂盒,还包含交联蛋白质和多核苷酸的试剂。20.项目19的试剂盒,其中所述试剂包含甲醛。21.项目1的试剂盒,还包含特异性地或选择性地结合染色质的组分(例如,组蛋白)的亲和试剂(例如,抗体或单克隆抗体)。22.项目1的试剂盒,还包含将含有损伤的或不相容的5’-和/或3’-突出末端的dna转化为5’-磷酸化的平端dna的末端修复混合物。23.项目1的试剂盒,还包含dna连接酶(例如,t4连接酶)。24.项目1的试剂盒,还包含逆转蛋白质和多核苷酸的交联的试剂(例如,蛋白酶k)。25.项目1的试剂盒,还包含所述第一和/或第二限制性内切酶。26.项目1的试剂盒,还包含用于平端双链dna的pcr扩增的一对串联的连接体。27.项目1的试剂盒,还包含taqdna聚合酶。28.项目1的试剂盒,还包含逆转录酶。29.一种末端配对标签(pet)多核苷酸,包含含有项目1的第一和第二双链区域的中心区域,所述中心区域:(1)在接近所述第一双链区域的位点处侧接非编码rna(ncrna)的序列标签;和(2)在接近所述第二双链区域的位点处侧接基因组dna的序列标签。30.项目29的pet多核苷酸,其中所述非编码rna(ncrna)的序列标签具有由所述第一限制性内切酶的消化产生的自由端。31.项目29的pet多核苷酸,其中所述非编码rna(ncrna)的序列标签唯一地鉴别转录所述ncrna的基因组区域。32.项目29的pet多核苷酸,其中所述非编码rna(ncrna)的序列标签长度为约8-30个碱基对。33.项目29的pet多核苷酸,其中所述基因组dna的序列标签具有由所述第二限制性内切酶的消化产生的自由端。34.项目29的pet多核苷酸,其中所述基因组dna的序列标签唯一地鉴别所述基因组dna所在的基因组区域。35.项目29的pet多核苷酸,其中所述基因组dna的序列标签长度为约8-30个碱基对。36.一种末端配对标签(pet)文库,包含两个或更多个项目29的pet多核苷酸的成员,其中所述pet文库的各成员包含相同的所述中心区域,及不同的项目29的所述非编码rna(ncrna)的序列标签或不同的项目29的所述基因组dna的序列标签或两者。37.一种载体,包含项目29的pet多核苷酸。38.项目37的载体,包含多个串联的项目29的pet多核苷酸。39.两个或更多个项目29的pet多核苷酸的串联体。40.一种鉴别基因组内基因组的非编码rna(ncrna)的功能性相互作用位点的方法,该方法包括:(1)提供包含交联的基因组dna片段和交联的ncrna的染色质;(2)使用项目1的所述rna接头和dna接头,在用于邻位连接的条件下,将交联的基因组dna片段的末端与交联的ncrna的cdna的末端连接,其中所述交联的基因组dna片段的末端连接于所述dna接头,且所述交联的ncrna的cdna的末端包含所述rna接头;(3)分离项目29的pet多核苷酸用于测序分析;和(4)将各所述pet多核苷酸内的所述基因组dna的序列标签和所述ncrna的序列标签映射到参照基因组,从而鉴别所述参照基因组内所述参照基因组的所述非编码rna(ncrna)的功能性相互作用位点。41.项目40的方法,其中所述ncrna和所述基因组dna在活细胞中通过甲醛介导的交联进行交联。42.项目40的方法,其中染色质片段通过超声处理生成。43.项目40的方法,其中所述交联的ncrna的cdna包含从所述rna接头的随机序列引物和所述ncrna模板逆转录的第一链cdna。44.项目40的方法,其中第二链cdna合成在邻位连接之后但在步骤(3)之前进行。45.项目40的方法,还包括在步骤(2)之前将交联的基因组dna片段的末端修复为5’-磷酸化的平端dna。46.项目40的方法,其中所述dna接头的第三多核苷酸是去磷酸化的且所述dna接头不自身连接。47.项目40的方法,还包括鉴别具有重叠的基因组dna的序列标签和重叠的ncrna的序列标签的两个或更多个pet多核苷酸的簇。48.项目47的方法,还包括排除包含rrna的序列标签的pet多核苷酸。49.项目40的方法,还包括在步骤(2)之前分离或富集染色质片段的亚集。50.项目49的方法,其中所述染色质片段的亚集使用对于所述染色质片段亚集的蛋白质组分特异性的抗体通过免疫沉淀进行分离或富集。51.项目50的方法,其中所述蛋白质组分是组蛋白、转录因子、多梳(pcg)家族蛋白;重组参与因子;染色质隔离子或染色质waver;甲基-cpg结合蛋白或rna结合蛋白。附图说明图1a显示使用rna接头和dna接头对的rich-pet方法的典型设置的示意流程图。ncrna与染色质的相互作用通过交联,接着将染色质纤维破裂成与rna、dna和蛋白质组分的系留复合物而捕获。在各染色质片段复合物中的系留rna和dna然后通过由特别设计的rna接头和dna接头寡核苷酸(其也具有用于定向和特异性的独特序列条码)介导的一系列连接反应进行连接。在各染色质复合物内,rna的3’-末端与rna接头的随机六聚体突出部分退火,接着进行逆转录用于cdna合成。同时,dna接头通过连接反应添加到系留的dna片段的平端。在洗掉过量接头寡聚物后,附接的rna和dna接头彼此连接,因此连接系留的rna和dna分子。在反交联后,杂合连接产物通过剪切或限制性消化碎裂成用于进一步扩增、测序和映射分析的所需大小以鉴别其中rna被转录的位置和其中其在基因组中相互作用的位置。图1b显示使用修饰的rna接头的rich-pet方法的典型设置的示意流程图。图1c显示使用直接rna接头的rich-pet方法的典型设置的示意流程图。“app”代表第一多核苷酸5’-末端的5’腺苷酸化。图2a-2c呈现了所选择的rich-pet文库的统计及测序和映射数据。图2a显示单态(singleton)pet(没有与其它pet序列的重叠)和pet簇的比例。使用pet簇数据,鉴别了大约700个rna位点和大约5000个dna位点。图2b显示通过rich-pet数据鉴别的rna和dna位点处的rna-seq数据强度。图2c显示限定ncrna相互作用的大多数rich-pet数据是反式相互作用的和染色体间的。图3显示所述方法的可重现性和灵敏度。该图显示代表性散点图,其显示了在技术和生物学重复中鉴别的rna相互作用位点的比较。已知的lncrnamalat1(pet计数174)和neat1(pet18)在rich-pet数据(未示出)中重复地检测。rnapiichia-pet数据也表明这两个ncrna也在可能用于共调节的相同rnapii转录复合物内空间连接。另外,rna-seq和rna-pet数据用于评估helas3中ncrna基因的表达水平(数据未示出)。两种数据都表明malat1高表达,neat1以中等水平表达和hotair以非常低的水平表达。映射在hotair位点的rich-pet在这一区域中显示差的rich-pet数据(数据未示出)。图4a-4b显示用于验证neat1和malat1rich-pet数据的数据。图4a显示neat1和malat1均在helas3细胞中表达,且在rich-pet数据中大量检测到。neat1仅限制于顺式作用,因为rna和dna标签两者映射在相同位点内的短距离中。malat1大部分是反式作用的,因为大多数dna标签映射在相同染色体的大距离中或在不同染色体(插入体)中。图4b显示人a549和helas3中的rna-fish实验。neat1探针产生很少的荧光斑(helas3细胞中每细胞核1-2个),而malat1探针产生多得多的斑点(helas3细胞中每细胞核13个)。计数是基于100个细胞核/探针/实验。图5a-5b表征rich-pet数据。图5a显示基因组中rna标签簇位置的类别的饼图,表明绝大多数的rna标签在推定的ncrna区域中发现,仅3%与蛋白质编码外显子重叠。检测到许多已知的ncrna,且鉴别许多新的ncrna。图5b显示基因组中dna标签簇位置的类别的饼图,表明大多数的dna标签簇映射到蛋白质编码区域,大部分在启动子或内含子中。图6a-6b显示malat1相互作用的多靶标和多功能。图6a是与59个基因组位点相互作用的malat1的关联性图谱。图6b是显示在其启动子区域中存在malat1的基因比在其内含子区域具有malat1相互作用的基因具有更高的rna-seq阅读的箱图。在rnapiichip-seq强度(未示出)的聚集图(aggregationplot)中,在其启动子区域中存在malat1的基因比在其内含子区域具有malat1相互作用的基因具有更高的rna-seq阅读。图7显示ccat1及其lncrna转录物作为几种靶基因的转录激活因子或共激活因子发挥作用的示意图。图8a显示人类染色体8上对应于seqidno:9的基因组ccat1和cdna序列的位置。图8b显示人类染色体8上对应于seqidno:1-8(分别ccat1_jax_1至_8)的八个另外的ccat1基因组和cdna序列的位置。填充的框代表外显子序列,而连接外显子序列的线代表内含子序列。图9a显示通过rna-seq数据测量的xist(其特别地靶向女性细胞中的x染色体)的计数,其以每百万阅读每kb的阅读数(rpkm)计。图9b显示xist结合覆盖的各染色体的比例。具体实施方式1.概述本文描述的本发明部分地基于以下认识:如果ncrna在核空间中具有表观遗传调控作用,则其在染色体中的特定位置处必然直接或间接地与染色质相互作用,其中发生调节染色质状态和靶基因活性的功能。因此,本文描述的本发明提供通过rna-dna连接,接着末端配对测序(rich-pet)来整体映射ncrna-染色质相互作用的新途径。简而言之,本文描述的组合物可用于包括三个主要部分的方法中:1)染色质交联以捕获活细胞(如体外培养的一种活细胞或从组织样品获得的原代细胞)中rna、dna和蛋白质之间(优选所有的)分子相互作用事件;2)系留的相互作用rna与染色质dna片段的连接(例如,通过特别设计的接头,如rna接头和dna接头对,或通过连接rna3’末端到5’腺苷酸化ssdna或5’腺苷酸化悬端);和3)rna-dna连接产物或由其来源的标签序列(例如,pet多核苷酸)的测序和映射分析以定位基因组中ncrna的转录位点和它们的染色质靶位点。因此,本发明的一个方面提供鉴别基因组内基因组的非编码rna(ncrna)的功能性相互作用位点的方法,该方法包括(1)提供包含交联的基因组dna片段和交联的ncrna(或其片段)的染色质片段;(2)在用于邻位连接的条件下连接交联的基因组dna片段的末端到交联的ncrna的末端;(3)分离用于测序分析的末端配对标签(pet)多核苷酸,其中pet多核苷酸包含非编码rna(ncrna)的序列标签和基因组dna的序列标签;和(4)映射基因组dna的序列标签和ncrna的序列标签到参照基因组,从而鉴别参照基因组内参照基因组的非编码rna(ncrna)的功能性相互作用位点。这一rna-dna连接途径不仅应用于所有ncrna-染色质相互作用的整体研究,而且可应用于研究特定染色质位置的rna-蛋白质相互作用。因此,基于染色体免疫沉淀(chip)的rich-pet方法可提供rna-蛋白质-染色质相互作用信息的额外特异性。本发明的试剂和方法具有广泛的研究、开发、药物靶标鉴别、药物筛选、诊断、治疗/功效监测、预后等中的潜在用途。例如,本发明的试剂和方法可用于综合地表征多种建立的细胞系、干细胞、ips细胞和来自初级组织的细胞(如源自癌症和健康组织对照的细胞)的ncrna-染色质相互作用组;和显著地提高我们研究调节基因组输出的rna功能的极其复杂的世界的能力。成功地完成rna-染色质相互作用组的表征会提供大多数(如果不是所有)ncrna种类的全面染色质地址薄,其增加了基因组信息的另一维度以帮助理解在健康和疾病状况中基因组如何发挥功能。本发明的几种特定实施方式以下更详细地描述。a)rna接头和dna接头对在第一特定实施方式中,本发明的方法可以使用rna接头和dna接头对完成以连接相同染色质片段中的交联的rna和染色体dna。因此,本发明的一个方面提供试剂盒,包含(1)rna接头,其包含:(i)第一多核苷酸和(ii)第二多核苷酸,其中第一和第二多核苷酸形成侧接第一连接相容末端和第一多核苷酸3’-末端的3’-悬端的第一双链区域,其中3’-悬端包含随机序列引物;和(2)dna接头,其包含:(iii)第三多核苷酸和(iv)第四多核苷酸,其中第三和第四多核苷酸形成侧接平端和第二连接相容末端第二双链区域,其中第一和第二连接相容末端彼此连接或可适应于彼此连接。在某些实施方式中,第一连接相容末端是第二多核苷酸3’-末端的3’-悬端,且第二连接相容末端是第三多核苷酸3’-末端的3’-悬端,其中两个3’-悬端彼此退火用于连接。在某些实施方式中,第一连接相容末端是第一多核苷酸5’-末端的5’-悬端,且第二连接相容末端是第四多核苷酸5’-末端的5’-悬端,其中两个5’-悬端彼此退火用于连接。在某些实施方式中,第一和/或第二连接相容末端可适应于连接。例如,代替具有用于连接的必要3’或5’悬端,第一和/或第二连接相容末端可以包含限制性内切酶(re)位点,其可以被re切割以产生连接所需要的必要3’或5’悬端。但是,在通过限制性内切酶切割之前,连接相容性末端可以是平端的(例如,去磷酸化的平端以防止自身连接),或具有防止自身连接或与其它连接相容末端连接的不相容悬端。在某些实施方式中,相容连接末端处的两个5’-或3’-悬端不自身退火且不彼此退火。这可以例如通过设计悬端的序列以使得悬端序列不自身退火且不彼此退火(至少在使用接头的条件下)来完成。这一设计在其中例如下游步骤包括pcr扩增的某些实施方式中可能是有利的。一种频繁观察到的非特异性扩增产物的类型是称为“引物二聚体”的扩增反应的非模板依赖性的人工制品,其是长度通常接近于两个引物长度的总和并表现为在一个引物延伸超出另一引物时出现的双链片段。所得的延伸产物形成不需要的模板,其由于其短的长度而有效地扩增。第一、第二、第三和第四多核苷酸各自可以在单独的容器中提供,如合成的多核苷酸,其为冷冻干燥的、冻干形式或在水或合适的缓冲溶液中。可选地,第一和第二多核苷酸可以组合在相同容器中(冻干的或在溶液中),例如,以1:1的摩尔比,以使得它们可以作为预退火的rna接头使用。类似地,第三和第四多核苷酸可以组合在相同容器中(冻干的或在溶液中),例如,以1:1的摩尔比,以使得它们可以作为预退火的rna接头使用。第二、第三和第四多核苷酸是基本上同质的或纯的(例如,同一容器中的单个多核苷酸分子是相同的),而3’-悬端区域中第一多核苷酸3’-末端包含随机序列引物(例如,同一容器中的单个第一多核苷酸分子是相同的,除了各自在3’-悬端区域内可以具有不同的随机序列引物)。因此,第一多核苷酸可以是独特的,因为它事实上是仅在单个多核苷酸的随机序列引物区域处不同的多核苷酸的混合物。但是,在相关的实施方式中,当对具有限定的3’-末端序列的特定ncrna感兴趣时,本发明的第一多核苷酸可以在随机序列引物区域处同质地包含相同的匹配序列,以特别地从具有限定的3’-末端序列的特定ncrna启动第一链cdna合成。随机序列引物一般具有足够的长度(例如,六聚体),从而能够指导从非编码rna的3’-末端的第一链cdna合成。虽然可以使用六聚体随机序列,但也可以使用其它长度,如4、5、7、8、9、10、11、12随机序列引物。在某些实施方式中,随机序列引物的大多数3’-末端不是脱氧胸苷(t)或尿苷(u),或者可以与mrna的多聚a尾中的腺嘌呤(a)碱基配对的其它核苷酸类似物。这种设计可以进一步帮助避免mrna的多聚a尾的逆转录。第二和第三多核苷酸3’-末端的5’-或3’-悬端(第一和第二连接相容末端)设计为互补的以使得它们彼此退火。第二和第三多核苷酸中悬端区域的长度可以是相同的,但不是必须相同的。在某些实施方式中,两个多核苷酸的悬端区域中的约2、3、4、5、6、7、8或更多个核苷酸是互补的且可以形成碱基对(watson-crick或摆动碱基对)。在某些实施方式中,rna接头上第一双链区域的长度是约6、7、8、9、10、15、20、25、30、35、40、50、60或更多个碱基对。在某些实施方式中,dna接头上第二双链区域的长度是约6、7、8、9、10、15、20、25、30、35、40、50、60或更多个碱基对。在某些实施方式中,连接的rna-dna接头中第一和第二双链区域的总长度是约10、15、20、25、30、35、40、45、50、55、60、65、70、75、80或更多个碱基对。在某些实施方式中,第一双链区域可以包含用于第一限制性内切酶如ii型限制性内切酶(re)的第一识别位点。re识别位点可以策略地布置以使得当re切割时,它在re位点之外、随机序列引物的3’侧进行切割。这允许产生与rna接头连接的rna标签。例如,mmei识别位点可以设置在第一双链区域的末端,第一双链区域另一端的远端(在此处rna接头和dna接头通过其相应3’-悬端区域连接)。mmei位点设计为其定向使得在mmei切割时,在源自连接的ncrna的cdna中产生具有2bp悬端的包含18-bp片段的rna标签。但是,re位点的设置不需要在第一双链区域的末端处。更内部的设置相应地产生较短的rna标签序列。在某些实施方式中,第一识别位点(用于第一(ii型)限制性内切酶)的最后一个核苷酸是随机序列引物5’侧的最后一个碱基配对的核苷酸。同样地,在某些实施方式中,第二双链区域可以包含用于第二限制性内切酶如ii型限制性内切酶(re)的第二识别位点,其可以切割第二re识别位点的3’侧和第三多核苷酸的5’侧。re识别位点的定向以使得其基于连接的基因组dna的末端序列产生dna标签的方式排列。在某些实施方式中,re位点的设置不需要在第二双链区域的末端处。更内部的设置相应地产生较短的dna标签序列。在某些实施方式中,第二识别位点(用于第二(ii型)限制性内切酶)的最后一个核苷酸是平端处的碱基配对的核苷酸。在某些实施方式中,第一和第二(ii型)限制性内切酶是相同的。在其它实施方式中,第一和第二(ii型)限制性内切酶是不同的。对于产生相对长的标签序列的re如i型或iii型re,第一和第二re识别序列的定向可以是相反的,以使得rna接头中的re位点指导dna标签的产生,而dna接头中的re位点指导rna标签的产生。对于识别两个识别位点的re(如iib型re),re位点中的一个可以在rna接头中,且另一个可以在dna接头中,以使得re仅在rna和dna接头如设计的正确连接以构成完全re识别位点时进行切割。可以根据本发明使用的合适限制性内切酶在以下更详细地描述。在某些实施方式中,第一或第二限制性内切酶的切割位点是识别位点的最后一个核苷酸3’侧的至少约10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30或更多个核苷酸。在某些实施方式中,rna接头、dna接头或两者不具有用于产生rna标签或dna标签的限制性内切酶识别位点。在某些实施方式中,第一、第二、第三和第四多核苷酸中的一个或多个是dna(例如,全部是dna),或包含dna和rna核苷酸两者。在其它实施方式中,它们中任一个可以是rna。在某些实施方式中,第一、第二、第三和第四多核苷酸中的一个或多个可以包含修饰的核苷酸。修饰的核苷酸可以是在5’-端、3’-端和/或内部位置处。在某些实施方式中,修饰的核苷酸是生物素化的核苷酸如生物素化的dt(脱氧胸苷)。生物素化的核苷酸的存在允许例如通过使用与生物素结合伴体(如抗生物素蛋白或链霉亲和素)偶联的树脂、琼脂糖、纳米颗粒、金属或磁珠来亲和纯化包含一个或多个这种生物素化的核苷酸的多核苷酸。这类珠然后可以通过磁体分离。生物素化的核苷酸可以存在于rna接头、dna接头或两者中。这一技术也可以与高通量下一代测序如单分子实时测序(pacificbio)、离子半导体(iontorrent测序)、焦磷酸测序(454)、通过合成测序(illumina)、通过连接测序(solid测序)、聚合酶克隆测序(polony测序)、大规模平行签名测序(mpss)、dna纳米球测序、heliscope单分子测序组合,或者可以采用有色珠或用于基于激光或facs的分选的其它抗体与luminex-型系统一起使用。在某些实施方式中,修饰的核苷酸增强随机序列引物通过逆转录合成第一链cdna的能力,如通过增强随机引物与ncrna的3’-末端之间杂交的稳定性和/或特异性。在某些实施方式中,随机引物序列可以包括至少一个含有天然存在的dna和rna中发现的常规2’-脱氧-d-核糖或d-核糖以外的糖的核苷酸,如其中糖通过侧基的添加或取代修饰的或者其中糖是天然存在的dna和rna中发现的常规2’-脱氧-d-核糖或d-核糖的立体异构体的核苷酸或这两者。参见美国专利no.6,794,142(通过引入并入本文)。这样的修饰的核苷酸可以在随机引物序列的3’-末端处或靠近随机引物序列的3’-末端。在一个实施方式中,修饰的随机引物序列基本上由其中三个3′末端核苷酸中的至少一个是选自2′-o-甲基-核苷酸、2′-氨基-核苷酸和2′-氟-核苷酸的修饰核苷酸的寡核苷酸组成。在一个实施方式中,修饰的引物序列基本上由其中三个3′末端核苷酸中的至少一个是选自2′-o-甲基-核糖核苷酸、2′-脱氧-2′-氨基-核苷酸和2′-脱氧-2′-氟-核苷酸的修饰核苷酸的寡核苷酸组成。这些修饰代表向2′oh添加一个部分或者2′-oh被可选的部分替代。在某些实施方式中,随机引物序列包含一个或多个lna或pna。rna中通常热力学稳定的结构片段如发夹的存在可能使得几乎不可能完成引物延伸。用lna-修饰的引物替代dna引物可以克服这一限制(参见fratczak等,biochemistry,48(3):514-6,2009;uppuladinne等,biomol.struct.dyn.,31(6):539-60,2013)。其它修饰的核苷酸,如赋予核苷酸间键对核酸酶降解的抗性的硫代磷酸酯(或磷硫酰,具有一般化学式ps4-xox3-(x=0、1、2或3)的化合物和阴离子家族)修饰、吗啉代寡核苷酸、2’f-ana、2’-o-alkyl等,也可以引入接头中以增强接头的稳定性和耐核酸酶能力。参见verma&eckstein,“modifiedoligonucleotide:synthesisandstrategyforusers,”annu.rev.biochem.,67:99-134,1998(通过引入并入本文)。在某些实施方式中,rna接头和/或dna接头可以包含区分rna接头与dna接头或者区分rna/dna接头与其它rna/dna接头(例如,当一起使用两组或更多组rna接头时)的独特序列(例如,“条码”)。例如,第一和/或第二双链区域可以包含区分rna接头与dna接头的独特序列。这种条码可以简单地是独特序列的小延伸片段,如2-、3-、4-、5-、6-、7-、8-、9-、10-核苷酸的序列(或更多)。在某些实施方式中,rna接头和dna接头的序列中的差异可能足以区分rna接头与dna接头。在某些实施方式中,仅rna接头或仅dna接头具有独特序列/条码。在某些实施方式中,rna接头和dna接头两者具有其相应的独特序列/条码。在某些实施方式中,第一多核苷酸是去磷酸化的。在某些实施方式中,第二多核苷酸是去磷酸化的。在某些实施方式中,第三多核苷酸是去磷酸化的。在某些实施方式中,第四多核苷酸是去磷酸化的。去磷酸化可以帮助避免多核苷酸或dna/rna接头的自身连接,如通过两个dna接头的平端的自身连接,其各自可以连接于相同染色质片段中的染色体dna片段。另外,如果接头或接头的可连接末端去磷酸化,则预期接头不太可能连接以形成接头的二聚体或串联体。此外,预期dna接头可以连接于染色体dna分子的磷酸化末端,但不能连接以将染色体dna分子的末端连接到一起,除非它们磷酸化。在可选的实施方式中,第一和第二多核苷酸可以杂交并形成在一端具有包含第一多核苷酸的随机引物序列的3’-悬端和在另一端具有包含限制性内切酶的识别位点的第一连接相容位点的rna接头。类似地,第三和第四多核苷酸可以杂交并形成在一端具有用于连接于染色体片段的自由端的平端和在另一端具有包含相同限制性内切酶的识别位点或者产生相容的可连接末端的相容限制性内切酶的识别位点的第二连接相容末端的dna接头。因此,限制性内切酶和/或其相容性re的消化产生可用于连接dna和rna接头的悬端(可以是3’或5’悬端)。在这一实施方式中,在限制性内切酶消化之前,dna和rna接头的末端可能不是可连接的(例如,rna接头可以具有5’悬端且dna接头可以具有3’悬端的平端,反之亦然),且这样的末端可以进一步去磷酸化。在re消化后,在dna和rna接头末端处产生可连接的末端,具有适当的磷酸化。dna和rna接头的可连接末端然后可以连接。限制性消化后的可连接末端可以是平端,或具有带5'或3'悬端的粘端。特别地,可以使用很少切割的限制性内切酶以降低在不希望的位置处切割核酸材料和/或产生非常短的片段的可能性。所述多核苷酸可以通过任何合适的方法制备,包括通过如narang等,1979,meth.enzymol.,68:90-99的磷酸三酯方法、如brown等,1979,meth.enzymol.,68:109-151的磷酸二酯方法、如beaucage等,1981,tetrahedronlett.,22:1859-1862的二乙基亚磷酰胺方法和美国专利no.4,458,066的固相载体方法的方法进行的直接化学合成,各文献通过引入并入本文。寡核苷酸和修饰的核苷酸的偶联物的合成方法的综述提供在goodchild,1990,bioconjugatechemistry,1(3):165-187中,其通过引入并入本文。用于实施本发明的方法的一种或多种另外的试剂也可以包括在本发明的试剂盒中。在某些实施方式中,试剂盒还包含交联蛋白质和多核苷酸的试剂,如甲醛(例如,1%甲醛)。在某些实施方式中,试剂盒还包含特异性或选择性地结合染色质的组分(例如,组蛋白或特定的目标crna)的亲和试剂。例如,亲和试剂可以是抗体(如单克隆抗体)或其任何功能性抗原结合片段或衍生物。亲和试剂也可以是与染色质的多核苷酸组分杂交/结合的多核苷酸(如反义多核苷酸)。反义多核苷酸可以被标记以利于后续捕获反义多核苷酸和其互补靶序列之间形成的杂交复合物。例如,标记可以是可由抗生物素蛋白或链霉亲和素包被的珠捕获的生物素标记(如生物素化的u或t)。反义多核苷酸也可以固定在可包装在柱中或用于亲和捕获互补靶序列的批混合物中的固相载体上,如微珠或纳米颗粒的表面上。在某些实施方式中,试剂盒还包含将含有损伤的或不相容的5’-和/或3’-突出端的dna转化为5’-磷酸化的平端dna的末端修复混合物。这类试剂容易商购获得,如来自epicentre的end-ittmdnaend-repairkit。在某些实施方式中,试剂盒还包含dna连接酶(例如,各种商业来源的t4dna连接酶,如newenglandbiolabs(neb))。在某些实施方式中,试剂盒还包含逆转蛋白质和多核苷酸的交联的试剂(例如,各种商业来源的蛋白酶k,如newenglandbiolabs(neb))。在某些实施方式中,试剂盒还包含第一和/或第二限制性内切酶,及任选地re消化所需的任何合适的缓冲剂或辅因子。在某些实施方式中,试剂盒还包含用于pcr扩增平端双链dna的一对串联的连接体。连接体可以包含可用于串联化的限制性内切酶位点,且可以包含适合用于pcr扩增的pcr引物序列。在某些实施方式中,试剂盒还包含用于pcr扩增的taqdna聚合酶或其它形式的扩增(例如,滚环扩增)需要的其它dna聚合酶。在某些实施方式中,试剂盒还包含用于第一链cdna合成的逆转录酶。本发明的另一方面提供包含含有通过第一和第二连接相容末端连接的第一和第二双链区域的中心区域的末端配对标签(pet)多核苷酸,所述中心区域(1)在接近第一双链区域的位点处侧接非编码rna(ncrna)的序列标签;和(2)在接近第二双链区域的位点处侧接基因组dna的序列标签。这样的pet多核苷酸包含rna标签和dna标签两者。其各源自相应ncrna和基因组dna的末端序列(末端配对标签)。总之,末端配对标签代表所观察到的其中ncrna和基因组dna片段在染色体片段中彼此紧密接近的事件或事项。在某些实施方式中,非编码rna(ncrna)的序列标签具有由第一限制性内切酶的消化产生的自由端。限制性内切酶可以是以上描述的任一种,如ii型re(iis、iib、iig型等)、i型re或iii型re,其可以在它们的识别位点之外消化。可选地,自由端可以通过对应于ncrna的cdna上天然存在的re位点产生。优选地,re基于中心区域的序列进行选择以使得re不切割中心区域内部而破坏连接的dna接头和rna接头的结构。在某些实施方式中,ncrna的rna序列标签或基因组dna的dna序列标签具有由物理剪切产生的自由端,如通过超声处理、水力剪切(hydroshearing)、经皮下注射针的重复抽吸等的剪切。在某些实施方式中,ncrna的rna序列标签或基因组dna的dna序列标签具有由非特异性核酸内切酶的有限消化产生的自由端,如micrococcalnuclease(neb目录m0247s)、dnasei(neb目录m0303s)或从双链dna的一端逐步消化的核酸外切酶,或者核酸内切酶和核酸外切酶的组合(例如,exonucleaseiii和mungbeannuclease)以减小交联的基因组dna或ncrna的cdna的平均长度。消化的程度可以通过限制酶或底物浓度、消化的温度和/或ph、辅因子的可用性或其组合进行控制。合适的消化条件可以使用限定长度的标准底物和在消化之前和之后检验消化产物(通过ce(毛细管电泳)的电泳等)来预测试。rna或dna序列标签的长度应当足以唯一地鉴别ncrna从其转录的或基因组dna位于其中的基因组区域。例如,非编码rna(ncrna)的rna序列标签和/或dna序列标签长度对于高等真核生物的相对复杂的基因组可以为约10-100碱基对(或15-50bp、20-40bp、20-30bp、20-25bp),但对于细菌或低等真核生物的相对简单的基因组可以较短(例如,6-10bp、8-10bp、8-12bp)。在相关的方面中,本发明提供末端配对标签(pet)多核苷酸文库,包含两个或更多个所述pet多核苷酸的成员,其中pet文库的各成员包含相同的中心区域及非编码rna(ncrna)的不同rna序列标签、基因组dna的不同dna序列标签或两者。在再另一相关的方面中,本发明提供包含所述pet多核苷酸的载体或重组载体。在某些实施方式中,载体包含多个串联的所述pet多核苷酸。本发明的另一方面提供鉴别基因组内基因组的非编码rna(ncrna)的功能性相互作用位点的方法,该方法包括:(1)提供包含交联的基因组dna片段和交联的ncrna的染色质片段;(2)在用于邻位连接的条件下使用本发明的rna接头和dna接头连接交联的基因组dna片段的末端与交联的ncrna的cdna的末端,其中交联的基因组dna片段的末端连接于dna接头,且交联的ncrna的cdna的末端包含rna接头;(3)分离本发明的pet多核苷酸用于测序分析;和(4)将各pet多核苷酸内的基因组dna的序列标签和ncrna的序列标签映射到基准基因组,从而鉴别基准基因组内基准基因组非编码rna(ncrna)的功能性相互作用位点。在某些实施方式中,本发明的方法使用活细胞进行,如组织培养细胞或从新切割的组织分离的细胞。在某些实施方式中,活细胞中的ncrna和基因组dna通过甲醛-和/或egs(乙二醇双[琥珀酰亚胺基琥珀酸酯])-介导的交联进行交联。也可以使用适合于交联蛋白质-dna、蛋白质-rna和/或蛋白质-蛋白质的其它相似的双官能交联试剂(例如,具有两个或更多个适合于与酰胺和/或巯基基团反应的反应性化学基团的那些)。如果使用egs,两个nhs-酯之间的间隔体区域可以是12-原子的间隔体,虽然也可以使用更长或更短的间隔体(例如,6、7、8、9、10、11、12、13、14、15、16、17、18、19、20个原子的间隔体)。如果使用甲醛或egs(通常约1-2mm或1.5mm),可以首先添加egs,接着添加(约1%)甲醛。反应可以通过甘氨酸淬灭。可选地,可以使用约1%甲醛或约1%戊二醛。在其它实施方式中,核酸通过uv交联与染色质交联。例如,组织培养细胞可以在254nm下以约150mj/cm2uv交联(例如,通过使用uv交联剂,如交联剂)。例如,约1-2×108活组织培养细胞或分离的细胞可以首先收集并在振摇条件下用egs进行交联40min,然后用甲醛(约1%终浓度;sigma)在室温下交联10分钟。可以添加蛋白酶抑制剂和/或rnase抑制剂以防止非特异性的蛋白酶或rnase消化。细胞然后在合适的裂解缓冲液(例如,50mmhepes,1mmedta,0.15mnacl,1%sds,1%tritonx-100,0.1%脱氧胆酸钠,均来自ambion)中裂解。一旦交联步骤完成,各种方法可以用于产生包含交联的基因组dna和ncrna的染色质片段。例如,在某些实施方式中,染色质片段通过物理剪切产生,如超声处理、水力剪切、经皮下注射针的重复抽吸。超声处理对于将染色质纤维破裂成与rna、dna和蛋白质组分的系留复合物而同时“摆脱”欺骗的、随机的或弱的ncrna-染色质-dna相互作用可能是有利的。可选地,在某些实施方式中,染色质片段可以通过限制性内切酶消化或者在受控的条件下的部分或有限的核酸内切酶和/或核酸外切酶消化来产生,以产生合适长度的rna和dna标签。为产生包含交联的基因组dna片段和交联的ncrna的染色质片段,染色质可以通过超声处理溶解(例如,使用以20%占空功率输出运行的branson450超声波细胞粉碎机,30秒,5-8次;或者使用以35%功率运行的探头超声波破碎仪1.5min,20sec开/30sec关的循环)。其它商业可得的仪器可用于超声处理。例如,来自covaris,inc.的s220focused-超声发生器利用adaptivefocusedacousticstm(afa)技术用于dna、rna和染色质剪切。按照制造商,其软件整合了用于标准方法如dna剪切到特定片段长度的各种预设的方案。可选地,(lifetechnologiescorp.),一种台式超声设备,也可以用于超声剪切。该设备由位于水浴之下的高功率超声发生元件组成,并以20khz频率(类似于探头超声破碎仪)运行以提供适合于标准化方案如chip、medip等的自动化超声处理步骤。一旦剪切,染色质稀释(例如,10倍)以降低sds浓度(例如,到约0.1-0.5%)。提取物然后通过离心(例如,在4℃下以14,000rpm离心10分钟)澄清。这一提取物可以储存在-80℃直到使用。如果希望免疫沉淀,约2μg的单克隆抗体(对于染色质组分特异性的)可以结合于蛋白质g琼脂糖(pharmacia)。抗体包被的珠然后在4℃下与染色质提取物孵育16小时。珠然后洗涤(例如,用来自sigmachemicalcompany的以下试剂:洗涤缓冲液1(50mmhepes,1mmedta,0.15mnacl,0.1%sds,1%tritonx-100,0.1%脱氧胆酸钠);2倍洗涤缓冲液2(50mmhepes,1mmedta,0.5mnacl,0.1%sds,1%tritonx-100,0.1%脱氧胆酸钠);1倍洗涤缓冲液3(20mmtris.hclph8.0,1mmedta,0.25mlicl,0.5%np40,0.5%脱氧胆酸钠);1倍洗涤缓冲液4(20mmtris.hclph8.0,1mmedta)。蛋白质-dna复合物然后在65℃下用洗脱缓冲液(例如,50mmtris.hclph8.0,1mmedta,1%sds)从珠洗脱20min。洗脱液然后在pbs(ambion)中透析以除去sds(例如,4℃下3小时)。任选地,染色质片段也可以生物素化(例如,通过使用ezlinkiodoacetyl-peg2-biotin(ipb)(thermoscientific,cat.21334)),并作为链霉亲和素珠-结合的染色质片段分离。例如,具有链霉亲和素的(myonetm链霉亲和素c1/t1)可以用于富集生物素化的染色质片段。另外,具有二氧化硅样涂层的珠可以用于富集染色质片段上的交联的核酸。在剪切或re消化后,染色质片段可以具有损伤的末端或另外地不适合与dna接头连接的末端。因此,末端修复可以按照制造商的建议使用例如来自epicentre的end-it试剂盒或t4聚合酶(promega,r0191)进行。第一链cdna合成可以使用逆转录酶和rna接头(或在以下第二特定实施方式中的修饰的rna接头)进行,如superscriptiiifirststrandsynthesissystem(lifetechnologies,cat.18080051)。在其平端具有5’磷酸化的修复的染色质dna然后可以用于与dna接头连接。这可以使用rna接头在用于逆转录的相同容器中完成,条件是提供用于dna连接的适当缓冲液和其它反应条件。dna连接酶如t4dna连接酶可以用于这一反应。如果需要,去磷酸化的dna接头然后可以磷酸化(例如,通过t4多核苷酸激酶)。在某些实施方式中,使用rna接头进行第一链cdna合成(在dna接头连接之前或之后或者与之同时)。在某些实施方式中,交联的ncrna的cdna包含从rna接头的随机序列引物和ncrna模板逆转录的第一链cdna。由于rna接头的存在,这种第一链cdna和ncrna模板杂合分子可以连接于dna接头,该dna接头早已与染色体dna片段的自由端连接。一旦rna接头和dna接头已与其相应的靶核酸末端连接,可以进行邻位连接以连接相同染色质片段上的dna接头和rna接头。邻位连接通常在稀释的环境下进行以使得相同染色质片段上的rna和dna接头的连接与不同染色质片段上的rna和dna接头相比由于其彼此接近而可能性高得多。在某些实施方式中,邻位连接相对于接头连接步骤用约2、3、4、5、6、7、8、9、10、12、13、14、15、16、70、18、19、20-倍或更高倍稀释进行。在某些实施方式中,邻位连接对于源自约1×108个人细胞的各等同量的捕获染色质片段在约5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20ml或更大的总连接体积中进行。连接体积可以基于细胞类型(例如,来源物种或基因组大小)相应地调整。邻位连接条件可以按照需要改变或调整以最大化dna和rna接头的连接。任何连接条件可以改变或调整,包括但不限于增加或减少连接反应的时间和/或试剂的浓度。换句话说,连接反应进行调整或改变以最大化与相同染色质片段交联的单独核酸分子的分子间连接。特别地,连接可以在非常稀的核酸分子的条件下进行以最大化不同核酸分子的末端的连接和减少循环多聚体的形成。在某些实施方式中,该方法包括评估与不同染色质片段交联的基因组dna和ncrna之间的不希望的或假阳性连接事件的程度或频率。在理想的邻位连接条件下,仅与相同染色质片段交联的基因组dna和ncrna会连接。例如,一组dna和rna接头(例如,接头组a)可以用于分别连接于一个反应容器中的基因组dna和rna末端。同时,第二组dna和rna接头(例如,接头组b)可以用于分别连接于第二反应容器中的基因组dna和rna末端。两个反应容器的内容物然后汇合用于邻位连接。如果接头组a中的rna接头可以连接于两个接头组中的dna接头(且接头组a中的dna接头可以连接于两个接头组中的rna接头),则如果在组a和b的接头之间没有或有非常少见的连接(例如,组a中的rna接头连接于组b中的dna接头),邻位连接条件是最佳的。相反,如果组a和b的接头之间存在显著的连接,邻位连接条件不是最佳的。在某些实施方式中,接头组a和b中的rna和dna接头的比率可以进一步调节(例如,不必然是1:1)。例如,接头组a中rna和dna接头的摩尔比与接头组b中的摩尔比相比可以是2:1、3:1、4:1、5:1、6:1、7:1、8:1、9:1、10:1,反之亦然。在某些实施方式中,本发明的第一、第二、第三和/或第四多核苷酸是去磷酸化的且dna接头或rna接头不自身连接。第二链cdna合成可以使用例如superscriptdouble-strandedcdnasynthesiskit(lifetechnologies,cat.1197-020)在rna接头-dna接头连接之前或之后完成。在某些实施方式中,第二链cdna合成在邻位连接之后但在步骤(3)之前进行。在某些实施方式中,dna聚合酶如t4dna聚合酶可以在第二链cdna合成后添加。接下来,染色质片段的交联的核酸和蛋白质组分可以用蛋白酶k反交联。在典型的反应条件中,例如,样品可以作为20μl等分试样通过在15μl的20mg/ml蛋白酶k(ambion)和任选地0.3%sds(ambion)存在下在65℃下孵育过夜来反交联。第二天,约1μl的10mg/mlrnasea(qiagen)可以添加以降解rna(例如,37℃下45min),接着进行dna的酚提取和乙醇沉淀。任选地,至少一种连接的和反交联的核酸分子的纯化或富集可以使用包含至少两种组分的结合系统来进行,其中至少一种第一组分与接头偶联(例如,并入例如rna或dna接头中的生物素化的核苷酸),且至少第二组分结合第一组分。该组分包括但不限于链霉亲和素-生物素、抗生物素蛋白-生物素、蛋白质-抗体和/或磁体/磁性材料。特别地,生物素化接头连接的核酸材料可以使用链霉亲和素珠纯化,如链霉亲和素偶联的磁性dynabeadstm(lifetechnologies,cat.11206d-10ml)。仅含有生物素化接头的核酸材料被固定在链霉亲和素珠上。如果另一组分与所使用的接头结合,可以使用适合于该组分的纯化核酸分子的其它系统。可选地,可以改为使用链霉亲和素柱来捕获生物素化的珠。在再另一替代方式中,珠可以是彩色的或荧光涂覆的以使得它们可以通过facs等在基于流动的检测仪器(例如,或型分析仪)上分选或收集。最终释放的dna可以用于通过例如re酶消化产生具有配对的dna和rna标签的pet多核苷酸。任选地,释放的pet多核苷酸可以在测序分析之前进一步通过pcr扩增。pcr连接体可以在进行pcr扩增之前连接于pet多核苷酸的两个末端(如通过t4dna连接酶)。仅平端的非环化核酸可以连接于连接体。自身连接的核酸分子和环状多聚体不能连接于连接体。pcr连接体也可以包含用于pcr产物纯化的修饰的核苷酸。类似地,链霉亲和素-生物素、抗生物素蛋白-生物素、蛋白质-抗体和/或磁体/磁性材料可以用于这一目的。pet多核苷酸(具有或不具有扩增)可以直接测序,如按照用于各种下一代测序的方案,如使用454多重测序分析仪(454lifesciences)的454测序分析。该技术在margulies等(2005)和美国申请no.20030068629中教导(两者通过引入并入本文)。任何其它高通量或下一代测序分析(ngs)方法可以用于确定pet多核苷酸的序列。所获得的rna/dna标签序列到其相应的基因组位置的映射可使用多种商业可得的工具、软件或服务中的任一种来进行。一旦pet多核苷酸的rna和dna标签被测序和映射到基准基因组,各连接的rna标签和dna标签代表推定的ncrna-染色质相互作用。所有这类观察到的相互作用的集合构成基准基因组内基准基因组的非编码rna(ncrna)的功能性相互作用位点。在某些实施方式中,该方法进一步包括鉴别具有基因组dna的重叠序列标签和ncrna的重叠序列标签的两个或更多个pet多核苷酸的簇。pet簇被认为是高可信度的数据,反映了更可靠的ncrna-染色质相互作用事件的重复检测。相反,没有rna标签和dna标签两者上与其它pet序列的重叠的单态pet可以代表弱连接信号,且可以与随机背景噪声区别开。在某些实施方式中,该方法进一步包括排除包含rrna的序列标签的pet多核苷酸。虽然一些rrna-染色质-gdna(基因组dna)相互作用可以具有真正的生物学意义,大量(在一些数据集中约1/4)的rrna-染色质-dna相互作用的存在可能掩盖其它较不丰富的相互作用。因此,在进一步的数据分析之前的这种数字减除(digitalsubtraction)对于分析较低频率的ncrna-染色质相互作用可能是希望的。在某些实施方式中,该方法进一步包括在邻位连接步骤之前分离或富集染色质片段的亚集。例如,染色质片段的亚集可以通过使用对于染色质片段亚集的蛋白质组分特异性的抗体的免疫沉淀或通过使用对于染色质片段亚集的核酸组分特异性的(标记的)多核苷酸的杂交来分离或富集。这可能可用于鉴别已知染色质组分和ncrna之间的特定相互作用。在某些实施方式中,蛋白质组分是组蛋白、转录因子(如一般转录因子rnapii、rnapi、rnapiii)、重构染色质的多梳(pcg)家族蛋白(如ezh2及来自昆虫、哺乳动物和植物的其它因子);重组参与因子(如prdm9);染色质隔离子或染色质waver(如ctcf);甲基-cpg-结合蛋白质(如mecp2)或rna结合蛋白。在该方法的变型中,特定的标记ncrna(如生物素化(biotyinylation))可以在交联之前添加到细胞。这类标记的ncrna可以通过使用抗生物素蛋白或链霉亲和素包被的磁珠来分离或富集。在该方法的再另一变型中,一种或多种特定的目标ncrna的互补序列可以用于分离或富集与染色质片段交联的这类特定ncrna(使用阵列或柱)。一旦分离或富集,这样的染色质片段可以经受该方法的剩余步骤以鉴别与特定ncrna相互作用的基因组dna的区域。在某些实施方式中,该方法进一步包括通过例如,dna/rnafish和免疫荧光分析验证一种或多种观察到的ncrna-染色质相互作用。例如,如果特定ncrna与特定基因组位点连接,dna/rnafish和免疫荧光分析可以使用ncrna进行以确认观察(参见,例如图4b)。b)修饰的rna接头在另一/第二特定实施方式中,本发明的方法可以使用一种修饰的rna接头(和无dna接头)进行以连接相同染色质片段中的交联的rna和染色体dna。因此,本发明的另一方面提供修饰的rna接头,其包含:(i)第一多核苷酸和(ii)第二多核苷酸,其中第一和第二多核苷酸形成侧接基因组dna连接相容末端和第一多核苷酸3’-末端的3’-悬端的双链区域,其中3’-悬端包含随机序列引物。按照本发明的这一方面,第一多核苷酸3’-末端的3’-悬端具有与小节a)中描述的特定实施方式(rna和dna接头对)中的rna接头相似的功能,而基因组dna连接相容末端可用于连接与相同染色质片段交联的平端基因组dna。在某些实施方式中,连接相容末端可以是平端的用于直接连接于交联的基因组dna片段的平端。在另一实施方式中,连接相容末端可以包含限制性内切酶位点,其可以被re切割以产生与交联的基因组dna片段的平端连接所需的必要平端。但是,在通过限制性内切酶切割之前,连接相容末端可以是平端的(例如,去磷酸化的平端以防止自身连接),或具有防止自身连接的不相容悬端。在某些实施方式中,修饰的rna接头不通过其3’-悬端或其连接相容末端自身连接。第一和第二多核苷酸可以提供在单独容器中,如合成的多核苷酸,其为冷冻干燥的、冻干的形式或者在水或合适的缓冲溶液中。可选地,第一和第二多核苷酸可以组合在相同容器中(冻干的或在溶液中),例如,以1:1的摩尔比,使得它们可以作为预退火的修饰的rna接头使用。第二多核苷酸基本上是同质的或纯的(例如,相同容器中的单个多核苷酸分子是相同的),而3’-悬端区域中第一多核苷酸的3’-末端包含随机序列引物。在相关的实施方式中,第一多核苷酸可以在随机序列引物区域处同质地包含相同的匹配序列,以特别地启动从具有限定的3’-末端序列的特定ncrna的第一链cdna合成。在某些实施方式中,双链区域可以包含第一限制性内切酶如ii型限制性内切酶(re)的第一识别位点。re识别位点可以策略地布置以使得当re切割时,它在re位点之外,随机序列引物的3’侧进行切割。这允许产生与rna接头连接的rna标签。例如,mmei识别位点可以设置在双链区域的末端处,接近于包含随机序列引物的3’悬端。mmei位点设计为其定向使得在mmei切割时,在源自连接的ncrna的cdna中产生具有2bp悬端的包含18-bp片段的rna标签。但是,re位点的设置不需要在第一双链区域的末端处。更内部的设置产生相应更短的rna标签序列。在某些实施方式中,第一识别位点(用于第一(ii型)限制性内切酶)的最后一个核苷酸是随机序列引物5’侧的最后一个碱基配对的核苷酸。在某些实施方式中,双链区域可以在连接相容末端处或靠近连接相容末端包含用于第二限制性内切酶如ii型限制性内切酶(re)的第二识别位点。re可以切割第二re识别位点的3’侧和第一多核苷酸的5’侧(例如,在连接的基因组dna中)。re识别位点的定向以使得其基于连接的基因组dna的末端序列产生dna标签的方式设置。在某些实施方式中,re位点的设置不需要在双链区域的末端处。更内部的设置产生相应更短的dna标签序列。在某些实施方式中,第二识别位点(用于第二(ii型)限制性内切酶)的最后一个核苷酸是连接相容末端/平端处的碱基配对的核苷酸。在某些实施方式中,修饰的rna接头不具有用于产生rna标签或dna标签的限制性内切酶识别位点。在某些实施方式中,修饰的rna接头可以包含区分修饰的rna接头与其它修饰的rna接头的独特序列(例如,“条码”)。在某些实施方式中,第一和/或第二多核苷酸是去磷酸化的。本发明的另一方面提供包含中心区域的末端配对标签(pet)多核苷酸,中心区域包含(修饰的rna接头的)双链区域,其:(1)在接近随机序列引物的位点处侧接非编码rna(ncrna)的序列标签和(2)在接近连接相容末端的位点处侧接基因组dna的序列标签。在相关的方面中,本发明提供末端配对标签(pet)多核苷酸文库,其包含两个或更多个所述pet多核苷酸的成员,其中pet文库的各成员包含相同的中心区域及非编码rna(ncrna)的不同rna序列标签、基因组dna的不同dna序列标签或两者。在再另一相关的方面中,本发明提供包含所述pet多核苷酸的载体或重组载体。本发明的另一方面提供鉴别基因组内基因组的非编码rna(ncrna)的功能性相互作用位点的方法,该方法包括:(1)提供包含交联的基因组dna片段和交联的ncrna的染色质片段;(2)使用本发明的修饰的rna接头在用于邻位连接的条件下连接交联的基因组dna片段的末端与交联的ncrna的cdna的末端,其中交联的基因组dna片段的末端连接于修饰的rna接头的连接相容末端,且交联的ncrna的cdna的末端包含修饰的rna接头;(3)分离本发明的pet多核苷酸用于测序分析;和(4)映射各pet多核苷酸内的基因组dna的序列标签和ncrna的序列标签到基准基因组,由此鉴别基准基因组内基准基因组的非编码rna(ncrna)的功能性相互作用位点。在某些实施方式中,交联的ncrna的cdna包含从修饰的rna接头的随机序列引物和ncrna模板逆转录的第一链cdna。由于修饰的rna接头的存在,这种第一链cdna和ncrna模板杂合分子可以连接于染色体dna片段的自由端。在某些实施方式中,修饰的rna接头上双链区域的长度为约6、7、8、9、10、15、20、25、30、35、40、50、60或更多个碱基对。如小节a)中描述的第一特定实施方式中所述的其它实施方式(rna和dna接头对)一般是可应用的,且在此引入(但不重述)。c)直接rna-dna连接在另一/第三特定实施方式中,本发明的方法可以使用将ncrna的3’-oh基团直接连接于5’腺苷酸化单链dna(5’app-ssdna)(如随后与互补多核苷酸杂交的ssdna接头)或具有用于直接连接于ncrna的3’-oh基团的可用作酶的底物的5’腺苷酸化悬端的dsdna的某些酶(如截短的rna连接酶2或rnl2)来进行。因此,本发明还提供连接相同染色质片段中交联的ncrna的3’-末端和交联的基因组dna片段的自由端的可选方式。根据本发明的这一方面,提供其5’预腺苷酸化的单链dna寡核苷酸(5’appssdna)。rna-dna连接酶(如热稳定的5’appdna/rna连接酶,neb目录m0319s或m0319l)然后可以用于直接连接ncrna的3’-oh与5’appssdna。根据制造商,热稳定的5’appdna/rna连接酶是来自methanobacteriumthermoautotrophicum的rna连接酶的催化性赖氨酸的点突变体(zhelkovsky和mcreynolds,bmcmol.biol.,13:24,2012)。这种酶是非atp依赖性的,但需要5’预腺苷酸的接头用于连接于rna或单链dna(ssdna)的3’-oh末端。酶也具有将具有2’-o-甲基化3’末端的rna连接于5’-腺苷酸化接头的活性(zhelkovsky和mcreynolds,同上)。突变体连接酶不能使rna或ssdna的5’-磷酸腺苷酸化,这减少不希望的连接产物(串联体和圆环)的形成。连接酶在65℃下发挥功能的能力可以进一步减少rna连接反应中rna二级结构的限制。用于本发明的这一实施方式的另一种合适的连接酶是rna连接酶2,如来自biooscientific(austin,tx)的airtmrna连接酶2(rnl2),其特异性地将连接体的腺苷酸化5’末端连接于rna的3’末端。类似地,酶不需要atp来进行连接,但确实需要腺苷酸化的底物,这急剧地减少随机rna分子之间连接的量。连接酶是t4rna连接酶2的截短形式。与全长rna连接酶2不同,airtm连接酶在不存在腺苷酸化底物的情况下不连接rna或dna的磷酸化的5′末端。可选地,t4rna连接酶1(nebcat.no.m0204s或m0204l)可以用于连接ncrna3’-oh与5’磷酰基封端的ssdna。一旦ncrna的3’-末端连接于ssdna,互补ssdna可以与连接的ssdna退火以启动第二链cdna合成和/或形成适合与相同染色质片段中交联的基因组dna片段的自由端连接的平端。在可选的实施方式中,在一端具有平端(或连接相容末端)和在另一端具有5’腺苷酸化悬端(其可以用作上述各种rna连接酶的单链底物)的dsdna接头可以首先连接于交联的基因组dna片段的自由端,之后突出的腺苷酸化5’末端直接连接于ncrna的3’-oh。同样地,以上针对连接的rna接头-dna接头或修饰的rna接头描述的所有实施方式或变型一般可应用于在5’appssdna和其互补序列之间形成的双链区域。例如,在某些实施方式中,在5’appssdna和其互补序列之间形成的双链区域可以包含一个或多个re识别位点以利于产生rna和dna标签序列。两个mmei位点可以位于双链区域的两个末端且指导双链区域之外的切割以产生双链区域侧翼的18-20bprna和dna标签。可选地,一个re位点可以产生rna标签(或dna标签),且dna标签(或rna标签)可以通过物理剪切或有限的非特异性酶消化产生(参见上文)。因此,本发明的另一方面提供直接rna接头,其包含:(i)第一多核苷酸和(ii)第二多核苷酸,其中第一和第二多核苷酸形成侧接基因组dna连接相容末端和第一多核苷酸5’-末端的5’-悬端的双链区域。5’-悬端任选地5’腺苷酸化,或可以通过5’dna腺苷酸化试剂盒(cat.no.e2610s或e2610l)中合适的酶如mthrna连接酶来腺苷酸化。如果rna连接对于5’-悬端进行,如与作为ssdna的第一多核苷酸(在其与第二多核苷酸退火之前)相反的,5’-悬端具有足够长度(例如,4、5、6、7、8、9、10、11、12、13、14、15个或更多个碱基)以用作用于直接rna连接的酶的底物。在某些实施方式中,连接相容末端可以是平端的以用于与交联基因组dna片段的平端直接连接。在另一实施方式中,连接相容末端可以包含限制性内切酶位点,其可以通过re切割以产生用于连接于交联基因组dna片段的平端所需的必要平端。但是,在通过限制性内切酶切割之前,连接相容末端可以是平端的(例如,去磷酸化的平端以防止自身连接),或具有防止自身连接的不相容悬端。在某些实施方式中,直接rna接头不自身连接。例如,第一多核苷酸的3’末端可以通过双脱氧核苷酸或其它修饰的核苷酸封闭以防止第一多核苷酸的自身连接(自身环化)。在rna-dna连接完成时,封闭的第一多核苷酸的3’末端变成连接相容末端的部分,且可以通过re消化切除掉以产生用于基因组dna连接的平端。在某些实施方式中,双链区域可以包含用于第一限制性内切酶如ii型限制性内切酶(re)的第一识别位点。re识别位点可以策略地布置以使得当re切割时,它在re位点之外,第一多核苷酸的5’腺苷酸化末端的5’侧进行切割。这允许产生与直接rna接头连接的rna标签。例如,mmei识别位点可以设置在双链区域的末端处,接近第一多核苷酸5’-悬端的5’末端。mmei位点设计为其定向使得在mmei切割时,在源自连接的ncrna的cdna中产生具有2bp悬端的包含18-bp片段的rna标签。但是,re位点的设置不需要在第一多核苷酸的末端处。更内部的设置相应地产生较短的rna标签序列。如果第一多核苷酸用作ssdna底物(与其5’-悬端用作底物相反),可以产生较长的rna标签序列,因为re位点可以设置在第一多核苷酸的5’-端处。因此,在某些实施方式中,第一识别位点(用于第一(ii型)限制性内切酶)的最后一个核苷酸是第一多核苷酸的5’-端。在某些实施方式中,双链区域可以在连接相容末端处或靠近连接相容末端包含用于第二限制性内切酶如ii型限制性内切酶(re)的第二识别位点。re可以切割第二re识别位点3’侧和第一多核苷酸的3’侧(例如,在连接的基因组dna中)。re识别位点的定向以使得其基于连接的基因组dna的末端序列产生dna标签的方式排列。在某些实施方式中,re位点的设置不需要在双链区域的末端处。更内部的设置相应地产生较短的dna标签序列。在某些实施方式中,第二识别位点(用于第二(ii型)限制性内切酶)的最后一个核苷酸是连接相容末端/平端处的碱基配对的核苷酸。在某些实施方式中,直接rna接头不具有用于产生rna标签或dna标签的限制性内切酶识别位点。在某些实施方式中,直接rna接头可以包含区分直接rna接头与其它直接rna接头的独特序列(例如,“条码”)。在某些实施方式中,第二多核苷酸是去磷酸化的。根据本发明的这一方面产生的pet多核苷酸包含对应于在5’appssdna和其互补序列(即,第二多核苷酸)之间形成的双链区域的中心区域。对于该区域不具有特别的序列要求,且区域长度是灵活的(例如,短到几个bp,足够长以支持rna-dna连接酶的底物要求和逆转录酶的底物要求),虽然较长的序列可以用于并入任何希望的re识别位点、条码序列或修饰的核苷酸(例如,用于亲和纯化的生物素化的核苷酸)。因此,本发明的另一方面提供包含中心区域的末端配对标签(pet)多核苷酸,该中心区域包含(直接rna接头的)双链区域,其:(1)在接近第一多核苷酸(5’腺苷酸化的或适合于5’腺苷酸化的)的5’末端的位点处侧接非编码rna(ncrna)的序列标签;和(2)在接近连接相容末端的位点处侧接基因组dna的序列标签。在相关的方面中,本发明提供末端配对标签(pet)多核苷酸文库,包含两个或更多个所述pet多核苷酸的成员,其中pet文库的各成员包含相同的中心区域及非编码rna(ncrna)的不同rna序列标签、基因组dna的不同dna序列标签或两者。在再另一相关的方面中,本发明提供包含所述pet多核苷酸的载体或重组载体。本发明的再另一方面提供鉴别基因组内基因组的非编码rna(ncrna)的功能性相互作用位点的方法,该方法包括:(1)提供包含交联的基因组dna片段和交联的ncrna的染色质片段;(2)连接ncrna的3’-oh与5’预腺苷酸化ssdna;(3)提供ssdna的互补序列以形成ssdna和互补序列之间的双链区域;(4)如果需要,在双链区域的末端处产生平端;(5)在用于邻位连接的条件下连接平端与交联的基因组dna片段的末端;(6)分离用于测序分析的pet多核苷酸,其中pet多核苷酸包含侧接交联的基因组dna片段的dna标签和ncrna的rna标签的双链区域;和(7)映射dna标签和rna标签到基准基因组,由此鉴别基准基因组内基准基因组的非编码rna(ncrna)的功能性相互作用位点。本发明的可选方面提供鉴别基因组内基因组的非编码rna(ncrna)的功能性相互作用位点的方法,该方法包括:(1)提供包含交联的基因组dna片段和交联的ncrna的染色质片段;(2)连接ncrna的3’-oh与具有双链区域的dsdna的5’预腺苷酸化悬端;(4)如果需要,在5’预腺苷酸化悬端远端的双链区域末端处产生平端;(5)在用于邻位连接的条件下连接平端与交联的基因组dna片段的末端;(6)分离用于测序分析的pet多核苷酸,其中pet多核苷酸包含侧接交联的基因组dna片段的dna标签和ncrna的rna标签的双链区域;和(7)映射dna标签和rna标签到基准基因组,由此鉴别基准基因组内基准基因组的非编码rna(ncrna)的功能性相互作用位点。在某些实施方式中,ssdna的互补序列(即,第二多核苷酸)具有与ssdna相同的长度。在某些实施方式中,互补序列比ssdna更长或更短,且形成具有突出的3’或5’末端的双链区域。在后一情况中,悬端可以通过酶填补以产生适合连接的平端,或通过产生平端的限制性内切酶从末端切除。re位点可以工程化到ssdna的序列中。在某些实施方式中,直接rna接头的第一多核苷酸的长度为约6、7、8、9、10、15、20、25、30、35、40、50、60个或更多个碱基。如分别在小节a)(rna和dna接头对)和小节b)(修饰的rna接头)中描述的第一和第二特定实施方式中描述的其它实施方式一般是适用的,且在此并入(但不重述)。对于如此描述的本发明的一般方面,以下章节提供与本发明的特定实施方式相关的额外细节及特定量和参数。对于本领域技术人员显而易见的是,本发明可以在不具有这样的细节或具有少量改变的情况下实施而不脱离本发明的一般范围。2.定义“非编码rna(ncrna)”包括不翻译成蛋白质的rna分子。较少见地,它也可以称为非蛋白质编码rna(npcrna)、非信使rna(nmrna)和功能性rna(frna)。它通常是具有编码蛋白质以外的功能的功能性rna,但一些可以是非功能性的或没有已知的功能。有时,术语小rna(srna)经常用于短的细菌ncrna。非编码rna由其转录的dna序列通常称为rna基因。非编码rna基因包括高度丰富的和功能上重要的rna,如转移rna(trna)和核糖体rna(rrna),以及如snorna(包括scrna;用于rna的核苷酸修饰)、snrna(用于剪接和其它功能)、grna(向导rna;用于mrna核苷酸修饰)、rnasep(用于trna成熟)、rnasemrp(用于rrna成熟和/或dna复制)、yrna(用于rna加工和/或dna复制)、端粒酶rna(用于端粒合成)、剪接前导序列rna、smyrna(用于mrna反式剪接)、反义rna、顺式天然反义转录物、microrna(用于基因调节)、sirna(包括反式作用sirna;用于基因调节)、exrna和pirna(包括重复相关sirna;用于转座子防御及可能的其它功能)的rna,7skrna(用于负向调节cdk9/细胞周期蛋白t复合体)及包括如xist和hotair的实例的长ncrna。人基因组内编码的ncrna的数目是未知的,但最近的转录组和生物信息学研究表明数千个ncrna的存在。由于许多新鉴别的ncrna未验证其功能,因此有可能许多是非功能性的。在某些实施方式中,本发明的ncrna不包括一种或多种上面提及的种类。例如,在某些实施方式中,本发明的ncrna不包括rrna。在某些实施方式中,本发明的ncrna不包括trna。在某些实施方式中,本发明的ncrna不包括trna。“限制性内切酶(re)”和“限制性核酸内切酶”在本文中可互换使用以包括切割双链dna的酶。酶通常在称为“限制性位点”或“re识别位点”的特定识别核苷酸序列处、之内或附近(例如,约几个碱基至约几千碱基)形成两个切口,一个切口通过双重螺旋的磷酸酯骨架中每一个而不损伤碱基。限制性内切酶一般分类为三个类型,其不同在于其结构及其是否在其识别位点处切割其dna底物,或者是否识别和切割位点彼此分离。迄今超过3000种限制性内切酶已详细研究,且它们中超过600个是商业可得的,其中许多常规地用于分子生物学中的dna修饰和操作。i型限制性内切酶切割与其识别位点不同且距其识别位点随机距离(至少1000bp)的位点。i型限制性内切酶识别位点是不对称的,且由被非特异性的约6-8个核苷酸的间隔体分隔的两个特定部分(一个包含3-4个核苷酸和另一个包含4-5个核苷酸)构成。这些酶是多功能的且能够具有限制性内切和修饰活性(取决于靶dna的甲基化状态)。水解腺苷三磷酸的辅因子s-腺苷甲硫氨酸(adomet)和镁(mg2+)因子是其完全活性所需要的。典型的ii型限制性内切酶是同型二聚体,具有通常未分开的、回文结构的4-8核苷酸长度的识别位点。它们在相同位点识别和切割dna,且它们不使用atp或adomet以获得其活性–它们通常仅需要mg2+作为辅因子。最近,产生了新的亚族命名(使用字母后缀定义)以基于与ii型酶的典型特征的偏离将这一大的家族分成亚类。例如,iib型限制性内切酶(例如,bcgi和bpli)是需要adomet和mg2+辅因子两者的多聚体,且它们在其识别位点的两侧切割dna以切开识别位点。iie型限制性核酸内切酶(例如,naei)在与两个拷贝的其识别序列相互作用后切割dna。一个识别位点用作切割的靶标,而另一个用作加速或提高酶切割效率的变构效应子。与iie型酶相似,iif型限制性核酸内切酶(例如,ngomiv)与两个拷贝的其识别序列相互作用,但同时切割两个序列。iig型限制性核酸内切酶(eco57i)确实具有单一亚基,如同典型的ii型限制性内切酶,但需要辅因子adomet来使其成为活性的。iim型限制性核酸内切酶如dpni能够识别和切割甲基化的dna。iis型限制性核酸内切酶(例如,foki)在距其非回文的不对称识别位点的限定距离处切割dna。也就是说,iis型酶在其识别序列之外在一侧切割。mmei以及大多数iis型限制性内切酶产生可变的末端长度。dunn等(2002)证明mmei可以1:1的大致比例切割18/20或19/21个碱基。因此,当18/20用于描述mmei限制性切割位点时,也应考虑19/21。iit型限制性内切酶(例如,bpu10i和bsli)由两个不同亚基构成。一些识别回文序列,而其它的具有不对称识别位点。iii型限制性内切酶(例如,ecop15)识别两个相反定向的单独的非回文序列。它们切割识别位点后约20-30个碱基对的dna。这些酶包含超过一个亚基并需要adomet和atp辅因子以分别实现其在dna甲基化和限制性内切中的作用。iii型酶识别短的5-6bp长度的不对称dna序列并切割下游的25-27bp而留下短的单链5’突出物。它们需要存在两个相反定向的非甲基化识别位点来发生限制性内切。限制性内切酶切割产物可以是平端的或具有带5’或3’悬端的粘性末端,该粘性末端片段可以不仅连接该粘性末端片段原先从其切割的片段,而且也连接具有相容的粘端或粘性末端的任何其它片段。如本文所用的“核苷酸”包括核苷的磷酸酯-核酸(dna或rna)的基础结构单元。两个或更多个核苷酸(例如,2-30、5-25、10-15个核苷酸)的短链有时称为“寡核苷酸”,而较长的链称为多核苷酸,虽然这两个术语之间没有明确的长度限制。术语核苷酸可以与术语“核酸”互换地使用。多核苷酸可以是单链的,或者可以是各条链具有5’末端和3’末端的双链。核酸延伸片段的末端区域可以分别称为5’端和3’端。多核苷酸中的核苷酸可以是天然核苷酸(对于dna是脱氧核糖核苷酸a、t、c或g,且对于rna是核糖核苷酸a、u、c、g),或可以包括修饰的核苷酸,其可以通过例如化学合成并入多核苷酸中。这样的修饰核苷酸可以赋予天然核苷酸中不存在或缺乏的另外的所需性质,且包含修饰的核苷酸的多核苷酸可以用于本发明的组合物和方法中。术语“引物”或“启动序列”指的是能够在诱导与核酸链互补的引物延伸产物的合成的条件(即在适宜缓冲液中存在四种不同的核苷三磷酸和用于延伸的试剂(例如,dna聚合酶或逆转录酶)和在合适的温度下)下用作dna合成的启动点的寡核苷酸。引物可以是单链dna。引物的适宜长度取决于引物的预期用途,但通常范围为10-50个核苷酸,如15-35个核苷酸。短的引物分子一般需要较低的温度以形成足够稳定的与模板的杂交复合物。引物不需要反映模板核酸的精确序列,但必须充分互补以与模板杂交。用于扩增给定靶序列的合适引物的设计是本领域中公知的且描述于例如本文中引用的文献中。“探针”一般指的是用于检测靶序列(如ccat1ncrna序列或其cdna)的cdna或mrna的至少一部分的存在的核酸分子或与其互补的序列。检测可以通过鉴别探针和分析的靶序列之间的杂交复合物来完成。探针可以连接于固体载体或可检测标记。探针一般是单链的。探针通常包含10-200个核苷酸。探针的特定性质取决于特定用途且在本领域技术人员的测定技能范围内。一般地,探针在高严格性杂交条件下与至少一部分靶cdna或rna杂交。“连接体”指的是待连接的寡核苷酸分子,或其连接于核酸分子的末端。连接体可以用于扩增(具有pcr引物序列的pcr连接体)、测序分析(具有测序引物序列)和/或插入核酸片段到载体(具有合适的克隆序列,如re识别位点)中。“串联体”通常由末端-末端连接的至少两个核苷酸单体序列构成,任选地由接头或间隔体分隔。单体可以是序列上相同的或不相同的,但可以具有相似的结构元件(如本发明的rna和dna接头)。单体也可以处于相同或不同的定向(例如,串联体内的单体以头对头、头对尾或其混合的方式相互连接。本发明的串联体包含至少两个按照本发明的方法制备的寡核苷酸(例如,pet多核苷酸)。“文库”包括类似核酸、寡核苷酸或多核苷酸的集合,其中文库的各成员共有一种或多种限定的特征。例如,本发明的pet多核苷酸的文库包含两个或更多个(例如,数万、数十万、数百万、数千万,等等)本发明的pet多核苷酸,其中各pet多核苷酸共有相似或相同的结构但具有不同的dna和/或rna标签序列。“载体”或“重组载体”是指称噬菌体、质粒或能够从一个细胞到另一个细胞转移或扩增其内包含的遗传材料(例如,克隆的遗传信息或克隆的dna)的其它试剂的本领域公认的术语。这样的载体根据特定的性质和特征可以通过转染和/或转化(如脂质转染、磷酸钙沉淀、逆转录病毒传送、电穿孔和基因枪转化)及本领域可得的任何其它分子生物学技术被引入不同的宿主细胞中。合适的载体可以包括已通过插入或并入异源遗传序列操作的质粒、病毒载体或本领域中已知的其它媒介。这样的载体可以包含用于合适的宿主扩增的复制原点、可以促进克隆序列的有效转录的启动子序列、用于克隆序列的直接扩增的侧翼pcr引物。载体也可以包含允许转化细胞的表型选择的特定基因。适合用于本发明中的载体包括例如,pbluescript(stratagene,lajolla,calif.);pbc、pzero-1(invitrogen,carlsbad,ca)和pgem3z(promega,madison,wi),或其修饰的载体以及本领域技术人员已知的其它相似载体。参见,例如美国专利no.4,766,072中公开的pgem载体,其通过引用并入本文。“染色质”用于描述细胞核中核酸和蛋白质(主要是组蛋白)的复合物,其容易用碱性染料染色且在细胞分裂期间凝缩以形成染色体。染色质是核酸-蛋白质复合物的实例。如本文中使用的“标签”包括可以唯一地鉴别基准基因组内的序列起源的可鉴别的核酸序列的延伸片段。标签可以具有唯一地或毫无疑义地将标签映射到基准基因组中的一个或几个位置(如一个基因的重复拷贝或具有高序列同一性的相关基因)的足够长度(通常18-20bp,但根据序列组成及基准基因组大小和复杂度等可以更短)。本发明的dna标签来源于基因组dna序列。它可以通过例如本发明的dna接头和rna接头(或本发明的修饰的rna接头,或本发明的直接rna接头)连接于ncrna或ncrna的cdna。本发明的rna标签来源于ncrna或从ncrna逆转录的cdna。rna标签可以通过例如本发明的dna接头和rna接头(或本发明的修饰的rna接头,或本发明的直接rna接头)连接于基因组dna。本发明的rna或dna标签可以是任何尺寸,但对于其所来源的亲本序列的大小需要是有意义的和有利的。在某些实施方式中,dna和rna标签的大小按照基因组复杂性决定。对于细菌基因组,约8bp至约16bp的标签可能是足够的,而对于复杂基因组如人基因组,可以考虑16-20bp的标签。“接头”通常是对于特定目的设计的人工核酸序列,如将两个多核苷酸连接在一起。本发明的“rna接头”设计为连接于本发明的dna接头和连接于从rna(如交联的非编码rna)的自由3’-末端合成的cdna。本发明的“dna接头”设计为连接于本发明的rna接头和连接于dna(如与染色质片段交联的染色体dna)的自由端。本发明的“修饰的rna接头”设计为在一端(例如,平端或能够产生平端的连接相容末端)连接于基因组dna片段和在另一端连接于从rna(如交联的非编码rna)的自由3’-末端合成的cdna。本发明的“直接rna接头”设计为通过预腺苷酸化5’-末端直接连接于ncrna的3’-oh和在另一端(例如,平端或能够产生平端的连接相容末端)连接于基因组dna片段。“测序”指的是用于确定生物聚合物(在这种情况下,核酸)中成分的顺序的各种方法。可以用于本发明的合适的测序分析技术包括传统的链终止sanger方法以及可从多个商业来源获得的所谓的下一代(高通量)测序分析,如大规模平行签名测序(或mpss,lynxtherapeutics/solexa/illumina)、聚合酶克隆测序(lifetechnologies)、焦磷酸测序或“454测序”(454lifesciences/rochediagnostics)、通过连接的测序(solid测序,appliedbiosystems/lifetechnologies)、通过合成的测序(solexa/illumina)、dna纳米球测序、heliscope测序(helicosbiosciences)、离子半导体或iontorrent测序(iontorrentsystemsinc./lifetechnologies)和单分子实时(smrt)测序(pacificbio),等等。还开发或完善了多种其它的高通量测序方法,其也可用于测序本发明的pet多核苷酸,包括纳米孔dna测序、通过杂交的测序分析、利用质谱的测序、微流体sanger测序、透射电子显微镜dna测序、rnap测序和体外病毒高通量测序,等等。在某些实施方式中,测序方法能够从所述pet多核苷酸两侧测序标签,因此提供末端配对标签信息。在某些实施方式中,测序方法能够在可变长度的长dna片段(如所述pet多核苷酸的串联体)上执行阅读。“基准基因组”指的是目标生物体的基因组或者ncrna和基因组dna所来源的基因组。本发明的方法和组合物应用于其中完整的或基本上完整的序列可得的任何基准基因组,包括多种古细菌或真细菌、原生生物、真菌(例如,酿酒酵母(s.cerevisae)或裂殖酵母(s.pombe))、植物、动物基因组。例如,人、小鼠及多种其它哺乳动物和非哺乳动物物种的基因组序列现在容易地在公开领域中可得。参见,例如venter等,“thesequenceofthehuamngenome,”science,291(5507):1304-1351,2001。其它非限制性的基准基因组包括多种非人灵长类、哺乳动物、啮齿动物(大鼠、小鼠、仓鼠、兔等)、家畜(牛、猪、马、绵羊、山羊)、鸟类(鸡)、爬行动物、两栖动物(爪蟾)、鱼(斑马鱼(daniorerio)、河豚)、昆虫(果蝇、蚊子)、线虫类、寄生虫、真菌(例如,酵母如酿酒酵母或裂殖酵母)、各种植物、病毒(如整合到宿主基因组中的那些)等等的那些。锁核酸(lna)是其中lna核苷酸的核糖部分用连接2'氧和4'碳的额外的桥进行修饰的修饰rna核苷酸。桥“锁定”核糖在3'-内向构象中。lna核苷酸可以在任何需要时与寡核苷酸中的dna或rna残基混合。这样的寡聚体是化学合成的且商业可得。锁定的核糖构象增强碱基堆叠和骨架预组织化。这显著提高寡核苷酸的杂交性能(解链温度)。肽核酸(pna)是与dna或rna类似的人工合成的聚合物。pna寡聚体在与互补dna的结合中显示更高的特异性,其中pna/dna碱基的失配比dna/dna双链体中的类似失配导致更高的不稳定。这种结合强度和特异性也适用于pna/rna双链体。本发明的“末端配对标签(pet)多核苷酸”是在一个末端处或靠近一个末端具有源自ncrna的rna标签和地另一末端处或靠近另一末端具有源自基因组dna的dna标签的多核苷酸,其中ncrna和基因组dna优选与相同染色质片段交联。在这个意义上,pet多核苷酸的两个末端处的rna和dna标签配对并反映在交联时ncrna和基因组dna之间物理接近的事件。“邻位连接条件”指的是用于多核苷酸连接反应的条件,在该条件下紧密接近的可连接的多核苷酸末端,如与相同染色质片段交联的那些基因组dna和ncrna,优先地连接。同时,非紧密接近的可连接多核苷酸末端,如与不同染色质片段交联的那些基因组dna和ncrna,不连接或基本上不连接。这样的连接条件包括大体积连接,以使得相同染色质片段上的可连接末端由于其彼此物理接近而使其连接比不同染色质片段上可连接末端之间的连接可能性高得多。“映射(序列标签到基因组)”包括鉴别基因组中序列标签的基因组位置。“双功能交联剂/试剂”或“交联剂/试剂”包括具有两个或更多个反应性基团的修饰试剂,各基团能够与一个部分(dna、rna或蛋白质)反应,因此在两个部分代表单独的分子时将两个部分交联到一起。这样的双功能交联剂是本领域中公知的(参见,例如isalm和dent,bioconjugation,chapter5,pp.218-363,grovesdictionariesinc.,newyork,1999)。例如,甲醛、戊二醛或其它具有醛反应性基团的类似试剂可以通过亚甲基(-ch2-)连接来交联蛋白质中的伯氨基与蛋白质或dna中的其它邻近氮原子。使得能够通过硫醚键实现连接的其它双功能交联剂包括n-琥珀酰亚胺基-4-(n-马来酰亚胺基甲基)-环己烷-1-羧酸酯(smcc)以引入马来酰亚胺基基团,或者n-琥珀酰亚胺基-4-(碘代乙酰基)-氨基苯甲酸酯(siab)以引入碘代乙酰基基团。引入马来酰亚胺基或卤代乙酰基基团到多肽上的其它双功能交联剂是本领域中公知的(参见美国专利申请2008/0050310、2005/0169933,可获自piercebiotechnologyinc.p.o.box117,rockland,il61105,usa)并包括但不限于双-马来酰亚胺基聚乙二醇(bmpeo)、bm(peo)2、bm(peo)3、n-(β-马来酰亚胺基丙氧基)琥珀酰亚胺酯(bmps)、γ-马来酰亚胺基丁酸n-琥珀酰亚胺酯(gmbs)、ε-马来酰亚胺基己酸n-羟基琥珀酰亚胺酯(emcs)、5-马来酰亚胺基戊酸nhs、hbvs、n-琥珀酰亚胺基-4-(n-马来酰亚胺基甲基)-环己烷-1-羧基-(6-酰胺基己酸酯)(其是smcc的“长链”类似物(lc-smcc))、m-马来酰亚胺基苯甲酰基-n-羟基琥珀酰亚胺酯(mbs)、4-(4-n-马来酰亚胺基苯基)-丁酸酰肼或hcl盐(mpbh)、3-(溴代乙酰氨基)丙酸n-琥珀酰亚胺酯(sbap)、n-琥珀酰亚胺基碘乙酸酯(sia)、κ-马来酰亚胺基十一烷酸n-琥珀酰亚胺酯(kmua)、4-(p-马来酰亚胺基苯基)-丁酸n-琥珀酰亚胺酯(smpb)、琥珀酰亚胺基-6-(β-马来酰亚胺基丙酰胺基)己酸酯(smph)、琥珀酰亚胺基-(4-乙烯砜基)苯甲酸酯(svsb)、二硫代双-马来酰亚胺基乙烷(dtme)、1,4-双-马来酰亚胺基丁烷(bmb)、1,4双马来酰亚胺基-2,3-二羟基丁烷(bmdb)、双-马来酰亚胺基己烷(bmh)、双-马来酰亚胺基乙烷(bmoe)、4-(n-马来酰亚胺基-甲基)环己烷-1-羧酸磺基琥珀酰亚胺基酯(sulfo-smcc)、磺基琥珀酰亚胺基(4-碘代乙酰基)氨基苯甲酸酯(sulfo-siab)、m-马来酰亚胺基苯甲酰基-n-羟基磺基琥珀酰亚胺酯(sulfo-mbs)、n-(γ-马来酰亚胺基丁酰氧基)磺基琥珀酰亚胺酯(sulfo-gmbs)、n-(ε-马来酰亚胺基己酰氧基)磺基琥珀酰亚胺酯(sulfo-emcs)、n-(κ-马来酰亚胺基十一酰氧基)磺基琥珀酰亚胺酯(sulfo-kmus)和4-(p-马来酰亚胺基苯基)丁酸磺基琥珀酰亚胺酯(sulfo-smpb)。可以用于交联的异源双功能交联剂可以包含胺反应性的n-羟基琥珀酰亚胺基团(nhs基团)和/或羧基反应性的肼基团。这类商业可得的异源双功能交联剂的实例包括琥珀酰亚胺基6-肼基尼克酰胺丙酮腙(sanh)、4-肼基对苯二甲酸琥珀酰亚胺酯盐酸盐(shth)和琥珀酰亚胺基烟酸肼盐酸盐(shnh)。携带酸不稳定键的偶联物也可以使用本发明的携带肼的苯并二氮衍生物制备。可以使用的双功能交联剂的实例包括琥珀酰亚胺基-p-甲酰基苯甲酸酯(sfb)和琥珀酰亚胺基-p-甲酰基苯氧基乙酸酯(sfpa)。使得能够通过二硫键交联的其它双功能交联剂是本领域中已知的且包括n-琥珀酰亚胺基-3-(2-吡啶基二硫)丙酸酯(spdp)、n-琥珀酰亚胺基-4-(2-吡啶基二硫)戊酸酯(spp)、n-琥珀酰亚胺基-4-(2-吡啶基二硫)丁酸酯(spdb)、n-琥珀酰亚胺基-4-(2-吡啶基二硫)2-磺基丁酸酯(sulfo-spdb)以引入二硫吡啶基基团。可以用于引入二硫基团的其它双功能交联剂是本领域中已知的且公开于美国专利6,913,748、6,716,821及美国专利公开2009/0274713和2010/0129314中,其全部通过引入并入本文。可选地,也可以使用引入硫醇基基团的交联剂如2-亚氨基四氢噻吩、同型半胱氨酸硫代内酯或s-乙酰基琥珀酸酐。上述双功能交联试剂中的两种或更多种可以一起使用以交联染色质片段中的dna、rna和蛋白质。3.限制性内切酶不需要本发明的dna和/或rna接头包含限制性内切酶识别位点。事实上,在某些实施方式中,可能更加希望的是本发明的dna和/或rna接头不包含限制性内切酶识别位点。但是,在某些实施方式中,本发明的dna和/或rna接头可以包含至少一个re识别位点如ii型re识别位点(例如,iis型re位点)。一般地,如果re切割的结果产生所需长度(如10-20bp)的dna或rna标签,可以使用本领域中已知的任何re及其识别位点。识别核酸分子内的至少一个识别位点且可以用于本发明的这类限制性内切酶对于本领域技术人员是明显的,特别是参照本文中提供的指导和说明性实施例时。参见,例如currentprotocolsinmolecularbiology,vol.2,1995,ed.ausubel等,greenepublish.assoc.&wileyinterscience,unit3.1.15和最新的newenglandbiolabscatalog或网站信息,2005及其以后。可能的限制性内切酶识别位点和识别该限制性内切酶识别位点的相应限制性内切酶的非排他性列表在以下报告。举例来说,iis型re如mmei可以用于产生侧邻连接的rna-dna接头的固定长度dna或rna标签。特别地,mmei识别位点可以设置在rna或dna接头的双链区域的末端处,以使得在mmei切割时,来源于rna或dna序列的17-21bp标签序列连接于当前连接的rna接头和dna接头。如果一个mmei位点出现于各rna和dna接头中,则两个产生的标签-一个是dna标签,另一个是rna标签-侧邻当前连接的rna接头和dna接头。两个标签可以另外地通过钝化进行处理以使得可以进行进一步的下游操作如pcr扩增、串联或测序。可以用于本发明的一些非穷尽的ii型限制性内切酶的实例包括:aari、aceiii、aloi、baei、bbr7i、bbvi、bbvii、bcci、bce83i、bceai、bcefi、bcgi、bcivi、bfii、bini、bpli、bsaxi、bscai、bsemii、bseri、bsgi、bsmi、bsmai、bsmfi、bsp24i、bspcni、bspmi、bsri、bsrdi、bstf5i、btgzi、btsi、cjei、cjepi、ecii、eco31i、eco57i、eco57mi、esp3i、fali、faui、foki、gsui、haeiv、hgai、hin4i、hphi、hpyav、ksp632i、mboii、mlyi、mmei、mnli、plei、ppii、psri、rleai、sapi、sfani、sspd5i、sth132i、stsi、taqii、tspdti、tspgwi、tspri和tth111ii(参见rebaseenzymes:rebasedotnebdotcomslashcgi-binslashoutsidelist网站中的列表;也参见szybalski,w.,1985,gene,40:169)。也可以使用本领域中已知的或以后发现的其它合适的re酶来实施本发明,其具有能够产生具有所需长度(例如,10-25bp至数百bp)的标签序列的类似性能。在某些实施方式中,限制性内切酶是iis型酶。在某些实施方式中,re产生约10-25bp或15-20bp的dna或rna标签序列。在某些实施方式中,re是mmei或gsui。几种ii类限制性内切酶的识别位点和切割位点的其它实例包括(括号中是识别位点和切割位点):bbvi(gcagc8/12)、hgai(gacgc5/10)、bsmfi(gggac10/14)、sfani(gcatc5/9)和bspi(acctgc4/8)。也可以使用人工限制性核酸内切酶。这些核酸内切酶可以通过蛋白质工程制备。例如,核酸内切酶foki已经通过插入工程化以使得其切割在dna底物的两条链上更远离其识别位点的一个核苷酸。参见li和chandrasegaran,proc.nat.acad.sciencesusa,90:2764-8,1993。这样的技术可以应用于制备具有所需识别序列和所需的从识别位点至切割位点的距离的限制性核酸内切酶。因此,在某些实施方式中,可用于本发明的组合物和方法的re酶包括人工限制性核酸内切酶,如能够在识别位点之外产生iis型切割片段的那些。但是,在其它某些实施方式中,可用于本发明的组合物和方法的re酶不包括人工限制性核酸内切酶。在某些实施方式中,iib型限制性内切酶识别位点可以并入设计的dna和/或rna接头中。iib型限制性内切酶(例如,bcgi和bpli)是需要adomet和mg2+辅因子两者的多聚体,且它们在其识别位点的两侧切割dna以切开识别位点。因此,iib型re位点可以工程化以跨越或跨过连接的rna和dna接头(例如,re位点的部分在rna接头上,且re位点的其余部分在dna接头上,以使得连接的dna和rna接头重构完整的iib型re位点),或完全在rna接头或dna接头内。在用iib型re消化时,可以产生rna和dna标签两者。在某些实施方式中,可以使用iig型re(如acui)识别位点代替iis型re位点。这类iig型re识别连续序列并仅在一侧切割(acui)。所有合适的ii型re识别位点的列表,例如,在一侧或两侧上其识别序列以外切割的ii型re,可以从各种来源获得。参见,例如restrictionendonucleases(nucleicacidsandmolecularbiology),a.pingoud编辑,springer;2004版(december1,2004),其通过引入并入本文。也参见newenglandbiolabs’2010目录及后续更新(通过引入并入本文)。在某些实施方式中,i型限制性内切酶也可以用于产生rna或dna标签,特别是dna标签。例如,i型re识别位点可以包括在dna接头中以使得re在连接的染色体dna中随机距离处切割。在某些实施方式中,iii型re识别位点(例如,ecop15i位点)可以用于rna和/或dna接头中。iii型re酶在其识别序列之外切割并需要相同dna分子内相反定向的两个这样的序列来完成切割。每次切割的两个需要的识别位点可以完全包含在dna接头内或完全包含在rna接头内,或者在两个接头中以使得仅正确连接的rna-dna接头再生re识别位点。iii型限制性位点和iii型酶的实例描述于例如,matsumura等,supersage,proc.natl.acad.sci.,usa100(26):15718-23(dec.2003;moencke-buchner等,j.biotechnol.,114:99-106,2004;mucke等,j.mol.biol,.312:687-698,2001;rao等,j.mol.biol.,209:599-606,1989;hadi等,j.mol.biol,.134:655-666,1979中,其全部通过引入并入本文。iii型限制性内切酶也可以从newenglandbiolabs(neb)购买。特别地,用于完成本发明的实施方式的示例性iii型re是iii型酶ecop15i。ecop15i的识别位点是cagcag(25/27)。任何上述限制性位点可以一起用于dna或rna接头中。例如,rna接头可以包含iis型re位点,且相应的dna接头可以不具有re位点,iig型re位点或iii型re位点等。4.串联体和文库在某些实施方式中,本发明的分离的pet多核苷酸可以与其它分离的pet多核苷酸接合或串联以形成pet多核苷酸的串联体。任意数目的pet多核苷酸可以接合在一起以用于测序分析的目的或用于克隆到合适的质粒或载体中的目的。因此,在另一方面,本发明提供包含至少两个pet多核苷酸的pet多核苷酸的串联体,各pet多核苷酸包含至少dna标签和至少一个rna标签,其中dna标签从染色体或基因组dna获得和rna标签从ncrna的cdna获得,其中dna和ncrna的cdna使用本发明的rna/dna接头和方法从交联的核酸-蛋白质复合物获得。pet多核苷酸串联体的各pet多核苷酸因此可以具有rna标签-rna接头-dna接头-dna标签(或相反定向)的一般结构。串联体可以通过许多本领域公认的方法中的任一种形成。特别地,可以使用长度控制的串联方法(ruan等,美国专利申请公开us2008/0124707a1,其通过引入并入本文)。在另一实施例中,如果需要,分离的pet多核苷酸可以在末端连接于可以被(ii型)限制性内切酶消化的一个或多个连接体寡核苷酸之前在两个末端进行修整。消化产物可以具有可帮助单个pet多核苷酸的串联化的相容性粘性末端。如果re位点对于连接于pet多核苷酸末端的所有连接体是相同的,则所有粘性末端对于连接和串联化是相容的,且单个pet多核苷酸可以独立地以头对尾方式或头对头方式连接在一起。如果连接体不同,例如,具有第一re位点的第一连接体可以连接于rna标签,而具有第二(不同)re位点的第二连接体可以连接于dna标签。在串联时,所有pet多核苷酸以头对头方式连接。因此,pet多核苷酸串联体的各pet多核苷酸可以以头对尾方式或头对头方式独立地连接于一个(对于末端pet多核苷酸)或两个(对于中间pet多核苷酸)另外的pet多核苷酸。在某些实施方式中,串联体内的所有pet多核苷酸以头对头方式连接。pet多核苷酸的dna和/或rna接头可以包含至少一个限制性内切酶识别位点,如iis型限制性内切酶(例如,mmei或gsui)的re识别位点。pet多核苷酸的串联体可以插入或克隆到载体或细胞中;细胞可以是细菌细胞。克隆的pet多核苷酸串联体可以通过re消化并在需要时单独地分离。明显的是,可以串联的本发明pet多核苷酸的数目取决于pet多核苷酸的长度,这可以由本领域技术人员容易地确定而无需过度的试验。在形成串联体后,多个标签可以克隆到载体中用于序列分析,或串联体可以在没有克隆的情况下通过本领域技术人员已知的方法直接测序,如通过本文中描述或本领域中已知的所谓下一代高通量测序方法中的任何一种,包括单分子测序方法。因此,pet多核苷酸的串联允许通过测序单一载体或克隆中的多个pet多核苷酸以串联方式高效地分析核酸分子。在相关的方面中,本发明提供包含至少两个pet多核苷酸的pet多核苷酸的文库,各pet多核苷酸包含至少dna标签和至少一个rna标签,其中dna标签从染色体或基因组dna获得且rna标签从ncrna的cdna获得,其中dna和ncrna的cdna使用本发明的rna/dna接头和方法从交联的核酸-蛋白质复合物获得。在某些实施方式中,文库可以包含最多一千万pet多核苷酸,或者最多一百万、十万、一万、一千、一百或10个pet多核苷酸。在某些实施方式中,文库未经过任何扩增,如pcr扩增。在某些实施方式中,文库进行扩增以使得文库内的至少两个成员来源于扩增,如pcr扩增、滚环扩增、克隆的遗传材料的生物学扩增或任何其它已知的扩增方法。pcr引物和探针序列可以基于连接于pet多核苷酸的末端的pcr连接体的信息或基于克隆载体上侧邻克隆的pet多核苷酸或其串联体的引物序列来制备。包含pet多核苷酸的pcr或其它扩增产物然后可以用识别侧翼re限制性位点(连接体内)的酶分离以导致扩增的文库,其可用于许多下游分析中的任一种。在某些实施方式中,pet多核苷酸串联体在扩增之前或之后通过任何标准方法选择合适的大小,包括凝胶电泳和凝胶切除。合适大小的选择中的主要考虑是该大小应当高于引物二聚体和未退火连接体的大小且低于特定的长线性多聚体的大小。特别地,可以选择具有大约100-1000bp或200-500bp的大小的串联体。因此,通过大小选择,优势是可以消除长的线性多聚体,因为它们的大小高于该尺寸范围。类似地,过短的未退火连接体和引物二聚体的片段也可以消除。5.染色质免疫沉淀(chip)在某些实施方式中,本发明的方法可以用于鉴别特定的ncrna-染色质/蛋白质-dna相互作用。例如,在某些实施方式中,可能有意义的是测定与特定染色质组分或蛋白质相关的任何ncrna-dna-染色质相互作用。本发明的方法可以进一步包括使用chip来免疫沉淀目标蛋白质。chip已用于富集并因而允许鉴别与特定蛋白质如组蛋白和核酸蛋白质复合物中与核酸结合的其它蛋白质相关的基因组区域(综述于taverner等,genomebiol.,2004,5(3):210中)。目的是在其相互作用的位点处交联蛋白质与dna。这可以通过直接添加合适的固定剂如甲醛、仲甲醛、戊二醛、丙酮、甲醇或其它双功能交联试剂(或其混合物)到培养中的活细胞来快速和高效地完成。然后制备这些固定的细胞的粗提取物,并按照本发明的方法破裂染色质。例如,破裂可以通过物理剪切(例如,通过超声处理、水力剪切、经皮下注射针的重复抽吸的剪切)或通过酶促消化(如限制性内切酶消化或具有受控的时机、酶浓度、温度、ph等的核酸内切酶的消化)实现以获得希望的平均尺寸(例如,通常约1kb)。交联的和剪切的染色质片段然后用于采用针对特定目标蛋白质(例如,转录因子或组蛋白)产生的抗体进行的免疫沉淀反应中。在各免疫沉淀中富集的交联ncrna和dna片段随后使用本发明的dna和rna接头通过邻位连接来连接,然后从蛋白质组分去连接或反交联(例如,通过热和/或proteasek消化),并纯化以允许通过本发明的方法进行它们的鉴别。使用chip的优势是这一途径能够通过染色质和其它非组蛋白蛋白质的快速交联在活细胞中“冷冻”ncrna或基因调控网络,如同这样的相互作用以其天然状态存在,由此在理论上表现在任何时间点特定ncrna或基因调控系统的“真实”图像,例如,没有由异源表达施加的潜在伪像。6.应用本发明的方法和组合物允许在无偏的总体水平或在感兴趣的特定ncrna或特定染色质组分水平上鉴别ncrna和基因组位点之间的相互作用。使用本方法获得的信息可以用于广泛的研究和开发环境中。例如,本发明提供鉴别特定的染色质靶标的方法,该ncrna可以具有先前未知的或不完全理解的功能,该方法包括使用本发明的方法和组合物测定特定ncrna及其基因组靶标序列之间的相互作用。鉴别的基因组靶标序列代表ncrna在其上发挥其生物学功能的候选靶标。在相关的方面中,本发明提供鉴别与特定基因或基因组区域(如携带肿瘤抑制基因或致癌基因的基因或基因组区域)相互作用的ncrna的方法,该方法包括使用本发明的方法和组合物测定特定基因或基因组区域与基因组的ncrna之间的相互作用。鉴别的ncrna代表基因功能的候选调节子(例如,抑制子、增强子或共激活剂)。在某些实施方式中,该方法进一步包括在两个或更多个样品之间比较ncrna和基因/基因组区域之间相互作用的存在/不存在或程度。这种比较可以帮助进一步解译相互作用和任何观察到的样品间差异的生物学意义。例如,样品之一可以是健康对照样品,且其它样品可以是疾病样品,如来自动物模型(例如,小鼠或大鼠模型)的疾病样品、在特定治疗之前和之后的疾病样品、在不同治疗阶段的疾病样品、来自对特定治疗有反应的患者或对治疗具有抗性的患者或在治疗后复发的患者的疾病样品。在某些实施方式中,样品之一是来自患者的干细胞或诱导的多能干细胞(ips),且任选地其它样品可以是从这类干细胞或ips细胞分化的细胞系。此时,特定ncrna-染色质相互作用可以与发育或分化程序的启动相关。在某些实施方式中,样品可以来自人、非人灵长类/哺乳动物、家畜(牛、马、猪、绵羊、山羊、鸡、骆驼、驴、猫和狗)、哺乳动物模型生物体(小鼠、大鼠、仓鼠、豚鼠、兔或其它啮齿动物)、两栖动物(例如,爪蟾)、鱼(例如,斑马鱼)、昆虫(果蝇)、线虫(例如,秀丽隐杆线虫(c.elegans))、植物、藻类、真菌(酵母,如酿酒酵母或裂殖酵母)。样品可以是建立的细胞系的组织培养物、培养的原代细胞、组织活检样品(新解剖的或冷冻的)等。如实施例9中所示的,本发明的方法鉴别ncrna-ccat1(结肠癌相关转录物1)–如这一位点中具有非常复杂的转录物亚型。rich-pet数据提供ccat1的潜在功能和基础机制的重要认识。具体地,发现ccat1位点本身具有显著的增强子特征,ccat1位点在宫颈癌细胞系hela细胞中高度表达,且rich-pet数据显示这一位点的转录产物靶向其它增强子和启动子区域。例如,对于ccat1ncrna转录物靶向的122个位点(各具有≥3rna标签),88个位点是增强子区域,包括六个具有rnapii相互作用的增强子位点。另外34个位点在启动子区域内。这与ccat1靶基因平均来说比随机选择的基因组更高地表达的观察一致。因此,lncrnaccat1可以用作转录辅因子以激活基因网络,包括致癌基因c-myc。因此,本发明的另一方面提供治疗表达ccat1的癌症的方法,该方法包括施用ccat1编码的lncrna的拮抗剂。在相关的方面中,本发明提供破坏由ccat1的基因产物(例如,转录的lncrna)介导的转录激活或共激活的方法,包括将基因产物与ccat1编码的lncrna的拮抗剂接触。在某些实施方式中,癌细胞中存在转录激活或共激活。在某些实施方式中,转录激活或共激活是对于c-myc、fan84b和/或snx14。在某些实施方式中,转录激活或共激活通过使ccat1基因组位点与靶基因位点物理接近来实现。在某些实施方式中,癌症是结肠癌(例如,结肠的腺癌)、直肠癌、宫颈癌、肺癌、胃癌、肝癌及其转移。在某些实施方式中,癌症以与匹配或对照样品相比高2-倍、3-、5-、10-、15-、20-、30-、40-、50-、60-、70-、80-、90-、100-、120-、150-、175-、200-、250-、300-、500-、1000-倍的水平表达ccat1转录物。在某些实施方式中,拮抗剂是可以任选地包含修饰的核苷酸的反义多核苷酸以例如改善血清稳定性、药理或药代动力学特性等。修饰的核苷酸可以包含pna、lna、2’-o-烷基或其它2’修饰和/或糖-磷酸酯骨架上的修饰。在某些实施方式中,拮抗剂是靶向编码的ccat1lncrna的sirna或mirna构建体。本发明还提供ccat1lncrna的拮抗剂(反义、sirna、mirna或其编码/表达载体)。在另一方面中,本发明提供药物筛选的方法,该方法包括建立药物功效与通过本发明的方法鉴别的所观察的特定ncrna-染色质相互作用(如在反应性患者中但非在抗性患者中鉴别的相互作用)之间的统计学显著的相关性或关联性,从而确定多种候选药物对统计学显著的相关性或关联性的影响并鉴别促进统计学显著的相关性或关联性的候选药物。在某些实施方式中,候选药物的效果使用来自抗性患者的样品进行测试。这可以允许鉴别恢复抗性患者中的统计学显著的相关性的候选药物。在另一方面中,本发明提供鉴别用于治疗疾病的靶基因的方法,该方法包括:(1)使用本发明的方法鉴别(观察的ncrna-基因组dna相互作用中)药物的功效与特定ncrna-基因组dna(基因)相互作用之间的统计学显著的相关性(例如,在对治疗有反应的患者中观察到功效的任何时候,观察到特定的ncrna-基因组dna(基因)相互作用;在对治疗没有反应的患者中没有观察到功效的任何时候,未观察到特定的ncrna-基因组dna(基因)相互作用),(2)测定涉及的ncrna和/或dna(基因)的表达水平;其中dna(基因)在药物疗效与增加的ncrna表达和dna(表达)表达的抑制相关时鉴别为用于治疗疾病的潜在靶基因。本发明的组合物和方法也可以用于鉴别特定基因组中迄今仍然未知的ncrna,因为本发明的方法是用于鉴别这类ncrna的无偏途径。如果pet多核苷酸簇始终鉴别不编码任何蛋白质的基因组一个区域中的rna标签簇且始终将这些rna标签与由对应dna标签代表的(远程,例如,染色体间)位点关联,很可能rna标签揭示ncrna。通过本发明的筛选方法鉴别的任何候选治疗剂或靶基因可以使用与疾病或病症相关的公知试验模型体外和/或体内验证。例如,如果特定ncrna鉴别为促进致癌基因的表达(或鉴别为抑制肿瘤抑制基因的表达),因此变为候选药物靶标,则使用ncrna的拮抗剂如sirna、mirna反义等的潜在疗法可以进一步体外和/或体内验证,后者可以在建立的癌症模型(例如,模型动物中,如待治疗癌症的小鼠模型)中进行。小鼠是用于药物发现和开发的公认的模型,其中可获得许多不同的种系。例如,用于研究癌症的大量可用模型可以在mousemodelsofhumancancersconsortium找到,其已经开发了几个数据库,例如,emice(emicedotncidotnihdotgov)、cancermodels数据库(cancermodelsdotncidotnihdotgov)和cancerimages数据库(cancerimagesdotncidotnihdotgov),或者在其它来源找到,如由thejacksonlaboratory发布的癌症研究模型(参见jaxmicedotjaxdotorgslashlistslashrax3dothtml)。使用原发癌症活检样品或细胞系的进一步的异种移植模型可用于研究癌症。例如,为开发其中可以验证针对候选ncrna的潜在拮抗剂的功效的肺癌模型,六至八只8周龄的雌性免疫缺陷小鼠如cb17-scidbeige小鼠(taconic,cat.no.cbscbg)或nod/scid(thejacksonlaboratorycat.001303)或nodscidgamma小鼠(也称为nsg)(thejacksonlaboratorycat.5557)经由左肺皮下或经胸注射(常位的;104/sup细胞/25μl)人肺癌a549细胞携带肿瘤的小鼠腹膜内注射中和抗-cxcl12或免疫前血清,或不接受处理。可选地,携带肿瘤的小鼠可以用platinol(顺铂)或abitrexate(甲氨喋呤)或紫杉醇或者其它化合物处理。肿瘤在各个时间点进行分离,其经处理或未处理。非编码rna按照之前描述的方法鉴别。7.ccat1转录物、拮抗剂及其用途在另一方面中,本发明提供通过本发明的方法鉴别的各种ccat1转录物、其cdna序列(两条链)、拮抗剂(例如,拮抗这些ccat1ncrna转录物的功能的反义序列、sirna或mirna构建体)。代表ccat1ncrna的不同亚型的八个鉴别的cdna序列在以下seqidno:1-8中提供。>ccat1_jax_1转录物序列;基因组位置:chr8:128128655-128241571链:-<128241571>3'-tataggtataaccaatatatatgtataacatatatatgtccatatatatgtataaccaaaccacaggtgtttttttggaagtcatattatacagggagttgacagaggtgtgagctggactttaagaagctgcacataagatgctagtatgatcaagctggaatggacttagacaatttgaaacaacttttctcagttttcagatgaggaaactgacgggtaccaagcttaaatgacttgacgaagctcatagaagattagcaggtagtagaataatgactgctgactcctaattcagtggatcttccctggccaccgttttgtattgagctgcaatgcttccttgactgttctccatgccagattcttatcaatgatctttcacctaagaaacagcaaagattctggcaagcacacgatctagagatacatcttattgcgatttttcacaaaaatcaaaagaagaaagaaggcttagctggtgtttaattattgttatttttttcaatagggaaatctgtacacaatgatttatctccagtgatttgccattgatcaatttttttctcatttcattttctatttttttgttttttgtttttctttattttttatttttttctcctttttctttttttaaattttctgtttatcacaaatgatcatgtaattatatgttaatactatgtaaccccagtgttttcaactgtttgtgattcaatgttacccagttttcttttcttaattttaaataaatttgaaaaattatcttagagtgttttgagcctgtgttggtacatttagttctagttcattgtggtaaatccacttcagtttctaagtttccaccctttagtaaagacatatttctaaatttggtttatatcctcagttacaaaagattctaactgctagttttgtgacagcaatcacacataccatcaagggatggggcaggcagttttggaatgtgctgatgctagcatttttataagcctatggcttttatagtctaaattgttcttatttctattaatgcttcaatttttgacaaacacacaaccatagaaacaaataataaatttgttttttgggaattatccaggattcttggttaagtggggaatttaggctttgacagcataaaggatcacggataagtatttttcacggtggctctaattataatgctgagcatgtggcaggcacagaaatatttactcattgactgaatatagcacatcgtaatgttgatttttttccaacataattttagagctaggcatattgtattctattacactagactatatatcattcttaaatagaaccagccttgctagataacacatgttggaggagaggcccttcttcttagccctcagtgtttccatctatggggaagaagttccaccatactaacattactatcgtctctccacctgctcactcacttctccccaagggaggggtgttcgatatggtttctgagcttggaaagaaaactcaggcatgtgtaacatggttccttcagtcccatgacccactgtccacaaatgggctgctcacagagtgcatgccttcacccttgttcctggccatgcaggaaattgtatgaaacagtcctagctgaagcctgagattttcctgcattgcctagtcctggtgggtatctgtctactcctggagtttggattggaaagtccacatgcctgaaggtataaacctattctacaaaggggtgttttctagaatgaaggtaatatttttatcttacatttgcagaaagagacagaacaatgttataggtgagtgcatggacaatgacctcaaacagctaagattcaaacccctgcgttgaatgattgaattgaaatgattcaatgagctaatgtacataaagcatccagaatgttgcctggcacaagggactgtattgtctgctagaccatttattcaaagtgggaggatgatgttctaaaagccaatgataaagctcatggcaatgcagggtatatctgatggcatggaatgctttaggatggccaagattgcccatcaaatgccaagtcaccgaagggttctgggaaataagaaatcattagaaaaagatttctgccttctagaagtacacagtctaatggtgagataggcaggttattaatggctctcctactaggagcctgacatcatggtgagcatcgagaaaggaataacctaagctgaagacacgccttttcaggaggccaagttccacgttctgtgcatgcttttggcgaaagtcaggtaaagcctccagaaatgcatggtctattctctcggaccatatggctgtgggcaaactctggcttcttggagcctccattagccacctagggaaaggtaattggctttatgtttggctccatcactgctggcgacaagctccactgcattctcaagcagtagatgaaatacaactgtgctcctgaatgcagcaaaggaaagagaacccaactggaccagtgtgaaattgggacctttggaggcaagaatgcacatatttataaatgaataaacataatttttctctcttcttcccattggcagcatatctgaaggccctgactcaacagtagtgggtcttaatgcttgtgcttgtccacccttctcggcagtgattgatttcattgttggcttgttttcatgacttgaatgtgcgatcttcagagggcctaattccttgcaagttccatttgagggtggaagagctagaacaagctataaccagtagacaactccgtgactcaggagcttaagcatgtgactaattagtaagaaaaaatgtggtgaagatttgtagttaataagaaggaaagaagaatcactggggctagaattatgcaagcttttgtttccttttgggactatatcagaactatgagagaagaaaggctaccttttacctttgaggaattttcaaagcctttttttttttttttggttggtttgtgactgacaaagggcacaatttcaacacctcagaaaaatgcctcatcatttcctcttgtgaaatctgggtgcttctgaatgaatccatgttaggaatgtgtacttccatccattaaagtcaatgtccagtctcattttgggccagaggctgttactaagttgtagtactggtgagaaaaggacagagcatttaccttccctgggtatcctgtgacctacatgtgtccttgcaaggcagggaaatgttactaattaagagcatagcctttcgtcagatgggttcagatcctaacacatcctctttctggttacaggacagtcctctctgagcctcagttttctcatctctaagatgaggctaattatacctacttcccaggggggtactaaggataaagtaacaacacaagaaaattttttatccttgattttcttagctggaaaattaggctagaaagacctccatccttggggtatttaaaataaaaggacaaacatatgatgagtctaagtgatcaatacattgtggcttttgtacttactattctgaaacatgggtgtggcctagatactttccaaaattctgccctctccacctagcaaaatgacaaccagacttacagatatgccaacaagctagtgtttaataatggtgtttgatgataaatggcatttcttttcgacatttgtctctttttaaaacttggtgctcttaaatgcatcattggataagtgatgactgttcccattcgcaagaagagacctggaatctaagcatgaaggacctgtcctgatgttgagaagtgtggatcacatttatttgtaaacttagcttcttgctcgtctcatggttgcttttttttctttttcctttttcttttttttttttttttttttttttttttttttgagagggagtctcactctgtcacccaggctggagtgcagtggtgtgatcccggctcactgcaacctccacctcccggtttcaagcaattctcctgcttcagcctccccagaagctgggattacaggcacgtgccaaggatttaatggcaagatgccattatagacaagaacaggattcagacactttcgtgttatgtgttcttgtccaaatactgtgtcactctgcactggcatcccaatcccaccaacacctttagcaggaacttcctgtttcaataacatttctcaatactctacctgattgcttagaatccatgagacagccatattctccatgtctaggtccctatcttatttttgttgagatggtgttaagactttatttgtgaggcttctggggaagaggagtaaggtattgatcccactgactggataatttgggtctcaaaatggataataaataagcattacatattttgaccacttccttggaggagaacttcttggaatgtgcaccatgtccgctgcactttttttgcacagatatctaagttggagaaacatactactagataaatcaatttgttctcttagtactcatgatatggttcctgggaacttctgattcaccaaattaatcttggccaggtacatacctggcaggaatccaaaaattccccaagtctccttgaagttcaggatcatcattcttaataaatacaccgagggaaaaaccatggagagtttgtcccagatgctgtgaatctggcccggggtacatgaagaagtccttaattgcagtcatttacatggtagattctctataatcatttaatttgctataggtctatgatttttagtccttcttctctaaatgattgaacatgtataattcccatttcaatcatattacctggatgaacaaaagtaacgctagactcattcatgcattctggttgccaaggaaaaggaaaaaaaaacaaaacaatcaacaggatgtttaaactgtcttagggcaacttcaggccatagtcactggtgttcttgcagactatgagatattttacattctgataagggataaaaattcgtgcctcacatggctcccatcacactaagatcttgcaacaataacactactgattcagacattaatcttaagtatccagggagccctaaaacattgtatcccactagcaaggaccatggtaattgccacgtaaatcccctccattatgtggcccttattatgaccagccagccaaggcttgcctttaaatcataccaattgaaccgagccttgtagaaacactatcacctacgcatacctctgcttcttttcattaacctgctatcctctttacaaatgggattcttcacccactcccttcttctagattagcaatgccctgttaagtaaacgaacacgaaattcaaagggaaacaggagcaatcatcattaccagctgccgtgttaagcattgcgaaaacgctcacgattcacagaaaaatccatgctgttctttgaaggcattcaagccttaatagctagctggatgaatgtttaacttctaggccaggcactactctgtcccaacaataagccctgtacattgggaaaggtgccgagacatgaactttggtcttctctgcaatccatctggagcattcactgacaacatcgactttgaagttgcactgacctggccagccctgccacttaccaggttgggctctgtatggctaagcgttttctcctaaaatcccttgaaaactgtgagaagaccataagaagatcatatctttaattctatttcacaagtcacacaatattccaatcaaatacagatggttgagaaaagtcatccatcttccctccccaccctcccacagcccctcaaccactgccctgaaacttatatgctgttatccgcagctccatctggagcatcacagctactgtcaaccctgacgctctttctgaaaaaacaccggatggacatcagaactatttctttaaggatgttactgagccacacaggaaaacttgccttatgattttgaatgcacggatctgatttgactaaacatgataactagagaatcacccaatctactcccattttcaactctaaatcatcagagtgtctcaaatccaaagcacacacagaccagcctggccaacacggtgaaactccacccctactaaaagtataaaaattatccaggtgtggtggcgggcgcctgtaatccaagctacttgggagtctggaggcaggagaatcccttgaacctgggagatggaggttgcagtgagcagagatcacaccaccgcactctagcctgggccacaaatcaacaacaacaacaacaacaaaaaacaaagcgcacacagagactgaggtcctctttggcattgagaagatggctatgcaagtcccaactagcaagtgcaaacttcccagcttcacttctgccagtgtcccttcaccccttctcaaccccactgggaggcaggagggtgcttgacaataacagccttggcatcactctgccagggtgtaataggaactgttacaattctgagattctgtgtaagcactggcctttctgcctagaatgccttctcctctcttttttaactgcatgctcctatttatctttcaaagcccggaaaaaataacactgcacacgggaaatgctcccttcctactgcagtcatttagatgactctatgccattccattcatttctctttcctaccacagaagtgctttgagattttggagtcagactgcttgaacttgaatcctggccctctcatcagagacttgacttattttaggcaagttatataaccaattttacctcagttccttacccataaaatgggtctaatgagagtacctaccacacagaattttgatgaaaactgaatgagatgaaggcctttaaggcagtggtccccaaccctggggacacagacaggtaccattttgtggcctgttaggaactgggccacacagcaggaggtgagcagtgggtgagtgagatcagcgttatttacagctgctccccattgctcaccttactgcctgagctccacctcctgtcagatcagcagtggcattaaattctcatagcagcacaaaccctgtcatgaactgcacatgcgagggatctaggttgtgcgctccttatgagaatctaatgcctaatgacctgtcaccgtctcccatcacccctagatgggagtgtctagttgcaggaaacaagctcagggcttccactgattctacattatggtgagttgtataattatttcattatataatacaatgtaataataatagaaacacagtgcacaacaaatgtaatgtgcttgaatcatccccaaaccatcccagtccacggtcttccacattttgtcttttcacaaaattgtcttccacaaaactggtccctggtgccaaaaaggcttgggaccactgctttaaagcctttgcatagtgcttagaattgagggggaaaaaaaaaacaaaaacaatgtagctagttgctacaatcactatattggtgagtttcaaaaggaaaagaattctgtcccatttatgcttgagccttgagttgctaaccaagcctgacacaaaattactgttgaagggatgtgtgagtcctaattgaaatgaggcctcttaagggaattgtggaccaaaccccaagcaggcagaaagccgtatcttaattattgcaagtatttcaggcaaggtgtggatggccatttgaattcaagcagactaggacctgggatgagaaagaaggtgtgtacgtgacttgatctttgaactttagctcaccatctggaagaaggctgagtattctctgcactcacatagtagctaatgcctactccccagccacccacaattctttctgtaggaaggctcgctagaatactttgtgatattggatattagttccatattctactgtgtatcttagttcaaccaaattgtaatcatctgatatttatttcttttaatataaatataagtatattaagtcttggcatgcttgctcagtctctctctctctcccattcctccccgctcccctctctctttcccaacaggcttggaaagcaggcatcaccatgcctatttaacagttggggtcccttggccaccaggtgctggagtaggaatctgagcccggacatgcctgatctgtaaattttgtgttttccccactgtgctgggcagatcacagctatcagcgccaaattcatagaaggggcgccccctgtggtcaattgagggatttgtgtttgaggtagatctcaagaaggaatgggtggggaacttagcctaggacagagcagaaaggagccctcactccccaagcaccaacggcctcagtccttcctgctgactccagcctctagctctcacccagactatctgcatccttctctccaccacgctcctttggaacctgcgtaaaacacagattaaaggaattccgccttacttccctttccgcattatgaccaaatggttttacactatcattgaacagtttagtacaaaacatgccaccttttaatctattcattcatttaacaaatactttggagtgtttaccatgtgccaagtgctgttctaatagacataagctgtgaggttatgcttatctgattctcacagcaacagctttcgagatatgaattggtatactcatttgacagatgaggaaattgaattcatgtagtgagaaggaagagctgcaattcagggttactggtttctcctgcactaagcactgagccacactagaagagaaggcatgaggaagacaaaagtgaggctgtggcttgccttttcacttcttgtgtcctgtttaagaaatatctgctatcccaaggtaagatactatactttttttaacatgttattttgttttacctttcacatttggtgtatactacatttgtaattaatttgtccatattatatgacatatagccaagatttattttttaccatacagatactcaatattgccatttacgtagaacatcgttctcttcctactcaattgccttggcacctttgtaataaatcagatgatcgtgtatgtgtagttcagtttctggactctgtcttctgtttctttagtctatttgcctattcttgtaccaatatgcactgtgttaataatcgtagctttgtagtaggtctcgaaatctgacagtgtaattcttttagtttctttcttctgcaaattttctttagctgttttacgtcctttgcatttgtatataaacttcagaatcagcttgtctattccaaaaacaacaacaacaaatgaaagtttcagaaactttaactgaaattgtattgaatctgtagacaattttggagtaaattgcatcttagcaatgtagagtctttcgaaccataaccatggtaagtctctccatttgcttaaatcttctttaatttatttcaacaatggcttccaatttccagcgggagctcttggaaactacaatttacatgaacttctaatttgatatttttcggtgtcattataaacattgttgttttaaaagttgtcttcaattttttgttgtcaggcacagaaatacaattattgataacatttatatataaactgtatccagtgatcttgctagattcactgatgagtctgatgttgtagattctttgggattttctccgtacataatcatatcctttctgaataagatagttttacttcttcatttttaatatctatgccttttatttatttttcttttgaactttttgctgacttcattattcactctcatgtttttctctttcattagactatgactcctcgatggtagcaatttgtagtaatcaaatttttgtattttattttagcatctggcatctttcttgacatatgtagcagttgctttttgacagcttgcttcttcagtgaatgaataaattaataaagagaaatgtgatgttcagtgatccattttgcaggtaagaaaactgaggcagacagaggatgttagcaagcaagaggccttggcctacaatttagatcactggactcttactccagatgcaatctgcagaacccacatacttttaattagtccctttgtctatgttctgccactgtcacttctaaggaaggtgtgtcatcccaaatggggtagtatcttattggtagacctaaatctgctgtgttcgccatctcacctacatgagtatctatgtgtagcattctgcatattcatcttttccaccttctggaggttttgtctttttataggcagcatgtgaataacaatggggccaaactggggaccagaaggggccattttctagttctgaacatagataagcatcactaacttttccctcctggcagtaatggcctcaaaagttccaacttaggagaaaaaggcaaaaccgtctgccaaagtgtgtgaaaagttagagcaaaccttggttttaccaagaacctgtgtccctcttatggaaattcacactttcacacttttagacaaatattaaatgtgtgacattctattacgtacagtgcctggcacatctagacacgcagcacactttagcccccttctttcttcttctaactccaagttctaaactagaaaaagccccacttgagtctgagatttgctttttgaactagtttatttcagattgtaatcatgctatctagggttgtgacagtgtttgctatttctagggcactttgacctgattctttttgcacaggaaacttgttctacccttttgcccacttcactgaagtgaggactgagacagagaaggattaagtcactttttatttaacaaatatttgttccacattcctcagcatttattaaatactggtgcataatgatggaataaattttataccgtaaggataaaccagtattctggactaagccaacgtgggagaccctaggaggcctgtttgagaaagtgacatttaaattgagacttggcggtggctgtggctacatatctagtaagtggctgggttgggatttgaactcatgcctgcttagctctaaagatgatgcttttggctttgtactctgctctctctctagacaaactctggtccaaaatcgttaaagctaacatttatccctgcccaactggaattgtcatgttatgacaaatggctctgtggtctcagatgcccagcagacccattagtggaattctatgtgctacagacctgggcaaaatgccagagccttatacacccatcacatttcgtctggcaaaggtcttcaacaaagagaagtaattacagcaatgaaaagcaacaggtccagcaacaccataagaacaaaataattaatttccctaaaatagaagaaaccatttatagagtaagagccgatacaatcaataattggaagaaatagaagaggctttagtattctagccttctttatttgtagatgtaaatgtcgagcctcagagaagttatatatctaattagtgtcactcaggtagataacaacagaattaggattagaacttaattcttatgactcccagagcagggaaaagacaggatgaagtcccaaaacattgcgtgtgaactcacatctgactctgaattgagagtctgctatttactccctatgtgaccagaatccttcagagcccatgagattccctgtcatagagtagattttgatccacactaggcatttttacctcttgctctttgagttggtgccccatgtttactcagaaatattccaaaggtgttacatctattggttttacatgttgagcacagatcattataagacaaattgaaatgaaaacatcaacaagtctcattcattgtctaacttacgctgagcaatatttaataactagaatatcaagagagtccaaagtgtttgcccatcccctcaagaccaatgtaatggaattttactcttatcacctgctcagggtagtggcaattcaggatacagaggacagaaataaagaatcatgacacacaatccacagaattcacagatgccaaacatctacccttctctgtcaccacacattggactcacatggtggaaataggcaacacaagcagagaggtggcttaacctttcataatttttcaacgacctatgggaagagagttttcttggttcaaatcccagcttagccacacagagtgtggtaatattgggcaagtcaaccaagctctctgtgcttcagtttcctcattattaaaatgggggaaataatagtgcctgcatcagagggttgttgtgagaactaaacgaaataatttatctgagctttagaacccacaccatattagttaaaaattcatgcattttcttttattatatttctctaccttagactgcaaactctaagaggaaaggccggactgttatattcataaagcattacaggaacagtaattagaactaggagcttttcaatggcctgcctgaaatctgaaaaataggtatattatttgaaattttgaaaaaatcaaataattaaaaattaatagatgttaataaaatatctgtaatatgtaatatcaaggtcaactcaactcttaattgtttatataaaatatagtgaagtttaaattgcaaaatcttacagaaaatgtgctattaaaactcaaaagtataattctttctaatatgtatatatatgtatatagttttattttaagttcaggggcacatgtgcaagtttgttacctagggaaactcacgtcacaggactttgttatacagatactttcatcacccaggtattaagcctagtgctcattagttatttttcctgatcctctccctcctcccaacctccaccctcaggtaggcctcagtatctcttgttcccctctatgtgttcatgagttcttatcatttagctcccacttacaagtgagaacatgtggcatttggttttctgttcctgccttagtttgctaaggaaagcactgtggcaatttctcaaaggactaaaaacggaattaccattcaacccagcaatcccattactagttacgtactcaaatatttttaaggcaaaacaaagctgcaaccagaacacctggactccctgaaaccccttccactgatgttgttgttgttgtttctttttccccagcttctcaggccaaaatactggatcatcttgggcactgttctctcctgcccaccctttcccatatgcagagtgttgtcacttctctctgcttccactgctaactccctggtccaagccgctgcaccacttttcgtgattattgccacagtctcctcactggttccctgcccccactcttgctctgaactatccagttaaaacctgaattagatcatctcatcctcatctcagagctttctcgtggctcctctgccctctcaggaaaaaatctaaattctagatgacctaaaaatcccttgtctcttactgtttatctgacctcatttactaccacctttttctttgatcattctgttccagccacactggcctccttaccactcctcaaatatgccaagcacagcccccaccccccagggctttgaactggctgatcccccttcctggaatgccttaccccaaatatcaacttagccaactccctcctctcctccaagtgtctgtttaaacatggcttcagtaggagctgtcttaacatcctattaatattgtaattcctctcatgacactttacacccccttccctgatatgctttccatataccatgcaatatctgctgagataatatataattcacttattttctttattgtcatttcaaagagggcggtgtgttctgtgttttatttagtgccaaaatacttgctgatgaagagagttcctgccacatagtaggtgctcaatatgtgcttgttgaataaatgtgtcaatgtttgatgtacagaccttttattatgtttgatttgctgccagtgctgcctccaaacacaggagtgcttcatgagatgttcacaaaagctcttaaaatattccacaaaaatcttaaaatatttcatgagttttctttcctgtatttttatagcagcatctggaatttagcctgcataggaccctctgtaagctgaccctgtttatctattcagctttacttctcccctctctccactttgtattttattctctactacttccaactgattgtaatttgaccagactccagactatcttatgcctctttgcttttgtttacctgttacttctctctggaattccctgccccttcttaatttttctggccaattctcactctctaggactcagaggtttctcctcaggagacttccatgagtctcatgttgagttaggtgaccccaatcctctgttcttcatagtcattcgcgcatttatctagctcagcatttgccatactacattgaaattatttccttatgtgcccatcactccccgtagattgcaaactcctagagaagggctcaacagtgagtgctgaggctgcacagaggaggaaggcagcacaatgatggaaggcttcctaaagaggtatgttccaagagcccccacttcctttcatgggagactcatgctgttacacctagactatctagggatacatctaatgtagtcgtggaaagaacagaggacttgagtactagactgacgtgattttgaatcctggctccctattgaccagatgtgtgtcttgaacaagtccctgagcctcagtgtcttcatctgcacagtgaggataatgataccacactgcatatatgaggtatccggcacatgtaaatgtccactacatgctgatttcttcacccgctactcacccctgggaaagaagtagactcacctactcttggtacccattcattccccctcagttggaagcatgaggtgtgcagctgcctgacctgggggaagggctgcaagcagtaggtgttgtcagatgtggtggagcttgttgacttcctcccaggggcccagctaacaacctgcctctgttccttgatagtcaagttcaactttcacttcttagcaccacaggaagttgactgaacattaactgaagtctctctcaaacaggagacatctttgccaggtccctgtacttcctagcctcattcctgctctccctagtgagcaggctgccctcccttctcgccccagcaccactgataggcaagggtactcagaactactaccttgtgggccatgtcatgtgccaggagctgcacccaggactttaatacagtagttggctcccactgaatgttcattgttaccccaggataaaaaggggacactgtgatcattttctattttgctgtgatcaggcttggtgagctaaagtcacctaccttcccagtctctactaatagaagtcatggatcagtcctattggttcttctgttacaaggattcagaattcataatcatggagctgcatttacaggcagaagtttctttcatagttttctaagtgttccttttagcaacaatggagaaaatcaaagagggcaaaggtgaggggagaaaataacatttccctttctgtcctttgctcttgtagtcttttgctttagtttctttactatgactgtgagggtgaaactagtgatcagagtggtccagaatgggtttgatgaatctgattctggtgacacaagatgaattgggtatatgtttccctaaagatagagagacaatataacatagtctttacattaatagactctggagccaatttttttaggttcactctctttcctttcatgtgtgttgattttcaacaaacatcttgcactcgaattccatctcactgttttattttcaaaaaatttaatttgagaaagttagctgtattaattttttctttttctaaaattcttttactaattgcaattatttccattgatgctattccattgaaaccattttaacatggactcaataacttcattgtatgttaatgtttaattttcatttctttaccttcttggttttctagctgtgtttaatgtggttgaccactcattctttgaagctctattcctctggctactacagtatgacacattttgtctccttcttcagtctctgtcttctccacagtcttctcttccttttatatacctttaaatattaatgtttcccagagatattttcttaactcacttctctactgattctaggtacttttcttgatccaactcttctgattttacccatctcgatgattctataatttgtattttctgttttgatctcttttcagtcttccagacctaaatatccaaatgcctgatggatagtgctttcttttttactaccaagccctcaaaggcactatgttcaaaaggaatttgtcatcagtctcacggcacataagcttcctcttgtgttcaatctggagacttgagagtcttcctgttcccttcttctccttattttctccataatcaatcacaaagtcatgtggattttgctcctaaatatgttaacttctttcctctctcactttatatcccttacatctaggtatttcagaccctcagtctctctcacatagactttggcaataaccttctaatatcagtcaacctgaccaataggccaccagtgcttcatgtagaatctggacaatgtagagcactgagaatgctcacactggtcatatatgtatgagttggtatgacatctagggaagttgaagacttacatagcctttggcccagcaatatacaccataatacattagagaaactctagcatgtgtacacagtgatatacacacaagaatgttcacaatgccattattttaatagcaaaattgtggaaacaacacaaatgtttatcaataacagaatggataagtgagccatggcatagtcatacaatgaaaaataatataatagtcaaaatgaatgatctgaagagatatcattattggcaatcttataaaagactgagttaaaaatgcaatttgtgaaaatttttaattatttgatattatttaatgcaaagttttagaacatgcaaacaactgtatatattatttatgtatatatgcaaattcagcaatagcatttaatcatgcctgggaatgataagtatcaaagtcagaaagtggttacccttgggaagagaggtatgtatcagcggtggggcacataggatgttgcagccatatctgtaatgtttctttgctttaaaaaatttgaatcaagcttggcaaagtgtgacatttgattaagcaggatagtgagtgcatatctgttacttatattgttctttataattttctctatgctaaagcattttgtaatttaaaaaacctgacagtgttactcccatgcttaaaatatgccagtggtcaaaccaaatccagcagcacatcaaaaagcttatccaccatgatcaagtgggcttcatccctgggatgcaaggctggttcaatataagcaaatcaataaatgtaatccagcatataaacagaaccaaagacaaaaaccacatgattatctcaatagatgcagaaaaggcctttgacaaaattcaacaactcttcatgccaaaaactctcaataaattaagtattgatgggacgtatctcaaaataataagagttatctatgaaaaacccacagccaatatcatactgaatgggcaaaaactggaagcattccctttgaaaactggcacaagacagggatgccctctctcaccactcctattcaacatggtgttggaagttctggccagggcaattaggcaggagaaggaaataaagggtattcaattaggaaaagaggaaatcaaattgtccctgtttgcagatgacatgtatatctagaaaaccccattgtctcagcccaaaatctccttaagctgataagcaacttcagcaaagtctcaggatataaaaatcaatgtacaaaaatcagaagcattcttatacaccaacaacagacaaacagagagccaaatcatgagtgaactcccattcacaattgcttcaaagagaataaaatacctaggaatccaacttacaagggacatgaaggaactcttcaaggagaactacaaaccactgctcaatgaaataaaagaggatacaaacaaatggaagaacattccatgctcatgggtaggaagaatcaatatcgtgaaaatggccatactgcccaaggtaatttatagattcaatgccatccccatcaagctaccaatgactttcttcacagaattggaaaaaactgctttaaagttcatatggcaccaaaaaagagcccgcatcaccaagtcaatcctaagccaaaagaacaaagctggaggcatcacactacctgacttcaaactatactacaaggctacagtaacccaaacagcatggtactggtaccaaaacagagatatagatcaatggaacagaacagagccctcagaaataacgccacatatctacaactctctgatctttgacaaacctgagaaaaacaagcaatggggaaaggattccctatttaataaatggtgctgggaaaactggctagccatatggagaaagctgaaactggatcccttccttacaccttatacaaaaattaattcaagatggattaaagacttaaatgttagacctaaaaccataaaaaccctagaagaaaacctaggcattaccattcaggacataggcatgggcaaggacttcatgtctaaaacaccaaaagcaatggcaacaaaagacaaaattgacaaaggggatctaattaaactgaagagcttctgcacagcaaaagaaactaccatcagagtgaacaggcaacctacaaaatgggagaaaattttcacaacctactcatctgacaaagggctaatatccagaatctacaatgaactcaaacaaatttacaagaaaaaaacaaacaaccccatcaaaaagtgggcaaaggacatgaacagacacttctcaaaagaagacatttatgcagccaaaaaacacatgaaaaaatgctcatcatcactggccatcagagaaatgcaaatcaaaaccacaatgagataccatctcacaccagttagaatggcaatcattaaaaagtcaggaaacaacaggtgctcgagaggatgtggagaaataggaacacttttacactgttagtgggactgtaaactagttcaaccattgtggaagtcagtgtggcgattcctcagggatctagaactagaaataccatttgacccagccatcccattactgggtatatacccaaaggactataaatcatgctgctataaagacacatgcatacgtatgtttattgtggcactattcacaatagcaaagacttggaaccaagccaaatgtccaacaatgatagactggattaagagaatgtggcacatatacaccatggaatactatgcagccataaaaaatgatgagttcatgtcctttgtagggacatggatgaaattggaaatcatcattctcagtaaactatcgcaaggacaaaaaccaaacaccgcatgttctcactcataggtgggaactgaacaatgaaaacacatggacacaggaaggggaacatcacactctggggactgttgtggggtggggggaggggggagggatagcattaggagatatacctaatgctaaatgatgagttaatgggtgcagcacaccagcatggcacacgtatacatatgtaactaacctgcacattgtgcacatgtaccctaaaacttaaagtataataataattaaaaaaaccaatagtttatgaaaccccccccaaaaaaaatatatgccagtggcctccagttgcccaccaggtagcatccacattctttaatggaaagcccttccttgcttcgaacttgccaactgggattggacatttgtagttgcatttctaagaactgttcccttttgtcaatggagcctgatttccacttggatatctgggtgatttagggaaactgacctcaaaacccaattctacatttcgaccatgtgaccttggcttaatcaattcacgcatctttttccctcacctcaggggatgatcatatgaactaagccagttgcaatagagtaaacctcatgttcctaatgagaaatccagaacaaaatgctttatttttcttcaattttttattaggtcatctcctgaatcaattaaaaaaaaaaccaacagtgacaacaaaactaaaaaatatgaagaagctgaaacatgaaagctctgccaactgcaatatgtagctgctaaggttgctgtatttattggaatcaagcaagtgttccagtaaagagcacagaagatgtgtctgggagcctttatgtgtaggtctgcaagtggtggatatcactactactcacacgccattggctagaactgagttgcatggatacacctaattgtaaaagaggctgggaaatagagactattgtgcccagaaagaagagaaaattcatttatggaagaggtagctagtctctcacagccatgaaaagagaagtgtttagctaattgaagtgaatagcagccatcttgggtccctaaggcaagttagactaatattgaagtggaaaccatgaggaaagcagtgatactgaaagtaaccgcatctttgagaacatgcattcatttcctacaacatgaatttattgaggacctaccttaatacaggcagcgtgctagacaccaagagaactgatgtcctcttccttcctgcctgcctggagcctgtattctggaggggacagagctagcagatcagacctaactggaaatctgctgtgccagtatatatttcagtgatgtgagccaatatatccccttgattgctcaaagtagtttggtcgatatattttgttgctttaaattgaacacattcttatgtacagcctctgtctcctcatctccaaccaagcaaaatagcttgttctctttatgcagggacacatgacatttcccacgtggctttgtgcatatctccacctcaatttaaaatgccttccaatccctgctcaaagtcaaacagcttaataatggtagacatagaatttgacttattctaataataggtcttttaaacaatgccttcttctcttcattctttccttcttagagtgggtattctttctggtgcatcatgtaaaggaaggtaactacatgcatgtaatgatgagaatatttatatgtatttatgattatcacaaaaaaacaaagattctaccattcaagaggaacatttattttatttttttatttgagaaaagtataattttatttatttatttatttgtgcaaatttatggggtacttgagaaaatgtgttacatgtatataatgtgtagtgatccaatcaggatactaagggtgtccatcacctgagtgtattacatttttgttaagtataatcatcctactccaggagaacattttaaaaactgttctgtagagatactactcaaattaagttctcagtcctgaaacatcagatcagctaggaatctgacaaaaatgcaagttctcagatgacagatgagaccacttcaatcagaatttctggagtggagcccacacatttgtatttttgcaacctttccaatgatacttatgtacatgctcaagcttgaaaaccactttcctaggacattagttcctcgacaagatttgtgagtaaccttgtttcatgaaaaagtgtttaggagatactgattcaataaaaactaatcaggcttttattgtttgcagggctttcaaaacttgcaataggccactgtgcattgttaatttctaagaggaagatgcttatgtcctcaatgaatatcttcccaccatgaagtactcttcttcccccactttttaaaacaattactaacacctggcagaagtaggcagacagcttacagcttagaaaaagttggcctaagataatggctaattttcatacattatttatttgtcatcatgcttatctttctctctctaaattgtatatttcatctctgtgatcacagattgagcctcatatttcatatctgcccctggcctaatggttgtttacagaatgagctcaatgaatattgttaagtgagtaggatttaatttatttgataaatagataaccttaagttttaaacggtggatttcacatgaggacatttacttactattgttgagctgtaattaatttttaatactgtttaggtactcataataaagaacaggatatttggagaaggaagacagtatcattcctggttcttagtcttaccagcttattgatcatgagtatataacctctctgtggctcagtgcctttctctgtaaaatgggaacacagtgatgttcacctcacaggattgatttgtaaaagggctggataaggttatgagaatgttttgcaaagtgatatcgaaagattaattgcaaacttcatttgaatcttaaattgtttgagataggtcatgctatgaatcaactatgaagtgcagatattgtcaagattcaatatttctttcccaagagctgagaggaggggctgcttgtttgtttgtttctttctttttagaaacatgccaggacaggctcattttcgggttttcctctcacttgctcatcttactttttctttagtttctctattcattaggatacagtactgtaaagctttatggcatttttattttgtgggagatgaatctgaataaagaattacagttaaatcattgctaagtttgatgaatgagcaccaaagaactcttcaagatgtcatttttaaagttttgtaaatgattggctttcagtggtttcctctaaggaattttaattttgaataatgcatagaaaaatgtgcgcacacacaaatcattcagtatccacctcgaagggaaatcaaagtgcctgtgaagtgaaactttacctttctatatcaccagcttcctgttagagcagactttttctttgctcaaagtctaagcattgaagaacttctttttagtaggtagatttttgtgtttttttgtttgtttttgagacggagtctcactctgtcgcccaggctgtggtgcagtggcacgatctcggcccactgcaagctctgcctcccgggttcacaccattctcctgcctcagcctcccaagtagctgggactacaggtgccagccaccacgcctggctaatttttttgcattttttagtagagatggggtttcatcgtgttagccaggatggtcttgatctcctgacctcatgatccacccgccttggcctgccagagtgctggaattacaggcgtgatttagtaggtagttttgagtagggagtatacatttaaaatgctgaaactcagttaaggaataatctaatactgtattcaactgaaactcagttgagaaatttctttccaataataaaggaaaatcaactgcagtaatgagggagatgattttgctgctaattacaacaaatatttactacagacctgttatgcaccaggaactgtgctaaatgttttatacatataactttatcttgtgctccaacaacttattacatagaaattgctattattcccattttctagattaataaattggtttagaggggtcgtataggtgaaacaactcactcaatatcacaagctgttatgtggtgaagtttgcatgatcagtacagggttctggtcatcccactcattgagtggtgctagtcaagatctggaagctcttctggtcttagtttctctagccgtgaagtgacaatgattaggtctaatcatagaacatgagagtacatgtgaaaaaatgcctttttaaagagtatgaaaaacttgagttgtaaaattttcttgtggataatttattattgcttttcttttttagataacactaacaaagttgaccttagaattggagtgcctgggttagaaccctgctggtaccacctgcttactggcatgcttctgatgtgagttcaggagaagacactggcaaggacagcaaagaacaggagaacactctagcttccctgatagcattcaaggtgctgtccaaagctgactgtgatggcaccctccagacagacagcgatgccacatgttcaagatggcagaatcactatcagcttcaattcctgaatgactgcagagcaaaatttcttacctgcaacatacactctattttcagcctccctggactgttacataatgatacataaaaatatttcttgtgttgaggcatcccaaatttgatttatttgtcatcacagtctatcctatgaatatatttctgatccaaataatgctagattcatgagagtttatagtccagagattttttttttttttttgagacagaatctgtctcccaggctggggtgcagtggcataatcttagctcactgcaacctctgcctcccgggctcaagtgatcctccctccttagcctccatgagtagctgggaccacaggtgtgcaccaccacaccccgctaattattttgtattttttgcagaggggggtttcaccacattgcccaagctggtctcaaactcctagacttaagggatctgcctgccttggcctcccaaaatgctgaaattacacttgttagccatagtaatagttcctagccatagtccggagatattttaaaaactttagtgtgagttatttctctttgctataattctctcatgcattttgaaaaccttttctgccatacagtggtttttgggaagtccttttggacagacatatcacaggatggagaaactaaaaagagagaatgcattaaagaaaaaatagtacttcgtatttcagaaattacaaaagggtgtcatgccccacagcatggtgggcagtcatttgtaacccacagaacattgagtttctaaaatttgagtgtttttaacttacgaaggccaactttactgatgaagtgattaccccaaatatgtgaaatccatatttcaagtacaatagtaggaaattggaaatggggctcttgagtttctgctttcaagtgaccctcagaaattcctccattcactgcagaggttctgtttctccttgctttgttctgactttacggcagaactaagctaatgagttagttactatgggttatcacttggatttgaagaaccatcatttctaggcattgctgctcagagtggggtctgccctcaggggaagctgtctaaccagagcctaacccactagggttttatcagaacctaactgacctgaggaaaagaaatactcaactccagcaagctccagccttccaaatggaggaggagaaaacccaactccagcccacttcagctatcttgtcccacataaaggggaagtggaagaaacctgagaagcaggcatgaagttcacatgtattccacaccatttcatattccatcgcattccattctacacaattccacatctccccggccacatcagcctgagagtaataaacagaccttgccatcgtatccccttttggagacacaagccaggaaggaagctcttcagtgcagctgtgaatagagaaatgctggctgaggttttggagacactgggtgatcattcccagtgaaatttttagatccctggggacatgagctgctttgtcttttccaaggtcaggataaacagaataacttcaggcttctctacccagaaagaacatgtggcataaatatcaactgcagaataaatatgattaatctggtacatggacgaagatgttttctaggagatgcttatcctgggatgagagctttcataagcattgatatttacatgactcttaccgtgtgtcaggaactgttctaattgttttacctatgtcaattcatttgatagtcacaaccactgaaaggagtaagtactcttattacttccattttacagatagggaaactgaggtatagagaagtcaagtgacttgcccgaggttattaaactacttaatgtcaacaccaggatttgaacccagatcatttgtttctgaagtacatgctcacaatcactgcattactgatacactgttttgtctttgcatacttaagtggtcataacttagtctgaaacactttgtgagagcaggaagcaaactgtccccttattaggtggaccagtatagtgataatacaaaagtgtattgcatttgaattacttgctaatatcttctaattgaggcaattttgaacaggaatatacatatctagcctctatttttctagctagaagttctgaaatccctgggcttaatattgtatggcaacaattggctggagttgagttgctgccactctcttttaactgagccatgctctctctagtttgctacaggccccaccactccctattgcctctccaataccaggtcatttggcatcttactcagctcatttcatgcacatgtgttccacaattggtaacataaccctaaaagtatttgagtttgtggcttctgctcttgtgacagaagacttttctcgaattccaaggtcaacatataccatattgactctgggccacattttttaatgtgctcaagctcagtttttctccttaaaaaatgaggggttaaaaataacacttcgtagttgcctcatctgtggattggaagaatgaatgcctgtcatttctagagttgtagtaagggccagttggggcagtgcctgtgaatatacgcaatgggccatcaagcaatctcagggcttcaggcaatgctggggtttataaagcactttatgttttaagttcacttttatttctaaagtctcattgactgctctgaaaatctctcaagtaaagtgggcactaaaggctttatactctctccaattataccttccattgtatagatttggaagctggggtccaaatgtgttaaatgacttgcctaacatggtccattactgtaagtacagaaacggaatgagaccccaaatctgcttcatggagggacactcttctaagatactgtgatgcttcttcccaagtaattccgtcttcagacttcaaggtctcaattcgaatgacaattcaatataggactttcataatcttaaaagcaacctgacagtcattacagtgggctgtgagaaatactaacgcagagccacatactctgggcttcatgctaggttttgccactcactatctttgctgaaaaagttttggaagccctcctaagcaggtgccagacccttttttggccagaggacaggatcttacgctgttgcccaggctggagtgcaatagtgtcatcatggctcactgtagcctggaactgctgggctcaagggatcctcccagctcagcctcccaagtagctaggagtaaaggtacatgccatcatgcctagctttttttttttttaatttttggagagataagttcccatcatgttgcccaggctggtctctaactcttggactcaagtggtcctcccacatcagcctctcaaagtgctgagattgcaggtgttagccactgcacttggcctggccccagtattctttgggatctggagtttgctgttgaatgagaaggcaagatgaaattccatgtagtcagactcctacgctgctgttctaagcagggttgggcctgattagtaggttatagatgatgtttttctgtggtgctatctggacctagtgctctttggcatctgggaaggtatggcctttaaaaagcaaactgccatgagaactgctctaccccaaattttggttcacagccttcatttgattatgtattggggcaaaaatagtttagccatgtgaacctgtttgtaaactggtgagtttctattgctatttcatagctaaagttttgaggtaaatgctattggatctttgtgtctgtgtgtgtatacatatttagattttttttttttttttttttttggttctttgaaacattgctgattctttttgttttgtttttcagagtctggagaacactttttcttttgagctgtttacaacctttagcaattgagtagagtgtactcttgtcaatagaatttgaagcacatttctctctctgcctgatttctgtagaatttgtaaactatttgtgaatattcttaatttatggcaatatggttgcttacataagttcaataataatctgttttcttttacaatgagacacagttggaggaactggttattttcccagggctttgactgaaatggccttgtgaatggttccaggaaagccaattttggagaccctatgtggatgatgatgcttgctgtactttctgtgggtaatcgggccaagtatatgggactgaagctaattttgcagggcaacatagagagacttgagttccagggggaaagttttaggatggagagaagccctcactggactgtgatgtggggtgagaaaatggaggtccagagagaaaagtgactcacccaagctgaaaacagttgcaggaagaaaagccaggacagaactgggctgttagaaaccttggtagtgcacttttgatttcactcttaaaatgctcaaatgtctttttctgacaacatgtagggagacctgaggttggacctaaagacaccatatattggattcagtgcttcacacaagttaatctaaattatgaaggagtacactaaagatgaggaactgagtcaccgcaatgccaaatgtaacacgctgtgggtcaaaatttggatggttgcgttggaaattattctgaagccacagacagggctaaaaggagtctagaatgtcctactagctgttggaacctgctggagttctaaaggcatgcgatgtcctgaatgtgcttgaccaatggacacagcaggtatttgaagagttttgatgcttcctttctgttttcagtgctatttttttgctttatttcttttaaaaaactttccccacagaggctatttattacacagcagcagccagcatgcaccaagcagaactgcttttcataataaacctatttatcttctggcatgtcagaaatttctcaaattgatacttataataaatatgattaataacacattgtatttcagttgttgccgtaaaataattgggaataaatatcgtattgtgatgttgacagtgtcaggttggttggtgccaaacagcgggagcagccaactgtatttcattaacagccacattgctacaatgctattaagtttttcataatctcttctgctcaaggttgatcattcattctgttagacaaattcttgcaacaatcacactggaagtaaatctgccagcgacaatctcagcaagatgggttctcac-5'<128128655>(seqidno:1)>ccat1_jax_2转录物序列;基因组位置:chr8:128128655-128232653链:-<128232653>3'-aaggatttaatggcaagatgccattatagacaagaacaggattcagacactttcgtgttatgtgttcttgtccaaatactgtgtcactctgcactggcatcccaatcccaccaacacctttagcaggaacttcctgtttcaataacatttctcaatactctacctgattgcttagaatccatgagacagccatattctccatgtctaggtccctatcttatttttgttgagatggtgttaagactttatttgtgaggcttctggggaagaggagtaaggtattgatcccactgactggataatttgggtctcaaaatggataataaataagcattacatattttgaccacttccttggaggagaacttcttggaatgtgcaccatgtccgctgcactttttttgcacagatatctaagttggagaaacatactactagataaatcaatttgttctcttagtactcatgatatggttcctgggaacttctgattcaccaaattaatcttggccaggtacatacctggcaggaatccaaaaattccccaagtctccttgaagttcaggatcatcattcttaataaatacaccgagggaaaaaccatggagagtttgtcccagatgctgtgaatctggcccggggtacatgaagaagtccttaattgcagtcatttacatggtagattctctataatcatttaatttgctataggtctatgatttttagtccttcttctctaaatgattgaacatgtataattcccatttcaatcatattacctggatgaacaaaagtaacgctagactcattcatgcattctggttgccaaggaaaaggaaaaaaaaacaaaacaatcaacaggatgtttaaactgtcttagggcaacttcaggccatagtcactggtgttcttgcagactatgagatattttacattctgataagggataaaaattcgtgcctcacatggctcccatcacactaagatcttgcaacaataacactactgattcagacattaatcttaagtatccagggagccctaaaacattgtatcccactagcaaggaccatggtaattgccacgtaaatcccctccattatgtggcccttattatgaccagccagccaaggcttgcctttaaatcataccaattgaaccgagccttgtagaaacactatcacctacgcatacctctgcttcttttcattaacctgctatcctctttacaaatgggattcttcacccactcccttcttctagattagcaatgccctgttaagtaaacgaacacgaaattcaaagggaaacaggagcaatcatcattaccagctgccgtgttaagcattgcgaaaacgctcacgattcacagaaaaatccatgctgttctttgaaggcattcaagccttaatagctagctggatgaatgtttaacttctaggccaggcactactctgtcccaacaataagccctgtacattgggaaaggtgccgagacatgaactttggtcttctctgcaatccatctggagcattcactgacaacatcgactttgaagttgcactgacctggccagccctgccacttaccaggttgggctctgtatggctaagcgttttctcctaaaatcccttgaaaactgtgagaagaccataagaagatcatatctttaattctatttcacaagtcacacaatattccaatcaaatacagatggttgagaaaagtcatccatcttccctccccaccctcccacagcccctcaaccactgccctgaaacttatatgctgttatccgcagctccatctggagcatcacagctactgtcaaccctgacgctctttctgaaaaaacaccggatggacatcagaactatttctttaaggatgttactgagccacacaggaaaacttgccttatgattttgaatgcacggatctgatttgactaaacatgataactagagaatcacccaatctactcccattttcaactctaaatcatcagagtgtctcaaatccaaagcacacacagaccagcctggccaacacggtgaaactccacccctactaaaagtataaaaattatccaggtgtggtggcgggcgcctgtaatccaagctacttgggagtctggaggcaggagaatcccttgaacctgggagatggaggttgcagtgagcagagatcacaccaccgcactctagcctgggccacaaatcaacaacaacaacaacaacaaaaaacaaagcgcacacagagactgaggtcctctttggcattgagaagatggctatgcaagtcccaactagcaagtgcaaacttcccagcttcacttctgccagtgtcccttcaccccttctcaaccccactgggaggcaggagggtgcttgacaataacagccttggcatcactctgccagggtgtaataggaactgttacaattctgagattctgtgtaagcactggcctttctgcctagaatgccttctcctctcttttttaactgcatgctcctatttatctttcaaagcccggaaaaaataacactgcacacgggaaatgctcccttcctactgcagtcatttagatgactctatgccattccattcatttctctttcctaccacagaagtgctttgagattttggagtcagactgcttgaacttgaatcctggccctctcatcagagacttgacttattttaggcaagttatataaccaattttacctcagttccttacccataaaatgggtctaatgagagtacctaccacacagaattttgatgaaaactgaatgagatgaaggcctttaaggcagtggtccccaaccctggggacacagacaggtaccattttgtggcctgttaggaactgggccacacagcaggaggtgagcagtgggtgagtgagatcagcgttatttacagctgctccccattgctcaccttactgcctgagctccacctcctgtcagatcagcagtggcattaaattctcatagcagcacaaaccctgtcatgaactgcacatgcgagggatctaggttgtgcgctccttatgagaatctaatgcctaatgacctgtcaccgtctcccatcacccctagatgggagtgtctagttgcaggaaacaagctcagggcttccactgattctacattatggtgagttgtataattatttcattatataatacaatgtaataataatagaaacacagtgcacaacaaatgtaatgtgcttgaatcatccccaaaccatcccagtccacggtcttccacattttgtcttttcacaaaattgtcttccacaaaactggtccctggtgccaaaaaggcttgggaccactgctttaaagcctttgcatagtgcttagaattgagggggaaaaaaaaaacaaaaacaatgtagctagttgctacaatcactatattggtgagtttcaaaaggaaaagaattctgtcccatttatgcttgagccttgagttgctaaccaagcctgacacaaaattactgttgaagggatgtgtgagtcctaattgaaatgaggcctcttaagggaattgtggaccaaaccccaagcaggcagaaagccgtatcttaattattgcaagtatttcaggcaaggtgtggatggccatttgaattcaagcagactaggacctgggatgagaaagaaggtgtgtacgtgacttgatctttgaactttagctcaccatctggaagaaggctgagtattctctgcactcacatagtagctaatgcctactccccagccacccacaattctttctgtaggaaggctcgctagaatactttgtgatattggatattagttccatattctactgtgtatcttagttcaaccaaattgtaatcatctgatatttatttcttttaatataaatataagtatattaagtcttggcatgcttgctcagtctctctctctctcccattcctccccgctcccctctctctttcccaacaggcttggaaagcaggcatcaccatgcctatttaacagttggggtcccttggccaccaggtgctggagtaggaatctgagcccggacatgcctgatctgtaaattttgtgttttccccactgtgctgggcagatcacagctatcagcgccaaattcatagaaggggcgccccctgtggtcaattgagggatttgtgtttgaggtagatctcaagaaggaatgggtggggaacttagcctaggacagagcagaaaggagccctcactccccaagcaccaacggcctcagtccttcctgctgactccagcctctagctctcacccagactatctgcatccttctctccaccacgctcctttggaacctgcgtaaaacacagattaaaggaattccgccttacttccctttccgcattatgaccaaatggttttacactatcattgaacagtttagtacaaaacatgccaccttttaatctattcattcatttaacaaatactttggagtgtttaccatgtgccaagtgctgttctaatagacataagctgtgaggttatgcttatctgattctcacagcaacagctttcgagatatgaattggtatactcatttgacagatgaggaaattgaattcatgtagtgagaaggaagagctgcaattcagggttactggtttctcctgcactaagcactgagccacactagaagagaaggcatgaggaagacaaaagtgaggctgtggcttgccttttcacttcttgtgtcctgtttaagaaatatctgctatcccaaggtaagatactatactttttttaacatgttattttgttttacctttcacatttggtgtatactacatttgtaattaatttgtccatattatatgacatatagccaagatttattttttaccatacagatactcaatattgccatttacgtagaacatcgttctcttcctactcaattgccttggcacctttgtaataaatcagatgatcgtgtatgtgtagttcagtttctggactctgtcttctgtttctttagtctatttgcctattcttgtaccaatatgcactgtgttaataatcgtagctttgtagtaggtctcgaaatctgacagtgtaattcttttagtttctttcttctgcaaattttctttagctgttttacgtcctttgcatttgtatataaacttcagaatcagcttgtctattccaaaaacaacaacaacaaatgaaagtttcagaaactttaactgaaattgtattgaatctgtagacaattttggagtaaattgcatcttagcaatgtagagtctttcgaaccataaccatggtaagtctctccatttgcttaaatcttctttaatttatttcaacaatggcttccaatttccagcgggagctcttggaaactacaatttacatgaacttctaatttgatatttttcggtgtcattataaacattgttgttttaaaagttgtcttcaattttttgttgtcaggcacagaaatacaattattgataacatttatatataaactgtatccagtgatcttgctagattcactgatgagtctgatgttgtagattctttgggattttctccgtacataatcatatcctttctgaataagatagttttacttcttcatttttaatatctatgccttttatttatttttcttttgaactttttgctgacttcattattcactctcatgtttttctctttcattagactatgactcctcgatggtagcaatttgtagtaatcaaatttttgtattttattttagcatctggcatctttcttgacatatgtagcagttgctttttgacagcttgcttcttcagtgaatgaataaattaataaagagaaatgtgatgttcagtgatccattttgcaggtaagaaaactgaggcagacagaggatgttagcaagcaagaggccttggcctacaatttagatcactggactcttactccagatgcaatctgcagaacccacatacttttaattagtccctttgtctatgttctgccactgtcacttctaaggaaggtgtgtcatcccaaatggggtagtatcttattggtagacctaaatctgctgtgttcgccatctcacctacatgagtatctatgtgtagcattctgcatattcatcttttccaccttctggaggttttgtctttttataggcagcatgtgaataacaatggggccaaactggggaccagaaggggccattttctagttctgaacatagataagcatcactaacttttccctcctggcagtaatggcctcaaaagttccaacttaggagaaaaaggcaaaaccgtctgccaaagtgtgtgaaaagttagagcaaaccttggttttaccaagaacctgtgtccctcttatggaaattcacactttcacacttttagacaaatattaaatgtgtgacattctattacgtacagtgcctggcacatctagacacgcagcacactttagcccccttctttcttcttctaactccaagttctaaactagaaaaagccccacttgagtctgagatttgctttttgaactagtttatttcagattgtaatcatgctatctagggttgtgacagtgtttgctatttctagggcactttgacctgattctttttgcacaggaaacttgttctacccttttgcccacttcactgaagtgaggactgagacagagaaggattaagtcactttttatttaacaaatatttgttccacattcctcagcatttattaaatactggtgcataatgatggaataaattttataccgtaaggataaaccagtattctggactaagccaacgtgggagaccctaggaggcctgtttgagaaagtgacatttaaattgagacttggcggtggctgtggctacatatctagtaagtggctgggttgggatttgaactcatgcctgcttagctctaaagatgatgcttttggctttgtactctgctctctctctagacaaactctggtccaaaatcgttaaagctaacatttatccctgcccaactggaattgtcatgttatgacaaatggctctgtggtctcagatgcccagcagacccattagtggaattctatgtgctacagacctgggcaaaatgccagagccttatacacccatcacatttcgtctggcaaaggtcttcaacaaagagaagtaattacagcaatgaaaagcaacaggtccagcaacaccataagaacaaaataattaatttccctaaaatagaagaaaccatttatagagtaagagccgatacaatcaataattggaagaaatagaagaggctttagtattctagccttctttatttgtagatgtaaatgtcgagcctcagagaagttatatatctaattagtgtcactcaggtagataacaacagaattaggattagaacttaattcttatgactcccagagcagggaaaagacaggatgaagtcccaaaacattgcgtgtgaactcacatctgactctgaattgagagtctgctatttactccctatgtgaccagaatccttcagagcccatgagattccctgtcatagagtagattttgatccacactaggcatttttacctcttgctctttgagttggtgccccatgtttactcagaaatattccaaaggtgttacatctattggttttacatgttgagcacagatcattataagacaaattgaaatgaaaacatcaacaagtctcattcattgtctaacttacgctgagcaatatttaataactagaatatcaagagagtccaaagtgtttgcccatcccctcaagaccaatgtaatggaattttactcttatcacctgctcagggtagtggcaattcaggatacagaggacagaaataaagaatcatgacacacaatccacagaattcacagatgccaaacatctacccttctctgtcaccacacattggactcacatggtggaaataggcaacacaagcagagaggtggcttaacctttcataatttttcaacgacctatgggaagagagttttcttggttcaaatcccagcttagccacacagagtgtggtaatattgggcaagtcaaccaagctctctgtgcttcagtttcctcattattaaaatgggggaaataatagtgcctgcatcagagggttgttgtgagaactaaacgaaataatttatctgagctttagaacccacaccatattagttaaaaattcatgcattttcttttattatatttctctaccttagactgcaaactctaagaggaaaggccggactgttatattcataaagcattacaggaacagtaattagaactaggagcttttcaatggcctgcctgaaatctgaaaaataggtatattatttgaaattttgaaaaaatcaaataattaaaaattaatagatgttaataaaatatctgtaatatgtaatatcaaggtcaactcaactcttaattgtttatataaaatatagtgaagtttaaattgcaaaatcttacagaaaatgtgctattaaaactcaaaagtataattctttctaatatgtatatatatgtatatagttttattttaagttcaggggcacatgtgcaagtttgttacctagggaaactcacgtcacaggactttgttatacagatactttcatcacccaggtattaagcctagtgctcattagttatttttcctgatcctctccctcctcccaacctccaccctcaggtaggcctcagtatctcttgttcccctctatgtgttcatgagttcttatcatttagctcccacttacaagtgagaacatgtggcatttggttttctgttcctgccttagtttgctaaggaaagcactgtggcaatttctcaaaggactaaaaacggaattaccattcaacccagcaatcccattactagttacgtactcaaatatttttaaggcaaaacaaagctgcaaccagaacacctggactccctgaaaccccttccactgatgttgttgttgttgtttctttttccccagcttctcaggccaaaatactggatcatcttgggcactgttctctcctgcccaccctttcccatatgcagagtgttgtcacttctctctgcttccactgctaactccctggtccaagccgctgcaccacttttcgtgattattgccacagtctcctcactggttccctgcccccactcttgctctgaactatccagttaaaacctgaattagatcatctcatcctcatctcagagctttctcgtggctcctctgccctctcaggaaaaaatctaaattctagatgacctaaaaatcccttgtctcttactgtttatctgacctcatttactaccacctttttctttgatcattctgttccagccacactggcctccttaccactcctcaaatatgccaagcacagcccccaccccccagggctttgaactggctgatcccccttcctggaatgccttaccccaaatatcaacttagccaactccctcctctcctccaagtgtctgtttaaacatggcttcagtaggagctgtcttaacatcctattaatattgtaattcctctcatgacactttacacccccttccctgatatgctttccatataccatgcaatatctgctgagataatatataattcacttattttctttattgtcatttcaaagagggcggtgtgttctgtgttttatttagtgccaaaatacttgctgatgaagagagttcctgccacatagtaggtgctcaatatgtgcttgttgaataaatgtgtcaatgtttgatgtacagaccttttattatgtttgatttgctgccagtgctgcctccaaacacaggagtgcttcatgagatgttcacaaaagctcttaaaatattccacaaaaatcttaaaatatttcatgagttttctttcctgtatttttatagcagcatctggaatttagcctgcataggaccctctgtaagctgaccctgtttatctattcagctttacttctcccctctctccactttgtattttattctctactacttccaactgattgtaatttgaccagactccagactatcttatgcctctttgcttttgtttacctgttacttctctctggaattccctgccccttcttaatttttctggccaattctcactctctaggactcagaggtttctcctcaggagacttccatgagtctcatgttgagttaggtgaccccaatcctctgttcttcatagtcattcgcgcatttatctagctcagcatttgccatactacattgaaattatttccttatgtgcccatcactccccgtagattgcaaactcctagagaagggctcaacagtgagtgctgaggctgcacagaggaggaaggcagcacaatgatggaaggcttcctaaagaggtatgttccaagagcccccacttcctttcatgggagactcatgctgttacacctagactatctagggatacatctaatgtagtcgtggaaagaacagaggacttgagtactagactgacgtgattttgaatcctggctccctattgaccagatgtgtgtcttgaacaagtccctgagcctcagtgtcttcatctgcacagtgaggataatgataccacactgcatatatgaggtatccggcacatgtaaatgtccactacatgctgatttcttcacccgctactcacccctgggaaagaagtagactcacctactcttggtacccattcattccccctcagttggaagcatgaggtgtgcagctgcctgacctgggggaagggctgcaagcagtaggtgttgtcagatgtggtggagcttgttgacttcctcccaggggcccagctaacaacctgcctctgttccttgatagtcaagttcaactttcacttcttagcaccacaggaagttgactgaacattaactgaagtctctctcaaacaggagacatctttgccaggtccctgtacttcctagcctcattcctgctctccctagtgagcaggctgccctcccttctcgccccagcaccactgataggcaagggtactcagaactactaccttgtgggccatgtcatgtgccaggagctgcacccaggactttaatacagtagttggctcccactgaatgttcattgttaccccaggataaaaaggggacactgtgatcattttctattttgctgtgatcaggcttggtgagctaaagtcacctaccttcccagtctctactaatagaagtcatggatcagtcctattggttcttctgttacaaggattcagaattcataatcatggagctgcatttacaggcagaagtttctttcatagttttctaagtgttccttttagcaacaatggagaaaatcaaagagggcaaaggtgaggggagaaaataacatttccctttctgtcctttgctcttgtagtcttttgctttagtttctttactatgactgtgagggtgaaactagtgatcagagtggtccagaatgggtttgatgaatctgattctggtgacacaagatgaattgggtatatgtttccctaaagatagagagacaatataacatagtctttacattaatagactctggagccaatttttttaggttcactctctttcctttcatgtgtgttgattttcaacaaacatcttgcactcgaattccatctcactgttttattttcaaaaaatttaatttgagaaagttagctgtattaattttttctttttctaaaattcttttactaattgcaattatttccattgatgctattccattgaaaccattttaacatggactcaataacttcattgtatgttaatgtttaattttcatttctttaccttcttggttttctagctgtgtttaatgtggttgaccactcattctttgaagctctattcctctggctactacagtatgacacattttgtctccttcttcagtctctgtcttctccacagtcttctcttccttttatatacctttaaatattaatgtttcccagagatattttcttaactcacttctctactgattctaggtacttttcttgatccaactcttctgattttacccatctcgatgattctataatttgtattttctgttttgatctcttttcagtcttccagacctaaatatccaaatgcctgatggatagtgctttcttttttactaccaagccctcaaaggcactatgttcaaaaggaatttgtcatcagtctcacggcacataagcttcctcttgtgttcaatctggagacttgagagtcttcctgttcccttcttctccttattttctccataatcaatcacaaagtcatgtggattttgctcctaaatatgttaacttctttcctctctcactttatatcccttacatctaggtatttcagaccctcagtctctctcacatagactttggcaataaccttctaatatcagtcaacctgaccaataggccaccagtgcttcatgtagaatctggacaatgtagagcactgagaatgctcacactggtcatatatgtatgagttggtatgacatctagggaagttgaagacttacatagcctttggcccagcaatatacaccataatacattagagaaactctagcatgtgtacacagtgatatacacacaagaatgttcacaatgccattattttaatagcaaaattgtggaaacaacacaaatgtttatcaataacagaatggataagtgagccatggcatagtcatacaatgaaaaataatataatagtcaaaatgaatgatctgaagagatatcattattggcaatcttataaaagactgagttaaaaatgcaatttgtgaaaatttttaattatttgatattatttaatgcaaagttttagaacatgcaaacaactgtatatattatttatgtatatatgcaaattcagcaatagcatttaatcatgcctgggaatgataagtatcaaagtcagaaagtggttacccttgggaagagaggtatgtatcagcggtggggcacataggatgttgcagccatatctgtaatgtttctttgctttaaaaaatttgaatcaagcttggcaaagtgtgacatttgattaagcaggatagtgagtgcatatctgttacttatattgttctttataattttctctatgctaaagcattttgtaatttaaaaaacctgacagtgttactcccatgcttaaaatatgccagtggtcaaaccaaatccagcagcacatcaaaaagcttatccaccatgatcaagtgggcttcatccctgggatgcaaggctggttcaatataagcaaatcaataaatgtaatccagcatataaacagaaccaaagacaaaaaccacatgattatctcaatagatgcagaaaaggcctttgacaaaattcaacaactcttcatgccaaaaactctcaataaattaagtattgatgggacgtatctcaaaataataagagttatctatgaaaaacccacagccaatatcatactgaatgggcaaaaactggaagcattccctttgaaaactggcacaagacagggatgccctctctcaccactcctattcaacatggtgttggaagttctggccagggcaattaggcaggagaaggaaataaagggtattcaattaggaaaagaggaaatcaaattgtccctgtttgcagatgacatgtatatctagaaaaccccattgtctcagcccaaaatctccttaagctgataagcaacttcagcaaagtctcaggatataaaaatcaatgtacaaaaatcagaagcattcttatacaccaacaacagacaaacagagagccaaatcatgagtgaactcccattcacaattgcttcaaagagaataaaatacctaggaatccaacttacaagggacatgaaggaactcttcaaggagaactacaaaccactgctcaatgaaataaaagaggatacaaacaaatggaagaacattccatgctcatgggtaggaagaatcaatatcgtgaaaatggccatactgcccaaggtaatttatagattcaatgccatccccatcaagctaccaatgactttcttcacagaattggaaaaaactgctttaaagttcatatggcaccaaaaaagagcccgcatcaccaagtcaatcctaagccaaaagaacaaagctggaggcatcacactacctgacttcaaactatactacaaggctacagtaacccaaacagcatggtactggtaccaaaacagagatatagatcaatggaacagaacagagccctcagaaataacgccacatatctacaactctctgatctttgacaaacctgagaaaaacaagcaatggggaaaggattccctatttaataaatggtgctgggaaaactggctagccatatggagaaagctgaaactggatcccttccttacaccttatacaaaaattaattcaagatggattaaagacttaaatgttagacctaaaaccataaaaaccctagaagaaaacctaggcattaccattcaggacataggcatgggcaaggacttcatgtctaaaacaccaaaagcaatggcaacaaaagacaaaattgacaaaggggatctaattaaactgaagagcttctgcacagcaaaagaaactaccatcagagtgaacaggcaacctacaaaatgggagaaaattttcacaacctactcatctgacaaagggctaatatccagaatctacaatgaactcaaacaaatttacaagaaaaaaacaaacaaccccatcaaaaagtgggcaaaggacatgaacagacacttctcaaaagaagacatttatgcagccaaaaaacacatgaaaaaatgctcatcatcactggccatcagagaaatgcaaatcaaaaccacaatgagataccatctcacaccagttagaatggcaatcattaaaaagtcaggaaacaacaggtgctcgagaggatgtggagaaataggaacacttttacactgttagtgggactgtaaactagttcaaccattgtggaagtcagtgtggcgattcctcagggatctagaactagaaataccatttgacccagccatcccattactgggtatatacccaaaggactataaatcatgctgctataaagacacatgcatacgtatgtttattgtggcactattcacaatagcaaagacttggaaccaagccaaatgtccaacaatgatagactggattaagagaatgtggcacatatacaccatggaatactatgcagccataaaaaatgatgagttcatgtcctttgtagggacatggatgaaattggaaatcatcattctcagtaaactatcgcaaggacaaaaaccaaacaccgcatgttctcactcataggtgggaactgaacaatgaaaacacatggacacaggaaggggaacatcacactctggggactgttgtggggtggggggaggggggagggatagcattaggagatatacctaatgctaaatgatgagttaatgggtgcagcacaccagcatggcacacgtatacatatgtaactaacctgcacattgtgcacatgtaccctaaaacttaaagtataataataattaaaaaaaccaatagtttatgaaaccccccccaaaaaaaatatatgccagtggcctccagttgcccaccaggtagcatccacattctttaatggaaagcccttccttgcttcgaacttgccaactgggattggacatttgtagttgcatttctaagaactgttcccttttgtcaatggagcctgatttccacttggatatctgggtgatttagggaaactgacctcaaaacccaattctacatttcgaccatgtgaccttggcttaatcaattcacgcatctttttccctcacctcaggggatgatcatatgaactaagccagttgcaatagagtaaacctcatgttcctaatgagaaatccagaacaaaatgctttatttttcttcaattttttattaggtcatctcctgaatcaattaaaaaaaaaaccaacagtgacaacaaaactaaaaaatatgaagaagctgaaacatgaaagctctgccaactgcaatatgtagctgctaaggttgctgtatttattggaatcaagcaagtgttccagtaaagagcacagaagatgtgtctgggagcctttatgtgtaggtctgcaagtggtggatatcactactactcacacgccattggctagaactgagttgcatggatacacctaattgtaaaagaggctgggaaatagagactattgtgcccagaaagaagagaaaattcatttatggaagaggtagctagtctctcacagccatgaaaagagaagtgtttagctaattgaagtgaatagcagccatcttgggtccctaaggcaagttagactaatattgaagtggaaaccatgaggaaagcagtgatactgaaagtaaccgcatctttgagaacatgcattcatttcctacaacatgaatttattgaggacctaccttaatacaggcagcgtgctagacaccaagagaactgatgtcctcttccttcctgcctgcctggagcctgtattctggaggggacagagctagcagatcagacctaactggaaatctgctgtgccagtatatatttcagtgatgtgagccaatatatccccttgattgctcaaagtagtttggtcgatatattttgttgctttaaattgaacacattcttatgtacagcctctgtctcctcatctccaaccaagcaaaatagcttgttctctttatgcagggacacatgacatttcccacgtggctttgtgcatatctccacctcaatttaaaatgccttccaatccctgctcaaagtcaaacagcttaataatggtagacatagaatttgacttattctaataataggtcttttaaacaatgccttcttctcttcattctttccttcttagagtgggtattctttctggtgcatcatgtaaaggaaggtaactacatgcatgtaatgatgagaatatttatatgtatttatgattatcacaaaaaaacaaagattctaccattcaagaggaacatttattttatttttttatttgagaaaagtataattttatttatttatttatttgtgcaaatttatggggtacttgagaaaatgtgttacatgtatataatgtgtagtgatccaatcaggatactaagggtgtccatcacctgagtgtattacatttttgttaagtataatcatcctactccaggagaacattttaaaaactgttctgtagagatactactcaaattaagttctcagtcctgaaacatcagatcagctaggaatctgacaaaaatgcaagttctcagatgacagatgagaccacttcaatcagaatttctggagtggagcccacacatttgtatttttgcaacctttccaatgatacttatgtacatgctcaagcttgaaaaccactttcctaggacattagttcctcgacaagatttgtgagtaaccttgtttcatgaaaaagtgtttaggagatactgattcaataaaaactaatcaggcttttattgtttgcagggctttcaaaacttgcaataggccactgtgcattgttaatttctaagaggaagatgcttatgtcctcaatgaatatcttcccaccatgaagtactcttcttcccccactttttaaaacaattactaacacctggcagaagtaggcagacagcttacagcttagaaaaagttggcctaagataatggctaattttcatacattatttatttgtcatcatgcttatctttctctctctaaattgtatatttcatctctgtgatcacagattgagcctcatatttcatatctgcccctggcctaatggttgtttacagaatgagctcaatgaatattgttaagtgagtaggatttaatttatttgataaatagataaccttaagttttaaacggtggatttcacatgaggacatttacttactattgttgagctgtaattaatttttaatactgtttaggtactcataataaagaacaggatatttggagaaggaagacagtatcattcctggttcttagtcttaccagcttattgatcatgagtatataacctctctgtggctcagtgcctttctctgtaaaatgggaacacagtgatgttcacctcacaggattgatttgtaaaagggctggataaggttatgagaatgttttgcaaagtgatatcgaaagattaattgcaaacttcatttgaatcttaaattgtttgagataggtcatgctatgaatcaactatgaagtgcagatattgtcaagattcaatatttctttcccaagagctgagaggaggggctgcttgtttgtttgtttctttctttttagaaacatgccaggacaggctcattttcgggttttcctctcacttgctcatcttactttttctttagtttctctattcattaggatacagtactgtaaagctttatggcatttttattttgtgggagatgaatctgaataaagaattacagttaaatcattgctaagtttgatgaatgagcaccaaagaactcttcaagatgtcatttttaaagttttgtaaatgattggctttcagtggtttcctctaaggaattttaattttgaataatgcatagaaaaatgtgcgcacacacaaatcattcagtatccacctcgaagggaaatcaaagtgcctgtgaagtgaaactttacctttctatatcaccagcttcctgttagagcagactttttctttgctcaaagtctaagcattgaagaacttctttttagtaggtagatttttgtgtttttttgtttgtttttgagacggagtctcactctgtcgcccaggctgtggtgcagtggcacgatctcggcccactgcaagctctgcctcccgggttcacaccattctcctgcctcagcctcccaagtagctgggactacaggtgccagccaccacgcctggctaatttttttgcattttttagtagagatggggtttcatcgtgttagccaggatggtcttgatctcctgacctcatgatccacccgccttggcctgccagagtgctggaattacaggcgtgatttagtaggtagttttgagtagggagtatacatttaaaatgctgaaactcagttaaggaataatctaatactgtattcaactgaaactcagttgagaaatttctttccaataataaaggaaaatcaactgcagtaatgagggagatgattttgctgctaattacaacaaatatttactacagacctgttatgcaccaggaactgtgctaaatgttttatacatataactttatcttgtgctccaacaacttattacatagaaattgctattattcccattttctagattaataaattggtttagaggggtcgtataggtgaaacaactcactcaatatcacaagctgttatgtggtgaagtttgcatgatcagtacagggttctggtcatcccactcattgagtggtgctagtcaagatctggaagctcttctggtcttagtttctctagccgtgaagtgacaatgattaggtctaatcatagaacatgagagtacatgtgaaaaaatgcctttttaaagagtatgaaaaacttgagttgtaaaattttcttgtggataatttattattgcttttcttttttagataacactaacaaagttgaccttagaattggagtgcctgggttagaaccctgctggtaccacctgcttactggcatgcttctgatgtgagttcaggagaagacactggcaaggacagcaaagaacaggagaacactctagcttccctgatagcattcaaggtgctgtccaaagctgactgtgatggcaccctccagacagacagcgatgccacatgttcaagatggcagaatcactatcagcttcaattcctgaatgactgcagagcaaaatttcttacctgcaacatacactctattttcagcctccctggactgttacataatgatacataaaaatatttcttgtgttgaggcatcccaaatttgatttatttgtcatcacagtctatcctatgaatatatttctgatccaaataatgctagattcatgagagtttatagtccagagattttttttttttttttgagacagaatctgtctcccaggctggggtgcagtggcataatcttagctcactgcaacctctgcctcccgggctcaagtgatcctccctccttagcctccatgagtagctgggaccacaggtgtgcaccaccacaccccgctaattattttgtattttttgcagaggggggtttcaccacattgcccaagctggtctcaaactcctagacttaagggatctgcctgccttggcctcccaaaatgctgaaattacacttgttagccatagtaatagttcctagccatagtccggagatattttaaaaactttagtgtgagttatttctctttgctataattctctcatgcattttgaaaaccttttctgccatacagtggtttttgggaagtccttttggacagacatatcacaggatggagaaactaaaaagagagaatgcattaaagaaaaaatagtacttcgtatttcagaaattacaaaagggtgtcatgccccacagcatggtgggcagtcatttgtaacccacagaacattgagtttctaaaatttgagtgtttttaacttacgaaggccaactttactgatgaagtgattaccccaaatatgtgaaatccatatttcaagtacaatagtaggaaattggaaatggggctcttgagtttctgctttcaagtgaccctcagaaattcctccattcactgcagaggttctgtttctccttgctttgttctgactttacggcagaactaagctaatgagttagttactatgggttatcacttggatttgaagaaccatcatttctaggcattgctgctcagagtggggtctgccctcaggggaagctgtctaaccagagcctaacccactagggttttatcagaacctaactgacctgaggaaaagaaatactcaactccagcaagctccagccttccaaatggaggaggagaaaacccaactccagcccacttcagctatcttgtcccacataaaggggaagtggaagaaacctgagaagcaggcatgaagttcacatgtattccacaccatttcatattccatcgcattccattctacacaattccacatctccccggccacatcagcctgagagtaataaacagaccttgccatcgtatccccttttggagacacaagccaggaaggaagctcttcagtgcagctgtgaatagagaaatgctggctgaggttttggagacactgggtgatcattcccagtgaaatttttagatccctggggacatgagctgctttgtcttttccaaggtcaggataaacagaataacttcaggcttctctacccagaaagaacatgtggcataaatatcaactgcagaataaatatgattaatctggtacatggacgaagatgttttctaggagatgcttatcctgggatgagagctttcataagcattgatatttacatgactcttaccgtgtgtcaggaactgttctaattgttttacctatgtcaattcatttgatagtcacaaccactgaaaggagtaagtactcttattacttccattttacagatagggaaactgaggtatagagaagtcaagtgacttgcccgaggttattaaactacttaatgtcaacaccaggatttgaacccagatcatttgtttctgaagtacatgctcacaatcactgcattactgatacactgttttgtctttgcatacttaagtggtcataacttagtctgaaacactttgtgagagcaggaagcaaactgtccccttattaggtggaccagtatagtgataatacaaaagtgtattgcatttgaattacttgctaatatcttctaattgaggcaattttgaacaggaatatacatatctagcctctatttttctagctagaagttctgaaatccctgggcttaatattgtatggcaacaattggctggagttgagttgctgccactctcttttaactgagccatgctctctctagtttgctacaggccccaccactccctattgcctctccaataccaggtcatttggcatcttactcagctcatttcatgcacatgtgttccacaattggtaacataaccctaaaagtatttgagtttgtggcttctgctcttgtgacagaagacttttctcgaattccaaggtcaacatataccatattgactctgggccacattttttaatgtgctcaagctcagtttttctccttaaaaaatgaggggttaaaaataacacttcgtagttgcctcatctgtggattggaagaatgaatgcctgtcatttctagagttgtagtaagggccagttggggcagtgcctgtgaatatacgcaatgggccatcaagcaatctcagggcttcaggcaatgctggggtttataaagcactttatgttttaagttcacttttatttctaaagtctcattgactgctctgaaaatctctcaagtaaagtgggcactaaaggctttatactctctccaattataccttccattgtatagatttggaagctggggtccaaatgtgttaaatgacttgcctaacatggtccattactgtaagtacagaaacggaatgagaccccaaatctgcttcatggagggacactcttctaagatactgtgatgcttcttcccaagtaattccgtcttcagacttcaaggtctcaattcgaatgacaattcaatataggactttcataatcttaaaagcaacctgacagtcattacagtgggctgtgagaaatactaacgcagagccacatactctgggcttcatgctaggttttgccactcactatctttgctgaaaaagttttggaagccctcctaagcaggtgccagacccttttttggccagaggacaggatcttacgctgttgcccaggctggagtgcaatagtgtcatcatggctcactgtagcctggaactgctgggctcaagggatcctcccagctcagcctcccaagtagctaggagtaaaggtacatgccatcatgcctagctttttttttttttaatttttggagagataagttcccatcatgttgcccaggctggtctctaactcttggactcaagtggtcctcccacatcagcctctcaaagtgctgagattgcaggtgttagccactgcacttggcctggccccagtattctttgggatctggagtttgctgttgaatgagaaggcaagatgaaattccatgtagtcagactcctacgctgctgttctaagcagggttgggcctgattagtaggttatagatgatgtttttctgtggtgctatctggacctagtgctctttggcatctgggaaggtatggcctttaaaaagcaaactgccatgagaactgctctaccccaaattttggttcacagccttcatttgattatgtattggggcaaaaatagtttagccatgtgaacctgtttgtaaactggtgagtttctattgctatttcatagctaaagttttgaggtaaatgctattggatctttgtgtctgtgtgtgtatacatatttagattttttttttttttttttttttggttctttgaaacattgctgattctttttgttttgtttttcagagtctggagaacactttttcttttgagctgtttacaacctttagcaattgagtagagtgtactcttgtcaatagaatttgaagcacatttctctctctgcctgatttctgtagaatttgtaaactatttgtgaatattcttaatttatggcaatatggttgcttacataagttcaataataatctgttttcttttacaatgagacacagttggaggaactggttattttcccagggctttgactgaaatggccttgtgaatggttccaggaaagccaattttggagaccctatgtggatgatgatgcttgctgtactttctgtgggtaatcgggccaagtatatgggactgaagctaattttgcagggcaacatagagagacttgagttccagggggaaagttttaggatggagagaagccctcactggactgtgatgtggggtgagaaaatggaggtccagagagaaaagtgactcacccaagctgaaaacagttgcaggaagaaaagccaggacagaactgggctgttagaaaccttggtagtgcacttttgatttcactcttaaaatgctcaaatgtctttttctgacaacatgtagggagacctgaggttggacctaaagacaccatatattggattcagtgcttcacacaagttaatctaaattatgaaggagtacactaaagatgaggaactgagtcaccgcaatgccaaatgtaacacgctgtgggtcaaaatttggatggttgcgttggaaattattctgaagccacagacagggctaaaaggagtctagaatgtcctactagctgttggaacctgctggagttctaaaggcatgcgatgtcctgaatgtgcttgaccaatggacacagcaggtatttgaagagttttgatgcttcctttctgttttcagtgctatttttttgctttatttcttttaaaaaactttccccacagaggctatttattacacagcagcagccagcatgcaccaagcagaactgcttttcataataaacctatttatcttctggcatgtcagaaatttctcaaattgatacttataataaatatgattaataacacattgtatttcagttgttgccgtaaaataattgggaataaatatcgtattgtgatgttgacagtgtcaggttggttggtgccaaacagcgggagcagccaactgtatttcattaacagccacattgctacaatgctattaagtttttcataatctcttctgctcaaggttgatcattcattctgttagacaaattcttgcaacaatcacactggaagtaaatctgccagcgacaatctcagcaagatgggttctcac-5'<128128655>(seqidno:2)>ccat1_jax_3转录物序列;基因组位置:chr8:128152989-128231094链:-<128231094>3'-gttgcactgacctggccagccctgccacttaccaggttggggctttgactgaaatggccttgtgaatggttccaggaaagccaattttggagaccctatgtggatgatgatgcttgctgtactttctgtgggtaatcgggccaagtatatgggactgaagctaattttgcaggcaacatagagagacttgagttccagggggaaagttttaggatggagagaagccctcactggactgtgatgtgggtgagaaaatggaggtccagagagaaaagtgactcacccaagctgaaaacagttgcaggaagaaaagccaggacagaactgggctgttagaaaccttgatgaaggagtacactaaagatgaggaactgagtcaccgcaatgccaaatgtaacacgctgtgggtcaaaatttggatggttgcgttggaaattattctgaagccacagacagggctaaaag-5'<128152989>(seqidno:3)>ccat1_jax_4转录物序列;基因组位置:chr8:128160497-128232653链:-<128232653>3'-aaggatttaatggcaagatgccattatagacaagaacaggattcagacactttcgtgttatgtgttcttgtccaaatactgtgtcactctgcactggcatcccaatcccaccaacacctttagcaggaacttcctgtttcaataacatttctcaatactctacctgattgcttagaatccatgagacagccatattctccatgtctaggtccctatcttatttttgttgagatggtgttaagactttatttgtgaggcttctggggaagaggagtaaggtattgatcccactgactggataatttgggtctcaaaatggataataaataagcattacatattttgaccacttccttggaggagaacttcttggaatgtgcaccatgtccgctgcactttttttgcacagatatctaagttggagaaacatactactagataaatcaatttgttctcttagtactcatgatatggttcctgggaacttctgattcaccaaattaatcttggccaggtacatacctggcaggaatccaaaaattccccaagtctccttgaagttcaggatcatcattcttaataaatacaccgagggaaaaaccatggagagtttgtcccagatgctgtgaatctggcccggggtacatgaagaagtccttaattgcagtcatttacatggtagattctctataatcatttaatttgctataggtctatgatttttagtccttcttctctaaatgattgaacatgtataattcccatttcaatcatattacctggatgaacaaaagtaacgctagactcattcatgcattctggttgccaaggaaaaggaaaaaaaacaaaacaatcaacaggatgtttaaactgtcttagggcaacttcaggccatagtcactggtgttcttgcagactatgagatattttacattctgataagggataaaaattcgtgcctcacatggctcccatcacactaagatcttgcaacaataacactactgattcagacattaatcttaagtatccagggagccctaaaacattgtatcccactagcaaggaccatggtaattgccacgtaaatcccctccattatgtggcccttattatgaccagccagccaaggcttgcctttaaatcataccattgaaccgagccttgtagaaacactatcacctacgcatacctctgcttcttttcattaacctgctatcctctttacaaatgggattcttcacccactcccttcttctagattagcaatgccctgttaagtaaacgaacacgaaattcaaagggaaacaggagcaatcatcattaccagctgccgtgttaagcattgcgaaaacgctcacgattcacagaaaaatccatgctgttctttgaaggcattcaagccttaatagctagctggatgaatgtttaacttctaggccaggcactactctgtcccaacaataagccctgtacattgggaaaggtgccgagacatgaactttggtcttctctgcaatccatctggagcattcactgacaacatcgactttgaagttgcactgacctggccagccctgccacttaccaggttgggctgtggcttgccttttcacttcttgtgtcctgtttaagaaatatctgctatcccaaggtaagatactatactttttttaacatgttattttgttttacctttcacatttggtgtatactacatttgtaattaatttgtccatattatatgacatatagccaagatttattttttaccatacagatactcaatattgccatttacgtagaacatcgttctcttcctactcaattgccttggcacctttgtaataaatcagatgatcgtgtatgtgtagttcagtttctggactctgtcttctgtttctttagtctatttgcctattcttgtaccaatatgcactgtgttaataatcgtagctttgtagtaggtctcgaaatctgacagtgtaattcttttagtttctttcttctgcaaattttctttagctgttttacgtcctttgcatttgtatataaacttcagaatcagcttgtctattccaaaaacaacaacaacaaatgaaagtttcagaaactttaactgaaattgtattgaatctgtagacaattttggagtaaattgcatcttagcaatgtagagtctttcgaaccataaccatggtaagtctctccatttgcttaaatcttctttaatttatttcaacaatggcttccaatttccagcgggagctcttggaaactacaatttacatgaacttctaatttgatatttttcggtgtcattataaacattgttgttttaaaagttgtcttcaattttttgttgtcaggcacagaaatacaattattgataacatttatatataaactgtatccagtgatcttgctagattcactgatgagtctgatgttgtagattctttgggattttctccgtacataatcatatcctttctgaataagatagttttacttcttcatttttaatatctatgccttttatttatttttcttttgaactttttgctgacttcattattcactctcatgtttttctctttcattagactatgactcctcgatggtagcaatttgtagtaatcaaatttttgtattttattttagcatctggcatctttcttgacatatgtagcagttgctttttgacagcttgcttcttcagtgaatgaataaattaataaagagaaatgtgatgttcagtgatccattttgcaggtaagaaaactgaggcagacagaggatgttagcaagcaagaggccttggcctacaatttagatcactggactcttactccagatgcaatctgcagaacccacatacttttaattagtccctttgtctatgttctgccactgtcacttctaaggaaggtgtgtcatcccaaatggggtagtatcttattggtagacctaaatctgctgtgttcgccatctcacctacatgagtatctatgtgtagcattctgcatattcatcttttccaccttctggaggttttgtctttttataggcagcatgtgaataacaatggggccaaactggggaccagaaggggccattttctagttctgaacatagataagcatcactaacttttccctcctggcagtaatggcctcaaaagttccaacttaggagaaaaaggcaaaaccgtctgccaaagtgtgtgaaaagttagagcaaaccttggttttaccaagaacctgtgtccctcttatggaaattcacactttcacacttttagacaaatattaaatgtgtgacattctattacgtacagtgcctggcacatctagacacgcagcacactttagcccccttctttcttcttctaactccaagttctaaactagaaaaagccccacttgagtctgagatttgctttttgaactagtttatttcagattgtaatcatgctatctagggttgtgacagtgtttgctatttctagggcactttgacctgattctttttgcacaggaaacttgttctacccttttgcccacttcactgaagtgaggactgagacagagaaggattaagtcactttttatttaacaaatatttgttccacattcctcagcatttattaaatactggtgcataatgatggaataaattttataccgtaaggataaaccagtattctggactaagccaacgtgggagaccctaggaggcctgtttgagaaagtgacatttaaattgagacttggcggtggctgtggctacatatctagtaagtggctgggttgggatttgaactcatgcctgcttagctctaaagatgatgcttttggctttgtactctgctctctctctagacaaactctggtccaaaatcgttaaagctaacatttatccctgcccaactggaattgtcatgttatgacaaatggctctgtggtctcagatgcccagcagacccattagtggaattctatgtgctacagacctgggcaaaatgccagagccttatacacccatcacatttcgtctggcaaaggtcttcaacaaagagaagtaattacagcaatgaaaagcaacaggtccagcaacaccataagaacaaaataattaatttccctaaaatagaagaaaccatttatagagtaagagccgatacaatcaataattggaagaaatagaagaggctttagtattctagccttctttatttgtagatgtaaatgtcgagcctcagagaagttatatatctaattagtgtcactcaggtagataacaacagaattaggattagaacttaattcttatgactcccagagcagggaaaagacaggatgaagtcccaaaacattgcgtgtgaactcacatctgactctgaattgagagtctgctatttactccctatgtgaccagaatccttcagagcccatgagattccctgtcatagagtagattttgatccacactaggcatttttacctcttgctctttgagttggtgccccatgtttactcagaaatattccaaaggtgttacatctattggttttacatgttgagcacagatcattataagacaaattgaaatgaaaacatcaacaagtctcattcattgtctaacttacgctgagcaatatttaataactagaatatcaagagagtccaaagtgtttgcccatcccctcaagaccaatgtaatggaattttactcttatcacctgctcagggtagtggcaattcaggatacagaggacagaaataaagaatcatgacacacaatccacagaattcacagatgccaaacatctacccttctctgtcaccacacattggactcacatggtggaaataggcaacacaagcagagaggtggcttaacctttcataatttttcaacgacctatgggaagagagttttcttggttcaaatcccagcttagccacacagagtgtggtaatattgggcaagtcaaccaagctctctgtgcttcagtttcctcattattaaaatgggggaaataatagtgcctgcatcagagggttgttgtgagaactaaacgaaataatttatctgagctttagaacccacaccatattagttaaaaattcatgcattttcttttattatatttctctaccttagactgcaaactctaagaggaaaggccggactgttatattcataaagcattacaggaacagtaattagaactaggagcttttcaatggcctgcctgaaatctgaaaaataggtatattatttgaaattttgaaaaaatcaaataattaaaaattaatagatgttaataaaatatctgtaatatgtaatatcaaggtcaactcaactcttaattgtttatataaaatatagtgaagtttaaattgcaaaatcttacagaaaatgtgctattaaaactcaaaagtataattctttctaatatgtatatatatgtatatagttttattttaagttcaggggcacatgtgcaagtttgttacctagggaaactcacgtcacaggactttgttatacagatactttcatcacccaggtattaagcctagtgctcattagttatttttcctgatcctctccctcctcccaacctccaccctcaggtaggcctcagtatctcttgttcccctctatgtgttcatgagttcttatcatttagctcccacttacaagtgagaacatgtggcatttggttttctgttcctgccttagtttgctaaggaaagcactgtggcaatttctcaaaggactaaaaacggaattaccattcaacccagcaatcccattactagttacgtactcaaatatttttaaggcaaaacaaagctgcaaccagaacacctggactccctgaaaccccttccactgatgttgttgttgttgtttctttttccccagcttctcaggccaaaatactggatcatcttgggcactgttctctcctgcccaccctttcccatatgcagagtgttgtcacttctctctgcttccactgctaactccctggtccaagccgctgcaccacttttcgtgattattgccacagtctcctcactggttccctgcccccactcttgctctgaactatccagttaaaacctgaattagatcatctcatcctcatctcagagctttctcgtggctcctctgccctctcaggaaaaaatctaaattctagatgacctaaaaatcccttgtctcttactgtttatctgacctcatttactaccacctttttctttgatcattctgttccagccacactggcctccttaccactcctcaaatatgccaagcacagcccccaccccccagggctttgaactggctgatcccccttcctggaatgccttaccccaaatatcaacttagccaactccctcctctcctccaagtgtctgtttaaacatggcttcagtaggagctgtcttaacatcctattaatattgtaattcctctcatgacactttacacccccttccctgatatgctttccatataccatgcaatatctgctgagataatatataattcacttattttctttattgtcatttcaaagagggcggtgtgttctgtgttttatttagtgccaaaatacttgctgatgaagagagttcctgccacatagtaggtgctcaatatgtgcttgttgaataaatgtgtcaatgtttgatgtacagaccttttattatgtttgatttgctgccagtgctgcctccaaacacaggagtgcttcatgagatgttcacaaaagctcttaaaatattccacaaaaatcttaaaatatttcatgagttttctttcctgtatttttatagcagcatctggaatttagcctgcataggaccctctgtaagctgaccctgtttatctattcagctttacttctcccctctctccactttgtattttattctctactacttccaactgattgtaatttgaccagactccagactatcttatgcctctttgcttttgtttacctgttacttctctctggaattccctgccccttcttaatttttctggccaattctcactctctaggactcagaggtttctcctcaggagacttccatgagtctcatgttgagttaggtgaccccaatcctctgttcttcatagtcattcgcgcatttatctagctcagcatttgccatactacattgaaattatttccttatgtgcccatcactccccgtagattgcaaactcctagagaagggctcaacagtgagtgctgaggctgcacagaggaggaaggcagcacaatgatggaaggcttcctaaagaggctgaaaaagttttggaagccctcctaagcaggtgccagacccttttttggccagagacaggatcttacgctgttgcccaggctggagtgcaatagtgtcatcatggctcactgtagcctggaactgctgggctcaagggatcctcccagctcagcctcccaagtagctaggagtaaaggtacatgccatcatgcctagctttttttttttttaatttttggagagataagttcccatcatgttgcccaggctggtctctaactcttggactcaagtggtcctcccacatcagcctctcaaagtgctgagattgcaggtgttagccactgcacttggcctggccccagtattctttgggatctggagtttgctgttgaatgagaaggcaagatgaaattccatgtagtcagactcctacgctgctgttctaagcagggttgggcctgattagtaggttatagatgatgtttttctgtggtgctatctggacctagtgctctttggcatctgggaaggtatggcctttaaaaagcaaactgccatgagaactgctctaccccaaattttggttcacagccttcatttgattatgtattggggcaaaaatagtttagccatgtgaacctgtttgtaaactggtgagtttctattgctatttcatagctaaagttttgaggtaaatgctattggatctttgtgtctgtgtgtgtatacatatttaga-5'<128160497>(seqidno:4)>ccat1_jax_5转录物序列;基因组位置:chr8:128172634-128231094链:-<128231094>3'-gttgcactgacctggccagccctgccacttaccaggttggtgtattccacaccatttcatattccatcgcattccattctacacaattccacatctccccggccacatcagcctgagagtaataaacaaccttgccatcgtatccccttttggagacacaagccaggaaggaagctcttcagtgcagctgtgaatagagaaatgctggctgaggttttggagacactgggtgatcattcccagtgaaatttttagatccctggggacatgagctgctttgtcttttccaaggtcaggataaacagaataacttcaggcttctctacccagaaagaacatgtggcataaatatcaactgcagaataaatatgattaatctggtacatggacgaagatgttttctaggagatgcttatcctgggatgagagctttcataagcattgatatttacatgactcttaccgtgtgtcaggaactgttctaattgttttacctatgtcaattcatttgatagtcacaaccactgaaaggagtaagtactcttattacttccattttacagatagggaaactgaggtatagagaagtcaagtgacttgcccgaggttattaaactacttaatgtcaacaccaggatttgaacccagatcatttgtttctgaagtacatgctcacaatcactgcattactgatacactgttttgtctttgcatacttaagtggtcataacttagtctgaaacactttgtgagagcaggaagcaaactgtccccttattaggtggaccagtatagtgataatacaaaagtgtattgcatttgaattacttgctaatatcttctaattgaggcaattttgaacaggaatatacatatctagcctctatttttctagctagaagttctgaaatccctgggcttaatattgtatggcaacaattggctggagttgagttgctgccactctcttttaactgagccatgctctctctagtttgctacaggccccaccactccctattgcctctccaataccaggtcatttggcatcttactcagctcatttcatgcacatgtgttccacaattggtaacataaccctaaaagtatttgagtttgtggcttctgctcttgtgacagaagacttttctcgaattccaaggtcaacatataccatattgactctgggccacattttttaatgtgctcaagctcagtttttctccttaaaaaatgaggggttaaaaataacacttcgtagttgcctcatctgtggattggaagaatgaatgcctgtcatttctagagttgtagtaagggccagttggggcagtgcctgtgaatatacgcaatgggccatcaagcaatctcagggcttcaggcaatgctggggtttataaagcactttatgttttaagttcacttttatttctaaagtctcattgactgctctgaaaatctctcaagtaaagtgggcactaaaggctttatactctctccaattataccttccattgtatagatttggaagctggggtccaaatgtgttaaatgacttgcctaacatggtccattactgtaagtacagaaacggaatgagaccccaaatctgcttcatggagggacactcttctaagatactgtgatgcttcttcccaagtaattccgtcttcagacttcaaggtctcaattcgaatgacaattcaatataggactttcataatcttaaaagcaacctgacagtcattacagtgggctgtgagaaatactaacgcagagccacatactctgggcttcatgctaggttttgccactcactatctt-5'<128172634>(seqidno:5)>_ccat1_jax_6转录物序列;基因组位置:chr8:128197810-128240377链:-<128240377>3'-gcatgtggcaggcacagaaatatttactcattgactgaatatagcacatcgtaatgttgatttttttccaacataattttagagctaggcatattgtattctattacactagactatatatcattcttaaatagaaccagccttgctagataacacatgttggaggagaggcccttcttcttagccctcagtgtttccatctatggggaagaagttccaccatactaacattactatcgtctctccacctgctcactcacttctccccaagggaggggtgttcgatatggtttctgagcttggaaagaaaactcaggcatgtgtaacatggttccttcagtcccatgacccactgtccacaaatgggctgctcacagagtgcatgccttcacccttgttcctggccatgcaggaaattgtatgaaacagtcctagctgaagcctgagattttcctgcattgcctagtcctggtgggtatctgtctactcctggagtttggattggaaagtccacatgcctgaaggtataaacctattctacaaaggggtgttttctagaatgaaggtaatatttttatcttacatttgcagaaagagacagaacaatgttataggtgagtgcatggacaatgacctcaaacagctaagattcaaacccctgcgttgaatgattgaattgaaatgattcaatgagctaatgtacataaagcatccagaatgttgcctggcacaaggactgtattgtctgctagaccatttattcaaagtgggaggatgatgttctaaaagccaatgataaagctcatggcaatgcagggtatatctgatggcatggaatgctttaggatggccaagattgcccatcaaatgccaagtcaccgaagttattaatggctctcctactaggagcctgacatcatggtgagcatcgagaaaggaataacctaagctgaagacacgccttttcaggaggccaagttccacgttctgtgcatgcttttggcgaaagtcaggccaggcactactctgtcccaacaataagccctgtacattgggaaaggtgccgagacatgaactttggtcttctctgcaatccatctggagcattcactgacaacatcgacttgaagttgcactgacctggccagccctgccacttaccaggttgggctgtggcttgccttttcacttcttgtgtcctgtttaagaaatatctgctatcccaagattgcaaactcctagagaagggctcaacagtgagtgctgaggctgcacagaggaggaaggcagcacaatgatggaaggcttcctaaagaggtatgttccaagagcccccacttcctttcatgggagactcatgctgttacacctagactatctagggatacatctaatgtagtcgtggaaagaacagaggacttgagtactagactgacgtgattttgaatcctggctccctattgaccagatgtgtgtcttgaacaagtccctgagcctcagtgtcttcatctgcacagtgaggataatgataccacactgcatatatgaggtatccggcacatgtaaatgtccactacatgctgatttcttcacccgctactcacccctgggaaagaagtagactcacctactcttggtacccattcattccccctcagttggaagcatgaggtgtgcagctgcctgacctgggggaagggctgcaagcagtaggtgttgtcagatgtggtggagcttgttgacttcctcccaggggcccagctaacaacctgcctctgttccttgatagtcaagttcaactttcacttcttagcaccacaggaagttgactgaacattaactgaagtctctctcaaacaggagacatctttgccaggtccctgtacttcctagcctcattcctgctctccctagtgagcaggctgccctcccttctcgccccagcaccactgataggcaagggtactcagaactactaccttgtgggccatgtcatgtgccaggagctgcacccaggactttaatacagtagttggctcccactgaatgttcattgttaccccaggataaaaaggggacactgtgatcattttctattttgctgtgatcaggcttggtgagctaaagtcacctaccttcccagtctctactaatagaagtcatggatcagtcctattggttcttctgttacaaggattcagaattcataatcatggagctgcatttacaggcagaagtttctttcatagttttctaagtgttccttttagcaacaatggagaaaatcaaagagggcaaaggtgaggggagaaaataacatttccctttctgtcctttgctcttgtagtcttttgctttagtttctttactatgactgtgagggtgaaactagtgatcagagtggtccagaatgggtttgatgaatctgattctggtgacacaagatgaattgggtatatgtttccctaaagatagagagacaatataacatagtctttacattaatagactctggagccaatttttttaggttcactctctttcctttcatgtgtgttgattttcaacaaacatcttgcactcgaattccatctcactgttttattttcaaaaaatttaatttgagaaagttagctgtattaattttttctttttctaaaattcttttactaattgcaattatttccattgatgctattccattgaaaccattttaacatggactcaataacttcattgtatgttaatgtttaattttcatttctttaccttcttggttttctagctgtgtttaatgtggttgaccactcattctttgaagctctattcctctggctactacagtatgacacattttgtctccttcttcagtctctgtcttctccacagtcttctcttccttttatatacctttaaatattaatgtttcccagagatattttcttaactcacttctctactgattctaggtacttttcttgatccaactcttctgattttacccatctcgatgattctataatttgtattttctgttttgatctcttttcagtcttccagacctaaatatccaaatgcctgatggatagtgctttcttttttactaccaagccctcaaaggcactatgttcaaaaggaatttgtcatcagtctcacggcacataagcttcctcttgtgttcaatctggagacttgagagtcttcctgttcccttcttctccttattttctccataatcaatcacaaagtcatgtggattttgctcctaaatatgttaacttctttcctctctcactttatatcccttacatctaggtatttcagaccctcagtctctctcacatagactttggcaataaccttctaatatcagtcaacctgaccaataggccaccagtgcttcatgtagaatctggacaatgtagagcactgagaatgctcacactggtcatatatgtatgagttggtatgacatctagggaagttgaagacttacatagcctttggcccagcaatatacaccataatacattagagaaactctagcatgtgtacacagtgatatacacacaagaatgttcacaatgccattattttaatagcaaaattgtggaaacaacacaaatgtttatcaataacagaatggataagtgagccatggcatagtcatacaatgaaaaataatataatagtcaaaatgaatgatctgaagagatatcattattggcaatcttataaaagactgagttaaaaatgcaatttgtgaaaatttttaattatttgatattatttaatgcaaagttttagaacatgcaaacaactgtatatattatttatgtatatatgcaaattcagcaatagcatttaatcatgcctgggaatgataagtatcaaagtcagaaagtggttacccttgggaagagaggtatgtatcagcggtggggcacataggatgttgcagccatatctgtaatgtttctttgctttaaaaaatttgaatcaagcttggcaaagtgtgacatttgattaagcaggatagtgagtgcatatctgttacttatattgttctttataattttctctatgctaaagcattttgtaatttaaaaaacctgacagtgttactcccatgcttaaaatatgccagtggtcaaaccaaatccagcagcacatcaaaaagcttatccaccatgatcaagtgggcttcatccctgggatgcaaggctggttcaatataagcaaatcaataaatgtaatccagcatataaacagaaccaaagacaaaaaccacatgattatctcaatagatgcagaaaaggcctttgacaaaattcaacaactcttcatgccaaaaactctcaataaattaagtattgatgggacgtatctcaaaataataagagttatctatgaaaaacccacagccaatatcatactgaatgggcaaaaactggaagcattccctttgaaaactggcacaagacagggatgccctctctcaccactcctattcaacatggtgttggaagttctggccagggcaattaggcaggagaaggaaataaagggtattcaattaggaaaagaggaaatcaaattgtccctgtttgcagatgacatgtatatctagaaaaccccattgtctcagcccaaaatctccttaagctgataagcaacttcagcaaagtctcaggatataaaaatcaatgtacaaaaatcagaagcattcttatacaccaacaacagacaaacagagagccaaatcatgagtgaactcccattcacaattgcttcaaagagaataaaatacctaggaatccaacttacaagggacatgaaggaactcttcaaggagaactacaaaccactgctcaatgaaataaaagaggatacaaacaaatggaagaacattccatgctcatgggtaggaagaatcaatatcgtgaaaatggccatactgcccaaggtaatttatagattcaatgccatccccatcaagctaccaatgactttcttcacagaattggaaaaaactgctttaaagttcatatggcaccaaaaaagagcccgcatcaccaagtcaatcctaagccaaaagaacaaagctggaggcatcacactacctgacttcaaactatactacaaggctacagtaacccaaacagcatggtactggtaccaaaacagagatatagatcaatggaacagaacagagccctcagaaataacgccacatatctacaactctctgatctttgacaaacctgagaaaaacaagcaatggggaaaggattccctatttaataaatggtgctgggaaaactggctagccatatggagaaagctgaaactggatcccttccttacaccttatacaaaaattaattcaagatggattaaagacttaaatgttagacctaaaaccataaaaaccctagaagaaaacctaggcattaccattcaggacataggcatgggcaaggacttcatgtctaaaacaccaaaagcaatggcaacaaaagacaaaattgacaaaggggatctaattaaactgaagagcttctgcacagcaaaagaaactaccatcagagtgaacaggcaacctacaaaatgggagaaaattttcacaacctactcatctgacaaagggctaatatccagaatctacaatgaactcaaacaaatttacaagaaaaaaacaaacaaccccatcaaaaagtgggcaaaggacatgaacagacacttctcaaaagaagacatttatgcagccaaaaaacacatgaaaaaatgctcatcatcactggccatcagagaaatgcaaatcaaaaccacaatgagataccatctcacaccagttagaatggcaatcattaaaaagtcaggaaacaacaggtgctcgagaggatgtggagaaataggaacacttttacactgttagtgggactgtaaactagttcaaccattgtggaagtcagtgtggcgattcctcagggatctagaactagaaataccatttgacccagccatcccattactgggtatatacccaaaggactataaatcatgctgctataaagacacatgcatacgtatgtttattgtggcactattcacaatagcaaagacttggaaccaagccaaatgtccaacaatgatagactggattaagagaatgtggcacatatacaccatggaatactatgcagccataaaaaatgatgagttcatgtcctttgtagggacatggatgaaattggaaatcatcattctcagtaaactatcgcaaggacaaaaaccaaacaccgcatgttctcactcataggtgggaactgaacaatgaaaacacatggacacaggaaggggaacatcacactctggggactgttgtggggtggggggaggggggagggatagcattaggagatatacctaatgctaaatgatgagttaatgggtgcagcacaccagcatggcacacgtatacatatgtaactaacctgcacattgtgcacatgtaccctaaaacttaaagtataataataattaaaaaaaccaatagtttatgaaaccccccccaaaaaaaatatatgccagtggcctccagttgcccaccaggtagcatccacattctttaatggaaagcccttccttgcttcgaacttgccaactgggattggacatttgtagttgcatttctaagaactgttcccttttgtcaatggagcctgatttccacttggatatctgggtgatttagggaaactgacctcaaaacccaattctacatttcgaccatgtgaccttggcttaatcaattcacgcatctttttccctcacctcaggggatgatcatatgaactaagccagttgcaatagagtaaacctcatgttcctaatgagaaatccagaacaaaatgctttatttttcttcaattttttattaggtcatctcctgaatcaattaaaaaaaaaaccaacagtgacaacaaaactaaaaaatatgaagaagctgaaacatgaaagctctgccaactgcaatatgtagctgctaaggttgctgtatttattggaatcaagcaagtgttccagtaaagagcacagaagatgtgtctgggagcctttatgtgtaggtctgcaagtggtggatatcactactactcacacgccattggctagaactgagttgcatggatacacctaattgtaaaagaggctgggaaatagagactattgtgcccagaaagaagagaaaattcatttatggaagaggtagctagtctctcacagccatgaaaagagaagtgtttagctaattgaagtgaatagcagccatcttgggtccctaaggcaagttagactaatattgaagtggaaaccatgaggaaagcagtgatactgaaagtaaccgcatctttgagaacatgcattcatttcctacaacatgaatttattgaggacctaccttaatacaggcagcgtgctagacaccaagagaactgatgtcctcttccttcctgcctgcctggagcctgtattctggaggggacagagctagcagatcagacctaactggaaatctgctgtgccagtatatatttcagtgatgtgagccaatatatccccttgattgctcaaagtagtttggtcgatatattttgttgctttaaattgaacacattcttatgtacagcctctgtctcctcatctccaaccaagcaaaatagcttgttctctttatgcagggacacatgacatttcccacgtggctttgtgcatatctccacctcaatttaaaatgccttccaatccctgctcaaagtcaaacagcttaataatggtagacatagaatttgacttattctaataataggtcttttaaacaatgccttcttctcttcattctttccttcttagagtgggtattctttctggtgcatcatgtaaaggaaggtaactacatgcatgtaatgatgagaatatttatatgtatttatgattatcacaaaaaaacaaagattctaccattcaagaggaacatttattttatttttttatttgagaaaagtataattttatttatttatttatttgtgcaaatttatggggtacttgagaaaatgtgttacatgtatataatgtgtagtgatccaatcaggatactaagggtgtccatcacctgagtgtattacatttttgttaagtataatcatcctactccaggagaacattttaaaaactgttctgtagagatactactcaaattaagttctcagtcctgaaacatcagatcagctaggaatctgacaaaaatgcaagttctcagatgacagatgagaccacttcaatcagaatttctggagtggagcccacacatttgtatttttgcaacctttccaatgatacttatgtacatgctcaagcttgaaaaccactttcctaggacattagttcctcgacaagatttgtgagtaaccttgtttcatgaaaaagtgtttaggagatactgattcaataaaaactaatcaggcttttattgtttgcagggctttcaaaacttgcaataggccactgtgcattgttaatttctaagaggaagatgcttatgtcctcaatgaatatcttcccaccatgaagtactcttcttcccccactttttaaaacaattactaacacctggcagaagtaggcagacagcttacagcttagaaaaagttggcctaagataatggctaattttcatacattatttatttgtcatcatgcttatctttctctctctaaattgtatatttcatctctgtgatcacagattgagcctcatatttcatatctgcccctggcctaatggttgtttacagaatgagctcaatgaatattgttaagtgagtaggatttaatttatttgataaatagataaccttaagttttaaacggtggatttcacatgaggacatttacttactattgttgagctgtaattaatttttaatactgtttaggtactcataataaagaacaggatatttggagaaggaagacagtatcattcctggttcttagtcttaccagcttattgatcatgagtatataacctctctgtggctcagtgcctttctctgtaaaatgggaacacagtgatgttcacctcacaggattgatttgtaaaagggctggataaggttatgagaatgttttgcaaagtgatatcgaaagattaattgcaaacttcatttgaatcttaaattgtttgagataggtcatgctatgaatcaactatgaagtgcagatattgtcaagattcaatatttctttcccaagagctgagaggaggggctgcttgtttgtttgtttctttctttttagaaacatgccaggacaggctcattttcgggttttcctctcacttgctcatcttactttttctttagtttctctattcattaggatacagtactgtaaagctttatggcatttttattttgtgggagatgaatctgaataaagaattacagttaaatcattgctaagtttgatgaatgagcaccaaagaactcttcaagatgtcatttttaaagttttgtaaatgattggctttcagtggtttcctctaaggaattttaattttgaataatgcatagaaaaatgtgcgcacacacaaatcattcagtatccacctcgaagggaaatcaaagtgcctgtgaagtgaaactttacctttctatatcaccagcttcctgttagagcagactttttctttgctcaaagtctaagcattgaagaacttctttttagtaggtagatttttgtgtttttttgtttgtttttgagacggagtctcactctgtcgcccaggctgtggtgcagtggcacgatctcggcccactgcaagctctgcctcccgggttcacaccattctcctgcctcagcctcccaagtagctgggactacaggtgccagccaccacgcctggctaatttttttgcattttttagtagagatggggtttcatcgtgttagccaggatggtcttgatctcctgacctcatgatccacccgccttggcctgccagagtgctggaattacaggcgtgatttagtaggtagttttgagtagggagtatacatttaaaatgctgaaactcagttaaggaataatctaatactgtattcaactgaaactcagttgagaaatttctttccaataataaaggaaaatcaactgcagtaatgagggagatgattttgctgctaattacaacaaatatttactacagacctgttatgcaccaggaactgtgctaaatgttttatacatataactttatcttgtgctccaacaacttattacatagaaattgctattattcccattttctagattaataaattggtttagaggggtcgtataggtgaaacaactcactcaatatcacaagctgttatgtggtgaagtttgcatgatcagtacagggttctggtcatcccactcattgagtggtgctagtcaagatctggaagctcttctggtcttagtttctctagccgtgaagtgacaatgattaggtctaatcatagaacatgagagtacatgtgaaaaaatgcctttttaaagagtatgaaaaacttgagttgtaaaattttcttgtggataatttattattgcttttcttttttagataacactaacaaagttgaccttagaattggagtgcctgggttagaaccctgctggtaccacctgcttactgcatgcttctgatgtgagttcaggagaagacactggcaaggacagcaaagaacaggagaacactctagcttccctgatagcattcaaggtgctgtccaaactgactgtgatggcaccctccagacagacagcgatgccacatgttcaagatggcagaatcactatcagcttcaattcctgaatgactgcagagcaaaatttcttacctgcaacatacactctattttcagcctccctggactgttacataatgatacataaaaatatttcttgtgttgaggcatcccaaatttgatttatttgtca-5'<128197810>(seqidno:6)>ccat1_jax_7转录物序列;基因组位置:chr8:128186443-128240377链:-<128240377>3'-gtaatgttgatttttttccaacataattttagagctaggcatattgtattctattacactagactatatatcattcttaaatagaaccagccttgctagataacacatgttggaggagaggcccttcttcttagccctcagtgtttccatctatggggaagaagttccaccatactaacattactatcgtctctccacctgctcactcacttctccccaagggaggggtgttcgatatggtttctgagcttggaaagaaaactcaggcatgtgtaacatggttccttcagtcccatgacccactgtccacaaatgggctgctcacagagtgcatgccttcacccttgttcctggccatgcaggaaattgtatgaaacagtcctagctgaagcctgagattttcctgcattgcctagtcctggtgggtatctgtctactcctggagtttggattggaaagtccacatgcctgaaggtataaacctattctacaaaggggtgttttctagaatgaaggtaatatttttatcttacatttgcagaaagagacagaacaatgttataggactgtattgtctgctagaccatttattcaaagtgggaggatgatgttctaaaagccaatgataaagctcatggcaatgcagggtatatctgatggcatggaatgctttaggatggccaagattgcccatcaaatgccaagtcaccgaagttattaatggctctcctactaggagcctgacatcatggtgagcatcgagaaaggaataacctaagctgaagacacgccttttcaggaggccaagttccacgttctgtgcatgcttttggcgaaagtcaggccaggcactactctgtcccaacaataagccctgtacattgggaaaggtgccgagacatgaactttggtcttctctgcaatccatctggagcattcactgacaacatcgacttgaagttgcactgacctggccagccctgccacttaccaggttgggctcttgagtttctgctttcaagtgaccctcagaaattcctccattcactgcagaggttctgtttctccttgctttgttctgactttacggcagaactaagctaatgagttagttactatgggttatcacttggatttgaagaaccatcatttctaggcattgctgc-5'<128186443>(seqidno:7)>ccat1_jax_8转录物序列;基因组位置:chr8:128218833-128240377链=-<128240377>3'-gcatgtggcaggcacagaaatatttactcattgactgaatatagcacatcgtaatgttgatttttttccaacataattttagagctaggcatattgtattctattacactagactatatatcattcttaaatagaaccagccttgctagataacacatgttggaggagaggcccttcttcttagccctcagtgtttccatctatggggaagaagttccaccatactaacattactatcgtctctccacctgctcactcacttctccccaagggaggggtgttcgatatggtttctgagcttggaaagaaaactcaggcatgtgtaacatggttccttcagtcccatgacccactgtccacaaatgggctgctcacagagtgcatgccttcacccttgttcctggccatgcaggaaattgtatgaaacagtcctagctgaagcctgagattttcctgcattgcctagtcctggtgggtatctgtctactcctggagtttggattggaaagtccacatgcctgaaggtataaacctattctacaaaggggtgttttctagaatgaaggtaatatttttatcttacatttgcagaaagagacagaacaatgttataggttctgggaaataagaaatcattagaaaaagatttctgccttctagaagtacacagtctaatggtgagataggcagttattaatggctctcctactaggagcctgacatcatggtgagcatcgagaaaggaataacctaagctgaagacacgccttttcaggaggccaagttccacgttctgtgcatgcttttggcgaaagtcaggccaggcactactctgtcccaacaataagccctgtacattgggaaaggtgccgagacatgaactttggtcttctctgcaatccatctggagcattcactgacaacatcgacttgaagttgcactgacctggccagccctgccacttaccaggttggctctgtatggctaagcgttttctcctaaaatcccttgaaaactgtgagaagaccataagaagatcatatctttaattctatttcacaagtcacacaatattccaatcaaatacagatggttgagaaaagtcatccatcttccctccccaccctcccacagcccctcaaccactgccctgaaacttatatgctgttatccgcagctccatctggagcatcacagctactgtcaaccctgacgctctttctgaaaaaacaccggatggacatcagaactatttctttaaggatgttactgagccacacaggaaaacttgccttatgattttgaatgcacggatctgatttgactaaacatgataactagagaatcacccaatctactcccattttcaactctaaatcatcagagtgtctcaaatccaaagcacacacagaccagcctggccaacacggtgaaactccacccctactaaaagtataaaaattatccaggtgtggtggcgggcgcctgtaatccaagctacttgggagtctggaggcaggagaatcccttgaacctgggagatggaggttgcagtgagcagagatcacaccaccgcactctagcctgggccacaaatcaacaacaacaacaacaacaaaaaacaaagcgcacacagagactgaggtcctctttggcattgagaagatggctatgcaagtcccaactagcaagtgcaaacttcccagcttcacttctgccagtgtcccttcaccccttctcaaccccactgggaggcaggagggtgcttgacaataacagccttggcatcactctgccagggtgtaataggaactgttacaattctgagattctgtgtaagcactggcctttctgcctagaatgccttctcctctcttttttaactgcatgctcctatttatctttcaaagcccggaaaaaataacactgcacacgggaaatgctcccttcctactgcagtcatttagatgactctatgccattccattcatttctctttcctaccacagaagtgctttgagattttggagtcagactgcttgaacttgaatcctggccctctcatcagagacttgacttattttaggcaagttatataaccaattttacctcagttccttacccataaaatgggtctaatgagagtacctaccacacagaattttgatgaaaactgaatgagatgaaggcctttaaggcagtggtccccaaccctggggacacagacaggtaccattttgtggcctgttaggaactgggccacacagcaggaggtgagcagtgggtgagtgagatcagcgttatttacagctgctccccattgctcaccttactgcctgagctccacctcctgtcagatcagcagtggcattaaattctcatagcagcacaaaccctgtcatgaactgcacatgcgagggatctaggttgtgcgctccttatgagaatctaatgcctaatgacctgtcaccgtctcccatcacccctagatgggagtgtctagttgcaggaaacaagctcagggcttccactgattctacattatggtgagttgtataattatttcattatataatacaatgtaataataatagaaacacagtgcacaacaaatgtaatgtgcttgaatcatccccaaaccatcccagtccacggtcttccacattttgtcttttcacaaaattgtcttccacaaaactggtccctggtgccaaaaaggcttgggaccactgctttaaagcctttgcatagtgcttagaattgagggggaaaaaaaaaacaaaaacaatgtagctagttgctacaatcactatattggtgagtttcaaaaggaaaagaattctgtcccatttatgcttgagccttgagttgctaaccaagcctgacacaaaattactgttgaagggatgtgtgagtcctaattgaaatgaggcctcttaagggaattgtggaccaaaccccaagcaggcagaaagccgtatcttaattattgcaagtatttcaggcaaggtgtggatggccatttgaattcaagcagactaggacctgggatgagaaagaaggtgtgtacgtgacttgatctttgaactttagctcaccatctggaagaaggctgagtattctctgcactcacatagtagctaatgcctactccccagccacccacaattctttctgtaggaaggctcgctagaatactttgtgatattggatattagttccatattctactgtgtatcttagttcaaccaaattgtaatcatctgatatttatttcttttaatataaatataagtatattaagtcttggcatgcttgctcagtctctctctctctcccattcctccccgctcccctctctctttcccaacaggcttggaaagcaggcatcaccatgcctatttaacagttggggtcccttggccaccaggtgctggagtaggaatctgagcccggacatgcctgatctgtaaattttgtgttttccccactgtgctgggcagatcacagctatcagcgccaaattcatagaaggggcgccccctgtggtcaattgagggatttgtgtttgaggtagatctcaagaaggaatgggtggggaacttagcctaggacagagcagaaaggagccctcactccccaagcaccaacggcctcagtccttcctgctgactccagcctctagctctcacccagactatctgcatccttctctccaccacgctcctttggaacctgcgtaaaacacagattaaaggaattccgccttacttccctttccgcattatgaccaaatggttttacactatcattgaacagtttagtacaaaacatgccaccttttaatctattcattcatttaacaaatactttggagtgtttaccatgtgccaagtgctgttctaatagacataagctgtgaggttatgcttatctgattctcacagcaacagctttcgagatatgaattggtatactcatttgacagatgaggaaattgaattcatgtagtgaaaggaagagctgcaattcagggttactggtttctcctgcactaagcactgagccacactagaagagaaggcatgaggaagacaaaagt-5'<128218833>(seqidno:8)对于seqidno:1-8中的每一个,具有与相应ccat1ncrna转录物亚型相同的序列(除了rna中u的被cdna中的t代替)的cdna序列“-”链从3’末端至5’末端显示。另外,也显示各cdna“-”链的第一个和最后一个核苷酸,如它们映射到基因组序列上的对应核苷酸(例如,在seqidno:1中,5’末端的第一个cdna核苷酸c对应于人基因组染色体8上的核苷酸128128655,且5’末端的最后一个cdna核苷酸t对应于人基因组染色体8上的核苷酸128241571)。此外,下表列出了8个转录物ccat1_jax_1至ccat1_jax_8(分别seqidno:1-8)的附加信息,包括由人染色体8上的核苷酸位置表示的各ccat1转录物的各外显子的开始和结束核苷酸位置、各外显子的长度和相应的基因组序列跨度。这些ccat1转录物与以下ncbi参照序列:xr_133500.3中描述的ccat1转录物不同:因此,在一个方面中,本发明提供ccat1ncrna转录物的cdna序列,其中cdna序列由选自seqidno:1-8的序列表示。在相关的方面中,本发明提供ccat1ncrna的拮抗剂序列,其中拮抗剂序列拮抗ccat1ncrna的功能。在某些实施方式中,拮抗序列不拮抗对应于seqidno:9的ccat1ncrna的功能。在某些实施方式中,拮抗剂序列是如seqidno:1-8中所示的任一“-”链cdna序列的反义序列。在某些实施方式中,反义序列在生理条件(例如,在细胞核中)下或在高严格性杂交条件(如通过引入并入本文的由coldspringharborlaboratorypress出版的sambrook和russell的molecularcloning:alaboratorymanual,thirdedition,2001中描述的高严格性杂交条件)下与如seqidno:1-8(但非seqidno:9)中所示的任一“-”链cdna序列杂交。一种这样的高严格性杂交条件可以包括约45℃下的6×氯化钠/柠檬酸钠(ssc),接着50℃、55℃、约60℃或约65℃或更高温度下0.2×ssc和0.1%sds中的一次或多次洗涤。在某些实施方式中,反义序列与如seqidno:1-8中所示的任一“-”链cdna序列在至少其中反义序列与cdna序列杂交的区域中至少约40%、50%、60%、70%、80%、85%、90%、95%、97%、99%或更高程度地相同。在某些实施方式中,反义序列与seqidno:9不超过约50%、40%、30%、20%相同。在某些实施方式中,反义序列长度为约10、12、14、16、20、22、24、26、28、30或更多个核苷酸。在某些实施方式中,拮抗剂序列是靶向于破坏如seqidno:1-8(但非seqidno:9)中所示的“-”链cdna序列代表的任何一个或多个ccat1ncrna亚型的sirna或mirna序列。在某些实施方式中,拮抗剂序列是编码sirna/mirna的载体,或可以被加工成sirna或mirna的rnaseiii(如dicer)的dsrna底物。在某些实施方式中,sirna或mirna包含靶向于破坏ccat1ncrna亚型的约20-25个核苷酸的指导序列。在相关的方面中,本发明提供诊断癌症或前癌性病变的方法,包括测量生物样品中seqidno:1-8任一种或其片段的表达水平,其中生物样品中seqidno:1-8任一种或其片段的表达指示癌症或前癌性病变。在某些实施方式中,片段不是seqidno:9的片段。在某些实施方式中,该方法进一步包括将生物样品中测量的表达水平与标准进行比较,其中生物样品中seqidno:1-8任一种或其片段的较高表达水平指示癌症或前癌性病变。在某些实施方式中,片段不是seqidno:9的片段。在某些实施方式中,该方法包括:(a)从获自受试者的生物样品分离核酸;(b)使能够识别seqidno:1-8中任一种的探针在允许形成杂交复合物的条件下与核酸杂交;和(c)将杂交复合物的形成与标准进行比较;其中生物样品中较高的杂交复合物水平指示癌症或前癌性病变。在某些实施方式中,探针不与seqidno:9杂交。在某些实施方式中,该方法包括:(a)从获自受试者的生物样品分离核酸;(b)扩增分离的核酸中seqidno:1-8任一种或其任一片段;(c)使扩增的ccat1产物可视化;和(d)将ccat1扩增产物的量与标准进行比较;其中较高水平ccat1扩增产物的存在指示癌症或前癌性病变。在某些实施方式中,片段不是seqidno:9的片段。在某些实施方式中,扩增使用对于seqidno:1-8中的一个或多个具有特异性的探针通过pcr(如实时定量pcr)进行。在某些实施方式中,标准通过测量未患癌症的受试者中ccat-1的表达水平来测定。在相关的实施方式中,标准通过测量相同受试者的非癌性组织中ccat-1的表达水平来测定。在某些实施方式中,癌症选自结肠癌(例如,结肠的腺癌)、直肠癌、宫颈癌、肺癌、胃癌、肝癌及其转移。在某些实施方式中,前癌性病变是腺瘤性息肉。在某些实施方式中,生物样品选自组织、血液、唾液、尿液、粪便和骨髓样品。本发明的相关方面提供可用作探针或引物的包含seqidno:1-8任一种或其互补序列的至少8个连续核苷酸的寡核苷酸。在某些实施方式中,寡核苷酸不与seqidno:9杂交。本发明的相关方面提供用于检测生物样品中ccat-1的表达的方法,该方法包括:(a)从生物样品分离核酸;(b)使本发明的ccat1寡核苷酸探针在允许形成杂交复合物的条件下与核酸杂交;和(c)将杂交复合物的形成与标准进行比较,其中生物样品中较高水平的杂交复合物指示样品中ccat-1的表达。本发明的另一相关的方面提供包含cdna或其片段的载体,其中cdna选自seqidno:1-8。在某些实施方式中,cdna片段不与seqidno:9杂交。本发明的另一相关的方面提供包含所述载体的宿主细胞。本发明的另一相关的方面提供成像癌症或前癌性病变的方法,包括:(a)向受试者施用本发明的ccat1探针,其中探针与指示分子偶联;和(b)通过成像设备检测与探针偶联的指示分子(例如,放射性同位素、荧光染料、可见光染料(visibledye)或纳米颗粒)。本发明的进一步相关的方面提供拮抗由seqidno:1-8中的任何一个或多个代表的ccat1ncrna转录物的功能的方法,包括使ccat1ncrna与ccat1的所述拮抗剂序列(例如,反义、mirna或sirna)接触。在某些实施方式中,该方法在体外进行,且ccat1ncrna转录物存在于来自组织培养样品的细胞中。在某些实施方式中,该方法在体内进行,包括向需要的受试者施用ccat1的所述拮抗剂序列(例如,反义、mirna或sirna)。本发明的再另一相关的方面提供包含ccat1的所述拮抗剂序列(例如,反义、mirna或sirna)和药学上可接受的赋形剂和/或载体的药物组合物。应当理解,本申请中描述的任何实施方式(包括仅在本发明的一个方面下描述的实施方式)可以与本发明的其它方面的其它实施方式结合。本领域技术人员应理解,本文中未具体教导的技术可以在标准的分子生物学参考书中找到,如sambrook和russell的molecularcloning:alaboratorymanual,thirdedition,2001,coldspringharborlaboratorypress出版;oligonucleotidesynthesis(m.j.gait编辑,1984);nucleicacidhybridization(b.d.hames和s.j.higgins编辑,1984);pcrtechnology-principlesandapplicationsfordnaamplification,1989,(h.a.erlich编辑)stocktonpress,newyork;pcrprotocols:aguidetomethodandapplications,1990,(m.a.innis等编辑)academicpress,sandiego;及pcrstrategies,1995,(m.a.innis等编辑)academicpress,sandiego;其全部通过引入并入本文。实施例以上一般描述的本发明通过参照以下仅用于说明目的的说明性实施例更容易理解,且说明性实施例不旨在在任何方面进行限制。实施例1一般rich-pet方法学使用rna-dna连接接着末端配对标签测序(rich-pet),申请人开发了以下描述的示例性方法来以无偏的和全基因组的方式研究ncrna(非编码rna)和染色质相互作用。该方法背后的主要概念是基于认识到大多数ncrna调控功能,特别是长ncrna(lncrna)具有的那些,很可能通过rna-蛋白质、rna-dna和/或rna-rna相互作用的任何组合在特定染色质位点中具有直接或间接接触。因此,整个基因组中染色质位置的ncrna接触地址的全面集合将提供大的结构框架和基因组元件的详细内容以理解整体影响以及由单个和/或集体ncrna介导的特定功能。通过交联,可以捕获rna-染色质相互作用。在染色质纤维通过超声处理破碎后,通过各染色质复合物中的蛋白质粘结系留在一起的ncrna和dna片段然后使用所述rna和dna接头经历rna-dna连接以建立rna分子和dna片段的人工连接关系用于具有特异性的高通量分析。本发明的rna接头可以包含随机寡核苷酸序列,例如,随机六聚体核苷酸(hexonucleotide),用于与任何系留rna分子的3’-末端退火,和作为用于逆转录的引物以将rna模板转化成第一链cdna分子。同时,本发明的dna接头连接于平端的染色质dna片段。rna接头和dna接头各自具有彼此互补但不与自身互补的粘性末端。因此,一旦接头相应地连接于其预期靶标,rna和dna片段可以通过连接反应共价地连接。杂合连接产物然后经历末端配对标签(pet)文库构建和后续高通量测序分析。这一方法的示意图描绘于图1a中。可选地,修饰的rna接头可以用于完成rna-dna连接步骤。这一方法的示意图描绘于图1b中。另外,直接rna接头可以用于利用可以将rna3’-末端直接连接于5’腺苷酸化ssdna或5’腺苷酸化悬端的特定酶(如截短的rnl2)进行rna-dna连接步骤,后一方法的示意图描绘于图1c中。为进一步从其作为rna或dna的原始特性区分标签序列,特定的核苷酸条码可以合并到rna和/或dna接头序列设计中,其然后允许精确地调用rich-pet文库数据集中配对的rna-标签和dna-标签。处理的rna-标签和dna-标签序列然后映射到基准基因组(例如,对于人来源的序列的基准人基因组)以鉴别ncrna及其染色质靶位点(数据未示出)。特定试验细节在下面提供用于说明目的。i.细胞培养和交联helas3细胞在补充5%胎牛血清(fbs)(lifetechnologies,cat.10082147)的ham'sf-12nutrientmix(lifetechnologies,cat.11765-054)中生长。对于各批交联的细胞,egs(间隔臂:16.1a;thermoscientific,cat.21565)和甲醛(间隔臂:2.0a;merck–calbiochem,cat.344198-250ml)用于处理细胞以获得蛋白质-dna、蛋白质-rna和蛋白质-蛋白质的双重交联,其可以提供比仅使用甲醛更好的连接性。245mm方形板(corning,cat.431110)中的约1×108个细胞在预热的dpbs(lifetechnologies,cat.14190250)中用45ml的1.5mmegs交联,首先以75rpm振摇40min,然后添加1%甲醛(merck–calbiochem,cat.344198-250ml)并保持振摇20min,接着用0.125m甘氨酸(promega,cat.h5071)淬灭10min,然后用冰冷的dpbs洗涤两次。然后添加包含蛋白酶抑制剂(roche,cat.11873580001)和rnase抑制剂(如superase·intmrnase抑制剂,lifetechnologies,cat.am2696)的3-5ml的冰冷dpbs,随后刮取细胞并转移到15ml-falcon管(lifetechnologies,cat.am1250)。这一过程必要时重复以确保收集所有细胞。细胞在4℃下以2000rpm旋转5min,然后细胞团储存在-80℃直到使用。ii.细胞裂解和染色质生物素化如之前所述进行细胞裂解(goh等,j.vis.exp.,(62),e3770,doi:10.3791/3770,2012;fullwood等,nature,462:58-64,2009,两者通过引用并入本文)。简而言之,核团用冰冷的洗涤缓冲液(50mmtris-hclph=8.0,150mmnacl,1mmedta,1%tritonx-100,0.1%sds)洗涤两次,并悬浮在1ml的相同缓冲液中。染色质通过例如超声处理剪切至平均尺寸约500bp的片段。sds然后添加至剪切染色质达到约0.5%的终浓度,且混合物在如之前所述(kalhor等,nat.biotechnol.,30:90–98,2012,通过引入并入本文)与ezlink碘代乙酰基-peg2-生物素(ipb)(thermoscientific,cat.21334)混合和在室温下旋转60min之前在37℃下孵育15min。链霉亲和素珠-结合的染色质然后进行rich-pet文库构建。iii.rich-pet文库构建链霉亲和素珠-结合的染色质中存在的dna片段使用t4聚合酶(promega,r0191)末端修复,接着使用superscriptiiifirststrandsynthesissystem(lifetechnologies,cat.18080051)进行第一链cdna合成。简而言之,1μg的包含侧翼mmei位点(idt)的生物素化的rna接头a(管1)和rna接头b(管2)分别添加到含有退火混合物(5μl10mmdntp,40μldepc-处理的水)的两个管,并在65℃下孵育5min,然后置于冰上至少约1min,随后与cdna合成混合物(10μl10×rt(逆转录)缓冲液,20μl25mmmgcl2,10μl0.1mdtt,5μlrnaseout,5μlsuperscriptiiirt)混合用于在25℃下孵育10min,接着在50℃下孵育30min。过夜连接在16℃下使用5μl的t4dna连接酶在连接混合物(140μl具有peg的5×t4dna连接酶缓冲液,3.5μlrnase抑制剂,546.5μl无核酸酶水)中分别使用1μg的dna接头a(管1)和dna接头b(管2)进行。接头添加的dna片段然后在pnk主混合物缓冲液(70μl10×t4dna连接酶缓冲液,3.5μlrnase抑制剂,612.5μl无核酸酶水)中用14μl的t4多核苷酸激酶(neb)磷酸化,接着两个管在16℃下用反应缓冲液(1000μl10×t4dna连接酶缓冲液,50μlrnase,8916μl无核酸酶水)中的34μlt4dna连接酶邻位连接过夜。具有接头的染色质dna片段用superscriptdouble-strandedcdnasynthesiskit(lifetechnologies,cat.1197-020)进行第二链cdna合成。具体地,染色质片段与第二链cdna混合物(111μldepc-处理的水,30μl5×第二链反应缓冲液,3μl10mmdntp混合物,1μle.colidna连接酶,4μle.colidna聚合酶i,1μle.colirnaseh)混合,并在16℃下孵育2小时。在反应后,添加2μl的t4dna聚合酶用于在16℃下继续孵育5min。dna/rna/蛋白质复合物中的交联然后通过在65℃下用0.3%sds(ambion)和蛋白酶k(ambion)孵育过夜逆转。cdna-dna片段通过苯酚/氯仿异丙醇沉淀来纯化。纯化的cdna-dna然后在37℃下通过合适的缓冲液(5μl10×nebuffer4,5μlhalf接头非生物素化的以淬灭过量mmei,5μl10×sam)中的1μl的mmei(neb)消化至少2小时以释放cdna标签-rna接头-dna接头-dna标签结构(末端配对标签,pet)。生物素化的pet然后固定在50μl的2×b&w缓冲液(10mmtris-hclph7.5,1mmedta,1mnacl)中的链霉亲和素偶联的磁性dynabeads(lifetechnologies,cat.11206d-10ml)上,在室温下振荡45min。各pet结构的末端然后在16℃下伴随混合通过在连接体连接缓冲液(4μl连接体a,4μl连接体b,5μl10×t4dna连接酶缓冲液,36μl无核酸酶水)中的1μl的t4dna连接酶(thermoscientific,cat.el0013)连接于连接体过夜。珠然后用1×b&w缓冲液(5mmtris-hclph7.5,0.5mmedta,1mnacl)洗涤三次。缺口翻译用反应混合物(38.5μl无核酸酶水,10×nebuffer2,2.5μl10mmdntp)中的4μl的e.colidna聚合酶i进行,其在室温下在intelli-mixer(f8,30rpm,u=50,u=60;elmiltd.,riga,latvia)上伴随搅拌孵育2小时。之后进行16轮的pcr以扩增pet。rich-pet文库在illuminahiseq2000(2×36bp阅读)上测序。所有步骤在具有蛋白酶抑制剂和rnase-抑制剂的缓冲液中进行以防止或最小化蛋白质和rna降解。本文中使用的各种多核苷酸或引物在下面列出:实施例2rich-pet文库统计三个rich-pet文库数据集使用来自helas3细胞的技术和生物学重复产生。helas3rich-pet数据映射结果文库重复阅读独特pet簇(≥pet2)chh24301(tec)52,254,1302,367,8985,371jchh24302(tec)211,837,2042,920,3699,089jchh24313(bio)83,143,9992,049,9423,128rich-pet数据分类为单态pet(即,rna-标签和dna-标签两者上没有与其它pet序列的重复)或为具有2个或更多个pet序列的pet簇(即,配对的rna-标签和dna-标签序列两者与其它pet重叠)。pet簇被认为更可靠,或认为是反映更可靠的ncrna-染色质相互作用事件的重复检测的高可信数据,而单态pet可以代表弱连接信号,但可与随机背景噪音区分开。使用簇集标准,鉴定了与约5000个染色质位点关联的大约700个推定的rna位点(图2a)。作为快速证实,检验了这些rna和dna位点的rna-seq信号,且发现rna位点事实上具有比dna位点显著更高的rna计数,表明rich-pet数据如所预期的(图2b)。约五分之一(约22%)的所获得的rna-dna关联性数据可以被认为性质上是顺式作用的(即,从rna至dna映射位点<100kb),而大多数rna-dna关联性数据是反式作用的(图2c)。一个关注点是染色质rna-dna连接途径可以在转录仍在进行时捕获大部分新生的mrna。令人惊异的是,数据显示大多数新生mrna转录物表现为使其3’-末端隐藏在rna聚合酶复合物的中心内,以使得本发明的方法(其部分地基于使用ncrna分子的假定自由的3’-末端)很大程度上避免新生mrna的干扰。具体地,映射配对的rich-pet数据揭示了配对的rna和dna标签之间的距离,因此表明可能的相互作用模式,顺式或反式。映射结果显示仅小的数据集是顺式作用的,而大多数是反式作用的和染色体间的,表明在rich-pet方案中捕获新生转录物的可能性是低的。rna标签簇的进一步注释分析(参见以下)显示仅3%的rna标签映射于mrna外显子,而绝大多数映射于ncrna。另一个关注点是细胞中rrna的丰度,这是rna相关分析的一般问题,因为在一些细胞中,rrna可以高达总rna的80%。处理rrna的一个策略包括在开始特定分析之前使用的回避方式(avoidanceapproach),如mrna的polya+选择方式和rrna的负向耗尽。我们评估在rich-pet文库之一中rrna序列的丰度水平,并发现rrna序列占总rna标签的约26%。相反,几乎没有(0.23%)dna标签对应于rrna序列。因此,数字耗尽(digitaldepletion)方式可以用于在任何进一步的分析之前除去所有rrna序列以降低由于rrna导致的数据噪音。实施例3rich-pet方法的可重现性和灵敏度为评估rich-pet数据的可重现性,可以进行两个技术重复(分成两个等分试样用于平行文库构建和测序分析的相同的细胞制剂)和两个生物学重复(在不同时间收集的用于采用具有轻微改变的几乎相同的过程的文库构建中的不同细胞制剂)。所得的重复结果显示出真正的可重现性(图3)。例如,已知牵涉在癌症中的两种充分研究的lncrnaneat1和malat1在所有三个文库中可再现地检测到(数据未示出)。值得注意的是,这两种lncrna基因发现在rna聚合酶ii(rnapii或rnapol2)介导的广泛染色质相互作用环结构中空间组织化,表明它们的表达最可能在共同的转录机构复合体下共调节。在本文中获得的rich-pet数据中,malat1和neat1两者在helas3细胞中高度表达,且在所有三个rich-pet数据集中丰富地检测。具体地,neat1与细胞中的malat1相比相对较低地表达,因此,对于neat1的rich-pet数据计数低于malat1(数据未示出)。作为对照,hotair是在helas3细胞中以低水平表达的另一个已知lncrna,且它在所得的rich-pet数据中未检测到(数据未示出)。因此,表现出的是rich-pet数据中ncrna的检测与ncrna表达水平良好相关。实施例4rich-pet数据的验证基于获得的rich-pet映射数据,有趣的是,即使这两个ncrna在相同的转录机构中共转录,它们的相互作用性质是非常不同的。具体地,neat1rna限制性地处于顺式,仅结合于其被转录处;而malat1大部分处于反式,与基因组中的许多位点相互作用(图4a)。为验证这一观察,使用neat1和malat1rna作为荧光进行rna-fish试验以检查hela细胞核(图4b)。如所预期的,neat1探针仅产生每细胞核1或2个斑点,而malat1探针在所有核空间上均产生斑点,与rich-pet数据中观察到的一致。a549细胞中对于neat1和malat1也获得相似的rna-fish结果。这一验证表明rich-pet数据是定性的且在检测和区分真正的顺式和反式相互作用中是准确的。实施例5rich-pet数据的表征rna和dna标签簇基于人基因组的genecodev14注释进行表征。仅3%的rna标签簇与蛋白质编码外显子重叠,且绝大多数rna标签簇映射到非编码区,其中许多是先前已知的ncrna(172,24%)。其余是位于蛋白质编码内含子区域、反义和基因间(inter-genetic)区域中的潜在的新ncrna(图5a)。在rich-pet数据中鉴别的所有推定ncrna具有rna-seq数据支持,表明它们在hela细胞中活跃地转录。相反,rich-pet数据的dna标签簇主要地映射于蛋白质编码基因且主要的部分映射于基因启动子(图5b)。在rna和dna标签簇周围的一组染色质活性标志进行进一步分析。有趣的是注意到rna标签簇的中心偏离对于开放染色质状态通过rnapol2和dhs信号限定的转录活性峰,且这种“中心偏离”性质是链特异性的(数据未示出)。这种链特异性的“中心偏离”性质与rich-pet方法一致,因为其被设计为捕获rna的3’-末端。因此,rna标签簇预期在转录开始位点的下游。相反,染色质活性信号在dna标签簇中心周围对称地成峰(数据未示出),反映了超声处理对染色质纤维的随机剪切。实施例6.malat1与许多基因组特征相互作用且可以发挥基因激活和基因抑制的两种功能使用与malat1关联的所有rich-pet数据(包括单态pet),申请人产生全染色体的和全基因组的malat1-相互作用谱,表明malat1具有与基因组中的大区域相互作用的潜能(数据未示出)。超过50个高可信度的相互作用(标签计数≥2的pet簇)位点中,大约一半位于启动子中且四分之一在已知基因的内含子区域中(图6a)。来自相同细胞的rna-seq和rnapol2chip-seq数据显示在其启动子中存在malat1的基因具有比在其内含子区域处具有malat1相互作用的那些基因显著更高的转录活性(图6b;数据未示出)。已经报告,malat1通过与多个剪接因子(包括srsf2)相互作用参与调节剪接功能(tripathi等,2011)。申请人还发现malat1rna可以通过与其启动子相互作用而直接参与调节srsf2的表达(数据未示出)。这些观察表明malat1可以具有调节基因激活和抑制的多种功能性作用。实施例7xist在x-染色体以外的功能最良好表征的lncrna是xist,其从一个拷贝的x染色体转录并结合(顺式作用)于另一拷贝的x染色体中的相同位点,且进一步延伸以覆盖整个染色体用于灭活(未示出)。rich-pet映射数据显示与xist的rna标签配对的dna标签在x染色体中高度富集,而背景噪音分散在整个基因组中,表明xist如预期的特异性地结合x染色体。有趣的是,它也表现出在一个非x染色体中一定水平的xist-结合的富集及对于另一非x染色体的某种程度的耗尽。获得了更多的数据和进一步的分析以进一步验证这一观察。实施例8ncrna的复杂相互作用网络此处给出的rich-pet数据提供了关于复杂的ncrna相互作用网络系统的第一印象。除了一个ncrna在基因组中可以具有多个靶标(malat1)的经典观点,已发现许多推定的ncrna位点具有“波动(in-and-out)”的rich-pet数据,因为发现位点通过ncrna发生相互作用,且由此相互作用的ncrna也检测到与另一位点相互作用。在许多意义上,这一ncrna相互作用网络类似于转录因子(tf)结合网络,其中许多tf结合用于转录调节的彼此的基因。更多数据有助于进一步阐明ncrna如何发挥功能,且ncrna相互作用网络如何影响基因组系统。实施例9通过ccat1编码的lncrna是转录共激活因子rich-pet方法用于鉴别总体ncrna-基因组dna相互作用。在鉴别的相互作用中,一种ncrna-结肠癌相关转录物1-具有特别的意义。结肠癌相关转录物1(ccat1)是目前使用代表性差异分析(rda)、cdna克隆和cdna末端的快速扩增(race)发现的2628核苷酸长的非编码rna(nissan等,“coloncancerassociatedtranscript-1:anovelrnaexpressedinmalignantandpre-malignant人tissues,”int.j.cancer,13:1598–1606,2012)。其最近发现在结肠癌(cc)中超表达,但不在正常组织中超表达,从而使其成为潜在的疾病特异性生物标志物(nissan等,int.j.cancer,130(7):1598-606,2012;alaiyan等,bmccancer,13:196,2013)。基于rich-pet数据的仔细分析揭示这一位点中亚型转录物的新的复杂模型(数据未示出)。另外,ccat1在宫颈癌细胞系hela细胞中高度转录。rich-pet数据也揭示ccat1lncrna转录物靶向基因组中的许多其它位点(数据未示出),包括所有人染色体,除染色体15、16、20、x和y。在具有至少2个ccat1标签的ccat1染色质靶标中,许多在增强子或启动子中显示出最强的lncrna-基因组dna相关性(数据未示出)。例如,对于与至少3个ccat1rna标签相关的122个ccat1基因组靶位点,88个靶位点在增强子区域中,包括具有rnapii相互作用的增强子位点中的6个。ccat1的另外34个基因组靶位点在启动子中。这些ccat1靶基因具有比随机选择的对照基因集合高几倍的平均表达水平,表明ccat1lncrna促进靶基因表达。这些ccat1靶基因中的一种是c-myc,在广泛的癌症是过表达的致癌基因,包括约80%的乳腺癌、70%的结肠癌、90%的妇科癌症、50%的肝细胞癌和具有异常myc表达的多种血液肿瘤(如伯吉特淋巴瘤)。另外的数据表明ccat1lncrna通过结合ccat1位点本身以及myc位点而发挥功能,因此使得ccat1和myc位点密切地物理接近并允许ccat1位点中的增强子刺激myc转录。另外,ccat1转录的lncrna可以结合蛋白质因子并用作转录共激活因子,因此直接增强myc以及其它ccat1靶基因如fam84b和snx14的转录。实施例10人b-类淋巴母细胞gm12878和果蝇s2细胞中另外的应用使用如上所述基本上相同的rich-pet方法,申请人从人b-类淋巴母细胞gm12878和果蝇s2细胞获得另外的数据以进一步支持rich-pet方法的一般适用性。具体地,人gm12878细胞用于rich-pet分析,因为ncrna基因xist在这一细胞系中高表达,而之前用于rich-pet分析的hela细胞具有低水平的xist表达,且hct116源自男性,因此不具有xist表达。因此,在使用xist作为模型来评价rich-pet分析检测ncrna与染色质的相互作用的性能时,gm12878是用于rich-pet分析的好得多的细胞类型。如之前所述的,xist特异性地或优先地结合x-染色体。参见图9a,其显示以每百万阅读每kb的阅读(rpkm)计的通过rna-seq数据测量的xist计数;和图9b,其显示xist结合覆盖的各染色体的比例。在gm12878细胞中,大部分染色体仅在10-20%的总染色体空间中被xist覆盖,而x-染色体接近90%被xist覆盖。这一覆盖率代表xist对于其靶向染色体的特异性高于其它非特异性染色体几乎6-倍(5.9-倍)的特异性。相反,在hela细胞中,覆盖率代表xist对于其靶向染色体的特异性高于其它非特异性染色体约3.4-倍的特异性,和在hct116细胞中,如所预期的,没有观察到x-染色体富集。类似地,在果蝇s2细胞中,ncrna基因rox2-与人的xist等同-显示rox2结合x-染色体的类似富集:高于其它染色体5-倍(数据未示出)。具体地,获得了全果蝇基因组中的rox2结合数据。超过80%的rox2-连接的dna-标签结合于x-染色体,代表对于x-染色体的5-倍富集。具有在通过chart-seq和通过rich-pet方法在x-染色体上的rox2映射之间观察到的合理强的相关值(0.6),证明rich-pet方法的适合性。大部分的rich-pet数据的rna-标签映射到非编码区,而仅约26%在编码区中,表明该方法具有ncrna的富集(数据未示出)。rich-pet数据的rna-标签与来自果蝇s2细胞的rna-seq数据的比较显示对于已知ncrna的显著富集(数据未示出)。总之,以上实施例给出的数据证明本发明的方法(例如,rich-pet方法)如设计的发挥作用。rich-pet数据中绝大多数的rna标签映射到非编码区,且它们中的一些映射到已知lncrna如malat1和neat1。这是这一方法如所预期的运行的强烈指示。更重要的是,通过rna-dna关联性映射数据,申请人能够鉴别全基因组的潜在ncrna-染色质相互作用位点。迄今为止进行的几条线的初步验证已经表明rich-pet鉴别的ncrna相互作用是真实的。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1