生产胞嘧啶的重组微生物及生产胞嘧啶的方法与流程
生产胞嘧啶的重组微生物及生产胞嘧啶的方法
[0001]
本申请是申请号为:201910602231.2、发明创造名称为:生产胞嘧啶的重组微生物及生产胞嘧啶的方法、申请日为:2019年07月05 日的发明专利申请的分案申请。
技术领域
[0002]
本发明属于生物技术领域,具体地,涉及生物发酵法和酶法生产胞嘧啶的方法。主要是生产胞嘧啶的重组微生物的构建和发酵生产方法的建立,其次是利用基因工程微生物生产胞苷水解酶液,然后利用该酶液来水解胞苷制造胞嘧啶的方法。
背景技术:
[0003]
胞嘧啶是核酸中嘧啶型碱基之一,学名为4-氨基-2-羰基嘧啶, cas rn:71-30-7,分子式:c4h5n3o。白色或类白色结晶性粉末,常温下在水中的溶解度低,水微溶于乙醇,不溶于乙醚。
[0004]
胞嘧啶是精细化工、农药和医药的重要中间体,特别在医药领域,主要用于合成抗艾滋病药物及抗乙肝药物拉米夫定,抗癌药物吉西他宾、依诺他宾、5-氟胞嘧啶等等。目前国内外对胞嘧啶是通过化学法来合成的,主要的生产集中在国内。胞嘧啶是一个含碳和氮的杂环化合物,在化学合成过程中需要使用到强碱和有机溶剂,导致废水处理难度大。随着国家对化工合成环保要求的提高,国内企业开始停止化工合成法生产胞嘧啶,或将生产转移到对环保要求不高的地区。因此需要开发一种能替代化学合成、降低污染的胞嘧啶生产方法。
[0005]
人类已经利用微生物来发酵生产核苷类的产品,如β-胸苷、鸟苷、肌苷和腺苷,但胞苷、胞嘧啶等没有实现微生物法工业生产。本公司在前期研究中,利用基因工程改造技术,首次报道了利用生物法生产胞苷(专利申请号:201710003833.7),构建得到的重组菌株在5l 发酵罐可以生产胞苷35g/l。本发明是在前期工作的基础上,对胞苷生产菌进行进一步的改造,期望实现胞嘧啶的生物法生产。
技术实现要素:
[0006]
发明目的:(1)能够生产胞嘧啶的基因工程菌种的构建;(2) 提供转化胞苷来生产胞嘧啶的酶,构建基因工程菌种生产相关酶液和转化胞苷生产胞嘧啶的方法。
[0007]
技术方案:
[0008]
申请人在国内专利申请(申请号201710003833.7)中提供了能够发酵生产胞苷的重组微生物,以大肠杆菌基因工程菌为例,本发明是在该菌的基础上改造成新的重组大肠杆菌生产胞嘧啶,满足工业发酵生产胞嘧啶的需要,提供了一条最佳的胞嘧啶生物合成途径,达到胞嘧啶的工业发酵生产的最低成本。既然上述专利申请(申请号 201710003833.7)中大肠杆菌基因工程菌可以发酵生产胞苷,那么最简单的方法是利用上述重组微生物进行发酵,在发酵结束时,或获得纯化的胞苷后,通过核苷水解酶水解胞苷生成胞嘧啶和核糖。然而,所产生的核糖的市场小,核糖没有销量,副产物核糖还会导致处理的成本和时间增
加。因此,本发明考虑在重组大肠杆菌发酵时直接产生胞嘧啶,同时使副产物进一步被利用而不积累副产物核糖。
[0009]
上面提到胞苷可以经过核苷水解酶生成胞嘧啶和核糖,如图1所示,本发明还考虑在产胞苷的大肠杆菌中恢复编码核苷水解酶的基因,或在细胞中超表达编码核苷水解酶的基因直接转化胞苷生成胞嘧啶和核糖,然而在以葡萄糖为碳源时核糖很难被大肠杆菌利用(在以甘油为碳源时核糖也有一定量的积累),这样就会造成胞嘧啶的产率低下,发酵液中积累的核糖也会增加发酵废液的处理时间和成本,因此需要改变细胞的代谢调控,使细胞在利用葡萄糖或甘油的时候,同时使生产胞嘧啶过程中产生的核糖回到嘧啶核苷的合成中。第二条胞苷生产胞嘧啶的途径是由核苷磷酸化酶在无机磷酸盐参与下产生胞嘧啶和1-磷酸核糖,后者可以经由异构酶生成5-磷酸核糖回到嘧啶核苷的发酵途径中,然而这一反应是可逆的,它是否能在细胞内将胞苷完全转化到胞嘧啶还是未知的。
[0010]
在上述国内专利申请(申请号201710003833.7)中披露了增加细胞内从三磷酸胞苷(ctp)合成单磷酸胞苷(cmp)代谢流,那么如图2所示,本发明可以直接以单磷酸胞苷(cmp)为底物,通过核苷酸5
’-
磷酸核苷酶产生胞嘧啶和5
’-
磷酸核糖(r5p),后者在5
’-
磷酸核糖二磷酸激酶(prs)的催化下合成5-磷酸核糖-1-焦磷酸(prpp)后回到核苷酸的合成途径中。
[0011]
大肠杆菌不仅具有从头合成嘧啶核苷的能力,而且拥有补救途径来通过尿嘧啶磷酸核糖转移酶(upp)利用尿嘧啶生成嘧啶核苷酸。细胞中的胞嘧啶先被脱氨生成尿嘧啶后参与嘧啶核苷酸合成的补救途径。因此无论是以胞苷还是cmp为胞嘧啶的直接前体物,所产生的胞嘧啶都不能降解成尿嘧啶,这就需要将大肠杆菌中的编码胞嘧啶脱氨酶的基因破坏。
[0012]
1.大肠杆菌中胞嘧啶的代谢途径
[0013]
在申请人的国内专利申请(申请号201710003833.7)中,在嘧啶核苷酸的从头合成途径中,三磷酸胞苷酸(ctp)被超表达的ctp 焦磷酸水解酶(大肠杆菌自身的nudg)水解,生成单磷酸胞苷酸 (cmp)。同时细胞中的rna在胞内经rna酶解后产生的cmp和被nudg合成的cmp一起被cmp水解酶,如phm8(kuznetsova 2015) 和大肠杆菌自身的核苷酸酶(usha,μmpg,μmph(www.ecocyc.org) 等等)降解生成胞苷。虽然大肠杆菌是被研究的最多最深的微生物,但具体的哪一个核苷酸对cmp的水解贡献最大至今没有知晓,这可能是为什么国内专利申请(申请号201710003833.7)中借用了酿酒酵母来源的phm8。胞苷在大肠杆菌中产生后由多种途径使其参与嘧啶核苷酸补救合成途径中,首先胞苷可以被脱氨生成尿苷,胞苷和尿苷在尿苷激酶(udk)的作用下生成相应的cmp和μmp,cmp被cmp 激酶(cmk)磷酸化生产cdp,cdp和μmp都是嘧啶核苷酸合成途径中的中间体。其次胞苷可以被细胞中的各种胞苷水解酶(见2a 中的介绍)降解成胞嘧啶,后者在脱氨酶(coda)作用下生成尿嘧啶。尿嘧啶可以在尿苷磷酸化酶(udp)或尿嘧啶磷酸核糖转移酶 (upp)的作用下分别生成尿苷和μmp。在国内专利申请(申请号 201710003833.7)中,大部分胞苷水解酶,尿苷磷酸化酶(udp)和尿苷激酶(udk)的编码基因都被用基因工程的手段敲除了。
[0014]
大肠杆菌能表达不同转运蛋白,有些负责将胞内产生的多余的物质转运出来(外转运蛋白),有些负责将胞外的营养物质从胞外转运到胞内(内转运蛋白)。已知的核苷外转运蛋白有nupg,nupc,tsx 和codb是胞嘧啶内转运蛋白等等,有关胞嘧啶外转运蛋白还不为
人知。如果能够找到胞嘧啶外转运蛋白,那么超表达胞嘧啶外转运蛋白将减少在基因工程菌种(同时codb基因被敲除)中胞嘧啶的积累,这样不仅可能提高胞嘧啶提取和纯化的收率,还可能进一步提高胞嘧啶的产量。
[0015]
2a.胞外水解胞苷合成胞嘧啶
[0016]
在生产胞苷菌株基础上进行菌种改造来生产胞嘧啶有两条路径可供选择(如图1所示),一条路径是通过胞苷的水解来生产胞嘧啶,另一条是从cmp水解来生产胞嘧啶。
[0017]
大多数野生型的生物自身都能针对不同类型的核苷底物产生很多核苷水解酶和核苷磷酸化酶。例如大肠杆菌可合成数种核苷水解酶,如riha、rihb、rihc(petersen 2001)和核苷磷酸化酶,如 ppnp(sevin 2017)。其中rhia、rihb和rihc可以水解各种核苷生成相应的嘌呤或嘧啶与核糖,而ppnp则需要磷酸根离子的参与,除了生成相应的嘌呤或嘧啶外,还产生1-磷酸核糖。在其它生物中能够水解胞苷的核苷水解酶很多,比如酿酒酵母中的urh1(ydr400w, mitterbauer 2002),产氨帮杆菌来源的rih1和rih2(kim 2006) 等等,它们都可以用来在体外酶催化水解胞苷制备胞嘧啶。
[0018]
2b.胞内水解胞苷合成胞嘧啶
[0019]
前述2a中在胞外水解胞苷的不足之处是胞苷水解后产生大量的核糖,每公斤的胞苷生产0.54公斤的核糖,而核糖的市场很小。因此在胞内直接水解胞苷生成胞嘧啶和核糖,后者被循环利用到核苷酸的合成中,这将是降低胞嘧啶生产成本的关键。另外,前期的研究结果显示,在同样的发酵条件下,基因工程菌种生产胞苷速率比菌种只产胞嘧啶要快得多,产量(mole/l)也高的多,这可能是核糖或胞嘧啶的积累对胞嘧啶的生成不利。众所周知,野生型大肠杆菌在葡萄糖和核糖同时存在时,野生型大肠杆菌不会利用核糖,一直要到葡萄糖被消耗完后再利用核糖。在本发明中,我们发现大肠杆菌在以甘油为碳源生产胞嘧啶时,也会积累d-核糖。
[0020]
改造大肠杆菌使其同时利用葡萄糖和其它糖类有很多先例和文献,但没有文献报道过基因工程菌种利用葡萄糖的同时消耗核糖。大肠杆菌的基因组上有一个编码核糖调控rbsr的基因。当培养基以核糖为唯一碳源时,核糖和rbsr结合,从而启动核糖转运蛋白酶和核糖激酶等四个和核糖代谢相关的酶。因为在胞嘧啶的生产过程中,核糖已经在胞内,我们通过(1)表达核糖激酶将核糖磷酸化成 5-磷酸核糖,后者可以直接由5-磷酸核糖焦磷酸激酶(prs)生成 prpp进入核苷酸的从头合成途;(2)将大肠杆菌中的rbsdacbk操纵子(www.ecocyc.org)置入一个不受核糖调控的启动子下,这样操纵子的基因在任何时候都能表达,从而使基因工程菌种能够利用核糖;(3)利用基因工程的方法将rbsr基因敲除,从而解除对rbsdabck的转录调控。在本发明中敲除rbsr基因的方法得到了证实,它可以有效的使基因工程菌种在产胞嘧啶的同时,不在发酵液中积累核糖。
[0021]
3.核苷酸5-磷酸核苷酶催化cmp合成胞嘧啶
[0022]
大肠杆菌中产生各种核苷酸5-磷酸核苷酶,虽然它们在细胞内的具体功能不是很清楚,但它们可能可以应用到嘌呤和嘧啶的合成之中。大肠杆菌中被研究到最多的核苷酸5-磷酸核苷酶是5-磷酸腺苷核苷酶(amn,uniprot id:p0ae12),它催化5-磷酸腺苷(amp)合成腺嘌呤和5-磷酸核糖(leung 1980)。大肠杆菌中的核苷酸磷酸核苷酶(核苷酶)(ppnn,uniprot id:p0adr8)据报道(sevin 2017)在体外催化各种单磷酸核苷酸(比如amp,tmp)产生相应的嘌呤或嘧啶。同amn的一样,与ppnn同源的蛋白不仅广泛的存在于各种革兰氏阴性
细菌中,而且同源性非常高(85%以上)。不同于amp核苷酶(amn),目前对ppnn在细胞体内的催化活性和功能研究则完全缺乏,我们曾尝试超表达ppnn或amn在大肠杆菌细胞内生成腺嘌呤,超表达amn 可以在培养基中积累腺嘌呤,但超表达ppnn则没有腺嘌呤积累,说明在胞内ppnn并不能以amp为底物或对amp的活性很低。因此在本发明之前,没有任何证据表明ppnn在细胞内能够以cmp,mmp和tmp 等嘧啶核苷酸为底物。在其它微生物中,类似的核苷酸5-磷酸核苷酶也有报道,例如在对灰色链霉菌中参与杀稻瘟菌素(blasticidin)合成的的blsm(grochowski 2006)和streptomyces remofaciens zju5119 来源的milb(zhao 2014)的体外研究中发现它们能够利用cmp为底物产生胞嘧啶。然而,大肠杆菌来源的ppnn同上面链霉菌来源的 milb和blsm没有过任何同源性。可以推测自然界中存在不同起源的核苷酶具有水解单磷酸核苷酸从而释放相应的嘌呤或嘧啶。可以预测如果在大肠杆菌中表达这些酶和它们的同源蛋白质,可以找到能够将胞内的cmp转化成胞嘧啶的酶。
[0023]
4.其它
[0024]
本发明中英文词缩写主要是大肠杆菌中的基因,酶及其代谢产物,它们都可以在www.eccocyc.org网页中查到,非大肠杆菌来源的英文词缩写则见相应的文献。
[0025]
现代的生物技术,特别与合成生物学和dna合成等相关的技术能够很容易地获得本发明权利要求的蛋白质的突变体,突变体不仅能够规避本发明权利要求,而且有可能获得酶活更高、更好的酶。同时随着人类对自然界生物基因组的系列测定,科学研究者能够在这些基因库中容易地找到同本发明中提到的酶同源的蛋白质,然后将编码同源蛋白质的基因进行异源表达,众所周知高度同源性的蛋白质常常具有一致的生物功能。所以无论是从基因序列库还是通过突变(通过基因合成或pcr的方法获得突变)应当视为对本发明的侵犯。
[0026]
本发明在世界上首次提供了基因工程大肠杆菌能够大量的发酵生产胞嘧啶,随着进一步的研发,胞嘧啶的产量会逐步的提高,生产胞嘧啶的成本也会进一步地降低。本发明中披露的胞嘧啶合成的途径和方法很容易地被仿效,用于在其它菌种-芽孢杆菌(bacillus),谷棒杆菌(corynebacterium),沙门菌(salmonella)和酵母等等中进行胞嘧啶的生产。
[0027]
有益效果:本发明具有以下优点:本发明提供了三种生物制造胞嘧啶的方法,特别是构建了基因工程菌种能够发酵单独生产胞嘧啶,或生产胞嘧啶和胞苷混合物(其中胞苷可以通过核苷水解酶来产生胞嘧啶)等等。本发明的胞嘧啶发酵生产完全可以替代目前的生产方法,从而实现环保、绿色的胞嘧啶生产。
附图说明:
[0028]
图1大肠杆菌胞嘧啶代谢路径示意图,其中orotate:乳清酸;omp:5
-ꢀ
单磷酸乳清酸苷;μmp:5-单磷酸尿苷;udp:5-二磷酸尿苷;utp: 5-三磷酸尿苷;ctp:5-三磷酸胞苷;cmp:5-单磷酸胞苷;cdr:胞苷;cyt:胞嘧啶;ribose:d-核糖;r5p-5磷酸核糖;uracil:尿嘧啶;coda:胞嘧啶脱氨酶;prs:5-磷酸核糖二磷酸激酶;prpp: 5-磷酸核糖-1-焦磷酸;upp:尿嘧啶磷酸核糖转移酶;(1):核糖核苷水解酶;(2):核苷磷酸化酶;(3):核苷酸5-磷酸核苷酶;
[0029]
图2为核苷酸5-磷酸核苷酶(核苷酶)水解cmp生成胞嘧啶示意图;
[0030]
图3为实施例8缓冲液浓度优化检测图;
[0031]
图4为实施例8ph条件优化检测图;
[0032]
图5为实施例8反应温度优化检测图。
具体实施方式
[0033]
下面将通过几个具体实施例,进一步阐明本发明,这些实施例只是为了说明问题,并不是一种限制。
[0034]
本发明的技术方案可以应用大肠杆菌,枯草杆菌,谷氨酸棒杆菌,乳杆菌或者其他的微生物,下面以大肠杆菌为例,对技术方案进行进一步说明。
[0035]
实施例1在大肠杆菌中基因敲除的方法
[0036]
本发明采用datsenko的方法在大肠杆菌中进行基因敲除 (datsenko 2000),相应的基因敲除引物见baba 2006。
[0037]
实施例2摇瓶发酵验证重组菌株的方法
[0038]
验证重组菌株在摇瓶发酵中生产胞嘧啶的发酵培养基,具体为每升培养基中yc溶液100ml,5倍的盐溶液200ml,tm2溶液1ml,柠檬酸铁10mg,无水硫酸镁120mg,氯化钙111mg,硫胺素1ug,以去离子水定容,其中5倍的盐溶液为每升中含磷酸氢二钠30g,磷酸二氢钾15g,氯化钠2.5g,氯化铵5.0g,以离子水定容;tm2溶液中每升含四水氯化锌2.0g,六水氯化钙2.0g,两水钼酸钠2.0g,五水硫酸铜1.9g,硼酸0.5g,盐酸100ml。发酵培养基中yc溶液为甘油 2g,蛋白胨0.6g,酵母粉0.2g,去离子水100ml;上面溶液灭菌为115 ℃,20-30分钟。
[0039]
摇瓶发酵过程如下:首先接种重组菌株至含有50mg/l壮观霉素的lb培养基(green 2012)中,置34℃摇床中,250rpm培养过夜;取过夜种子200μl转移到至2ml含有抗生素的lb,于34℃摇床中, 250rpm培养5小时至od 600
值为1左右;随后将2ml二级种子全部转入装有18ml发酵培养基m11的摇瓶中,置34℃摇床中,250rpm 培养4-6小时,添加iptg至终浓度0.1mm后,继续培养20小时左右,取1毫升的发酵液离心后(7000rmp,4分钟),取上清检测,检测方法见实施例3。
[0040]
实施例3hplc测定发酵液中的胞苷和胞嘧啶
[0041]
吸取1ml发酵液于2ml离心管中,100℃加热5分钟,冷却至室温,稀释一定的倍数,离心过0.22μm的滤膜,用高效液相色谱(hplc) 检测,hplc的参数如下:采用agilent sb c18 4.6*150mm 5μm,流动相为甲醇和10mm pbs(ph4.0),流动相比例0.01-2.80分钟甲醇比例为2%,2.80-3.50分钟甲醇比例由2%上升到10%,3.50-3.60分钟甲醇比例由10%降至2%,3.60-8.5分钟甲醇比例为2%,利用紫外检测器检,测波长260nm;初始流动相的流速为1.0ml/min,发酵液的上样量为5μl,柱温30℃。
[0042]
实施例4构建不能降解和利用胞嘧啶的重组大肠杆菌
[0043]
微生物中的胞嘧啶主要是在胞嘧啶脱氨酶coda的催化下转化成尿嘧啶,之后再回到嘧啶核苷酸的合成中。因此,为了使微生物能够积累胞嘧啶,首先要对细胞中利用胞嘧啶降解为尿嘧啶参与嘧啶核苷酸合成补救路径的胞嘧啶脱氨酶的编码基因coda进行阻断或敲除,这样胞嘧啶就不能脱氨降解为尿嘧啶,微生物就能积累胞嘧啶。此外,为了积累更多的胞嘧啶,需要将胞嘧啶运输到胞内的转运蛋白基因 codb进行敲除,使胞嘧啶在胞外进行积累,有助于提高产量,也有助于简化后期的产物纯化。codb和coda位于同一个操纵子下,
因此同时进行敲除。
[0044]
为了使重组微生物能够积累胞嘧啶,胞苷菌种 hf9/phs01-nudg-phm8-pyre-pyrhm(cgmcc no.13427)的母本菌hf9 中的codba基因被一起敲除掉,得到了缺失产生胞嘧啶脱氨酶和胞嘧啶转运蛋白的菌株hf11k。具体实验过程如下:
[0045]
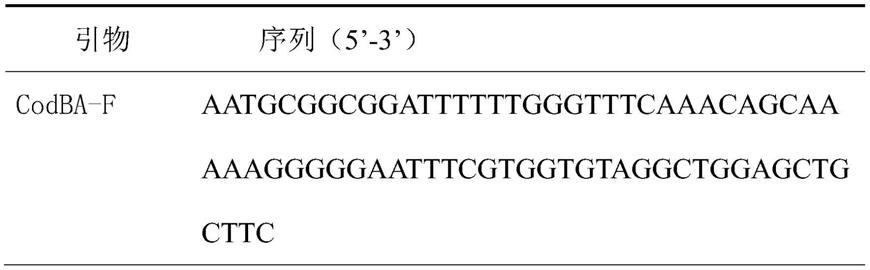
在敲除codab时以pkd4为模板,用下表(引物表格1)引物对 codba-f/codba-r中扩增含50bp同源臂的kan-frt敲除片段,扩增得到的片段电泳无杂带,直接进行过柱回收纯化(捷瑞胶回收纯化试剂盒),得到的片段电转化约200ng加入hf9/pkd46感受态细胞,混匀,转入0.1cm电击杯中,用电击仪作电转化。电击条件:200ω, 25μf,电击电压1.8kv,电击时间为5ms,电击后迅速加入1ml 的lb,37℃,200rpm培养1h,之后涂于含50ng/ml卡那霉素的 lb平板,37℃过夜培养。次日,将电转化得到的转化子进行菌液pcr 验证,引物分别为codba-200f/kan-r、kan-f/codba-200r,确认构建成功hf11k,即hf9(δcodba::kan-frt)。
[0046]
随后对hf11k是否降解胞嘧啶进行了验证。在验证时2g/l的胞嘧啶被添加到接种hf11k摇瓶中,在34℃生长24小时后,经 hplc检测剩余,培养液上清中胞嘧啶的含量2g/l,从而证明敲除 codab得到的重组菌株hf11k不能利用和降解胞嘧啶。
[0047]
表1 codba敲除和验证引物
[0048][0049][0050]
实施例5构建用于核苷水解酶和核苷磷酸化酶表达的重组大肠杆菌
[0051]
使用bl21(de3)作为表达宿主时发现,胞苷在核苷水解酶水解成为胞嘧啶的同时,反应液中的尿嘧啶含量变高,分析可能是由于野生型菌中的胞嘧啶脱氨酶coda没有敲除,从而将由胞苷产生的胞嘧啶降解为尿嘧啶。因此,在本发明中bl21(de3)菌中编码胞嘧啶脱
氨酶的基因(coda)和胞嘧啶转运蛋白的基因(codb)同时被敲除。由于bl21 (de3)和w3110在此段基因簇的序列相同,所以可以利用实施例4 中的引物codba-f/codba-r以及类似方法进行敲除,得到的敲除菌株按照如下过程进行消抗:bl21(de3,
△
codba::kanfrt)接种抗性试管培养过夜进行化转感受态制备,并转化质粒pcp20,涂布含amp抗性的平板。30℃过夜培养后得到的单克隆接种于含20mg/l cm的 lb试管,30℃培养4h后转接到lb无抗性液体培养基试管继续培养4h后升温42℃,培养过夜,然后进行涂布lb平板,得到的单克隆分别点板于氨苄霉素、氯霉素、卡那霉素抗性的lb平板以及无抗lb平板,确认抗性片段消除成功,最后通过引物codba-200f/codba-200r进行菌液pcr验证,得到重组菌株bl21(de3,
△
codba),用于核苷水解酶和核苷磷酸化酶的表达和制备的宿主。
[0052]
实施例6筛选大肠杆菌中的核苷水解酶和核苷磷酸化酶来生产胞嘧啶
[0053]
以大肠杆菌w3110基因组为模板,分别利用下表引物对(表2) 中对应的引物,将riha、rihb、rihc以及ppnp等基因进行扩增,得到的片段分别与酶切的pet28a载体进行连接,转化宿主tg1,通过菌液pcr和酶切验证得到构建成功的表达质粒pet28a-riha、 pet28a-rihb、pet28a-rihc、pet28a-ppnp。这些质粒被通过电转化的方法转化到大肠杆菌bl21(de3,δcodba)菌株中,分别进行过表达。其中,以riha为例,其基因序列详见seq id no 3,其氨基酸序列详见seq id no 4。
[0054]
上述重组菌株过表达流程如下:含50mg/l卡那霉素的lb培养基(green 2012)中活化上述重组大肠杆菌,将培养过夜的菌种分别转接0.8%至50ml含50mg/l卡那霉素的lb培养基,于37℃、200rpm 培养,当生长到od为0.8左右时,添加iptg使其终浓度0.4mm诱导,同时将温度降低到30℃培养,培养过夜后离心收集菌液,用5ml 的50mm ph7.0的pbs缓冲液悬浮细胞。对上述得到的各种酶的重组菌菌体用超声法破碎,破碎条件:开2s,关4s,8min,30%功率,破碎后离心,上清和沉淀分别进行蛋白电泳检测,经过离心后上清酶液直接用于胞苷转化生成胞嘧啶的反应。
[0055]
表2.核苷水解酶构建引物
[0056][0057]
实施例7利用核苷水解酶酶液转化胞苷生成胞嘧啶
[0058]
上述得到的各类核苷水解酶酶液按照如下反应体系和时间进行反应:上述破碎处理的酶液稀释50倍,取100ul加入放有3ml底物的反应管,底物为溶于10mm、ph7.0的pbs缓冲液的浓度为15g/l 的胞苷,总反应体系3.1ml,35度反应过夜,取样进行检测,结果如下表。
[0059]
结果表明riha表达酶液相对酶活最好,对胞苷的水解最彻底,接下来的几个实施例以riha表达酶液为主,来证明胞外酶反应生产胞嘧啶的可行性。
[0060]
表3核苷水解酶水解相对酶活
[0061][0062]
实施例8核苷水解酶(riha)反应条件的优化
[0063]
上述核苷水解酶反应过夜(反应18h)完全转化为胞嘧啶,为了进一步缩短反应时间,提高转化效率,选取核苷水解酶(riha)酶液,对缓冲液浓度、反应ph以及反应温度等进行进一步优化。详细考察如下:
[0064]
1)缓冲液浓度优化:
[0065]
考察pbs浓度在0mm、2mm、10mm、50mm条件下不同时间点的转化率。
[0066]
表4反应体系
[0067][0068]
方法:按上述体系,加入底物a溶液,35℃温浴3min后,加入100ul酶液b,开始计时,搅拌速度1200rpm,不同时间点取100ul反应液加至900ul的0.2%甲酸水中终止反应,hplc检测结果如图3 所示:如图3上表结果所示,缓冲液pbs浓度在2-10mm之间较为适宜。
[0069]
同时自来水代替pbs缓冲液进行实验也成功地将胞苷全部转化为胞嘧啶。
[0070]
2)ph条件优化:
[0071]
考察ph5.2、ph5.5、ph5.7、ph6.0、ph6.2、ph6.5、ph7.0、ph7.5、 ph8.0条件下不同时间点的转化率。反应体系见1)中表4及方法说明。反应结果如图4所示:如图4上表结果所示,反应ph在5.7-6.5 时较为适宜。
[0072]
3)反应温度条件优化:
[0073]
考察不同温度条件下,不同时间点的转化率,考察温度35℃、 37℃、40℃、43℃、45℃、47℃、50℃、53℃、55℃、60℃、65℃和 70℃。反应体系见1)中表4及方法说明。反应结果如图5下:如图 5上表结果所示,反应温度在60-65℃较为适宜。
[0074]
综上,核苷水解酶(riha)较佳的反应条件是:缓冲液pbs浓度在2-10mm,反应ph在6.0,反应温度在60-65℃。
[0075]
实施例9在胞苷发酵结束后进行加入核苷水解酶(riha)生产胞嘧啶
[0076]
按照实施例2方式进行上罐发酵生产胞苷,发酵结束后直接向发酵罐中加入一定量的核苷水解酶(riha)酶液,用氨水调节罐中ph 到6.0,升高发酵液的温度到60℃,反应10h后进行取样检测。检测结果显示发酵液中的胞苷已完全水解成胞嘧啶。
[0077]
实施例10构建生产胞嘧啶的重组质粒
[0078]
上述关于核苷水解酶riha酶液的体外反应实施例显示此酶的活力很高,因此,设计将riha基因构建到phs01-nudg-phm8-pyre-pyrhm 上,然后转化宿主hf11进行摇瓶发酵生产胞嘧啶。
[0079]
此外,之前的专利(申请号201710003833.7)中的菌株 hf9/phs01-nudg-phm8-pyre-pyrhm(cgmcc no.13427)可以在上罐发酵生产胞苷达到20g/l以上,这里的胞苷主要是通过cmp水解得到,说明此菌株生产核苷产品过程中cmp是足够的,因此设计将核苷酸磷酸核苷酶ppnn(基因序列详见seq id no 1,其氨基酸序列详见seqid no 2)构建到phs01-nudg-phm8-pyre-pyrhm上,期望可以通过直接在发酵过程中将cmp水解生成胞嘧啶,不再产生胞苷。
[0080]
例如,分别以w3110基因组为模板,引物对ppnn-f1/ppnn-r1、 riha-f/riha-r(见下表5)扩增片段ppnn和riha,得到的pcr产物电泳检测无杂带,直接进行过柱回收纯化(捷瑞胶回收纯化试剂盒),得到的纯化片段分别与sali酶切回收的 phs01-nudg-phm8-pyre-pyrhm载体片段进行重组克隆构建(苏州神洲基因gbclonart无缝克隆试剂盒),重组克隆反应液于45度水浴锅 30min,然后转移到冰上,加入tg1感受态细胞,42度热击2min,冰浴2min加入复苏培养基lb,复苏培养1h后离心涂布含100mg/l壮观酶素抗性的lb平板,次日挑去克隆培养过夜,提取质粒进行酶切验证,最终构建得到质粒phs01-nudg-phm8-pyre-pyrhm-ppnn和 phs01-nudg-phm8-pyre-pyrhm-riha。
[0081]
表5 riha和ppnn-1扩增引物
[0082][0083]
实施例11构建基因重组菌种直接发酵生产胞嘧啶
[0084]
将表达质粒phs01-nudg-phm8-pyre-pyrhm-riha转化宿主 hf11k,得到重组菌株hf11k/phs01-nudg-phm8-pyre-pyrhm-riha。按照实施例2进行摇瓶发酵,发酵24h后进行取样检测,可以检测到胞嘧啶0.37g/l,胞苷0.02g/l。重组菌株 hf11k/phs01-nudg-phm8-pyre-pyrhm-riha简写为菌株hf11k145,其分类命名为大肠埃希氏菌(escherichia coli.),该菌株已于2019 年05月08日保藏于中国微生物菌种保藏管理委员会普通微生物中心,地址:北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所,邮编:100101,保藏编号为cgmcc no.17729。
[0085]
将表达质粒phs01-nudg-pyrhm-pyre-ppnn转化宿主hf11k,得到重组菌株hf11k/phs01-nudg-pyrhm-pyre-ppnn。按照实施例2进行摇瓶发酵,发酵24h后进行取样检测,可以检测到胞嘧啶0.08g/l,胞苷0.58g/l,分析是由于此质粒上的phm8表达过高,与ppnn竞争同一个底物,且酶活较高,所以积累的胞嘧啶很少,因此构建质粒 phs01-nudg-pyrhm-pyre-ppnn,然后转化宿主hf11进行摇瓶发酵生产胞嘧啶。
[0086]
例如,以w3110基因组为模板,引物对ppnn-f2/ppnn-r2(见下表6)扩增片段ppnn-2,得到的pcr产物电泳检测无杂带,直接进行过柱回收纯化(捷瑞胶回收纯化试剂盒),得到的纯化片段分别与 hindiii酶切回收的phs01-nudg-pyrhm-pyre载体片段进行重组克隆构建(苏州神洲基因gbclonart无缝克隆试剂盒),重组克隆反应液于45度水浴锅30min,然后转移到冰上,加入tg1感受态细胞,42 度热击2min,冰浴2min加入复苏培养基lb,复苏培养1h后离心涂布含100mg/l壮观酶素抗性的lb平板,次日挑去克隆培养过夜,提取质粒进行酶切验证,最终构建得到质粒 phs01-nudg-pyrhm-pyre-ppnn。
[0087]
将表达质粒phs01-nudg-pyrhm-pyre-ppnn转化宿主hf11k,得到重组菌株hf11k/
edition)isbn 978-1-936113-42-2
[0098]
grochowski l.l.(2006)characterization of blsm,a nucleotide hydrolase involved in cytosine production for the biosynthesis of blasticidin s.chembiochem 7:957-964.
[0099]
kim,h.,et al(2006)genes encoding ribonucleoside hydrolase 1 and 2 from corynebacterium ammoniagenes.microbiology 152:1169-1172.
[0100]
kuznetsova,e.,et al(2015)functional diversity of haloacid dehalogenase superfamily phosphatases from saccharomyces cerevisiae biochemical,structural,and evolutionary insights.j boil.chem. 290(30):18678-18698.
[0101]
leung,h.b.et al(1980)adenylate degradation in escherichia coli.the role of amp nucleosidase and properties of the purified enzyme.j bio chem 255(22):10867-10874.
[0102]
petersen,c.,et al(2001)the riha,rihb,and rihc ribobucleoside hydrolase of escherichia coli.j.bio.chem 276(2):884-894.
[0103]
mitterbauer,r.,et al(2001)saccharomyces cerevisiae urh1(encoding uridine-cytidine n-ribohydrolase):functional complementation by a nucleoside hydrolase from a protozoan parasite and by a mammalian uridine phosphorylase.appl environ microbiol 68(3):1336-1343.
[0104]
sevin,d.c,et al 2017 nontargeted in vitro metabolomics for high-throughput identification of novel enzymes in escherichia coli。 nature methods 14(2)187-194.
[0105]
zhao,g.,et al(2014)structure of the n-glycosidase milb in complex with hydroxymethyl cmp reveals its arg23 specificially recognizes the substrate and control its entry.nucleic acids research 42(12):8115-8124.
[0106]
seq id no 1
[0107]
atgattacacatattagcccgcttggctccatggatatgttgtcg cagctggaagtggatatgcttaaacgcaccgccagcagcgacc tctatcaactgtttcgcaactgttcacttgccgtactgaactcc ggtagtttgaccgataacagcaaagaattgctgtctcgttttga aaatttcgatattaacgtcttgcgccgtgaacgcggcgtaaagc tggaactgattaatcccccggaagaggcttttgtcgatgggcga attattcgcgctttgcaggccaacttgttcgcggtcctgcgtga cattctcttcgtttacgggcaaatccataacaccgttcgttttcc caacctgaatctcgacaactccgtccacatcactaacctggtct tttccatcttgcgtaacgctcgcgcgctgcatgtgggtgaagcg ccaaatatggtggtctgctggggcggtcactcaattaacgaaa acgagtatttgtatgcccgtcgcgtcggaaaccagctgggcctg cgtgagctgaatatctgcaccggctgtggtccgggagcgatgg aagcgccgatgaaaggtgctgcggtcggacacgcgcagcagc gttacaaagacagtcgttttattggtatgacagagccgtcgatt atcgccgctgaaccgcctaacccgctggtcaacgaattgatcat catgccagatatcgaaaaacgtctggaagcgtttgtccgtatcg ctcacggtatcattatcttccctggcggtgtgggtacggcagaa gagttgctctatttgctgggaattttaatgaacccggccaacaa agatcaggttttaccattgatcctcaccggcccgaaagagagc gccgactacttccgcgtactggacgagtttgtcgtgcatacgct gggtgaaaacgcgcgccgccattaccgcatcatcattga
tgacg ccgctgaagtcgctcgtcagatgaaaaaatcgatgccgctggt gaaagaaaatcgccgtgatacaggcgatgcctacagctttaact ggtcaatgcgcattgcgccagatttgcaaatgccgtttgagccg tctcacgagaatatggctaatctgaagctttacccggatcaacc tgttgaagtgctggctgccgacctgcgccgtgcgttctccggta ttgtggcgggtaacgtaaaagaagtcggtattcgcgccattga agagtttggtccttacaaaatcaacggcgataaagagattatgc gtcgtatggacgacctgctacagggttttgttgcccagcatcgt atgaagttgccaggctcagcctacatcccttgctacgaaatctg cacgtaa
[0108]
seq id no 2
[0109]
mithisplgsmdmlsqlevdmlkrtassdlyqlfrncslavlnsg sltdnskellsrfenfdinvlrrergvklelinppeeafvdgriira lqanlfavlrdilfvygqihntvrfpnlnldnsvhitnlvfsilrna ralhvgeapnmvvcwgghsineneylyarrvgnqlglrelnict gcgpgameapmkgaavghaqqrykdsrfigmtepsiiaaeppnpl vneliimpdiekrleafvriahgiiifpggvgtaeellyllgilmnpa nkdqvlpliltgpkesadyfrvldefvvhtlgenarrhyriiidda aevarqmkksmplvkenrrdtgdaysfnwsmriapdlqmpfeps henmanlklypdqpvevlaadlrrafsgivagnvkevgiraieef gpykingdkeimrrmddllqgfvaqhrmklpgsayipcyeict
[0110]
seq id no 3
[0111]
atggcactgccaattctgttagattgcgacccaggtcatgacga cgctatcgcaatagttctcgccctcgcctcaccagagcttgatg tcaaagcaattacgtcttccgccggaaaccagacaccagaaaa aaccttacgcaatgttctgcgtatgctgaccttgcttaatcgcac cgatattccggtagcaggcggcgcggtaaaaccgttaatgcgtg agttgattatcgcggacaatgtgcacggcgaaagcggtctcga cggcccggcattaccggaaccgacattcgcaccgcaaaactgt acggcggtagagctgatggcgaaaacgctgcgtgaaagtgcg gaacctgtcaccattgtgtctaccggaccgcaaactaacgttgc cttgctgctcaatagccatccggaactgcatagcaaaattgccc gtatcgtgattatgggtggcgctatggggctgggtaactggacg cctgcggctgaatttaatatttacgttgacccggaagcggcaga aattgtcttccagtcagggatcccggtggtgatggccggtctgg atgttactcataaagcacaaatccacgttgaagacaccgagcg tttccgcgcgattggtaaccctgtttcaaccattgttgccgaac tgctggatttcttcctcgaatatcataaagacgaaaaatggggc tttgtcggcgcaccactgcatgacccatgcaccatcgcctggct gttgaaaccggagttatttacctctgttgagcgctgggttggcg tggaaacacaggggaaatatacccagggtatgacggttgttgat tattattatctgacaggcaataaaccgaatgccaccgtaatggtc gatgttgatcgtcagggctttgttgatttactggctgatcgtctg aaattttacgcttaa
[0112]
seq id no 4
[0113]
malpilldcdpghddaiaivlalaspeldvkaitssagnqtpektl rnvlrmltllnrtdipvaggavkplmreliiadnvhgesgldgpal peptfapqnctavelmaktlresaepvtivstgpqtnvalllnshp elhskiarivimggamglgnwtpaaefniyvdpeaaeivfqsgipv vmagldvthkaqihvedterfraignpvstivaelldffleyhkd ekwgfvgaplhdpctiawllkpelftsverwvgvetqgkytqgm tvvdyyyltgnkpnatvmvdvdrqgfvdlladrlkfya 。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1