一种香菇L952菌株的InDel标记指纹图谱及其构建方法与流程
本发明属于香菇菌种的检测领域,具体涉及一种香菇l952菌种的indel标记指纹图谱及其构建方法。
背景技术:
香菇(lentinulaedodes(berk.)pegler)因具有较高的营养价值和药用价值,而备受消费者欢迎。中国是世界上最早进行香菇人工栽培的国家,栽培历史已有800多年,目前香菇是国内栽培最广、产量最高的菇种。据中国食用菌协会统计,2018年中国食用菌总产量为3842.04万吨,其中香菇总产量为1043.12万吨,占国内食用菌总产量的27.15%,占世界香菇总产量的90%以上。近年来香菇产业成为国内精准扶贫的重要抓手,据统计全国超过50%以上的国家级贫困县选择香菇作为重要产业,我国香菇栽培规模进一步扩大,但随着我国香菇产业的快速发展,香菇生产用种“同种异名,异种同名”的问题日益突出,这不仅损害了香菇育种者的知识产权更为生产者造成了极大地风险,为香菇产业进一步发展造成了隐患。为保障育种者的权益和降低生产者的风险,促进我国香菇产业健康、稳定和可持续发展,建立一种简便、快速、可靠的香菇菌种特异性鉴定技术迫在眉睫。
随着生物信息学的快速发展,dna测序技术的不断成熟,测序的通量在不断增加而成本在降低。dna分子标记技术能够从基因水平上检测香菇菌种的遗传特异性,香菇全基因组测序的完成为indel标记的开发提供了便利。indel分子标记是一种基于高通量测序技术的应用,降低了indel标记开发的成本,适用于香菇全基因组测序分子标记的开发。且indel标记具有数量丰富、准确性高、稳定性好、快捷简便等优点,不仅用于分子标记辅助育种,而且也可用于香菇菌株鉴定。
技术实现要素:
本发明所要解决的技术问题是提供一种香菇l952菌种的indel标记指纹图谱及其构建方法与应用,该指纹图谱与常规形态学检测、拮抗试验、出菇试验相比,具有检测时间短,准确性高,重复性好的优点。
本发明的一种香菇l952菌种的indel标记指纹图谱,该指纹图谱由7对indel标记组成,是基于香菇基因组插入/缺失片段开发的indel标记引物,扩增带型好,重复性高,标记详细信息如表1。
表1indel标记引物信息列表
本发明还提供了一种香菇l952菌种的indel标记指纹图谱的应用,是利用香菇基因组插入/缺失片段开发的7对indel标记引物,对香菇菌种进行indel标记扩增,将得到的带型与l952菌种的带型对照,与该带型一致即为香菇l952菌种;
其中香菇l952菌种的带型编号组合为:12(1+2)1(1+2)1(1+2)。
通过对收集的44个香菇品种的indel标记引物带型扩增,本发明确定了7对indel标记引物在44个香菇品种中扩增出的等位片段的数量并编号(表2),通过不同indel等位位点的编号组合可有效识别l952菌种(图2-8)。通过dna分子量对照d2000bpdnaladder可确定各indel标记引物扩增的等位位点的相对分子量,存在l952菌种特异indel等位片段组合的菌种,即是香菇l952菌种,该菌种的编号组合为:12(1+2)1(1+2)1(1+2)。
表2indel引物扩增的等位片段信息汇总表
本发明还提供了上述香菇l952菌种的indel标记指纹图谱的构建方法,该方法包括如下步骤:
(1)菌丝培养:将香菇菌丝转接到马铃薯葡萄糖培养基pda中,23~25℃下避光培养,10d后收集菌丝;
(2)基因组dna的提取:用ctab法提取上述菌丝的基因组dna,紫外分光光度法检测总基因组dna浓度和纯度,调整样品dna的浓度为20~30ng/ul;
ctab法提取菌丝的基因组dna工艺包括:
①取冷冻干燥后的香菇菌丝于2ml离心管中,并放入2-3颗钢珠;
②将离心管放入多样品组织研磨机中,设置频率60hz,时间30s研磨至均匀粉末;
③加入65℃预热1h的2×ctab抽提液,于65℃保温60min,间隔20min轻摇混匀一次;
④12000rpm、4℃离心20min,分装上清液,分装两管各800ul;
⑤在上述上清液中加入体积比为25:24:1的酚、氯仿和异戊醇的混合液,轻轻混匀10min,12000rpm、4℃离心10min,取上清液移入新的离心管中;
⑥在上述离心管中加入体积比为24:1的氯仿和异戊醇混合液,轻轻混匀10min,12000rpm、4℃离心10min,取上清液(记录体积)移入另一离心管中;
⑦在上述离心管中加入相对于步骤⑥中的上清液2/3体积的-20℃预冷的异丙醇,混匀,-20℃下静置沉淀1h,之后8000rpm、4℃离心10min,弃上清;
⑧将得到的沉淀加入1ml浓度为70%乙醇洗涤1次,8000rpm,室温离心5min;
⑨弃乙醇,超净工作台中吹干;加入100μl10×te缓冲液,轻轻敲打使沉淀溶解,加入1μl10mg/ml的rnasea于37℃水浴1h,去除rna;
⑩将得到的dna提取物于-20℃冰箱贮藏备用;
indel分子标记的检测:对上述提取的dna进行全基因组indel标记的pcr扩增;pcr扩增体系为:总体积20μl,包括:premixtaqtm(1.25u/25μltaqdna酶,0.4mm/ldntp,0.3mm/lpcrbuffer)10μl,10μmol/lindel标记正向引物和反向引物总体积各1μl,浓度20~30ng/μl提取的模板dna2μl,ddh2o6μl;
pcr反应条件:94℃5min;94℃45second,55-60℃45second,72℃30second,35个循环;72℃7min;10℃保存;
电泳检测:将上述pcr扩增得到的产物,点样7μl于加入核酸染料的琼脂糖凝胶上电泳,琼脂糖凝胶的体积百分比浓度为2.5%,电泳缓冲液为1×tae,电压90v,电流300ma,功率100w,电泳5h,拍照,分析结果;
采用7对indel标记引物对香菇菌株进行pcr扩增,通过dna分子量对照d2000bpdnaladder可确定各indel标记引物扩增的等位片段的数量和相对分子量,找到符合编号组合为:12(1+2)1(1+2)1(1+2)的菌种,即可确定该菌种为香菇l952菌种。
本发明的有益效果
本发明的香菇l952菌种的indel标记指纹图谱可用于l952菌种的鉴别,与常规形态学检测、拮抗试验、出菇试验相比,具有检测时间短,准确性高,重复性好的优点。检测所需时间只需要3-4天,而常规的拮抗试验所需时间至少需要两周时间,出菇试验则需要至少3个月的时间;该方法在所收集的我国44个香菇菌种中具有l952菌种的专一性,具有良好的应用前景。
附图说明
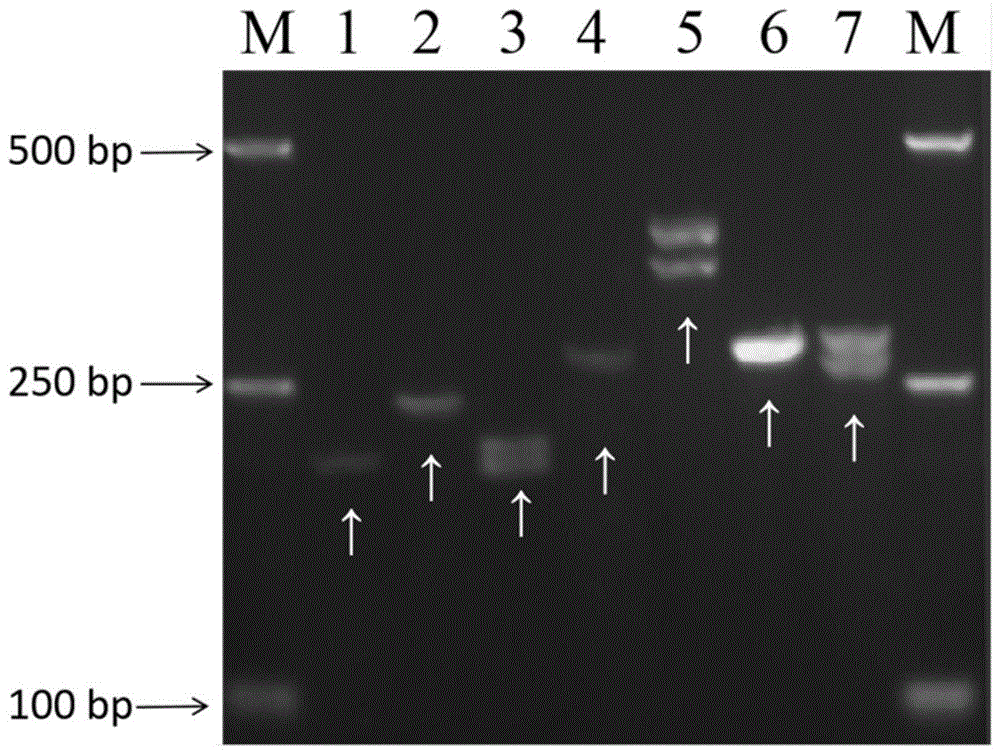
图1为香菇l952菌种indel标记指纹图谱,其中m是d2000bpdnaladder,数字1-7代表的是所用的7对indel标记引物,箭头所指的是香菇l952菌种的特异性indel等位片段组合;
图2为引物lefp_id001在所选44个香菇材料中的扩增图谱;
图3为引物lefp_id002在所选44个香菇材料中的扩增图谱;
图4为引物lefp_id003在所选44个香菇材料中的扩增图谱;
图5为引物lefp_id004在所选44个香菇材料中的扩增图谱;
图6为引物lefp_id005在所选44个香菇材料中的扩增图谱;
图7为引物lefp_id006在所选44个香菇材料中的扩增图谱;
图8为引物lefp_id007在所选44个香菇材料中的扩增图谱。
具体实施方式
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。此外应理解,在阅读了本发明讲授的内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
marker、premixtaqtm均购自:takalabio宝日医生物技术有限公司
其余材料和试剂均为普通市售产品。
44个菌株来源:
1、cv108菌株,来源上海市农业科学院食用菌研究所;2、香九菌株,来源广东省微生物研究所;3、l7402菌株,来源松阳县食药用菌研究所;4、8404菌株,来源湖北省宜昌森源食用菌有限公司;5、华香5号菌株,来源华中农业大学;6、cv442菌株,来源上海市农业科学院食用菌研究所;7、广香菌株,来源广东省微生物研究所;8、l135菌株,来源福建省三明真菌研究所;9、l9319菌株,来源浙江省丽水市大山菇业研究开发有限公司;10、闽丰1号菌株,来源福建省三明真菌研究所;11、森源1号菌株,来源湖北省宜昌森源食用菌有限责任公司;12、申香18号菌株,来源上海市农业科学院食用菌研究所;13、香如菌株,来源上海市农业科学院食用菌研究所;14、申香15号菌株,来源上海市农业科学院食用菌研究所;15、cv105菌株,来源上海市农业科学院食用菌研究所;16、l241菌株,来源上海市农业科学院食用菌研究所;17、香杂26菌株,来源广东省微生物研究所;18、l9608菌株,河南西峡县食用菌科研中心;19、z3244菌株,来源上海市农业科学院食用菌研究所;20、申香16号菌株,来源上海市农业科学院食用菌研究所;21、华香8号菌株,来源华中农业大学;22、n7菌株,来源上海市农业科学院食用菌研究所;23、rbl1菌株,来源上海市农业科学院食用菌研究所;24、z3210菌株,来源上海市农业科学院食用菌研究所;25、沪香f2菌株,来源上海市农业科学院食用菌研究所;26、l241-4菌株,来源浙江省庆元县食用菌科学技术研究中心;27、l26菌株,来源福建省三明真菌研究所;28、菌兴8号菌株,来源浙江省丽水市食用菌研究开发中心;29、cv501菌株,来源上海市农业科学院食用菌研究所;30、苏香菌株,来源上海市农业科学院食用菌研究所;31、l952菌株,来源华中农业大学;32、z3239菌株,来源上海市农业科学院食用菌研究所;33、z3243菌株,来源上海市农业科学院食用菌研究所;34、hk3161菌株,来源上海市农业科学院食用菌研究所;35、l212菌株,来源上海市农业科学院食用菌研究所;36、l868菌株,来源上海市农业科学院食用菌研究所;37、tw1151菌株,来源上海市农业科学院食用菌研究所38、l9105菌株,来源浙江省庆元县食用菌科学技术研究中心;39、申香12号菌株,来源上海市农业科学院食用菌研究所;40、j868菌株,来源上海市农业科学院食用菌研究所;41、森源10号菌株,来源湖北省宜昌森源食用菌有限责任公司;42、wd4204菌株,来源上海市农业科学院食用菌研究所;43、wd5140菌株,来源上海市农业科学院食用菌研究所;44、931菌株,来源上海市农业科学院食用菌研究所。
其中7对indel标记引物序列(5′-3′)如下:
lefp_id001正向引物:acggatggagagacacagga
反向引物:tgccccacttactctcaacc
lefp_id002正向引物:cctttctcaaaagcggactg
反向引物:gggagtgggttgtttggata
lefp_id003正向引物:cggatgttatgcactggaag
反向引物:cgtacggttggacatttgaa
lefp_id004正向引物:agcctttcacagtcagctcg
反向引物:gtcaacggagggaaacagag
lefp_id005正向引物:gtagcactcctcatacaacc
反向引物:accaaatgtcacagcacagg
lefp_id006正向引物:acattggcgaaggctgtacg
反向引物:cccttttgccctataaggcg
lefp_id007正向引物:aacagtaacctgtgcactgc
反向引物:catgatcagatcacacagcg。
引物由上海生工生物工程有限公司合成。
实施例1
(1)菌丝培养:将香菇菌丝转接到马铃薯葡萄糖培养基pda中,23~25℃下避光培养,10d后收集菌丝;
(2)基因组dna的提取:用ctab法提取上述菌丝的基因组dna,紫外分光光度法检测总基因组dna浓度和纯度,调整样品dna的浓度为20~30ng/ul;
ctab法提取菌丝的基因组dna工艺包括:
①取冷冻干燥后的香菇菌丝于2ml离心管中,并放入2-3颗钢珠;
②将离心管放入多样品组织研磨机中,设置频率60hz,时间30s研磨至均匀粉末;
③加入65℃预热1h的2×ctab抽提液,于65℃保温60min,间隔20min轻摇混匀一次;
④12000rpm、4℃离心20min,分装上清液,分装两管各800ul;
⑤在上述上清液中加入体积比为25:24:1的酚、氯仿和异戊醇的混合液,轻轻混匀10min,12000rpm、4℃离心10min,取上清液移入新的离心管中;
⑥在上述离心管中加入体积比为24:1的氯仿和异戊醇混合液,轻轻混匀10min,12000rpm、4℃离心10min,取上清液(记录体积)移入另一离心管中;
⑦在上述离心管中加入相对于步骤⑥中上清液2/3体积的-20℃预冷的异丙醇,混匀,-20℃下静置沉淀1h,之后8000rpm、4℃离心10min,弃上清;
⑧将得到的沉淀加入1ml浓度为70%乙醇洗涤1次,8000rpm,室温离心5min;
⑨弃乙醇,超净工作台中吹干。加入100μl10×te缓冲液,轻轻敲打使沉淀溶解,加入1μl10mg/ml的rnasea于37℃水浴1h,去除rna;
⑩将得到的dna提取物于-20℃冰箱贮藏备用;
(3)indel分子标记的检测:对上述提取的dna进行全基因组indel标记的pcr扩增;
pcr扩增体系为:总体积20μl,包括:premixtaqtm(1.25u/25μltaqdna酶,0.4mm/ldntp,0.3mm/lpcrbuffer)10μl,10μmol/lindel标记正向引物和反向引物总体积各1μl,浓度20~30ng/μl提取的模板dna2μl,ddh2o6μl;
pcr反应条件:94℃5min;94℃45second,55-60℃45second,72℃30second,35个循环;72℃7min;10℃保存;
(4)电泳检测:将上述pcr扩增得到的产物,点样7μl于加入核酸染料的琼脂糖凝胶上电泳,琼脂糖凝胶的体积百分比浓度为2.5%,电泳缓冲液为1×tae,电压90v,电流300ma,功率100w,电泳5h,拍照,分析结果;
采用7对indel标记引物分别对香菇菌株进行pcr扩增,通过dna分子量对照d2000bpdnaladder可确定各indel标记引物扩增的等位片段的数量和相对分子量,如图1所示,找到符合编号组合为:12(1+2)1(1+2)1(1+2)的菌种,即可确定该菌种为香菇l952菌种,图2-8中的编号31即为l952菌种,其条带与图1的一致。
序列表
<110>上海市农业科学院
<120>一种香菇l952菌株的indel标记指纹图谱及其构建方法
<160>14
<170>siposequencelisting1.0
<210>1
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>1
acggatggagagacacagga20
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
tgccccacttactctcaacc20
<210>3
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>3
cctttctcaaaagcggactg20
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
gggagtgggttgtttggata20
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
cggatgttatgcactggaag20
<210>6
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>6
cgtacggttggacatttgaa20
<210>7
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>7
agcctttcacagtcagctcg20
<210>8
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>8
gtcaacggagggaaacagag20
<210>9
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>9
gtagcactcctcatacaacc20
<210>10
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>10
accaaatgtcacagcacagg20
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
acattggcgaaggctgtacg20
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
cccttttgccctataaggcg20
<210>13
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>13
aacagtaacctgtgcactgc20
<210>14
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>14
catgatcagatcacacagcg20
- 还没有人留言评论。精彩留言会获得点赞!