与玉米大斑病抗性相关的ZmGNAT19基因SNP分子标记及应用
与玉米大斑病抗性相关的zmgnat19基因snp分子标记及应用
技术领域
1.本发明属于作物遗传育种分子标记应用技术领域,具体涉及与玉米大斑病抗性相关的zmgnat19基因snp分子标记及应用。
背景技术:
2.玉米大斑病(northern corn leaf blight)是由大斑突脐蠕孢菌(exserohilum turcicum)引起的一种产生大型病斑症状为主的严重叶枯性病害。病斑可以沿叶脉迅速扩展并不受叶脉限制,形成条形或长梭形病斑,严重时病斑融合,造成叶片大面积枯死,导致产量大幅度降低。一般流行年份大斑病导致玉米减产20%,严重发生年份的易感品种减产50%以上。玉米大斑病抗性品种的选育与推广不仅可降低发病率,减少产量损失,也可以减少化学药剂防治对生态环境的破坏,还可以减少生产成本投入,提高玉米生产的经济效益。过往对玉米大斑病质量抗性基因ht1、ht2、ht3、htn的qtl定位研究取得了一定的进展,将它们分别定位于玉米染色体的特定区域即2.07、8.05、7.04、8.06,但ht基因至今没有被克隆的报道(王贺等,玉米科学,2011)。
3.常规育种中将分散于各个种质中的优良基因聚合于同一个体的选择针对性不强,培育优良新品种的过程缓慢、难度较大。分子标记由于能对基因型进行直接的选择,具有快速、准确的优点。将分子标记技术与常规育种相结合,即分子标记辅助选择培育作物新品种正引起广泛的重视。随着玉米基因组测序的完成,玉米遗传图谱精度的提高,分子标记辅助基因聚合正日益体现出巨大的优势和应用前景。但目前与玉米大斑病抗性相关的分子标记在数量与质量上都无法满足生产实践的需求。单核苷多态性(snp)在玉米基因组中广泛存在,snp遗传稳定,检测方便,基于snp的全基因组关联分析(genome
‑
wide association study,gwas)广泛应用于作物重要农艺性状遗传位点的检测,可以满足gwas对于大样本、高密度标记的要求。与传统的qtl相比,gwas具有更高的分辩率,可以更准确的识别和定位与目标性状相关联的新的基因,精确的筛选出相关农艺性状的分子标记从而应用于作物遗传育种领域。
4.蛋白质乙酰化是蛋白质翻译后修饰的一种,由乙酰转移酶和去乙酰化酶共同调控,细胞核内,组蛋白的乙酰化与去乙酰化过程处于动态平衡之中,可以精确地调控基因的转录和表达,新近的研究还证明,非组蛋白的乙酰化也广泛参与了机体生理或病理过程。zmgnat19基因(zea mays gcn5
‑
related n
‑
acetyltransferase)即gcn5相关的n端乙酰基转移酶超家族成员,编号zm00001d033620。最初在酵母中发现gcn5(general control non
‑
derepressible5)可以常规调控氨基酸合成信号通路而得名,后来证明gcn5是一种与基因转录相关的组蛋白乙酰转移酶(hats),在不同的物种中gcn5进化上相当保守(xin liu et al.,jbc,2013)。gnat催化乙酰基从乙酰辅酶a转移到多种底物上,广泛的参与生命的各种活动。zhongze li等(frontiers in plant science,2020)研究表明,在莱茵衣藻中crgnat19基因的过表达可以显著的提高微藻淀粉含量和生物学产量,表明该基因在生物技术方面具有巨大的应用潜力。孙淑豪等(广西植物,2017)研究表明,拟南芥中组蛋白去乙酰
化酶athda19基因的缺失突变体对灰葡萄孢(botrytis cineara)的基础防御显著增强,抗病相关基因表达上调。liang kong等(current biology,2017)研究表明,疫霉菌(phytophthora)的效应因子psavh23可以降低gcn5的酶活性,减弱对寄主植物h3k9的乙酰化,使防卫基因表达量下降进而削弱植物抵御病害的能力。bo ding等(plant cell,2012)研究表明,水稻中去乙酰化酶oshdt701基因的过量表达,转基因株系中组蛋白h4的乙酰化水平降低,导致对稻瘟病菌的(magnaporthe oryzae)抗性降低。
5.但至今为至,人们对玉米zmgnat19基因的作用与功能了解是非常有限的,zmgnat19基因参与玉米大斑病的病程进展相关的文献更是鲜见报道。通过gwas研究自然群体中突变位点与性状的关联,进而探究未知基因的功能是一个非常有效的技术手段。
技术实现要素:
6.本发明的目的在于克服现有技术的缺陷,提供两个与玉米大斑病抗性极显著相关的zmgnat19基因snp分子标记,作为玉米育种过程中大斑病抗性种质筛选的辅助选择分子标记,以提高选择的准确性、加快育种进程。
7.本发明的与玉米大斑病抗性相关的zmgnat19基因snp分子标记,其位于玉米第1号染色体zmgnat19基因的3’utr区域,其核苷酸序列如seq id n01所示,163位的碱基y为c或t。
8.本发明的与玉米大斑病抗性相关的zmgnat19基因snp分子标记,其位于玉米第1号染色体zmgnat19基因的3’utr区域,其核苷酸序列如seq id n01所示,178位碱基s为g或c。
9.本发明的与玉米大斑病抗性相关的zmgnat19基因snp分子标记,能在玉米大斑抗病品种辅助选择中应用,其基因型c/c为大斑病极显著的抗病分子标记,其基因型c/t和t/t为大斑病极显著的易感分子标记。
10.本发明的与玉米大斑病抗性相关的zmgnat19基因snp分子标记,能在玉米大斑抗病品种辅助选择中应用,其基因型g/g为大斑病极显著的抗病分子标记,其基因型c/c和c/g为大斑病极显著的易感分子标记。
11.本发明的与玉米大斑病抗性相关的zmgnat19基因snp分子标记的操作过程如下:
12.1.提取玉米基因组dna;
13.2.简化基因组测序,检出高质量的候选标记;
14.3.使用软件gapit(3.1.0)中的cmlm模型(compressed mixed linear model)对玉米大斑病病情进展曲线下的面积(audpc)进行关联分析;
15.4.检出的显著位点不同基因型组间比较。
16.本发明与玉米大斑病抗性相关的zmgnat19基因snp分子标记,运用全基因组关联分析的方法,研究证明zmgnat19基因的序列多态性与玉米大斑病audpc存在极显著的关联,即该基因的3’utr区域,位于玉米第1号染色体chr1:269012623和chr1:269012638位置存在与玉米大斑病抗性相关的两个zmgnat19基因snp分子标记。
17.chr1:269012623位点基因型c/c与(c/t+t/t)之间audpc差异极显著(p<0.0001),组间表型均值差为3.573,基因型c/c为大斑病极显著的抗病分子标记,基因型c/t和t/t为大斑病极显著的易感分子标记。
18.chr1:269012638位点基因型g/g与(c/c+c/g)之间audpc差异极显著(p<0.0001),
组间表型均值差为3.573,基因型g/g为大斑病极显著的抗病分子标记,基因型c/c和c/g为大斑病极显著的易感分子标记。
19.以此作为玉米大斑病抗性选择的辅助分子标记,能提高选择的准确性、加快育种进程。
附图说明
20.图1为玉米大斑病audpc次数分布的方柱形图
21.图2为玉米基因组dna检测的部分电泳图
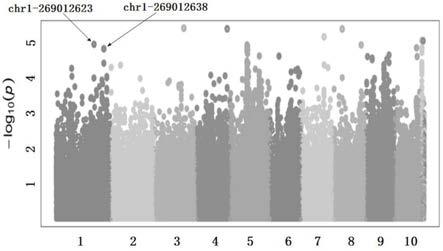
22.图3为玉米大斑病audpc全基因组关联分析的曼哈顿图
23.其中:横坐标代表每个snp所在的基因组位置;纵坐标代表cmlm模型下每个snp位点p值的以10为底的负对数。
24.图4为玉米大斑病audpc的全基因组关联分析的qq图
25.其中:横坐标代表假设p值服从均匀[0,1]分布,所预期观察的p值以10为底的负对数;纵坐标代表观察到p值以10为底的负对数。
[0026]
图5为zmgnat19基因chr1:269012623位点不同基因型audpc的箱形图
[0027]
图6为zmgnat19基因chr1:269012638位点不同基因型audpc的箱形图
具体实施方式
[0028]
实施例1
[0029]
1.1gwas群体的构建
[0030]
玉米全基因组关联分析群体包括431份不同亲缘关系的自交系。所有的自交系都由吉林大学植物科学学院提供,种植于吉林大学植物科学学院教学与科研实验基地(吉林省长春市绿园区)。小区排列遵循随机区组设计,区组3次重复,单行小区,行长3m,行距65厘米,株距20厘米,田间管理措施与大田相同。
[0031]
1.2样品的采集
[0032]
小区玉米幼苗出苗后的5
‑
6叶期,采集3株幼苗,清洗干净后置于
‑
20℃冰箱冻存。
[0033]
1.3病叶病情分级
[0034]
玉米病叶病情分级的鉴定依据地方标准:db52t1501.9
‑
2020
[0035]
0:叶片上无病斑
[0036]
1:叶片上有零星病斑,病斑占叶面积少于或等于5%
[0037]
3:叶片上有少量病斑,占叶面积6%
‑
10%
[0038]
5:叶片上有较多病斑,占叶面积11%
‑
30%
[0039]
7:叶片上有大量病斑,病斑相连,占叶面积31%
‑
70%
[0040]
9:叶片病斑占叶面积70%以上,叶片枯死。
[0041]
在8月上旬玉米授粉后每个小区调查3株植株,每株调查果穗上下各2片叶的发病情况,病情指数的计算方法如下:
[0042][0043]
1.4病情进展曲线下面积(audpc)
[0044]
病情进展曲线的计算方法参考yang等(bio
‑
protocol,2017),并进行一定改良。
[0045][0046]
primdla:ine primary aiseasea leaf area,即初生病叶的病斑面积,p1、p2…
p
n
为同一处理中各玉米植株的病情指数,n为一个处理调查的总株数即3株。
[0047][0048]
audpc:the area under the disease progress curve,病情进展曲线下面积,用以评价玉米种质的抗病性。y
i
=primdla,t
i+1
‑
t
i
=两次调查间隔天数,n为调查次数,本试验中t
i+1
‑
t
i
=7天;n=4次。以区组三次重复的平均值作为gwas分析的表型数据,调查数据采用excel2013软件进行整理。
[0049]
1.5玉米基因组dna的提取与检测
[0050]
(1)取50
‑
100mg玉米幼苗用液氮研磨成粉末,转移至1.5ml离心管中,加入400ul buffer pcl,8ul β
‑
巯基乙醇,震荡混匀。65℃水浴45min至样品完全裂解。
[0051]
(2)加入200ul buffer pp,充分颠倒混匀,
‑
20℃冰箱放置5min,室温10000rpm离心5min,将上清液(500
‑
550u1)转移到新的1.5ml离心管中。若上清液混浊,可再加入等体积氯仿混匀,12000rpm离心取上清。
[0052]
(3)加入等体积的异丙醇,颠倒5
‑
8次使之充分混匀,室温放置2
‑
3min。室温10000rpm离心5min,弃上清。
[0053]
(4)加入1ml75%乙醇,颠倒漂洗1
‑
3min,10000rpm离心2min,弃上清。重复上述操作一次,开盖室温倒置5
‑
10min至残留的乙醇完全挥发。
[0054]
(5)得到的dna用50
‑
100ul te buffer溶解。提取的dna可立即进行下一步实验或
‑
20℃保存。
[0055]
(6)1%琼脂糖凝胶(200v电泳30min)检测dna完整性,qubit2.0定量检测dna样本浓度。
[0056]
1.6研究结果
[0057]
供试群体玉米大斑病audpc样本均值5.59,中位数为5.26,平均偏差2.94,极差17.92,标准差3.71,变异系数0.664,95%置信区间为5.22
‑
5.96级,audpc次数分布见图1。
[0058]
玉米幼苗基因组dna采用rapid p1ant genomic dna isolation kit提取后,在裂解液作用下裂解消化rna,经有机相洗脱蛋白等杂质,得到纯净dna,dna样本经电泳检测合格后(>10ng/ul),可用于基因组文库构建,电泳检测结果见图2。
[0059]
实施例2
[0060]
2.1测序文库的构建
[0061]
(1)200ng基因组dna用限制性内切酶ecori酶切,酶切消化完全后磁珠纯化回收。
[0062]
(2)酶切后纯化的dna用t4 dna ligase连接barcode adapters pi,连接产物磁珠纯化回收,记录合格的p1引物标签。
[0063]
(3)所有样本按要求等比例混合为一个总dna mix,使用covaris220将dna片段化,打断后的dna的碎片长度约为200
‑
500bp。
[0064]
(4)end repair&da
‑
tailing后,连接adaptor p2,连接产物磁珠纯化回收。
[0065]
(5)利用kapa 2g robust pcr kit扩增并富集接头连接产物,利用磁珠对pcr反应产物进行纯化分选,通过2%琼脂糖凝胶电泳检测构建完成的文库pcr纯化产物质量,合格的pcr产物进行二代测序。
[0066]
2.2简化基因组测序(restriction site
‑
associated dna sequencing,rad)
[0067]
(1)采用illumina novaseq6000测序平台进行双末端测序(pe150),读长(reads)为2
×
150bp。
[0068]
(2)产出数据的质控与过虑:对碱基检出的错误发生几率进行预测,如果质量分值(q
‑
score)为低质量(q≤5(e))的碱基数占整条read的一半以上,则去掉该read,同时还去除用于样品识别的标签序列。
[0069]
(3)利用stacks
‑
1.08(http://creskolab.uoregon.edu/stacks/)分别对所有样品进行stack聚类分析,检测获得各样本tag序列,以及snp信息。
[0070]
(4)数据比对以及snp
‑
callling:采用bwa软件(版本0.7.17
‑
r1188)进行基因组比对,将质控合格的数据比对b73参考基因组序列。
[0071]
2.3基因型检测与gwas
[0072]
(1)变异检测和筛选:使用picard软件标记重复,samtools(1.9)对bam进行排序,bcftools(1.9)进行cail snp。
[0073]
(2)snp质控:使用质控软件vcf tools(0.1.16)对snp进行质控,质控标准为maf<0.05,缺失率>0.8,hw(哈迪
‑
温伯格指数)>0.0001。
[0074]
(3)snp注释:软件snp eff(版本4.3t)利用基因组结构注释数据(gtf文件)对vcf文件中的snp/indel信息进行注释,即是否能够对基因编码蛋白造成影响,包括snp的突变类型和突变位置,氨基酸的变异类型和效应等。
[0075]
(4)gwas:使用软件gapit(3.1.0)中的cmlm模型(compressed mixed linear model)对性状进行关联分析。
[0076]
2.4研究结果
[0077]
snp质控后,获得549211个合格的snp用于全基因组关联分析,关联分析时基于贝叶斯信息准则(bayesian information criterion,bic)的模型选择,找到gwas模型中最优的pcs数量,用以拟合最优的性状因子。玉米大斑病audpc的gwas分析结果的见图3和图4,图3是关联分析结果的曼哈顿图,x轴是每个snp所在的基因组上的位置,y轴是cmlm模型下每个snp位点p值的以10为底的负对数。图4是关联分析的qq图,y轴是观察到p值以10为底的负对数,x轴是假设p值服从均匀[0,1]分布,所预期观察的p值以10为底的负对数;
[0078]
2.5全基因组关联分析。
[0079]
当检出的snp的p值小于10
‑4时,我们认为这个snp达到了基因组水平的显著,zmgnat19基因(zm00001d033620)的3’utr区域检出2个snp位点符合上述条件,位于玉米第1号染色体chr1:269012623和chr1:269012638位置。生物信息学功能注释表明该基因编码产物是gcn5相关的n端乙酰基转移酶超家族成员(zea mays gcn5
‑
related n
‑
acetyltransferase)。gnat催化乙酰基从乙酰辅酶a转移到多种底物上,广泛的参与生命的各种活动,包括机体的生理与病理过程。
[0080]
结果详见表1。
[0081][0082]
表2zmgnat19基因chr1∶269012623位点大斑病audpc的组间均值比较
[0083][0084]
3.2zmgnat19基因chr1:269012638位点组间比较
[0085]
该位点包括三种基因型即g/g、c/c、c/g,不同基因型样本的病情指数分布的箱形图见图6。注意,由于zmgnat19基因chr1∶269012623和chr1∶269012638两个位点完全连锁,故不同基因型的组间箱形图图5和图6图形一致,表2和表3中表型均值的组间比较数据也一致。g/g组中位数为5.02、下四分位数q1为2.38、上四分位数q3为7.33;c/c组中位数为7.92、下四分位数q1为5.54、上四分位数q3为11.38;c/g组中位数为8.63、下四分位数q1为5.6、上四分位数q3为5.6。
[0086]
不同基因型组间表型均值差异的t测验结果见表3,由表3可知基因型g/g与c/c组间差异极显著(p=0.0003),组间均值差为3.59;c/c与c/g之间差异不显著(p=0.935),c/g与g/g之间差异也不显著(p=0.184);g/g与(c/c+c/g)之间差异极显著(p<0.0001),组间均值差为3.573。因此,基因型g/g是大斑病极显著的抗病分子标记,基因型c/c和c/g是大斑病极显著的易感分子标记。
[0087]
表3zmgnat19基因chr1:269012638位点大斑病audpc的组间均值比较
[0088][0089]
序列:
[0090][0091][0092]
上述序列中163位的碱基y为c或t,178位碱基s为g或c,导致供试玉米自交系群体基因多态性及玉米大斑病病情进展极显著差异。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1