一种钻喙兰属SSR分子标记引物组合物及其应用
一种钻喙兰属ssr分子标记引物组合物及其应用
技术领域
1.本发明涉及分子生物学技术领域,具体涉及一种钻喙兰属ssr分子标记引物组合物、指纹码及其在钻喙兰属种质资源鉴定中的应用。
背景技术:
2.钻喙兰属(rhynchostylisblume)是隶属兰科、兰亚科、万代兰族、指甲兰亚族的一类单轴生长型附生兰花,具花多且密的总状花序,因此又得名狐狸尾兰,主要分布于我国的云南省、贵州省和海南岛,以及印度、斯里兰卡和东南亚各国。钻喙兰属植物株型紧凑、花繁色艳且绝大多数具浓烈芳香,栽培管理简单,耐热性强且易杂交,具有很高的观赏价值和开发应用前景。从19世纪中叶开始,就陆续有钻喙兰属的种间杂交种、与近源兰科植物之间的属间杂交种在英国皇家园艺学会进行登录,截止2020年11月钻喙兰属原生种登录的f1代杂交组合已达381个。
3.除传统杂交育种工作外,钻喙兰属植物相关研究较少,目前主要集中在组培快繁、栽培管理和野生资源分布及鉴定等方面。莫饶等(2009)研究发现海南钻喙兰(rhy.gigantea)的核型公式为2n=2x=36m+2sm(莫饶,等.兰科11属14种植物核型分析.云南植物研究,2009,31(6):504
‑
508)。颜平等(2014)利用srap标记技术分析了来自海南岛22个居群地的144份海南钻喙兰野生资源的遗传多样性(颜平,等.基于srap标记的海南钻喙兰种质资源遗传多样性分析.热带作物学报,2014,35(9):1677
‑
1682)。junitakurniawati等(2019)利用2个rapd引物对包括钻喙兰(rhy.retusa)在内的9个不同的兰科原生种进行了遗传相似性分析,发现种内相似性远高于种间的相似性(junitakurniawati,etal.molecular identification of several orchid species based on opa10 and opa18 rapd marker.journal of physics:conference series,2019,1397(1):012042)。atsushi matsuba等(2015)构建了包含10600个bac克隆的立梗蓝狐尾(rhy.coelestis)人工细菌染色体(atsushi matsuba,etal.molecular cytogenetic use of bac clones in neofinetia falcate and rhynchostylis coelestis.nucleus,2015,58(3):207
‑
210)。subhas chandra roy等(2017)利用ddrad技术对包括钻喙兰(rhy.retusa)在内的4个兰花原生种进行简化基因组测序和遗传多样性分析(subhas chandra roy,etal.assessment of genetic diversity among four orchids based on ddrad sequencing data for conservation purposes.physiology and molecular biology of plants,2017,23(1):169
‑
183)。
4.微卫星标记(microsatellite)也称为短串联重复序列(str)或简单重复序列(ssr),是由几个核苷酸(2
‑
5个)为重复单位组成的长达几十个核苷酸的重复序列,广泛分布在真核生物整个基因组的不同位置上,由于重复次数的不同及重复程度的不完全造成了每个位点多态性。利用某个微卫星dna两端的保守序列设计一对特异引物,扩增这个位点的微卫星dna序列,通过电泳检测即可显示不同基因型个体在这个ssr位点上的多态性。tp
‑
m13
‑
ssr(simple sequence repeatwith tailed primer m13)技术结合了ssr分子标记技
术和荧光测序技术,具有重复性高,结果准确等优点,在较大程度上解决了分析通量较低,扩增产物检测流程繁琐,数据记录的工作量大等一系列问题(李会勇等,2005)。
5.因此,利用钻喙兰属的基因组序列开发新的ssr分子标记及其引物组合物,并利用tp
‑
m13
‑
ssr技术进行遗传多样性的分析及分子身份证的构建,将会对钻喙兰属的种质资源鉴定、亲缘关系分析、性状基因的定位、分子标记辅助育种及钻喙兰属相关的分子研究起重要的作用。
技术实现要素:
6.针对上述不足,本发明提供了一种钻喙兰属ssr分子标记引物组合物、指纹码及其在钻喙兰属种质资源鉴定中的应用。本发明利用简化基因组测序技术提供了钻喙兰属ssr引物组合物,并利用tp
‑
m13
‑
ssr技术对属内3个种10份不同的种质资源进行遗传多样性的分析及分子身份证的构建,可准确、高效、稳定地鉴定钻喙兰属种质资源,为钻喙兰属的种质资源鉴定、亲缘关系分析、性状基因的定位、分子标记辅助育种及钻喙兰属相关的分子研究等奠定了基础。
7.为了实现上述发明目的,本发明的技术方案如下:
8.一方面,本发明提供了一种钻喙兰属ssr分子标记的引物组合物,所述的ssr分子标记包括5
‑
n1001614、7
‑
n1001895、14
‑
n1007590、25
‑
n101022、28
‑
n1012378、37
‑
n3601419、45
‑
n1438823、82
‑
n100023、88
‑
n1001036、89
‑
n1001051、91
‑
n1001523、96
‑
n1007386和97
‑
n1844635。
9.具体地,所述的引物组合物包括:
10.(1)扩增ssr分子标记5
‑
n1001614的引物:
11.seq id no:1:5
‑
n1001614
‑
f:5'
‑
tttggtggaggatggagaag
‑
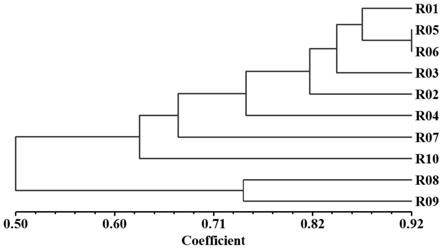
3'
12.seq id no:2:5
‑
n1001614
‑
r:5'
‑
gcttccttgaggtgaagagc
‑
3';
13.(2)扩增ssr分子标记7
‑
n1001895的引物:
14.seq id no:3:7
‑
n1001895
‑
f:5'
‑
atcgccatttttgtgctctt
‑
3'
15.seq id no:4:7
‑
n1001895
‑
r:5'
‑
attgaaatgatggctttcgc
‑
3';
16.(3)扩增ssr分子标记14
‑
n1007590的引物:
17.seq id no:5:14
‑
n1007590
‑
f:5'
‑
tgacatggaagttatggcattc
‑
3'
18.seq id no:6:14
‑
n1007590
‑
r:5'
‑
tgtcgcaattctatgtcagga
‑
3';
19.(4)扩增ssr分子标记25
‑
n101022的引物:
20.seq id no:7:25
‑
n101022
‑
f:5'
‑
tggaccacctatctcaaggc
‑
3'
21.seq id no:8:25
‑
n101022
‑
r:5'
‑
acgacatgctcatgattcca
‑
3';
22.(5)扩增ssr分子标记28
‑
n1012378的引物:
23.seq id no:9:28
‑
n1012378
‑
f:5'
‑
ggaagaaggaggaggtgagg
‑
3'
24.seq id no:10:28
‑
n1012378
‑
r:5'
‑
tcctagagaaagagcttggagc
‑
3';
25.(6)扩增ssr分子标记37
‑
n3601419的引物:
26.seq id no:11:37
‑
n3601419
‑
f:5'
‑
aatggaaaatctggcagcac
‑
3'
27.seq id no:12:37
‑
n3601419
‑
r:5'
‑
tctccatcacttcaatggca
‑
3';
28.(7)扩增ssr分子标记45
‑
n1438823的引物:
29.seq id no:13:45
‑
n1438823
‑
f:5'
‑
atttggctcaaatccaccaa
‑
3'
30.seq id no:14:45
‑
n1438823
‑
r:5'
‑
atacctgaaagcatggtgcc
‑
3';
31.(8)扩增ssr分子标记82
‑
n100023的引物:
32.seq id no:15:82
‑
n100023
‑
f:5'
‑
agggtgggaagggagataaa
‑
3'
33.seq id no:16:82
‑
n100023
‑
r:5'
‑
ctctgccttctccatctctca
‑
3';
34.(9)扩增ssr分子标记88
‑
n1001036的引物:
35.seq id no:17:88
‑
n1001036
‑
f:5'
‑
agggagggagtgagagagga
‑
3'
36.seq id no:18:88
‑
n1001036
‑
r:5'
‑
tcccgctttctatcgctatc
‑
3';
37.(10)扩增ssr分子标记89
‑
n100105的引物:
38.seq id no:19:89
‑
n100105
‑
f:5'
‑
tttcctgctctccattcctg
‑
3'
39.seq id no:20:89
‑
n100105
‑
r:5'
‑
tgtcatcttctcacactccca
‑
3';
40.(11)扩增ssr分子标记91
‑
n1001523的引物:
41.seq id no:21:91
‑
n1001523
‑
f:5'
‑
tgatggaatgaagttgaggga
‑
3'
42.seq id no:22:91
‑
n1001523
‑
r:5'
‑
ggcacatatgctttgtggtg
‑
3';
43.(12)扩增ssr分子标记96
‑
n1007386的引物:
44.seq id no:23:96
‑
n1007386
‑
f:5'
‑
aagtcagccactatgccagg
‑
3'
45.seq id no:24:96
‑
n1007386
‑
r:5'
‑
caagcgacatttgcgtctaa
‑
3';
46.(13)扩增ssr分子标记97
‑
n1844635的引物:
47.seq id no:25:97
‑
n1844635
‑
f:5'
‑
gtggatataccgcttcgtcg
‑
3'
48.seq id no:26:97
‑
n1844635
‑
r:5'
‑
gaagaaagtggctgcgtctc
‑
3'。
49.另一方面,本发明提供了一种钻喙兰属ssr分子标记指纹码,所述的指纹码包括指纹图谱和分子身份证,所述的分子身份证ssr分子标记顺序为:5
‑
n1001614、7
‑
n1001895、14
‑
n1007590、25
‑
n101022、28
‑
n1012378、37
‑
n3601419、45
‑
n1438823、82
‑
n100023、88
‑
n1001036、89
‑
n1001051、91
‑
n1001523、96
‑
n1007386、97
‑
n1844635。
50.具体地,所述的分子身份证如下表1所示。
51.表1
52.编号拉丁名分子身份证信息r01rhy.gigantea
‘
shen's suns’23334514342344121112448833r02rhy.gigantea24331414242344121112440033r03rhy.gigantea14332414342344122113442613r04rhy.gigantea
‘
jh#11’24004414352344121112444533r05rhy.gigantean var.petoniana24334444242344121111445733r06rhy.giganteaf.rubra23331444242344121111441133r07rhy.coelestis11244414121313121611443323r08rhy.coelestisf.alba11334422121215113611131322r09rhy.coelestis11123422121215114622120022r10rhy.retusa22442433332224125111443333
53.进一步具体地,所述的分子身份证按照二倍体标准构建,依据ssr检测结果,对指纹数据进行数据化编码(各位点扩增片段按分子量大小排列,扩增片段(等位基因)从小到
大以阿拉伯数字1
‑
9标注,9个以上的等位基因,以大写英文字母a
‑
z标注),若该位点在某个品种中无扩增则记为0,每个位点占两位。
54.具体地,所述的指纹图谱如下表2所示。
55.表2
56.位点r01r02r03r04r05r06r07r08r09r105
‑
n1001614100100981001001009898981005
‑
n10016141031061061061061039898981007
‑
n1001895182182182
‑
1821821721821691927
‑
n1001895182182182
‑
18218219218217219214
‑
n100759012411511712412411512412412011714
‑
n100759012812412412412412412412412412425
‑
n10102212812812812813313312813113113225
‑
n10102213313313313313313313313113113228
‑
n101237817417117417417117116716716717428
‑
n101237817617617617917617617117117117437
‑
n360141918718718718718718718118118118737
‑
n360141919019019019019019019018718718745
‑
n143882319319319319319319316316316318245
‑
n143882319319319319319319318419419419382
‑
n10002316716716716716716716716716716782
‑
n10002317117117117117117117116716717188
‑
n100103614314313514314314314315315713788
‑
n100103614314314314314314316116116114389
‑
n10010519797979797979797999789
‑
n100105199991019997979797999791
‑
n100152313613613613613613613611611613691
‑
n100152313613613613613613613613412613696
‑
n1007386150
‑
1111161249211392
‑
11396
‑
n1007386150
‑
12712413092113113
‑
11397
‑
n184463512812812212812812812412412412897
‑
n1844635128128128128128128128124124128
57.又一方面,本发明提供了一种包含上述ssr分子标记扩增引物组合物或ssr分子标记指纹码的试剂盒。
58.又一方面,本发明提供了上述ssr分子标记扩增引物组合物、ssr分子标记指纹码或试剂盒在钻喙兰属筛选或种质资源鉴定中的应用。
59.又一方面,本发明提供了上述ssr分子标记筛选方法,所述的方法包括以下步骤:以钻喙兰属总dna为模板,用超声波破碎仪随机打断成长度约350bp的片段,经末端修复、加a尾、加测序接头、纯化、pcr扩增等步骤完成文库制备后,使用qubit 2.0进行初步定量,稀释文库至2ng/μl,随后使用agilent 2100对文库的插入片段进行检测,使用q
‑
pcr方法对文
库的有效浓度进行准确定量,以保证文库质量。使用illuminahiseq 2500进行测序,原始下机数据首先采用fastqc软件进行质量检测,去除接头及低质量碱基序列后,将双端reads按照重叠碱基使用flash软件进行拼接,使用misa筛选测序结果,保留有微卫星标记ssr的序列。
60.又一方面,本发明提供了一种钻喙兰属筛选或种质资源鉴定的方法,所述的方法包括以下步骤:
61.(1)待测植株总dna提取;
62.(2)以步骤(1)提取的总dna作为模板进行ssr分子标记pcr扩增;
63.(3)毛细管电泳检测步骤(2)的pcr扩增产物,收集数据;
64.(4)根据步骤(3)获得的数据进行遗传多样性指标、聚类及多态信息含量pic计算分析。
65.具体地,步骤(2)中所述的pcr扩增体系为:反应体系总体积为10μl,包括1.2μl dna模板(50ng/μl),1.0μl 10
×
bufferι缓冲液,0.1μl takara hs taq酶(5u/μl),引物(5μm)0.6μl,0.8μl2.5mm dntp,tp
‑
m13(5μm)0.5μl,去离子水补足至10μl。
66.进一步具体地,所述的pcr扩增反应程序:95℃5min;95℃30s,60℃30s,72℃30s,30个循环;95℃30s,53℃30s,72℃30s,10个循环;60℃30min;4℃保存。
67.进一步具体地,所述的pcr扩增体系引物包括如seq id no:1
‑
seq id no:26所示的序列。
68.又一方面,本发明还提供了上述ssr分子标记扩增引物组合物、ssr分子标记指纹码或试剂盒在钻喙兰属亲缘关系分析、性状基因的定位和分子标记辅助育种中的应用。
69.与现有技术相比,本发明的积极和有益效果在于:
70.1、本发明提供了一种钻喙兰属ssr分子标记及引物组合物,且利用tp
‑
m13
‑
ssr技术对钻喙兰属内3个种10份不同的种质资源进行遗传多样性的分析及分子身份证的构建,为钻喙兰属的筛选和种质资源鉴定提供了基础。
71.2、本申请采用所述的钻喙兰属ssr分子标记及引物组合物,可准确、高效、稳定地鉴定钻喙兰属种质资源,操作简便。
72.3、本申请所述的钻喙兰属ssr分子标记可用于辅助选择育种,实现苗期提前选择,从而加快钻喙兰属的育种进程。
附图说明
73.图1为ssr分子标记引物扩增结果峰图。
74.图2为遗传分析及聚类图。
具体实施方式
75.下面结合具体实施例,对本发明作进一步详细的阐述,下述实施例不用于限制本发明,仅用于说明本发明。以下实施例中所使用的实验方法如无特殊说明,实施例中未注明具体条件的实验方法,通常按照常规条件,下述实施例中所使用的材料、试剂等,如无特殊说明,均可从商业途径得到。
76.除非另外定义,否则本文中所用的全部技术与科学用语均具有本领域技术人员通
常理解的含义。
77.实施例1钻喙兰属种质资源
78.钻喙兰属目前已发现和鉴定的原生种共5个,其中3个分布广泛、发现较早、杂交利用率较高,3个种之间均已有杂交后代登录,另2个原生种分布范围狭窄、发现较晚,尚未有育种利用和针对性研究。除种间杂交外,钻喙兰属已与万代兰属、蝴蝶兰属、指甲兰属、火焰兰属、槽舌兰属等兰科其他20个属的不同种质资源进行跨属杂交培育出新种质。
79.本发明采用广东省农业科学院环境园艺研究所收集保存的3个不同原生种共10份钻喙兰属种质资源,具体信息见下表3。所有实验材料均种植于广东省农业现代化科技示范区广东省农业科学院环境园艺研究所温室大棚内,于2020年5月采集根尖液氮速冻15min后放入超低温冰箱备用。
80.表3 3个种10份种质资源基本信息
[0081][0082][0083]
实施例2钻喙兰属种植资源鉴定
[0084]
1.总dna提取
[0085]
按照天根生物科技(北京)有限公司的新型植物基因组dna提取试剂盒操作步骤进行钻喙兰植株总dna的提取,具体步骤如下:
[0086]
(1)取钻喙兰植株幼嫩根尖或嫩叶组织加入液氮充分研磨,称取植物新鲜组织约100mg。
[0087]
(2)向研磨好的粉末中迅速加入400μl缓冲液gps和10μl rnase a(10mg/ml),迅速涡旋混匀后,将离心管放在65℃水浴15min,水浴过程中颠倒离心管以混合样本数次。
[0088]
(3)加入100μl缓冲液gpa,涡旋震荡1min,12000rpm离心5min,转移上清至过滤柱cs中(过滤柱cs放在收集管中),然后12000rpm离心1min,转移滤液至新的离心管中。
[0089]
(4)加入等体积的无水乙醇,充分混匀,此时可能会出现絮状沉淀。
[0090]
(5)将上一步所得溶液和絮状沉淀都转移到rnase
‑
free吸附柱cr2中(吸附柱cr2放在收集管中),12000rpm离心1min,倒掉废液,将rnase
‑
free吸附柱cr2放入收集管中。
[0091]
(6)向rnase
‑
free吸附柱cr2中加入550μl去蛋白液rd(使用前请先检查是否已加入无水乙醇),12000rpm离心1min,倒掉废液,将rnase
‑
free吸附柱cr2放入收集管中。
[0092]
(7)向rnase
‑
free吸附柱cr2中加入700μl漂洗液pw(使用前请先检查是否已加入无水乙醇),12000rpm离心1min,倒掉废液,将rnase
‑
free吸附柱cr2放入收集管中。
[0093]
(8)重复步骤(7)。
[0094]
(9)将rnase
‑
free吸附柱cr2放回收集管中,12000rpm离心2min,弃收集管,然后将rnase
‑
free吸附柱cr2转移到新的离心管中,室温晾干5
‑
10min。
[0095]
(10)在rnase
‑
free吸附柱cr2中加入50
‑
100μl洗脱缓冲液tb,室温放置3
‑
5min,12000rpm离心2min,将溶液收集到离心管中。
[0096]
(11)取2μl dna用于1.2%琼脂糖凝胶电泳检测,取2μl dna用于nanodrop分光光度计测浓度。
[0097]
2.ssr分子标记及引物组合物
[0098]
2.1.将检测合格的10份钻喙兰属种质资源dna样品等量混匀后,用bioruptor超声波破碎仪随机打断成长度约为350bp的片段,按照rapid dna
‑
seq kit(bioo scientific,5144
‑
08)试剂盒步骤,经末端修复、加a尾、加测序接头、纯化、pcr扩增等步骤完成文库制备后,使用qubit 2.0进行初步定量,稀释文库至2ng/μl,随后使用agilent 2100对文库的插入片段进行检测,使用q
‑
pcr方法对文库的有效浓度进行准确定量,以保证文库质量。使用illuminahiseq 2500进行测序,原始下机数据首先采用fastqc软件进行质量检测,去除接头及低质量碱基序列后,将双端reads按照重叠碱基使用flash软件进行拼接,使用misa筛选测序结果,保留有微卫星标记的序列作为ssr分子标记。
[0099]
2.2.根据ssr分子标记设计引物,所述引物如下表4所示。
[0100]
表4 ssr分子标记引物
[0101]
[0102][0103]
3.样品扩增
[0104]
使用表4中的引物进行样品的pcr扩增。
[0105]
反应体系总体积为10μl,包括1.2μl dna模板(50ng/μl),1.0μl 10
×
buffer ι缓冲液,0.1μl takara hs taq酶(5u/μl),引物(5μm)0.6μl,0.8μl2.5mm dntp,tp
‑
m13(5μm)0.5μl,去离子水补足至10μl。
[0106]
反应程序:95℃5min;95℃30s,60℃30s,72℃30s,30个循环;95℃30s,53℃30s,72℃30s,10个循环;60℃30min;4℃保存。
[0107]
4.检测
[0108]
1.2%琼脂糖凝胶电泳检测扩增产物。96孔板中每孔加入扩增产物1.0μl,rox
‑
500分子量内标和甲酰胺混合液(体积比0.5:8.5)9μl,95℃变性3min后,上abi 3730xl检测仪进行检测,1kv电压进样10s,电泳15kv,30min。将data colletion软件收集的原始数据文件导入到genemapper 3.2软件中进行分析,将各峰值的位置与其泳道中的分子量内标予以比较,计算目标dna片段的准确大小。各荧光标记座位上独立进行3次重复的毛细管电泳检测,取3次重复的平均值并四舍五入取整,作为实验材料在该座位上的数据。
[0109]
5.数据分析
[0110]
将整理好的数据,运用ntsys软件进行遗传多样性指标、聚类及多态信息含量pic计算分析。
[0111]
实施例3引物扩增结果
[0112]
本发明所述的ssr分子标记引物扩增结果详见下表5。因ssr分子标记引物及钻喙兰属种类较多,以5
‑
n1001614扩增结果峰图为例进行展示,5
‑
n1001614扩增结果峰图如图1所示。由表5及图1可知,本发明所述的ssr分子标记引物扩增效果好,检出率高,且可扩增出稳定的dna条带。
[0113]
表5引物扩增结果
[0114][0115]
实施例4遗传分析及聚类
[0116]
遗传多样性指标、聚类及多态信息含量pic计算分析结果如下表6及图2所示。
[0117]
表6
[0118]
[0119][0120]
实施例5分子身份证构建
[0121]
按照二倍体标准构建,依据ssr检测结果,对指纹数据进行数据化编码(各位点扩增片段按分子量大小排列,扩增片段(等位基因)从小到大以阿拉伯数字1
‑
9标注,9个以上的等位基因,以大写英文字母a
‑
z标注),若该位点在某个品种中无扩增则记为0,每个位点占两位。其中,分子身份证ssr分子标记顺序为:5
‑
n1001614、7
‑
n1001895、14
‑
n1007590、25
‑
n101022、28
‑
n1012378、37
‑
n3601419、45
‑
n1438823、82
‑
n100023、88
‑
n1001036、89
‑
n1001051、91
‑
n1001523、96
‑
n1007386、97
‑
n1844635。分子身份证信息如下表7所示,指纹图谱如下表8所示。
[0122]
表7
[0123]
编号拉丁名分子身份证信息r01rhy.gigantea
‘
shen's suns’23334514342344121112448833r02rhy.gigantea24331414242344121112440033r03rhy.gigantea14332414342344122113442613r04rhy.gigantea
‘
jh#11’24004414352344121112444533r05rhy.gigantean var.petoniana24334444242344121111445733r06rhy.giganteaf.rubra23331444242344121111441133r07rhy.coelestis11244414121313121611443323r08rhy.coelestisf.alba11334422121215113611131322r09rhy.coelestis11123422121215114622120022r10rhy.retusa22442433332224125111443333
[0124]
表8指纹图谱
[0125]
[0126][0127]
实验例1准确性检测
[0128]
根据本发明所述的ssr分子标记引物组合物和种质鉴定方法,选取r01、r02、r03、r04、r05、r06、r07、r08、r09、r10的钻喙兰植株各10个样品进行检测,检测结果如下表9所示。
[0129]
表9准确性检测结果
[0130]
[0131][0132]
由表9可知,采用本发明所述的ssr分子标记引物组合物和种质鉴定方法,可准确鉴定10份钻喙兰属,准确率为100%。
[0133]
本发明所述的ssr分子标记可用于辅助选择育种,实现苗期提前选择,从而加快钻喙兰属的育种进程。
[0134]
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1