一种花生野生种基因组特异探针和使用方法
1.本发明涉及一种花生细胞遗传学研究方法,特别涉及一种花生野生种基因组特异探针的设计和使用方法。
背景技术:
2.花生野生种中存在丰富的生物、非生物胁迫的抗源和高含油量等优质性状,而且抗性机制与栽培种花生有所不同,是改良花生栽培种的重要基因资源。国内外研究发现,危害花生的病虫害有120多种,引起产量损失5%
‑
100%;花生野生种对许多花生病害、虫害和生长逆境具有高水平抗性,几乎所有的花生病虫害都能在花生野生种中找到抗源,部分野生种甚至对一些病虫害免疫,例如a.diogoi和a.glabrata抗花生芽坏死病毒病和番茄斑萎病毒免疫,a.kuhlmannii、a.duranensis和a.ipaensis对花生丛簇病毒病免疫,a.magna和a.cardenasii对锈病免疫等,而这些免疫抗源在花生栽培种中是找不到的。
3.种间杂交是将花生野生种优异基因导入到栽培种的重要手段。然而,花生属物种由于染色体倍性、基因组组成等方面的差异,使得同一基因组内或不同基因组间的物种杂交出现不同程度的生殖隔离,例如:杂交不亲和、杂种夭亡、杂种不育等杂交障碍,导致将不同基因组野生种的基因导入栽培种难度不一。研究发现,花生栽培种(基因组aabb)与花生组(section arachis)中a、b、k、f、g等基因组的物种杂交几乎都是可亲和的,与拥有e基因组的部分直立组(section erectoides)物种和拥有r基因组的部分根茎组(section rhizomatosae)物种杂交也比较容易;而与拥有c基因组的大根组(section caulorrhizae)、拥有p基因组的匍匐组(section procumbentes)、拥有te基因组的三叶组(section trierectoides)、拥有t基因组的三籽组(section triseminatae)、拥有ex基因组的围脉组(section extranervosae)和拥有h基因组的异形花组(section heteranthae)的物种杂交很难成功。因此,明确花生野生种基因组间相互关系对克服花生种间杂交不亲和障碍,高效利用优异野生种质至关重要。
4.植物基因组在进化过程中,其重复序列存在不同程度的扩增和收缩,这种扩增与收缩使物种之间及物种内部出现重复序列类型及含量差异,这种差异能够反映物种间的亲缘关系远近。串联重复序列是一种在基因组中成簇状分布重复序列,串联重复序列的扩增为基因组进化提供了丰富的变异来源。植物特异串联重复序列,通常只在近缘物种或基因组中扩增,而不存在于其它基因组中,因此,常被用来确定物种或基因组间的亲缘关系。
5.花生野生种经历了漫长的演化过程,形成了a、b、k、f、g、c、p、t、te、ex和h等不同的基因组类型。然而,目前对于花生野生种基因组特异重复序列的研究还未见报道。
技术实现要素:
6.针对现有技术的不足,本发明的目的是提供一种花生野生种基因组特异探针和使用方法,以h基因组物种a.dardonoi和a.pusilla,以及b基因组物种a.ipaensis为研究对象,通过基因组测序、生物信息学分析建立了花生野生种基因组特异探针,并将其应用到花
生野生种亲缘关系分析,为花生远缘杂交研究和野生种利用提供了新方法和理论依据。
7.为了实现上述目的,本发明所采用的技术方案是:
8.一种花生野生种基因组特异探针,包括h基因组特异探针和b基因组特异探针;所述h基因组特异探针为dard
‑
127和pus
‑
187,所述b基因组特异探针为ipa
‑
163,具体序列为:
9.探针dard
‑
127:
10.fam
‑5′‑
taaactatggtattttcatgagttttgaggcatgccgga
‑3′
;
11.探针pus
‑
187:
12.tamra
‑5′‑
tcactaggcatataatgccactcgatggcgttgaaacgcggagct
‑3′
;
13.探针ipa
‑
163:
14.tamra
‑5′‑
tagggtttatgatttaggctttagggtttgt
‑3′
。
15.所述的花生野生种基因组特异探针的设计方法,包括以下步骤:
16.(a)首先对花生属物种进行illumina双端基因组测序,测序深度为20
×
;然后利用trimmomatic软件对基因组测序原始序列进行过滤,关键参数为:illuminaclip:trim_home/adapters/truseq3
‑
pe.fa:2:30:10、leading:5、trailing:5slidingwindow:4:20minlen:150,获得高质量“clean reads”;
17.(b)使用花生栽培种tifrunner基因组作为参考基因组,用bowtie2软件构建索引文件,而后将“clean reads”比对到tifrunner参考基因组上,过滤出双端均未比对到tifrunner参考基因组的“clean reads”;
18.(c)利用linux的shuf命令,从过滤出的“clean reads”中随机取样100万个序列,利用tarean软件,对基于随机取样得到的“clean reads”进行串联重复序列发掘,发掘“high confidence”的串联重复序列集合;
19.(d)利用b2dsc将串联重复序列定位到花生栽培种tifrunner参考基因组上,排除在栽培种中高度富集的串联重复序列;利用ncbi
‑
blast排除候选串联重复序列可能的基因序列,剩余串联重复序列用于探针设计;
20.(e)选择串联重复序列最保守的区段截取30
‑
45bp的序列,进行5
′‑
tamra或fam人工合成修饰,获得了基因组特异探针。
21.所述花生属物种为h基因组物种a.dardonoi和a.pusilla,对应的基因组特异探针为h基因组特异探针dard
‑
127和pus
‑
187。
22.所述花生属物种为b基因组物种a.ipaensis,对应的基因组特异探针为b基因组特异探针ipa
‑
163。
23.所述的花生野生种基因组特异探针的使用方法,包括以下步骤:
24.(a)通过染色体压片法,获得分别拥有花生a基因组、b基因组、e基因组、h基因组和k基因组二倍体野生种根尖细胞有丝分裂中期染色体制片,在
‑
80℃冰冻揭片,用无水乙醇脱水,风干;
25.(b)在染缸内加入40ml 2
×
ssc缓冲液、h基因组特异探针干粉或/和b基因组特异探针干粉,以及5μl的dapi母液,将野生种染色体制片置于染缸中染色5h,荧光显微镜拍照;所述h基因组特异探针干粉为0.1od dard
‑
127和0.1od pus
‑
187,b基因组特异探针干粉为0.1od ipa
‑
163。
[0026]2×
ssc缓冲液由0.3mol/l的柠檬酸三钠c6h5na3o7·
2h2o和3mol/l的nacl组成。
[0027]
dapi母液为浓度100μg/ml的dapi染色液。
[0028]
a基因组、b基因组、e基因组、h基因组、k基因组二倍体野生种分别为a基因组物种a.duranensis、a物种基因组a.diogoi、b基因组物种a.ipaensis、e基因组物种a.paraguariensis、e基因组物种a.stenophylla、h基因组物种a.pusilla、h基因组物种a.dardonoi、k基因组物种a.batizocoi。
[0029]
本发明有益效果:
[0030]
本发明通过对花生野生种进行illumina双端基因组测序,利用基因组序列和生物信息学分析,成功开发了花生野生种h和b基因组的特异探针标记,丰富了野生种细胞学特征,建立了一种花生野生种基因组特异探针的设计方法,为获得花生野生种基因组特异细胞学标记提供了新的有效方法。
[0031]
本发明利用新设计的花生野生种h和b基因组特异探针对多个花生基因组染色体染色分析,发现探针pus
‑
187是h基因组特异探针,可以特异识别h基因组物种;探针dard
‑
127是a.dardonoi特异探针,可以特异识别a.dardonoi;探针ipa
‑
163为b和k基因组特异探针,证实了b和k基因组可能存在很近的亲缘关系。本发明特异探针可用于分析不同花生野生种基因组,能够有效识别花生野生种或基因组,确定花生野生种或基因组间的亲缘关系,为花生种间杂交研究和野生种利用提供了新方法和理论依据。
附图说明
[0032]
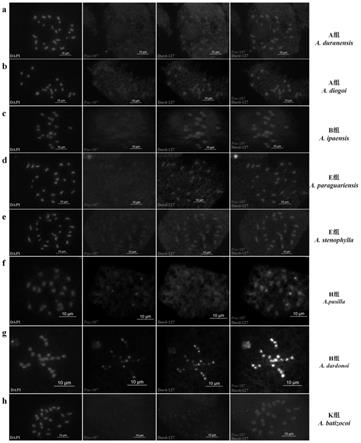
图1为以特异探针pus
‑
187和dard
‑
127在野生种a.duranensis(a,a基因组)、a.diogoi(b,a基因组)、a.ipaensis(c,b基因组)、a.paraguariensis(d,e基因组)、a.stenophylla(e,e基因组)、a.pusilla(f,h基因组)、a dardonoi(g,h基因组)和a.batizocoi(h,k基因组)上的染色信号结果。
[0033]
其中,(a
‑
h)中从左到右第一列为dapi染色,第二列为pus
‑
187探针红色信号在野生种染色体上的分布,第三列为dard
‑
127探针绿色信号在野生种染色体,第四列为dapi、pus
‑
187和dard
‑
127探针信号合成图。
[0034]
图2为以特异探针ipa
‑
163在野生种a.duranensis(a,a基因组)、a.diogoi(b,a基因组)、a.ipaensis(c,b基因组)、a.paraguariensis(d,e基因组)、a.stenophylla(e,e基因组)、a.dardonoi(f,h基因组)和a.batizocoi(g,k基因组)上的染色信号结果。
[0035]
其中,(a
‑
h)中从左到右第一列为dapi染色,第二列为ipa
‑
163探针红色信号在野生种染色体上的分布,第三列dapi和ipa
‑
163探针信号合成图。
具体实施方式
[0036]
以下结合实施例对本发明的具体实施方式作进一步详细说明。
[0037]
实施例1、一种花生野生种h基因组特异探针的设计方法
[0038]
(a)首先对两个h基因组物种a.dardonoi和a.pusilla进行illumina双端基因组测序,测序深度为20
×
。然后利用trimmomatic(v0.38)软件对基因组测序原始序列进行过滤,关键参数为:
[0039]
illuminaclip:trim_home/adapters/truseq3
‑
pe.fa:2:30:10、leading:5、trailing:5slidingwindow:4:20minlen:150,获得高质量的“clean reads”。
[0040]
(b)使用花生栽培种tifrunner基因组(https://www.peanutbase.org/download)作为参考基因组,用bowtie2(v2.3.5)软件构建索引文件,而后将a.dardonoi和a.pusilla的“clean reads”比对到tifrunner参考基因组上,过滤出双端均未比对到tifrunner参考基因组上的“clean reads”。
[0041]
(c)利用linux的shuf命令,从过滤出的“clean reads”中随机取样100万个序列,利用tarean软件(http://www.repeatexplorer.org),对基于随机取样得到的“clean reads”进行串联重复序列发掘,发掘“high confidence”的串联重复序列集合。
[0042]
(d)利用b2dsc(http://mcgb.uestc.edu.cn/b2dsc)将串联重复序列定位到花生栽培种tifrunner参考基因组上,排除在栽培种中高度富集的串联重复序列;利用ncbi
‑
blast(https://www.ncbi.nlm.nih.gov/)排除候选串联重复序列可能的基因序列,剩余串联重复序列用于探针设计。
[0043]
(e)选择a.dardonoi和a.pusilla串联重复序列最保守的区段截取30
‑
45bp的序列,进行5
′‑
tamra或者5
′‑
fam人工合成修饰,获得了h基因组特异探针dard
‑
127和pus
‑
187。探针dard
‑
127:
[0044]
fam
‑5′‑
taaactatggtattttcatgagttttgaggcatgccgga
‑3′
;(seq id no:1)探针pus
‑
187:
[0045]
tamra
‑5′‑
tcactaggcatataatgccactcgatggcgttgaaacgcggagct
‑3′
(seq id no:2)。
[0046]
实施例2、一种花生野生种b基因组特异探针的设计方法
[0047]
(a)首先对b基因组物种a.ipaensis进行illumina双端基因组测序,测序深度为20
×
。然后利用trimmomatic(v0.38)软件对基因组测序原始序列进行过滤,关键参数为:illuminaclip:trim_home/adapters/truseq3
‑
pe.fa:2:30:10、leading:5、trailing:5slidingwindow:4:20minlen:150,获得高质量的“clean reads”。
[0048]
(b)使用花生栽培种tifrunner基因组(https://www.peanutbase.org/download)作为参考基因组,用bowtie2(v2.3.5)软件构建索引文件,而后将a.ipaensis的“clean reads”比对到tifrunner参考基因组上,过滤出双端均未比对到tifrunner参考基因组上的“clean reads”。
[0049]
(c)利用linux的shuf命令,从过滤出的“clean reads”中随机取样100万个序列,利用tarean软件(http://www.repeatexplorer.org),对基于随机取样得到的“clean reads”进行串联重复序列发掘,发掘“high confidence”的串联重复序列集合。
[0050]
(d)利用b2dsc(http://mcgb.uestc.edu.cn/b2dsc)将串联重复序列定位到花生栽培种tifrunner参考基因组上,排除在栽培种中高度富集的串联重复序列;利用ncbi
‑
blast(https://www.ncbi.nlm.nih.gov)排除候选串联重复序列可能的基因序列,剩余串联重复序列用于探针设计。
[0051]
(e)选择a.ipaensis串联重复序列最保守的区段截取30
‑
45bp的序列,进行5
′‑
tamra人工合成修饰,获得了b基因组特异探针ipa
‑
163。
[0052]
探针ipa
‑
163:tamra
‑5′‑
tagggtttatgatttaggctttagggtttgt
‑3′
(seq id no:3)。实施例3、一种花生野生种h基因组特异探针的使用方法
[0053]
首先,通过染色体压片法,获得花野生种a.duranensis(a基因组)、a.diogoi(a基
因组)、a.ipaensis(b基因组)、a.paraguariensis(e基因组)、a.stenophylla(e基因组)、a.pusilla(h基因组)、a.dardonoi(h基因组)和a.batizocoi(k基因组)二倍体野生种根尖细胞有丝分裂中期制片,在
‑
80℃冰冻揭片,无水乙醇脱水,风干。
[0054]
其次,在染缸内加入40ml 2
×
ssc缓冲液(由0.3m的柠檬酸三钠c6h5na3o7·
2h2o和3m的nacl组成)、0.1od的dard
‑
127和0.1od的pus
‑
187探针,以及5μl的dapi母液(100μg/ml的dapi染色液),将野生种染色体制片置于染缸中染色5h,荧光显微镜拍照,获得染色图片(图1)。
[0055]
结果发现,两种探针在a基因组物种a.duranensis和a.diogoi、b基因组物种a.ipaensis、e基因组物种a.paraguariensis和a.stenophylla、k基因组物种a.batizocoi的染色体上均没有染色信号。探针pus
‑
187在h基因组物种a.pusilla和a.dardonoi染色体上均产生红色信号(图中亮点),表明该探针是h基因组特异探针,可以特异识别h基因组物种。探针dard
‑
127只在a.dardonoi染色体上产生绿色信号(图中亮点),表明该探针是h基因组物种a.dardonoi的特异探针,可以特异识别a.dardonoi。
[0056]
实施例4、一种花生野生种b基因组特异探针的使用方法
[0057]
首先,通过染色体压片法,获得花野生种a.duranensis(a基因组)、a.diogoi(a基因组)、a.ipaensis(b基因组)、a.paraguariensis(e基因组)、a.stenophylla(e基因组)、a.pusilla(h基因组)、a.dardonoi(h基因组)和a.batizocoi(k基因组)二倍体野生种根尖细胞有丝分裂中期制片,在
‑
80℃冰冻揭片,无水乙醇脱水,风干。
[0058]
其次,在染缸内加入40ml 2
×
ssc缓冲液(由0.3m的柠檬酸三钠c6h5na3o7·
2h2o和3m的nacl组成)、0.1od的ipa
‑
163,以及5μl的dapi母液(100μg/ml的dapi染色液),将野生种染色体制片置于染缸中染色5h,荧光显微镜拍照,获得染色图片(图2)。
[0059]
结果发现,两种探针在a基因组物种a.duranensis和a.diogoi、e基因组物种a.paraguariensis和a.stenophylla、h基因组物种a.pusilla和a.dardonoi的染色体上均没有染色信号。探针ipa
‑
163只在b基因组物种a.ipaensis、k基因组物种a.batizocoi染色体上产生红色信号(图中亮点),表明该探针是b和k基因组特异探针,同时暗示了与其它基因组相比,b与k基因组可能存在更近的亲缘关系。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1