一种RNA测序文库的构建方法及其应用与流程
一种rna测序文库的构建方法及其应用
技术领域
1.本发明涉及高通量基因测序技术领域,尤其涉及一种rna测序文库的构建方法及其应用。
背景技术:
2.随着高通量测序技术(ngs)的发展,自2004年问世以来,高通量测序成本已降低了几个数量级,使其在肿瘤检测、基因筛查等领域得到了广泛应用。自2014年首次报道了其在病原微生物检测领域的成功应用,在随后的几年里其在临床病原检测领域得到了广泛应用,尤其是在常规检测方法存在局限性的领域。ngs在临床微生物检测中的应用是多种多样的,包括宏基因组ngs,即mngs,它可以无偏性的检测病原体,可用于从患者的临床样本中直接鉴定病原体,而无需依赖于传统的培养法,为难以培养或不能实验室培养的病原体检测提供了可靠的检测平台。
3.mngs虽能无假设或无偏的检测微生物并发现新的微生物,但其仍存在一定的缺点,如患者样本中宿主背景核酸的干扰,研究表明临床样本中用于鉴定的病原微生物的序列是相对较少的,绝大多数为宿主(>99%)的核酸序列,这为mngs在病原体检测中的应用带来了很大的挑战。宿主序列可在样本制备和生物信息学分析阶段进行宿主剔除,目前在宿主去除方面已取得了较好的效果。mngs另一方面的缺点是检测样本中存在背景微生物的污染,包括用于提取、建库等步骤的试剂或实验室环境中的微生物污染。实验室环境中的微生物污染可通过净化实验室环境得到有效改善。但来源于检测试剂中的污染很难通过常规手段去除,为后续测序数据分析带来极大的困难。
4.目前,ngs已经广泛应用于rna测序,在rna测序过程中通常需要构建rna测序文库,现有技术中构建rna测序文库一般包括以下步骤:将rna片段采用机械法或酶法打断到一定长度、纯化打断的rna、利用随机引物进行反转录、合成cdna二链、末端修复和加a加接头、纯化、index pcr扩增和index pcr产物纯化。这样的建库方法包括三次繁琐的纯化步骤,对实验试剂、耗材、时间和人力造成了很大的浪费。同时,建库过程中需要采用随机引物,随机引物有时会带来反转录的不平衡,给测序结果带来一定偏差。
5.因此,开发一种新型rna测序文库的构建方法并对其进行合理利用显得非常有必要,对提高了rna文库构建的质量和效率,解决或降低试剂中微生物核酸对病原体检测造成的污染具有非常重要的意义。
技术实现要素:
6.本发明的主要目的在于提供一种rna测序文库的构建方法及其应用,该构建方法通过在文库构建前为rna模板加一段标签标记,有效降低了试剂盒中的背景微生物核酸的噪音污染,优化了生信分析流程,提高了rna测序检测的灵敏度;该方法不仅可标记rna模板,还可有效保留rna模板的相关信息,将rna模板转化为dna进行文库构建,同时将rna模板转化为可用于文库构建的小片段,长度为50~1000bp;通过该技术标记的核酸与dna模板一
样,一起进行文库构建,通过生物信息学分析能有效的将两组核酸进行区分。
7.为达到以上目的,本发明采用的技术方案为:一种rna测序文库的构建方法,其特征在于,包括在文库构建前通过标签对靶标rna序列进行标记步骤,所述标签命名为rx,由两部分组成:5'端为特定的标签序列tx;3'端为随机序列nx;标签rx中的x为数字,用户可根据需求进行命名,如r1、r2、r3
……
;标签序列tx为一段固定的核酸序列,由5
‑
18个碱基组成,用户可根据需要进行选取,但应避免与靶标序列相同;随机序列nx由一段随机的碱基序列组成,n可为a、t、c、g中的任一个,x表示含有n的个数,其碱基数量可为5
‑
15个。
8.进一步地,所述通过标签对靶标rna序列进行标记步骤,具体为:
9.步骤s1、利用标签序列tx为rna模板进行标记,其碱基序列是固定的,在数据分析时可根据该序列对模板进行筛选区分,去除试剂中的背景噪音;
10.步骤s2、所述随机序列nx与模板上互补的区域结合形成rna
‑
dna双链结构,该结构可被逆转录酶识别并与之结合生成cdna的第一条链,生成该cdna链的5'端即标记有标签tx;
11.步骤s3、当rx中的随机序列nx与合成第一链cdna中互补的区域结合时,其可与cdna第一链形成cdna
‑
cdna双链结构,该结构可被dna聚合酶识别,并与之结合合成cdna的第二条链,随着cdna第二链的合成,所生成的cdna第二链的5'端和3'端均含有固定的碱基标签序列,5'端为rx,3'端为rx的反向互补序列,生成rx标记的序列。
12.进一步地,步骤s3中所述rx标记的序列为rna模板的一部分,其长度为为50~1000bp。
13.进一步地,所述rna测序文库的构建方法还可以将长的rna打断为短的rna序列,即将rna进行片段化。
14.进一步地,所述rna测序文库的构建方法还包括将通过该方法所形成的核酸序列可直接按相应测序平台建库流程进行文库构建,或将该方法所形成的核酸序列与同一样本制备的经片段化后的dna混合,然后按相应测序平台建库流程进行文库构建。
15.优选地,所述测序平台为illumina平台、ion torrent平台中的一种,但不限于这两种测序平台。
16.进一步地,一种rna测序文库的构建方法的应用,将该方法所形成的核酸序列直接按相应测序平台建库流程进行文库构建,其数据分析流程包括如下步骤:
17.(1)对测序数据进行质量评估,去除低质量、长度小于50bp的短序列、接头等低质量的reads;
18.(2)筛选数据中5'含有tx的序列或3'含有tx的反向互补序列的序列;
19.(3)将上步筛选的序列与宿主基因组序列进行比对,去除可与宿主基因组匹配的序列,此处宿主为人基因组;
20.(4)将筛选的数据与相应数据库进行比对分析,确定其所属物种;
21.(5)生成相应的检测报告。
22.进一步地,一种rna测序文库的构建方法的应用,将该方法所形成的核酸序列与同一样本制备的经片段化后的dna混合,然后按相应测序平台建库流程进行文库构建,其数据分析流程包括如下步骤:
23.步骤c1:对测序数据进行质量评估,去除低质量、长度小于50bp的短序列、接头等
低质量的reads;
24.步骤c2:将符合要求的数据中按5'端含有tx的序列或3'端含有tx的反向互补序列的序列分为一组,记为rna组;5'端不含有tx的序列且3'端不含有tx的反向互补序列的序列分为一组,记为dna组;
25.步骤c3:将rna组数据与宿主人基因组序列进行比对,去除可与宿主基因组匹配的序列;
26.步骤c4:将上步筛选的rna组剩余数据与病原体基因组数据库进行比对分析,确定其所含有的rna病原体;
27.步骤c5:将dna组数据与宿主人基因组序列进行比对,去除可与宿主基因组匹配的序列;
28.步骤c6:将上步筛选的dna组剩余数据与病原体基因组数据库进行比对分析,确定其所含有的dna病原体;
29.步骤c7:根据rna组和dna组的分析结果确定样本中所含有的病原体;
30.步骤c8:生成相应的检测报告。
31.进一步地,所述标签序列rx为r1,由t8和n6组成;r1核酸序列为5'
‑
cagatatcnnnnnn
‑
3';所用的t8含有8个固定碱基,其序列为cagatatc;n6含有6个随机碱基,其序列为nnnnnn。
32.进一步地,所述cdna的第一条链合成方法,包括如下步骤:将待建库的靶标rna与标签r1混匀,r1终浓度为2μm,加入rnase抑制剂、dtt、dntp、amv逆转录酶及其相应buffer,各组分浓度分别为:rnase抑制剂1u/μl,amv逆转录酶0.05
‑
0.5u/μl(优选0.1u/μl),dtt为5mm,dntp为1mm,混合均匀,将反应管置于pcr仪中,运行下述程序:25℃10min,42℃10
‑
60min(优选30min),70℃5min,4℃hold。
33.进一步地,所述cdna的第二条链合成方法,包括如下步骤:待上述反应程序结束后,将反应管取出,加入终浓度为0.05~0.5u/μl(优选0.1u/μl)的klenow大片段(3
′→5′
exo
‑
),混合均匀,将反应管置于pcr仪中,运行下述程序:25℃10~60min(优选30min),75℃5min,4℃hold。
34.进一步地,所述rna测序文库的构建方法,还包括反应结束后,将反应产物用1.8
×
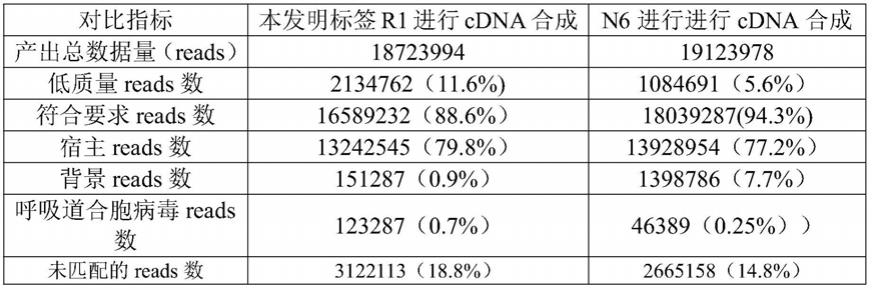
磁珠进行纯化,纯化产物直接用于文库构建或与片段化后的dna核酸混合进行文库构建。
35.由于上述技术方案运用,本发明与现有技术相比具有下列优点:
36.(1)本发明的一种用于rna文库构建的方法,在文库构建前为rna模板加一段标签标记,可有效降低试剂盒中的背景微生物核酸的噪音污染,优化生信分析流程。
37.(2)本发明的一种用于rna文库构建的方法,该方法不仅可标记rna模板,还可有效保留rna模板的相关信息,将rna模板转化为dna进行文库构建,同时将rna模板转化为可用于文库构建的小片段,长度为50~1000bp。
38.(3)本发明的一种用于rna文库构建的方法,通过其标记的核酸与dna模板一样,一起进行文库构建,通过生物信息学分析可有效的将两组核酸进行区分。该技术可明显减少试剂盒的背景噪音,可有效提高rna测序检测的灵敏度,可用于rna病毒测序检测的研究。
39.(4)本发明的一种用于rna文库构建的方法,可有效降低rna文库构建过程中试剂核酸的污染,提高rna的检出灵敏度,同时可将rna模板进行片段化,直接用于后续的文库构
建。
40.(5)本发明的一种用于rna文库构建的方法,该方法还可用于rna病原体与dna病原体核酸的同时检测,能有效检出样本的rna病原体和dna病原体,提高rna病原体检出灵敏度和特异性,适用于病原体检测领域。
具体实施方式
41.以下描述用于揭露本发明以使本领域技术人员能够实现本发明。以下描述中的优选实施例只作为举例,本领域技术人员可以想到其他显而易见的变型。
42.下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
43.下述实施例涉及的仪器包括:超净工作台、离心机、qubit4.0,pcr仪、移液器、磁力架、测序仪等。
44.下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
45.实施例1
46.本实施例公开了一种用于rna文库构建的方法,包括如下步骤:
47.(一)rna逆转录为cdna
48.(1)取提取的呼吸道合胞病毒核酸,分为2份,每份13μl,第一份核酸加入4μl 5
×
buffer,只含有n6序列的随机引物(终浓度2μm)、dntp(终浓度1mm)、dtt(终浓度5mm)、rnase抑制剂(终浓度1u/μl)、amv逆转录酶(终浓度0.1u/μl),无核酸酶水补至20μl;第二份核酸加入4μl 5
×
buffer,标签序列r1(终浓度2μm)、dntp(终浓度1mm)、dtt(终浓度5mm)、rnase抑制剂(终浓度1u/μl)、amv逆转录酶(终浓度0.1u/μl),无核酸酶水补至20μl。
49.(2)将配制好的2份体系,漩涡混匀,瞬时离心,置于pcr仪中进行反应;反应程序为:25℃5min;42℃30min;70℃5min;4℃hold;
50.(3)反应结束后,向反应体系体系中加入1μlklenow酶,混合均匀,瞬时离心,置于pcr仪中进行反应;反应程序为:25℃30min;75℃5min;4℃hold
51.(4)反应结束后用1.8
×
的ampure xp磁珠进行纯化,得到逆转录后的病毒核酸。
52.(二)文库构建
53.本发明采用的ion proton测序平台,建库方法采用ion proton平台建库流程。所用试剂盒为ion xpress fragment library kit和ion xpress barcode adapters 1
–
16kit操作流程按照操作说明书进行。
54.(1)取12.5μl上述逆转录的核酸,依次加入24.5μl无核酸酶水、5μl连接酶buffer、1μl dntp、1μldna连接酶、4μl缺壳修复酶、1μl通用接头、1μl含有barcode x的接头(每个样本采用不同的barcode);混合均匀,瞬时离心,将反应管置于pcr仪中,反应程序:25℃,20min;72℃,5min;4℃,hold。
55.(2)反应结束后,用1.5
×
ampure xp磁珠进行纯化,用14μlte洗脱核酸,取12.5μl加接头的核酸溶液,加入50μl pcr混合液,2.5μl扩增引物;混合均匀,瞬时离心,将反应管置于pcr仪中;反应程序:预变性95℃,5min;循环相(10个循环)95℃15s,58℃15s,70℃1min;4℃,hold;
56.(3)反应结束后,用1.5
×
ampure xp磁珠进行纯化,用20μl te洗脱核酸。
57.(4)按要求进行文库混合、文库模板制备和上机测序。
58.(三)数据分析及结果对比
59.1、采用标签r1进行逆转录的样本测序数据的分析
60.对测序数据进行质量评估,去除低质量、长度小于50bp的段序列、接头等低质量的reads。筛选数据中5'含有t8:cagatatc的序列或3'含有t8的反向互补序列gatatctg的序列。将上步筛选的数据与宿主人基因组序列进行比对,去除可与宿主基因组匹配的序列。将筛选的数据与微生物病原体数据库进行比对分析,确定其所属物种。生成相应的检测报告。
61.2、采用n6引物进行逆转录样本的数据分析
62.对测序数据进行质量评估,去除低质量、长度小于50bp的段序列、接头等低质量的reads。将上步筛选的数据与宿主人基因组序列进行比对,去除可与宿主基因组匹配的序列。将筛选的数据与微生物病原体数据库进行比对分析,确定其所属物种。生成相应的检测报告。
63.3、检测结果对比。
64.测序数据中各数据所占比例对比如下表1所示。在数据量产出相差不大的情况下,采用本发明标签r1进行cdna合成最终产出数据背景噪音序列仅占有用数据的0.9%,而采用常规引物进行cdna合成最终产出数据背景噪音序列占有用数据的7.7%,采用本发明的方法其背景噪音减少接近90%(88.2%)。采用本发明检出的呼吸道合胞病毒核酸reads数明显多于常规方法检出的reads数123287vs 46389。说明采用该技术可有效的降低试剂中的背景核酸,并提高rna病毒的检出灵敏度。表1本发明与普通rna文库构建方法数据对比
65.实施例2
66.本实施例公开了一种rna病原体核酸与dna病原体核酸进行共测序的方法,包括如下步骤:
67.(一)样本来源及核酸提取
68.1.样本来源:经临床确诊为rna病毒、dna病毒和细菌共感染的肺泡灌洗液样本3例。
69.2.取300μl样本,12000rpm离心2min,取上清液采用qiagen的病毒核酸提取试剂盒进行病毒核酸的提取。操作流程按操作说明书进行。
70.3.剩余沉淀采用金麦格细菌基因组核酸提取试剂盒进行细菌基因组核酸的提取。操作流程按提取说明书进行。
71.(二)rna病原体提取核酸的cdna合成
72.将每个样本提取的病毒核酸取13μl,采用标签r1进行cdna的合成,具体方法参照
实施例1,另取病毒核酸13μl,采用n6引物进行cdna的合成,具体方法参照实施例1进行。
73.(三)rna病原体与dna病原体核酸共建库及上机测测序
74.1、dna核酸片段化取20μl提取的病毒核酸和20μl提取的细菌核酸进行混合,然后采用kapa片段化试剂盒进行核酸片段化。具体操作流程如下:取混合核酸40μl,加入5μl片段化酶和5μl片段化buffer;混合均匀,瞬时离心,将反应管置于pcr仪中进行反应;反应程序为:4℃,1min;37℃,40min;4℃,hold;反应结束后,加入5μl终止缓冲液,旋涡混匀,瞬时离心;用1.8
×
ampure xp磁珠进行纯化;用无核酸酶水洗脱核酸
75.2、分别将r1合成的cdna与经片段化的dna核酸按等体积混合,同时将用n6合成的cdna与经片段化的dna核酸按等体积混合。然后,将每个样本的两组核酸采用ion xpress fragment library kit和ion xpress barcodeadapters 1
–
16kit进行文库构建,操作流程参照实施例1进行。
76.3、将构建好的文库按照要求进行文库混样、模板制备和上机测序。
77.(四)数据分析
78.1、含有标签r1的测序数据的生物信息学分析:
79.(1)对测序数据进行质量评估,去除低质量、长度小于50bp的短序列、接头等低质量的reads;
80.(2)将符合要求的数据中按5’含有t8:cagatatc的序列或3’含有tx的反向互补序列gatatctg的序列分为一组,记为rna组;5’端不含有t8:cagatatc的序列且3’不含有tx的反向互补序列的序列分为一组,记为dna组;将rna组数据与宿主人基因组序列进行比对,去除可与宿主基因组匹配的序列;
81.(3)将上步筛选的rna组剩余数据病原体基因组数据库进行比对分析,确定其所含有的rna病原体基因;将dna组数据与宿主人基因组序列进行比对,去除可与宿主基因组匹配的序列;将上步筛选的dna组剩余数据与病原体基因组数据库进行比对分析,确定其所含有的dna病原体基因;根据rna组和dna组的分析结果确定样本所含有的病原体;生成相应的检测报告。
82.2、采用n6引物进行逆转录样本的数据分析
83.(1)对测序数据进行质量评估,去除低质量、长度小于50bp的段序列、接头等低质量的reads。
84.(2)将上步筛选的数据与宿主人基因组序列进行比对,去除可与宿主基因组匹配的序列。
85.(3)将筛选的数据与微生物病原体数据库进行比对分析,确定其所属物种。
86.(4)生成相应的检测报告。
87.3、检测结果分析
88.用于测序的3个样本检测结果如表2、3、4所示,由下表检测结果可以看出,采用本发明的rna建库方法,可有效减少共建库的背景噪音,噪音减少在80%~90%,对数据分析具有正面影响。且在共感染样本中rna病毒的检出效果明显优于传统rna建库技术,rna检出量提高30%~150%不等。且对dna的检出无明显影响。由此可见该rna建库技术可用于rna病原体与dna病原体的共测序检测。表2样本balf001检测结果
表3样本balf002检测结果表4样本balf003检测结果表4样本balf003检测结果
89.以上所述仅是本发明的部分实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进,这些改进应视为本发明的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1