高热稳定性高活性异丁香酚单加氧酶突变体及其应用
本发明属于生物工程技术领域,具体涉及一种高热稳定性高活性异丁香酚单加氧酶突变体及其应用。
背景技术:
异丁香酚单加氧酶(isoeugenolmonooxygenase)简称iem,以异丁香酚为底物,在无需任何辅酶和辅因子的存在下,通过氧化裂解连接苯环的丙烯基从而生成香草醛。而香草醛(4-羟基-3-甲氧基苯甲醛)又名香兰素,由于其独特的香气和性质,被广泛应用于食品、化工和医药等领域。香兰素在食品中可以作为调味剂加入至蛋糕、冰激凌中以增强其香气和风味;在化工行业中常用于检验间苯二酚、单宁酸的存在;在制药行业中也是许多药物的生产原料和重要中间体,例如其可用于制备多巴胺、抗癫痫药等(黄希顺,黄丽娟,魏建科.香草醛治疗癫的疗效观察[j].实用神经疾病杂志.2005,8(4):78.)。
目前市场上的香兰素大多数都是化学法合成的,经研究发现,使用大剂量化学法合成的香兰素会导致头痛、呕吐、呼吸困难等不良反应,对人体危害较大。因此近年来,消费者们对于天然香料的呼声越来越高,导致天然香兰素的需求量也在大幅增长。而天然香兰素主要存在于香荚兰豆中,可通过超临界co2萃取技术进行提取,但该方法提取出的天然香兰素价格非常昂贵,无法满足市场需求(符史良,周江,黄茂芳,等.超临界co2萃取香草兰的工艺研究和成分分析[j].食品与机械.2002,(2):12-14.)。随着生物催化技术的迅速发展,其可在较短的时间内进行高效、快速的反应,并且拥有绿色环保、选择性和专一性强等优点,使得该方法更适合商业化生产。
异丁香酚是合成香兰素的最佳前体物质,而iem是生物转化异丁香酚的唯一关键酶,该方法合成出来的香兰素在香气上最接近于天然香兰素,因此引起广泛的关注。
蛋白质工程中的理性设计是基于对目的蛋白的序列、结构、催化机制等酶学信息有一定的了解下,借助各类生物信息学软件,预测不同位点的突变对于目的蛋白稳定性和催化性能等方面的影响。全质粒pcr法是目前应用最广泛的定点突变方法之一,具有操作简便、快速、高效等优点。对已报道微生物来源的iem研究发现,主要围绕外源表达、酶学性质的研究居多(yamadam,okaday,yoshiday,etal.vanillinproductionusingescherichiacolicellsover-expressingisoeugenolmonooxygenaseofpseudomonasputida[j].biotechnologyletters.2008,30(4):665-670.),而针对分子改造方面的研究较少。中国专利申请cn106754802b公开了一种异丁香酚单加氧酶突变体及其应用,属于生物技术领域,针对产物抑制的问题,对异丁香酚单加氧酶基因进行定点突变,最终获得了催化活性比亲本至少高出117%的突变酶,然而,该异丁香酚单加氧酶突变体不具有高热稳定性。因此,本发明首次针对该酶热稳定性差的问题,借助生物信息学软件对其进行定点突变,并且在提高热稳定性的同时还进一步提高了异丁香酚单加氧酶的催化活性。
发明人在前期的研究中从ncbi库中报道的硝基还原假单胞菌(pseudomonasnitroreducens)jin1基因组中获得编码异丁香酚单加氧酶(iem)的基因序列,并将其克隆至大肠杆菌bl21(de3)中成功实现表达,但该酶存在一定的缺陷,特别是热稳定性较差,只能在较低温度下具有较好的催化效果,使得其在工业应用中有一定的局限性。
技术实现要素:
本发明的目的就是为了解决上述问题而提供一种异丁香酚单加氧酶突变体及其应用,本发明以表面氨基酸(gly-ala,lys-arg)替换和共识理论为指导思想,借助生物信息学软件getarea和consensusfinder的辅助,获得了具有高热稳定性、高活性的异丁香酚单加氧酶突变体。
为了解决上述技术问题,本发明先基于嗜热蛋白表面氨基酸具有gly替换为ala,lys替换为arg的原则,利用getarea软件找到iem表面总共有6个gly和14个lys,并对其分别进行定点突变,获得了2个热稳定性有所提升的突变酶;再基于同源序列中保守氨基酸较非保守氨基酸更为稳定的原则,利用consensusfinder软件对iem进行多序列比对,筛选出阈值大于70%的氨基酸总共有9个,将其分别定点突变成相应的保守氨基酸后,获得了1个酶活有所提升的突变酶。
将上述三个氨基酸组合突变之后,得到了一个热稳定性显著提升以及比活也有所提高的三位点突变体,解决了现有的iem热稳定性较差,无法满足工业化生产的问题。
一种高热稳定性高活性异丁香酚单加氧酶突变体,其包括如seqidno.1所示的氨基酸序列,其具有选自第83位、第95位或第273位中的至少一个突变。
进一步地,所述第83位的赖氨酸k突变为精氨酸r。
进一步地,所述第95位的赖氨酸k突变为精氨酸r。
进一步地,所述第273位的亮氨酸l突变为苯丙氨酸f。
优选地,对如seqidno.1所示的氨基酸序列中的第83位的赖氨酸k、第95位的赖氨酸k、第273位的亮氨酸l分别突变为精氨酸r、精氨酸r、苯丙氨酸f。
本发明所述的比活及热稳定性提高的异丁香酚单加氧酶突变体,是对亲本硝基还原假单胞菌(pseudomonasnitroreducens)jin1来源的异丁香酚单加氧酶iem(其氨基酸序列如ncbi登录号acp17973.1所示,核苷酸序列如ncbi登录号fj851547所示)进行定点突变,通过全质粒pcr技术对iem第83位的赖氨酸k、第95位的赖氨酸r、第273位的亮氨酸l引入突变,用于表达所述突变体的载体为pet21a,将含突变后基因的重组质粒转化至宿主大肠杆菌(escherichiacoli)bl21(de3)中进行表达。
所述突变是将第83位的赖氨酸k突变为精氨酸r、第95位的赖氨酸k突变为精氨酸r、第273位的亮氨酸l突变为苯丙氨酸f。采用“原始氨基酸缩写+突变位置+替换氨基酸缩写”的命名方式来表示所述的异丁香酚单加氧酶突变体k83r/k95r/l273f。
与亲本硝基还原假单胞菌jin1的异丁香酚单加氧酶相比(纯酶的比活为3.4u/mg),所述异丁香酚单加氧酶突变体k83r、k95r、l273f、k83r/k95r和k83r/k95r/l273f的比活分别为6.1u/mg、5.3u/mg、5.5u/mg、6.0u/mg和7.0u/mg,其中k83r/k95r/l273f显示出最佳的比活,较亲本相比提高了2.1倍。
与亲本硝基还原假单胞菌jin1的异丁香酚单加氧酶相比(25℃、30℃、35℃下的半衰期t1/2分别为16.1h、49.8min、7.8min),所述异丁香酚单加氧酶突变体k83r、k95r、l273f、k83r/k95r和k83r/k95r/l273f在25℃下的半衰期分别为亲本的1.1倍、1.1倍、0.6倍、2.3倍和3.0倍(t1/2分别为18.2h、18.5h、10.0h、37.1h和47.5h),在30℃下的半衰期较亲本分别提高了5.3倍、4.0倍、2.7倍、9.9倍和12.0倍(t1/2分别为262.2min、198.0min、133.2min、491.4min和594.0min),而在35℃下的半衰期较亲本分别提高了4.1倍、3.7倍、1.6倍、12.2倍和24.7倍(t1/2分别为31.8min、28.8min、12.6min、94.8min和192.6min)。
本发明还提供了上述异丁香酚单加氧酶突变体的应用,应用于以异丁香酚为底物制备香兰素,在底物浓度为100mm(助溶剂为10%的dmso),反应温度为30℃,ph为9.0,细胞量为15g/l,转化12h后,香兰素的转化率可达87%,而亲本在相同条件下的转化率为75%,提高了1.16倍。
与现有技术相比,本发明有益效果为:
以表面氨基酸的替换和共识理论为指导思想,对异丁香酚单加氧酶基因进行定点突变,获得了热稳定性和比活均提高的iem突变酶k83r/k95r/l273f,在30℃下半衰期可达野生型酶的12.0倍,比活为野生型酶的2.1倍。该突变酶能以异丁香酚为底物,在较高的温度下高效催化转化异丁香酚合成香兰素,在30℃和35℃下香兰素的转化率分别比亲本高出16%和35%。
附图说明
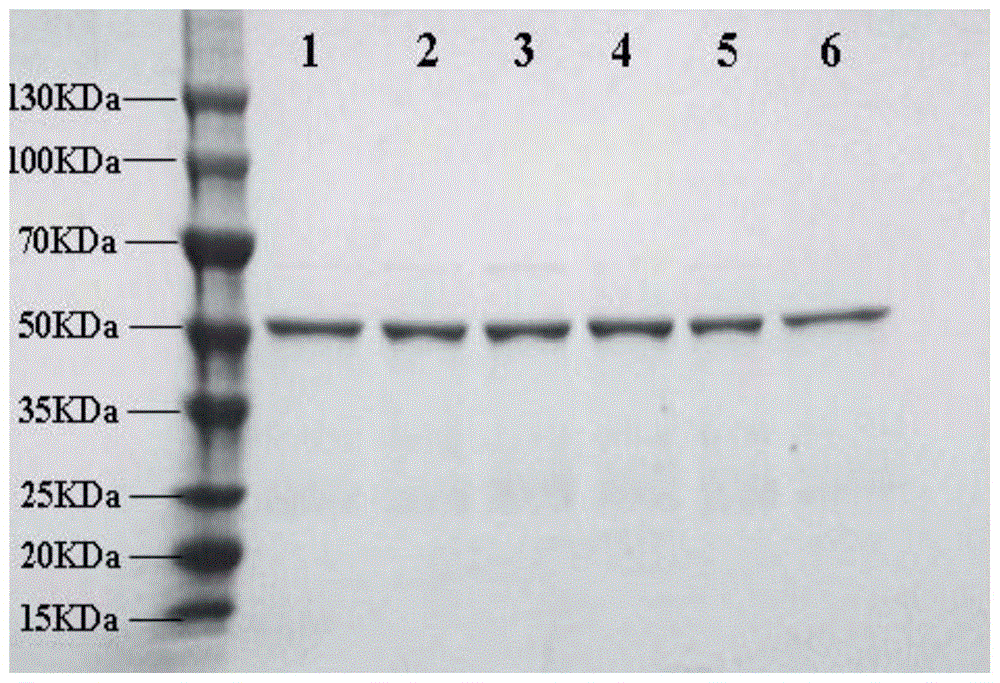
图1为本发明构建的突变体以及野生型iem的纯酶蛋白电泳图;
其中,泳道1代表亲本,泳道2代表突变体k83r,泳道3代表突变体k95r,泳道4代表突变体l273f,泳道5代表突变体k83r/k95r,泳道6代表突变体k83r/k95r/l273f。
图2为本发明构建的突变体以及野生型iem的比活比较;
图3a、3b、3c为本发明构建的突变体以及野生型iem在不同温度下的失活半衰期比较。
具体实施方式
下面结合附图和具体实施例对本发明进行详细说明。
实施例中涉及到的培养基配方如下:
lb液体培养基:胰蛋白胨10g/l,酵母浸粉5g/l,nacl10g/l,ph7.0。
lb固体培养基:在lb液体培养基的基础配方上添加15g/l琼脂。
发酵培养基:na2hpo47g/l,kh2po43g/l,酵母浸粉10g/l,nacl0.5g/l,葡萄糖14g/l,ph7.0。
异丁香酚单加氧酶的酶活测定:
测活反应体系为(1ml):纯酶50μg与0.1mtris-hcl(ph8.0)缓冲液混合,再与200mm底物异丁香酚在30℃下预热3min,随即加入50μl底物至反应体系中,使得其终浓度为10mm,在恒温混匀仪上30℃,1000rpm反应10min后,加入1ml液相甲醇进行淬灭,于涡旋仪混匀后置于离心机中12000rpm离心2min,用无菌注射器吸取上清进
hplc分析。
分析方法(hplc法):色谱柱为反相柱c18(diamonsilplus,4.6mm*250mm*5μm);流动相(梯度洗脱):1-8min甲醇:水(ph2.5)=35:65,9-16min甲醇:水(ph2.5)=65:35;17-24min甲醇:水(ph2.5)=35:65;检测波长为280nm;柱温设定为25℃;进样量为20μl。
酶活定义:在30℃,ph8.0的条件下,单位时间内生成1μmol产物香兰素所需要的酶量定义为一个单位u。
热稳定性的测定:将稀释好的纯酶分装,并分别置于25℃、30℃、35℃的金属水浴锅中进行保温,毎隔一段时间取样测定残余酶活,以保温时间为x轴,残余酶活的对数为y轴拟合线性关系图,再根据t1/2=ln2/kd得出半衰期。
全细胞催化异丁香酚合成香兰素:取15g/l异丁香酚单加氧酶及其突变体的细胞,与100mm底物异丁香酚(溶剂:dmso)在ph9.0的条件下,反应温度为20~35℃,反应时间为2、4、8、12h,从而观察香兰素转化率的变化。
产物香兰素的转化率计算如下:
转化率α=(0.8823×(a-a0)+0.0286)×b/c×100%;
其中a代表外标法积分峰面积,a0代表底物自发氧化峰面积,b代表总反应体系的稀释倍数,c代表底物异丁香酚的浓度。关系式y=0.8823x+0.0286为香兰素的外标法标准曲线。
下面以具体实施例对本发明进一步说明。
实施例1
异丁香酚单加氧酶基因的克隆以及重组工程菌的构建
根据蛋白质ncbi库,从硝基还原假单胞菌jin1中获取一段异丁香酚单加氧酶基因(其氨基酸序列如ncbi登录号acp17973.1所示,核苷酸序列如ncbi登录号fj851547所示),并交由上海捷瑞生物工程有限公司进行合成,这段异丁香酚单加氧酶iem的氨基酸序列如seqidno.1所示,其核苷酸序列如seqidno.7所示。通过上游引物5’-cgccatatgatggcgagactcaaccgcaac-3’(下划线碱基为限制性内切酶ndei识别位点)和下游引物5’-ccgctcgagttaaggtctgggtacccagca-3’(下划线碱基为限制性内切酶xhoi识别位点)扩增目的基因,pcr扩增使用宝日医生物技术(北京)有限公司(takara中国)的primestarmax高保真聚合酶,pcr反应体系如下:(引物浓度为10μmol/l):
pcr扩增程序为:98℃预变性2min;98℃变性10s,55℃退火15s,72℃延伸60s,30个循环;72℃延伸7min;4℃保存。
反应结束后用1%的琼脂糖凝胶电泳检测pcr产物,得到1.5kb的条带,符合预期目标蛋白长度。按照试剂盒操作,割胶回收、纯化该目的片段,使用限制性内切酶ndei和xhoi对回收片段和pet21a质粒进行双酶切,ligationmix用于连接,连接产物转化至大肠杆菌bl21(de3)感受态细胞中,涂布于含氨苄霉素(100μg/ml)的lb平板上,提取阳性克隆质粒送样测序,结果显示插入的iem基因序列正确,命名重组菌为e.colibl21(de3)/pet21a-iem。
实施例2
基于全质粒pcr法构建异丁香酚单加氧酶突变体
以重组质粒pet21a-iem为模板,利用pcr体外扩增含突变基因的质粒。
用于定点突变的引物为(下划线为突变位点):
k83rprimer-f:5’-catcagtcgctgggttaggaccgctcgattcacg-3’
k83rprimer-r:5’-cgtgaatcgagcggtcctaacccagcgactgatg-3’
k95rprimer-f:5’-gaacgactagcgcgaagatcgctatttggcatg-3’
k95rprimer-r:5’-catgccaaatagcgatcttcgcgctagtcgttc-3’
l273fprimer-f:5’-gtcagattcgctggttcaaggcaccggcgctc-3’
l273fprimer-r:5’-gagcgccggtgccttgaaccagcgaatctgac-3’
pcr反应体系如下:(引物浓度为10μmol/l)
pcr扩增程序为:95℃预变性10min;98℃变性10s,56℃退火15s,72℃延伸75s,24个循环;72℃延伸5min;4℃保存。
取10μlpcr得到的产物经核酸电泳验证正确后,用通用型dna纯化回收试剂盒进行反应液的回收。再利用dpni内切酶消化dna模板,酶切反应温度为37℃,反应时间为4h,然后将酶切产物转化至大肠杆菌bl21(de3)感受态细胞中。涂布lb抗性固体平板(内含100μg/mlamp),37℃培养12-14h,挑取1-3个转化子转移至lb液体培养基中(内含100μg/mlamp)培养6h待液体浑浊后,取菌液送样测序。
通过优势菌株筛选获得热稳定性较原始菌有提高的菌株e.colibl21(de3)/pet21a-iemk83r(氨基酸序列如seqidno.2所示,其核苷酸序列如seqidno.8所示)、e.colibl21(de3)/pet21a-iemk95r(氨基酸序列如seqidno.3所示,其核苷酸序列如seqidno.9所示),和酶活较原始菌有提高的菌株e.colibl21(de3)/pet21a-ieml273f(氨基酸序列如seqidno.4所示,其核苷酸序列如seqidno.10所示)。
第二轮以pet21a-iemk83r质粒为模板,以k95r-f和k95r-r为引物,经过全质粒pcr、转化、涂平板后,获得热稳定性有进一步提升的优势菌株e.colibl21(de3)/pet21a-iemk83r-k95r(氨基酸序列如seqidno.5所示,其核苷酸序列如seqidno.11所示)。第三轮以pet21a-iemk83r-k95r质粒为模板,以l273f-f和l273f-r为引物,经过全质粒pcr、转化、涂平板后,获得酶活和热稳定性较原始菌均有所提高的优势菌株e.colibl21(de3)/pet21a-iemk83r-k95r-l273f(氨基酸序列如seqidno.6所示,其核苷酸序列如seqidno.12所示)。
实施例3
亲本和突变菌株的诱导表达
平板活化:用接种环蘸取甘油管菌液,于lb抗性固体平板(内含100μg/mlamp)四区划线,在恒温培养箱中37℃倒置培养12h。
种子培养:从固体平板上挑取单菌落接种至lb液体培养基中(内含100μg/mlamp),装液量为50ml/250ml,37℃,200rpm摇床上振荡培养12h。
摇瓶发酵:将培养12h的种子液以1%的接种量接入发酵培养基中(内含100μg/mlamp),装液量为50ml/250ml,先37℃,200rpm培养至菌体浓度od600=0.6-0.8,待液体冷却至室温后,再加入终浓度为0.1mmiptg进行诱导,25℃下诱导表达12h。发酵完之后,在4℃离心机中10000rpm离心10min收集所有菌体,用0.8%生理盐水洗涤细胞两次。
实施例4
异丁香酚单加氧酶亲本和突变体的纯化
向收集完的菌体加入相应缓冲液重悬细胞至菌体浓度为10g/l,置于含冰水的烧杯中,用超声波破碎仪进行细胞破碎,破碎条件为:超声2s,间歇4s,功率38%,时间10min,共两次,破碎完再12000rpm离心30min,上清即粗酶液。利用苏州海狸his-tag蛋白纯化磁珠对异丁香酚单加氧酶的亲本和阳性突变体k83r、k95r、l273f、k83r/k95r和k83r/k95r/l273f进行纯化,磁珠表面ni2+能与蛋白his标签中的组氨酸形成配位键,将目标蛋白结合到磁珠上,然后先用100mm咪唑去除吸附上的杂蛋白,再用500mm咪唑洗脱目标蛋白,最后将目标蛋白置于millipore超滤离心管中进行浓缩和置换缓冲液。纯化后,取相同蛋白量和相同体积上样进行sds-page验证,结果如附图1所示,其中泳道1代表异丁香酚单加氧酶的亲本,泳道2代表突变体k83r,泳道3代表突变体k95r,泳道4代表突变体l273f,泳道5代表突变体k83r/k95r,泳道6代表突变体k83r/k95r/l273f。
实施例5
不同突变体与亲本酶的活性比较
将浓缩后的纯酶稀释,按照上述方法测定酶活,用bradford法测定蛋白浓度。如附图2所示,亲本iem的比活为3.4±0.15u/mg,突变体k83r、k95r、l273f、k83r/k95r和k83r/k95r/l273f的比活分别为6.1±0.04u/mg、5.3±0.02u/mg、5.5±0.09u/mg、6.0±0.14u/mg和7.0±0.04u/mg,较亲本分别提高了1.8、1.6、1.6、1.8和2.1倍。
实施例6
不同突变体与亲本酶的热稳定性比较
不同突变体与亲本酶的热稳定性比较主要通过测定不同温度下的半衰期来确定。如附图3a所示,25℃下亲本的半衰期为16.1h,而突变体k83r、k95r、l273f、k83r/k95r和k83r/k95r/l273f的半衰期为18.2h、18.5h、10.0h、37.1h和47.5h,分别为亲本的1.1倍、1.1倍、0.6倍、2.3倍和3.0倍。如附图3b所示,30℃下亲本的半衰期为49.8min,而突变体k83r、k95r、l273f、k83r/k95r和k83r/k95r/l273f的半衰期为262.2min、198.0min、133.2min、491.4min和594.0min,较亲本相比分别提高了5.3倍、4.0倍、2.7倍、9.9倍和12.0倍。如附图3c所示,35℃下亲本的半衰期为7.8min,而突变体k83r、k95r、l273f、k83r/k95r和k83r/k95r/l273f的半衰期为31.8min、28.8min、12.6min、94.8min和192.6min,分别较亲本提高了4.1倍、3.7倍、1.6倍、12.2倍和24.7倍。由此可见,三位点突变体k83r/k95r/l273f无论在低温还是高温下,都显示出最佳的热稳定性,说明组合突变之后,可以进一步提升酶的热稳定性。
实施例7
突变体iemk83r-k95r-l273f与亲本酶在不同温度下合成香兰素转化率的比较
在50ml离心管中,加入15g/l细胞量(亲本和突变酶)、100mmph9.0的gly-naoh缓冲液9ml、底物异丁香酚100mm(助溶剂:10%dmso)1ml,分别于20、25、30、35℃,180rpm下转化12h。反应完加入等体积的液相甲醇以淬灭反应,混匀后离心吸取上清进hplc分析。结果发现,突变酶和亲本在20℃和25℃下香兰素的转化率均接近100%,而在30℃和35℃时突变酶表现出比亲本更佳的催化效率,香兰素转化率分别为87%和70%,较亲本转化率分别提高16%和35%(30℃:75%;35℃:52%)。
序列表
<110>上海应用技术大学
<120>高热稳定性高活性异丁香酚单加氧酶突变体及其应用
<160>12
<170>siposequencelisting1.0
<210>1
<211>478
<212>prt
<213>人工序列(isoeugenolmonooxygenase)
<400>1
metalaargleuasnargasnaspproglnleuvalglythrleuleu
151015
prothrargileglualaaspleupheaspleugluvalaspglyglu
202530
ileprolysserileasnglythrphetyrargasnthrproglupro
354045
glnvalthrproglnlysphehisthrpheileaspglyaspglymet
505560
alaseralaphehisphegluaspglyhisvalasppheileserarg
65707580
trpvallysthralaargphethralagluargleualaarglysser
859095
leupheglymettyrargasnprotyrthraspaspthrservallys
100105110
glyleuaspargthrvalalaasnthrserileileserhishisgly
115120125
lysvalleualavallysgluaspglyleuprotyrgluleuasppro
130135140
argthrleugluthrargglyargpheasptyraspglyglnvalthr
145150155160
serglnthrhisthralahisprolystyraspprogluthrglyasp
165170175
leuleuphepheglyseralaalalysglyglualathrproaspmet
180185190
alatyrtyrilevalasplyshisglylysvalthrhisgluthrtrp
195200205
phegluglnprotyrglyalaphemethisaspphealailethrarg
210215220
asntrpserilepheproilemetproalathrasnserleuserarg
225230235240
leulysalalysglnproiletyrmettrpgluprogluleuglyser
245250255
tyrileglyvalleuproargargglyglnglyserglnileargtrp
260265270
leulysalaproalaleutrpvalphehisvalvalasnalatrpglu
275280285
valglythrlysiletyrileaspleumetglusergluileleupro
290295300
phepropheproasnserglnasnglnprophealaproglulysala
305310315320
valproargleuthrargtrpgluileaspleuaspserserserasp
325330335
gluilelysargthrargleuhisaspphephealaglumetproile
340345350
metasppheargphealaleuglncysasnargtyrglyphemetgly
355360365
valaspaspproarglysproleualahisglnglnalaglulysile
370375380
phealatyrasnserleuglyiletrpaspasnhisargglyasptyr
385390395400
aspleutrptyrserglyglualaseralaalaglngluproalaphe
405410415
valproargserprothralaalagluglyaspglytyrleuleuthr
420425430
valvalglyargleuaspgluasnargseraspleuvalileleuasp
435440445
thrglnaspileglnserglyprovalalathrilelysleuprophe
450455460
argleuargalaalaleuhisglycystrpvalproargpro
465470475
<210>2
<211>478
<212>prt
<213>人工序列(isoeugenolmonooxygenase)
<400>2
metalaargleuasnargasnaspproglnleuvalglythrleuleu
151015
prothrargileglualaaspleupheaspleugluvalaspglyglu
202530
ileprolysserileasnglythrphetyrargasnthrproglupro
354045
glnvalthrproglnlysphehisthrpheileaspglyaspglymet
505560
alaseralaphehisphegluaspglyhisvalasppheileserarg
65707580
trpvalargthralaargphethralagluargleualaarglysser
859095
leupheglymettyrargasnprotyrthraspaspthrservallys
100105110
glyleuaspargthrvalalaasnthrserileileserhishisgly
115120125
lysvalleualavallysgluaspglyleuprotyrgluleuasppro
130135140
argthrleugluthrargglyargpheasptyraspglyglnvalthr
145150155160
serglnthrhisthralahisprolystyraspprogluthrglyasp
165170175
leuleuphepheglyseralaalalysglyglualathrproaspmet
180185190
alatyrtyrilevalasplyshisglylysvalthrhisgluthrtrp
195200205
phegluglnprotyrglyalaphemethisaspphealailethrarg
210215220
asntrpserilepheproilemetproalathrasnserleuserarg
225230235240
leulysalalysglnproiletyrmettrpgluprogluleuglyser
245250255
tyrileglyvalleuproargargglyglnglyserglnileargtrp
260265270
leulysalaproalaleutrpvalphehisvalvalasnalatrpglu
275280285
valglythrlysiletyrileaspleumetglusergluileleupro
290295300
phepropheproasnserglnasnglnprophealaproglulysala
305310315320
valproargleuthrargtrpgluileaspleuaspserserserasp
325330335
gluilelysargthrargleuhisaspphephealaglumetproile
340345350
metasppheargphealaleuglncysasnargtyrglyphemetgly
355360365
valaspaspproarglysproleualahisglnglnalaglulysile
370375380
phealatyrasnserleuglyiletrpaspasnhisargglyasptyr
385390395400
aspleutrptyrserglyglualaseralaalaglngluproalaphe
405410415
valproargserprothralaalagluglyaspglytyrleuleuthr
420425430
valvalglyargleuaspgluasnargseraspleuvalileleuasp
435440445
thrglnaspileglnserglyprovalalathrilelysleuprophe
450455460
argleuargalaalaleuhisglycystrpvalproargpro
465470475
<210>3
<211>478
<212>prt
<213>人工序列(isoeugenolmonooxygenase)
<400>3
metalaargleuasnargasnaspproglnleuvalglythrleuleu
151015
prothrargileglualaaspleupheaspleugluvalaspglyglu
202530
ileprolysserileasnglythrphetyrargasnthrproglupro
354045
glnvalthrproglnlysphehisthrpheileaspglyaspglymet
505560
alaseralaphehisphegluaspglyhisvalasppheileserarg
65707580
trpvallysthralaargphethralagluargleualaargargser
859095
leupheglymettyrargasnprotyrthraspaspthrservallys
100105110
glyleuaspargthrvalalaasnthrserileileserhishisgly
115120125
lysvalleualavallysgluaspglyleuprotyrgluleuasppro
130135140
argthrleugluthrargglyargpheasptyraspglyglnvalthr
145150155160
serglnthrhisthralahisprolystyraspprogluthrglyasp
165170175
leuleuphepheglyseralaalalysglyglualathrproaspmet
180185190
alatyrtyrilevalasplyshisglylysvalthrhisgluthrtrp
195200205
phegluglnprotyrglyalaphemethisaspphealailethrarg
210215220
asntrpserilepheproilemetproalathrasnserleuserarg
225230235240
leulysalalysglnproiletyrmettrpgluprogluleuglyser
245250255
tyrileglyvalleuproargargglyglnglyserglnileargtrp
260265270
leulysalaproalaleutrpvalphehisvalvalasnalatrpglu
275280285
valglythrlysiletyrileaspleumetglusergluileleupro
290295300
phepropheproasnserglnasnglnprophealaproglulysala
305310315320
valproargleuthrargtrpgluileaspleuaspserserserasp
325330335
gluilelysargthrargleuhisaspphephealaglumetproile
340345350
metasppheargphealaleuglncysasnargtyrglyphemetgly
355360365
valaspaspproarglysproleualahisglnglnalaglulysile
370375380
phealatyrasnserleuglyiletrpaspasnhisargglyasptyr
385390395400
aspleutrptyrserglyglualaseralaalaglngluproalaphe
405410415
valproargserprothralaalagluglyaspglytyrleuleuthr
420425430
valvalglyargleuaspgluasnargseraspleuvalileleuasp
435440445
thrglnaspileglnserglyprovalalathrilelysleuprophe
450455460
argleuargalaalaleuhisglycystrpvalproargpro
465470475
<210>4
<211>478
<212>prt
<213>人工序列(isoeugenolmonooxygenase)
<400>4
metalaargleuasnargasnaspproglnleuvalglythrleuleu
151015
prothrargileglualaaspleupheaspleugluvalaspglyglu
202530
ileprolysserileasnglythrphetyrargasnthrproglupro
354045
glnvalthrproglnlysphehisthrpheileaspglyaspglymet
505560
alaseralaphehisphegluaspglyhisvalasppheileserarg
65707580
trpvallysthralaargphethralagluargleualaargargser
859095
leupheglymettyrargasnprotyrthraspaspthrservallys
100105110
glyleuaspargthrvalalaasnthrserileileserhishisgly
115120125
lysvalleualavallysgluaspglyleuprotyrgluleuasppro
130135140
argthrleugluthrargglyargpheasptyraspglyglnvalthr
145150155160
serglnthrhisthralahisprolystyraspprogluthrglyasp
165170175
leuleuphepheglyseralaalalysglyglualathrproaspmet
180185190
alatyrtyrilevalasplyshisglylysvalthrhisgluthrtrp
195200205
phegluglnprotyrglyalaphemethisaspphealailethrarg
210215220
asntrpserilepheproilemetproalathrasnserleuserarg
225230235240
leulysalalysglnproiletyrmettrpgluprogluleuglyser
245250255
tyrileglyvalleuproargargglyglnglyserglnileargtrp
260265270
phelysalaproalaleutrpvalphehisvalvalasnalatrpglu
275280285
valglythrlysiletyrileaspleumetglusergluileleupro
290295300
phepropheproasnserglnasnglnprophealaproglulysala
305310315320
valproargleuthrargtrpgluileaspleuaspserserserasp
325330335
gluilelysargthrargleuhisaspphephealaglumetproile
340345350
metasppheargphealaleuglncysasnargtyrglyphemetgly
355360365
valaspaspproarglysproleualahisglnglnalaglulysile
370375380
phealatyrasnserleuglyiletrpaspasnhisargglyasptyr
385390395400
aspleutrptyrserglyglualaseralaalaglngluproalaphe
405410415
valproargserprothralaalagluglyaspglytyrleuleuthr
420425430
valvalglyargleuaspgluasnargseraspleuvalileleuasp
435440445
thrglnaspileglnserglyprovalalathrilelysleuprophe
450455460
argleuargalaalaleuhisglycystrpvalproargpro
465470475
<210>5
<211>478
<212>prt
<213>人工序列(isoeugenolmonooxygenase)
<400>5
metalaargleuasnargasnaspproglnleuvalglythrleuleu
151015
prothrargileglualaaspleupheaspleugluvalaspglyglu
202530
ileprolysserileasnglythrphetyrargasnthrproglupro
354045
glnvalthrproglnlysphehisthrpheileaspglyaspglymet
505560
alaseralaphehisphegluaspglyhisvalasppheileserarg
65707580
trpvalargthralaargphethralagluargleualaargargser
859095
leupheglymettyrargasnprotyrthraspaspthrservallys
100105110
glyleuaspargthrvalalaasnthrserileileserhishisgly
115120125
lysvalleualavallysgluaspglyleuprotyrgluleuasppro
130135140
argthrleugluthrargglyargpheasptyraspglyglnvalthr
145150155160
serglnthrhisthralahisprolystyraspprogluthrglyasp
165170175
leuleuphepheglyseralaalalysglyglualathrproaspmet
180185190
alatyrtyrilevalasplyshisglylysvalthrhisgluthrtrp
195200205
phegluglnprotyrglyalaphemethisaspphealailethrarg
210215220
asntrpserilepheproilemetproalathrasnserleuserarg
225230235240
leulysalalysglnproiletyrmettrpgluprogluleuglyser
245250255
tyrileglyvalleuproargargglyglnglyserglnileargtrp
260265270
leulysalaproalaleutrpvalphehisvalvalasnalatrpglu
275280285
valglythrlysiletyrileaspleumetglusergluileleupro
290295300
phepropheproasnserglnasnglnprophealaproglulysala
305310315320
valproargleuthrargtrpgluileaspleuaspserserserasp
325330335
gluilelysargthrargleuhisaspphephealaglumetproile
340345350
metasppheargphealaleuglncysasnargtyrglyphemetgly
355360365
valaspaspproarglysproleualahisglnglnalaglulysile
370375380
phealatyrasnserleuglyiletrpaspasnhisargglyasptyr
385390395400
aspleutrptyrserglyglualaseralaalaglngluproalaphe
405410415
valproargserprothralaalagluglyaspglytyrleuleuthr
420425430
valvalglyargleuaspgluasnargseraspleuvalileleuasp
435440445
thrglnaspileglnserglyprovalalathrilelysleuprophe
450455460
argleuargalaalaleuhisglycystrpvalproargpro
465470475
<210>6
<211>478
<212>prt
<213>人工序列(isoeugenolmonooxygenase)
<400>6
metalaargleuasnargasnaspproglnleuvalglythrleuleu
151015
prothrargileglualaaspleupheaspleugluvalaspglyglu
202530
ileprolysserileasnglythrphetyrargasnthrproglupro
354045
glnvalthrproglnlysphehisthrpheileaspglyaspglymet
505560
alaseralaphehisphegluaspglyhisvalasppheileserarg
65707580
trpvalargthralaargphethralagluargleualaargargser
859095
leupheglymettyrargasnprotyrthraspaspthrservallys
100105110
glyleuaspargthrvalalaasnthrserileileserhishisgly
115120125
lysvalleualavallysgluaspglyleuprotyrgluleuasppro
130135140
argthrleugluthrargglyargpheasptyraspglyglnvalthr
145150155160
serglnthrhisthralahisprolystyraspprogluthrglyasp
165170175
leuleuphepheglyseralaalalysglyglualathrproaspmet
180185190
alatyrtyrilevalasplyshisglylysvalthrhisgluthrtrp
195200205
phegluglnprotyrglyalaphemethisaspphealailethrarg
210215220
asntrpserilepheproilemetproalathrasnserleuserarg
225230235240
leulysalalysglnproiletyrmettrpgluprogluleuglyser
245250255
tyrileglyvalleuproargargglyglnglyserglnileargtrp
260265270
phelysalaproalaleutrpvalphehisvalvalasnalatrpglu
275280285
valglythrlysiletyrileaspleumetglusergluileleupro
290295300
phepropheproasnserglnasnglnprophealaproglulysala
305310315320
valproargleuthrargtrpgluileaspleuaspserserserasp
325330335
gluilelysargthrargleuhisaspphephealaglumetproile
340345350
metasppheargphealaleuglncysasnargtyrglyphemetgly
355360365
valaspaspproarglysproleualahisglnglnalaglulysile
370375380
phealatyrasnserleuglyiletrpaspasnhisargglyasptyr
385390395400
aspleutrptyrserglyglualaseralaalaglngluproalaphe
405410415
valproargserprothralaalagluglyaspglytyrleuleuthr
420425430
valvalglyargleuaspgluasnargseraspleuvalileleuasp
435440445
thrglnaspileglnserglyprovalalathrilelysleuprophe
450455460
argleuargalaalaleuhisglycystrpvalproargpro
465470475
<210>7
<211>1437
<212>dna
<213>人工序列(isoeugenolmonooxygenase)
<400>7
atggcgagactcaaccgcaacgacccgcaattagtaggaacacttctccccacccgtatc60
gaggcagacttgttcgatctagaggttgacggcgaaatcccaaaatcaataaatggaacg120
ttctaccgtaatacgccagaacctcaagttaccccgcaaaaattccacaccttcatagat180
ggagatggaatggcctctgccttccacttcgaagatggtcatgtcgacttcatcagtcgc240
tgggttaaaaccgctcgattcacggccgaacgactagcgcgaaaatcgctatttggcatg300
tacagaaacccctataccgacgacaccagtgtaaaaggactagaccgcaccgttgccaat360
acaagcatcattagccatcacggcaaggtgctggcggtgaaggaagacggcctaccgtac420
gaactggatcctcgtacacttgaaactcgcggacgcttcgactacgacggccaagttacc480
agccaaacccacaccgcccatccaaaatatgacccggaaacgggtgacttgttgttcttc540
ggttcggcagctaagggcgaagcaactccagacatggcctattacattgtcgacaagcac600
ggcaaggtgacacatgaaacttggtttgagcagccctatggcgcattcatgcacgacttt660
gccattacccgaaattggtccattttcccaattatgccggccaccaacagcctgtcccgc720
ctcaaggcgaaacagccaatttatatgtgggagccggaactgggcagctacattggcgta780
ctgccgcgccgcggccagggcagtcagattcgctggctcaaggcaccggcgctctgggta840
tttcatgttgtgaatgcttgggaagtcggaaccaagatttatatcgaccttatggaaagt900
gaaatcctgccgttccccttccccaactcacaaaaccaacccttcgcccctgagaaagcc960
gtaccacgcctgactcgttgggaaattgacctcgatagcagcagcgacgagatcaagcga1020
acccggctacacgatttctttgcggaaatgccaatcatggattttcgcttcgccctgcaa1080
tgcaaccgctatggctttatgggggtggacgatccacgcaaaccacttgcgcatcagcag1140
gccgagaagatatttgcgtacaactcactcggcatctgggacaaccaccgaggtgactac1200
gacctctggtactccggagaagcctcggcggcccaggagccggccttcgtccctagaagt1260
ccgaccgccgccgaaggtgatgggtacttgctgaccgtggttggtcgtctcgatgaaaat1320
cgcagtgatctggtaattctcgacactcaagacatccagtctggtcccgtggcaaccatc1380
aagctgccattccggttaagggccgctctccatggctgctgggtacccagaccttaa1437
<210>8
<211>1437
<212>dna
<213>人工序列(isoeugenolmonooxygenase)
<400>8
atggcgagactcaaccgcaacgacccgcaattagtaggaacacttctccccacccgtatc60
gaggcagacttgttcgatctagaggttgacggcgaaatcccaaaatcaataaatggaacg120
ttctaccgtaatacgccagaacctcaagttaccccgcaaaaattccacaccttcatagat180
ggagatggaatggcctctgccttccacttcgaagatggtcatgtcgacttcatcagtcgc240
tgggttaggaccgctcgattcacggccgaacgactagcgcgaaaatcgctatttggcatg300
tacagaaacccctataccgacgacaccagtgtaaaaggactagaccgcaccgttgccaat360
acaagcatcattagccatcacggcaaggtgctggcggtgaaggaagacggcctaccgtac420
gaactggatcctcgtacacttgaaactcgcggacgcttcgactacgacggccaagttacc480
agccaaacccacaccgcccatccaaaatatgacccggaaacgggtgacttgttgttcttc540
ggttcggcagctaagggcgaagcaactccagacatggcctattacattgtcgacaagcac600
ggcaaggtgacacatgaaacttggtttgagcagccctatggcgcattcatgcacgacttt660
gccattacccgaaattggtccattttcccaattatgccggccaccaacagcctgtcccgc720
ctcaaggcgaaacagccaatttatatgtgggagccggaactgggcagctacattggcgta780
ctgccgcgccgcggccagggcagtcagattcgctggctcaaggcaccggcgctctgggta840
tttcatgttgtgaatgcttgggaagtcggaaccaagatttatatcgaccttatggaaagt900
gaaatcctgccgttccccttccccaactcacaaaaccaacccttcgcccctgagaaagcc960
gtaccacgcctgactcgttgggaaattgacctcgatagcagcagcgacgagatcaagcga1020
acccggctacacgatttctttgcggaaatgccaatcatggattttcgcttcgccctgcaa1080
tgcaaccgctatggctttatgggggtggacgatccacgcaaaccacttgcgcatcagcag1140
gccgagaagatatttgcgtacaactcactcggcatctgggacaaccaccgaggtgactac1200
gacctctggtactccggagaagcctcggcggcccaggagccggccttcgtccctagaagt1260
ccgaccgccgccgaaggtgatgggtacttgctgaccgtggttggtcgtctcgatgaaaat1320
cgcagtgatctggtaattctcgacactcaagacatccagtctggtcccgtggcaaccatc1380
aagctgccattccggttaagggccgctctccatggctgctgggtacccagaccttaa1437
<210>9
<211>1437
<212>dna
<213>人工序列(isoeugenolmonooxygenase)
<400>9
atggcgagactcaaccgcaacgacccgcaattagtaggaacacttctccccacccgtatc60
gaggcagacttgttcgatctagaggttgacggcgaaatcccaaaatcaataaatggaacg120
ttctaccgtaatacgccagaacctcaagttaccccgcaaaaattccacaccttcatagat180
ggagatggaatggcctctgccttccacttcgaagatggtcatgtcgacttcatcagtcgc240
tgggttaaaaccgctcgattcacggccgaacgactagcgcgaagatcgctatttggcatg300
tacagaaacccctataccgacgacaccagtgtaaaaggactagaccgcaccgttgccaat360
acaagcatcattagccatcacggcaaggtgctggcggtgaaggaagacggcctaccgtac420
gaactggatcctcgtacacttgaaactcgcggacgcttcgactacgacggccaagttacc480
agccaaacccacaccgcccatccaaaatatgacccggaaacgggtgacttgttgttcttc540
ggttcggcagctaagggcgaagcaactccagacatggcctattacattgtcgacaagcac600
ggcaaggtgacacatgaaacttggtttgagcagccctatggcgcattcatgcacgacttt660
gccattacccgaaattggtccattttcccaattatgccggccaccaacagcctgtcccgc720
ctcaaggcgaaacagccaatttatatgtgggagccggaactgggcagctacattggcgta780
ctgccgcgccgcggccagggcagtcagattcgctggctcaaggcaccggcgctctgggta840
tttcatgttgtgaatgcttgggaagtcggaaccaagatttatatcgaccttatggaaagt900
gaaatcctgccgttccccttccccaactcacaaaaccaacccttcgcccctgagaaagcc960
gtaccacgcctgactcgttgggaaattgacctcgatagcagcagcgacgagatcaagcga1020
acccggctacacgatttctttgcggaaatgccaatcatggattttcgcttcgccctgcaa1080
tgcaaccgctatggctttatgggggtggacgatccacgcaaaccacttgcgcatcagcag1140
gccgagaagatatttgcgtacaactcactcggcatctgggacaaccaccgaggtgactac1200
gacctctggtactccggagaagcctcggcggcccaggagccggccttcgtccctagaagt1260
ccgaccgccgccgaaggtgatgggtacttgctgaccgtggttggtcgtctcgatgaaaat1320
cgcagtgatctggtaattctcgacactcaagacatccagtctggtcccgtggcaaccatc1380
aagctgccattccggttaagggccgctctccatggctgctgggtacccagaccttaa1437
<210>10
<211>1437
<212>dna
<213>人工序列(isoeugenolmonooxygenase)
<400>10
atggcgagactcaaccgcaacgacccgcaattagtaggaacacttctccccacccgtatc60
gaggcagacttgttcgatctagaggttgacggcgaaatcccaaaatcaataaatggaacg120
ttctaccgtaatacgccagaacctcaagttaccccgcaaaaattccacaccttcatagat180
ggagatggaatggcctctgccttccacttcgaagatggtcatgtcgacttcatcagtcgc240
tgggttaaaaccgctcgattcacggccgaacgactagcgcgaaaatcgctatttggcatg300
tacagaaacccctataccgacgacaccagtgtaaaaggactagaccgcaccgttgccaat360
acaagcatcattagccatcacggcaaggtgctggcggtgaaggaagacggcctaccgtac420
gaactggatcctcgtacacttgaaactcgcggacgcttcgactacgacggccaagttacc480
agccaaacccacaccgcccatccaaaatatgacccggaaacgggtgacttgttgttcttc540
ggttcggcagctaagggcgaagcaactccagacatggcctattacattgtcgacaagcac600
ggcaaggtgacacatgaaacttggtttgagcagccctatggcgcattcatgcacgacttt660
gccattacccgaaattggtccattttcccaattatgccggccaccaacagcctgtcccgc720
ctcaaggcgaaacagccaatttatatgtgggagccggaactgggcagctacattggcgta780
ctgccgcgccgcggccagggcagtcagattcgctggttcaaggcaccggcgctctgggta840
tttcatgttgtgaatgcttgggaagtcggaaccaagatttatatcgaccttatggaaagt900
gaaatcctgccgttccccttccccaactcacaaaaccaacccttcgcccctgagaaagcc960
gtaccacgcctgactcgttgggaaattgacctcgatagcagcagcgacgagatcaagcga1020
acccggctacacgatttctttgcggaaatgccaatcatggattttcgcttcgccctgcaa1080
tgcaaccgctatggctttatgggggtggacgatccacgcaaaccacttgcgcatcagcag1140
gccgagaagatatttgcgtacaactcactcggcatctgggacaaccaccgaggtgactac1200
gacctctggtactccggagaagcctcggcggcccaggagccggccttcgtccctagaagt1260
ccgaccgccgccgaaggtgatgggtacttgctgaccgtggttggtcgtctcgatgaaaat1320
cgcagtgatctggtaattctcgacactcaagacatccagtctggtcccgtggcaaccatc1380
aagctgccattccggttaagggccgctctccatggctgctgggtacccagaccttaa1437
<210>11
<211>1437
<212>dna
<213>人工序列(isoeugenolmonooxygenase)
<400>11
atggcgagactcaaccgcaacgacccgcaattagtaggaacacttctccccacccgtatc60
gaggcagacttgttcgatctagaggttgacggcgaaatcccaaaatcaataaatggaacg120
ttctaccgtaatacgccagaacctcaagttaccccgcaaaaattccacaccttcatagat180
ggagatggaatggcctctgccttccacttcgaagatggtcatgtcgacttcatcagtcgc240
tgggttaggaccgctcgattcacggccgaacgactagcgcgaagatcgctatttggcatg300
tacagaaacccctataccgacgacaccagtgtaaaaggactagaccgcaccgttgccaat360
acaagcatcattagccatcacggcaaggtgctggcggtgaaggaagacggcctaccgtac420
gaactggatcctcgtacacttgaaactcgcggacgcttcgactacgacggccaagttacc480
agccaaacccacaccgcccatccaaaatatgacccggaaacgggtgacttgttgttcttc540
ggttcggcagctaagggcgaagcaactccagacatggcctattacattgtcgacaagcac600
ggcaaggtgacacatgaaacttggtttgagcagccctatggcgcattcatgcacgacttt660
gccattacccgaaattggtccattttcccaattatgccggccaccaacagcctgtcccgc720
ctcaaggcgaaacagccaatttatatgtgggagccggaactgggcagctacattggcgta780
ctgccgcgccgcggccagggcagtcagattcgctggctcaaggcaccggcgctctgggta840
tttcatgttgtgaatgcttgggaagtcggaaccaagatttatatcgaccttatggaaagt900
gaaatcctgccgttccccttccccaactcacaaaaccaacccttcgcccctgagaaagcc960
gtaccacgcctgactcgttgggaaattgacctcgatagcagcagcgacgagatcaagcga1020
acccggctacacgatttctttgcggaaatgccaatcatggattttcgcttcgccctgcaa1080
tgcaaccgctatggctttatgggggtggacgatccacgcaaaccacttgcgcatcagcag1140
gccgagaagatatttgcgtacaactcactcggcatctgggacaaccaccgaggtgactac1200
gacctctggtactccggagaagcctcggcggcccaggagccggccttcgtccctagaagt1260
ccgaccgccgccgaaggtgatgggtacttgctgaccgtggttggtcgtctcgatgaaaat1320
cgcagtgatctggtaattctcgacactcaagacatccagtctggtcccgtggcaaccatc1380
aagctgccattccggttaagggccgctctccatggctgctgggtacccagaccttaa1437
<210>12
<211>1437
<212>dna
<213>人工序列(isoeugenolmonooxygenase)
<400>12
atggcgagactcaaccgcaacgacccgcaattagtaggaacacttctccccacccgtatc60
gaggcagacttgttcgatctagaggttgacggcgaaatcccaaaatcaataaatggaacg120
ttctaccgtaatacgccagaacctcaagttaccccgcaaaaattccacaccttcatagat180
ggagatggaatggcctctgccttccacttcgaagatggtcatgtcgacttcatcagtcgc240
tgggttaggaccgctcgattcacggccgaacgactagcgcgaagatcgctatttggcatg300
tacagaaacccctataccgacgacaccagtgtaaaaggactagaccgcaccgttgccaat360
acaagcatcattagccatcacggcaaggtgctggcggtgaaggaagacggcctaccgtac420
gaactggatcctcgtacacttgaaactcgcggacgcttcgactacgacggccaagttacc480
agccaaacccacaccgcccatccaaaatatgacccggaaacgggtgacttgttgttcttc540
ggttcggcagctaagggcgaagcaactccagacatggcctattacattgtcgacaagcac600
ggcaaggtgacacatgaaacttggtttgagcagccctatggcgcattcatgcacgacttt660
gccattacccgaaattggtccattttcccaattatgccggccaccaacagcctgtcccgc720
ctcaaggcgaaacagccaatttatatgtgggagccggaactgggcagctacattggcgta780
ctgccgcgccgcggccagggcagtcagattcgctggttcaaggcaccggcgctctgggta840
tttcatgttgtgaatgcttgggaagtcggaaccaagatttatatcgaccttatggaaagt900
gaaatcctgccgttccccttccccaactcacaaaaccaacccttcgcccctgagaaagcc960
gtaccacgcctgactcgttgggaaattgacctcgatagcagcagcgacgagatcaagcga1020
acccggctacacgatttctttgcggaaatgccaatcatggattttcgcttcgccctgcaa1080
tgcaaccgctatggctttatgggggtggacgatccacgcaaaccacttgcgcatcagcag1140
gccgagaagatatttgcgtacaactcactcggcatctgggacaaccaccgaggtgactac1200
gacctctggtactccggagaagcctcggcggcccaggagccggccttcgtccctagaagt1260
ccgaccgccgccgaaggtgatgggtacttgctgaccgtggttggtcgtctcgatgaaaat1320
cgcagtgatctggtaattctcgacactcaagacatccagtctggtcccgtggcaaccatc1380
aagctgccattccggttaagggccgctctccatggctgctgggtacccagaccttaa1437
- 还没有人留言评论。精彩留言会获得点赞!