胰腺癌预后标志物及其应用的制作方法
1.本发明属于生物医药领域,涉及胰腺癌预后标志物及其应用。
背景技术:
2.胰腺癌在中国是一种较为高发且难治的癌症,据2015年的中国癌症数据统计显示,胰腺癌的发病率在癌症领域排名第9,而死亡率排名第6。为了提高胰腺癌的精准诊疗水平以及治愈率,如何对胰腺癌患者的预后状态进行判断就显得尤为重要。
3.目前,针对胰腺癌的预后判断存在很多技术方案,但这些技术方案存在不同的缺陷。首先,一类技术方案是基于单一临床因素对胰腺癌患者的预后进行判断,包括ecog表现状态(eastern cooperative oncology group performance status)、肿瘤标志物ca 19
‑
9水平和年龄等,但是基于单一临床因素进行的预后判断效能不够,易受其他因素影响。第二,一类技术方案是基于多个临床因素计算预后分数,根据分数的高低对患者预后进行判断,但该类方案在针对不同临床因素对应分数的设定上存在较大的主观因素,影响预后判断的准确性。因此,通过对现有技术的分析可以看出,目前对胰腺癌的预后判断技术并不理想。
技术实现要素:
4.技术问题:针对现有对胰腺癌预后判断的技术不佳的问题,本技术提供胰腺癌预后标志物及其应用,从而能够对胰腺癌患者进行准确的预后判断。
5.本技术一方面提供了与胰腺癌预后相关的标志分子,所述标志分子包括ereg、s100a2、sema7a、stat1的至少一个基因。
6.本技术另一方面提供了评价胰腺癌预效果的检测试剂盒,所述试剂盒包括检测前面所述的标志分子的试剂。
7.进一步,用于检测所述标志分子的试剂包括针对所述标志分子的引物对或特异性荧光探针。
8.本发明的引物可以通过化学合成来制备,通过使用本领域技术人员知道的方法参考已知信息来适当地设计,并通过化学合成来制备。
9.本发明的探针可以通过化学合成来制备,通过使用本领域技术人员知道的方法参考已知信息来恰当设计,并通过化学合成来制备,或者可以通过从生物材料制备含有期望核酸序列的基因,并使用设计用于扩增期望核酸序列的引物扩增它来制备。
10.与基因的核酸序列杂交的探针可以是dna、rna、dna
‑
rna嵌合体、pna或其它衍生物。所述探针的长度没有限制,只要完成特异性杂交、与目的核苷酸序列特异性结合,任何长度都可以。所述探针的长度可短至25、20、15、13或10个碱基长度。同样,所述探针的长度可长至60、80、100、150、300个碱基对或更长,甚至整个基因。由于不同的探针长度对杂交效率、信号特异性有不同的影响,所述探针的长度通常至少是14个碱基对,最长一般不超过30个碱基对,与目的核苷酸序列互补的长度以15
‑
25个碱基对最佳。所述探针自身互补序列最
好少于4个碱基对,以免影响杂交效率。
11.本技术又一方面提供了胰腺癌预后效果的评价装置,所述评价装置包括:
12.数据输入模块,用于将与胰腺癌预后相关的标志分子的含量检测结果输入模型计算模块;所述标志分子包括ereg、s100a2、sema7a、stat1中的至少一个基因。
13.进一步,所述模型计算模块用于对输入的含量检测结果进行计算处理,得到被测患者的预后效果数据;所述模型计算模块采用的模型具体为:
14.risk score=0.21738327*ereg基因表达水平+0.01655056*s100a2基因表达水平+0.38376746*sema7a基因表达水平+0.21867157*stat1基因表达水平。
15.进一步,所述评价装置还包括结果输出模块,用于根据胰腺癌预后效果评价标准对被测患者的预后效果数据进行评价,输出评价结果。
16.所述结果输出模块,根据risk score的值的高低进行风险高低评估,据此对被测患者的预后效果数据进行评价。
17.在进行临床实际评估之前,可通过大数据的方式采集大量患者数据,并将大量数据汇总后统计得到的中位数为截断值,将样本分为高、低风险组,据此对被测患者的预后效果数据进行评价。即如果患者的risk score值高于该截断值,则为高风险;如果患者的riskscore值低于该截断值,则为低风险。
18.本技术又一方面提供了前面所述的模型的构建方法,所述构建方法包括以下步骤:
19.(1)候选基因分析:在癌症表达图谱数据库中筛选免疫相关基因;
20.(2)筛选预后相关基因:利用单因素cox分析获得与胰腺癌患者生存相关的免疫相关基因;
21.(3)建立模型:利用lasso cox分析步骤(2)获得的预后相关基因,最终确定组成预后基因标签的基因构建所述模型,所述基因包括ereg、s100a2、sema7a、stat1中的至少一个。
22.进一步,所述构建方法还包括:验证所述模型的效能,验证方法包括:
23.基于所述模型及测试集中胰腺癌患者样本中前面所述的标志分子的表达量,计算每位患者的risk score;
24.基于所述risk score截断值,将测试集分为高风险组和低风险组,并比较两组患者之间的预后差异。
25.本技术又一方面提供了一种应用,所述应用包括以下任一项:
26.1)前面所述的标志分子在制备评价胰腺癌预效果的检测试剂盒中的应用;
27.2)前面所述的标志分子在制备胰腺癌预后效果的评价装置中的应用;
28.3)前面所述的标志分子在制备前面所述的模型中的应用;
29.4)前面所述的标志分子在制备前面所述的计算设备中的应用;
30.5)前面所述的标志分子在制备前面所述的计算机可读存储介质中的应用;
31.6)前面所述的模型在制备胰腺癌预后效果的评价装置中的应用;
32.7)前面所述的模型在制备前面所述的计算设备中的应用;
33.8)前面所述的模型在制备前面所述的计算机可读存储介质中的应用。
34.本技术一方面提供一种胰腺癌预后判定方法,包括
35.检测胰腺癌患者样本中前面所述的标志分子的表达量;
36.根据基因表达量及前面所述的模型,确定胰腺癌患者的截断值;
37.根据所述的截断值确定胰腺癌患者预后情况。
38.进一步,根据所述的截断值确定胰腺癌患者预后情况的方法包括:
39.所述风险分数大于或等于截断值,则判定胰腺癌患者预后情况不好;
40.所述风险分数小于risk score截断值则判定胰腺癌患者预后情况良好。
41.进一步,所述risk score截断值的确定方法包括:
42.基于前面所述的模型及训练集中样本中前面所述的标志分子的表达量,计算每位患者的风险分数;
43.基于每位患者的风险分数,选择一个数值能够将患者分为高风险组和低风险组,当两组患者之间具有最大的预后差异时,所选择的数值确定为risk score截断值。
44.进一步,所述risk score截断值的确定方法包括:
45.在进行临床实际评估之前,可通过大数据的方式采集大量患者数据,并将大量数据汇总后统计得到的中位数为截断值,将样本分为高、低风险组,据此对被测患者的预后效果数据进行评价。即如果患者的risk score值高于该截断值,则为高风险;如果患者的riskscore值低于该截断值,则为低风险。
46.本发明通首先收集基因表达综合数据库(geo)和癌症基因组图谱数据库(tcga)中的胰腺癌公共基因表达数据,根据immport数据库中的免疫相关基因信息确定在tcga及geo数据集中存在的免疫相关基因,对免疫相关基因进行单因素cox回归分析,根据单因素p值选择相关基因进行lasso回归分析。根据lasso回归结果,选择基因构建生存相关的线性风险评估模型,计算各个样本的风险值(risk score),取risk score的中位数为截断值,将样本分为高、低风险组。采用时间依赖的roc曲线评估模型1、3、5年的生存期的预测能力,同时分析高、低组的生存曲线。
47.本技术又一方面提供了一种计算设备,包括存储器和处理器,所述存储器存储有程序,所述处理器执行所述程序时实现前面所述的模型或前面所述的胰腺癌预后判断方法。
48.本技术又一方面提供了一种计算机可读存储介质,其上存储有程序,所述程序被处理器执行时实现前面所述的模型或前面所述的胰腺癌预后判断方法。
49.本发明的计算设备可以是,但不限于任何一种可与用户通过键盘、触摸板或声控设备等方式进行人机交互的个人电脑、服务器等终端。本文中的计算设备还可以包括移动终端,其可以是,但不限于任何一种可与用户通过键盘、触摸板或声控设备等方式进行人机交互的电子设备,例如,平板电脑、智能手机、个人数字助理(personal digital assistant,pda)、智能式穿戴式设备等终端。计算设备所处的网络包括,但不限于互联网、广域网、城域网、局域网、虚拟专用网络(virtual private network,vpn)等。
50.本发明的存储器包括非易失性和/或易失性存储器。非易失性存储器可包括只读存储器(rom)、可编程rom(prom)、电可编程rom(eprom)、电可擦除可编程rom(eeprom)或闪存。其上存储有操作系统的代码。例如,存储器上还存储有代码或指令,通过运行这些代码或指令,可以实现本公开实施例提供的胰腺癌预后模型。易失性存储器可包括随机存取存储器(ram)或者外部高速缓冲存储器。作为说明而非局限,ram以多种形式可得,诸如静态
ram(sram)、动态ram(dram)、同步dram(sdram)、双数据率sdram(ddrsdram)、增强型sdram(esdram)、同步链路(synchlink)dram(sldram)、存储器总线(rambus)直接ram(rdram)、直接存储器总线动态ram(drdram)、以及存储器总线动态ram(rdram)等。
51.进一步,本发明的计算设备可以包括通过系统总线连接的处理器、存储器、外界接口、显示器和输入装置。其中,处理器用于提供计算和控制能力。该计算设备的显示器可以是液晶显示屏或者电子墨水显示屏,输入装置可以是显示屏上覆盖的触摸层,也可以是例如计算设备外壳上设置的按键、轨迹球或触控板,还可以是外接的键盘、触控板或鼠标等。
52.所述处理器可以包括一个或者多个微处理器、数字处理器。所述处理器可调用存储器中存储的程序代码以执行相关的功能。所述处理器又称中央处理器(cpu,central processing unit),可以是一块超大规模的集成电路,是运算核心(core)和控制核心(control unit)。
53.计算机可读存储介质存储的程序在被处理器执行时可以实现前面所述的模型或前面所述的胰腺癌预后判断方法。另外,计算机可读存储介质也可以以单独的物理形式存在,例如一u盘,当其与一处理器连接时,u盘上存储的程序被执行可以实现前面所述的模型或前面所述的胰腺癌预后判断方法。本发明的方法,也可以实现为苹果或安卓应用市场中的一个app(应用程序),供用户下载到各自的移动终端运行。
54.如上所述,本领域普通技术人员可以理解实现上述判断方法中的全部或部分流程,是可以通过计算机程序来指令相关的硬件来完成,所述的计算机程序可存储于计算机可读存储介质中。
附图说明
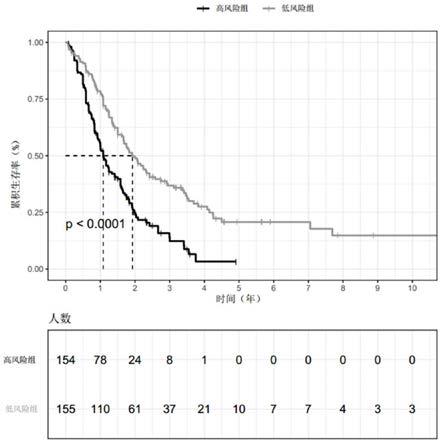
55.图1显示geo数据集的生存曲线图;
56.图2显示tcga的生存曲线图;
57.图3显示geo数据集的roc曲线图;
58.图4显示tcga的roc曲线图。
具体实施方式
59.以下实施例用于说明本发明,但不用来限制本发明的范围。
60.实施例1胰腺癌预后评分模型构建
61.1、数据下载
62.在基因表达综合数据库(geo)和癌症基因组图谱数据库(tcga)中搜索公共基因表达数据和完整的临床注释。没有生存信息的患者从进一步的评估中删除。本研究共收集了5个合格的pdac队列(gse28735、gse62452、gse71729、gse85916和tcga
‑
paad)进行进一步分析。对于的微阵列数据,我们下载了原始的“cel”文件,在affy软件包中使用rma算法进行背景调整和分位数归一化。对于其他平台的微阵列数据,直接下载归一化矩阵文件。对于tcga中的数据集,基因表达的rna测序数据(fpkm值)和临床信息从ucsc xena(https://gdc.xenahubs.net)下载。然后将fpkm值转化为每千碱基百万(tpm)值的转录本。采用sva软件包的“combat”算法对非生物技术偏差造成的批量效应进行了修正。所有合格的pdac数据集的信息汇总在表1中,其中geo数据集作为训练集,tcga数据集作为验证集。
63.表1本研究中数据集的基本信息
[0064][0065]
2、免疫相关基因
[0066]
免疫相关基因来自immport数据库(https://www.immport.org/home)。我们在immport数据库共纳入了1793个免疫相关基因。其中,有1116个免疫相关基因在tcga及geo数据集中存在。
[0067]
3、单因素cox分析
[0068]
对1116个免疫相关基因进行单因素cox分析,p<0.01的基因被认为是对胰腺癌患者的生存有影响。在此标准下,tcga数据库中有156个基因,geo数据库中有100个基因。两者进行取交集处理后,共26个基因。
[0069]
4、lasso cox分析
[0070]
在geo数据集中对26个基因进行lasso cox分析,筛选出基因组成预后gene signature。根据公式计算每个样本的风险评分,根据风险评分的中位数,将所有样本分为高风险组与低风险组。
[0071][0072]
注:风险评分的计算公式,n为预后基因数,expi为基因i的表达值,βi为基因i的回归系数。
[0073]
最终筛选确定的用于构建风险评分模型的基因包括以下四个基因:ereg、s100a2、sema7a、stat1。表2列出了用于构建风险评分模型的4个基因的相关信息和参数。单因素cox回归分析中的hr用于表征相对危险度,其中,hr值大于1表示对应的基因的表达值与风险评分呈正相关关系,从而相应的lasso系数大于0,hr值小于1表示对应的基因的表达值与风险评分呈负相关关系,从而相应的lasso系数小于0。另外,在表2中,95%ci表示95%的置信区间(confidence interval)。
[0074]
表2风险评分模型中的4个基因
[0075][0076]
根据表2的结果可知,4个基因对应的风险评分模型表示为:
[0077]
risk score(风险评分)=
‑
0.21738327*ereg基因表达水平+0.01655056*s100a2基因表达水平+0.38376746*sema7a基因表达水平+0.21867157*stat1基因表达水平
[0078]
注:风险评分的计算公式,n为预后基因数,expi为基因i的表达值,βi为基因i的回归系数
[0079]
生存分析结果表明,高风险评分组患者的生存时间明显短于低风险评分组(图1)。为了评估由上述基因组成的预后模型在预测胰腺癌预后的准确性,我们进行了1年、3年和5年受试者工作特征(roc)曲线分析,比较各自的auc值。结果表明,1年、3年和5年的auc分别是0.65、0.74、0.82(图3)。auc值表明,由上述基因组成的预后模型对胰腺癌患者的预后具有较好的区分性能。其次,我们在tcga验证集中利用相同的公式,计算了每个样本的风险评分,并进行了生存分析及受试者工作特征(roc)曲线分析,结果与训练集表现出相同的趋势(图2和图4)。这些结果表明,基于4个风险特征计算的风险评分可以较好的预测胰腺癌患者的预后。
[0080]
虽然,上文中已经用一般性说明、具体实施方式及试验,对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1