一种基因在促进骨形成中的应用
1.本发明属于基因工程技术领域,具体涉及一种基因在促进骨形成中的应用。
背景技术:
2.骨质疏松症是一种全身性的骨代谢疾病,表现为骨密度降低和骨质量下降,可加剧骨折的风险,目前预防和治疗骨质疏松的药物往往效果较差且副作用大。人类的成熟骨骼通过成骨细胞引起骨形成,并且成骨细胞会经历如增殖、基质形成、基质矿化等一系列分化阶段,而相关标志基因的上调或下调,会显著影响成骨细胞的增殖速率,减少骨形成,最终导致骨质疏松症的发生。
3.因此,如果能研究出影响成骨细胞增殖的相关因素,在骨质疏松药物制备上获得特异性强、副作用弱的新型药物,对骨质疏松疾病患者会是极大的帮助。
4.因此,如何提供一种基因在促进骨形成中的应用是本领域亟待解决的问题。
技术实现要素:
5.本发明公开了一种基因在促进骨形成中的应用。
6.本发明的目的是研究etv4基因在表达下调的情况下,对成骨细胞增殖的影响,进而研究etv4基因在骨质疏松中所起的作用,为临床上治疗骨质疏松疾病提供可靠的理论依据和实验数据。
7.为了实现上述目的,本发明采用如下技术方案:
8.一种基因在骨形成中的应用,所述基因cds序列如seq id no:1所示;
9.优选的,所述基因为鼠源etv4基因;
10.优选的,所述骨形成中的应用包括促进骨形成中的应用和抑制骨形成中的应用;
11.优选的,所述促进骨形成中的应用和抑制骨形成中的应用包括促进骨形成中的应用为制备促进骨形成制剂中的应用和抑制骨形成中的应用为制备促进骨形成制剂中的应用;
12.优选的,其特征在于,所述抑制骨形成中的应用通sirna技术实现,sirna序列为:
13.etv4-sirna-2:
[0014]5’ꢀ
ggaccucagucacuuccaadtdt 3’,seq id no:3;
[0015]3’
dtdtccuggagucagugaagguu 5’,seq id no:4;
[0016]
或
[0017]
etv4-sirna-3:
[0018]5’ꢀ
gcucgcugcgauacuauuadtdt 3’,seq id no:5;
[0019]3’
dtdtcgagcgacgcuaugauaau 5’,seq id no:6;
[0020]
中的一种或多种;
[0021]
优选的,骨细胞为mc3t3-e1细胞;
[0022]
优选的,所述基因可增强成骨细胞增殖能力;
[0023]
seq id no:1:
[0024]
atggag cggaggatga aaggcggata cttggaccag cgagtgccct acaccttctg cagcaaatct cccggaaatg ggagcttggg cgaagcgctg atggtcccgc agggaaagct catggacccg ggctccctgc cgccttccga ctcagaagat ctcttccagg acctcagtca cttccaagag acgtggctcg cagaagctca ggtaccggac agtgatgagc agtttgttcc tgatttccat tcagaaaact tagctttcca tagccccacc accaggatca agaaggaacc ccagagtccc cgcacagacc ccgccctgtc ctgcagcagg aagccaccac tcccctacca ccatggagag cagtgccttt actccagtgc ctatgactcc cccagacaaa tcgccatcaa gtcccccgcc cccggtgccc ctggacagtc gcccctgcag cccttttcca gggcagaaca gcagcagagc ctcctgagag cctccagctc ttcccagtcc caccctggcc acgggtacct tggtgagcac agctccgtct tccagcagcc cgtggacatg tgccactcct tcacatctcc tcagggaggg ggccgggaac ctctcccagc cccctatcaa caccaactgt cggagccctg cccaccctac ccccagcaga acttcaagca ggagtaccat gaccccctgt acgaacaggc tggccagccc gcttcaagcc agggtggggt cagtgggcac aggtacccag gggcgggggt ggtgatcaaa caggagcgca cagacttcgc ctacgactca gatgtccctg gatgtgcatc aatgtacctc cacccagagg gcttctctgg accctctcca ggtgatggag tgatgggtta tggctatgaa aaatcccttc gaccattccc agatgatgtc tgcattgtcc ctgaaaaatt tgaaggagac atcaagcagg aagggattgg agctttccgg gaggggccac cctaccagcg ccggggtgcc ttacaactgt ggcagtttct ggtggccctg ctggatgacc caacaaatgc tcatttcatt gcttggacag gccggggaat ggagtttaaa ctaattgaac ctgaagaggt tgccaggctc tggggtatcc agaagaaccg gccagccatg aattatgaca agctgagccg ctcgctgcga tactattatg agaaaggcat catgcagaag gtggctggcg aacgctacgt gtacaagttt gtgtgcgagc cggaggccct gttctctctg gccttcccag ataatcaacg tccagctctg aaggctgagt ttgaccggcc agtcagtgag gaggacacag tccctttgtc ccacttggat gagagtcctg cctacctccc agaactcact ggccccgctc cgcccttcgg ccacagaggt ggatattctt actag。
[0025]
cds对应的mrna序列如seq id no:2所示,具体如下:
[0026]
1 ggaggccaag gcaaaggaaa tgcaccaatc agctgctccc ccgggctcac aactgtctgc
[0027]
61 tgcgcccgga aaacaagccg gggctctggg gacccggggc tcaggccgcc tcgctccggc
[0028]
121 ctagccccgc caccttagtt gtgtcatccc ccgggcatgc tgagcatccc cccgcggctc
[0029]
181 cggcacagac gcccggacct caggtctctg cctccgcgcg ggggcccggc cctgtggccg
[0030]
241 gagggagcgg ccggatggag cggaggatga aaggcggata cttggaccag cgagtgccct
[0031]
301 acaccttctg cagcaaatct cccggaaatg ggagcttggg cgaagcgctg atggtcccgc
[0032]
361 agggaaagct catggacccg ggctccctgc cgccttccga ctcagaagat ctcttccagg
[0033]
421 acctcagtca cttccaagag acgtggctcg cagaagctca ggtaccggac agtgatgagc
[0034]
481 agtttgttcc tgatttccat tcagaaaact tagctttcca tagccccacc accaggatca
[0035]
541 agaaggaacc ccagagtccc cgcacagacc ccgccctgtc ctgcagcagg aagccaccac
[0036]
601 tcccctacca ccatggagag cagtgccttt actccagtgc ctatgactcc cccagacaaa
[0037]
661 tcgccatcaa gtcccccgcc cccggtgccc ctggacagtc gcccctgcag cccttttcca
[0038]
721 gggcagaaca gcagcagagc ctcctgagag cctccagctc ttcccagtcc caccctggcc
[0039]
781 acgggtacct tggtgagcac agctccgtct tccagcagcc cgtggacatg tgccactcct
[0040]
841 tcacatctcc tcagggaggg ggccgggaac ctctcccagc cccctatcaa caccaactgt
gccctgaaca
[0062]
2161 ggggggtcct gtgttcttgg tgctgtgctc tgggaggcag gagcatgtgg gcggcagctg
[0063]
2221 gggggggggg ttgggtggaa gagtagggat ggctctctgc cctaggccta cccaggccta
[0064]
2281 attccacctt tgcctcttat gccagacctt aataaagcct ctgcttctcc ggtaaaaaaa
[0065]
2341 aaaaaaaaaa aa。
[0066]
综上所述,本发明公开了一种基因在促进骨形成中的应用,通过对mc3t3-e1的etv4基因进行抑制发现,etv4基因能够促进了骨细胞的增殖和分化,促进骨的形成,为临床上治疗骨质疏松等疾病提供可靠的理论依据和实验数据。
附图说明
[0067]
图1为qrt-pcr检测sirna对etv4基因在成骨细胞系的敲低效果;
[0068]
图2为cck-8细胞增殖检测etv4基因敲低后成骨细胞的增殖活性;
[0069]
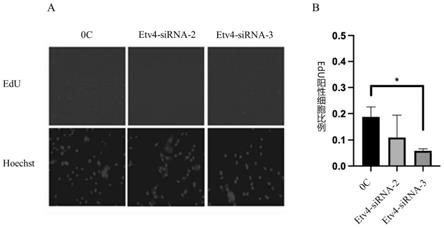
图3为edu标记法检测etv4敲低后成骨细胞相对细胞数量的标记图;
[0070]
图4为人源etv4蛋白和鼠源etv4蛋白氨基酸序列保守性分析。
具体实施方式
[0071]
下面对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
[0072]
实施例1
[0073]
生物信息学分析
[0074]
通过生物信息学分析发现etv4在人和小鼠间很保守,如图4所示蛋白的一致性高达92%。
[0075]
成骨细胞(mc3t3-e1)的培养
[0076]
配置含10%fbs(胎牛血清)、1%p/s(双抗)的mem-α培养基,水浴锅预热20min,按照对照组和实验组分别均匀种于10cm培养皿中,摇匀,放入37℃,5%的培养箱中进行培养。24h首次换液,pbs清洗一次,加入新鲜培养液继续培养,以后每2-3天换液一次,定期观察细胞状态。
[0077]
细胞转染
[0078]
(1)弃培养皿内旧培养基,pbs清洗细胞后加入胰蛋白酶,待细胞变圆飘起后终止消化,转移细胞消化液至15ml离心管中,室温下1000rpm,离心5min,可明显观察到离心管底部的细胞沉淀,使用真空泵吸取上清,加入6ml培养基重悬细胞,吸取20μl细胞悬液滴入细胞计数板,通过自动细胞计数仪检测细胞悬液的浓度(选取三个不同的视野进行计数,最终取平均数),按照预期细胞种植数量稀释细胞悬液,接种于培养皿中,预计第二天转染时细胞密度达到40%-50%,细胞培养板置于细胞培养箱中。
[0079]
(2)根据转染试剂lipofectamine
tm
3000 reagent的使用说明书,不同规格的培养皿使用不同剂量的lipofectamine
tm
3000 reagent、sirna及opti-mem
tm
medium,计划好所需试剂的量。首先取两个1.5ml离心管加入等量opti-mem
tm
medium,取其中一个离心管加入适量lipofectamine
tm
3000 reagent,吹匀后于室温下静置5min;另外一个离心管加入sirna(具体sirna oligos信息如表1),移液器吹匀,再将含转染试剂的opti-mem
tm
medium溶液加入到含sirna或质粒的opti-mem
tm
medium溶液中,吹匀混合液于室温下静置5min。
[0080]
表1 sirna oligo序列
[0081][0082]
rna提取
[0083]
(1)取细胞:取转染48h后的细胞,真空泵抽取培养皿中的旧培养基,使用pbs洗涤细胞后弃液,重复两次。
[0084]
(2)裂解细胞:加入适量buffer rlt plus溶液覆盖在培养皿底面,涡旋震荡15s。
[0085]
(3)去除基因组dna:将gdna eliminator spin column放置在新的2ml收集管中,步骤(2)产生的细胞裂解液转移到gdna eliminator spin column中,8000rpm离心1min,丢弃gdna eliminator spin column,保留收集管中的滤液。
[0086]
(4)向滤液中加入350μl 70%的酒精,移液器吹打均匀混合液,加入700μl混合液至rneasy spin column,8000rpm离心15s,弃液,保留rneasy spin column。若混合液体积大于700μl,可分多次转移至同一个rneasy spin column中,重复上述操作。
[0087]
(5)向rneasy spin column中加入700μl buffer rw1溶液,8000rpm离心15s,丢弃过滤液,保留rneasy spin column。
[0088]
(6)向rneasy spin column中加入500μl buffer rpe溶液,8000rpm离心15s,丢弃过滤液,保留rneasy spin column。
[0089]
(7)向rneasy spin column中加入500μl buffer rpe溶液,8000rpm离心2min,丢弃过滤液,保留rneasy spin column。
[0090]
(8)为rneasy spin column更换2ml收集管,12 000rpm离心1min。
[0091]
(9)将rneasy spin column放入未使用的1.5ml离心管中,向rneasy spin column中加入50μl去离子水洗脱rna,先在室温下孵育1min使rneasy spin column过滤膜中的rna充分溶于去离子水,全速(14 000rpm)离心1min,丢弃rneasy spin column,保留过滤液。
[0092]
(10)rna浓度测定:使用nanodrop 2000进行rna浓度测定。首先打开nanodrop 2000软件,选择浓度检测模式(核酸—》rna)。抬起样品臂使用去离子水清洗nanodrop 2000检测基座,加入1μl去离子水放下样品臂进行空白定量,然后加入1μl待测样品测量吸光度,每个样品检测结束后用干净的无尘纸把检测基座擦拭干净,每次操作相同。观察图像面板显示的样品光谱,记录下相应的样品浓度及吸光度。
[0093]
反转录
[0094]
rna反转录成cdna借助于high-capacity cdna reverse transcription kit with rnase inhibitor试剂盒,反转录反应体系如表2(反应体系的配制全程冰上操作),反转录体系配制完成后于pcr仪上设置程序(如表3),完成反转录。
[0095]
表2反转录反应体系
[0096][0097]
表3反转录程序
[0098][0099]
实时荧光定量pcr
[0100]
实时荧光定量pcr是通过荧光信号实时检测pcr扩增过程。pcr指数扩增时期,荧光信号ct值与目的基因的起始量存在线性关系,故使用ct值对目的基因进行定量。设计目的基因的引物(如表4),配制实时荧光定量pcr的反应体系如表5(反应体系配制全程冰上操作),于biorad cfx96 pcr仪上运行相应的反应程序(如表6)。
[0101]
表4 qrt-pcr引物序列
[0102][0103][0104]
表5 qrt-pcr反应体系
[0105]
[0106]
表6 qrt-pcr程序
[0107][0108]
qrt-pcr结果显示,靶向etv4的三个sirna中,etv4-sirna-2和etv4-sirna-3在成骨细胞中mrna水平显著降低,确定sirna敲低成功,因此接下来的验证实验选用etv4-sirna-2和etv4-sirna-3。
[0109]
细胞增殖检测
[0110]
(1)cck-8检测细胞增殖
[0111]
a)接种细胞:通过细胞计数将细胞接种于96孔板内(根据不同细胞类型决定接种细胞数量),每个样品重复6个孔,每孔终体积100μl,“8”字晃动细胞培养皿保证细胞接种均匀,96孔板置于细胞培养箱中,接种24小时后进行转染。
[0112]
b)cck-8孵育:转染3天后,取96孔板细胞,每孔加入10μl cck-8(操作应避光,cck-8应加入细胞培养基液面以下),“8”字摇匀,细胞培养箱内避光孵育1-4h(肉眼可观察到溶液颜色加深),使用酶标仪读取450nm波长处的吸光度。
[0113]
cck-8结果表明,和对照组相比,两组实验组etv4-sirna-2和etv4-sirna-3的成骨细胞增殖能力均受到明显抑制(p《0.01),差异具有统计学意义。
[0114]
(2)edu标记法检测细胞增殖
[0115]
a)接种细胞:通过细胞计数将细胞接种于96孔板内(根据不同细胞类型决定接种细胞数量),每个样品重复6个孔,每孔终体积100μl,“8”字晃动细胞培养皿保证细胞接种均匀,将96孔板放置在细胞培养箱中,接种24小时后进行转染,转染3天后进行edu标记实验。
[0116]
b)edu标记细胞:用细胞培养基配制一份2
×
edu工作液,96孔板每孔加入100μl 2
×
edu工作液,终体积200μl,edu工作液终浓度为1
×
,在细胞培养箱孵育合适时间(mc3t3-e1孵育4h)。此处使用的为绿色荧光标记。
[0117]
c)细胞固定及促渗:
[0118]
edu工作液孵育完成后,吸取旧培养基,每孔加入50μl 4%的多聚甲醛,室温下固定15min后,弃液。每孔加入50μl 2mg/ml甘氨酸溶液中和多聚甲醛,室温下孵育20min后,弃液;3%bsa in pbs清洗两次,弃液;加入0.5%tritonx-100 in pbs促渗,室温下孵育20min,弃液;3%bsa in pbs清洗两次,弃液。
[0119]
d)edu检测
[0120]
每孔加入100μl click-it反应混合物,放置摇床上,室温下避光孵育30min,弃液;3%bsa in pbs清洗两次,弃液。
[0121]
e)dna复染
[0122]
每孔加入100μl pbs清洗细胞,弃液;加入100μl 5μg/ml的hoechst溶液,室温下避光孵育20min;再加入pbs清洗两次,弃液。
[0123]
f)成像及分析
[0124]
使用荧光显微镜,在紫外光下观察hoechst标记,在蓝色荧光下观察edu绿色标记,在同一焦平面下用紫外光和蓝色荧光观察同一视野,使用imagej处理图片,计算出同一视野下edu/hoechst的比值。
[0125]
edu检测结果表明,和对照组相比,两组实验组etv4-sirna-2和etv4-sirna-3的edu标记的细胞核数量显著减少,进一步证实了成骨细胞的增殖受到明显抑制,差异具有统计学意义。
[0126]
上述实验均进行了3次独立实验,展示结果基于3次代表性独立实验,结果以均值与标准差表示,两组间均数比较用t检验,多组间的比较用单因素方差分析(anova),以p<0.05为有统计学差异。
[0127]
etv4(ets variant 4)是属于ets家族中新发现的一种转录因子,与细胞的增殖和转移密切相关。本发明通过sirna技术在成骨细胞中特异性敲低etv4基因,观察etv4基因对成骨细胞增殖的影响,结果显示,etv4敲低后,成骨细胞的增殖相较对照组明显受到抑制,且具有统计学意义。
[0128]
通过cck-8和edu两种细胞增殖检测方法,验证etv4基因对成骨细胞增殖能力的影响。cck-8和edu结果显示,etv4基因敲低后,成骨细胞的增殖受到明显抑制,且具有统计学意义,以上结果提示,etv4基因在成骨细胞的增殖过程中发挥了促进作用。
[0129]
本说明书中各个实施例采用递进的方式描述,每个实施例重点说明的都是与其他实施例的不同之处,各个实施例之间相同相似部分互相参见即可。
[0130]
对所公开的实施例的上述说明,使本领域专业技术人员能够实现或使用本发明。对上述实施例的多种修改对本领域的专业技术人员来说将是显而易见的,本文中所定义的一般原理可以在不脱离本发明的精神或范围的情况下,在其它实施例中实现。因此,本发明将不会被限制于本文所示的这些实施例,而是要符合与本文所公开的原理和新颖特点相一致的最宽的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1