基于T细胞受体信号传导基因对前列腺癌对象的放疗反应的预测的制作方法
基于t细胞受体信号传导基因对前列腺癌对象的放疗反应的预测
技术领域
1.本发明涉及预测前列腺癌对象对放疗的反应的方法。此外,本发明涉及诊断试剂盒、该试剂盒在预测前列腺癌对象对放疗的反应的方法中的用途、针对八种或更多t细胞受体信号传导基因中的每种t细胞受体信号传导基因的基因表达谱在对前列腺癌对象的放疗预测中的用途,以及对应的计算机程序产品。
背景技术:
2.癌症是这样一类疾病:其中一组细胞表现出不受控制的生长、侵袭并且有时还表现出不受控制的转移。癌症的这三种恶性特性将它们与良性肿瘤区分开来,良性肿瘤具有自限性且不会发生侵袭或转移。前列腺癌(pca)是男性的第二常见的非皮肤恶性肿瘤,2018年全球新诊断出的病例估计有130万,并且有360000人死亡(参见bray f.等人的“global cancer statistics 2018:globocan estimates of incidence and mortality worldwide for 36cancers in 185countries”(ca cancer j clin,第68卷,第6期,第394-424页,2018年))。在美国,大约90%的新病例涉及局限性癌症,这意味着尚未形成转移(参见acs(美国癌症协会)“cancer facts&figures 2010”,2010年)。
3.对于原发性局限性前列腺癌的处置,有几种根治性治疗可用,其中,最常用的是手术(根治性前列腺切除术,rp)和辐射治疗(rt)。rt经由外部射束或经由将放射性种子植入前列腺(近距离放疗)或这两者的组合来进行。对于不适合手术或已被诊断患有晚期局限性或区域性肿瘤的患者来说尤其优选。在美国,多达50%的被诊断患有局限性前列腺癌的患者接受了根治性rt(参见acs,2010年,出处同上)。
4.在处置后,测量血液中的前列腺癌抗原(psa)水平以进行疾病监测。血液psa水平的增大提供了针对癌症复发或进展的生化替代指标。然而,报告的无生化进展生存率(bpfs)的差异很大(参见grimm p.等人的“comparative analysis of prostate-specific antigen free survival outcomes for patients with low,intermediate and high risk prostate cancer treatment by radical therapy.results from the prostate cancer results study group”(bju int,增刊1,第22-29页,2012年))。对于许多患者,在根治性rt后有5年或甚至10年的bpfs可以高于90%。遗憾的是,对于处于中等复发风险并且特别是处于较高复发风险的患者组,5年的bpfs能够下降到40%左右,这取决于所使用的rt的类型(参见grimm p.等人,2012年,出处同上)。
5.大量未经rt处置的原发性局限性前列腺癌患者将经受rp(参见acs,2010年,出处同上)。在rp后,在最高风险组中平均有60%的患者在5年和10年后经历生化复发(参见grimm p.等人,2012年,出处同上)。在rp后发生生化进展的情况下,主要挑战之一是不确定这是由于复发的局限性疾病,还是由于一个或多个转移,甚至是由于不会导致临床疾病进展的惰性疾病(参见dal pra a.等人的“contemporary role of postoperative radiotherapy for prostate cancer”(transl androl urol,第7卷,第3期,第399-413页,
2018年)和herrera f.g.和berthold d.r.的“radiation therapy after radical prostatectomy:implications for clinicians”(front oncol,第6卷,第117期,2016年))。rt根除前列腺床中的剩余的癌细胞是在rp后psa增大之后挽救生存率的主要处置选项之一。挽救性放疗(srt)的有效性使18%至90%的患者达到了5年的bpfs,具体取决于多种因素(参见herrera f.g.和berthold d.r.,2016年,出处同上,以及pisansky t.m.等人“salvage radiation therapy dose response for biochemical failure of prostate cancer after prostatectomy
–
a multi-institutional observational study”(int j radiat oncol biol phys,第96卷,第5期,第1046-1053页,2016年))。
6.很明显,对于某些患者组,根治性rt或挽救性rt是无效的。rt能够导致的严重副作用(例如,肠道炎症和功能障碍、尿失禁和勃起功能障碍)甚至使他们的情况更加恶化(参见resnick m.j.等人的“long-term functional outcomes after treatment for localized prostate cancer”(n engl j med,第368卷,第5期,第436-445页,2013年)和hegarty s.e.等人的“radiation therapy after radical prostatectomy for prostate cancer:evaluation of complications and influence of radiation timing on outcomes in a large,population-based cohort”(plos one,第10卷,第2期,2015年))。另外,基于医疗保险报销的一个rt疗程的成本的中位值为18000美元,该成本的差异很大,最高达到约40000美元(参见paravati a.j.等人的“variation in the cost of radiation therapy among medicare patients with cancer”(j oncol pract,第11卷,第5期,第403-409页,2015年))。这些数字不包括在根治性rt和挽救性rt后的后续护理的可观的纵向成本。
7.对每个患者的rt有效性的改进预测(无论是在根治性背景中还是在挽救性背景中)都将改进治疗选择和提高潜在生存率。这能够通过以下操作来实现:1)为那些预测rt有效的患者优化rt(例如通过剂量递增或不同的开始时间),以及2)将预测rt无效的患者引导到可能更有效的处置形式的替代方案。另外,这将减少那些免于无效治疗的患者的痛苦并减少在无效治疗上花费的成本。
8.对根治性rt的反应预测指标已经进行了大量的研究(参见hall w.a.等人的“biomarkers of outcome in patients with localized prostate cancer treated with radiotherapy”(semin radiat oncol,第27卷,第11-20页,2016年)和raymond e.等人的“an appraisal of analytical tools used in predicting clinical outcomes following radiation therapy treatment of men with prostate cancer:a systematic review”(radiat oncol,第12卷,第1期,第56页,2017年)和srt(参见herrera f.g.和berthold d.r.,2016年,出处同上))。这些指标中的许多指标取决于基于血液的生物标志物psa的浓度。为在rt(根治性以及挽救性)开始前预测反应而研究的度量包括psa浓度的绝对值、psa相对于前列腺体积的绝对值、一定时间内的绝对增大量和倍增时间。其他经常考虑的因素是gleason评分和临床肿瘤分期。对于srt背景,其他因素是相关的,例如,手术切缘状态、rp后复发时间、手术前/围手术期psa值和临床病理参数。
9.虽然这些临床变量在各个风险组中的患者分层方面提供了有限的改进,但是仍然需要更好的预测工具。
10.已经研究了在组织和体液中种类众多的候选生物标志物,但是验证通常很有限并
且通常证明预后信息并且不是预测(治疗特异性)值(参见hall w.a.等人,2016年,出处同上)。当前,商业组织正在验证少数基因表达板。这些基因表达板中的一种或多种基因表达板可以示出对未来的rt的预测值(参见dal pra a.等人,2018年,出处同上)。
11.总之,对于原发性前列腺癌以及术后环境,仍然强烈需要对rt的反应的更好预测。
12.wo2019/028285a2公开了用于对象中的前列腺癌的诊断、预后和癌症进展确定的方法、系统和试剂盒。特别地,本公开涉及免疫细胞特异性基因表达在确定预后和识别将对辐射治疗有反应的需要处置前列腺癌的个体中的用途。
13.wo2017/216559a1公开了一种预测患有前列腺癌的对象对有丝分裂抑制剂和/或dna损伤治疗剂的反应性的方法,其包括测量来自对象的样本中的多种给定基因中的至少一种基因的表达水平。
技术实现要素:
14.本发明的目的是提供预测前列腺癌对象对放疗的反应的方法,该方法允许做出更好的处置决定。本发明的另外的目的是提供诊断试剂盒、该试剂盒在预测前列腺癌对象对放疗的反应的方法中的用途、针对八个或更多个t细胞受体信号传导基因中的每种t细胞受体信号传导基因的基因表达谱在对前列腺癌对象的放疗预测中的用途,以及对应的计算机程序产品。
15.在本发明的第一方面,提出了一种预测前列腺癌对象对放疗的反应的方法,包括:
[0016]-确定或接收对针对从包括以下各项的组中选择的八个或更多个,例如,8、9、10、11、12、13、14、15、16个或所有,t细胞受体信号传导基因中的每个t细胞受体信号传导基因的基因表达谱的确定的结果:cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和zap70,所述基因表达谱是在从所述对象获得的生物样本中确定的,
[0017]-优选地由处理器基于针对所述八个或更多个t细胞受体信号传导基因的所述基因表达谱来确定对放疗反应的预测;并且
[0018]-任选地,将所述预测或基于所述预测的治疗推荐提供给医护人员或所述对象。
[0019]
近年来,免疫系统在癌症抑制以及癌症起始、促进和转移中的重要作用已经变得非常明显(参见mantovani a.等人的“cancer-related inflammation”(nature,第454卷,第7203期,第436-444页,2008年)以及giraldo n.a.等人的“the clinical role of the tme in solid cancer”(br j cancer,第120卷,第1期,第45-53页,2019年))。免疫细胞及其分泌的分子构成了肿瘤微环境的重要组成部分,并且大多数免疫细胞能够浸润肿瘤组织。免疫系统和肿瘤相互影响和塑造。因此,抗肿瘤免疫能够防止肿瘤形成,而炎症性肿瘤环境可以促进癌症的起始和增殖。同时,可能以独立于免疫系统的方式起源的肿瘤细胞将通过募集免疫细胞来塑造免疫微环境,并且能够具有促炎作用,同时也抑制抗癌免疫。
[0020]
肿瘤微环境中的一些免疫细胞要么具有一般的肿瘤促进作用,要么具有一般的肿瘤抑制作用,而另一些免疫细胞则表现出可塑性,并且同时显示出肿瘤促进潜力和肿瘤抑制潜力。因此,肿瘤的整体免疫微环境是以下这些项目的混合物:存在的各种免疫细胞、这些免疫细胞产生的细胞因子,以及这些免疫细胞与肿瘤微环境中的肿瘤细胞和其他细胞的相互作用(参见giraldo n.a.等人,2019年,出处同上)。
[0021]
上述关于免疫系统在癌症中的作用原理通常也适用于前列腺癌。慢性炎症与良性和恶性前列腺组织的形成有关(参见hall w.a.等人,2016年,出处同上),并且大多数前列腺癌组织样本显示出免疫细胞浸润。具有肿瘤促进作用的特定免疫细胞的存在与较差的预后相关,而其中自然杀伤细胞更活跃的肿瘤显示出更好的对治疗的反应和更长的无复发期(参见shiao s.l.等人的“regulation of prostate cancer progression by tumor microenvironment”(cancer lett,第380卷,第1期,第340-348页,2016年))。
[0022]
虽然治疗会受到肿瘤微环境的免疫成分的影响,但是rt本身会广泛地影响这些成分的构成(参见barker h.e.等人的“the tumor microenvironment after radiotherapy:mechanisms of resistance or recurrence”(nat rev cancer,第15卷,第7期,第409-425页,2015年))。因为抑制细胞类型对辐射不太敏感,所以它们的相对数量会增大。相反,造成的辐射损伤会激活细胞存活途径并刺激免疫系统,从而引发炎症反应和免疫细胞募集。净效应是促进肿瘤还是抑制肿瘤尚不确定,但是正在研究其增强癌症免疫治疗的潜力。本发明基于这样的构思:由于免疫系统和免疫微环境的状态对治疗有效性有影响,因此识别预测这种效果的标志物的能力可能有助于更好地预测总体rt反应。
[0023]
可以在不同水平下引发针对病原体的免疫反应:存在物理屏障,诸如皮肤,以防止入侵者进入。如果被破坏,先天免疫发挥作用;第一个快速的非特异性反应。如果这不够,则引发适应性免疫反应。这是更具体的,并且在第一次遇到病原体时需要时间来发展。淋巴细胞通过与来自先天免疫系统的激活的抗原呈递细胞相互作用而被激活,并且还负责维持记忆以便在下次遇到相同病原体时进行更快的反应。
[0024]
由于淋巴细胞在活化时是高度特异性的且有效的,因此它们因其识别自身的能力而受到阴性选择,这是一种称为中枢耐受的过程。由于并非所有自身抗原都在选择位点处表达,因此也进化了外周耐受机制,诸如在不存在共刺激的情况下tcr的连接、抑制性共受体的表达和通过treg的抑制。活化和抑制之间的平衡紊乱可能分别导致自身免疫病症或免疫缺陷和癌症。
[0025]
t细胞活化可以具有不同的功能后果,这取决于所涉及的t细胞类型的位置。cd8+t细胞分化成细胞毒性效应细胞,而cd4+t细胞可以分化成th1(ifnγ分泌和促进细胞介导的免疫)或th2(il4/5/13分泌和促进b细胞和体液免疫)。向其他最近识别的t细胞亚群的分化也是可能的,例如treg,其对免疫活化具有抑制作用(参见mosenden r.和tasken k.的“cyclic amp-mediated immune regulation
–
overview of mechanisms of action in t-cells”(cell signal,第23卷,第6期,第1009-1016页,2011年)(特别是的图4,其是t细胞活化及其通过pka的调节)、以及tasken k.和ruppelt a.的“negative regulation of t-cell receptor activation by the camp-pka-csk signaling pathway in t-cell lipid rafts”(front biosci,第11卷,第2929-2939页,2006年))。
[0026]
pka和pde4调节的信号传导两者与tcr诱导的t细胞活化相交以微调其调节,具有相反的效应(参见abrahamsen h.等人的“tcr-and cd28-mediated recruitment of phosphodiesterase 4to lipid rafts potentiates tcr signaling”(j immunol,第173卷,第4847-4848页,2004年),特别是图6,其示出了pka和pde4对tcr活化的相反效应)。连接这些效应物的分子是环amp(camp),细胞外配体作用的细胞内第二信使。在t细胞中,其介导前列腺素、腺苷、组胺、β-肾上腺素能激动剂、神经肽激素和β-内啡肽的作用。这些细胞外分
子与gpcr的结合导致其构象变化、刺激性亚基的释放和腺苷酸环化酶(ac)的随后活化,所述腺苷酸环化酶将atp水解为camp(参见abrahamsen h.等人(2004年,出处同上)的图6)。尽管不是唯一的一个,但pka是camp信号传导的主要效应物(参见mosenden r.和tasken k.(2011年,出处同上)以及tasken k.和ruppelt a.(2006年,出处同上)。在功能水平处,camp的增加的水平导致t细胞中的减少的ifnγ和il-2产生(参见abrahamsen h.等人(2004年,出处同上))。除了干扰tcr活化之外,pka具有更多得多的效应物(参见torheim e.a.的“immunity leashed
–
mechanisms of regulation in the human immune system”(thesis for the degree of philosophiae doctor(phd),the biotechnology centre of ola,university of oslo,norway,2009年)的图15)。
[0027]
在幼稚t细胞中,过度磷酸化的pag将csk靶向脂筏。经由ezrin-ebp50-pag支架复合体pka靶向csk。通过pka的特异性磷酸化,csk可以负调节lck和fyn以抑制其活性并下调t细胞活化(参见abrahamsen h.等人(2004年,出处同上)的图6)。在tcr活化后,pag被去磷酸化,并且csk从筏中释放。进行t细胞活化需要csk的解离。在相同的时间过程中,形成csk-g3bp复合物,并且似乎将csk隔离在脂筏外(参见mosenden r.和tasken k.(2011,出处同上)以及tasken k.和ruppelt a.(2006,出处同上))。
[0028]
相比之下,组合的tcr和cd28刺激介导环核苷酸磷酸二酯酶pde4向脂质筏的募集,这增强了camp降解(参见abrahamsen h.等人(2004年,出处同上)的图6)。这样一来,tcr诱导的camp产生被抵消,并且t细胞免疫反应增强。在单独的tcr刺激后,pde4募集可能太低而不能完全降低camp水平,并且因此不能发生最大t细胞活化(参见abrahamsen h.等人(2004年,出处同上)的图6)。
[0029]
因此,通过主动抑制近端tcr信号传导,经由camp-pka-csk的信号传导被认为设定了t细胞活化的阈值。pdes的募集能够遇到这种抑制。通过ac、pka和pde的多种同种型的表达来实现组织或细胞类型特异性调节。如上所述,需要严格调节活化和抑制之间的平衡以防止自身免疫病症、免疫缺陷和癌症的发展。
[0030]
识别的t细胞受体信号传导基因cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和zap70被识别如下:用rp处置一组538名前列腺癌患者,并将前列腺癌组织与临床(例如,病理gleason分级组(pggg)、病理状态(pt阶段))以及相关结果参数(例如,生化复发(bcr)、转移性复发、前列腺癌特异性死亡(pca死亡)、补救性辐射处置(srt)、补救性雄激素剥夺治疗(sadt)、化学治疗(ctx))一起储存。对于这些患者中的每个,计算pde4d7评分并将其分类为四个pde4d7评分类别,如alves de inda m.等人的“validation of cyclic adenosine monophosphate phosphodiesterase-4d7 for its independent contribution to risk stratification in a prostate cancer patient cohort with longitudinal biological outcomes”(eur urol focus,第4卷,第3期,第376-384页,2018年)中所述。pde4d7评分类别1表示pde4d7表达水平最低的患者样本,而pde4d7评分类别4表示pde4d7表达水平最高的患者样本。然后针对pde4d7评分类别1和4之间的差异基因表达研究538名前列腺癌对象的rnaseq表达数据(tpm-每百万转录物)。特别地,针对约20000种蛋白质编码转录物确定pde4d7评分1类患者的平均表达水平是否是pde4d7评分4类患者的平均表达水平的两倍以上。该分析导致637个基因,其中,pde4d7评分类别1/pde4d7评分类别4的比率》2,其中,在四个pde4d7评分类别的每个中最小
平均表达为1tpm。这637个基因然后进一步经受分子途径分析(www.david.ncifcrf.gov),这产生一系列富集的注释簇。注释簇#6证明了17种基因中的富集(富集评分:5.9),其在原发性免疫缺陷和t细胞受体信号传导的活化中具有功能。进一步的热图分析证实,这些t细胞受体信号传导基因在来自pde4d7评分类别1的患者的样本中的表达通常高于来自pde4d7评分类别4的患者的表达。
[0031]
术语“cd2”是指分化簇2基因(ensembl:ensg00000116824),例如是指ncbi参考序列nm_001767中定义的序列,特别是指seq id no:1中显示的核苷酸序列,其对应于cd2转录物的上述ncbi参考序列的序列,并且术语“cd2”还指例如seq id no:2中显示的对应的氨基酸序列,其对应于编码cd2多肽的ncbi蛋白质加入参考序列np_001758中定义的蛋白质序列。
[0032]
术语“cd2”还包括针对cd2显示出高度同源性的核苷酸序列,例如,与seq id no:1中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:2中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:2中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:1中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0033]
术语“cd247”是指分化簇247基因(ensembl:ensg00000198821),例如是指ncbi参考序列nm_000734或ncbi参考序列nm_198053中定义的序列,特别是指seq id no:3或seq id no:4中显示的核苷酸序列,其对应于cd247转录物的上述ncbi参考序列的序列,并且术语“cd247”还指例如seq id no:5或seq id no:6中显示的对应的氨基酸序列,其对应于编码cd247多肽的ncbi蛋白质加入参考序列np_000725和ncbi蛋白质加入参考序列np_932170中定义的蛋白质序列。
[0034]
术语“cd247”还包括针对cd247显示出高度同源性的核苷酸序列,例如,与seq id no:3或seq id no:4中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:5或seq id no:6中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:5或seq id no:6中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:3或seq id no:4中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0035]
术语“cd28”是指分化簇28基因(ensembl:ensg00000178562),例如是指ncbi参考序列nm_006139或ncbi参考序列nm_001243078中定义的序列,特别是指seq id no:7或seq id no:8中显示的核苷酸序列,其对应于cd28转录物的上述ncbi参考序列的序列,并且术语“cd28”还指例如seq id no:9或seq id no:10中显示的对应的氨基酸序列,其对应于编码cd28多肽的ncbi蛋白质加入参考序列np_006130和ncbi蛋白质加入参考序列np_001230007中定义的蛋白质序列。
[0036]
术语“cd28”还包括针对cd28显示出高度同源性的核苷酸序列,例如,与seq id no:7或seq id no:8中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:9或seq id no:10中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:9或seq id no:10中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:7或seq id no:8中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0037]
术语“cd3e”是指分化簇3e基因(ensembl:ensg00000198851),例如是指ncbi参考序列nm_000733中定义的序列,特别是指seq id no:11中显示的核苷酸序列,其对应于cd3e转录物的上述ncbi参考序列的序列,并且术语“cd3e”还指例如seq id no:12中显示的对应的氨基酸序列,其对应于编码cd3e多肽的ncbi蛋白质加入参考序列np_000724中定义的蛋白质序列。
[0038]
术语“cd3e”还包括针对cd3e显示出高度同源性的核苷酸序列,例如,与seq id no:11中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:12中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:12中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:11中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0039]
术语“cd3g”是指分化簇3g基因(ensembl:ensg00000160654),例如是指ncbi参考序列nm_000073中定义的序列,特别是指seq id no:13中显示的核苷酸序列,其对应于cd3g转录物的上述ncbi参考序列的序列,并且术语“cd3g”还指例如seq id no:14中显示的对应的氨基酸序列,其对应于编码cd3g多肽的ncbi蛋白质加入参考序列np_000064中定义的蛋白质序列。
[0040]
术语“cd3g”还包括针对cd3g显示出高度同源性的核苷酸序列,例如,与seq id no:13中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:14中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:14中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:13中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0041]
术语“cd4”是指分化簇4基因(ensembl:ensg00000010610),例如是指ncbi参考序列nm_000616中定义的序列,特别是指seq id no:15中显示的核苷酸序列,其对应于cd4转录物的上述ncbi参考序列的序列,并且术语“cd4”还指例如seq id no:16中显示的对应的氨基酸序列,其对应于编码cd4多肽的ncbi蛋白质加入参考序列np_000607中定义的蛋白质
序列。
[0042]
术语“cd4”还包括针对cd4显示出高度同源性的核苷酸序列,例如,与seq id no:15中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:16中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:16中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:15中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0043]
术语“csk”是指c-terminal src kinase基因(ensembl:ensg00000103653),例如是指ncbi参考序列nm_004383中定义的序列,特别是指seq id no:17中显示的核苷酸序列,其对应于csk转录物的上述ncbi参考序列的序列,并且术语“csk”还指例如seq id no:18中显示的对应的氨基酸序列,其对应于编码csk多肽的ncbi蛋白质加入参考序列np_004374中定义的蛋白质序列。
[0044]
术语“csk”还包括针对csk显示出高度同源性的核苷酸序列,例如,与seq id no:17中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:18中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:18中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:17中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0045]
术语“ezr”是指ezrin基因(ensembl:ensg00000092820),例如是指ncbi参考序列nm_003379中定义的序列,特别是指seq id no:19中显示的核苷酸序列,其对应于ezr转录物的上述ncbi参考序列的序列,并且术语“ezr”还指例如seq id no:20中显示的对应的氨基酸序列,其对应于编码ezr多肽的ncbi蛋白质加入参考序列np_003370中定义的蛋白质序列。
[0046]
术语“ezr”还包括针对ezr显示出高度同源性的核苷酸序列,例如,与seq id no:19中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:20中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:20中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:19中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0047]
术语“fyn”是指fyn原癌基因(ensembl:ensg00000010810),例如是指ncbi参考序列nm_002037或ncbi参考序列nm_153047或ncbi参考序列nm_153048中定义的序列,特别是指seq id no:21或seq id no:22或seq id no:23中显示的核苷酸序列,其对应于fyn转录物的上述ncbi参考序列的序列,并且术语“fyn”还指例如seq id no:24或seq id no:25或
seq id no:26中显示的对应的氨基酸序列,其对应于编码fyn多肽的ncbi蛋白质加入参考序列np_002028和ncbi蛋白质加入参考序列np_694592和ncbi蛋白质加入参考序列xp_005266949中定义的蛋白质序列。
[0048]
术语“fyn”还包括针对fyn显示出高度同源性的核苷酸序列,例如,与seq id no:21或seq id no:22或seq id no:23中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:24或seq id no:25或seq id no:26中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:24或seq id no:25或seq id no:26中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:21或seq id no:22或seq id no:23中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0049]
术语“lat”是指t细胞活化衔接因子基因(ensembl:ensg00000198821),例如是指ncbi参考序列nm_001014987或ncbi参考序列nm_014387中定义的序列,特别是指seq id no:27或seq id no:28中显示的核苷酸序列,其对应于lat转录物的上述ncbi参考序列的序列,并且术语“lat”还指例如seq id no:29或seq id no:30中显示的对应的氨基酸序列,其对应于编码lat多肽的ncbi蛋白质加入参考序列np_001014987和ncbi蛋白质加入参考序列np_055202中定义的蛋白质序列。
[0050]
术语“lat”还包括针对lat显示出高度同源性的核苷酸序列,例如,与seq id no:27或seq id no:28中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:29或seq id no:30中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:29或seq id no:30中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:27或seq id no:28中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0051]
术语“lck”是指lck原癌基因(ensembl:ensg00000182866),例如是指ncbi参考序列nm_005356中定义的序列,特别是指seq id no:31中显示的核苷酸序列,其对应于lck转录物的上述ncbi参考序列的序列,并且术语“lck”还指例如seq id no:32中显示的对应的氨基酸序列,其对应于编码lck多肽的ncbi蛋白质加入参考序列np_005347中定义的蛋白质序列。
[0052]
术语“lck”还包括针对lck显示出高度同源性的核苷酸序列,例如,与seq id no:31中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:32中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:32中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:
31中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0053]
术语“pag1”是指具有鞘糖脂微结构域1的磷蛋白膜锚定物基因(ensembl:ensg00000076641),例如是指ncbi参考序列nm_018440中定义的序列,特别是指seq id no:33中显示的核苷酸序列,其对应于pag1转录物的上述ncbi参考序列的序列,并且术语“pag1”还指例如seq id no:34中显示的对应的氨基酸序列,其对应于编码pag1多肽的ncbi蛋白质加入参考序列np_060910中定义的蛋白质序列。
[0054]
术语“pag1”还包括针对pag1显示出高度同源性的核苷酸序列,例如,与seq id no:33中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:34中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:34中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:33中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0055]
术语“pde4d”是指磷酸二酯酶4d基因(ensembl:ensg00000113448),例如是指ncbi参考序列nm_001104631或ncbi参考序列nm_001349242或ncbi参考序列nm_001197218或ncbi参考序列nm_006203或ncbi参考序列nm_001197221或ncbi参考序列nm_001197220或ncbi参考序列nm_001197223或ncbi参考序列nm_001165899或ncbi参考序列nm_001197219中定义的序列,特别是指seq id no:35或seq id no:36或seq id no:37或seq id no:38或seq id no:39或seq id no:40或seq id no:41或seq id no:42或seq id no:43中显示的核苷酸序列,其对应于pde4d转录物的上述ncbi参考序列的序列,并且术语“pde4d”还指例如seq id no:44或seq id no:45或seq id no:46或seq id no:47或seq id no:48或seq id no:49或seq id no:50或seq id no:51或seq id no:52中显示的对应的氨基酸序列,其对应于编码pde4d多肽的ncbi蛋白质加入参考序列np_001098101和ncbi蛋白质加入参考序列np_001336171和ncbi蛋白质加入参考序列np_001184147和ncbi蛋白质加入参考序列np_006194和ncbi蛋白质加入参考序列np_001184150和ncbi蛋白质加入参考序列np_001184149和ncbi蛋白质加入参考序列np_001184152和ncbi蛋白质加入参考序列np_001159371和ncbi蛋白质加入参考序列np_001184148中定义的蛋白质序列。
[0056]
术语“pde4d”还包括针对pde4d显示出高度同源性的核苷酸序列,例如,与seq id no:35或seq id no:36或seq id no:37或seq id no:38或seq id no:39或seq id no:40或seq id no:41或seq id no:42或seq id no:43中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:44或seq id no:45或seq id no:46或seq id no:47或seq id no:48或seq id no:49或seq id no:50或seq id no:51或seq id no:52中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:44或seq id no:45或seq id no:46或seq id no:47或seq id no:48或seq id no:49或seq id no:50或seq id no:51或seq id no:52中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码
氨基酸序列的核酸序列,或与seq id no:35或seq id no:36或seq id no:37或seq id no:38或seq id no:39或seq id no:40或seq id no:41或seq id no:42或seq id no:43中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0057]
术语“prkaca”是指蛋白激酶camp活化的催化亚基α基因(ensembl:ensg00000072062),例如是指ncbi参考序列nm_002730或ncbi参考序列nm_207518中定义的序列,特别是指seq id no:53或seq id no:54中显示的核苷酸序列,其对应于prkaca转录物的上述ncbi参考序列的序列,并且术语“prkaca”还指例如seq id no:55或seq id no:56中显示的对应的氨基酸序列,其对应于编码prkaca多肽的ncbi蛋白质加入参考序列np_002721和ncbi蛋白质加入参考序列np_997401中定义的蛋白质序列。
[0058]
术语“prkaca”还包括针对prkaca显示出高度同源性的核苷酸序列,例如,与seq id no:53或seq id no:54中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:55或seq id no:56中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:55或seq id no:56中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:53或seq id no:54中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0059]
术语“prkacb”是指蛋白激酶camp活化的催化亚基β基因(ensembl:ensg00000142875),例如是指ncbi参考序列nm_002731或ncbi参考序列nm_182948或ncbi参考序列nm_001242860或ncbi参考序列nm_001242859或ncbi参考序列nm_001242858或ncbi参考序列nm_001242862或ncbi参考序列nm_001242861或ncbi参考序列nm_001300915或ncbi参考序列nm_207578或ncbi参考序列nm_001242857或ncbi参考序列nm_001300917中定义的序列,特别是指seq id no:57或seq id no:58或seq id no:59或seq id no:60或seq id no:61或seq id no:62或seq id no:63或seq id no:64或seq id no:65或seq id no:66或seq id no:67中显示的核苷酸序列,其对应于prkacb转录物的上述ncbi参考序列的序列,并且术语“prkacb”还指例如seq id no:68或seq id no:69或seq id no:70或seq id no:71或seq id no:72或seq id no:73或seq id no:74或seq id no:75或seq id no:76或seq id no:77或seq id no:78中显示的对应的氨基酸序列,其对应于编码prkacb多肽的ncbi蛋白质加入参考序列np_002722和ncbi蛋白质加入参考序列np_891993和ncbi蛋白质加入参考序列np_001229789和ncbi蛋白质加入参考序列np_001229788和ncbi蛋白质加入参考序列np_001229787和ncbi蛋白质加入参考序列np_001229791和ncbi蛋白质加入参考序列np_001229790和ncbi蛋白质加入参考序列np_001287844和ncbi蛋白质加入参考序列np_997461和ncbi蛋白质加入参考序列np_001229786和ncbi蛋白质加入参考序列np_001287846中定义的蛋白质序列。
[0060]
术语“prkacb”还包括针对prkacb显示出高度同源性的核苷酸序列,例如,与seq id no:57或seq id no:58或seq id no:59或seq id no:60或seq id no:61或seq id no:62或seq id no:63或seq id no:64或seq id no:65或seq id no:66或seq id no:67中显
示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:68或seq id no:69或seq id no:70或seq id no:71或seq id no:72或seq id no:73或seq id no:74或seq id no:75或seq id no:76或seq id no:77或seq id no:78中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:68或seq id no:69或seq id no:70或seq id no:71或seq id no:72或seq id no:73或seq id no:74或seq id no:75或seq id no:76或seq id no:77或seq id no:78中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:57或seq id no:58或seq id no:59或seq id no:60或seq id no:61或seq id no:62或seq id no:63或seq id no:64或seq id no:65或seq id no:66或seq id no:67中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0061]
术语“ptprc”是指蛋白酪氨酸磷酸酶受体c型基因(ensembl:ensg00000081237),例如是指ncbi参考序列nm_002838或ncbi参考序列nm_080921中定义的序列,特别是指seq id no:79或seq id no:80中显示的核苷酸序列,其对应于ptprc转录物的上述ncbi参考序列的序列,并且术语“ptprc”还指例如seq id no:81或seq id no:82中显示的对应的氨基酸序列,其对应于编码ptprc多肽的ncbi蛋白质加入参考序列np_002829和ncbi蛋白质加入参考序列np_563578中定义的蛋白质序列。
[0062]
术语“ptprc”还包括针对ptprc显示出高度同源性的核苷酸序列,例如,与seq id no:79或seq id no:80中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:81或seq id no:82中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:81或seq id no:82中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:79或seq id no:80中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0063]
术语“zap70”是指t细胞受体相关蛋白激酶70基因(ensembl:ensg00000115085),例如是指ncbi参考序列nm_001079或ncbi参考序列nm_207519中定义的序列,特别是指seq id no:83或seq id no:84中显示的核苷酸序列,其对应于zap70转录物的上述ncbi参考序列的序列,并且术语“zap70”还指例如seq id no:85或seq id no:86中显示的对应的氨基酸序列,其对应于编码zap70多肽的ncbi蛋白质加入参考序列np_001070和ncbi蛋白质加入参考序列np_997402中定义的蛋白质序列。
[0064]
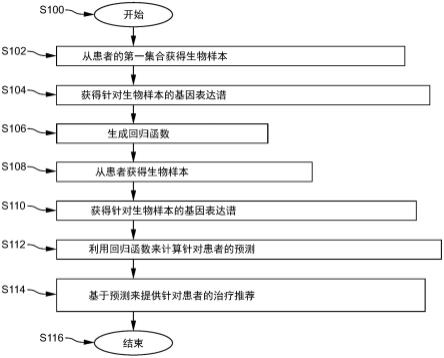
术语“zap70”还包括针对zap70显示出高度同源性的核苷酸序列,例如,与seq id no:83或seq id no:84中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的核酸序列,或与seq id no:85或seq id no:86中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的氨基酸序列,或与seq id no:85或seq id no:86中显示的序列的
相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的编码氨基酸序列的核酸序列,或与seq id no:83或seq id no:84中显示的序列的相同程度为至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的被核酸序列编码的氨基酸序列。
[0065]
术语“生物样本”或“从对象获得的样本”是指经由本领域技术人员已知的合适方法从对象(例如,前列腺癌患者)获得的任何生物材料。可以以临床上可接受的方式(例如以保存核酸(特别是rna)或蛋白质的方式)收集所使用的生物样本。
[0066]
(一个或多个)生物样本可以包括身体组织和/或体液,例如但不限于血液、汗液、唾液和尿液。此外,生物样本可以包含来源于上皮细胞或包括上皮细胞的细胞群(例如,癌性上皮细胞或来源于疑似癌性的组织的上皮细胞)的细胞提取物。生物样本可以包含来源于腺体组织的细胞群,例如,样本可以来源于男性对象的前列腺。另外,如果有必要的话,可以从获得的身体组织和体液纯化细胞,然后将经纯化的细胞用作生物样本。在一些实现方式中,样本可以是组织样本、尿液样本、尿沉渣样本、血液样本、唾液样本、精液样本、包括循环肿瘤细胞的样本、细胞外囊泡、含有前列腺分泌的外泌体的样本或细胞系或癌细胞系。
[0067]
在一个特定的实现方式中,可以获得和/或使用活检或切除样本。这样的样本可以包括细胞或细胞裂解物。
[0068]
还可以设想将生物样本的内容物提交给富集步骤。例如,样本可以与针对细胞膜或某些细胞类型的细胞器特异性的配体(例如,(例如用磁性颗粒功能化的)前列腺细胞)接触。通过磁性颗粒浓缩的材料随后可以用于如上文或下文所述的检测步骤和分析步骤。
[0069]
此外,可以经由对体液或液体样本(例如,血液、尿液等)的过滤过程来富集细胞(例如,肿瘤细胞)。这种过滤过程也可以与基于如上文所述的配体特异性相互作用的富集步骤进行组合。
[0070]
术语“前列腺癌”是指男性生殖系统中的前列腺的癌症,这种癌症是在前列腺的细胞发生突变并开始失控增殖时发生的。通常,前列腺癌与前列腺特异性抗原(psa)水平升高有关。在本发明的一个实施例中,术语“前列腺癌”是指显示出高于3.0的psa水平的癌症。在另一实施例中,该术语是指显示出高于2.0的psa水平的癌症。术语“psa水平”是指血液中psa的浓度(以ng/ml为单位)。
[0071]
术语“非进展性前列腺癌状态”意指个体的样本并未显示出指示“生化复发”和/或“临床复发”和/或“转移”和/或“去势抵抗性疾病”和/或“前列腺癌或疾病特异性死亡”的参数值。
[0072]
术语“进展性前列腺癌状态”意指个体的样本显示出指示“生化复发”和/或“临床复发”和/或“转移”和/或“去势抵抗性疾病”和/或“前列腺癌或疾病特异性死亡”的参数值。
[0073]
术语“生化复发”通常是指psa升高的复发生物学值,其指示样本中存在前列腺癌细胞。然而,也可以使用能够用于检测前列腺癌细胞存在或引起对这种存在的怀疑的其他标志物。
[0074]
术语“临床复发”是指存在指示以下信息的临床症状:通过测量(例如使用体内成像)而指示存在肿瘤细胞。
[0075]
术语“转移”是指在除了前列腺以外的器官中存在转移性疾病。
[0076]
术语“去势抵抗性疾病”是指存在对激素不敏感的前列腺癌;即,前列腺中的癌症
不再对雄激素阻断治疗(adt)产生反应。
[0077]
术语“前列腺癌特异性死亡或疾病特异性死亡”是指患者死于前列腺癌。
[0078]
优选的是,所述八个或更多个t细胞受体信号传导基因包括t细胞受体信号传导基因中的九个或更多个。
[0079]
进一步优选的是,所述八个或更多个t细胞受体信号传导基因包括t细胞受体信号传导基因中的十二个或更多个。
[0080]
更进一步优选的是,所述八个或更多个t细胞受体信号传导基因包括t细胞受体信号传导基因中的十五个或更多个,优选地包括所有t细胞受体信号传导基因。
[0081]
优选的是,对放疗反应的预测的确定包括将针对八个或更多个(例如,8、9、10、11、12、13、14、15、16个或所有)t细胞受体信号传导基因的基因表达谱与已经从前列腺癌对象的群体中导出的回归函数进行组合。
[0082]
cox比例风险回归允许分析若干风险因素对到测试事件的时间(如生存期)的影响。因此,风险因素可能是二分或离散变量,如风险评分或临床分期,但也可能是连续变量,如生物标志物测量结果或基因表达值。终点(例如,死亡或疾病复发)的概率被称为风险。仅次于关于患者队列中的对象是否到达测试终点的信息(例如,患者是否确实死亡),在回归分析中还考虑了到终点的时间。该风险被建模为:h(t)=h0(t)
·
exp(w1·v1
+w2·v2
+w3·v3
+
…
),其中,v1、v2、v3…
是预测变量,并且h0(t)是基线风险,而h(t)是任何时间t时的风险。风险比率(或到达事件的风险)由ln[h(t)/h0(t)]=w1·v1
+w2·v2
+w3·v3
+
…
来表示,其中,系数或权重w1、w2、w3…
是通过cox回归分析来估计的,并且能够以与逻辑回归分析类似的方式进行解读。
[0083]
在一个特定的实现方式中,放疗反应的预测被确定如下:
[0084][0085]
其中,w1至w
17
是权重,并且cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和zap70是t细胞受体信号传导基因的表达水平。
[0086]
在一个示例中,w1可以是大约-0.5到0.5,诸如-0.07388,w2可以是大约-3.5到-2.5,诸如-3.1496,w3可以是大约-1.0到0.0,诸如-0.4443,w4可以是大约-0.5到0.5,诸如-0.005986,w5可以是大约-1.0到0.0,诸如-0.2943,w6可以是大约0.0到1.0,诸如0.5738,w7可以是大约-0.5到0.5,诸如0.1856,w8可以是大约0.0到1.0,诸如0.3914,w9可以是大约-0.5到0.5,诸如0.02173,w
10
可以是大约0.5到1.5,诸如0.7811,w
11
可以是大约-0.5到0.5,诸如0.2116,w
12
可以是大约-0.5到0.5,诸如0.2432,w
13
可以是大约-5.0到-3.0,诸如-4.0934,w
14
可以是大约0.5到1.5,诸如1.176,w
15
可以是大约-1.0到0.0,诸如-0.4655,w
16
可以是大约-1.5到-0.5,诸如-1.153,并且w
17
可以是大约-1.0到0.0,诸如-0.2942。
[0087]
基于放疗反应的预测的值,还可以将放疗反应的预测分类或归类到至少两个风险组之一。例如,可能有两个风险组或三个风险组或四个风险组或四个以上的预定义风险组。每个风险组覆盖放疗反应的预测的(非交叠)值的相应范围。例如,风险组可以指示特定临床事件发生的概率,其范围从0至《0.1或从0.1至《0.25或从0.25至《0.5或从0.5至1.0等。
[0088]
优选的是,所述生物样本是在开始所述放疗之前从所述对象获得的。可以以前列腺癌的组织中的mrna或蛋白质的形式来确定基因表达谱。替代地,如果t细胞受体信号传导基因以可溶形式存在,则可以在血液中确定基因表达谱。
[0089]
进一步优选的是,所述放疗为根治性放疗或挽救性放疗。
[0090]
优选的是,对所述放疗反应的所述预测是针对所述放疗的有效性为负面的或正面的,其中,治疗是基于所述预测来推荐的,并且如果所述预测是负面的,则所推荐的治疗包括以下各项中的一项或多项:(i)作为标准的较早提供的放疗;(ii)具有增大的辐射剂量的放疗;(iii)辅助治疗,例如,雄激素阻断治疗;以及iv)作为非辐射治疗的替代治疗。预测为负面的程度可以决定所推荐的治疗偏离标准形式的放疗的程度。
[0091]
在本发明的另一方面中,提出了一种用于预测前列腺癌对象对放疗的反应的装置,包括:
[0092]-输入部,其适于接收数据,所述数据指示针对从包括以下各项的组中选择的八个或更多个,例如,8、9、10、11、12、13、14、15、16个或所有,t细胞受体信号传导基因中的每个t细胞受体信号传导基因的基因表达谱:cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和zap70,所述基因表达谱是在从所述对象获得的生物样本中确定的,
[0093]-处理器,其适于基于针对所述八个或更多个t细胞受体信号传导基因的所述基因表达谱来确定对放疗反应的预测,并且
[0094]-任选地,提供单元,其适于将所述预测或基于所述预测的治疗推荐提供给医护人员或所述对象。
[0095]
在本发明的另一方面中,提出了一种包括指令的计算机程序产品,当所述程序由计算机执行时,所述指令使所述计算机执行包括以下操作的方法:
[0096]-接收数据,所述数据指示针对从包括以下各项的组中选择的八个或更多个,例如,8、9、10、11、12、13、14、15、16个或所有,t细胞受体信号传导基因中的每个t细胞受体信号传导基因的基因表达谱:cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和zap70,所述基因表达谱是在从前列腺癌对象获得的生物样本中确定的,
[0097]-基于针对所述八个或更多个t细胞受体信号传导基因的所述基因表达谱来确定对放疗反应的预测,以及
[0098]-任选地,将所述预测或基于所述预测的治疗推荐提供给医护人员或所述对象。
[0099]
在本发明的另一方面中,提出了一种诊断试剂盒,包括:
[0100]-至少八个引物和/或探针,其用于确定针对从包括在从对象获得的生物样本中的以下各项的组中选择的八个或更多个,例如,8、9、10、11、12、13、14、15、16个或所有,t细胞受体信号传导基因中的每个t细胞受体信号传导基因的基因表达谱:cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和zap70,并且
[0101]-任选地,根据权利要求9所述的装置或根据权利要求10所述的计算机程序产品。
[0102]
在本发明的另一方面中,提出了一种根据权利要求11所述的试剂盒的用途。
[0103]
优选的是,根据权利要求12所述的用途,其用在预测前列腺癌对象对放疗的反应的方法中。
[0104]
在本发明的另一方面中,提出了一种方法,包括:
[0105]-接收从前列腺癌对象获得的生物样本,
[0106]-使用根据权利要求10所述的试剂盒来确定针对从包括在从所述对象获得的生物样本中的以下各项的组中选择的八个或更多个,例如,8、9、10、11、12、13、14、15、16个或所有,t细胞受体信号传导基因中的每个t细胞受体信号传导基因的基因表达谱:cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和zap70。
[0107]
在本发明的另一方面中,提出了一种针对从包括以下各项的组中选择的八个或更多个,例如,8、9、10、11、12、13、14、15、16个或所有,t细胞受体信号传导基因中的每个t细胞受体信号传导基因的基因表达谱在预测前列腺癌对象对放疗的反应的方法中的用途:cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和zap70,所述用途包括:
[0108]-优选地由处理器基于针对所述八个或更多个t细胞受体信号传导基因的所述基因表达谱来确定对放疗反应的预测,并且
[0109]-任选地,将所述预测或基于所述预测的治疗推荐提供给医护人员或所述对象。
[0110]
应当理解,根据权利要求1所述的方法、根据权利要求9所述的装置、根据权利要求10所述的计算机程序产品、根据权利要求11所述的诊断试剂盒、根据权利要求12所述的诊断试剂盒的用途、根据权利要求14所述的方法以及根据权利要求15所述的(一个或多个)基因表达谱的用途具有相似和/或相同的优选实施例,特别是如在从属权利要求中所定义的实施例。应当理解,本发明的优选实施例也能够是从属权利要求或上述实施例与相应的独立权利要求的任意组合。
[0111]
参考下文描述的实施例,本发明的这些方面和其他方面将变得明显并且得到阐明。
附图说明
[0112]
在以下附图中:
[0113]
图1示意性且示例性地示出了预测前列腺癌对象对放疗的反应的方法的实施例的流程图。
[0114]
图2示出了两个预测模型的roc曲线分析。
[0115]
图3示出了t细胞受体信号传导模型(tcr_信号传导_模型)的kaplan-meier曲线分析。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0116]
图4示出了t细胞受体信号传导8基因模型(tcr_8.1_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0117]
图5示出了另一t细胞受体信号传导8基因模型(tcr_8.2_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0118]
图6示出了另一t细胞受体信号传导8基因模型(tcr_8.3_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特
异性死亡(pca death)的时间。
[0119]
图7示出了另一t细胞受体信号传导8基因模型(tcr_8.4_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0120]
图8示出了另一t细胞受体信号传导8基因模型(tcr_8.5_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0121]
图9示出了另一t细胞受体信号传导8基因模型(tcr_8.6_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0122]
图10示出了另一t细胞受体信号传导8基因模型(tcr_8.7_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0123]
图11示出了另一t细胞受体信号传导8基因模型(tcr_8.8_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0124]
图12示出了t细胞受体信号传导9基因模型(tcr_9.1_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0125]
图13示出了另一t细胞受体信号传导9基因模型(tcr_9.2_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0126]
图14示出了另一t细胞受体信号传导9基因模型(tcr_9.3_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
[0127]
图15示出了另一t细胞受体信号传导9基因模型(tcr_9.4_模型)的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。
具体实施方式
[0128]
放疗反应预测概述
[0129]
图1示意性且示例性地示出了预测前列腺癌对象对放疗的反应的方法的实施例的流程图。
[0130]
该方法开始于步骤s100。
[0131]
在步骤s102处,从被诊断患有前列腺癌的患者(对象)的第一集合中的每个患者(对象)获得生物样本。优选地,在获得生物样本后的一段时间内(例如,至少一年,或至少两年,或大约五年),对这些前列腺癌患者进行了前列腺癌监测。
[0132]
在步骤s104处,例如通过对从每个生物样本中提取的rna执行rt-qpcr(实时定量pcr),针对从患者的第一集合获得的生物样本中的每个生物样本获得针对从包括以下各项
的组中选择的八个或更多个,例如,8、9、10、11、12、13、14、15、16个或所有,t细胞受体信号传导基因中的每个t细胞受体信号传导基因的基因表达谱:cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和zap70。示例性基因表达谱包括针对这一个或多个t细胞受体信号传导基因中的每个t细胞受体信号传导基因的表达水平(例如,值)。
[0133]
在步骤s106处,基于针对患者的第一集合获得的生物样本中的至少一些生物样本获得的这八个或更多个t细胞受体信号传导基因(cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和/或zap70)的基因表达谱并且根据监测获得的相应结果来确定用于分配对放疗反应的预测的回归函数。在一个特定的实现方式中,如上述方程式(1)中所指定的那样确定回归函数。
[0134]
在步骤s108处,从患者(对象或个体)获得生物样本。患者能够是新的患者或第一集合中的一个患者。
[0135]
在步骤s110处,例如通过对生物样本执行pcr来获得针对八个或更多个(例如,8、9、10、11、12、13、14、15、16个或所有)t细胞受体信号传导基因中的每个t细胞受体信号传导基因的基因表达谱。
[0136]
在步骤s112处,使用回归函数来确定针对患者的基于针对八个或更多个t细胞受体信号传导基因的基因表达谱的放疗反应的预测。这将在后面的描述中更详细地描述。
[0137]
在s114处,可以基于预测向例如患者或其监护人员、医生或另一医疗保健工作人员提供治疗建议。为此目的,可以基于预测的值将预测分类到风险组的预定义集合中的一个风险组中。在一个特定的实现方式中,对放疗反应的预测是针对放疗的有效性为负面的或正面的。如果预测是负面的,则所推荐的治疗包括以下各项中的一项或多项:(i)比标准更早提供的放疗;(ii)具有增大的辐射剂量的放疗;(iii)辅助治疗,例如,雄激素阻断治疗;以及iv)作为非辐射治疗的替代治疗。
[0138]
该方法结束于s116。
[0139]
在一个实施例中,通过使用八个或更多个引物和/或探针和/或它们的八个或更多个集合检测mrna表达来确定在步骤s104和s110处的基因表达谱。
[0140]
免疫系统在全身水平上和在肿瘤微环境中都以强烈方式与前列腺癌相互作用。t细胞受体信号传导基因在免疫活动的调节中起核心作用。因此,t细胞受体信号传导基因可以提供关于rt的有效性的信息。然而,由于影响t细胞受体信号传导基因的确切功能的因素很多,因此很难从现有文献中推断出哪些t细胞受体信号传导基因在该应用中可能具有预测价值。
[0141]
我们研究了前列腺癌组织中的t细胞受体信号传导基因的表达与根治性rt或srt后疾病复发的相关程度。
[0142]
我们已经在151个前列腺癌患者的队列中识别出17个t细胞受体信号传导基因,这17个t细胞受体信号传导基因在前列腺癌组织中的表达程度与srt后的死亡率显著相关。
[0143]
基于与rt后的结果的显著相关性,我们预计所识别的分子将提供关于根治性rt和/或srt的有效性的预测价值。
[0144]
结果
[0145]
cox回归分析
the european association of urology biochemical recurrence risk groups to predict metastasis and mortality after radical prostatectomy in a european cohort”(eur urol,第75卷,第6期,第896-900页,2019年))。
[0157]
图2示出了两个预测模型的roc曲线分析。tcr_信号传导_模型(auc=0.88)是基于17个t细胞受体信号传导基因的cox回归模型。eau_bcr_risk(auc=0.77)是eau_bcr_risk组(european association of urology biochemical recurrence risk groups)。
[0158]
kaplan-meier生存分析
[0159]
对于kaplan-meier曲线分析,基于截止值将风险模型(tcr_信号传导_模型)的cox函数归类到两个子队列中(参见下面对附图的描述)。目标是通过将具有如根据每个患者的t细胞受体信号传导模型计算的中值风险评分的类别分开来创建患者类别,其中在个体组内具有一定程度上相似数量的患者。
[0160]
患者类别表示:经历自针对创建的风险模型(tcr_信号传导_模型)的挽救性挽救性辐射治疗(srt)的开始以来到前列腺癌特异性死亡(图3)的时间的测试临床终点的风险增加。
[0161]
图3示出了tcr_信号传导_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr回归模型使用值-6.5作为截止值(对数秩p《0.0001;hr=6.4;95%ci=2.9-14.1)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_信号传导_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:76、76、73、72、61、48、45、24、3、2、0;高风险:75、68、57、42、34、28、26、13、3、1、0。
[0162]
如图3所示的kaplan-meier曲线分析展示出不同患者风险组的存在。通过遭受通过风险模型tcr_信号传导_模型计算的相应临床终点(例如,前列腺癌特异性死亡)的概率来确定患者风险组。取决于预测的患者风险(即,取决于患者可能属于风险组1还是风险组2),可能指示不同类型的介入。
[0163]
进一步的结果
[0164]
本节示出了针对分别仅基于经识别t细胞受体信号传导基因中的八个t细胞受体信号传导基因和九个t细胞受体信号传导基因的cox回归模型的额外结果。总共测试了八个不同的8基因模型和四个不同的9基因模型。在下面的表2和表3中分别示出了权重的详细信息。
[0165]
表2:针对8基因cox回归模型的变量和权重,即,八个t细胞受体信号传导8基因模型(tcr_8.1_模型至tcr_8.8_模型);na
–
不可用。
[0166][0167]
表3:针对9基因cox回归模型的变量和权重,即,四个t细胞受体信号传导9基因模型(tcr_9.1_模型至tcr_9.4_模型);na
–
不可用。
[0168][0169]
对于kaplan-meier曲线分析,基于截止值将十二个风险模型(tcr_8.1_模型至
tcr_8.8_模型和tcr_9.1_模型至tcr_9.4_模型)的cox回归函数归类到两个子队列中(参见下面对附图的描述),如上所述。
[0170]
患者类别表示经历自针对所创建的风险模型开始挽救性rt以来到前列腺癌特异性死亡的时间的测试临床终点的风险提高。
[0171]
图4示出了tcr_8.1_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_8.1_模型使用值0.08作为截止值(对数秩p=0.005;hr=4.1;ci=1.5-10.8)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_8.1_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:79、72、62、49、27、17、8、4、0;高风险:106、91、69、49、33、16、7、4、0。
[0172]
图5示出了tcr_8.2_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_8.2_模型使用值-0.02作为截止值(对数秩p=0.003;hr=3.2;ci=1.5-7.1)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_8.2_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:69、62、52、41、23、15、6、3、0;高风险:116、101、79、57、37、18、9、5、0。
[0173]
图6示出了tcr_8.3_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_8.3_模型使用值-0.1作为截止值(对数秩p=0.005;hr=3.1;ci=1.4-6.8)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_8.3_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:60、54、47、36、23、15、6、3、0;高风险:125、109、84、62、37、18、9、5、0。
[0174]
图7示出了tcr_8.4_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_8.4_模型使用值0.25作为截止值(对数秩p=0.0002;hr=4.5;ci=2.0-9.8)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_8.4_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:92、82、68、54、31、20、10、5、0;高风险:93、81、63、44、29、13、5、3、0。
[0175]
图8示出了tcr_8.5_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_8.5_模型使用值-0.3作为截止值(对数秩p=0.004;hr=3.2;ci=1.5-7.0)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_8.5_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:68、60、50、39、24、17、7、3、0;高风险:117、103、81、59、36、16、8、5、0。
[0176]
图9示出了tcr_8.6_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_8.6_模型使用值0.1作为截止值(对数秩p=0.0002;hr=4.5;ci=2.9-9.8)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_8.6_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:89、78、66、49、31、20、7、3、0;高风险:96、85、65、49、29、13、8、5、0。
[0177]
图10示出了tcr_8.7_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_8.7_模型使用值0.1作为截止值(对数秩p=0.004;hr=3.2;ci=1.5-7.0)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_8.7_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:92、81、68、51、33、21、8、4、0;高风险:93、82、63、47、12、7、4、0。
[0178]
图11示出了tcr_8.8_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_8.8_模型使用值0.2作为截止值(对数秩p《0.0001;hr=5.3;ci=2.4-11.8)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_8.8_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:92、81、68、51、33、21、8、4、0;高风险:93、82、63、47、27、12、7、4、0。
[0179]
图12示出了tcr_9.1_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_9.1_模型使用值0.14作为截止值(对数秩p=0.0002;hr=4.4;ci=2.0-9.7)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_9.1_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:85、76、63、50、28、18、9、5、0;高风险:100、87、68、48、32、15、6、3、0。
[0180]
图13示出了tcr_9.2_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_9.2_模型使用值0.06作为截止值(对数秩p=0.0007;hr=3.9;ci=1.8-8.6)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_9.2_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:90、79、65、50、26、15、7、3、0;高风险:95、84、66、48、34、18、8、5、0。
[0181]
图14示出了tcr_9.3_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_9.3_模型使用值0.0作为截止值(对数秩p=0.0006;hr=4.0;ci=1.8-8.7)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对
分析的tcr_9.3_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:87、77、63、48、24、15、7、3、0;高风险:98、86、68、50、36、18、8、5、0。
[0182]
图15示出了tcr_9.4_模型的kaplan-meier曲线。被测试的临床终点是由于术后疾病复发而开始挽救性辐射治疗(srt)后到前列腺癌特异性死亡(pca death)的时间。根据患者经历如通过tcr_9.4_模型使用值0.1作为截止值(对数秩p=0.0003;hr=4.3;ci=2.0-9.6)预测的临床终点的风险将患者分层到两个队列(低vs高)中。以下补充列表指示了针对分析的tcr_9.4_模型类别的处于风险的患者的数量,即,示出了在手术后+20个月的任何时间区间处于风险的患者:低风险:89、82、69、55、29、19、10、5、0;高风险:96、81、62、43、31、14、5、3、0。
[0183]
如图4至15所示的kaplan-meier分析展示出也能够使用仅基于经识别的t细胞受体信号传导基因的子集(例如,基因中的八个基因或九个基因)的风险模型来区分不同患者风险组。
[0184]
讨论
[0185]
根治性rt和srt这两者对局限性前列腺癌的有效性是有限的,从而导致患者的疾病进展和最终死亡,特别是对于高复发风险的患者。随着许多因素在治疗有效性和疾病复发中发挥作用,对治疗的预测变得非常复杂。很可能尚未识别出重要因素,同时无法精确确定其他因素的效果。当前正在研究多种临床病理措施并将其应用于临床环境中以改进反应预测和治疗选择,从而提供一定程度的改进。尽管如此,仍然强烈需要更好的对根治性rt和srt的反应的预测,以便提高这些治疗的成功率。
[0186]
我们已经识别出一些分子,其表达显示出与根治性rt和srt后死亡具有显著关系,因此预计将改进对这些处置的有效性的预测。无论是在根治性背景中还是在挽救性背景中,改进的对rt对每个患者的有效性的预测都将改进治疗选择并将潜在地提高生存率。这能够通过以下操作来实现:1)为那些预测rt有效的患者优化rt(例如通过剂量递增或不同的开始时间),以及2)将预测rt无效的患者引导到可能更有效的处置形式的替代方案。另外,这将减少那些免于无效治疗的患者的痛苦并减少在无效治疗上花费的成本。
[0187]
本领域技术人员通过研究附图、公开内容以及权利要求,在实践请求保护的发明时能够理解并实现对所公开的实现方式的其他变型。
[0188]
在优选的实施例中,一个或多个t细胞受体信号传导基因从包括以下各项的组中选择:cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、prkaca、prkacb、ptprc和zap70。例如,可以选择来自该组的1、2、3、4、5、6、7、8、9、10、11、12、13、14、15或所有t细胞受体信号传导基因,并且可以基于针对一个或多个t细胞受体信号传导基因的(一个或多个)基因表达谱来确定放疗反应的预测。同样地,针对从该组中选择的八个或更多个,例如,2、3、4、5、6、7、8、9、10、11、12、13、14、15或所有,t细胞受体信号传导基因的基因表达谱可以与已经从前列腺癌对象的群体中导出的回归函数进行组合。
[0189]
在权利要求中,“包括”一词并不排除其他元件或步骤,并且词语“一”或“一个”并不排除多个。
[0190]
图1显示的方法的一个或多个步骤可以被实施在可以在计算机上执行的计算机程序产品中。计算机程序产品可以包括在其上记录(存储)控制程序的非瞬态计算机可读记录
介质,例如,磁盘、硬盘驱动器等。非瞬态计算机可读介质的常见形式包括例如软盘、软磁盘、硬盘、磁带或任何其他磁性存储介质、cd-rom、dvd或任何其他光学介质、ram、prom、eprom、flash-eprom或其他存储器芯片或盒式存储器,或计算机能够从中读取和使用的任何其他非瞬态介质。
[0191]
替代地,该方法的一个或多个步骤可以被实施在瞬态介质中,例如,可传输的载波,其中,控制程序被体现为使用传输介质(例如,(例如在无线电波和红外数据通信等期间生成的那些)声波或光波)的数据信号。
[0192]
示例性方法可以被实施在一个或多个通用计算机、(一个或多个)专用计算机、编程的微处理器或微控制器和外围集成电路元件、asic或其他集成电路、数字信号处理器、硬连线的电子或逻辑电路(例如,分立元件电路)、可编程逻辑器件(例如,pld、pla、fpga、图形卡cpu(gpu)或pal)等上。一般而言,任何能够实施有限状态机的设备(该有限状态机反过来能够实施图1显示的流程图)都能够用于实施风险分层方法的一个或多个步骤,以便提供对前列腺癌患者的治疗选择,如图显示。如将意识到的,虽然该方法的步骤都可以由计算机来实施,但是在一些实施例中,这些步骤中的一个或多个步骤也可以至少部分地手动执行。
[0193]
计算机程序指令也可以被加载到计算机、其他可编程数据处理装置或其他设备上,以引起要在计算机、其他可编程装置或其他设备上执行的一系列操作步骤,从而产生计算机实施的过程,使得在计算机或其他可编程装置上运行的指令提供用于实施在本文中指定的功能/动作的过程。
[0194]
权利要求中的任何附图标记都不应被解释为对范围的限制。
[0195]
本发明涉及一种预测前列腺癌对象对放疗的反应的方法,包括确定或接收对针对从包括以下各项的组中选择的八个或更多个,例如,8、9、10、11、12、13、14、15、16个或所有,t细胞受体信号传导基因中的每个t细胞受体信号传导基因的基因表达谱的确定的结果:cd2、cd247、cd28、cd3e、cd3g、cd4、csk、ezr、fyn、lat、lck、pag1、pde4d、prkaca、prkacb、ptprc和zap70,所述基因表达谱是在从所述对象获得的生物样本中确定的,优选地由处理器基于针对所述八个或更多个t细胞受体信号传导基因的所述基因表达谱来确定对放疗反应的预测,并且任选地,将所述预测或基于所述预测的治疗推荐提供给医护人员或所述对象。由于免疫系统和免疫微环境的状态对治疗有效性有影响,因此识别预测这种效果的标志物的能力可能有助于更好地预测总体rt反应。t细胞受体信号传导基因在免疫活动的调节中起核心作用。发现经识别的t细胞受体信号传导基因与rt后的结果具有显著相关性,因此我们预计经识别的t细胞受体信号传导基因将提供关于根治性rt和/或srt的有效性的预测价值。
[0196]
所附的序列表的标题为2019pf00712_sequencelisting_st25,通过引用将其整体并入本文。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1