用于进行提高谷氨酰胺生产力的转化的重组载体和使用其的菌株的制作方法
1.本发明涉及用于进行提高谷氨酰胺生产力的转化的重组载体,以及引入了该重组载体的菌株。
背景技术:
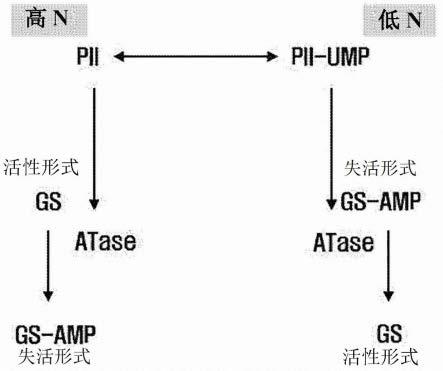
2.谷氨酰胺合成酶(glutamine synthetase,gs)的活性由谷氨酰胺合成酶腺苷酸转移酶(glutamine synthetase adenylyltransferase,atase,glne)调节。atase通过催化gs的腺苷酸化和脱腺苷酸化来调节gs的活性,并受到培养基中氮浓度的影响。atase的活性受pii(氮调节蛋白p-ii基因,glnb)的调节。图1示出了这样的机制:其中当氮浓度高时,gs的活性受到pii和atase活化的抑制。因此,当供应氮源来产生谷氨酰胺时,会出现这样的问题:gs的活性受到所供应的氮源反馈抑制,从而导致谷氨酰胺产量降低。
3.因此,为了提高谷氨酰胺产生的效率,需要抑制由atase引起的对gs的反馈抑制。日本专利no.jp4898441公开了这样的菌株:在所述菌株中,通过抑制参与gs活性抑制的glnb和glne基因的活性来提高谷氨酰胺产生,但仍有改进的空间。
4.[现有技术文献]
[0005]
(专利文献0001)日本专利no.jp 4898441 b2(2012年1月6日)
[0006]
公开内容
[0007]
技术问题
[0008]
一个实施方案提供了产生谷氨酰胺的菌株,其包含含有编码由seq id no:1的氨基酸序列组成的谷氨酰胺合成酶(gs)的核苷酸序列的载体。
[0009]
技术方案
[0010]
一个方面提供了用于进行转化的载体,其含有编码由seq id no:1的氨基酸序列组成的谷氨酰胺合成酶(gs)的核苷酸序列。
[0011]
谷氨酰胺合成酶是由谷氨酸和氨合成谷氨酰胺的酶。由于由seq id no:1的氨基酸序列组成的谷氨酰胺合成酶的活性受atase抑制的程度低于由另一序列组成的谷氨酰胺合成酶的活性受atase抑制的程度,因此具有seq id no:1的氨基酸序列的谷氨酰胺合成酶可提高谷氨酰胺生产力。
[0012]
根据一个实施方案,编码谷氨酰胺合成酶的核苷酸序列可由seq id no:2的核苷酸序列组成。
[0013]
根据一个实施方案,由seq id no:1的氨基酸序列组成的谷氨酰胺合成酶和由seq id no:2的核苷酸序列组成的glna基因可源自以登记号kfcc10694保藏的谷氨酸棒状杆菌(corynebacterium glutamicum)菌株。根据一个实施方案,作为比较源自kfcc10694的glna序列与源自另一谷氨酸棒状杆菌菌株atcc13032的glna序列之间的同源性的结果,确定了核苷酸序列同源性仅为88.2%,并且由所述基因表达的谷氨酰胺合成酶之间的氨基酸序列同源性仅为93.7%。由于这种序列差异,源自kfcc10694的谷氨酰胺合成酶的活性受atase
for general bacteriology,american society for bacteriology,washington d.c.,usa,1981)。此外,培养基可包含多种碳源、氮源和微量元素组分。可使用的碳源的一些实例包括:糖类和碳水化合物,例如葡萄糖、蔗糖、乳糖、果糖、麦芽糖、淀粉和纤维素;油和脂肪,例如大豆油、葵花籽油、蓖麻油和椰子油;脂肪酸,例如棕榈酸、硬脂酸和亚油酸;醇,例如甘油和乙醇;以及有机酸,例如乙酸。这些物质可单独使用或作为混合物使用。可使用的氮源的一些实例包括蛋白胨、酵母提取物、肉提取物、麦芽提取物、玉米浆、大豆粉、尿素,或者无机化合物例如硫酸铵、氯化铵、磷酸铵、碳酸铵和硝酸铵。氮源也可单独使用或作为混合物使用。可使用的磷源的一些实例包括磷酸二氢钾或磷酸氢二钾、或者相应的含钠盐。培养基可包含生长所需的金属盐,例如硫酸镁或硫酸铁。另外,培养基可包含必需的生长物质,例如氨基酸和维生素。此外,可在培养基中使用合适的前体。可在培养期间将培养基或单独的组分通过合适的方法分批或以连续方式添加至培养基中。
[0030]
培养基的ph可通过在培养期间以适当方式向微生物培养基中添加化合物例如氢氧化铵、氢氧化钾、氨、磷酸和硫酸来调节。另外,在培养期间,可使用消泡剂例如脂肪酸聚乙二醇酯来抑制发泡。另外,为了将培养基保持在有氧条件下,可将氧气或含氧气体(例如,空气)注入培养基中。培养基的温度通常可以是20℃至45℃,例如25℃至40℃。可继续培养直至产生所期望量的l-谷氨酰胺。例如,培养时间可以是10小时至160小时。
[0031]
用于产生谷氨酰胺的方法可包括从所培养的微生物或培养基中回收l-谷氨酰胺的步骤。用于回收l-谷氨酰胺的方法没有特别限制,并且可以根据培养方法使用本领域中已知的适当方法回收l-谷氨酰胺。用于回收l-谷氨酰胺的方法的一些实例包括离心、过滤、阴离子交换色谱、结晶、hplc等。
[0032]
有益效果
[0033]
由于根据一个实施方案的菌株表达的谷氨酰胺合成酶较少地受到atase反馈抑制,因此其可显著提高谷氨酰胺产量。
附图说明
[0034]
图1示出了其中谷氨酰胺合成酶的活性受调节的机制。
[0035]
图2示出了根据一个实施方案的pk19ms/
△
glna载体。
[0036]
图3示出了根据一个实施方案的pk19ms/
△
glne载体。
[0037]
图4示出了根据一个实施方案的pa
’‑
glna(atcc13032)载体。
[0038]
图5示出了根据一个实施方案的pa
’‑
glna(kfcc10694)载体。
[0039]
发明实施方式
[0040]
在下文中,将参考实施例更详细地描述一个或更多个实施方案。然而,这些实施例用于举例说明一个或更多个实施方案,并且本发明的范围不限于这些实施例。
[0041]
实施例1:用于使kfcc-10694染色体上glna和glne基因缺失的载体的构建
[0042]
作为用于载体构建的材料,使用了wizard基因组dna纯化试剂盒(promega,usa)、primestar max dna聚合酶(takara,japan)、dna连接试剂盒(takara,japan)以及hindiii和bamhi(neb,england)。
[0043]
1-1.用于使glna缺失的载体的构建
[0044]
使用kfcc-10694菌株(谷氨酸棒状杆菌mwm-891020)的染色体dna作为模板,用引
物1和引物2进行pcr以获得kfcc-10694的glna左臂的扩增产物。类似地,用引物3和引物4进行pcr以获得glna右臂的扩增产物。
[0045]
用引物1和引物4的组合对glna的左臂和右臂的扩增产物进行交叉pcr以获得左臂和右臂连接在一起的扩增产物。将所获得的扩增产物插入到pk19mobsacb载体的bamhi位点中。将用于使glna缺失的所构建的载体命名为pk19ms/
△
glna(参见图1)。
[0046]
1-2.用于使glne缺失的载体的构建
[0047]
以与构建用于使glna缺失的载体相同的方式,用引物5和引物6的组合进行pcr以获得kfcc-10694的glne左臂的扩增产物,并且用引物7和引物8的组合进行pcr以获得glne右臂的扩增产物。
[0048]
使用引物5和引物8的组合,通过交叉pcr来扩增glne的左臂和右臂的扩增产物以获得左臂和右臂连接在一起的扩增产物。将扩增产物插入到pk19mobsacb载体的hindiii位点中。将用于使glne缺失的所构建的载体命名为pk19ms/
△
glne(参见图2)。
[0049]
下表1示出了关于kfcc-10694的glna、kfcc-10694的glne以及引物1至8的核苷酸序列的信息。
[0050]
[表1]
[0051][0052]
实施例2:过表达glna的两种载体的构建
[0053]
使用atcc13032的glna基因dna作为模板,用引物9和引物10的组合进行pcr以获得glna(at)扩增产物。
[0054]
单独地,使用kfcc10694的glna基因dna作为模板,用引物11和引物12的组合进行pcr以获得glna(kf)扩增产物。
[0055]
作为检查glna(at)和glna(kf)之间的同源性的结果,确定了核苷酸序列同源性仅为88.2%,并且氨基酸序列同源性仅为93.7%。glna(at)和glna(kf)的氨基酸序列和核苷酸序列在下表2中示出。
[0056]
[表2]
[0057]
[0058][0059]
作为sod启动子,通过使用引物13(正向)和引物14(反向)的组合扩增atcc13032的染色体dna(作为模板)来获得第一sod启动子扩增产物,并且通过使用引物13(正向)和引物
15(反向)的组合扩增atcc13032的染色体dna(作为模板)来获得第二sod启动子扩增产物。分别使用引物14和15作为反向引物(反向)的原因在于glna序列不同,而sod启动子序列相同(参见下表2中的)。
[0060]
作为rrnbt1t2转录终止子序列,通过使用引物16(正向)和引物17(反向)的组合扩增大肠杆菌(e.coli)dh5a的染色体dna(作为模板)来获得第一rrnbt1t2扩增产物,并且通过使用引物18(正向)和引物17(反向)的组合扩增大肠杆菌dh5a的染色体dna来获得第二(2)rrnbt1t2扩增产物。分别使用引物16和18作为正向引物的原因在于glna序列不同,而转录终止子序列相同(参见下表2中的)。
[0061]
通过使用引物13和引物17的组合进行交叉pcr来将第一sod启动子扩增产物glna(at)和第一rrnbt1t2终止子扩增产物插入到pa’载体(其是大肠杆菌-棒状杆菌穿梭载体)中的bamhi位点中,从而构建pa
’‑
glna(at)载体(参见图4)。
[0062]
类似地,通过使用引物13和引物17的组合进行交叉pcr来将第二sod启动子扩增产物glna(kf)和第二rrnbt1t2终止子扩增产物插入到pa’载体(其是大肠杆菌-棒状杆菌穿梭载体)中的bamhi位点中,从而构建pa
’‑
glna(kf)载体(参见图5)。
[0063]
实施例2中使用的实验材料为primestar max dna聚合酶(takara,japan)、dna连接试剂盒(takara,japan)以及bamhi(neb,england)。
[0064]
引物9至18的核苷酸序列在下表3中示出。
[0065]
[表3]
[0066][0067]
实施例3:含有glna基因和/或glne基因的缺失的kfcc10694菌株的构建
[0068]
通过电穿孔将实施例1中构建的pk19ms/
△
glna载体引入到kfcc10694菌株的感受态细胞中,并将细胞平板接种于2yt km agar培养基上,并随后在30℃下在培养箱中培养4天以获得菌落。在其中诱导了第一同源重组的菌落中,将转化的菌落在2yt液体培养基中培养12小时,并随后平板接种到2yt蔗糖gm琼脂培养基上,并通过第二同源重组去除抗生素标志物。最后通过对选定的菌落进行pcr和测序来检查glna基因是否如预期地被去除。将通过上述过程构建的glna基因缺失菌株命名为d10694a。
[0069]
以同样的方式,将实施例1中构建的pk19ms/
△
glne载体引入到d10694a菌株中,从而构建包含glna和glne这两种基因的缺失的菌株。在本实验中使用的培养基包含浓度为100mg/l的谷氨酰胺。将通过上述过程构建的包含glna和glne这两种基因的缺失的菌株命名为d10694ae。
[0070]
实施例3中使用的实验材料为2yt琼脂(16g/l胰胨,10g/l酵母提取物,5g/lnacl,1.5%琼脂)、2yt km琼脂(2yt琼脂,15mg/l卡那霉素)、2yt蔗糖gm琼脂(2yt琼脂,100g/l蔗糖,100mg/l谷氨酰胺)以及电泳仪(bio-rad,usa)。
[0071]
实施例4:引入有pa
’‑
glna(at)载体或pa
’‑
glna(kf)载体的菌株的构建
[0072]
通过电穿孔将实施例2中构建的pa
’‑
glna(atcc13032)载体和pa
’‑
glna(kfcc10694)载体中的每一种引入到实施例3中构建的d10694a和d10694ae菌株中的每一种中。将已引入有每种载体的每种菌株平板接种在2yt km琼脂培养基上,并在30℃下在培养箱中培养3天以获得菌落。将通过上述过程构建的菌株分别命名为d10694a/pa-glna(at)、d10694a/pa-glna(kf)、d10694ae/pa-glna(at)以及d10694ae/pa-glna(kf)。
[0073]
实施例4中使用的实验材料为2yt琼脂(16g/l胰胨,10g/l酵母提取物,5g/lnacl,1.5%琼脂)、2yt km琼脂(2yt琼脂,15mg/l卡那霉素)以及电泳仪(bio-rad,usa)。
[0074]
实施例5:实施例4中构建的菌株的谷氨酰胺生产力的分析
[0075]
将20ml种子培养基分配到500ml锥形瓶中,并根据常规方法进行高压灭菌,并随后将每种菌株接种到培养基中,并在30℃下振荡培养24小时以获得种子培养物。将100ml生产培养基分配到500ml锥形瓶中,并根据常规方法进行高压灭菌,并随后将100ml的每种预先制备的种子培养物接种到培养基中,并在30℃下振荡培养72小时。为了比较d10694a和d10694ae菌株的生产力,将100mg/l谷氨酰胺添加到培养基中。在培养完成之后,通过常规的hplc法确定每种培养物中的l-谷氨酰胺含量。
[0076]
对于谷氨酰胺生产力的实验结果在下表4中示出。
[0077]
[表4]
[0078]
菌株名称od(1/100)δ(l-gln(%))kfcc106940.2563.56d10694a(glna缺失)0.135n.d.d10694ae(glna与glne缺失)0.185n.d.d10694a/pa-glna(at)0.1381.97d10694a/pa-glna(kf)0.2025.24d10694ae/pa-glna(at)0.2093.88d10694ae/pa-glna(kf)0.2175.31
[0079]
上表3中的实验结果表明,由于glna(gs表达基因)的缺失,d10694a菌株和d10694ae菌株未产生l-谷氨酰胺。
[0080]
关于表3中d10694a/pa-glna(at)和d10694a/pa-glna(kf)的谷氨酰胺生产力,与亲本菌株kfcc10694的l-谷氨酰胺生产力相比,d10694a/pa-glna(at)的l-谷氨酰胺生产力下降,而与亲本菌株kfcc10694的l-谷氨酰胺生产力相比,d10694a/pa-glna(kf)的l-谷氨酰胺生产力提高。虽然将相同的sod启动子引入到两种菌株中,但是与引入glna(at)时相比,引入glna(kf)时的l-谷氨酰胺生产力显著更高。这表明glna(at)和glna(kf)之间的反
馈抑制程度有差异。
[0081]
作为检查由于atase失活而没有进行反馈抑制的d10694ae/pa-glna(at)和d10694ae/pa-glna(kf)的l-谷氨酰胺生产力的结果,可以看出d10694ae/pa-glna(at)菌株的l-谷氨酰胺生产力比d10694a/pa-glna(at)菌株的l-谷氨酰胺生产力高约1.91%,这表明取决于atase活性的存在或不存在,l-谷氨酰胺生产力存在显著差异。然而,可以看出,与d10694a/pa-glna(kf)菌株的l-谷氨酰胺生产力相比,d10694ae/pa-glna(kf)菌株的l-谷氨酰胺生产力提高了0.07%,但提高的程度并不显著,这表明取决于atase活性的存在与不存在的l-谷氨酰胺生产力的差异是不显著的。这意味着glna(kf)较少地受到atase的反馈抑制。
[0082]
更具体地说,与亲本菌株相比,d10694a/pa-glna(kf)菌株显示出生产力提高了约47%,并且与亲本菌株相比,d10694ae/pa-glna(kf)菌株显示出生产力提高了约49%。值得注意的是,与亲本菌株相比,d10694a/pa-glna(kf)和d10694ae/pa-glna(kf)之间的生产力差异仅为2%,其是不显著的。虽然d10694ae/pa-glna(kf)额外缺少参与gs的反馈抑制的glne基因,但是与相比于亲本菌株的d10694a/pa-glna(kf)的谷氨酰胺生产力的提高相比,相比于亲本菌株的d10694ae/pa-glna(kf)的谷氨酰胺生产力的提高为仅2%,这表明取决于atase活性的存在或不存在的谷氨酰胺生产力的差异是不显著的。这表明通过glna(kf)的引入提高生产力不仅仅是由于sod启动子的贡献,而且还是由于glna(kf)对反馈抑制的抗性的贡献。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1