一种与黑鲷生长性状相关的SNP分子标记及其应用的制作方法
kinase2基因的snp分子标记的核苷酸序列如seqidno.1所示,该序列的第276位碱基为t或c;
8.所述位于cytochromep4503a基因的snp分子标记的核苷酸序列如seqidno.2所示,该序列的第364位碱基为a或c;
9.所述位于complementcomponent4基因的snp分子标记的核苷酸序列如seqidno.3所示,该序列的第6070位碱基为a或t。
10.上述与黑鲷生长性状相关的snp分子标记在筛选快速生长的黑鲷方面的应用,所述位于adenylatekinase2基因的snp分子标记的tt基因型个体生长速度显著高于cc和tc基因型个体;所述位于cytochromep4503a基因的snp分子标记的aa基因型个体生长速度显著高于cc和ac基因型个体;所述位于complementcomponent4基因的snp分子标记的tt基因型个体生长速度显著高于aa和at基因型个体。
11.用于检测上述与黑鲷生长性状相关的snp分子标记的引物组,所述引物组包括用于检测位于adenylatekinase2基因的snp分子标记的引物对、用于检测位于cytochromep4503a基因的snp分子标记的引物对和用于检测位于complementcomponent4基因的snp分子标记的引物对,所述用于检测位于adenylatekinase2基因的snp分子标记的引物对包括正向引物、反向引物和延伸引物,序列如seqidno.4、seqidno.5和seqidno.6所示;所述用于检测位于cytochromep4503a基因的snp分子标记的引物对包括正向引物、反向引物和延伸引物,序列如seqidno.7、seqidno.8和seqidno.9所示;所述用于检测位于complementcomponent4基因的snp分子标记的引物对包括正向引物、反向引物和延伸引物,序列如seqidno.10、seqidno.11和seqidno.12所示。
12.上述引物组在筛选快速生长的黑鲷方面的应用。
13.一种用于筛选快速生长黑鲷的试剂盒,其包括用于检测位于adenylatekinase2基因的snp分子标记的引物对、用于检测位于cytochromep4503a基因的snp分子标记的引物对和用于检测位于complementcomponent4基因的snp分子标记的引物对。所述用于检测位于adenylatekinase2基因的snp分子标记的引物对包括正向引物、反向引物和延伸引物,序列如seqidno.4、seqidno.5和seqidno.6所示;所述用于检测位于cytochromep4503a基因的snp分子标记的引物对包括正向引物、反向引物和延伸引物,序列如seqidno.7、seqidno.8和seqidno.9所示;所述用于检测位于complementcomponent4基因的snp分子标记的引物对包括正向引物、反向引物和延伸引物,序列如seqidno.10、seqidno.11和seqidno.12所示。
14.一种快速生长黑鲷的筛选方法,包括如下步骤:
15.(1)提取待检测黑鲷的基因组dna;
16.(2)以提取得到的dna为模板,利用上述引物组进行pcr扩增,得到pcr产物;
17.(3)采用sap(shrimpalikaiinephosphtase虾碱性硫酸酶)对pcr扩增产物进行碱性磷酸酶处理;
18.(4)以处理后的pcr扩增产物为模板,利用上述引物组进行延伸反应;
19.(5)对延伸反应产物进行飞行时间质谱检测,根据检测结果确定样品的snp分子标记的基因型,位于adenylatekinase2基因的snp分子标记的tt基因型个体、位于cytochromep4503a基因的snp分子标记的aa基因型个体或位于complementcomponent4
基因的snp分子标记的tt基因型个体为快速生长的黑鲷个体。
20.本发明的有益效果:
21.1)本发明提供了一种与黑鲷生长性状相关的snp分子标记,与现有技术相比,本发明可以实现从dna水平上进行选择育种,克服了传统方法受人为因素影响大的不利因素,提高了选择的准确性。
22.2)本发明可实现在早期鉴定出具有优良性状的个体,筛选出优良的后备亲本,从而缩短育种周期,加快育种进程。
23.3)本发明共采用三处snp分子标记位点,任意一个标记均可作为高生长率黑鲷个体的判断依据,也可配合使用,操作方便且结果准确可靠,实现从分子遗传学角度对黑鲷进行快速生长品系的选育的目的,提高黑鲷生长性能。
附图说明
24.图1为位于adenylate kinase 2基因的snp分子标记的tt基因型质谱图;
25.图2为位于cytochrome p4503a基因的snp分子标记的aa基因型质谱图;
26.图3为位于complement component 4基因的snp分子标记的tt基因型质谱图。
具体实施方式
27.下面结合附图和具体实施例对本发明的技术方案作进一步的说明,但并不局限于此。
28.实施例1
29.一、snp分子标记的获得
30.所用实验鱼为同批刚孵化的黑鲷,室外池塘培育,每日投喂饲料两次。培育期间,水温及盐度均在黑鲷养殖适应范围内。4月龄时,黑鲷存活率为100%。在所有黑鲷中挑选极大个体(生长速度快)、极小个体(生长速度慢)各5条,麻醉后立即剪取组织样品,然后提取每尾鱼的总rna,取等量的总rna构建快生长组和慢生长组的rna文库,并进行转录组测序,测序结果与黑鲷全基因组序列进行比对,经生物信息软件分析,获得了大量潜在的snp位点,其中在 adenylate kinase 2(ak2)、cytochrome p4503a(cyp3a40)和complementcomponent 4(c4)基因的编码区各存在一个snp位点。采用maldi-tof ms 方法对3个位点进行验证,经实验证实了这3个snp位点的存在。
31.第一个snp分子标记位于黑鲷ak2基因序列(seq id no:1)的第276 个碱基处,该snp位点的等位基因为t和c,有tt、ct和cc三种基因型。
32.第二个snp分子标记位于黑鲷cyp3a40基因序列(seq id no:2)的第364个碱基处,该snp位点的等位基因为a和c,有aa、ac和cc三种基因型。
33.第三个snp分子标记位于黑鲷c4基因序列(seq id no:3)的第6070个碱基处,该snp位点的等位基因为a和t,有aa、at和tt三种基因型。
34.二、黑鲷3个snp分子标记不同基因型与生长性状的关联分析
35.从同批培育的4月龄黑鲷群体中随机挑选158尾作为关联分析群体,并测量每一尾鱼的体质量、全长、体长和体高等生长数据。由于该群体为同一批繁殖、同池养殖,且采样时间一致,因此在建立分析模型时不考虑时间、环境及人工饲养条件的差异。
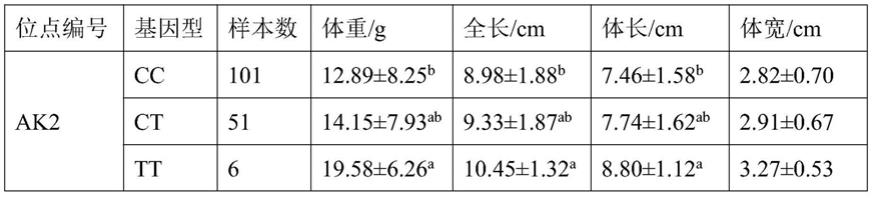
36.利用maldi-tof ms方法对ak2基因上突变位点在158尾黑鲷个体进行基因分型后,采用单因素方差分析法分析不同基因型对体质量、全长、体长和体高进行关联分析。结果见表1,可以看出,tt基因型个体在体质量、全长、体长和体高上的平均值均高于tc型和cc型个体的均值,且tt基因型个体在体质量、全长和体长上显著高于cc基因型个体(p《0.05)。
37.利用maldi-tof ms方法对cyp3a40基因上突变位点在158尾黑鲷个体进行基因分型后,采用单因素方差分析法分析不同基因型对体质量、全长、体长和体高进行关联分析。结果见表1,可以看出,aa基因型个体在体质量、全长、体长和体高上的平均值均高于ac型和cc型个体的均值,且aa基因型个体在体质量、全长和体长上显著高于cc基因型个体(p《0.05)。
38.利用maldi-tof ms方法对c4基因上突变位点在158尾黑鲷个体进行基因分型后,采用单因素方差分析法分析不同基因型对体质量、全长、体长和体高进行关联分析。结果见表1,可以看出,tt基因型个体在体质量、全长、体长和体高上的平均值均高于at型和aa型个体的均值,且tt基因型个体在体质量、全长、体长和体高上显著高于aa基因型个体(p《0.05)。
39.表1黑鲷3个snp位点不同基因型与生长形状的关联分析
[0040][0041][0042]
注:表中数值为平均值
±
标准误差,同一基因的同一列数值上表字母表示差异显著(p《0.05)。
[0043]
实施例2
[0044]
利用实施例1筛选出的snp分子标记对具有高生长率的黑鲷亲本进行筛选,包括如下步骤:
[0045]
(1)剪取待测黑鲷尾鳍样本,提取dna;
[0046]
(2)以提取的dna为模板,利用引物对进行pcr扩增,得到初次pcr扩增产物;
[0047]
其中,用于检测位于adenylate kinase 2基因的snp分子标记的引物对为:
[0048]
f1:3
’‑
acgttggatgtcgtggagctcatagagaag-5’(seq id no.4)
[0049]
r1:3
’‑
acgttggatgtctccgcttgtttgactgtg-5’(seq id no.5)
[0050]
延伸引物1:3
’‑
agaagaacttggacacgtc-5’(seq id no.6);
[0051]
用于检测位于cytochrome p4503a基因的snp分子标记的引物对为:
[0052]
f2:3
’‑
acgttggatgtaccctttggaaccacaagc-5’(seq id no.7)
[0053]
r2:3
’‑
acgttggatgtttgtaccccatccttccac-5’(seq id no.8)
[0054]
延伸引物2:3
’‑
cacaagcccatttatctc-5’(seq id no.9);
[0055]
用于检测位于complement component 4基因的snp分子标记的引物对为:
[0056]
f3:3
’‑
acgttggatgatgctgttgccattacagcc-5’(seq id no.10)
[0057]
r3:3
’‑
acgttggatgaagttgtccccaggttgatg-5’(seq id no.11)
[0058]
延伸引物3:3
’‑
cagcctattgcctttcagt-5’(seq id no.12)。
[0059]
pcr扩增的反应体系见表2:
[0060]
表2
[0061][0062][0063]
pcr扩增的反应程序为:94℃预变性3min;94℃变性30s,56℃退火25s, 72℃延伸30s,共40个循环;72℃延伸3min。
[0064]
(3)采用sap(虾碱性硫酸酶)对pcr扩增产物进行碱性磷酸酶处理;
[0065]
碱性磷酸酶处理的sap反应液配方见表3:
[0066]
表3
[0067]
组分浓度体积(1rxn/μl)供应商sapbuffer10x0.17thermofishersapenzyme1.7u/μl0.3thermofisherwater/1.53生工生物pcr产物/5 totalvolume 7 [0068]
sap程序:37℃40min;85℃5min;4℃保存。
[0069]
(4)以处理后的pcr扩增产物为模板,通过maldi-tof ms方法,使用延伸引物延伸一个碱基,不同基因型黑鲷个体的dna上对应的snp位点具有不同的碱基类型,因此,在延伸
引物的引导下,不同基因型延伸扩增出不同的碱基,而后用瞬时纳秒(10-9
s)强激光激发,在一非电场漂移区内按照其质荷比率得以分离,根据延伸碱基的质量不同在真空小管中飞行到达检测器时间不同来区分不同基因型,最后根据质谱图来确定待测样本的基因型,位于adenylate kinase 2基因的snp分子标记的tt基因型质谱图如图1所示;位于cytochrome p450 3a基因的snp分子标记的aa基因型质谱图如图2所示;位于complement component4基因的snp分子标记的tt基因型质谱图如图3所示。
[0070]
步骤(4)中,引物延伸的反应体系见表4:
[0071]
表4
[0072]
组分浓度体积(1rxn/μl)供应商iplexbuffer10x0.2sequenomiplexterminationmix10x0.2sequenomiplexenzyme2u/μl0.041sequenomsapenzyme1.70.3thermofisher延伸引物10μm0.804 water/0.755 纯化pcr产物/7 totalvolume 9 [0073]
延伸条件为:94℃预变性30s;94℃变性5s,52℃退火5s,80℃延伸5s,共 40个循环;72℃延伸3min。
[0074]
以上实施例仅为介绍本发明的优选案例,对于本领域技术人员来说,在不背离本发明精神的范围内所进行的任何显而易见的变化和改进,都应被视为本发明的一部分。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1