RNA介导的染色质构象捕获方法
rna介导的染色质构象捕获方法
技术领域
1.本发明涉及染色质构象捕获领域,特别是涉及一种rna介导的染色质构象捕获方法。
背景技术:
2.在哺乳动物中,rna、蛋白质和线性的dna在细胞核内有机地高度折叠组装,以确保基因的精确转录与调控,从而使得细胞功能正常执行。研究发现许多非编码rna可以直接作用于染色质,并富集在细胞核亚结构中。另外,有研究结果提示非编码rna在染色质空间结构和基因表达调控中可能发挥重要作用。
3.然而目前探索rna在核组织结构和基因组功能上的调控作用的方法,如chirp(chromatin isolation by rna purification)和chart(capture hybridization analysis of rna targets)等技术需要采用有生物素标记的目标rna的反义链,需要进行反义链合成和hplc纯化,成本高,且只是检查rna的靶标dna位点,无法知道这些dna靶标位点是否是以组合的形式执行功能或如何组合进而执行功能。
4.因此,采用传统的方法不仅不简便,而且无法获取rna介导的染色质空间构象信息。
技术实现要素:
5.基于此,有必要提供一种简便且有效地获取rna介导的染色质空间构象信息的rna介导的染色质构象捕获方法。
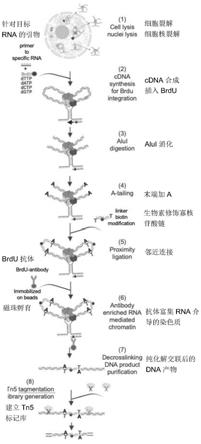
6.一种rna介导的染色质构象捕获方法,包括以下步骤:
7.对结合在染色质上的目标rna进行反转录,其中,在反转录所需的dntp中掺入brdu;
8.对染色质进行邻近连接;
9.采用带有brdu抗体的磁珠富集邻近连接后的染色质;
10.对染色质进行解交联,并纯化dna;及
11.对纯化后的dna进行测序,并进行数据分析,得到目标rna介导的染色质构象。
12.上述rna介导的染色质构象捕获方法,巧妙地利用反转录将brdu引入靶标位点,结合brdu抗体就能够简便有效地对靶标位点进行富集,并且能够广泛适用于rna介导的染色质构象的捕获中,不需要针对特定rna设计特定的带有标记的探针,极大地降低了成本。此外,通过将邻近连接技术引入rna介导的染色质构象捕获领域,将在同一染色质复合物中与rna密切相关的dna位点进行连接,可以获得目标rna介导的dna位点的交互信息和互作网络。
13.在其中一个实施例中,对染色质进行邻近连接的步骤包括:采用带有生物素标记的寡核苷酸片段对染色质进行邻近连接。
14.在其中一个实施例中,对纯化后的dna进行测序之前,还包括以下步骤:采用带有
链霉素标记的磁珠对带有生物素标记的纯化后的dna进行富集。
15.在其中一个实施例中,上述染色质的dna进行了3’末端加a,上述寡核苷酸片段的3’末端带有至少一个t。
16.在其中一个实施例中,对结合在染色质上的目标rna进行反转录之前,还包括步骤:将经过细胞核裂解的细胞的染色质进行片段化。
17.在其中一个实施例中,采用限制性核酸内切酶将经过细胞核裂解的细胞的染色质进行片段化,其中,上述限制性核酸内切酶包括aluⅰ、识别序列为4bp的限制性核酸内切酶或识别序列为6bp的限制性核酸内切酶。
18.在其中一个实施例中,将经过细胞核裂解的细胞的染色质进行片段化之前,还包括步骤:采用交联剂对细胞进行交联;其中,交联剂包括甲醛和egs的至少一种。
19.在其中一个实施例中,在采用带有brdu抗体的磁珠富集邻近连接后的染色质之前,还包括步骤:采用超声将邻近连接后的染色质进行破碎。
20.在其中一个实施例中,采用二代测序、三代测序或单分子测序对所述纯化后的dna进行测序。
21.在其中一个实施例中,对纯化后的dna进行测序的步骤包括:对纯化后的dna进行双端测序。
22.在其中一个实施例中,对纯化后的dna进行测序的步骤包括:采用转座酶对纯化后的dna进行切割,并引入测序的适配体。
23.一种药物靶点筛选方法,该方法包括采用上述任一实施例中的rna介导的染色质构象捕获方法筛选药物靶点,该药物靶点包括rna及/或与rna互作的染色质靶点。
24.一种药物设计方法,该方法包括采用上述任一实施例中的rna介导的染色质构象捕获方法设计药物。
附图说明
25.图1为一实施方式的rna介导的染色质构象捕获方法中反转录引物设计位置示意图;
26.图2为另一实施方式的rna介导的染色质构象捕获方法的流程示意图;
27.图3中c图为图2所示的rna介导的染色质构象捕获方法捕获得到的外源rna介导的在宿主体内的外源染色质自身的相互作用示意图;d图为c图的局部放大图;
28.图4中e图为图2所示的rna介导的染色质构象捕获方法捕获得到的外源rna介导的在宿主体内的外源染色质与宿主染色质的相互作用示意图;f图为本发明的rna介导的染色质构象捕获方法捕获得到的外源rna介导的宿主染色质之间的相互作用示意图;
29.图5为与鼻咽癌密切相关的eb病毒染色质示意图;其上标注为本发明的rna介导的染色质构象捕获方法捕获得到的eb病毒产生的rna介导的eb病毒染色质自身及与宿主染色质之间的相互作用示意;
30.图6为图5对应的eb病毒染色质的线性展开示意图;
31.图7为实施例4的rna介导的染色质构象捕获方法捕获得到的eb病毒产生的rna介导病毒-宿主之间的染色质远程交互,左侧虚线框中示意交互频率高的区域;
32.图8为实施例4的rna介导的染色质构象捕获方法捕获得到的eb病毒产生的rna介
导的病毒内染色质间的交互、病毒-宿主之间的染色质远程交互以及宿主内染色质之间的远程交互的频率统计图;
33.其中,图1~图8中,“e to e”或“e-e”表示外源eb病毒染色质间的相互作用;“e to h”或“e-h”表示外源eb病毒染色质与宿主染色质间的相互作用;“h to h”或“h-h”表示宿主染色质间的相互作用。
具体实施方式
34.为使本发明的上述目的、特征和优点能够更加明显易懂,下面对本发明的具体实施方式做详细的说明。在下面的描述中阐述了很多具体细节以便于充分理解本发明。但是本发明能够以很多不同于在此描述的其它方式来实施,本领域技术人员可以在不违背本发明内涵的情况下做类似改进,因此本发明不受下面公开的具体实施例的限制。
35.除非另有定义,本文所使用的所有的技术和科学术语与属于本发明的技术领域的技术人员通常理解的含义相同。本文中在本发明的说明书中所使用的术语只是为了描述具体的实施例的目的,不是旨在限制本发明。本文所使用的术语“和/或”包括一个或多个相关的所列项目的任意的和所有的组合。
36.本文所述的“dntp”是指脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate),n是指含氮碱基,代表变量指代a、t、g和c中的一种,在生物dna合成中,以及各种pcr(rt-pcr(reverse transcription pcr)、real-time pcr)中作为原料作用。所述的“brdu”是指溴化去氧尿苷(5-bromo-2'-deoxyuridine),是胸腺嘧啶的类似物,于合成dna时取代胸腺嘧啶。所述的“egs”是指乙二醇双[琥珀酰亚胺琥珀酸酯],是一种交联剂,能够将蛋白交联到dna上并捕获蛋白间相互作用。所述的“邻近连接”是指采用邻近连接技术(proximity ligation assay,pla)对在空间上距离很近的染色质进行连接,以检测染色质间的相互作用。所述的“二代测序”是指next generation sequencing(ngs),又称为高通量测序或深度测序,一次可对几十万到几百万条dna或rna片段进行序列测定和分析。所述的“双端测序”是指在构建待测dna或rna文库时在两端的接头上都加上测序引物结合位点,在第一轮测序完成后,去除第一轮测序的模板链,用对读测序模块(paired-end module)引导互补链在原位置再生和扩增,以达到第二轮测序所用的模板量,进行第二轮互补链的合成测序。所述的“药物”包括在体内或体外提供生理和/或药理作用的任何药剂、化合物、组合物或混合物;所述“药物”在体内产生生理和/或药理作用的范围没有特别限制,可以为全身效果,也可以只在局部产生效果。所述的“molecule grade water”是指不含核酸酶、蛋白酶和内毒素的可用于分子实验的水。所述的“w/v”是指质量与体积之比。所述的“v/v”是指体积之比。
[0037]
本发明聚焦开发新型的染色质构象捕获技术,对rna介导的染色质空间构象与基因组功能进行研究,为揭示rna功能提供新的工具和依据。具体地:
[0038]
本发明一实施方式提供了一种rna介导的染色质构象捕获方法,包括步骤s11、步骤s12、步骤s13、步骤s14和步骤s15,具体地:
[0039]
步骤s11:对结合在染色质上的目标rna进行反转录,其中,在反转录所需的dntp中掺入brdu。
[0040]
具体地,针对目标rna设计引物后对目标rna进行原位反转录,其中设计引物的规
则如图1所示,扩增产物需要覆盖外显子区域。
[0041]
在其中一个实施例中,dttp与brdu的摩尔量的比为1:1。在一些可选的实施例中,dttp与brdu的摩尔量的比为1:(0.2~5)。在一个可选的具体示例中,dttp与brdu的摩尔量的比为5:1、2:1、1:2、1:3、1:4或1:5。可以理解的是,brdu掺入量的选择标准为能够实现在反转录产生的cdna中替代一部分dttp即可。
[0042]
在其中一个实施例中,对结合在染色质上的目标rna进行反转录之前,还包括步骤:将经过细胞核裂解的细胞的染色质进行片段化。其中,细胞可来源于培养的细胞也可来源于组织。在其他一些实施例中,可以在对染色质进行邻近连接之前对染色质片段化,如图2所示。
[0043]
在其中一个实施例中,采用限制性核酸内切酶将经过细胞核裂解的细胞的染色质进行片段化,其中,上述限制性核酸内切酶包括但不限于aluⅰ、识别序列为4bp的限制性核酸内切酶或识别序列为6bp的限制性核酸内切酶。可以理解的是,还可以采用其他方式对染色质进行片段化,例如超声破碎。
[0044]
在其中一个实施例中,将经过细胞核裂解的细胞的染色质进行片段化之前,还包括步骤:采用交联剂对细胞进行交联;其中,交联剂包括甲醛和egs的至少一种。
[0045]
在一些实施例中,交联剂中甲醛的浓度为0.5%(w/v)~1.5%(w/v)。进一步地,在其他一些实施例中,交联剂中甲醛的浓度为0.7%(w/v)~1.3%(w/v)。更进一步地,在一些可选的具体示例中,交联剂中甲醛的浓度为0.8%(w/v)、0.9%(w/v)、1.0%(w/v)、1.1%(w/v)或1.2%(w/v)。
[0046]
在一些实施例中,交联剂中egs的浓度为1.5mmol/l~2.5mmol/l。进一步地,在其他一些实施例中,交联剂中甲醛的浓度为1.7mmol/l~2.3mmol/l。更进一步地,在一些可选的具体示例中,交联剂中甲醛的浓度为1.8mmol/l、1.9mmol/l、2.0mmol/l、2.1mmol/l或2.2mmol/l。
[0047]
在一些实施例中,交联剂中的甲醛溶解在水中。在其他一些实施例中,交联剂中的甲醛溶解在缓冲溶液中,例如磷酸缓冲液(pbs)。
[0048]
在一些实施例中,交联剂中的egs溶解在dmso中后再溶解于水中。在其他一些实施例中,交联剂中的egs溶解在dmso中后再溶解于缓冲溶液中,例如磷酸缓冲液(pbs)。
[0049]
具体地,甲醛是一种零长度的交联剂,egs是较长的交联剂,根据实际需要两者可以分开使用也可以结合使用。两者结合使用能够可以有效地将蛋白质-dna复合物锁在一起,即使是短暂的相互作用复合物也能被捕获并稳定下来进行分析。
[0050]
步骤s12:对染色质进行邻近连接。
[0051]
在其中一个实施例中,对染色质进行邻近连接的步骤包括:采用带有生物素标记的寡核苷酸片段对染色质进行邻近连接。
[0052]
在其中一个实施例中,上述染色质的dna进行了3’末端加a,上述寡核苷酸片段的3’末端带有至少一个t。可以理解的是,采用末端带有a尾的dna与末端带有t的寡核苷酸片段配对的方式能够提高邻近连接的效率。
[0053]
步骤s13:采用带有brdu抗体的磁珠富集邻近连接后的染色质。
[0054]
具体地,采用带有brdu抗体的磁珠富集邻近连接后的染色质,能够将含有brdu的染色质锚定到带有brdu抗体的磁珠上,即达到将与目标rna有相互作用的染色质筛选出来
的目的,后续洗脱步骤能够除去非目标片段。
[0055]
在其中一个实施例中,在采用带有brdu抗体的磁珠富集邻近连接后的染色质之前,还包括步骤:采用超声将邻近连接后的染色质进行破碎。在一些实施例中,采用超声仪对染色质进行破碎,所用的超声仪是sonics的超声仪,包括隔音箱(630-0451)和2mm探头(630-0423);或diagenode的超声仪bioruptor(b01060010);或covaris的超声仪covaris(2764)。可以理解的是,还可以采用其他超声仪对染色质进行破碎,能使破碎产物达到目标需求即可。
[0056]
在其中一个实施例中,在采用带有brdu抗体的磁珠富集邻近连接后的染色质之后,还包括步骤:去除与带有brdu抗体的磁珠非特异性结合的染色质。具体地,通过对磁珠-brdu抗体-brdu-染色质进行高盐、低盐和中和缓冲液的洗脱,采用磁力架吸附磁珠-brdu抗体-brdu-染色质,再进行换液,从而将非特异性结合的染色质去除。具体地,通过搭建“磁珠-brdu抗体-brdu-染色质”使得原先在溶液中难以操作的实验转化为固相操作,可以随意对缓冲液或反应体系进行置换,操作简单方便。
[0057]
步骤s14:对染色质进行解交联,并纯化dna。
[0058]
在其中一个实施例中,将染色质置于60℃~70℃中孵育4h~20h解交联。
[0059]
在另一个实施例中,将染色质置于90℃~98℃中孵育10min~20min解交联。
[0060]
在其中一个实施例中,采用蛋白酶k消化染色质中的蛋白质成分。
[0061]
步骤s15:对纯化后的dna进行测序,并进行数据分析,得到目标rna介导的染色质构象。
[0062]
在其中一个实施例中,采用二代测序、三代测序或单分子测序对所述纯化后的dna进行测序。可以理解的是,在其他一些实施例中,还可以采用其他方法对纯化后的dna进行测序,能够获得靶标dna序列信息的测序方法均可以采用。
[0063]
在其中一个实施例中,对纯化后的dna进行测序的步骤包括:对纯化后的dna进行双端测序。
[0064]
在其中一个实施例中,对纯化后的dna进行测序的步骤包括:采用转座酶对纯化后的dna进行切割,并引入测序的适配体。
[0065]
在其中一个实施例中,对纯化后的dna进行测序之前,还包括以下步骤:采用带有链霉素标记的磁珠对带有生物素标记的纯化后的dna进行富集。可以理解的是,通过带有链霉素标记的磁珠对带有生物素标记的纯化后的dna进行富集,即可获得带有邻近连接接头部分的dna,能够减轻测序的压力,避免大量非目标片段进入测序增大测序工作量以及后续数据分析的工作量。
[0066]
在其中一个实施例中,将测序获得的原始数据跑pipeline,具体pipeline可选择chia-pipe,并比对到基因组序列,最终获得交互的数据。
[0067]
本发明研究发现,将brdu和邻近连接引入rna介导的染色质空间构象捕获方法中,可以简便且有效地获取rna介导的染色质空间构象信息。
[0068]
本发明一实施方式还提供了一种药物靶点筛选方法,该方法包括采用上述任一实施例中的rna介导的染色质构象捕获方法筛选药物靶点,该药物靶点包括rna及/或与rna互作的染色质靶点。
[0069]
具体地,采用上述任一实施例中的rna介导的染色质构象捕获方法能够捕获得与
目标rna互作的染色质区域,从而能够有选择性地将某些互作靶点及/或该目标rna作为药物靶点进行药物干预,提高了药物干预的精确性和特异性。
[0070]
本发明一实施方式还提供了一种药物设计方法,该方法包括采用上述任一实施例中的rna介导的染色质构象捕获方法设计药物。
[0071]
具体地,该药物设计方法包括前期药物开发及/或后期药物验证。在前期药物开发阶段,采用上述任一实施例中的rna介导的染色质构象捕获方法可以获知目标rna与染色质的互作网络,进而能够根据该互作网络设计调控该互作网络的药物,例如:调控该目标rna的染色质靶点的药物、调控该目标rna的药物或调控该目标rna与染色质互作的药物。在后期药物验证阶段,可以对经待验证的药物处理过的样本与未经该药物处理过的样本采用上述任一实施例中的rna介导的染色质构象捕获方法进行染色质构象捕获,根据捕获结果判断药物干预的效果。
[0072]
上述rna介导的染色质构象捕获方法,巧妙地利用反转录将brdu引入靶标位点,结合brdu抗体就能够简便有效地对靶标位点进行富集,并且能够广泛适用于rna介导的染色质构象的捕获中,不需要每次针对特定rna设计和合成特定的带有标记的探针,只需要合成引物对目标rna进行反转录,大大降低了成本、减少了细胞或组织量、简化实验步骤和操作流程。此外,通过将邻近连接技术引入rna介导的染色质构象捕获领域,将在同一染色质复合物中与rna密切相关的dna位点进行连接,可以获得目标rna介导的dna位点的交互信息和互作网络,使得能够对rna介导的染色质空间构象与基因组功能进行研究,为宿主内源性rna的功能研究以及病原体病毒等外源性rna的功能研究提供了全新的手段,有望为新冠病毒产生的rna对宿主人类的致病机制提供全新的技术方法。
[0073]
具体实施例
[0074]
以下结合具体实施例进行详细说明。以下实施例如未特殊说明,则不包括除不可避免的杂质外的其他组分。实施例中采用试剂和仪器如非特别说明,均为本领域常规选择。实施例中未注明具体条件的实验方法,按照常规条件,例如文献、书本中所述的条件或者生产厂家推荐的方法实现。
[0075]
实施例1
[0076]
1.双交联固定细胞(细胞系:gm12878)
[0077]
先用1%(w/v)的甲醛水溶液在25℃条件下对细胞进行固定20min,后加入甘氨酸至终浓度为0.125mol/l,于25℃反应10min以终止甲醛交联反应。然后用dpbs洗涤2次,再加入2mmol/l egs溶液在25℃条件下反应40min进行固定,再加入甘氨酸至终浓度为0.125mol/l,在25℃条件下反应10min以终止egs交联反应,然后用dpbs洗涤2次,离心后去除上清,于-80℃保存备用。
[0078]
2.裂解细胞
[0079]
从-80℃中取出双交联固定后的细胞,在冰水混合物中解冻,用dpbs重悬后在25℃以2000rpm离心5min,去除上清,将细胞沉淀重悬在细胞裂解液(0.1%(w/v)sds fa裂解液)中,用移液枪吹打混合后,于25℃在摇床上旋转20min。
[0080]
3.裂解细胞核
[0081]
加入sds至终浓度为1%(w/v),混匀,于37℃放置20min,后于25℃在5000rpm条件下离心5min,去除上清。重复上述细胞核裂解步骤直至核裂解完全(在显微镜下观察到细胞
膜已完全脱离或细胞核明显发黑时说明核裂解完全)。然后用dpbs洗涤一次,于25℃在3000rpm条件下离心5min,去除上清。细胞核沉淀重悬在终浓度为1%(v/v)的tritonx-100中,于37℃放置30min,以终止多余的sds的反应。
[0082]
4.切割细胞质
[0083]
采用限制性内切酶alui,对裂解好的细胞核进行酶切,于37℃反应2小时,然后用tritonx-100洗涤2次,于25℃在5000rpm条件下离心5min,去除上清。
[0084]
5.反转录(采用反转录试剂盒,货号:18080051;厂家:invitrogen)
[0085]
1)先用1
×
rt缓冲液(采用10
×
rt缓冲液稀释得到)洗涤细胞核沉淀一次,然后将细胞核重悬在rna antisense mixture中(如表1所示),混匀,于65℃放置5min,后放置冰上至少1min。其中,目标rna为malat1,反转录引物的核苷酸序列如表5中malat1-1~malat1-28(seq id no:1~28)所示,每个反应体系中加入所有引物。
[0086]
表1
[0087]
rna antisense mixture组分终浓度体积(μl)反转录引物1.0μmol/l5牛血清白蛋白(bsa)20mg/ml5细胞核样本和ddh2o 90
[0088]
2)然后加入cdna synthesis mixture(如表2所示),于25℃10min,42℃30min,然后于25℃以5000rpm离心10min,除去上清。
[0089]
表2
[0090]
cdna synthesis mixture组分体积(μl)10
×
rt缓冲液2025mmol/l mgcl2200.1mol/l dtt1010mmol/l datp1.2510mmol/l dgtp1.2510mmol/l dctp1.2510mmol/l dttp0.62510mmol/l brdutp0.625ddh2o35rnaseout(40u/μl)5superscript iii rt(200u/μl)5
[0091]
6.末端加尾
[0092]
通过dna polymerase i(large(klenow)fragment(3’—》5’exo
—
))对染色质dna末端添加poly(a)。
[0093]
7.邻近连接
[0094]
将接头与带有poly(a)的染色质dna进行邻近连接,接头是带有生物素标记的寡核苷酸链linker,其核苷酸序列seq id no:42为:5
’‑
acgcgatggctactctgact-3’。
[0095]
8.超声破碎
[0096]
通过超声破碎仪(sonics;隔音箱:630-0451;2mm探头:630-0423)对邻近连接后的
染色质进行破碎,超声幅度设置为38%,超声持续30s后停止30s,总时间为3min。根据破碎结果再适当调整超声的时间和频率,最终获得dna大小片段在2kb左右的染色质。
[0097]
9.染色质免疫共沉淀
[0098]
将超声后的染色质与磁珠进行4℃孵育2h,去除一些非特异结合到磁珠上的非特异性物质。将brdu抗体与磁珠结合,然后与染色质进行4℃孵育16h,从而将含有brdu的染色质锚定到结合brdu抗体的磁珠上。
[0099]
10.去除非特异性染色质
[0100]
通过对磁珠-brdu抗体-brdu-染色质进行高盐、低盐和中和缓冲液的洗脱,采用磁力架吸附磁珠-brdu抗体-brdu-染色质,后进行换液,从而将非特异性结合的染色质去除。具体如下:
[0101]
1)采用0.1%sds fa溶液对磁珠-brdu抗体-brdu-染色质洗涤3次,每次在摇床上旋转5min;
[0102]
2)采用0.1%sds fa/350mmol/l nacl溶液(具体溶液组分和配比如表3所示)对磁珠-brdu抗体-brdu-染色质洗涤1次,在摇床上旋转5min;
[0103]
表3
[0104]
组分(原液)终浓度配制10ml目标溶液所需体积(ml)1mol/l tris-hcl(ph=7.0)50mmol/l0.51mol/l tris-hcl(ph=8.0)50mmol/l0.55mol/l nacl350mmol/l0.70.5mol/l edta1mmol/l0.0210%(w/v)sds0.1%(w/v)0.1molecular grade water 8.18
[0105]
3)采用te缓冲液(具体溶液组分和配比如表4所示)对磁珠-brdu抗体-brdu-染色质洗涤1次,在摇床上旋转5min。
[0106]
表4
[0107]
组分(原液)终浓度配制50ml目标溶液所需体积1mol/l tris-hcl(ph=8.0)10mmol/l500μl0.5mol/l edta0.1mmol/l10μlmolecular grade water 49.49ml
[0108]
11.解交联并纯化dna
[0109]
通过在65℃中孵育6h对染色质进行解交联,然后用蛋白酶k消化其中的蛋白成分,采用dna纯化试剂盒获得dna产物。
[0110]
12.切割dna
[0111]
采用转座酶tn5对邻近连接后的dna进行切割并加入测序的适配体。
[0112]
13.建库测序
[0113]
采用链霉素磁珠对转座酶切割后的dna进行富集,获得含有接头的邻近连接后的dna部分,然后采用atac-seq的引物(如表6所示,seq id no:43~67)进行pcr扩增,在pcr产物中筛选出300bp~600bp的片段进行双端测序。
[0114]
14.数据处理
[0115]
将测序获得的原始数据跑pipeline(chia-pipe),比对到人类基因组序列hg38,最终获得交互的数据。结果如表7所示。
[0116]
实施例2
[0117]
本实施例与实施例1的步骤大致相同,其不同在于本实施例的步骤5中目标rna为vsno-rna1,反转录引物的核苷酸序列如表5中vsno-rna1-r(seq id no:41)所示。
[0118]
实施例3
[0119]
本实施例与实施例1的步骤大致相同,其不同在于本实施例的步骤5中目标rna为mir-bart,反转录引物的核苷酸序列如表5中mir-bart-1~mir-bart-12(seq id no:29~40)所示。
[0120]
表5
[0121]
[0122][0123]
表6
[0124]
核苷酸序列序号pcr引物名称核苷酸序列(5'-3')seq id no:43ad1_nomxaatgatacggcgaccaccgagatctacactcgtcggcagcgtcagatgtgseq id no:44ad2.1caagcagaagacggcatacgagattcgccttagtctcgtgggctcggagatgtseq id no:45ad2.2caagcagaagacggcatacgagatctagtacggtctcgtgggctcggagatgtseq id no:46ad2.3caagcagaagacggcatacgagatttctgcctgtctcgtgggctcggagatgtseq id no:47ad2.4caagcagaagacggcatacgagatgctcaggagtctcgtgggctcggagatgtseq id no:48ad2.5caagcagaagacggcatacgagataggagtccgtctcgtgggctcggagatgtseq id no:49ad2.6caagcagaagacggcatacgagatcatgcctagtctcgtgggctcggagatgtseq id no:50ad2.7caagcagaagacggcatacgagatgtagagaggtctcgtgggctcggagatgtseq id no:51ad2.8caagcagaagacggcatacgagatcctctctggtctcgtgggctcggagatgtseq id no:52ad2.9caagcagaagacggcatacgagatagcgtagcgtctcgtgggctcggagatgtseq id no:53ad2.10caagcagaagacggcatacgagatcagcctcggtctcgtgggctcggagatgtseq id no:54ad2.11caagcagaagacggcatacgagattgcctcttgtctcgtgggctcggagatgtseq id no:55ad2.12caagcagaagacggcatacgagattcctctacgtctcgtgggctcggagatgtseq id no:56ad2.13caagcagaagacggcatacgagatatcacgacgtctcgtgggctcggagatgtseq id no:57ad2.14caagcagaagacggcatacgagatacagtggtgtctcgtgggctcggagatgtseq id no:58ad2.15caagcagaagacggcatacgagatcagatccagtctcgtgggctcggagatgtseq id no:59ad2.16caagcagaagacggcatacgagatacaaacgggtctcgtgggctcggagatgtseq id no:60ad2.17caagcagaagacggcatacgagatacccagcagtctcgtgggctcggagatgtseq id no:61ad2.18caagcagaagacggcatacgagataacccctcgtctcgtgggctcggagatgtseq id no:62ad2.19caagcagaagacggcatacgagatcccaacctgtctcgtgggctcggagatgtseq id no:63ad2.20caagcagaagacggcatacgagatcaccacacgtctcgtgggctcggagatgtseq id no:64ad2.21caagcagaagacggcatacgagatgaaacccagtctcgtgggctcggagatgtseq id no:65ad2.22caagcagaagacggcatacgagattgtgaccagtctcgtgggctcggagatgtseq id no:66ad2.23caagcagaagacggcatacgagatagggtcaagtctcgtgggctcggagatgtseq id no:67ad2.24caagcagaagacggcatacgagataggagtgggtctcgtgggctcggagatgt
[0125]
通过对测序数据进行分析,本发明的目标rna介导的染色质构象捕获技术(rna mediated chromatin dna-dna interactions,简称rdd)可以高效地获得rna介导的染色质交互数据。从表3中可以看出,实施例1~3的三种目标rna分别介导的染色质构象捕获实验中,获得的测序数据中含有连接接头的(fraction_read_pairs_with_linker)均在90%以上,说明连接效率高,获得大量的染色质交互数据,使用rdd进行的捕获是成功的。而且根据染色体内的交互数据是染色体间的2倍~3倍(ratio_of_intra/inter_pet),提示连接主要发生在染色体内,这与目前对染色体交互的实际情况的认识相符合,说明使用rdd进行的捕获获取的数据是可信的。此外,从“pets_number_2”~“pets_number》10”的数据可以看出,交互的数据在三个目标rna中均比较丰富,同时也捕获到高频交互(大于10)的数据,说明rdd的对染色质相互作用的捕获能力比较强。
[0126]
表7
[0127]
lib typerddrddrddcell_typegm12878gm12878gm12878factormalat1v-snorna1mir-barttotal_read_pairs34,641,68063,174,30153,807,867read_pairs_with_linker31,823,37257,161,93148,622,839fraction_read_pairs_with_linker92.0%90.0%90.0%one_tag22,201,48740,934,02535,241,937pet9,058,26514,952,44112,129,450uniquely_mapped_pet7,173,50211,538,6649,179,566non-redundant_pet1,289,0813,352,5852,427,430redundancy82%71%74%non-redundant_tag14,688,86640,077,33731,539,430peak161,160328,870266,140self-ligation_pet534,2141,482,4791,047,929inter-ligation_pet754,8671,870,1061,379,501intra-chr_pet552,8141,305,895965,354inter-chr_pet202,053564,211414,147ratio_of_intra/inter_pet2.742.312.33singleton607,6361,612,8671,178,122intra-chr_singleton439,6981,115,141816,460inter-chr_singleton167,938497,726361,662pet_cluster60,758111,29985,188ratio_of_intra/inter_cluster3.222.752.69intra-chr_pet_cluster46,37781,58762,111pets_number_236,77567,93450,773pets_number_35,5808,8056,984pets_number_41,8602,6352,261pets_number_59231,1841,029pets_number_6508518510pets_number_7317242243pets_number_8163119120pets_number_91044775pets_number_10644232pets_number》10836184
[0128]
实施例4
[0129]
通过rdd技术挖掘病毒rna介导宿主染色质dna-dna的远程交互
[0130]
通过本发明的rdd技术,发现鼻咽癌病毒产生的rna会介导病毒内染色质间的交互;同时介导病毒-宿主之间的染色质远程交互,而且也会介导宿主内染色质之间的远程交互,如图5~图8所示。图5为与鼻咽癌密切相关的eb病毒染色质示意图;其上标注为本发明
的rna介导的染色质构象捕获方法捕获得到的eb病毒产生的rna介导的eb病毒染色质自身及与宿主染色质之间的相互作用示意。图6为图5对应的eb病毒染色质的线性展开示意图。图7为本发明的rna介导的染色质构象捕获方法捕获得到的eb病毒产生的rna介导病毒-宿主之间的染色质远程交互,左侧虚线框中示意交互频率高的区域。图8为本发明的rna介导的染色质构象捕获方法捕获得到的鼻咽癌病毒产生的rna介导的病毒内染色质间的交互、病毒-宿主之间的染色质远程交互以及宿主内染色质之间的远程交互的频率统计图。以上结果提示病毒对宿主的致病中rna起到关键性的作用,通过调控远程交互作用,改变或影响三维结构,从而调控靶基因的表达,导致疾病的发生。
[0131]
以上实施例的各技术特征可以进行任意的组合,为使描述简洁,未对上述实施例中的各技术特征所有可能的组合都进行描述,然而,只要这些技术特征的组合不存在矛盾,都应当认为是本说明书记载的范围。
[0132]
以上实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。应当理解的是,本领域技术人员在本发明提供的技术方案的基础上,通过合乎逻辑的分析、推理或有限的试验得到的技术方案,均在本发明所附权利要求的保护范围内。因此,本发明专利的保护范围应以所附权利要求为准,说明书及附图可以用于解释权利要求的内容。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1