一种诺如病毒GI-GIX亚型全基因组捕捉试剂盒及应用的制作方法
本发明涉及诺如病毒检测领域,特别涉及一种诺如病毒gi-gix亚型全基因组捕捉试剂盒及应用。
背景技术:
1、诺如病毒(norovirus,nv)是一种带有包膜的单股正链rna病毒,属于人类杯状病毒科(caliciviridae),是成年人腹泻的第一大病原和≤5岁婴幼儿腹泻的第二大病原。它在环境中存活能力强,可在物体表面存活长达2周,在水中甚至可存活2个月以上。这一特性增加了疫情爆发的风险,尤其是通过粪口途径传播。临床上,nv感染通常导致轻度和自限性的疾病,表现为急性腹泻、发热和呕吐等症状,免疫脆弱人群症状较重,严重者可导致死亡。据估计,全球约有20%的急性胃肠炎病例与诺如病毒感染相关。疫情通常在人口密集的环境,如邮轮、学校、医院和养老院等地方爆发,但也可能在家庭和社区中传播。因此,nv对全球卫生和食品安全构成了重大挑战。
2、诺如病毒的基因组大小约为7.5至7.7kb,包括三个可读框(open reading frame,orf)。其中,orf2编码主要的衣壳蛋白(vp1),该蛋白决定了病毒的免疫原性和抗原性,也成为其分型和分类的关键依据。vp1蛋白由高度保守的s区和变异性较大的p区组成,特别是p2区域,包括免疫识别位点和受体结合位点,使nv表现出了极高的遗传多样性。为了更准确地分类和命名不同的nv毒株,目前采用了双命名系统,同时结合rdrp区域(如gⅱ.p4)和vp1区域(如gⅱ.4)对基因型进行标识。已经发现,nv至少分为10个基因组(gⅰ至gⅹ)和8个p组。其中,gⅰ包括了9个不同的基因型(gⅰ.1至gⅰ.9型),而gⅱ则至少包含了22个不同的基因型(gⅱ.1至gⅱ.22型),包含了大部分可感染人类的毒株。值得注意的是,gⅱ.4基因型一直占据主导地位,并且由于抗原漂移和基因重组的作用,每2~4年就会出现新的gⅱ.4变异株以替代既往流行的株型。此外,gⅳ、gⅷ及gⅸ也可感染人类,增加了对免疫系统和抗病毒药物产生抗性的风险。因此,深入了解不同亚型的分布和传播动态对于流行病学研究及预防与控制至关重要。
3、目前市面上针对于诺如病毒的鉴定主要有以下几种方式:
4、1、传统血清学方法:传统的对诺如病毒鉴定主要有分离病毒毒株,进行血清补体结合试验和中和试验,这种方法耗时久,且病毒分离培养有失败的可能,灵敏度不够;
5、2、常规定量pcr方法:主要以诺如病毒vp1为目标区域,根据不同分型基因核酸序列特点设计引物和探针,在特定的反应体系和反应条件下,通过扩增特异性目的片段进行分型,但是定量pcr只能鉴定vp1分型,无法进行更进一步的基因序列分析和rdrp分型且无法鉴定新的分型。比如cn108796128a提供的一种gii.8型诺如病毒基因组靶向引物和扩增方法,其是在保守区域设计了六组靶向引物和两条基因组末端测序引物,但仅针对于gii.8型诺如病毒基因组序列;cn108823331a提供了一种gii.6型诺如病毒基因组靶向引物和扩增方法,设计了仅针对于gii.6型诺如病毒基因组序列,然而不同分型的诺如病毒存在差异且不同引物套装的设计覆盖诺如病毒的不同区域,这就导致了并不能简单地将针对于不同分型的引物合并作为诺如病毒全基因测序的引物,这种方式会出现不同引物之间交叉杂交以及难以覆盖全基因组的问题。
6、3、测序技术:现用检测序列变异的技术是基于pcr结合一代测序检测1到2个基因区域的变异信息,存在低效、监测范围有限的局限,已有免培养的办法多是采用宏转录组的方法,但测序数据中存在大量的宿主的数据,存在大量的数据浪费、高成本和难以检测低丰度的病毒的局限。
7、综上所述,目前市面上缺乏针对于诺如病毒gi-gix亚型全基因组的测序和扩增方案,限制了对诺如病毒的检测和变异监测。
技术实现思路
1、本方案提供了一种诺如病毒gi-gix亚型全基因组捕捉试剂盒及应用,能从血液、拭子、感染组织等环境中提取的核酸样本中进行诺如病毒gi-gix亚型全基因组逆转录与富集扩增,得到的rna产物可用于各种分子生物学实验,包括二代和三代全基因组测序实验,以方便对诺如病毒的检测和变异监测。
2、本方案通过采用特异性引物基于多重pcr技术,对诺如病毒gi-gix亚型目标序列进行靶向富集,结合二代或三代纳米孔测序技术进行实时测序分析,可以研究诺如病毒的进化机制,包括分型检测、基因突变和选择压力等,能够更好地了解其抗性特性。诺如病毒的靶向扩增研究在疫情监测、病原体鉴定、流行病学研究、抗病毒药物研发等领域具有重要意义,有助于更深入地了解和有效应对由诺如病毒引发的疾病。
3、为实现以上目的,第一方面,本方案提供了一种诺如病毒gi-gix亚型全基因组捕捉试剂盒,包括:三组引物池,其中第一引物池内的引物含有引物序列如seq id no.1-seqid no.5所示的靶向引物;第二引物池内的引物含有引物序列如seq id no 4-seq id no.7所示的靶向引物,第三引物池内的引物含有引物序列如seq id no 8-seq id no.17所示的靶向引物。
4、具体的,第一引物池内的靶向引物序列如下表一所示:
5、表一第一引物池的靶向引物
6、 序列编号 引物名称 引物序列 seq id no.1 nv-2-1-f-1 taytatgtyarvtgyktbcarga seq id no.2 nv-2-1-f-3 gargghaagaarggbaaraacaa seq id no.3 nv-2-2-f ggyaagaarcacacwgch seq id no.4 nv-2-2-r-1 tcgacgccatcttcattca seq id no.5 nv-2-1-r-1 ckrggdganactgtraaytcwcc
7、。在本方案的实施例中,seq id no.1-seq id no.3是正向的靶向引物,引导反应中的dna合成或扩增的起始点;seq id no.4-seq id no.5是反向的靶向引物,用于扩增的dna链的反向方向。seq id no.1-seq id no.2的正向的靶向引物同seq id no.5的反向的靶向引物构成一组引物对,seq id no.3的正向的靶向引物同seq id no.4的反向的靶向引物构成一组引物对。
8、具体的,第二引物池内的靶向引物序列如下表二所示:
9、表二第二引物池的靶向引物
10、 序列编号 引物名称 引物序列 seq id no.4 nv-2-2-r-1 tcgacgccatcttcattca seq id no.5 nv-2-1-r-1 ckrggdganactgtraaytcwcc seq id no.6 nv-2-1-f-4 atgytbctsacwggrkcmaa seq id no.7 nv-2-2-f-1 ggmaacacygtcatmtgtgc
11、。
12、在本方案的实施例中,seq id no.6-seq id no.7是正向的靶向引物,引导反应中的dna合成或扩增的起始点;seq id no.4-seq id no.5是反向的靶向引物,用于扩增的dna链的反向方向。seq id no.6的正向的靶向引物同seq id no.5的反向的靶向引物构成一组引物对,seq id no.7的正向的靶向引物同seq id no.4的反向的靶向引物构成一组引物对。
13、具体的,第三引物池内的靶向引物序列如下表三所示:
14、表三第三引物池的靶向引物
15、 序列编号 引物名称 引物序列 seq id no.8 nv-1-1-f gtgaatgaagatggcgtctaac seq id no.9 nv-1-2-f tctaacgacgctwccgytg seq id no.10 nv-3-1-f gggagggcgatcgcaat seq id no.11 nv-3-1-r-1 gcacrgtygagacygtsc seq id no.12 nv-3-1-r-2 rsmhgtccaggagtccaa seq id no.13 nv-3-2-f tgaatgaagatggcgtcga seq id no.14 nv-3-2-r-1 strctwgakggwggvgtsac seq id no.15 nv-3-2-r-2 ctrgargmystrctwgakgg seq id no.16 nv-1-1-r-1 ttgwactcrtcrtactcytcatc seq id no.17 nv-1-2-r-2 tcdtcwggdytyctyttyctrat
16、在本方案的实施例中,seq id no.8-seq id no.10以及seq id no.13是正向的靶向引物,引导反应中的dna合成或扩增的起始点;seq id no.11-seq id no.12以及seq idno.14-seq id no.17是反向的靶向引物,用于扩增的dna链的反向方向。seq id no.8的正向的靶向引物同seq id no.16的反向的靶向引物构成一组引物对,seq id no.9的正向的靶向引物同seq id no.17的反向的靶向引物构成一组引物对,seq id no.10的正向的靶向引物同seq id no.11-seq id no.12的反向的靶向引物构成一组引物对,seq id no.13的正向的靶向引物同seq id no.14-seq id no.15的反向的靶向引物构成一组引物对。
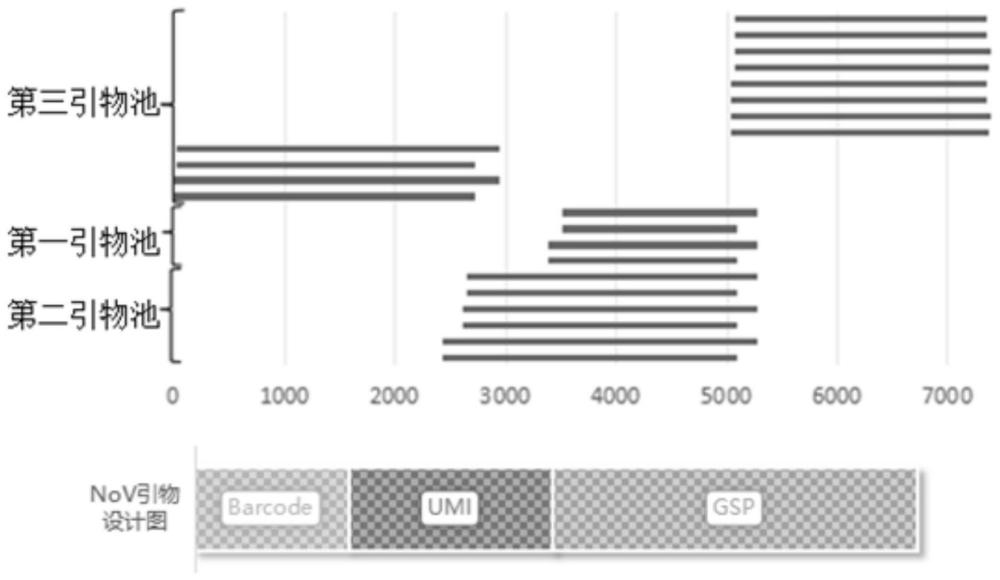
17、如图1所示,需要说明的是,本方案的诺如病毒gi-gix亚型的全基因组试剂盒中第一引物池、第二引物池、第三引物池内的每一引物由靶向引物、umi序列和barcdoe序列排列组成,其中umi序列连接在每一靶向引物的5’端,每一barcdoe序列连接在umi序列的5’端,故第一引物池、第二引物池、以及第三引物池内的引物表示为:“barcode-nx-靶向引物”的形式。其中umi序列含有6-8个n,表示为(n)x,x=6-8,其中barcode序列共有96条序列,每条序列对应于24bp(三代)或8bp(二代)。
18、这样编译的好处在于可以有效降低后续建库过程中连接加barcdoe序列的成本,起到降低成本的作用;可进行病原微生物的绝对定量,可提高检测准确性,避免假阳性和交叉污染;数据量充足的情况下,可提高检测灵敏度。
19、关于barcode序列的示意如下表四所示:
20、表四barcode序列
21、
22、
23、
24、本方案针对于诺如病毒gi-gix亚型的全基因组试剂盒按照叠瓦式原理设计了17个引物对应于8个引物对,分成三个引物池对诺如病毒进行扩增,扩增完的两管产物混合后就得到了诺如病毒gi-gix亚型的全基因组序列,通过软件把数据进行拼接成完整的一致性序列;且本方案在靶向引物的5’端引入umi序列和barcode序列。
25、在一些实施例中,三组引物池内的引物的总体积比相同。
26、在一些实施例中,三组引物池内的引物分别对诺如病毒逆转录产物进行多重pcr扩增,且三组引物池的多重pcr扩增条件相同。
27、在一些实施例中,多重pcr扩增条件如下表五所示:
28、表五多重pcr扩增条件
29、
30、需要说明的是,本方案之所以将17个引物分成三组引物池,是由于诺如病毒的全基因组可能相对较长,使用单个引物池可能导致某些区域的低覆盖或遗漏,通过将引物分成多个池,可以同时放大不同区域,以覆盖整个基因组,确保高质量的全基因组测序。且将引物分成多个池可以减轻引物之间的相互竞,提高每个引物的特异性,可以实现对不同区域的深度测序的控制。如图1所示,,本方案提供的第一引物池的靶向引物覆盖片段1-2946,5040-7350,针对于诺如病毒的p48,ntpase,p22,vpg和vp1,vp2分型,第二引物池的靶向引物覆盖片段2420-5281,针对于诺如病毒rdrp分型,第三引物池的靶向引物覆盖片段3376-5281,针对于诺如病毒rdrp分型,之所以这么设计是考虑到rdrp为重要分型基因,故设计内外两对引物扩增,即第二引物池和第三引物池,增强扩增效果和测序信号;vp1也是重要分型基因,与orf1部分区域靶向引物扩增效率相当,且无重叠,故放在同一引物池中,即第一引物池。
31、另外,本方案设计的针对于引物池的多重pcr扩增条件中的阶梯式温度条件可有效提高靶向捕获效率,针对ct临界值的样本也可实现高效捕获。
32、在一些实施例中,包括hifi高保真dna聚合酶预混液和无核酸酶纯水。
33、在一些实施例中,诺如病毒gi-gix亚型全基因组捕捉试剂盒进一步包括逆转录反应预混液与逆转录引物池,以用于从纯化的诺如病毒的总rna中一致且快速的合成cdna。
34、在一些实施例中,逆转录的反应程序如下表六所示:
35、表六逆转录的反应程序
36、 pcr步骤 温度 时间 循环 1 22℃ 10min 1 2 55℃ 20min 1 3 85℃ 5min 1 4 4℃ hold
37、。
38、第二方面,本方案提供了一种诺如病毒gi-gix亚型全基因组捕捉试剂盒的应用方法,包括:获取待测的诺如病毒的逆转录cdna链,分别利用第一引物池、第二引物池以及第三引物池中的引物对诺如病毒的逆转录cdna链进行扩增得到扩增产物,将扩增产物拼接后利用测序平台测序得到诺如病毒gi-gix亚型全基因序列,其中第一引物池内的引物含有引物序列如seq id no.1-seq id no.5所示的靶向引物;第二引物池内的引物含有引物序列如seq id no 4-seq id no.7所示的靶向引物,第三引物池内的引物含有引物序列如seqid no 8-seq id no.17所示的靶向引物
39、在一些实施例中,取待测诺如病毒提取rna后加入逆转录反应预混液与逆转录引物后进行逆转录得到逆转录cdna链。
40、关于逆转录的条件如表六所示:
41、表六逆转录的反应程序
42、
43、
44、在一些实施例中,分别利用第一引物池、第二引物池以及第三引物池中的引物对诺如病毒的逆转录cdna链进行多重pcr扩增,多重pcr扩增条件如表五所示:
45、表五多重pcr扩增条件
46、
47、在一些实施例中,利用二代测序平台和三代测序平台对扩增产物进行测序,可利用illumina、mgi、nanopore等在内的所有主流测序平台对扩增产物进行测序。
48、在一些实施例中,适用于血液、拭子、感染组织等环境中提取的核酸样本。
49、相较于现有技术,本技术方案具有以下特点和有益效果:优化设计了针对于诺如病毒gi-gix亚型全基因组的引物,以实现诺如病毒所有分型的全基因组扩增,扩增的序列能够覆盖诺如病毒gi-gix亚型98.45%的完整序列,具有覆盖度均一的优点,且扩增得到的扩增产物可适用于包括illumina、mgi、nanopore等在内的所有主流测序平台,具有适配好性的优点,可广泛应用于医疗卫生、检验检疫等具有诺如病毒检测需求的机构以及相应的科研领域。
- 还没有人留言评论。精彩留言会获得点赞!