一种细胞水平蛋白高通量检测方法与流程
本发明属于生物技术领域,具体涉及一种细胞水平蛋白高通量检测方法。
背景技术:
在常规癌症诊断中,组织活检有一定局限性,并且会给患者带来一定的创伤和痛苦。液态活检领域的循环肿瘤细胞ctc检测技术为癌症诊断提供了新思路。ctc是从肿瘤原位灶脱落并进入并进入外周循环血中的肿瘤细胞,对循环血中ctc进行富集、鉴定、分离,可提前优于影像学发现肿瘤。对ctc表型研究和功能信息研究可为肿瘤细胞的鉴定、分类、追踪用药和实时疗效监测提供手段。
目前对ctc蛋白质组研究的较少,主要是缺乏稀有细胞蛋白质高通量检测手段。流式细胞术是目前常用的单细胞多种蛋白同步检测方法,但是由于存在光谱重叠现象,检测蛋白种类有限。质谱检测法可一次检测超过30种蛋白,但是无法对稀有的少量细胞进行检测。与荧光标记或质谱信息相比,用寡核苷酸做标记信号,可更灵敏、更高通量的检测蛋白。
寡核苷酸做信号标记抗体,其优点在于,与目的细胞孵育并收集目的细胞单细胞后,加入引物进行pcr,可对微量的信号放大,更有利于检测到痕量蛋白,并且可通过设计不同的序列或不同长度的寡核苷酸链以区分不同的抗体,8个碱基即可形成65536(48)种信号,可以实现真正意义上的高通量检测。
然而,ctc在外周血中含量极少,处理操作过程中易造成丢失,增加ctc富集捕获的难度。
因此,本领域迫切需要开发一种高效快速捕获ctc单细胞,并且对大量单细胞蛋白质组进行同步高通量检测的方法。
技术实现要素:
本发明公开了一种能最大限度防止ctc丢失的,微控芯片辅助的基于寡核苷酸条形码和下一代测序技术的循环肿瘤细胞单细胞水平蛋白高通量检测方法。
在本发明的第一方面,提供了一种细胞水平蛋白高通量检测方法,其特征在于,包括以下步骤:
(a)对外周血中的细胞进行富集,从而得到富集的细胞;
(b)将步骤(a)中富集的细胞、偶联有寡核苷酸的待检蛋白抗体、细胞鉴定标志物进行孵育;
(c)将步骤(b)中孵育后的细胞加载到微孔芯片上,从而使得所述微孔芯片上每个孔中有不多于一个细胞;
(d)对所述细胞进行分选,获得带有细胞鉴定标志物的目的细胞;
(e)对抗体上偶联的寡核苷酸进行扩增,从而得到扩增产物;
(f)对步骤(e)的扩增产物进行测序;
(g)对测序结果进行分析,从而获得单细胞水平蛋白高通量的测序结果。
在另一优选例中,所述细胞选自下组:血液中循环肿瘤细胞、其他液体样本如胸腔积液、脑脊液中的肿瘤细胞、血液与体液样本中的其他与疾病相关的细胞。
在另一优选例中,所述寡核苷酸为barcode序列。
在另一优选例中,所述barcode序列为不同的序列。
在另一优选例中,所述细胞鉴定标志物包括循环肿瘤细胞鉴定标志物,包括能够鉴定循环肿瘤细胞的荧光标记抗体或分子。
在另一优选例中,所述细胞鉴定标志物选自下组:荧光素apc标记的cd45抗体(anti-cd45-apc)、荧光素pe标记的epcam抗体(anti-epcam-pe)、荧光葡萄糖类似物2-nbdg。
在另一优选例中,步骤(a)所述的富集的方法包括:去除外周血中非ctc的负选方法、密度梯度离心方法。
在另一优选例中,所述寡核苷酸链中可变区为n个随意碱基,n为4-100,较佳地为5-60,更佳地,6-20,最佳地,6-8。
在另一优选例中,步骤(c)所述的微孔芯片,每个芯片含800-10000个微孔,较佳地,1000-8000个微孔,更佳地,3000-7000个微孔,最佳地,6400个微孔。
在另一优选例中,步骤(c)所述的微孔芯片,微孔直径为10-200μm,较佳地,20-90μm,更佳地,30-80μm,最佳地,70μm。
在另一优选例中,步骤(c)所述的微孔芯片,孔深50-500μm,较佳地,80-450μm,更佳地,100-400μm,最佳地,360μm。
在另一优选例中,步骤(c)所述的微孔芯片,底部有0.5-30μm小孔,较佳地,1-20μm小孔,更佳地,2-10μm小孔,最佳地,5μm小孔。
在另一优选例中,步骤(c)中,所述循环肿瘤细胞加载到微孔芯片上后,施加5-30mbar压力,较佳地,8-25mbar,最佳地,20mbar。
在另一优选例中,步骤(f)中建库引物为人工合成单链核苷酸,包含p5接头(illumina)、p7接头(illumina)、测序引物、n个碱基单细胞标签,其中n个随意碱基,n为4-100,较佳地为5-60,更佳地,6-20,最佳地,6-8。
在另一优选例中,单细胞标签与p5接头合成在同一条链上。
在另一优选例中,单细胞标签与p7接头合成在同一条链上。
在另一优选例中,步骤(g)中,所述测序使用的测序平台为illuminamiseq测序平台。
应理解,在本发明范围内中,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。
附图说明
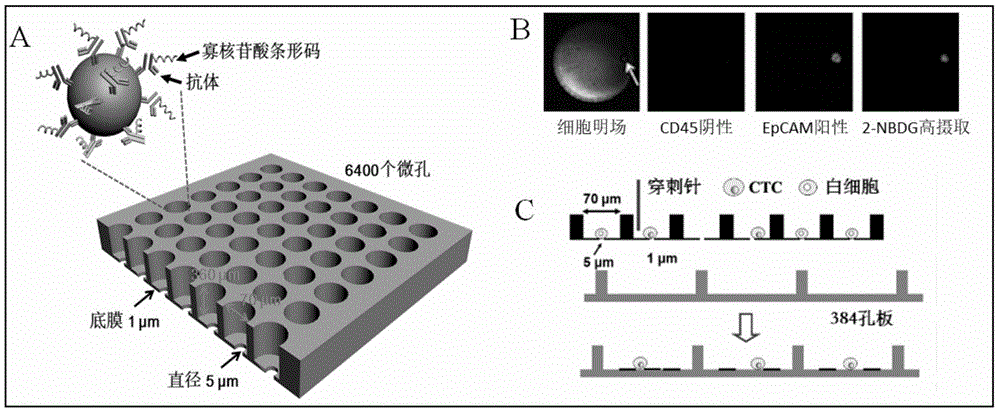
图1显示了本发明所述微孔芯片辅助ctc单细胞分离示意图。图1a为本发明中使用的微孔芯片;图1b为细胞明场和各荧光通道图片,anti-cd45-apc阴性,anti-epcam-pe阳性,2-nbdg高摄取的为ctc;图1c为穿刺针穿透微孔底膜,使目的细胞落入384孔板管中,而不损坏单细胞的完整性。
图2显示了细胞系单细胞表面蛋白高通量检测。图2a为十种蛋白在h1650细胞和h1975细胞表面表达情况;图2b为ck8、ck18、ck19在h1975细胞表面表达的荧光图。
图3显示了外周血中循环肿瘤细胞ctc单细胞表面蛋白高通量检测和基因组突变图谱。图3a为微孔中细胞明场及各通道荧光图;图3b为15种蛋白在90个单细胞表面表达情况;图3c为cd59表达量与her2表达量相关关系;图3d为基因组突变图谱。
具体实施方式
本发明人经过广泛而深入的研究,首次开发了一种灵敏度高、特异性好、通量高的循环肿瘤细胞单细胞水平蛋白检测方法。基于该方法,不仅可以同步检测不同单细胞中几十种至上百种蛋白,而且可以准确地检测到痕量蛋白。微孔芯片辅助循环肿瘤细胞ctc单细胞定位和挑选,ctc丢失率极少。在此基础上完成了本发明。
本发明的主要优点包括:
1)本发明所述的方法灵敏度高、特异性好、通量高;
2)本发明所述的方法不仅可以同步检测不同单细胞中几十种至上百种蛋白,而且可以准确地检测到痕量蛋白;
3)本发明所述的方法ctc丢失率极少。
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件,例如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。除非另外说明,否则百分比和份数是重量百分比和重量份数。
实施例1.
肿瘤细胞系单细胞水平蛋白高通量检测
方法:
(1)取预染anti-cd45-apc,anti-epcam-pe的肿瘤细胞系h1650和h1975各30个,分别混于5ml健康人(自愿者)外周血中,用rosetteseptmctc富集试剂盒(stemcelltechnologies)通过密度梯度离心对肿瘤细胞进行富集。
(2)富集后的细胞用0.1%-3%bsa和fc受体阻抗剂封闭细胞非特异性位点,室温孵育1小时。
(3)加入适当浓度的偶联了核苷酸条形码(见表2)的待检蛋白抗体(mcab),anti-pdgfβ-mcab,anti-egfr-mcab,anti-cytokeratin(ck)19-mcab,anti-ck8-mcab,anti-ck18-mcab,anti-pd-l1-mcab,anti-met-mcab,anti-epcam-mcab,anti-cd45-mcabandanti-cd44-mcab,各抗体终浓度均为1μg/ml,室温孵育1小时。
(4)上述细胞悬液用无糖的培养基dmem洗3-5次。用无糖的培养基dmem孵育细胞10分钟,对细胞进行饥饿处理。
(5)然后加入含0.4mm2-nbdg的无糖的培养基dmem于37度含5%二氧化碳的培养箱中培养15分钟。
(6)洗掉未被摄取的多余的2-nbdg,适当稀释制成细胞悬液,加载到微孔芯片上,施加20mbar压力,使肿瘤细胞进入微孔中,使每个微孔中最多有一个细胞。
(7)荧光成像系统扫描芯片,anti-cd45-apc阴性、anti-epcam-pe阳性,2-nbdg高摄取的细胞被鉴定为目的细胞,由穿测针穿透微孔底膜落入384孔板管中。
(8)每个孔中分别加入带有细胞标签的建库引物(见表1)和dna聚合酶预混物,进行16个pcr循环。
(9)将每个孔的pcr产物混合在一起,用miseq平台(illumina)进行下一代测序。
(10)统计测序得到的数据,单细胞标签序列超过2个碱基突变的,视为无效数据;寡核苷酸条形码序列超过2个碱基突变的,视为无效数据。单细胞标签序列代表某一具体的细胞,寡核苷酸条形码对应某一具体的抗体,根据单细胞标签序列,对寡核苷酸条形码进行分类,得到某个单细胞的蛋白表达数据。
结果
h1650细胞系实验中,检测到23个anti-cd45-apc阴性、anti-epcam-pe阳性,2-nbdg高摄取的细胞;h1975细胞系实验中,检测到28个anti-cd45-apc阴性、anti-epcam-pe阳性,2-nbdg高摄取的细胞。其中每个细胞系分别选出15个细胞进行蛋白检测。
结果如图2a所示,h1650细胞和h1975细胞中均未检测到cd45;与h1650细胞相比,h1975细胞epcam,egfr和pd-l1呈现高表达,而cd44表达相对较低。结果与已报道过的文献结果一致。
两种细胞系细胞中,ck8,ck18,ck19在细胞膜上均有一定程度的表达,图2b为ck8,ck18,ck19在h1975细胞表面表达的荧光图片。细胞膜表面的细胞角蛋白(如ck8,ck18,ck19)可以作为肿瘤细胞的潜在标志物。
实施例2.
外周血中循环肿瘤细胞ctc单细胞水平蛋白高通量检测
方法:
(1)5ml肺癌患者(自愿者)外周血全血样本,用rosetteseptmctc富集试剂盒(stemcelltechnologies)通过密度梯度离心对ctc进行富集。
(2)富集后的细胞用0.1%-3%bsa和fc受体阻抗剂封闭细胞非特异性位点,室温孵育1小时。
(3)加入适当浓度的anti-cd45-apc,anti-epcam-pe和偶联了核苷酸条形码(见表2)的待检蛋白抗体(mcab),anti-egfr-mcab,anti-ck19-mcab,anti-ck8-mcab,anti-ck18-mcab,anti-muc1-mcab,anti-her2-mcab,anti-trop2-mcab,anti-met-mcab,anti-pd-l1-mcab,anti-cd44-mcab,anti-cd46-mcab,anti-cd59-mcab,anti-cd133-mcab,anti-cd56-mcabandanti-pdgfβ-mcab,各抗体终浓度均为1μg/ml,室温孵育1小时。
(4)上述细胞悬液用无糖的培养基dmem洗3-5次。用无糖的培养基dmem孵育细胞10分钟,对细胞进行饥饿处理。
(5)然后加入含0.4mm2-nbdg的无糖的培养基dmem于37度含5%二氧化碳的培养箱中培养15分钟。
(6)洗掉未被摄取的多余的2-nbdg,适当稀释制成细胞悬液,加载到微孔芯片上,施加20mbar压力,使肿瘤细胞进入微孔中,使每个微孔中最多有一个细胞。
(7)荧光成像系统扫描芯片,anti-cd45-apc阴性、anti-epcam-pe阳性,2-nbdg高摄取的细胞被鉴定为ctc,由穿测针穿透微孔底膜落入384孔板管中。
(8)anti-cd45-apc阳性的单细胞为白细胞,选取50个作为对照,由穿测针穿透微孔底膜落入384孔板管中。
(9)365nm紫外光照射384孔板25分钟,使抗体上的寡核苷酸条形码释放。
(10)释放到孔中的寡核苷酸条形码可进行测序建库,分别加入带有细胞标签的建库引物(见表1)和dna聚合酶预混物,进行16个pcr循环。
(11)将每个单细胞的pcr产物混合在一起,用miseq平台(illumina)进行下一代测序。
(12)获取的ctc单细胞进行全基因组放大后,检测驱动基因突变,基因组驱动基因突变检测引物见表3。
(13)统计测序得到的数据,单细胞标签序列超过2个碱基突变的,视为无效数据;寡核苷酸条形码序列超过2个碱基突变的,视为无效数据。单细胞标签序列代表某一具体的细胞,寡核苷酸条形码对应某一具体的抗体,根据单细胞标签序列,对寡核苷酸条形码进行分类,得到某个单细胞的蛋白表达数据。
结果:在5个iv期肺腺癌患者外周血样本中,共找到61个anti-cd45-apc阴性、anti-epcam-pe阳性,2-nbdg高摄取的细胞,去除有白细胞干扰的微孔,共获得40个ctc单细胞。获取50个白细胞单细胞作为对照。建库过程中分别对每个单细胞引入了细胞标签,所以90个单细胞的建库产物可以混合在一起测序。
结果如图3b所示,ck19在细胞表面呈现高表达,说明ck19可以作为肿瘤细胞鉴定的潜在标志物;pd-l1不同细胞间表达水平不一致,揭示ctc的异质性;图3c显示,cd59的表达与her2表达高度相关,对单细胞蛋白质组表达情况进行研究,有利阐明蛋白之间的关系。
取1号样本中2个ctc单细胞,检测驱动基因突变。如图3d所示,在2个ctc中均检测到egfr的19外显子缺失,和患者原位肿瘤信息一致。
表1
表2
表3
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
序列表
<110>上海交通大学
<120>一种细胞水平蛋白高通量检测方法
<130>p2018-1696
<160>31
<170>patentinversion3.5
<210>1
<211>91
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(59)..(66)
<223>nisa,c,g,ort
<400>1
aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatctnn60
nnnnnnaatgctcgggaaggctactcagttt91
<210>2
<211>88
<212>dna
<213>人工序列(artificialsequence)
<400>2
caagcagaagacggcatacgagattggtcagtgactggagttcagacgtgtgctcttccg60
atctggtctgtggtgctgtattgagcgg88
<210>3
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>3
tggtctgtggtgctgtattgagcggtgcgatcacgacccaaactgagtagccttcccgag60
catt64
<210>4
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>4
tggtctgtggtgctgtattgagcggtgcgacagtggtccaaactgagtagccttcccgag60
catt64
<210>5
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>5
tggtctgtggtgctgtattgagcggtgcgcagatccaccaaactgagtagccttcccgag60
catt64
<210>6
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>6
tggtctgtggtgctgtattgagcggtgcgacaaacggccaaactgagtagccttcccgag60
catt64
<210>7
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>7
tggtctgtggtgctgtattgagcggtgcgacccagcaccaaactgagtagccttcccgag60
catt64
<210>8
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>8
tggtctgtggtgctgtattgagcggtgcgagggtcaaccaaactgagtagccttcccgag60
catt64
<210>9
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>9
tggtctgtggtgctgtattgagcggtgcgcccaacctccaaactgagtagccttcccgag60
catt64
<210>10
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>10
tggtctgtggtgctgtattgagcggtgcgcaccacacccaaactgagtagccttcccgag60
catt64
<210>11
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>11
tggtctgtggtgctgtattgagcggtgcggaaacccaccaaactgagtagccttcccgag60
catt64
<210>12
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>12
tggtctgtggtgctgtattgagcggtgcgtgtgaccaccaaactgagtagccttcccgag60
catt64
<210>13
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>13
tggtctgtggtgctgtattgagcggtgcgagactcgtccaaactgagtagccttcccgag60
catt64
<210>14
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>14
tggtctgtggtgctgtattgagcggtgcgcatgagtgccaaactgagtagccttcccgag60
catt64
<210>15
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>15
tggtctgtggtgctgtattgagcggtgcgaggatcacccaaactgagtagccttcccgag60
catt64
<210>16
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>16
tggtctgtggtgctgtattgagcggtgcgtcctagacccaaactgagtagccttcccgag60
catt64
<210>17
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>17
tggtctgtggtgctgtattgagcggtgcggtacagatccaaactgagtagccttcccgag60
catt64
<210>18
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>18
tggtctgtggtgctgtattgagcggtgcgacagtgcgccaaactgagtagccttcccgag60
catt64
<210>19
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>19
tggtctgtggtgctgtattgagcggtgcgctagtggtccaaactgagtagccttcccgag60
catt64
<210>20
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>20
ctcatcctaacacatttcaagccc24
<210>21
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>21
cagcatctcagggccaa17
<210>22
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>22
tgagagcctttagccgcc18
<210>23
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>23
taccctctcacgaaactctg20
<210>24
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>24
tggagaagctcccaaccaa19
<210>25
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>25
ttcccaaacactcagtgaaaca22
<210>26
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>26
gtggcaccatctcacaatt19
<210>27
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>27
atgctccaggctcaccaag19
<210>28
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>28
ctttatccaatgtgctcctc20
<210>29
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>29
tctcccttccctgattacct20
<210>30
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>30
ttcgccagccataagtcct19
<210>31
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>31
tcattcactgtcccagcaag20
- 还没有人留言评论。精彩留言会获得点赞!