一种具有抑制机制的流式生物数据隐私保护增量发布方法与流程
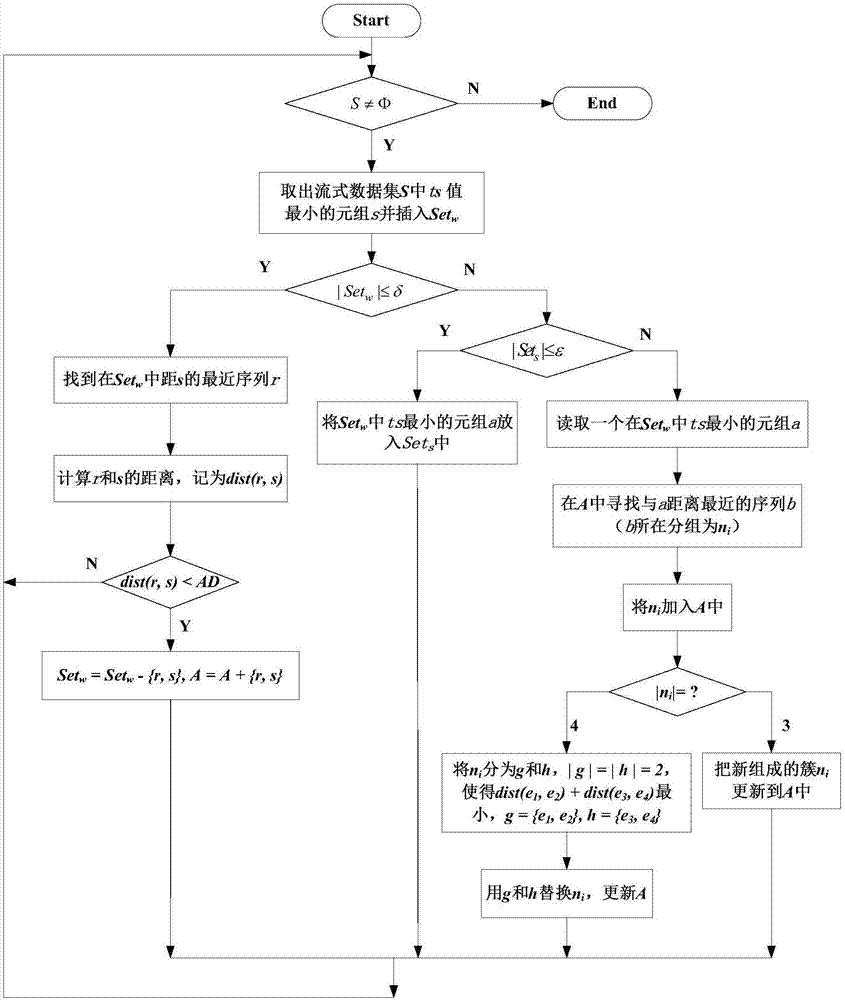
本发明涉及数据发布中的匿名隐私保护技术领域,具体是一种具有抑制机制的流式生物数据隐私保护增量发布方法。
背景技术:
随着DNA测序技术的进步,DNA测序向着高通量、低成本的方向不断发展,“人类基因组计划”得以完成。在此之后,大量以基因数据为主体的生物数据仍然不间断地产生,并且这些生物数据通过发布被广泛应用于医学研究和临床诊断。动态生物数据以数据流的形式到达收集方后,该数据会被及时更新到已发布的数据集中。然而,生物数据的发布具有潜在的隐私泄露风险,易造成数据提供者身份被识别问题。这将会阻碍生物数据的分享,导致生物数据难以被安全地应用于医学研究。因此,生物数据在发布时需要保护提供者身份不被识别,进行合理的隐私保护。
目前,针对生物数据隐私保护的方法主要为基于一种DNA泛化格的k-匿名算法——DNALA,如图2所示。该算法直接对基因组序列进行泛化操作,使发布的生物数据表满足2-匿名。在DNALA算法中,Malin已经证明了若k>2,则匿名后的基因组数据容易造成过度泛化,使得发布的数据集效用偏低。为保留数据的可用性,DNALA算法尽可能保证序列聚合成两两一组的簇,再对每个簇进行泛化。因此,DNALA算法在保证满足2-匿名的同时,保留了数据的可用性。此外,Li等人提出Hybrid算法解决流式生物数据的匿名问题。然而,Hybrid算法往往会形成大量三序列簇,导致发布的数据集可用性降低。
技术实现要素:
为了克服上述现有技术的缺点,本发明提供一种具有抑制机制的流式生物数据的隐私保护方法,大幅度提升了发布的生物数据集的实用性,使其具有较高的挖掘价值。
本发明是以如下技术方案实现的:一种具有抑制机制的流式生物数据隐私保护增量发布方法,输入:流式生物数据集S;抑制阈值ε;已发布数据集A;时延约束δ;已发布数据集A的平均距离AD(Average Distance);已发布数据集A的聚类结果m个簇(n1,n2,...,nm),其中,任意ni与nj不包含相同的元组,且任意一个元组簇ni中包含元组的数量为2或者3,已发布数据集A中的元组均存在于这m个簇;输出:更新后的匿名表A';具体步骤如下:
1)首先,设有空集合Setw用来存放等待发布的元组,空集合Sets用来存放被抑制的元组;
2)当流式生物数据集S非空时,取出流式生物数据集S中ts值最小的元组s,将其插入到Setw中,ts为元组达到收集方的时间;
3)若集合Setw中元组个数不大于δ,则执行步骤4);否则,执行步骤6);
4)找到集合Setw内距元组s最近的序列r,计算出r和s的距离dist(r,s);
5)如果,dist(r,s)小于已发布数据集A的平均距离AD时,从集合Setw取出元组r和s形成的簇放入已发布数据集A中,并泛化r和s,然后执行步骤7);否则,直接执行步骤7);
6)若集合Setw中元组个数大于δ,且集合Sets中元组个数小于ε,则将集合Setw中ts最小的元组a移入集合Sets中,然后执行步骤7);若集合Setw中元组个数大于δ,且Sets中元组个数大于ε,则获取集合Setw中ts值最小的元组a,找到已发布数据集A中距离a最近的序列b,将a添加到含b的元组簇ni中;针对新形成的元组簇ni所含元素个数的不同,采取相应处理方式:若此时的ni中元组个数为3时,则泛化ni;若ni中元组个数为4,则把ni划分为元素个数相等的g和h两个簇,并确保两个分组的内部元素距离之和最小,然后泛化g和h;
7)跳转到步骤2),直到流式生物数据集S为空;
8)得到更新后的匿名表A'。
本发明的有益效果是:通过抑制机制,有效地抑制了离群点的发布,在保障发布数据集隐私安全的同时保证了其具有较高的可用性,克服现存匿名流式数据的Hybrid算法容易数据集过度泛化的缺陷,另设置一个抑制阈值,可以控制抑制元组的数量,平衡了牺牲数据量和提高可用性之间的关系;本算法基于现存算法的框架,提高了匿名后的流式生物数据的可用性。与DNALA算法和改进后的Hybrid算法相比,本算法可形成较少的三元组簇,使待发布的数据集可用性得以显著提升,同时确保了基因数据的安全性,防止个人隐私的泄露。
附图说明
图1是本发明流程框图:
图2为DNALA算法下的DNA泛化格示意图;
图3为多序列比对机制(MSA)和两两序列比对机制(PSA)示意图;
图4为Hybrid算法下新到达的生物数据更新到已发布数据集中的示例图;
图5为WSPSGD方法下新到达的生物数据更新到已发布数据集中的示例图;
图6a为数据集I,δ=40,ε=20时WSPSGD的影响示例图;
图6b为数据集II,δ=40,ε=30时WSPSGD的影响示例图;
图6c为数据集III,δ=80,ε=40时WSPSGD的影响示例图;
图7a为数据集I,ε=20时流基因函数的平均距离示例图;
图7b为数据集II,ε=30时流基因函数的平均距离示例图;
图7c为数据集III,ε=40时流基因函数的平均距离示例图。
图8a为数据集I,δ=40时抑制阈值的影响示例图;
图8b为数据集II,δ=40时抑制阈值的影响示例图;
图8c为数据集III,δ=80时抑制阈值的影响示例图。
具体实施方式
本发明主要提出了一种具有抑制机制的流式生物数据的k-匿名隐私保护增量发布方法,以下为本发明方法使用到的k-匿名概念以及流式基因组数据的概念。
定义1 k-匿名模型定义:发布的数据集中每条元组至少具有k-1条不可区分的元组。根据这一原则,k-匿名模型确保重新确定一个人在发布数据集中的概率不超过1/k。具体参考表1的处理结果。表1为原数据集及其k-匿名的转换示意图。其中年龄和性别属性已被泛化,并且表中最后一条数据被抑制。从表中可以得出,处理后的数据集满足2-匿名。
表1
定义2 流式基因组数据的k-匿名:假设S为一个具有属性集AS=(pid,DNA sequence,at)的流式基因组数据集,其中pid标志个人序列号、DNA sequence表示基因序列,ts为S中元组的到达时间。假设S'为S匿名后的数据,则S'中不包含pid、ts属性。若S'满足k-匿名,则需满足以下条件:
(1)对于t'由t泛化而得,
(2)对于所有EQ(t')中的元组和t'相同,并且|EQ(t')|代表|EQ(t')|的数目,那么我们将S'命名为一个满足k-匿名的流式基因数据集。例如表2所示,表格中左边的数据集为原始流式基因数据,右边的数据则是匿名后满足2-匿名的数据集。其中pid为3201和3202的元组为一个EQ(t'),此时|EQ(t')|=2。
表2
定义3 延迟约束δ:设P是一个动态基因组数据集的匿名策略,如果由P输出的满足k-匿名的数据集S'满足:其中,t是S中与t'相对应的元组,δ为一个给定的实数且δ>0。那么,我们称P满足时延约束δ。
针对现存的DNALA算法和Hybrid算法处理动态基因数据的缺陷,我们提出了一种改进的k-匿名算法。首先,DNALA是一种静态基因组数据,其处理动态序列花费时间较长。其次,在DNALA中,已证明对包含三个元组的聚簇进行泛化时容易造成过度泛化,降低数据的可用性,而Hybrid算法在处理动态生物数据时会形成大量包含三元组的簇,造成数据集的过度泛化。为为解决这一问题,本发明中的算法尽可能地使得元组两两聚合成簇并进行泛化,使匿名后的数据表在满足k=2的同时,聚合更多的包含两元组的簇。
定义4 抑制阈值ε:一个待发布数据集定义为D。如果D中的元组d将不会被发布,那么称元组d被抑制。其中,给定参数ε是可抑制的最大数据量,该阈值用于限制不被发布的元组数量。
基于以上定义,一种具有抑制机制的流式生物数据隐私保护增量发布方法,输入:流式生物数据集S;抑制阈值ε;已发布数据集A;时延约束δ;已发布数据集A的平均距离AD(Average Distance);已发布数据集A的聚类结果m个簇(n1,n2,...,nm),其中,任意ni与nj不包含相同的元组,且任意一个元组簇ni中包含元组的数量为2或者3,已发布数据集A中的元组均存在于这m个簇;输出:更新后的匿名表A';具体步骤如下:
1)首先,设有空集合Setw用来存放等待发布的元组,空集合Sets用来存放被抑制的元组;
2)当流式生物数据集S非空时,取出流式生物数据集S中ts值最小的元组s,将其插入到Setw中,ts为元组达到收集方的时间;
3)若集合Setw中元组个数不大于δ,则执行步骤4);否则,执行步骤6);
4)找到集合Setw内距元组s最近的序列r,计算出r和s的距离dist(r,s);
5)如果,dist(r,s)小于已发布数据集A的平均距离AD时,从集合Setw取出元组r和s形成的簇放入已发布数据集A中,并泛化r和s,然后执行步骤7);否则,直接执行步骤7);
6)若集合Setw中元组个数大于δ,且集合Sets中元组个数小于ε,则将集合Setw中ts最小的元组a移入集合Sets中,然后执行步骤7);若集合Setw中元组个数大于δ,且Sets中元组个数大于ε,则获取集合Setw中ts值最小的元组a,找到已发布数据集A中距离a最近的序列b,将a添加到含b的元组簇ni中;针对新形成的元组簇ni所含元素个数的不同,采取相应处理方式:若此时的ni中元组个数为3时,则泛化ni;若ni中元组个数为4,则把ni划分为元素个数相等的g和h两个簇,并确保两个分组的内部元素距离之和最小,然后泛化g和h;
7)跳转到步骤2),直到流式生物数据集S为空;
8)得到更新后的匿名表A'。
该方法简称WSPSGD(With Suppression Publishing Streaming Genomic Data)方法,由以上步骤可知,WSPSGD方法的步骤2),取出S中最先到达的元组s,并将其插入一个等待发布的临时存储集合Setw。步骤3),判断Setw中是否有元组等待时间超过时延。步骤4)~5),若Setw中元组的等待时间均未超过时延,则从Setw找到离s最近的元组r,计算r和s的间距dist(r,s)。如果dist(r,s)小于AD,把r和s组成的簇更新到A中,这一步确保了已发布数据集新增簇时,不会增大其信息损失量。步骤6)~步骤7),若Setw中存在元组的等待时间超过时延,则判断Sets包含元组的数量与抑制阈值ε的关系。如果Sets小于ε,那么将Setw中的ts最小的元组a转移到用于存储不发布数据的集合Sets中,换言之,抑制掉超出时延的数据(即永远不发布这些数据);如果Sets数目不小于ε,则取出Setw中最先到达的元组a,在数据集A中找到距a最近的元组b,将a插入到包含b的簇中。如果a插入后的簇包括四个序列,将其分成两个小簇,使每个簇中仅包含两个元组,接着泛化这些序列;而新组成的簇包括三个序列时,则直接泛化这些序列。
WSPSGD方法尽管牺牲少量的数据,但通过抑制机制有效地减少三元组簇的形成,使发布的数据集具有较大的挖掘价值。此外,WSPSGD方法中的参数ε可以控制被抑制的元组数量,通过调节ε,可以在牺牲数据量和提高可用性之间得到平衡。图3为多序列比对机制(MSA)和两两序列比对机制(PSA)示意图,图5是WSPSGD方法处理流式数据的例图,图4是Hybrid算法处理流式数据的例图。由图可知,WSPSGD方法处理的流式数据集包含三序列的簇的个数比Hybrid算法少,因此,WSPSGD方法具有更高的精确度。
实验验证及结果分析
实验数据集及环境:为了评估WSPSGD方法,进行算法性能测试,实验使用来自NCBI的三个数据集,分别包含元组个数为:327、540和711。详情如表3所示。为模拟大数据流,实验将这些数据的1/3作为静态处理数据集,采用Hybrid和其他MWM-based算法对其进行匿名处理。此后将剩余的2/3作为动态更新数据,再通过WSPSGD方法进行动态匿名化处理。
表3
测试WSPSGD方法的实验平台配置如下:AMD Athlon(tm)II 2.1GHz CPU/4GB内存,Window 10系统。以下所得实验数据均为在运行10次实验的基础上取其结果的平均值。
实验结果分析
图6a、图6b以及图6c所示为WSPSGD方法中,不同数据集情景下平均距离随流基因序列更新数量的变化情况。从图6a中可以看出,WSPSGD方法处理后形成的平均距离小于Hybrid算法,WSPSGD方法隐匿的数据平均距离在[20-120]范围内不断缩减,[120-180]范围内出现上升,Hybrid算法总体呈现曲折下降。在这个过程中,Hybrid算法泛化产生了许多三序列簇,从而导致平均距离增大,而WSPSGD方法可以找到一些Setw中适当的二序列簇使得平均距离减小。因此,被WSPSGD方法隐匿的数据相比于Hybrid算法的处理结果具有更小的平均距离和IL。图6b和图6c也显示了相同结论:在处理流式数据时,WSPSGD方法比Hybrid算法有更高的精度。
图7a、图7b以及图7c主要展现WSPSGD方法本身所具有的参数与效果之间的评估,图中数据表示平均距离与发布序列量、时延δ之间的函数关系。从中可以看出普遍规律,随着时延的增大,在同一发布数据量的情况下,平均距离随之减小。
图8a、图8b以及图8c证明了平均距离随着流基因序列更新数量的增大逐渐增大。此外,抑制阈值越大,平均距离越小的实验现象说明,WSPSGD方法在抑制离群数据发布,提高数据实用性方面效果显著。
综上所述,相比于Hybrid算法,WSPSGD方法整体性能更优。尤其在数据处理量较少时,具有更大的优越性。同时,实验结果表明该算法遵循一般规则:在整个过程中抑制阈值越大,信息损失得越少。它能够保障生物数据隐私安全的同时,克服现存的Hybrid算法生成大量三序列聚类的缺陷,发布更精确的数据集,使得发布的生物数据集的实用性大大增强。
- 还没有人留言评论。精彩留言会获得点赞!