一种植物响应逆境胁迫的lncRNAs序列模块功能注释方法与流程
本发明涉及分子生物学技术领域,具体涉及一种植物响应逆境胁迫的lncrnas序列模块功能注释的方法。
背景技术:
高通量rna测序技术是现代基因组学研究最重要的实验技术,在整个生物学领域具有广泛的应用。随着大量测序数据的建立,基因组学研究得到快速发展。在利用高通量rna测序技术运用于差异表达基因即编码rna的研究基础上,越来越多的研究关注到基因组的非编码rna上。这些非编码rna包括mirnas、sirnas、lncrnas、circularrna等,其中mirnas由于其具有独特序列模块以及与靶基因的作用方式已经被广泛地证明在植物生长发育和响应逆境胁迫的转录调控方面具有重要的作用。而lncrnas是一类长度超过200nt,不具有蛋白编码能力的非编码rna,可以通过多种方式影响靶基因的转录调控。因此,高通量的开展lncrnas的功能注释工作对于今后非编码rna的功能研究具有重要意义。
目前,已有的研究主要通过计算非编码rna与编码rna共表达关系的方法将lncrnas与共表达的mrna分为一组,利用mrna的注释信息来完成lncrnas的功能注释。该方法一是需要大量的表达数据用于共表达分析,同时还将缺失胁迫响应特异表达lncrnas的功能注释信息。
技术实现要素:
有鉴于此,本发明的第一目的在于提供了一种植物响应逆境胁迫的lncrnas序列模块功能注释方法,包括以下步骤:
步骤s1,筛选植物响应逆境胁迫的lncrnas及其靶基因;
步骤s2,再次筛选植物响应逆境胁迫的lncrnas及其靶基因表达模式分析;
步骤s3,按照靶基因表达模式进行植物响应逆境胁迫的lncrnas分组;
步骤s4,响应逆境胁迫的lncrnas特异序列模块富集分析;
步骤s5,响应逆境胁迫的lncrnas特异序列模块功能注释。
优选地,本发明所述的植物响应逆境胁迫的lncrnas序列模块功能注释方法中,所述步骤s1中所述逆境胁迫为低温处理或高盐处理;优选地,所述低温处理为4℃处理6小时;所述高盐处理为150mmnacl处理6小时。
优选地,本发明所述的植物响应逆境胁迫的lncrnas序列模块功能注释方法中,所述步骤s1中lncrnas筛选标准为①长度大于200nt;②最小读长覆盖率为3;③开放阅读框小于300nt;④cpcscore<0,cnciscore<0。
优选地,本发明所述的植物响应逆境胁迫的lncrnas序列模块功能注释方法中,所述步骤s2中所述再次筛选响应逆境胁迫差异表达的lncrnas,筛选最小阈值为:差异倍数>2或<0.5,p-值<0.05,q-值<0.05。
优选地,本发明所述的植物响应逆境胁迫的lncrnas序列模块功能注释方法中,所述步骤s3中所述分组方法为根据响应逆境胁迫的lncrnas表达模式或靶基因功能富集结果,将响应逆境胁迫的lncrnas进行分组。
优选地,本发明所述的植物响应逆境胁迫的lncrnas序列模块功能注释方法中,所述步骤s4中响应逆境胁迫的lncrnas特异序列模块富集分析方法为利用meme(http://meme-suite.org/tools/meme)对分组后的响应逆境胁迫的lncrnas特异序列模块进行富集分析。
更优选地,本发明所述的植物响应逆境胁迫的lncrnas序列模块功能注释方法中,所述分析中筛选的参数设置为:①每个lncrnas上至少预测到一个序列模块;②预测序列模块数量3-5个;③模块长度为6bp-15bp;④分布模式为正反义两条链;⑤不允许软件对序列进行重排。
优选地,本发明所述的植物响应逆境胁迫的lncrnas序列模块功能注释方法中,所述步骤s5中所述响应逆境胁迫的lncrnas特异序列模块功能注释为利用gomo(http://meme-suite.org//tools/gomo),对富集的lncrnas特异序列模块功能进行功能预测。
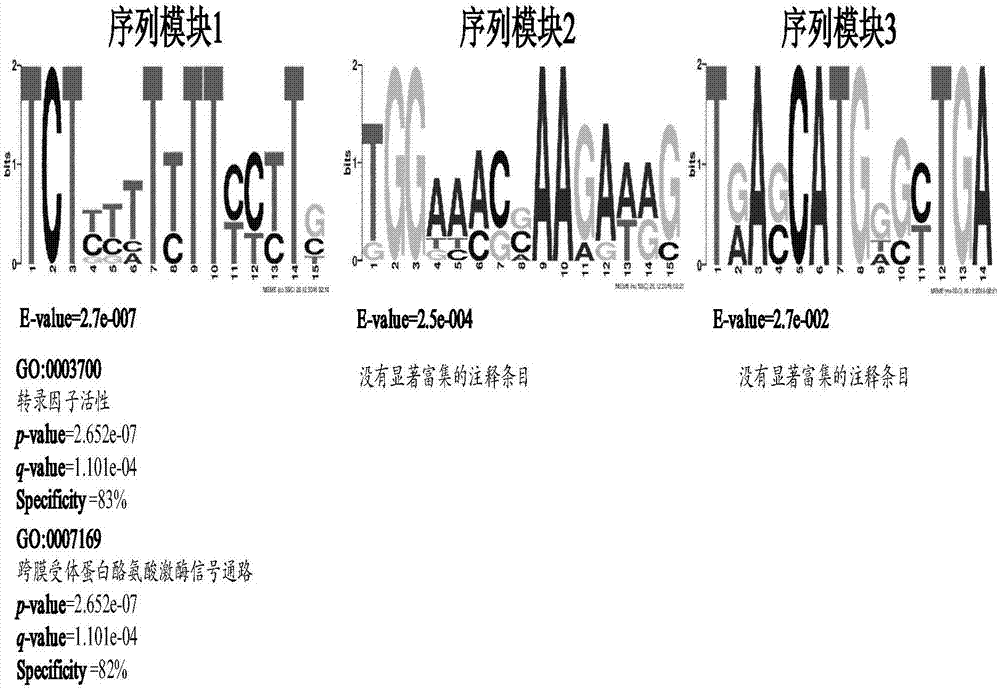
更优选地,期望值(e-value)表示由于随机性造成获得与数据库比对结果的可能次数。期望值越小,发生这一事件的概率越低,比对结果越显著。假设机率(p-value)代表给定原假设为真时样本结果出现的概率。p值越小代表原假设不成立的概率越大。q值(q-value)代表经过假阳性率校正之后的p值,该数值越低则代表假阳性率越小。特异性(specificity)代表该模块的独特性,数值越高则独特性越强。本发明所述的植物响应逆境胁迫的lncrnas序列模块功能注释方法中,所述注释方法中筛选参数设置如下:①显著性参数设置为p-value<0.01并且q-value<0.01;②计算次数5000次;③专一性(specificity)参数大于80%;④期望值e-value<0.001。序列模块1(e-value=2.7e-007;p-value=2.6e-07;q-value=1.1e-04;specificity=83%)获得goterm注释转录因子活性(见图2)。
因此,针对现有lncrnas功能预测方法依赖于需要大量的表达数据用于共表达分析,以及还没有响应逆境胁迫的特异表达lncrnas的功能注释信息。本方案提供一种响应逆境胁迫的植物lncrnas序列模块功能注释方法,结合生物信息学与差异表达分析对植物响应逆境胁迫的lncrnas序列模块进行注释,将为系统解析lncrns生物学功能以及构建其转录调控网络提供了技术支持。
即本发明提供一种植物lncrnas逆境胁迫响应序列模块功能注释的方法,结合生物信息学与差异表达分析对植物响应逆境胁迫的lncrnas序列模块进行注释,不但极大地提高了实验的效率、精准性以及灵活性,并显著地降低了实验成本。
附图说明
图1为响应逆境胁迫的植物lncrnas序列模块功能注释的方法流程示意图;
图2毛白杨响应低温lncrnas序列模块功能注释结果图;
图3小叶杨响应高盐胁迫lncrnas序列模块功能注释结果图。
具体实施方式
根据本发明一个典型的实施方式,待测样本为毛白杨1年生植株。对其进行低温(4℃,6小时)处理,立即收集叶片用于提取总rnas。利用ribo-zerorrna试剂盒对核糖体rna进行去除。利用smart试剂盒进行链特异性cdna文库构建。利用illuminahiseqtm2500测序平台完成cdna文库测序,测序深度为10×。去除接头以及冗余序列,通过cufflinks软件拼接转录本,筛选长度大于200nt、最小读长覆盖率为5、cpcscore<0、cnciscore<0以及与对照差异表达倍数大于2(p<0.05)的lncrnas。预测差异表达的lncrnas顺式及反式作用靶基因,并对靶基因的表达模式进行解析,筛选差异表达的靶基因作为候选基因(最小阈值为:差异倍数>2或<0.5,p-值<0.05,q-值<0.05)。对于候选基因,利用ncbi核酸数据库(https://blast.ncbi.nlm.nih.gov/blast.cgi)进行功能注释。利用agrigo(http://bioinfo.cau.edu.cn/agrigo/)对候选基因进行goterm富集分析。利用meme(http://meme-suite.org/tools/meme)对分组后的响应逆境胁迫的lncrnas特异序列模块进行富集分析。利用gomo(http://meme-suite.org//tools/gomo),对富集的lncrnas特异序列模块功能进行功能预测。
以下通过具体实施例进一步对本发明的技术方案进行说明,应理解以下仅为本发明的示例性说明,并不用于限制本发明权利要求的保护范围。
实施例1
对毛白杨1年生植株进行低温(4℃,6小时)处理,提取其总rna用于对响应低温胁迫的lncrnas序列模块进行功能注释。
s1,利用ribo-zerorrna试剂盒对核糖体rna进行去除。利用smart试剂盒进行链特异性cdna文库构建。利用illuminahiseqtm2500测序平台完成cdna文库测序,测序深度为10×。去除接头以及冗余序列,通过cufflinks软件拼接转录本,筛选长度大于200nt、最小读长覆盖率为5、cpcscore<0、cnciscore<0lncrnas,共获得4218个。筛选差异倍数小于0.35且大于0.2(p-值<0.05,q-值<0.05)的lncrnas,共17个(见表1)。
表1毛白杨响应低温逆境胁迫的lncrnas
在14个响应高温胁迫的lncrna上下游10kb范围内筛选顺式作用靶基因,共获得26个(见表2)。利用blast进行序列互补计算,参数设置为e-value=1e-10,identity=90%以及利用rnaplex进行热力学上的互补计算,参数设置为e=-70。共获得反式作用靶基因25个(见表3)。根据差异表达靶基因筛选标准(p-值<0.05,q-值<0.05),筛选获得候选靶基因42个(见表4)
表2毛白杨响应低温逆境胁迫的lncrnas顺式作用靶基因
表3毛白杨响应低温逆境胁迫的lncrnas反式作用靶基因
表4毛白杨响应低温逆境胁迫的lncrnas靶基因功能注释
s2,对于候选基因,利用ncbi核酸数据库(https://blast.ncbi.nlm.nih.gov/blast.cgi)进行功能注释(见表4)。利用agrigo(http://bioinfo.cau.edu.cn/agrigo/)对候选基因进行goterm富集分析(见表5)。
表5毛白杨响应低温逆境胁迫的lncrnas靶基因功能富集分析
s3,植物响应逆境胁迫的lncrnas分组。根据逆境胁迫响应lncrnas表达模式或靶基因功能富集结果,将响应逆境胁迫的lncrnas进行分组(见表6)。
表6毛白杨响应逆境胁迫的lncrnas进行分组
s4,利用meme(http://meme-suite.org/tools/meme)对分组后的响应逆境胁迫的lncrnas特异序列模块进行富集分析。筛选参数设置如下:①每个lncrnas上至少预测到一个序列模块;②预测序列模块数量3-5个;③模块长度为6bp-15bp;④分布模式为正反义两条链;⑤不允许软件对序列进行重排。第3分组共获得富集的lncrnas序列模块3个(见图2)
s5,利用gomo(http://meme-suite.org//tools/gomo),对富集的lncrnas特异序列模块功能进行功能预测。筛选参数设置如下:①显著性参数设置为q-value<0.01;②计算次数5000次;③专一性(specificity)参数大于80%。序列模块1获得goterm注释(见图2)。
如图2所示,序列模块1期望值e-value=2.7e-007;注释条目:go:0003700,功能预测结果:转录因子活性(p-value=2.6e-07;q-value=1.1e-04;specificity=83%);跨膜受体蛋白酪氨酸激酶信号通路(p-value=2.6e-07;q-value=1.1e-04;specificity=82%);序列模块2、序列模块3无显著富集注释条目。期望值(e-value)表示由于随机性造成获得与数据库比对结果的可能次数。期望值越小,发生这一事件的概率越低,比对结果越显著。假设机率(p-value)代表给定原假设为真时样本结果出现的概率。p值越小代表原假设不成立的概率越大。q值(q-value)代表经过假阳性率校正之后的p值,该数值越低则代表假阳性率越小。特异性(specificity)代表该模块的独特性,数值越高则独特性越强。
实施例2
对小叶杨1年生植株进行高盐处理(150mmnacl,6小时),提取其总rna用于对响应渗透胁迫的lncrnas序列模块进行功能注释。
s1,利用ribo-zerorrna试剂盒对核糖体rna进行去除。利用smart试剂盒进行链特异性cdna文库构建。利用illuminahiseqtm2500测序平台完成cdna文库测序,测序深度为10×。去除接头以及冗余序列,通过cufflinks软件拼接转录本,筛选长度大于200nt、最小读长覆盖率为5、cpcscore<0、cnciscore<0lncrnas,共获得4241个。筛选差异倍数大于3且小于13(p-值<0.05,q-值<0.05)共13个lncrnas(见表7)。
表7小叶杨响应高盐逆境胁迫lncrnas
在13个高盐胁迫响应的lncrna上下游10kb范围内筛选顺式作用靶基因,共获得44个(见表8)。利用blast进行序列互补计算,参数设置为e-value=1e-10,identity=90%以及利用rnaplex进行热力学上的互补计算,参数设置为e=-70。共获得反式作用靶基因59个(见表9)。根据差异表达靶基因筛选标准(p-值<0.05,q-值<0.05)筛选获得候选靶基因73个(见表10)
表8小叶杨响应高盐逆境胁迫的lncrnas顺式作用靶基因
表9小叶杨响应高盐逆境胁迫的lncrnas反式作用靶基因
表10小叶杨响应高盐逆境胁迫的lncrnas靶基因功能注释
s2,对于候选基因,利用ncbi核酸数据库(https://blast.ncbi.nlm.nih.gov/blast.cgi)进行功能注释(见表10)。利用agrigo(http://bioinfo.cau.edu.cn/agrigo/)对候选基因进行goterm富集分析(见表11)。
表11小叶杨响应高盐逆境胁迫的lncrnas靶基因功能富集分析
s3,植物响应逆境胁迫的lncrnas分组。根据响应逆境胁迫的lncrnas表达模式或靶基因功能富集结果,将响应逆境胁迫的lncrnas进行分组(见表12)。
表12小叶杨响应逆境胁迫的lncrnas分组
s4,利用meme(http://meme-suite.org/tools/meme)对分组后的响应逆境胁迫的lncrnas特异序列模块进行富集分析。筛选参数设置如下:①每个lncrnas上至少预测到一个序列模块;②预测序列模块数量3-5个;③模块长度为6bp-15bp;④分布模式为正反义两条链;⑤不允许meme软件对序列进行重排。第3组共获得富集的lncrnas序列模块3个(见图3)。
s5,利用gomo(http://meme-suite.org//tools/gomo),对富集的lncrnas特异序列模块进行功能预测。筛选参数设置如下:①显著性参数设置为q-value<0.01;②计算次数5000次;③专一性(specificity)参数大于80%。三个序列模块分别获得goterm注释(见图3)。
如图3所示,序列模块1期望值e-value=1.5e-004;注释条目:go:0009507,功能预测结果:叶绿体(p-value=3.447e-07;q-value=1.300e-02;specificity=85%);序列模块2期望值e-value=8.1e-003;注释条目:go:0010287,功能预测结果:质体球(p-value=1.856e-07;q-value=3.500e-03;specificity=100%);注释条目:go:0009535,功能预测结果:叶绿体类囊体膜(p-value=1.856e-07;q-value=3.500e-03;specificity=91%)。序列模块3期望值e-value=1.6e-004;注释条目:go:0005739,功能预测结果:线粒体(p-value=5.569e-06;q-value=1.050e-02;specificity=82%);期望值(e-value)表示由于随机性造成获得与数据库比对结果的可能次数。期望值越小,发生这一事件的概率越低,比对结果越显著。假设机率(p-value)代表给定原假设为真时样本结果出现的概率。p值越小代表原假设不成立的概率越大。q值(q-value)代表经过假阳性率校正之后的p值,该数值越低则代表假阳性率越小。特异性(specificity)代表该模块的独特性,数值越高则独特性越强。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!