小细环病毒及其获得方法和应用与流程
本发明涉及口腔医学和病毒学技术领域,尤其涉及一种从牙周炎患者口腔中分离获得的小细环病毒及其获得方法和应用。
背景技术:
牙周病是一组复杂的感染性疾病,通常认为其由细菌刺激引起、由人体免疫等多因素参与。牙周病以牙槽骨吸收、牙齿松动及脱落为主要临床症状,已经成为成年人失牙的主要原因。重度牙周炎不仅会影响口腔健康,还与糖尿病、心血管疾病、低体重新生儿及早产等系统性疾病相关,对全身健康产生严重的负面影响。传统的牙周病病因学研究通常认为牙周病的发病与特定种类的细菌感染相关,然而细菌致病理论却并不能完整地解释牙周病的各种临床特征。例如,umeda等研究发现,牙周致病菌广泛存在于唾液之中,接触所有牙齿,但是牙周病却常常不是全口发病,而仅影响有限数目的牙齿,具有位点特异性;尽管牙周环境中总有不同种类和数目的牙周致病菌持续感染和定植,但是牙周病却通常是短期发病,有自限性;病毒作为另一种潜在的牙周病致病因素逐渐受到关注,牙周病发病过程中,与病毒相关的牙周致病菌的粘附定植、病毒导致的宿主抗牙周致病菌的免疫功能抑制、与牙周病相关的病毒激活等机制可相对合理地解释菌斑量与牙周炎严重程度、进展速度的关系。病毒宏基因组学作为一项新兴技术能充分挖掘特定环境中的病毒群落和未知病毒。
ttv是一种无囊膜的单链环状dna病毒,于1997年首次被发现,属于指环病毒科(anelloviridae)。分为三属:torquetenovirus(ttv)s;torquetenominivirus(ttmv);torquetenomidivirus(ttmdv)。目前ttv的研究较多,但是,指环病毒是否致病仍是一个悬而未决的问题。
技术实现要素:
本发明要解决的技术问题是提供一种小细环病毒(ttmv),该病毒是从牙周炎患者牙周袋内壁牙龈上皮中分离获得的新病毒,为口腔疾病的治疗与预防提供新的解决方案。
此外,还需要提供一种上述小细环病毒的获得方法和应用。
为了解决上述技术问题,本发明通过如下技术方案实现:
在本发明的一个方面,提供了一种小细环病毒,该病毒包含:编码seqidno.1所示氨基酸序列的核苷酸序列。
优选的,所述病毒具有seqidno.2所示的全长核苷酸序列。
在本发明的另一方面,还提供了一种重组载体,包含上述小细环病毒的全部或部分核苷酸序列。
所述重组载体包括重组克隆载体或重组表达载体。
在本发明的另一方面,还提供了一种抗小细环病毒的抗体,该抗体与seqidno.1所示的氨基酸序列或其部分片段特异性结合。
在本发明的另一方面,还提供了一种检测小细环病毒的试剂盒,包含特异性针对seqidno.2所示全部或部分核苷酸序列设计的引物对。利用该试剂盒,通过简单的pcr反应就可以检测出本发明的小细环病毒。
在本发明的另一方面,还提供了一种检测小细环病毒的试剂盒,包含上述的抗体。利用该试剂盒,通过elisa等方法就可检测出本发明的小细环病毒。
在本发明的另一方面,还提供了一种小细环病毒的获得方法,包括以下步骤:
取牙周炎患者牙龈组织样品,提取该样品中的病毒基因组;
将所述病毒基因组反转录成病毒cdna,再用随机引物进行pcr扩增,扩增后的产物通过高通量测序,构建测序文库;
将高通量测序结果进行数据预处理后,把长度大于100bp的短序列拼接为重叠群;
将拼接的序列与数据库中的已知序列数据进行比对,找出序列之间的相似性;
分析比对结果,依据已知序列设计扩增引物,通过巢式pcr扩增,获得未知病毒的全长基因序列,该基因序列编码seqidno.1所示的氨基酸序列;
经系统进化树分析,对未知病毒进行物种分类,将该未知病毒归类为小细环病毒。
在本发明的另一方面,还提供了一种上述小细环病毒的应用,用于筛选预防或治疗口腔疾病的药物。
在本发明的另一方面,还提供了一种抗上述小细环病毒的药物。
在本发明的另一方面,还提供了一种上述药物的应用,用于制备预防或治疗口腔疾病的药物。
本发明的小细环病毒,是采用病毒宏基因组学方法从牙周炎患者牙周袋内壁牙龈上皮中分离获得的一株新病毒,为口腔疾病的病因、治疗及预防策略提供新的依据。
附图说明
下面结合附图和具体实施方式对本发明作进一步详细的说明。
图1是本发明实施例1小细环病毒ttmv-204的基因结构图;
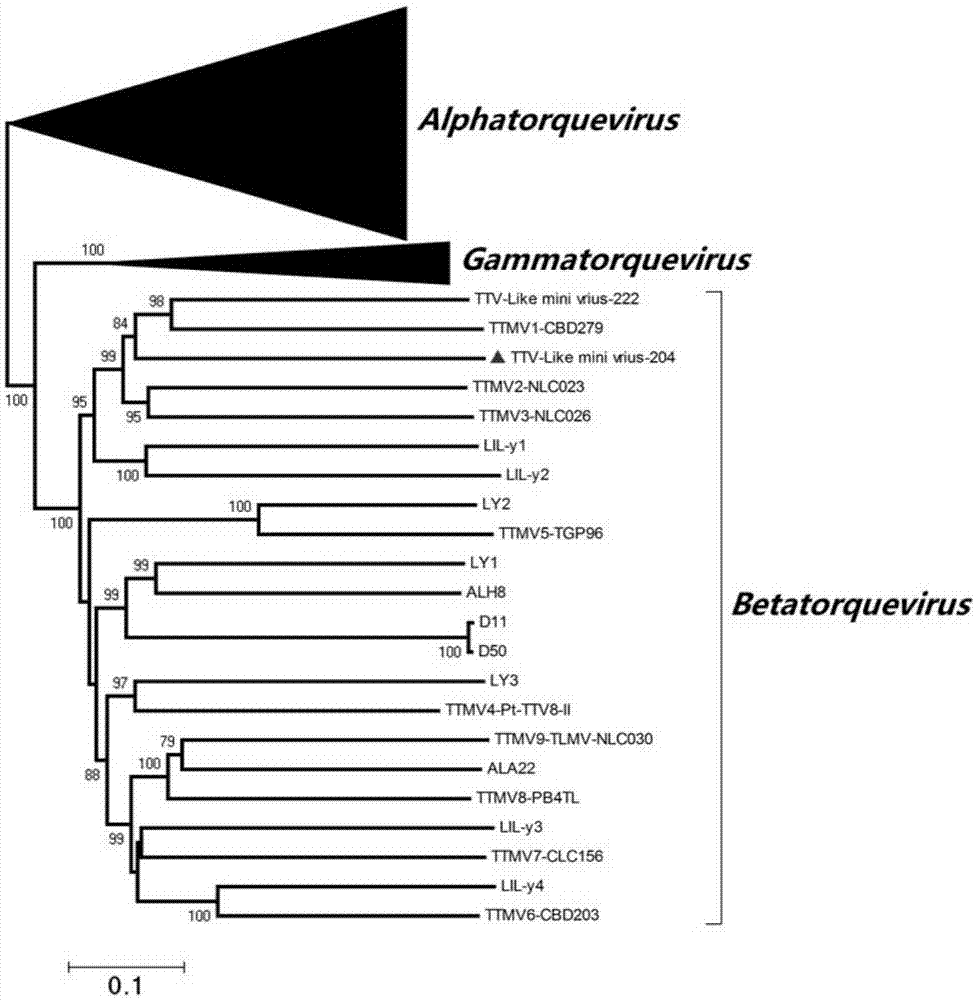
图2是本发明实施例1的ttmv-204系统进化树分析图。
具体实施方式
下列实施例中,未注明具体条件的实验方法,通常按常规条件,如《精编分子生物学实验指南》(f.m.奥斯伯,r.e.金斯顿,j.g.塞德曼等主编,马学军,舒跃龙的译.北京:科学出版社,2004)中所述的方法进行。
传统的牙周病病因学研究焦点多为牙周致病细菌,而细菌致病理论却不足以完整解释牙周病相关的临床症状,本发明将关注转移到牙周病发生、发展过程中的病毒因素,力图揭示牙周环境中的病毒群落,筛选出牙周病的可疑致病病毒,为后续治疗与预防牙周病提供基础。由于病毒特殊的生物学特性,传统的病毒鉴定方法已经无法满足要求,本发明首次采用病毒宏基因组学方法分析牙周环境中的病毒群落以及鉴定牙周环境中的未知病毒。通过钓取疑似新病毒的序列,克隆出新病毒的全基因组序列,分析该新病毒的全基因组,构建系统进化树,并将其命名为小细环病毒ttmv-204。
实施例1采用病毒宏基因组学方法从牙周炎患者口腔中分离获得小细环病毒的全长基因序列
取牙龈组织约2g,放入2ml离心管中,加入1mlpbs及3粒直径0.3cm钢珠后封口膜封口,后进行匀浆(反复冻融三次并匀浆5min)。4℃条件下,13000g离心10分钟,抽取上清样品,再次4℃条件下,12000g离心5分钟,抽取上清样品,两次离心去除细胞碎片和其它微生物;通过0.45m滤器处理上清样品,7500rpm离心1min过滤,再次去除悬液中的非病毒壳粒;37℃下水浴90min,应用turbodnase、rnase、baselinezerodnase和benzonase去除病毒颗粒包裹外的遗传物质。目前已处理牙周炎患者病例组66份样品,健康对照组32份样品。并且提取其遗传物质(包括dna、rna、dsdna、ssdna、dsrna和ssrna等)。用qiaampviralrnaextractionkit提取样品中的病毒基因组,通过dna酶去除dna病毒的遗传物质及反转录和klenowfragment酶,提取完病毒基因组后立刻使用superscriptⅲreversetranscriptase试剂盒进行反转录操作,反转录的引物为fixed-primer-8n,该引物的设计是在网站(http://www.changbioscience.com/primo/primor.html)上完成,在网站中产生随机引物,随后通过与ncbi的数据库进行blast比对,挑选其中不能够匹配到人或者细菌的引物作为fixed-primer;向提取的病毒基因组12μl中加入1μl100pmol的fixed-primer-8n,上下颠倒5-6次混合均匀;70℃水浴5min;冰上静置2min;后进行反转录;反转录产物置于94℃下5min变性,冰上冷却2min;后向上述产物中加入1μl的klenow酶进行第二链的合成反应,合成病毒双链cdna,同时将引物标签分别加在不同样品的病毒双链cdna上;通过单引物pcr扩增病毒遗传物质,pcr扩增的引物为fixed-primer-8n对应的fixed-primer引物,模板即为上一步中得到的双链cdna模板;pcr产物经过1%的琼脂糖凝胶电泳,切取满足高通量测 序的dna;设置48个复孔,并设定电压为100伏特。经90分钟电泳后得到smear条带,分别切取48个复孔对应长度范围在500-1000bp的凝胶,置于2ml离心管,称重。产物经pcrpurificationkit纯化,电泳后切胶回收;根据miseq高通量测序文库构建试剂盒的操作说明,构建miseq测序文库,送至测序公司测序。测序结果由斯坦福大学基因组工程中心进行数据处理和序列分析,依据引物标签将各条序列信息分类至各个样品,随后去除引物及标签。将长度大于100bp的短序列经newbler2.5软件拼接为重叠群(contigs)。下载ncbi数据库中的生物遗传信息,建立本地blast文库,并将拼接和翻译的核苷酸和氨基酸序列在本地文库和ncbi数据库中进行多重blast比对。设定e-value≤0.00001为临界参数,将比对结果依据e-value值的大小分为真核生物、细菌、病毒和未知序列。比对结果上传至可视化网站,分析病毒序列信息,发现疑似全新ttv序列。
分析比对结果,依据已有序列设计疑似ttv扩增引物,进行反向巢式pcr扩增后测序,第一轮引物为a*g*tctggtggaacgggcaag(seqidno.9)和g*a*attgccccttgactacgg(seqidno.10),第二轮引物为caaaacccaacatactctcc(seqidno.11)和tgactacggtggtttcactc(seqidno.12),引物中“*”标记的碱基指硫代修饰的碱基。pcr扩增后,产物经凝胶电泳、用axyprepdnagelextractionkit(axygene,siliconvalley,usa)切胶回收、克隆后测序。测序后的序列经过载体的剪切和拼接,找出插入碱基和突变位点,并进行纠错。克隆出的基因序列通过ncbi的orffinder工具寻找其开放阅读框,结果如图1所示,新病毒的全长基因序列为2824bp(seqidno.2),其编码的氨基酸序列如seqidno.1所示(925aa),包含3个开放阅读框(orf1、orf2、orf3)。三个开放阅读框中,orf1长1948bp,位于271-2211bp(核苷酸序列如seqidno.4所示),其编码的氨基酸序列如seqidno.3(646aa)所示;orf2长270bp,位于122-391bp(核苷酸序列如seqidno.6所示),其编码的氨基酸序列如seqidno.5(89aa)所示;orf3长228bp,位于1973-2200bp(核苷酸序列如seqidno.8所示),其编码的氨基酸序列如seqidno.7(75aa)所示。
通过mega5.1软件分析新病毒与其它代表株的同源性,并画出其系统进化树(结果见图2),予以命名并归类其种属信息。该新病毒命名为ttmv-204,经系统进化树分析和同源性分析,新病毒与指环病毒科(anelloviridae)、乙型细环病毒属(betatorquevirus)中的ttmv1-cbd279,ttmv2-nlc023及ttmv3-nlc026全核酸同源性最高,分别为57.6%,57.1%和55.9%。对orf1同源性分析得其与ttmv1-cbd279,ttmv2-nlc023及ttmv3-nlc026核酸同源性为55.8%,55.5%和54.3%,氨基酸同源性为38.7%,39.3%和37.2%。
以上所述实施例仅表达了本发明的实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
- 还没有人留言评论。精彩留言会获得点赞!