一种基于基因组LoF位点筛查间接关联猕猴桃种性的方法与流程
本发明属于植物遗传资源评价和生物技术领域,具体涉及一种基于基因组LoF位点筛查间接关联猕猴桃种性的方法及其育种应用,即用于可稳定遗传的猕猴桃种性特征的分子遗传基础分析鉴定,以提高猕猴桃资源评价和遗传育种利用效率,促进新品种的快速培育。
背景技术:
我国猕猴桃属(ActinidiaLindl.)植物野生资源丰富,该属植物约54个物种共75个分类单元中,绝大多数类群的自然分布为我国独有。如何充分利用丰富的自然资源,特别是形态各异的栽培猕猴桃野生近缘种资源,是当前猕猴桃遗传育种工作的重要研究内容。在对物种自然资源进行评价利用的过程中,识别各物种特有的可稳定遗传的性状特征(简称“种性”)并研究其内在的分子遗传基础是充分利用物种资源进行猕猴桃遗传育种工作的重要前提。由于猕猴桃为雌雄异株多年生植物且广布于从热带到亚热带甚至温带的大量地理区域,在多样化的自然生境中进化方式各异,包括频繁的种间杂交、适应辐射和多倍化,从而导致各种性状特征形成的分子遗传机制复杂多样,类似特征性状的可遗传稳定性也存在较大差异,筛选可稳定遗传的物种种性并进行育种应用十分困难。
在基因组测序技术飞速发展的今天,充分利用逐渐增加的基因组数据资源来解析基因组遗传位点变异的多样性并与潜在的特征性状进行关联分析已成为当前作物分子遗传育种工作的重要发展方向。在物种基因组呈现的各类遗传变异中,LoF变异位点,即基因功能失活变异位点,是形成可稳定遗传的种性特征的重要变异源泉之一。筛选物种整基因组范围的LoF位点信息,将有利于我们间接的理解物种种性的具体特征属性和可预期功能变异范围,在物种资源的遗传育种过程中形成焦点性状的定向资源选择和应用,大大提升资源遗传育种应用的效率。
技术实现要素:
本发明的目的是针对猕猴桃可稳定遗传的种性特征识别利用困难的问题,提供一种基于基因组LoF位点筛查间接关联猕猴桃种性的方法,该方法是从基因组LoF变异位点入手,筛查猕猴桃物种关联特异的LoF变异位点信息并进行相关的功能注释,从而间接关联物种种性特征变异属性和范围,并最终形成资源评价和育种应用。
本发明是通过公共数据库或者基因组低深度重测序分析获取相关猕猴桃物种的基因组短片段序列,再将序列比对到中华猕猴桃参考基因组的基础上,筛选功能基因的比对序列并寻找引起功能失活的突变位点,最终形成对这些位点的功能预判和相关种性的间接关联与育种应用。
本发明包括物种基因组序列片段的获取和参考基因组比对,在序列完全比对的基础上获取相关编码区基因的比对序列并对所呈现的相关变异位点进行变异类型划分,从中筛查LoF功能失活变异位点并进行相关的功能注释和变异预测,对所有完成功能注释的LoF位点进行功能家族的分类管理和物种种性间接关联预测,并最终形成潜在的育种应用指导实践。
本发明的基于基因组LoF位点筛查间接关联猕猴桃种性的方法,其特征在于,包括以下步骤:
A、物种基因组序列的获取:获取猕猴桃物种的基因组短片段序列
B、序列的参考基因组比对和变异发掘与分类:将各猕猴桃物种的基因组短片段序列与中华猕猴桃参考基因组进行比对,然后进行比对猕猴桃物种所有位点的基因型确定和单碱基变异位点SNPs的识别和信息收集;再下载获取中华猕猴桃基因组参考编码基因集合,根据序列比对的位置确定比对的基因数量和具体边界,并基于每一个基因在中华猕猴桃基因组的具体位置将所有比对到编码基因的序列形成的SNP变异划分为基因间变异、基因编码区域的编码序列变异和内含子区域变异三个大的类型;
C、LoF变异位点的筛查获取与类型划分:对于存在于基因编码区域的编码序列变异进行注释和效应预测,并区分同义和非同义变异两种类型,对于同义变异,进一步识别终止获取类型,对于非同义变异,进一步识别错配变异、抑制子变异和启动与终止密码丢失/获取变异;
根据基因编码区域的编码序列变异的注释和效应预测,在所有变异位点中按照如下原则进行LoF变异位点的筛查:基因编码区域的变异导致永久的终止密码获取、基因编码区域小的插入缺失引起基因读码框转移、变异存在于小于2bp的剪切位点中;
D、LoF变异位点的功能注释和归类:所有筛选出的LoF变异位点所在的基因编码区域的序列,在获取注释信息的基础上进行大的功能类别的富集分析;
E、物种特有LoF变异信息的筛选及种性间接关联分析:收集猕猴桃物种的可显著观察的生态和表型数据集,利用LoF变异位点所在的基因编码区域的序列的功能类别所注释的信息以及每个类别中富集的LoF位点对应的基因数量与相关的生态表型数据进行线性相关分析,找出显著相关的功能信息重叠区,从而将所对应的LoF位点指定为物种种性的代表标记位点。
所述的可显著观察的生态和表型数据集包括:所在地理分布区域的19个生物气候因子;相关猕猴桃花、叶和果的10个形态因子。
相比于现有技术,本发明有益效果如下:
①相比复杂多变的可观察表型性状,本发明利用的LoF变异位点信息由于直接相关于基因组序列的变异,具有更高的稳定性和重复性,从而使得物种种性的关联预测更加准确;
②利用方便的基因组序列数据,使用本方法可以快速获取并预测特定猕猴桃种质的潜在种性特征和可育种利用价值,大大提高了资源评价利用效率;
③本方法可利用各种类型的基因组序列数据,对LoF位点的筛查也具有高度的灵活性,因而可进一步演化到对特异功能性状遗传位点的变异发掘利用。
附图说明:
图1是本发明的流程示意图;
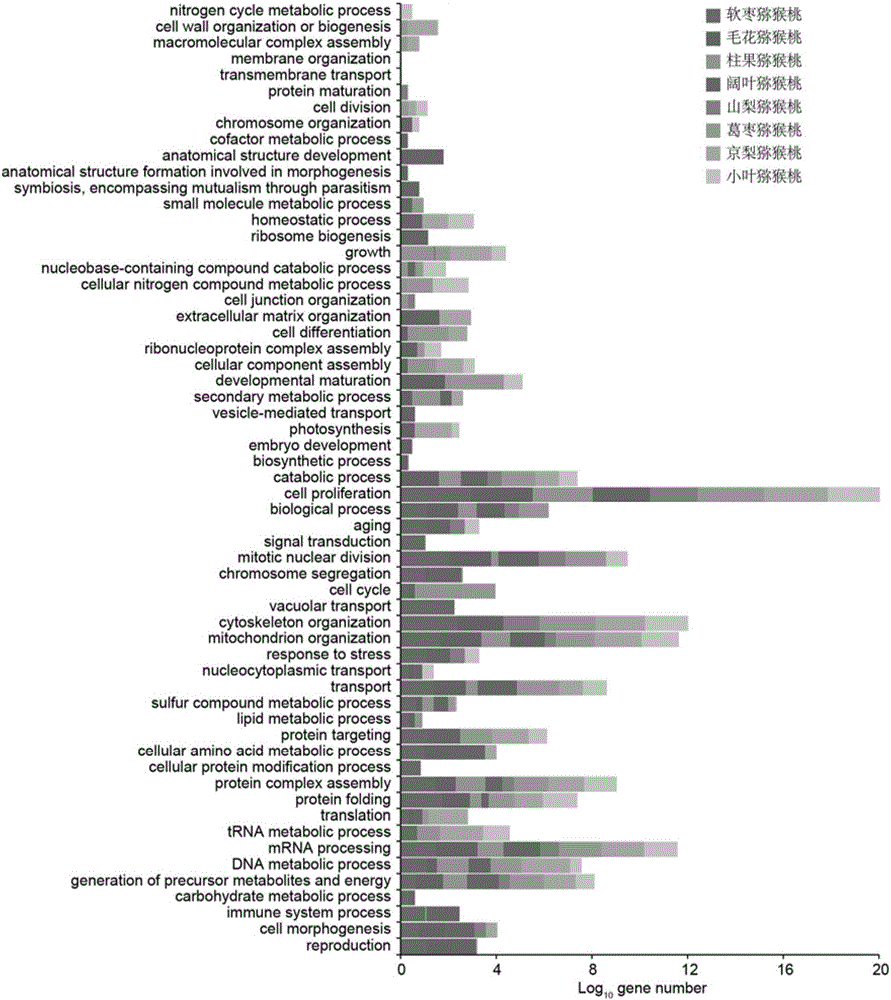
图2是不同猕猴桃物种LoF位点功能富集信息分布图。
具体实施方式:
以下实施例是对本发明的进一步说明,而不是对本发明的限制。
实施例1:本实施例的基于基因组LoF位点筛查间接关联猕猴桃种性的方法,包括以下步骤:
1、如图1步骤①物种基因组序列的获取。利用公共序列数据库资源,包括美国国家生物技术信息中心(National Center for Biotechnology Information,简称NCBI),猕猴桃参考基因组数据库(Kiwifruit Information Resource,简称KIR),国家猕猴桃种质资源圃(武汉)内部数据库资源,获取猕猴桃物种的基因组短片段序列。
对于未发布序列数据的猕猴桃物种,采用整基因组低深度测序的方式获取序列数据(基因组短片段序列)。根据中华猕猴桃参考基因组约700M的基因组大小,对待测序的猕猴桃物种样本进行约5倍低深度的二代基因组测序,达到总数据量约3.5Gb即可。样本基因组低深度测序的具体步骤包括:采集猕猴桃物种样本约5ug的基因组总DNA,将基因组打断构建180bp的小片段文库,然后在高通量二代测序仪如本实施例使用的Illumina公司的Hiseq2000测序仪完成簇的生成,最后对生成的片段进行双末端测序获得所有的碱基信息。将得到的原始测序小片段按标准进行过滤从而得到分析所用的基因组数据(基因组短片段序列)。过滤标准采用测序仪自带软件的缺省值。
2、如图1步骤②序列的参考基因组比对和变异发掘与分类。利用免费的Stampy软件基于缺省值将各猕猴桃物种基因组短片段序列比对到现有的中华猕猴桃参考基因组,然后利用免费的基因组分析工具箱GATK进行比对物种的所有位点的基因型确定和单碱基变异位点(SNPs)的识别和信息收集。为了保证SNPs变异位点的准确性,本实施例SNPs的收集过程中排除reads深度小于6或者大于70的SNPs,同时排除与邻近SNP的物理距离小于5bp的SNP位点来进一步提高精确性。
在KIB数据库中下载获取中华猕猴桃基因组参考编码基因集合,根据序列比对的位置确定比对的基因数量和具体边界,并基于每一个基因在中华猕猴桃基因组的具体位置将所有比对到编码基因的序列形成的SNP变异划分为基因间变异、基因编码区域的编码序列变异和内含子区域变异三个大的类型。
3、如图1步骤③LoF变异位点的筛查获取与类型划分。对于存在于基因编码区域的编码序列变异,利用免费的SnpEff软件对所有的基因编码区域的编码序列变异进行注释和效应预测,并区分同义(Synonymous)和非同义(Nonsynonymous)变异两种类型。对于同义变异,进一步识别终止获取(stop_retained)类型,对于非同义变异,进一步识别错配变异(missense_variant)、抑制子变异(initiator_variant)和启动与终止密码丢失/获取变异(start_lost、stop_gained和stop_lost)。
根据基因编码区域的编码序列变异的注释和效应预测,在所有变异位点中按照如下原则进行LoF变异位点的筛查:基因编码区域的变异导致永久的终止密码获取、基因编码区域小的插入缺失引起基因读码框转移、变异存在于小于2bp的剪切位点中。
4、如图1步骤④LoF变异位点的功能注释和归类。对所有筛选出的LoF变异位点所在的基因编码区域的序列,在获取注释信息的基础上利用免费的Goseq软件进行大的功能类别的富集分析。功能类别的富集显著水平P值设定为0.05(经过Benjamini-Hochberg矫正)。
5、如图1步骤⑤物种特有LoF变异信息的筛选及种性间接关联分析。收集猕猴桃物种的可显著观察的生态和表型数据集,包括:所在地理分布区域的19个生物气候因子(基于WorldClim公共数据库);相关猕猴桃花、叶和果的10个形态因子(基于通用的UPOV标准)。利用LoF变异位点所在的基因编码区域的序列的功能类别所注释的信息以及每个类别中富集的LoF位点对应的基因数量与相关的生态表型数据进行线性相关分析,找出显著相关(P值<0.05)的功能信息重叠区,从而将所对应的LoF位点指定为物种种性的代表标记位点,方便资源的育种应用。
二、应用
基于上述方法,本实施例随机选择了软枣猕猴桃、毛花猕猴桃、柱果猕猴桃、阔叶猕猴桃、山梨猕猴桃、葛枣猕猴桃、京梨猕猴桃和小叶猕猴桃等8种不同的猕猴桃物种,按照上述步骤一的方法,经过LoF变异位点的筛查,总共发现了28151个LoF位点,分属于59个不同的功能类别(图2),包括相关植物抗性、细胞分裂、色素调控和代谢等。
通过生态表型数据的相关分析,其中物种特异高度相关的LoF位点2638个,将其定义为猕猴桃物种种性LoF标记位点,具有重要的育种应用价值。例如软枣猕猴桃相关免疫防御和压力适应的11个LoF位点的富集,与其高抗病性、适应高海拔冷凉气候一致;毛花猕猴桃相关果实成熟发育的143个LoF位点的富集,与其高的耐贮藏特性高度相关;阔叶猕猴桃中594个相关细胞扩张分裂的LoF位点与其多花序表型一致。
- 还没有人留言评论。精彩留言会获得点赞!